Tot oder Lebendig – Studien zum programmierten
Werbung

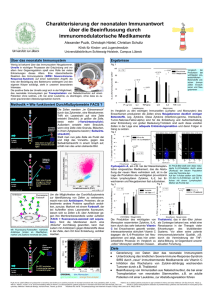
Überblick 143 Tot oder Lebendig – Studien zum programmierten Zelltod in C. elegans Nicole Wittenburg und Barbara Conradt Max-Planck-Institut für Neurobiologie, Martinsried Abb. 1: Darstellung der vier aufeinanderfolgenden Schritte des programmierten Zelltodprozesses und der an den einzelnen Schritten beteiligten C. elegans Gene. Während der Entwicklung, wie auch in adulten Tieren, fallen in multizellulären Organismen eine große Anzahl von „unerwünschten“ Zellen an. Unerwünschte Zellen sind Zellen, die entweder in zu großer Menge gebildet wurden, die ihre Funktion erfüllt haben oder die beschädigt und somit für den Organismus gefährlich sind. Unerwünschte Zellen werden in multizellulären Tieren durch den physiologischen und konservierten Prozess des programmierten Zelltods, auch Apoptose genannt, eliminiert. Wird die Regulation oder Durchführung des programmierten Zelltods im Menschen gestört, so kann dies Krankheiten zur Folge haben, wie z.B. neurodegenerative Krankheiten, Krebs oder Autoimmunkrankheiten. Dass Zellen gezielt eliminiert werden, wurde schon im 19. Jh. beobachtet. Der Bedeutung dieses Prozesses für die Entwicklung und die Aufrechterhaltung des zellulären Gleichgewichts wurde man sich allerdings erst Mitte des 20. Jh. bewusst. Und erst in den letzten 20 Jahren hat man begonnen, die molekularen Mechanismen dieses wichtigen Prozesses aufzudecken[1]. Von großer Bedeutung waren dabei Studien an dem Nematoden Caenorhabditis elegans. BIOspektrum · 2/02 · 8. Jahrgang Der Prozess des programmierten Zelltods kann in vier aufeinanderfolgende Schritte aufgeteilt werden: die Zelltodspezifizierung, die Zelltodexekution, die Phagocytose und die Degradation der toten Zelle. Während der Zelltodspezifizierung wird bestimmt, welche Zellen überleben und welche sterben; während der Zelltodexekution findet dann das eigentliche Töten der Zellen, die sterben sollen, statt. Während der Phagocytose werden die absterbenden Zellen durch andere Zellen phagocytiert und letztendlich von ihnen verdaut (Abbildung 1). Ein Organismus, der bei der Identifizierung von am Zelltod beteiligter Moleküle eine bedeutende Rolle gespielt hat – und immer noch spielt – ist C. elegans. Aufgrund seines im wesentlichen konstanten Zellstammbaums, seines kompakten, seit 1998 sequenzierten Genoms und seiner pflegeleichten Aufzucht, hat sich dieser 1mm große Fadenwurm als wichtiger genetischer Modelorganismus für die Analyse verschiedenster biologischer Prozesse etabliert[2]. Die Tatsache, dass programmierter Zelltod für das Überleben von C. elegans zumindest unter Laborbedingungen nicht lebensnotwendig ist, dass man sterbende Zellen im lebenden Tier mit Hilfe von Nomarski Optik beobachten kann, und dass man während der Entwicklung genau weiß, welche Zellen sterben werden, machen C. elegans für genetische Studien des programmierten Zelltods besonders geeignet. Das ermöglichte die Identifizierung von Genen, die für die Durchführung der verschiedenen Schritte des programmierten Zelltods notwendig sind (Abbildung 1). Programmierter Zelltod in C. elegans Programmierter Zelltod kommt in C. elegans während der Entwicklung in somatischen Geweben und im adulten Tier in der weiblichen Keimbahn vor[3, 4]. Der programmierte Tod von somatischen Zellen während der Entwicklung ist durch den weitgehend invarianten Zellstammbaum bestimmt: von den 1090 somatischen Zellen, die während der Entwicklung eines Hermaphroditen gebildet werden, werden genau 131 wieder durch programmierten Zelltod eliminiert. In adulten Hermaphroditen werden ungefähr 50% der in der Gonade gebildeten Keimzellen durch programmierten Zelltod eliminiert (physiologischer Keimzelltod). Die sterbenden Keimzellen dienen wahrscheinlich mit ihrem Cytoplasma als Nahrung für überlebende, sich differenzierende Oocyten. Die Beschädigung der DNS durch genotoxische Reagenzien oder verschiedene Formen von Stress, wie z.B. bakterielle Infektionen, können den programmierten Zelltod von zusätzlichen Keimzellen induzieren (nicht-physiologischer Keimzelltod)[5, 6]. Überblick 144 Abb: 2: Der genetische Zelltodexekutionsweg in C. elegans mit seinen konservierten Komponenten. Die aus ced-9, ced-4 und ced-3 bestehende zentrale Exekutionsmaschinerie wird in der weiblichen Keimbahn und im Soma von C. elegans auf verschiedene Weisen aktiviert. Die zentrale Zelltodexekutionsmaschinerie ist konserviert Die genetischen Untersuchungen in C. elegans führten zur Identifizierung von drei Genen, ced-3, ced-4 und ced-9 (ced, „cell death abnormal“), die, wenn mutiert, den programmierten Zelltod während der Entwicklung in somatischen Geweben, wie auch in der Keimbahn von adulten Hermaphroditen, generell blockieren können, und die somit in diesem Organismus Komponenten der zentralen Zelltodexekutionsmaschinerie darstellen (3). Im Fall von ced-3 und ced-4 sind es Mutationen, die zum Funktionsverlust des Gens führen, welche den programmierten Zelltod blockieren. ced-3 und ced-4 kodieren somit für Zelltodaktivatoren, die für programmierten Zelltod notwendig sind. Im Fall von ced-9 blockiert eine Mutation, die zur Überaktivität des Gens führt, den programmierten Zelltod. Dies bedeutet, dass ced-9 für einen Zelltodinhibitor kodiert. Mutationen, die das ced-9 Gen inaktivieren, führen zum gegensätzlichen Phänotyp und zwar zu ektopischem programmierten Zelltod. Dieser ektopische Zelltod ist von funktionalen ced-3 und ced-4 Genen abhängig, was darauf hindeutet, dass diese beiden Gene nach ced-9 während der Zelltodexekution wirken (Abbildung 2). Die Überexpression der Zelltodaktivatorgene ced-3 und ced-4 kann ebenfalls zu ektopischem Zelltod führen. Während dies im Fall von ced-3 unabhängig von einem funktionalen ced-4 Gen ist, hängt es im Fall von ced-4 von ced-3 ab, was daraus schließen lässt, dass ced-3 im Zelltodexekutionsschritt nach ced-4 wirkt (Abbildung 2). Die Klonierung und molekulare Charakterisierung von ced-9, ced-4, und ced-3 bestätigte, dass die molekularen Mechanismen der programmierten Zelltodexekution von C. elegans bis zum Menschen konserviert sind (2). ced-9 kodiert für ein strukturelles und funktionales Homolog des menschlichen Proto-Onkoproteins und Zelltodinhibitors Bcl-2. ced-3 kodiert für eine Cysteinprotease und ist der Prototyp der Familie der Zelltodproteasen genannt Caspasen. ced-4 kodiert für ein Homolog des menschlichen Adaptorproteins und Caspaseaktivators Apaf-1. Die Aktivierung von Pro-Caspasen durch Apaf-1-ähnliche Proteine ist ein wichtiges Ereignis während der Zelltodexekution und stellt oft den „point of no return“ dar. Während der frühen embryonalen Entwicklung von C. elegans, der Phase während der die meisten der 131 somatischen Zelltodereignisse stattfinden, scheinen die drei Komponenten der zentralen Zelltodexekutionsmaschinerie, die Proteine CED-9, CED-4, und pro-CED-3, in den meisten, wenn nicht sogar allen Zellen vorhanden zu sein. Die meisten Zellen haben somit das Potential, durch programmierten Zelltod zu Abb 3: Model für die Aktivierung der zentralen Zelltodexekutionsmaschinerie. Durch die Bindung von EGL-1 an CED-9, transloziert CED-4 an die perinuklearen Membranen und kann dann proCED-3 in aktives CED-3 überführen. BIOspektrum · 2/02 · 8. Jahrgang Überblick 145 Abb. 4: In den sexuell dimorphen HSNs wird die egl-1 Transkription direkt durch den Zelltyp-spezifischen Regulator TRA-1 reguliert. sterben. Das Zelltodinhibitorprotein CED-9 befindet sich an Mitochondrien und bildet einen Komplex mit dem CED-4 Protein (Abbildung 3). Die Interaktion zwischen CED-9 und CED-4 blockiert die Fähigkeit von CED-4, pro-CED-3 zu aktivieren und den Zelltod auszulösen. Zellen überleben demnach so lange die Interaktion zwischen CED-9 und CED-4 aufrechterhalten wird[7]. Initiierung der Zelltodexekution: egl-1 – der zentrale Regulator im Soma Wie die Interaktion zwischen CED-9 und CED-4 aufgehoben und somit die Exekution einer Zelle besiegelt wird, konnte durch die Charakterisierung eines weiteren Zelltodaktivators aufgeklärt werden. Mutationen, die zum Funktionsverlust des Gens egl-1 führen (egl, „egg laying defective“), blockieren programmierten Zelltod in somatischen Geweben. Der durch das Fehlen eines funktionalen ced-9 Gens ausgelöste ektopische Zelltod ist allerdings nicht von egl-1 abhängig, was bedeutet, dass das egl-1 Gen während der Zelltodexekution vor ced-9 wirkt (Abbildung 2). egl-1 kodiert ein Protein, welches zu einer Unterfamilie der Bcl-2-ähnlichen Zelltodregulatoren gehört, und zwar den „BH3-only“ Zelltodaktivatoren. „BH3only“ Proteine fungieren als wichtige Auslöser der Zelltodexekution, indem sie mit Bcl-2-ähnlichen Zelltodinhibitoren interagieren und diese negativ regulieren. Dementsprechend bindet das EGL-1 Protein von C. elegans an den Zelltodinhibitor CED-9, was dazu führt, dass die Interaktion zwischen CED-9 und CED-4 aufgehoben wird, CED-4 von Mitochondrien zu peri-nuklearen Membranen relokalisiert und den CED-3-abhängigen Zelltod induziert (Abbildung 3)[7]. Im Gegensatz zu Mutationen in ced-9, ced-4 und ced-3, die alle Formen von programmiertem Zelltod in C. elegans blockieren, blockieren Mutationen, die zum Funktionsverlust von egl-1 führen, den programmierten Zelltod in somatischen Geweben während der Entwicklung, nicht aber den BIOspektrum · 2/02 · 8. Jahrgang physiologischen Keimzelltod in der Gonade von Hermaphroditen (4, 8). Nicht-physiologischer Keimzelltod wird durch egl-1 Mutationen zu einem bestimmten Grad beeinträchtigt, was darauf hindeutet, dass es neben egl-1 einen weiteren Faktor gibt, der bei der Initiierung der Zelltodexekution nach Induktionen eine Rolle spielt (Abbildung 2) (5, 6). Es existieren somit in C. elegans mindestens drei Wege, auf denen die zentrale Exekutionsmaschinerie aktiviert werden kann. Die Faktoren, die neben egl-1 dafür in der Keimbahn notwendig sind, sind momentan noch nicht bekannt. Zelltyp-spezifische Regulation von egl-1 Die Aktivität von EGL-1 Homologen in Säugetieren wird durch verschiedene Mechanismen reguliert wie z.B. transkriptionale Regulation, Phosphorylierung, Proteolyse oder Sequestrierung. Ein Mechanismus, der bei der Regulation der Aktivität von EGL-1 eine Rolle zu spielen scheint, ist die transkriptionale Regulation. So konnte gezeigt werden, dass in den Hermaphroditenspezifischen Neuronen (HSNs), zwei serotonergen Neuronen, die in beiden Geschlechtern (Hermaphroditen und Männ- chen) gebildet werden, in Männchen allerdings durch programmierten Zelltod wieder eliminiert werden, das egl-1 Gen nur in Männchen transkriptional aktiv ist[9]. In Hermaphroditen wird die Transkription des egl-1 Gens durch das Zink-Finger DNS-Bindeprotein TRA-1 (tra, „transformer“) reprimiert (Abbildung 4). TRA-1, das zur Familie der Gli Transkriptionsfaktoren gehört, fungiert in C. elegans als terminaler Regulator des Geschlechts in somatischen Geweben. In Zellen, die durch programmierten Zelltod sexuellen Dimorphismus aufweisen, wird die zentrale Exekutionsmaschinerie somit durch egl-1 direkt von der Regulationskette kontrolliert, die das Geschlecht von somatischen Geweben determiniert. Mutationen im tra-1 Gen interferieren nur mit sexuell dimorphen Zelltodereignissen, wie dem programmiertem Zelltod der HSNs. Dies deutet darauf hin, dass der Schritt der Zelltodspezifizierung in somatischen Geweben während der Entwicklung von Zelltyp-spezifischen Faktoren bestimmt wird. Kandidaten für weitere Zelltyp-spezifische Regulatoren der zentralen Exekutionsmaschinerie und der egl-1 Transkription im speziellen, sind die Gene ces-1 und ces-2 (ces, „cell death selection abnormal“). Mu- Abb. 5: Die Zelltodspezifikation in den NSM Schwesterzellen von C. elegans kann als Model für aktue B-Zell Lymphomen dienen. Überblick 146 tationen, die zum Funktionsverlust von ces-2 oder der Überaktivierung des ces-1 Gens führen, blockieren den Zelltod einiger weniger Zellen, den NSM Schwesterzellen, zwei Zellen neuronaler Abstammung, die normalerweise durch programmierten Zelltod eliminiert werden[10, 11]. Der Phänotyp von ces-2 Mutationen hängt von einem funktionalen ces-1 Gen ab, was bedeutet, dass ces-1 nach ces-2 wirkt. Mutationen in beiden Genen sind von einem funktionalen egl-1 Gen abhängig, was weiterhin darauf hindeutet, dass sie vor egl-1 fungieren (Abbildung 5). Beide Gene kodieren DNS-Bindeproteine und somit potentielle Transkriptionsfaktoren. ces-2 kodiert ein b-ZIP und ces-1 ein Zink-Finger DNS-Bindeprotein. Ob CES-1 oder CES-2 wie TRA-1 direkte Regulatoren der egl-1 Transkription sind, ist allerdings noch nicht bekannt. Dass nicht nur die zentrale Zelltodexekutionsmaschinerie und einer ihrer wichtigen Aktivatoren, egl-1, von C. elegans bis zum Menschen konserviert ist, wurde durch Arbeiten am menschlichen Homolog von ces-2 demonstriert. Das menschliche Homolog von ces-2 ist das Proto-Onkogen HLF (HLF, „hepatic leukemia factor“), welches häufig die Ursache von akuten B-Zellen Lymphomen darstellt[12]. Die onkogene Form des HLF Proteins, das E2A-HLF Fusionsprotein, wirkt nicht wie HLF als transkriptionaler Repressor, sondern als konstitutiv aktiver transkriptionaler Aktivator. Die Expression dieses E2A-HLF Fusionsproteins in B Zellen blockiert den programmierten Zelltod von B Zellen, die normalerweise sterben sollten, und ermöglicht damit deren Transformation zu Tumorzellen. Aufgrund der Homologie zu CES-2 wurde postuliert, dass das E2A-HLF Fusionsprotein Zelltod blockiert, indem es die transkriptionale Aktivierung eines CES1-ähnlichen Zelltodinhibitors induziert (Abbildung 5). Die Suche nach Zielgenen von E2A-HLF resultierte tatsächlich in der Identifizierung des SLUG Gens, welches ein Homolog des C. elegans Transkriptionsfaktors CES-1 kodiert[13]. Phagocytose sterbender Zellen Die rasche Beseitigung von sterbenden oder toten Zellen und deren Phagocytose durch Nachbarzellen oder phagocytotischen Zellen ist ein wichtiger Aspekt des programmierten Zelltods[14]. Der Prozess umfasst spezifische Interaktionen zwischen zwei Zellen, der sterbenden Zelle und der phagocytierenden Zelle, und kann in folgende Schritte unterteilt werden: die Bildung eines „Fress mich“ Signals auf der Zelloberfläche der sterbenden Zelle, die Erkennung dieses Signals durch die phagocytierende Zelle und dessen Weiterleitung zur zel- Abb. 6: Darstellung der an der Phagocytose beteiligten Moleküle zur Ausbildung und Transduktion des „Fress mich“ Signals. lulären Maschinerie der phagocytierenden Zelle. Zwei partiell redundante Genkaskaden, definiert durch die Gene ced-1, ced-6 und ced-7 einerseits und ced-2, ced-5, ced-10 und ced-12 andererseits, sind in C. elegans für die Phagocytose sterbender oder toter Zellen notwendig (Abbildung 1)[15, 16]. Mutationen, die die Funktion einer Kaskade inaktivieren, blockieren den Prozess der Phagocytose nur im geringen Maße. Nur wenn beide Kaskaden inaktiviert sind, wird die Phagocytose der meisten sterbenden Zellen blockiert. ced-1, ced-6 und ced-7, wie auch ihre menschlichen Homologe, spielen bei der Bildung des „Fress mich“ Signals in der sterbenden Zelle und bei der Erkennung des Signals in der phagocytierenden Zelle eine wichtige Rolle. ced-1 kodiert für einen „Scavenger“ Rezeptor, der auf der Plasmamembran der phagocytierenden Zelle exprimiert wird. ced-6 kodiert für ein Adaptorprotein, das in der phagocytierenden Zelle agiert. ced-7 kodiert für einen ABC-Transporter, der während der Phagocytose in beiden Zellen, der sterbenden wie auch der phagocytierenden, notwendig ist (Abbildung 6). ced-2, ced-5, ced-10 und ced-12 sind an der Weiterleitung des „Fress mich“ Signals zur zellulären Maschinerie der phagocytierenden Zelle beteiligt (Abbildung 6). Alle vier Gene fungieren in der phagocytierenden Zelle und kodieren Komponenten einer konservierten Rac GTPase Signaltransduktionskette. Sie sind somit an der Regulation des Aktincytoskeletts und der Kontrolle von Zellformveränderungen beteiligt, die für die Phagocytose der sterbenden Zelle notwendig sind. ced-2 kodiert für ein Adaptorprotein, welches dem menschlichen CrkII Protein ähnelt. ced-5 kodiert für ein Mitglied der Dock180 Familie von Proteinen. ced-12 kodiert ebenfalls für ein konserviertes Adaptorprotein, ein ELMO-ähnliches Protein, und ced-10 kodiert für ein Rac-ähnliches Protein, das zur Familie der Rho/Rac/CDC42ähnlichen GTPasen gehört. Die Überexpression von ced-10 supprimiert den Phagocytosedefekt von Tieren mit Mutationen in ced-2, ced-5 oder ced-12, was bedeutet, dass diese drei Gene durch ced-10 und sein Genprodukt wirken. Das CED-2 Adaptorprotein interagiert mit CED-5, welches wiederum mit CED-12 interagiert. Es wurde postuliert, dass dieser aus CED-2, CED-5 und CED12 bestehende tertiäre Komplex nach der Aktivierung eines Rezeptors durch das „Fress mich“ Signal an die Plasmamembran der phagocytierenden Zelle rekrutiert wird und die Aktivierung der membranständigen, inaktiven CED-10 GTPase bewirkt. Aktives CED-10 induziert daraufhin Veränderungen im Aktincytoskelett, die für den Phagocytoseprozess und die Vereinleibung der sterbenden Zelle notwendig sind. Die Identität des „Fress mich“ Signals und dessen Rezeptors, sowie die Identität der Substrate von CED-10 sind bislang noch nicht bekannt. Studien in Säugetierzellen haben gezeigt, dass der Schritt der Phagocytose ebenfalls konserviert ist. Homologe der C. elegans Proteine CED-2, CED-5, CED-12 und CED10 sind auch in höheren Organismen an der Phagocytose von sterbenden Zellen beteiligt. Degradation von toten Zellen Die Fragmentierung des Chromatins der sterbenden Zelle zu einer typischen chromosomalen DNS Leiter ist ein Teil des Degradationsschritts und ein Charakteristikum des programmierten Zelltods. Mindestens zwei Nukleasen scheinen in C. elegans bei diesem Prozess eine Rolle zu spielen: NUC1 (nuc, „abnormal nuclease“), ein Homolog der DNase II von Säugern[17] und CPS-6 (cps, „CED-3 caspase suppressor“), ein Homolog der Endonuclease EndoG in Säugern[18]. Mutationen im nuc-1 oder cps-6 Gen interferieren mit der Fragmentierung des Chromatins. NUC-1 ist auch an der Fragmentierung von bakterieller DNS im Darm von C. elegans beteiligt. CPS-6 dagegen scheint speziell während der Degradation der chromosomalen DNS in sterbenden Zellen eine Rolle zu spielen. Offene Fragen Obwohl wir durch die Studien in C. elegans bereits viel über die einzelnen Schritte des programmierten Zelltods gelernt haben, bleiben noch eine Reihe von Fragen unbeantwortet. BIOspektrum · 2/02 · 8. Jahrgang Überblick 147 Wie bereits erwähnt, ist die Identität der Signale, die an der Zelltodspezifizierung in der weiblichen Keimbahn von C. elegans beteiligt sind, noch unbekannt. Des weiteren ist unklar, wie die Phagocytosemaschinerie aktiviert wird, d.h. wie die Aktivierung von CED-3 zur Bildung des „Fress mich“ Signals führt. Unbeantwortet ist auch die Frage, wie die Nukleasen in den sterbenden Zellen aktiviert werden. Einige vor kurzem gemachte Beobachtungen deuten außerdem darauf hin, dass der Schritt von Zelltodexekution und Phagocytose von sterbenden Zellen kein linearer Prozess ist. In Tieren, die homozygot für Mutationen sind, die zu einem partiellen Verlust von ced-3 oder ced-4 führen, ist der Zelltod einiger weniger Zellen blockiert. Tragen die Tiere allerdings zusätzlich eine Mutation, die die Phagocytose blockiert, überleben weit mehr Zellen als in den Einzelmutanten[19, 20]. Dies deutet darauf hin, dass die an der Phagocytose beteiligten Gene ebenfalls an der terminalen Zelltodexekution beteiligt sind. Die Experimente zeigten auch, dass es einen „halbtoten“ Zustand gibt, d.h. dass Zellen, die bereits die Charakteristika von sterbenden Zellen aufwiesen, sich von diesem Zustand erholen konnten und wieder funktional waren. [8] Conradt B, Horvitz HR. The C. elegans protein EGL-1 is required for programmed cell death and interacts with the Bcl-2-like protein CED-9. Cell. 1998 May 15; 93 (4): 519–29. [9] Conradt B, Horvitz HR. The TRA-1A sex determination protein of C. elegans regulates sexually dimorphic cell deaths by repressing the egl-1 cell death activator gene. Cell. 1999 Aug 6; 98 (3): 317–27. [10] Metzstein MM, Hengartner MO, Tsung N, Ellis RE, Horvitz HR. Transcriptional regulator of programmed cell death encoded by Caenorhabditis elegans gene ces-2. Nature. 1996 Aug 8; 382 (6591): 545–7. [11] Metzstein MM, Horvitz HR. The C. elegans cell death specification gene ces-1 encodes a snail family zinc finger protein. Mol Cell. 1999 Sep; 4 (3): 309–19. [12] Inukai T, Inoue A, Kurosawa H, Goi K, Shinjyo T, Ozawa K, Mao M, Inaba T, Look AT. SLUG, a ces-1-related zinc finger transcription factor gene with antiapoptotic activity, is a downstream target of the E2AHLF oncoprotein. Mol Cell. 1999 Sep ;4 (3): 343–52. Ashmun RA, Canman CE, Laken SJ, Kastan MB, Look AT. Reversal of apoptosis by the leukaemia-associ- ated E2A-HLF chimaeric transcription factor. Nature. 1996 Aug 8; 382 (6591): 541–4. [14] Savill J, Fadok V. Corpse clearance defines the meaning of cell death. Nature. 2000 Oct 12; 407 (6805): 784–8. [1] Lockshin RA, Zakeri Z. Programmed cell death and apoptosis: origins of the theory. Nat Rev Mol Cell Biol. 2001 Jul; 2 (7): 545–50. [2] The C. elegans Sequencing Consortium Genome sequence of the nematode C. elegans: a platform for investigating biology. Science. 1998 Dec 11; 282 (5396): 2012–8. [4] Gumienny TL, Lambie E, Hartwieg E, Horvitz HR, Hengartner MO. Genetic control of programmed cell death in the Caenorhabditis elegans hermaphrodite germline. Development. 1999 Feb; 126 (5): 1011–22. Nicole Wittenburg Jahrgang 1971, Biologiestudium an der Friedrich-Alexander Universität Erlangen, 1997 Diplomarbeit bei Prof. W. Hillen, 2000 Promotion bei Prof. R. Baumeister am Genzentrum in München, seitdem Postdoktorandin bei B. Conradt am Max-Planck Institut für Neurobiologie in Martinsried [5] Gartner A, Milstein S, Ahmed S, Hodgkin J, Hengartner MO. A conserved checkpoint pathway mediates DNA damage-induced apoptosis and cell cycle arrest in C. elegans. Mol Cell. 2000 Mar; 5 (3): 435–43. [6] Aballay A, Ausubel FM. Programmed cell death mediated by ced-3 and ced-4 protects Caenorhabditis elegans from Salmonella typhimurium-mediated killing. Proc Natl Acad Sci U S A. 2001 Feb 27; 98 (5): 2735–9. [7] Chen F, Hersh BM, Conradt B, Zhou Z, Riemer D, Gruenbaum Y, Horvitz HR. Translocation of C. elegans CED-4 to nuclear membranes during programmed cell death. Science. 2000 Feb 25; 287 (5457): 1485–9. BIOspektrum · 2/02 · 8. Jahrgang [16] Conradt B. Cell engulfment, no sooner ced than done. Dev Cell. 2001 Oct; 1 (4): 445–7. [17] Wu YC, Stanfield GM, Horvitz HR. NUC-1, a Caenorhabditis elegans DNase II homolog, functions in an intermediate step of DNA degradation during apoptosis. Genes Dev. 2000 Mar 1; 14 (5): 536–48. [18] Parrish J, Li L, Klotz K, Ledwich D, Wang X, Xue D. Mitochondrial endonuclease G is important for apoptosis in C. elegans. Nature. 2001 Jul 5; 412 (6842): 90–4. [19] Hoeppner DJ, Hengartner MO, Schnabel R. Engulfment genes cooperate with ced-3 to promote cell death in Caenorhabditis elegans. Nature. 2001 Jul 12; 412 (6843): 202–6. [20] Reddien PW, Cameron S, Horvitz HR. Phagocytosis promotes programmed cell death in C. elegans. Nature. 2001 Jul 12; 412 (6843): 198–202. [13] Inaba T, Inukai T, Yoshihara T, Seyschab H, Literaturverzeichnis [3] Horvitz, H.R. Genetic control of programmed cell death in the nematode Caenorhabditis elegans. Cancer Res. 1999 Apr 1; 59 (7 Suppl): 1701s-1706s. [15] Hengartner MO. Apoptosis: Corralling the Corpses. Cell 2001 104: 325–328. Barbara Conradt Jahrgang 1965, Studium der Agrar- und Molekularbiologie an der Universität Hohenheim und der University of Massachusetts, Amherst, USA, 1994 Promotion bei Prof. W.T. Wickner an der University of California, Los Angeles, USA, 1994–1999 Postdoktorandin bei Prof. H.R. Horvitz am Massachusetts Institute of Technology, Cambridge, USA, seitdem Leiterin einer selbständigen Nachwuchsgruppe am MaxPlanck Institut für Neurobiologie in Martinsried Korrespondenzadresse: Dr. Barbara Conradt Max-Planck Institut für Neurobiologie Am Klopferspitz 18a 82152 Martinsried Tel.: 089-8578-3625 Fax: 089-8578-2411 e-mail: [email protected]