Allgemeine Grundlagen Replikation - Ruhr
Werbung
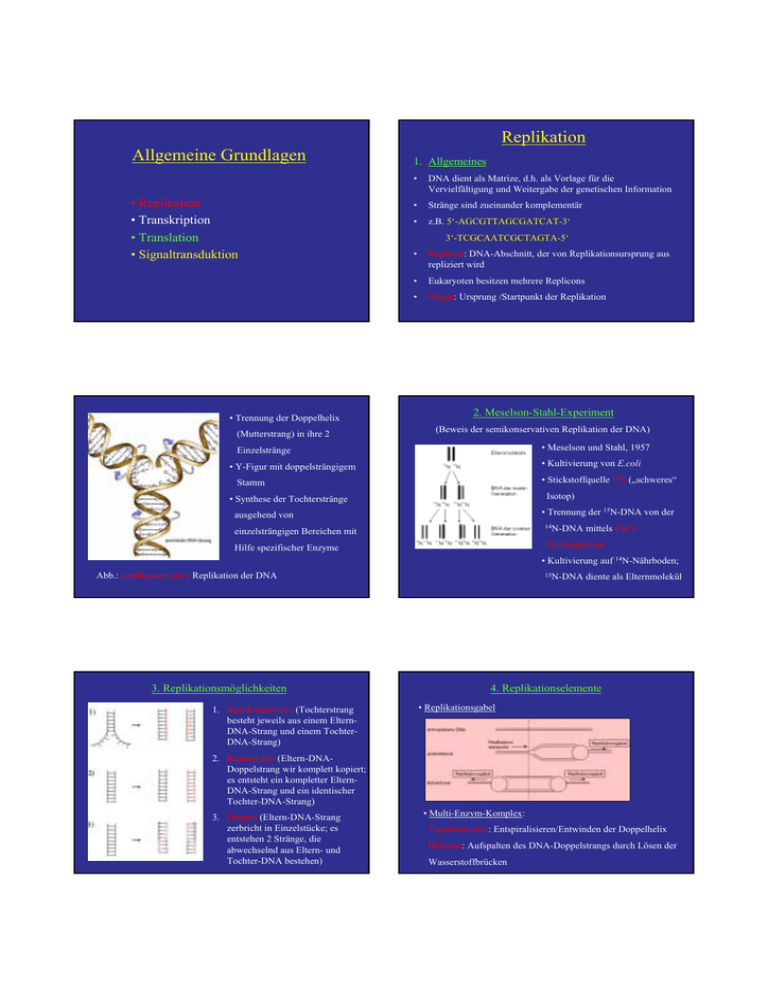
Replikation Allgemeine Grundlagen • Replikation • Transkription • Translation • Signaltransduktion • Trennung der Doppelhelix (Mutterstrang) in ihre 2 1. Allgemeines • DNA dient als Matrize, d.h. als Vorlage für die Vervielfältigung und Weitergabe der genetischen Information • Stränge sind zueinander komplementär • z.B. 5‘-AGCGTTAGCGATCAT-3‘ • Replicon: DNA-Abschnitt, der von Replikationsursprung aus repliziert wird • Eukaryoten besitzen mehrere Replicons • Origin: Ursprung /Startpunkt der Replikation 3‘-TCGCAATCGCTAGTA-5‘ 2. Meselson-Stahl-Experiment (Beweis der semikonservativen Replikation der DNA) • Meselson und Stahl, 1957 Einzelstränge • Kultivierung von E.coli • Y-Figur mit doppelsträngigem • Stickstoffquelle 15N („schweres“ Stamm Isotop) • Synthese der Tochterstränge • Trennung der 15N-DNA von der ausgehend von 14N-DNA einzelsträngigen Bereichen mit mittels CsCl- Dichtegradient Hilfe spezifischer Enzyme • Kultivierung auf 14N-Nährboden; Abb.: semikonservative Replikation der DNA 3. Replikationsmöglichkeiten 1. Semikonservativ (Tochterstrang besteht jeweils aus einem ElternDNA-Strang und einem TochterDNA-Strang) 15N-DNA diente als Elternmolekül 4. Replikationselemente • Replikationsgabel 2. Konservativ (Eltern-DNADoppelstrang wir komplett kopiert; es entsteht ein kompletter ElternDNA-Strang und ein identischer Tochter-DNA-Strang) 3. Dispers (Eltern-DNA-Strang zerbricht in Einzelstücke; es entstehen 2 Stränge, die abwechselnd aus Eltern- und Tochter-DNA bestehen) • Multi-Enzym-Komplex: Topoisomerase: Entspiralisieren/Entwinden der Doppelhelix Helicase: Aufspalten des DNA-Doppelstrangs durch Lösen der Wasserstoffbrücken •1 • ssBP: Einzelstrang-bindendes Protein (Stabilisierung) • Übersicht Replikationskomplex • Primase: Synthese des Primers (kurze RNA-Sequenz) • DNA-Polymerase: Synthese des komplementären Stranges in 5‘-3‘-Richtung • RNase: Abbau der RNA-Primer • Ligase: Verbinden der einzelnen DNA-Stücke • DNA-Polymerisation • Voraussetzungen für Polymerisation: Matrize (Eltern-DNAStrang) und Primer (mit freier 3‘-OH-Gruppe am Ende) • Korrekturlesefunktion: Entfernung falsch eingebauter Nucleotide •2 •3 •4 Dnarepli.swf 5. Mechanismus • Synthese kurzer Primersequenzen (ca. 10 nt) durch RNAPrimase; Verlängerung durch DNA-Polymerase zu Okazaki-Fragment (ca. 1000 nt) bis zum 5‘-Ende des vorherigen Fragmentes • Topoisomerase entspannt die superhelikale Struktur der DNA (Spaltung der DNA, Entwinden, Verschliessen) • Helicase trennt die Doppelhelix in 2 Einzelstränge durch Lösen der Wasserstoffbrücken zwischen A/T bzw. G/C • Abbau der Primer durch RNase; Auffüllen der Lücken durch DNA-Polymerase • Primase synthetisiert RNA-Primer als Startpunkt für die DNA-Polymerase (freies 3‘-OH-Ende) • DNA-Ligase verbindet aufeinandertreffende 3‘-und 5‘Enden zu kontinuierlichem Strang • Polymerase synthetisiert komplementären Tochterstrang ausgehend vom 3‘-Ende des Primers • • kontinuierliche Synthese am Leitstrang (5‘-3‘-Richtung) Beenden (Termination) der Replikation durch Bindung des Proteins Tus (terminator utilization substance) an DNA; Blockieren der Helicase • Folgestrang: Polymerase muss in entgegengesetzter Richtung zur Helicase arbeiten • Topoisomerase trennt den restlichen unreplizierten Abschnitt Übungsaufgaben: Replikation 1. Das menschliche Genom besteht aus ca. 3 x 109 bp. Die DNA-Polymerase arbeitet mit einer Geschwindigkeit von ca. 50 bp/s. Wie lange würde demzufolge ein Replikationszyklus dauern, wenn angenommen wird, dass nur ein Replicon existiert? 3x 109 bp / 50 bp x s-1 ⇒ 6 x 107 s ⇒ etwa 695 Tage (ca. 2 Jahre) 2. Aus der Lösung der Aufgabe 1 ist ersichtlich, dass die Replikation des menschlichen Genoms an mehreren Orten gleichzeitig starten muss, da sie für das gesamte Genom etwa 20 h dauert. Wieviele Origins müssen demnach auf dem Genom mindestens vorhanden sein? ⇒833 Origins 1 Origin: Replikation dauert 6 x 107 s = 1 Mio min = 16 666,7 h / 20 h = 833,3 Origins •5 3. Weshalb findet die Replikation ausschliesslich in 5‘-3‘Richtung statt? Elternmoleküle 15N ⇒ Die DNA-Polymerase benötigt das 3'-OH-Ende, um 15N DNA der ersten Nukleotide anhängen zu können. Damit ist die Richtung Generation der Replikation definiert. 14N 15N 15N 14N 4. Worin liegt der Unterschied in der Funktion einer Topoisomerase und einer Helicase? 2. Generation ⇒ Die Topoisomerase überwindet die Superwindung der 14N 14N 14N 15N 15N 14N 14N 14N DNA, indem sie den einen Strang spaltet, abwickelt und wieder verschliesst. Damit macht sie die DNA der Helicase 3. Generation zugänglich, die nun die Stränge voneinander trennen kann. 14N 14N 14N 14N/15N 14N/15N 14N 14N 14N Transkription Informationsfluss 1. Allgemeines Transkription DNA RNA Zellkern Prokaryoten • Translation 1 RNA-Polymerase bestehend aus Core (Kern) mit den Untereinheiten α2, β und β‘ sowie dem σFaktor Protein • erster Schritt der Proteinbiosynthese • Basensequenz der DNA wird in mRNA (Botenoder messenger-RNA) umgeschrieben • dabei auch Bildung von tRNA (Transfer- oder Transport-RNA) und rRNA (ribosomale RNA) • tRNA: Transport aktivierter Aminosäuren zu Ribosomen • rRNA: Bestandteil der Ribosomen Cytoplasma Eukaryoten • 3 RNA-Polymerasen aus bis zu 12 Untereinheiten • RNAP I synthetisiert rRNA • RNAP II und III synthetisieren mRNA und tRNA 2. Initiation (Start) • RNA-Polymerase bewerkstelligt Abschreiben eines Gens • Verwendung von DNA als Substrat sowie den RibonucleotidTriphosphaten ATP, UTP, CTP und GTP führt zu einem zum DNA-Strang komplementären mRNA-Strang • Polymerase bindet an bestimmte Nucleotidsequenz (Promotor) • Prokaryoten: 10 Nucleotide vor dem Transkriptionsstart (Pribnow-Schaller-Box); Erkennung dieser Sequenz durch σFaktor, der nach der Bindung an die DNA abdissoziiert • Eukaryoten: unmittelbar vor dem Startpunkt (GoldbergHognes-Box); Sequenz: TATAA RNA-Polymerasen benötigen keinen Primer und haben keine Reparaturfunktion! •6 3. Elongation (Kettenverlängerung) • • Polymerase synthetisiert mRNA-Strang, der komplementär zum Matrizen-DNA-Strang ist • Nucleotide: ATP, UTP (anstelle von TTP), CTP und GTP • Synthese erfolgt in 5‘-3‘-Richtung Entwindung der DNA durch RNA-Polymerase-ProteinKomplex, Synthese der mRNA und sofortiges Verschliessen der DNA 4. Termination (Ende der Transkription) • 3 Stop-Signalsequenzen in der DNA: ATT, ATC, ACT • vorgelagerte GC-reiche Sequenzen, die Haarnadelschleifen ausbilden können ⇒ Transkriptionsabbruch • Beteiligung von Proteinen: Rho-Protein führt unter ATPSpaltung zur Freisetzung der mRNAs Modifikationen der mRNA • Aufbereitung der mRNA für den Transport aus dem Zellkern durch die Kernporen in das Cytoplasma; erfolgt noch vor dem Spleißen • Capping (cap = Kappe): Anhängen einer Art Kappe aus mehreren Methylguaninmolekülen (Veränderung des Guanins) an das 5‘-Ende; wichtig für die Bindung an das Ribosom während der Translation • Polyadenylation: Anhängen einer bis zu 200 Nucleotide langen Sequenz aus Adenylatresten (Poly-A-Schwanz) an das 5‘-Ende; vermutlich Stabilisierung der mRNA • am Ende wird die mRNA mit Hilfe von Proteinen aus dem Zellkern in das Cytoplasma zu den Ribosomen transportiert Reifung der RNA (Processing) • Unterteilung eukaryotischer Gene in Exons (codierende Sequenzen) und Introns (nichtcodierende Sequenzen) • pro Gen bis zu 50 der 65 bis 100000 nt langen Introns möglich • Spleißen: Entfernen der Intronsequenzen, erfolgt im Zellkern • Beteiligung von Proteinen Codogen – Codon - Anticodon •7 Der Genetische Code • „Sprache der Gene“ wird mit Hilfe der Ribosomen in die „Sprache der Proteine“ übersetzt • deutsche Sprache: 26 Buchstaben; Computersprache: 2 Zeichen (0 und 1) ⇒ Speicherung von Informationen durch Kombination der Zeichen, d.h. Wörter und Sätze • genetische Sprache: 4 Buchstaben (A, T, C, G) • Proteinsprache: 20 Buchstaben (Aminosäuren) • Triplett-Code: Verschlüsselung der 20 Aminosäuren durch unterschiedliche Kombination der 4 Basen: 43 = 64 Möglichkeiten • 61 Tripletts codieren für Aminosäuren; 3 sind Stop-Codone, für die es keine Aminosäure gibt; Proteinsynthese bricht an der Stelle ab • eindeutig: eine bestimmte Nucleotidsequenz legt eine bestimmte Aminosäuresequenz fest • degeneriert: die Mehrzahl der AS wird durch mehr als ein Triplett codiert • kommafrei: die Tripletts schliessen lückenlos aneinander • nicht überlappend: ein Nucleotid ist immer nur Bestandteil eines Tripletts • universell: der genetische Code gilt für alle Organismen gleichermassen Translation 1. tRNA und Ribosomen • an der Translation beteiligt: Enzyme, Proteine, Ribosomen, mit Aminosäuren beladene tRNAs und die mRNA • tRNA = Übersetzermolekül, trägt an einem Ende die spezifische AS und am anderen Ende das Anticodon (passend zur mRNA) • Anticodon-Schleife zum Abtasten der mRNA; D- und T-Schleife dienen der Anheftung an das Ribosom Das Enzym Aminoacyl-tRNA-Synthase belädt die tRNA im Cytoplasma mit den Aminosäuren. •8 Ribosomen • Orte der Translation • Zellen besitzen tausende Ribosomen (E.coli ca. 15 000) • in Eukaryoten frei oder gebunden an das Endoplasmatische Reticulum oder die Kernmembran vorkommend, auch in Mitochondrien und Chloroplasten zu finden • Unterteilung in grosse und kleine Untereinheit • Prokaryoten: 70S Ribosomen (50S und 30S Untereinheit) • Eukaryoten: 80S Ribosomen (60S und 40S Untereinheit) Typischer Aufbau eines eukaryotischen Ribosoms 2. Ablauf der Translation 3D-Modell eines E.coli-Ribosoms mit kleiner (gelb) und grosser (blau) Untereinheit. Die mRNA ist braun, die beiden tRNA-Moleküle sind magenta und grün. • Die Codon-Sequenz der mRNA wird in die Aminosäuresequenz des Proteins übersetzt • mRNA enthält Start- und Stop-Codon (für Beginn und Ende der Translation wichtig) • mRNA fädelt sich in Ribosom ein • mit Aminosäuren beladene tRNAs tasten mit ihrem Anticodon mRNA Stück für Stück ab bis zum fertigen Protein • Bildung von Peptidbindungen zwischen den Aminosäuren • 3 Phasen: Initiation (Start), Elongation (Kettenverlängerung) und Termination (Ende) Initiation • kleine UE des Ribosoms und die Start-tRNA (trägt Methionin) binden an die mRNA • Startcodon: AUG • Zwischen Sartcodon AUG und der Start-tRNA kommt es zur Basenpaarung durch Wasserstoffbrücken • Methionin: erste AS, trägt NH2-Gruppe am Ende • Anlagerung der grossen UE an die kleine UE; grosse UE besitzt 2 Bindungsstellen für tRNA-Moleküle (Eingang und Ausgang) •9 Elongation • Start-tRNA-Komplex wandert ein Codon weiter an den P-Ort • A-Ort (Eingang) ist frei für neue tRNA (Abb. Prolin) • nach Bindung der Prolin-tRNA an die mRNA wird zwischen den AS Methionin und • Ribosom „rutscht“ ein Codon weiter in 5‘-3‘Richtung • verbliebene tRNA wandert dann an den POrt (Ausgang); A-Ort (Eingang) wird frei • Vorgang geht weiter in 5‘-3‘-Richtung • immer neue passende, beladene tRNA-Moleküle binden an AOrt, Peptidbindung wird geknüpft, tRNAs rutschen an P-Ort bis Stop-Codon auftritt • Abb.: Basenpaarung desAnticodons der tRNA mit dem Codon der mRNA sowie die Peptidbindung Prolin eine Peptidbindung geknüpft • tRNA für Methionin verlässt das Ribosom Termination (Ende) • Stop-Codons: UAA, UAG und UGA • Abbruch der Synthese, da keine passende tRNA vorhanden • Ribosomen-Untereinheiten fallen von der tRNA ab • Proteine tragen Signalsequenz am Ende für Transport Signaltransduktion Polysomen • eine mRNA fädelt sich oft in mehrere Ribosomen hintereinander • gleichzeitige Entstehung von Polypeptidketten (Proteinen) mit steigender Kettenlänge 2. Signalmoleküle • Hydrophile (wasserlösliche) Signalmoleküle (löslich oder auf Zelloberflächen) – Zelloberflächenrezeptoren • Hydrophobe (wasserunlösliche) Signalmoleküle (Steroide, Vitamine, Tyroxin) – intrazelluläre Rezeptoren • Beispiele: Proteine, Peptide, Aminosäuren, Nucleotide, Steroide (Cortisol, Sexualhormone, Vitamin D), Fettsäurederivate, Stickstoffmonoxid, Kohlenstoffmooxid 1. Definition • Übertragung extrazellulärer (von aussen) Signale ins Zellinnere bzw. Umwandlung extrazellulärer Signale in zellinterne Signale, Signalverstärkung und Vermittlung einer spezifischen Zellantwort •10 Prozesse im Inneren der Zellen besitzen ein ausgefeiltes System, um auf Signale von aussen Zelle: reagieren zu können: • GTP-bindende Proteine (wichtige Klasse von Transmembranrezeptoren) • Rezeptoren mit enzymatischer Wirkung (Transmembranproteine, die ausserhalb der Zelle das Signalmolekül binden und innen die enzymatische Reaktion auslösen) • 1. Anhängen von Phosphatgruppen an andere Proteine durch Proteinkinasen 2. Abspaltung von Phosphatgruppen an andere Proteine durch Proteinphosphatasen Intrazelluläre Rezeptorproteine (Signal gelangt durch die Zellmembran und verbindet sich mit dem Rezeptor) Erläuterung zur Abbildung Signaltransduktion: 3. Phasen der Signaltransduktion 1. Ein G-Protein-abhängiger Rezeptor wird durch ein Signal aktiviert. Dadurch wird ein second messenger aktiviert (Ca2+ oder cAMP). Dieser aktiviert seinerseits Kinasen. Deren Aktivierung führt zur Expression ganz bestimmter Gene. Bindung des Signalmoleküls an Membranrezeptoren 2. Durch ein äußeres Signal dimerisiert der Tyrosinkinaserezeptor. Im Inneren phosphoryliert er bestimmte Kinasen. Diese aktivieren dann ganz bestimmte Transkriptionsfaktoren, die bestimmte Gene exprimieren. Weiterleitung des Signals über die Zellmembran hinweg 3. Ein Steroidhormon diffundiert durch die Zellmembran. Im Inneren der Zelle verbindet es sich mit einem Rezeptor. Dieser Komplex kann nun in den Kern diffundieren und dort die Expression bestimmter Gene auslösen. Ligand (z.B.) Hormon durchdringt die Zellmembran Bindung des Liganden an einen Rezeptor innerhalb der Zelle, Weiterleitung des Signals in der Zelle Intrazelluläre Signaltransduktion Steuerung von Transkription, Translation, Stoffwechsel, etc. Abb.: TransmembranRezeptor 4. Signalübertragung durch Transmembranrezeptoren • Übertragung einer Information über die Zellmembran hinweg • äusseres Signal (primärer messenger) bindet an einen Rezeptor auf der Zellmembran und löst im Zellinneren eine Reaktion aus, z.B.: • Einleiten der Genexpression (Transkription, Translation) • Veränderung der Aktivität eines Enzyms • Umorganisieren des Cytoskeletts der Zelle • Veränderung der Durchlässigkeit der Membran • äusserer Teil ragt in Extrazellularraum und bindet Signalmoleküle • Einleiten der Mitose • • Einleiten der Apoptose (Selbstmord der Zelle) durch mittleren Teil erfolgt Verankerung des Proteins in der Zellmembran (z.T. bis zu 7 Membrandurchgänge) • innerer Teil ragt ins Cytoplasma und löst eine Reaktion aus •11 5. G-Protein-gekoppelte Rezeptoren • • cAMP-Weg: Das G-Protein aktiviert die Adenylatcyclase (Enzym), die cAMP bildet. Das entstandene cAMP (second messenger, 2. Botenstoff) aktiviert das Zielprotein. • Ca2+-Weg: Das G-Protein aktiviert ein Enzym, das seinerseits die Öffnung von Ionenkanälen für Ca2+(second messenger) bewirkt. Das freigesetzte Ca2+ aktiviert das Zielprotein. Wichtige Klasse von Rezeptoren, die einen „second messenger“ (2. Botenstoff im Inneren der Zelle) aktivieren •12