Modelle zur Kontrolle der Kopienzahl
Werbung

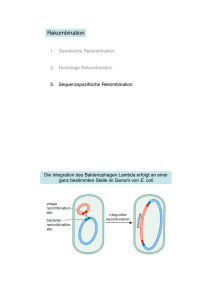
Timing, self-control and a sense of direction are the secrets of multicopy plasmid stability David Summers Department of Genetics, Downing Street, Cambridge, UK Ausgangssituation Fragen: Bekannt ist: multicopy plasmide werden bei der Zellteilung zufällig auf die Tochterzellen übertragen ● natürliche Plasmide sind extrem stabil hinsichtlich ihrer Kopienzahl ● künstliche Vektoren (pUC-Reihe) gehen unter nicht-selektiven Bedingungen schnell verloren ● multicopy-plasmide können durch Rekombination leicht multimerisieren ● ● wie wird die Kopienzahl reguliert? ● wie werden Multimere aufgelöst? Modelle zur Kontrolle der Kopienzahl A) Sufenmodell: hohe Replikationsrate bis zum Sollwert, dann kompletter Stop B) Exponentielle Kinetik: Mittelwert C) Hyperbolische Kinetik: umgekehrte Proportionalität zw.Kopienzahl und Replikationsrate Replikationskontrolle des Plasmids ColE1 ● ● ● ● ● RNAII Präprimertranskript wird transkribiert hybridisiert am OriV Spaltung durch RNAse H Spaltprodukt bildet Replikationsprimer Replikation startet Replikationskontrolle des Plasmids ColE1 ● ● ● RNAI-Repressortranskript interagiert mit RNAII Veränderte Faltung der RNA keine Primerbildung, keine Transkription Folgen der Plasmid-Multimerisierung Bereits kleiner Anteil an Dimeren wirkt sich aus ● Chance, dass plasmidfreie Zellen entstehen ● Dimer hat doppelte Chance, repliziert zu werden ● Dimere verdrängen Monomere ● Dimer Katastrophe ● ● Aber: Zellen mit Multimeren wachsen langsamer Auflösung von Plamid-Multimeren Replikationsstellen auf Plasmiden: cer-site aus 240 Bp ●Bindestelle für XerC- und XerD-Rekobinase ● Weitere Proteine: ArgR und PepA ●Rekombination an cer folgt topologischem Zwang ●XerCD könnte in cis und in trans rekombinieren ● Topologischer Zwang muss von anderen Proteinen stammen ArgR bildet in Gegenwart von Arginin eine Hexamer ● enthält DNA-Bindedomäne ● bindet an ARG-Boxen (z.B. cer-site) ● biegt DNA um 70-90° ● Mutation von ArgR senkt Zwang zum cis-Produkt ● Zustand im Plasmid-Monomer: ArgR, XerCD und PepA bilden Komplex an cer-site ArgR kann als „Dimer von Trimeren“ angesehen werden Ein Trimer kann die cer-site binden Zustand im Plasmid-Dimer ● ● ● 2 single-site Komplexe assoziieren Synaptischer Komplex ArgR bildet Brücke Zustand im Plasmid-Dimer ● ● ● ● Arg alleine ist zu schwach um den Komplex zu stabilisieren Stabilisierung durch Anlagerung von Supercoil-DNA Stabilisierung kann nur in cis funktionieren Topologisch einzigartiges Rekombinationsprodukt Verbindung zwischen Rekombination und Zellteilung ● ● ● ● ● Promotor Pcer an der cer-site Genprodukt (Rcd) inhibiert Wachstum der Zellen Stopp des Zell-Zyklus c(Rcd) ist in Zellen mit Multimeren erhöht Synaptische Komplexe verändern DNA-Struktur und ermöglichen Transkription von Pcer Rcd bildet eine Checkpoint bei der Replikation Der Rcd-Checkpoint ● ● ● ● ● Bei Zellteilung mit unvollständiger Multimerauflösung besteht die Gefahr plasmidloser Tochterzellen Problem: langsamer Rekombinationsmechanismus Erster Strangaustausch wird von XerC katalysiert Bildung der Holiday-junction Kein XerD-katalysierter Austausch den Gegenstrangs ● ● ● Komplex muss disoziieren, Auflösung der Holiday-junction erfolgt durch zelleigene Resolvase Verlangsamung des Gesamtvorgangs Erhöhung der Chance von Multimeren Zusammenfassung ● Plasmid-Multimere stellen die Zelle vor Probleme ● Xer-cer Rekombination löst Multimere auf ● Langsamer Vorgang ● Rcd-Checkpoint sichert Replikation ab