Translation - Werner-von-Siemens
Werbung
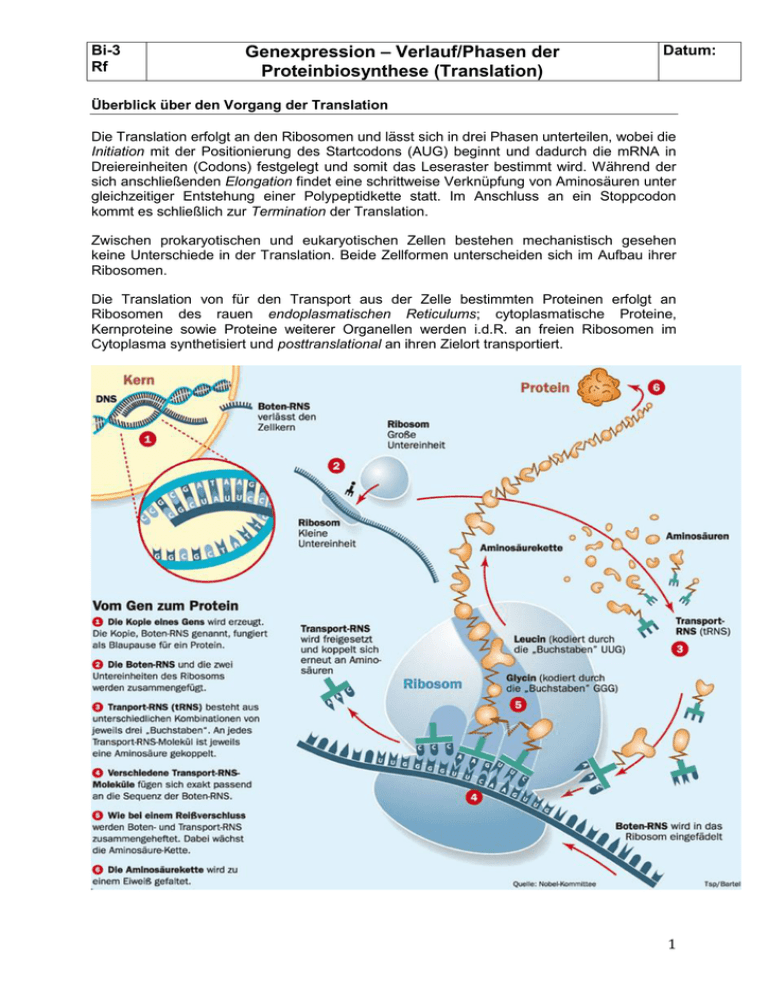
Bi-3 Rf Genexpression – Verlauf/Phasen der Proteinbiosynthese (Translation) Datum: Überblick über den Vorgang der Translation Die Translation erfolgt an den Ribosomen und lässt sich in drei Phasen unterteilen, wobei die Initiation mit der Positionierung des Startcodons (AUG) beginnt und dadurch die mRNA in Dreiereinheiten (Codons) festgelegt und somit das Leseraster bestimmt wird. Während der sich anschließenden Elongation findet eine schrittweise Verknüpfung von Aminosäuren unter gleichzeitiger Entstehung einer Polypeptidkette statt. Im Anschluss an ein Stoppcodon kommt es schließlich zur Termination der Translation. Zwischen prokaryotischen und eukaryotischen Zellen bestehen mechanistisch gesehen keine Unterschiede in der Translation. Beide Zellformen unterscheiden sich im Aufbau ihrer Ribosomen. Die Translation von für den Transport aus der Zelle bestimmten Proteinen erfolgt an Ribosomen des rauen endoplasmatischen Reticulums; cytoplasmatische Proteine, Kernproteine sowie Proteine weiterer Organellen werden i.d.R. an freien Ribosomen im Cytoplasma synthetisiert und posttranslational an ihren Zielort transportiert. 1 Bi-3 Rf Genexpression – Verlauf/Phasen der Proteinbiosynthese (Translation) Datum: Bau und Funktion der Ribosomen (Quelle: aus Wikipedia) 1. Allgemeines: Ribosomen sind makromolekulare Komplexe aus Proteinen und Ribonukleinsäuren (RNA), die im Cytoplasma, in den Mitochondrien und in den Chloroplasten vorkommen. An ihnen werden Proteine hergestellt, und zwar entsprechend der Basensequenz der DNA, die die Information zur Aminosäuresequenz der Proteine enthält. Hier werden die einzelnen Aminosäuren in genau der Reihenfolge, die das jeweilige Gen vorschreibt, zu einem Kettenmolekül zusammengesetzt. Die Information zur Aminosäuresequenz in der DNA wird durch Boten-RNA (mRNA) vermittelt. Die Umwandlung der in der mRNA gespeicherten Information in eine Abfolge von verknüpften Aminosäuren wird als Translation (lateinisch für Übersetzung) bezeichnet. Die Translation der mRNA am Ribosom ist ein zentraler Bestandteil der Proteinbiosynthese und kommt in allen Lebewesen vor. 2. Bau und Bildung der Ribosomen: Ribosomen sind granuläre Partikel mit einem Durchmesser von etwa 20–25 nm. Sie bestehen zu etwa zwei Dritteln aus RNA (rRNA) und einem Drittel aus ribosomalen Proteinen. Sie setzen sich in allen Organismen aus zwei unterschiedlich großen und funktionell verschiedenen Untereinheiten zusammen. Die Masse der Ribosomen wird durch ihr Sedimentationsverhalten charakterisiert, das in Svedberg-Einheiten (S) angegeben wird. Während der Translation vereinigen sie sich zu einem funktionalen Komplex, wobei die große Untereinheit in der Proteinbiosynthese die Aminosäuren zur Kette verknüpft (Peptidyltransferaseaktivität), und die kleine Untereinheit für die mRNA-Erkennung verantwortlich ist. Beide Untereinheiten bestehen aus Proteinen und rRNA, wobei die Proteine für den Zusammenhalt und die richtige Positionierung zuständig sind, die eigentlichen Reaktionen hingegen werden durch die rRNAs vorgenommen. Beide Untereinheiten werden bei Eukaryonten in den Kerkörperchen innerhalb der Zellkerne gebildet und werden dann durch die Kernporen ins Cytoplasma geleitet. 3. Funktion der Ribosomen 2 Bi-3 Rf Genexpression – Verlauf/Phasen der Proteinbiosynthese (Translation) Datum: Die Funktionsweise des Ribosoms während der Translation kann durch das Dreistellenmodell charakterisiert werden. Demnach besitzt das Ribosom drei tRNABindungsstellen, die A-(Aminoacyl-), P-(Peptidyl-) und E-(Exit-)Stelle. Während des Elongationszyklus oszilliert das Ribosom zwischen zwei Zuständen, dem präund dem post-translationalen Zustand, wobei zwei der drei tRNA-Bindungsstellen mit einer tRNA besetzt sind. Im prätranslationalen Zustand sind die A- und P-Stelle besetzt, wobei die P-Stelle die tRNA mit der Polypeptidkette trägt und die A-Stelle von der neu hinzugekommenen Aminoacyl-tRNA besetzt ist. Im Ribosom wird nun die Polypeptidkette mittels Peptidyltransferase von der P-Stellen-tRNA auf die A-Stellen-tRNA übertragen. Danach wechselt das Ribosom in den posttranslationalen Zustand und wandert um drei Basen auf der mRNA weiter, wodurch die vorherige A-Stellen-tRNA zur P-Stellen-tRNA wird und die nun leere ehemalige P-Stellen-tRNA über die E-Stelle (Exit) aus dem Ribosom geschleust wird. Dabei ist eine Translokase (EF-G) beteiligt. Die beiden Hauptzustände des Ribosoms (prä- und posttranslational) sind durch eine hohe Aktivierungsenergie-Barriere voneinander getrennt. Die zentrale Rolle der beiden Elongationsfaktoren besteht darin, diese Energiebarriere zu erniedrigen und so das Ribosom in den jeweils anderen Zustand zu versetzen. Häufig formieren sich mehrere prokaryotische Ribosomen an demselben mRNA-Molekül perlschnurartig zu einem Polysom. Nachdem ein Peptid im Ribosom verknüpft wurde, durchwandert es einen ribosomalen Tunnel. Dieser besteht größtenteils aus rRNA und tritt aus der großen ribosomalen Untereinheit aus. Er hat eine Länge von ca. 100 Å (10 nm) und einen durchschnittlichen Durchmesser von 15 Å (1,5 nm). An dessen engster Stelle wird der Kanal durch zwei konservierte ribosomale Proteine begrenzt, L4e und L22. Phasen der Translation 1. Initiation (Start) Für den Start der Translation benötigt die Zelle eine tRNA, an die das Startcodon AUG bindet. Die Translation beginnt mit der Erkennung des Start-Codons, an der bei Prokaryoten neben der mRNA und der Initiations-tRNA (transfer-RNA) für Formyl-Methionin (fMet-tRNAfMet) drei Initiationsfaktoren (IF) und die kleine ribosomale Untereinheit (30S-UE) beteiligt sind. Dabei erfolgt die Bindung der mRNA an die kleine ribosomale UE über die Shine-DalgarnoSequenz. Der so entstandene 30S-Initiationskomplex bildet unter Anlagerung der großen ribosomalen UE anschließend den 70S-Initiationskomplex. Bei Eukaryoten erfolgt die Ribosomenbindung und Erkennung des Startcodons anhand der 7-Methylguanosin-Cap; hier 3 Bi-3 Rf Genexpression – Verlauf/Phasen der Proteinbiosynthese (Translation) Datum: ist die Initiations-tRNA auch nicht formyliert. ( vgl. Abb. ) 2. Elongation (Kettenverlängerung) Während der Elongationsphase laufen mehrere Reaktionen (Aminoacyl-tRNA-Bindung, Peptidyltransfer, Translokation) zyklisch hintereinander ab. Die Ribosomen wandern dabei an der mRNA in 5'-3'-Richtung entlang und katalysieren die schrittweise Verknüpfung von Aminosäuren an wachsende Polypeptide. Zunächst bindet eine mit einer Aminosäure beladene tRNA, die Aminoacyl-tRNA (AA-tRNA) aufgrund ihres Anticodons an die Aminoacyl- oder Acceptor-Bindestelle (A-Stelle). Im Anschluss daran kommt es zur Übertragung der bereits bestehenden, als Peptidylrest bezeichneten Peptidkette von der sich in der P-Stelle (Peptidyl-Bindestelle) befindlichen Peptidyl-tRNA (PP-tRNA) auf die Aminoacylgruppe der AA-tRNA. Diese durch die PeptidylTransferase-Aktivität der Ribosomen katalysierte Reaktion führt somit zur Verlängerung des wachsenden Polypeptids um eine Aminosäure. Durch die sich anschließende Translokation des Ribosoms um drei Nucleotide gelangt die deacylierte tRNA in die E-Stelle (Exit) und löst sich vom Ribosom, um erneut mit einer Aminosäure beladen zu werden. Gleichzeitig wird die A-Stelle wieder frei, weil sich die verlängerte PP-tRNA nun in der P-Stelle befindet. Die Geschwindigkeit der Elongation liegt pro Sekunde und Ribosom zwischen zehn und 20 Aminosäuren bei Prokaryoten und bei ca. zwei Aminosäuren bei Eukaryoten, wobei eine Fehlerrate von einer falsch eingebauten Aminosäure pro Tausend erzielt wird, die als ein evolutionärer Kompromiss zwischen Genauigkeit und Energieaufwand angesehen wird. ( vgl. Abb. ) 4 Bi-3 Rf Genexpression – Verlauf/Phasen der Proteinbiosynthese (Translation) Datum: 3. Termination Trifft ein sich an der mRNA entlang bewegendes Ribosom auf eines der drei Stoppcodons, kommt es zunächst zum Stillstand der Translation, da keine passenden tRNA-Moleküle vorhanden sind (Suppression). An ihre Stelle treten so genannte Terminations- oder Release-Faktoren (RFs), die an die A-Stelle binden und die Substratspezifität der PeptidylTransferase dahingehend verändern, dass ein Wassermolekül anstelle einer AA-tRNA aktiviert wird. Durch dessen nucleophilen Angriff auf die Bindung zwischen Peptidkette und tRNA kommt es schließlich zur Freisetzung des synthetisierten Proteins und zur Trennung der mRNA vom Ribosom, das wiederum in seine zwei Untereinheiten dissoziiert. 5