Pflanzen- biotechnologie
Werbung

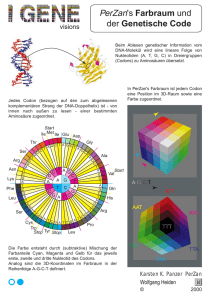
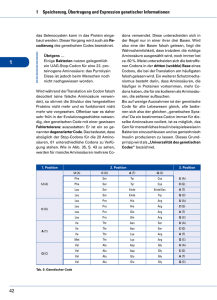
Pflanzenbiotechnologie ten Selektion konnten jetzt durch Pyramidisierung Linien erhalten werden, die wesentlich resistenter gegen die Schadpilze sind. Ist die Genstruktur aufgeklärt, folgt die ungleich schwierigere Aufgabe, dieser Struktur eine Funktion zuzuordnen. Im Fall der Resis- tenzgene sind hier in jüngerer Zeit deutliche Fortschritte erzielt worden. So kennt man heute die Funktionen, die viele der monogen wirkenden Resistenzgene, wie zum Beispiel die Resistenzgene der Kartoffel gegen PVX (Potato virus X), gegen Globodera (Kartoffelnemato- Synthetische Gene zur Expression in transgenen Pflanzen – Matthias Pfeiffer, Uwe Köhler Beispiel Signalpeptide oder Introns – nicht kodierende Sequenzen, die in Pflanzen oft regulatorische Funktionen aufweisen – mit relativ einfachen Mitteln in die Sequenz integriert werden. Drei Basen von innen nach außen gelesen ergeben die von dem Triplett kodierte Aminosäure, zum Beispiel das Triplett CAC die Aminosäure Histidin (His). Grafik: DHGP Der wichtigste Faktor ist jedoch die Anpassung des Gens an die sogenannte „Codon Präferenz“ des Zielorganismus. Der genetische Code ist ein Triplettcode (das heißt, drei Nukleotide auf der DNA kodieren für eine Aminosäure) und ist mit wenigen Ausnahmen in allen Organismen gleich. Es gibt vier verschiedene Nukleotide (G, A, T und C), daraus ergeben sich für einen Triplettcode insgesamt 64 (43) Codonvarianten, wovon drei nicht für Aminosäuren kodieren, sondern als Stoppsignale der Translation dienen. Insgesamt gibt es 20 unterschiedliche Aminosäuren, die von den 61 verbliebenen Tripletts kodiert werden. Das führt dazu, dass für die meisten Aminosäuren zwei, drei, vier oder sogar sechs Codons zur Verfügung stehen. Daher kann ein Gen, das die Information für ein definiertes Protein trägt, in seiner Codonzusammensetzung recht variabel sein. Die chemische Synthese von Nukleinsäuren erlaubt es, vollständige Gene de novo aufzubauen. In einem Ansatz werden eine Reihe von sequenziell überlappenden kurzen Nukleinsäurestücken, so genannte Oligonukleotide, auf chemischem Wege hergestellt und durch enzymatische Reaktionen, meist Ligase- oder Polymerase-Reaktionen, miteinander verknüpft. Auf diese Weise können DNAFragmente beliebiger Sequenz und Länge produziert werden, die nach Transfer in Pflanzenzellen in die entsprechenden Proteine übersetzt werden. Sequenzvergleiche haben gezeigt, dass prokaryotische, eukaryotische und virale Arten oft bestimmte Präferenzen bei der Verwendung von alternativen Tripletts haben. Viele Pflanzen besitzen im allgemeinen eher GC-reiche kodierende Sequenzen, während tierische Gene tendenziell eher AT-reich sind. Bakterien variieren abhängig vom jeweiligen Stamm. So ist zum Beispiel in E. coli K12 das Triplett AAA (und nicht AAG) das bevorzugte Codon für die Aminosäure Lysin. 75 % der für Lysin kodierenden Tripletts sind AAA. In der einkeimblättrigen Pflanze Zea mays (Mais) ist dagegen das Codon AAG das bevorzugte Triplett für Lysin: 72,5 % der Codons sind AAG. Vor der Synthese kann die Gensequenz in silico, also am Computer, an die Bedürfnisse des Wirtsorganismus angepasst werden. Hierbei werden alle Faktoren berücksichtigt, die die Effektivität der Proteinexpression negativ beeinflussen, beispielsweise Sekundärstrukturen, an denen die Proteinsynthese vorzeitig abbrechen könnte. Andererseits können auch wichtige positiv wirkende Elemente, zum Die Codonpräferenz spielt bei der Expression bakterieller oder tierischer Proteine in Pflanzen eine große Rolle. Ist das Fremdgen aus Codons aufgebaut, die in der Pflanze weniger bevorzugt sind, wird das Gen schlecht in Protein übersetzt. Die Pflanzenzellen besitzen nämlich nur einen begrenzten Vorrat an transferRNAs (tRNAs), die für den Transport einer bestimmten Aminosäure bei der Protein- 20 mensch+umwelt spezial 17. Ausgabe 2004/2005 den) oder gegen die Kraut- und Knollenfäule, im Stoffwechsel der Zelle haben: Sie übertragen mit Hilfe bestimmter Proteine Signale zwischen Zellkompartimenten, zum Beispiel aus dem Zellkern ins Zytoplasma, so dass dort Abwehrreaktionen aktiviert werden. Resistenzgene sind meist nicht zufällig im Pflanzengenom verteilt, sondern treten gehäuft in hot spots auf. Damit wird eine Suchstrategie möglich, mit der in der Nachbarschaft von bereits bekannten Resistenzgenen nach Erbanlagen für neue Resistenzeigenschaften ge- fahndet werden kann. Allerdings finden sich dabei oft auch so genannte Resistenzgen-Analoge, die zwar ähnliche DNA-Sequenzen besitzen, letztlich aber nicht für aktive Proteine kodieren. Daher muß die resistenzvermittelnde Wirkung solcher Kandidatengene durch Genüber- synthese verantwortlich sind. Wird nun ein von der Pflanze selten verwendetes Codon für die Translation des Fremdgens sehr oft benötigt, kommt es schnell zur Ausdünnung der entsprechenden tRNAMoleküle. In der Folge pausiert die Translation des Fremdgens oder es werden vermehrt falsche Aminosäuren in die Polypeptidkette eingebaut. Es ist daher sinnvoll – oder gegebenenfalls sogar notwendig –, die Sequenz eines Fremdgens an die Gegebenheiten des Wirtsorganismus anzupassen. Hierzu werden Codons mit einer geringen Präferenz durch Codons mit hoher Präferenz ersetzt, ohne dass sich dadurch die Aminosäuresequenz des resultierenden Proteins ändert. Diese Methode wurde bereits vielfach bei zweikeimblättrigen Pflanzen eingesetzt (zum Beispiel bei der Expression von bakteriellem Endotoxin in Kartoffeln). Einkeimblättrige Pflanzen sind im Vergleich zu den zweikeimblättrigen noch GCreicher. Daher ist zum Beispiel die Expression bakterieller oder tierischer Gene in Reis bisher nur selten geglückt. Die Verwendung Codon-angepasster synthetischer Gene hat nun aber auch in dieser wichtigen Nutzpflanze zum Erfolg geführt. Die Herstellung synthetischer Gene a) Chemisch synthetisierte Oligonukleotide mit zueinander komplementären Enden werden gemischt. b) Unter geeigneten Bedingungen hybridisieren die passenden Enden der Oligonukleotide zu kurzen Doppelsträngen. c) und d) Die einzelsträngigen Zwischenräume werden durch das Enzym DNA-Polymerase mit dem jeweiligen Gegenstrang als Matrize aufgefüllt. e) Die synthetische DNA ist nun doppelsträngig, allerdings sind die Rückgrate beider Stränge noch nicht kovalent geschlossen. f) Das Enzym Ligase verknüpft die freien Enden im Rückgrat durch kovalente Bindung. g) Das synthetische Gen ist nun vollständig und kann zur Expression des von ihm kodierten Proteins weiterverwendet werden. Grafik: Medigenomix/H. Guldner Dr. Matthias Pfeiffer, Biologe und Betriebswirt, und Dr. Uwe Köhler, Biologe, gehören der Martinsrieder Firma Medigenomix an. 21