Whitepaper lesen - GIT
Werbung

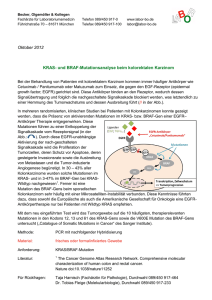
Pyrosequencing – eine neue und vielseitige Technologie Flexible DNA Sequenzierung in Kombination mit präziser Quantifizierung Nicht selten zeigen Medikamente bei verschiedenen Patienten unterschiedliche Reaktionen. So kann eine bei einer Person gut wirksame Behandlung bei einer anderen Person gar keinen Effekt haben oder sogar zu unerwünschten Nebenwirkungen führen. Dies liegt hauptsächlich an der Variabilität des menschlichen Genoms. Das Wissen um die individuellen genetischen Unterschiede soll Ärzten daher bei der Auswahl der geeigneten und auf den Patienten zugeschnittenen Therapie helfen. Dies verspricht die personalisierte Medizin. Die Grundlage der personalisierten Medizin ist die Pharmakogenomik – die Erforschung der physiologischen Reaktion einer Person auf Medikamente je nach genetischer Veranlagung. Sie ermöglicht es Medizinern, die Therapien bezüglich Effizienz und Effektivität individuell auf den Patienten anzupassen. Vor allem im onkologischen Bereich hat das Interesse an personalisierter Medizin jüngst stark zugenommen. Ein deutlicher Fokus liegt hierbei bei der Bestimmung von genetischen Parametern, anhand derer Patientenpopulationen unterteilt werden kön- nen, um spezifische Behandlungsmethoden anzubieten. Eine der vielversprechendsten Technologien auf diesem Gebiet ist Pyrosequencing. Pyrosequencing – eine schnelle, zuverlässige und sensitive Methode für die Krebsforschung Pyrosequencing beruht auf dem Prinzip der Sequenzierung durch Synthese (siehe Abbildung 1) und liefert Sequenzinformationen in Echtzeit. Im Gegensatz zu anderen Routinetechnologien der Molekulardiagnostik (wie z. B. PCR) liest Pyrosequencing die DNA Sequenz. Anders als PCR, die nur bekannte Sequenzen detektiert, erlaubt Pyrosequencing die Erfassung sämtlicher, d.h. bekannter und unbekannter Mutationen im betrachteten Genbereich. Während Mutations- und Methylierungsanalysen nach der traditionellen Sanger-Sequenzierungsmethode wenig sensitiv sind und spezielle quantitative Sanger-Sequenzierungen zeit- und arbeitsintensive Klonierungen von PCR Produkten erfordern, bietet Pyrosequencing einfaches und flexibles Sequenzieren (ohne notwendige Klonierungen) in Kombination mit hochgradig präziser Quantifizierung in kurzer Zeit. Pyrosequencing ist in der Krebsforschung sehr gut einsetzbar, es ermöglicht beispielsweise die Detektion und Quantifizierung mehrerer und/oder komplexer Mutationen in nur einem einzigen Test. Im Gegensatz zu Real-Time-PCR Methoden können dabei auch unbekannte Mutationen erkannt werden. Die Technologie ist darüber hinaus ideal für die Quantifizierung von Methylierungen an mehreren aufeinander folgenden CpG-Dinukleotiden. Des Weiteren können im gleichen Assay Single Nucleotide Polymorphismen (SNPs) bestimmt werden. Pyrosequencing ist ebenfalls wichtig für die Quantifizierung von Loss of Heterozygosity (LOH), die z. B. bei der Identifizierung von Tumorsuppressorgenen hilft. Große Aufmerksamkeit seitens der Krebsforschung/-therapie erhält momentan ein spezieller Anwendungsbereich rund um das KRAS Gen. KRAS und Krebs KRAS ist ein Onkogen, das eine entscheidende Rolle bei der Entstehung von Tumoren spielt. Mutationen innerhalb der RAS-Familie (diese beinhaltet HRAS, NRAS und KRAS) sind sehr häufig und Schritt 1: Nach Anlagerung eines Sequenzierprimers an das zu sequenzierende einzelsträngige DNA Fragment werden die Enzyme DNA Polymerase, ATP Sulfurylase, Luziferase und Apyrase sowie die Substrate Adenosin 5’ Phosphosulfat (APS) und Luziferin zugegeben Schritt 2: Das erste Desoxyribonukleosidtriphosphat (dNTP) wird automatisch in den Reaktionsansatz dispensiert. Die DNA Polymerase baut das dNTP in den DNA Strang ein, wenn es komplementär zur Base des Templates ist. Beim Einbau wird Pyrophosphat (PPi) in äquimolarer Menge zum eingefügten Nukleotid frei. Schritt 3: ATP Sulfurylase katalysiert die Umwandlung von PPi und APS in ATP. Dieses ATP wird von der Luziferase zur Umwandlung von Luziferin in Oxyluciferin benötigt. Die dabei frei werdende Lichtmenge ist proportional zur Menge an ATP. Das Licht aus der Luziferase-katalysierten Reaktion wird mit einem CCD-Chip quantifiziert, in einer Software verarbeitet und als sog. Pyrogramm grafisch dargestellt. Schritt 4: Apyrase baut kontinuierlich nicht eingebaute Nukleotide und ATP ab. Wenn der Abbau abgeschlossen ist, wird das nächste Nukleotid hinzugefügt und der Zyklus beginnt wieder bei Schritt 2. Schritt 5: Die Zugabe der dNTPs folgt entsprechend der vorgegebenen Sequenz nacheinander. Statt Desoxyadenosintriphosphat (dATP) wird dabei Desoxyadenosin alfa-thio triphosphat (dATP∙S) eingesetzt, da es von der DNA Polymerase verwendet, aber nicht von der Luziferase erkannt wird. Während des Pyrosequencing Laufes wird der zum Template komplementäre DNA Strang synthetisiert, und anschließend die Sequenz durch Auswertung der Peakhöhen im Pyrogramm bestimmt. Abb. 1: Das Prinzip des Pyrosequencing Pyrosequencing folgt einer Reihe verschiedener Schritte für jedes sequenzierte Nukleotid. Die Technologie basiert auf dem Prinzip der Sequenzierung durch Synthese und liefert quantitative Daten im Sequenzkontext. führen zu einem permanent aktiven RASProtein, welches in bis zu 30 % aller menschlichen Tumore auftritt. Es wird vermutet, dass solch eine onkogenetische Aktivierung in vielerlei Aspekte der Krebsentstehung und -progression involviert ist, wie z. B. entartetes Zellwachstum, Proliferation, Differenzierung sowie auch gesteigerte Invasion und Metastasierung (siehe Abb. 2). KRAS ist in ungefähr 35–45 % aller metastasierenden kolorektalen Krebserkrankungen (CRC), 15–50 % aller Lungenkrebse und 72–90 % aller Pankreaskarzinome mutiert. Die Mutationen wurden in verschiedenen Codons, wie zum Beispiel Codon 12,13 und 61 gefunden. Studien haben gezeigt, dass durch die Testung auf eine KRAS-Mutation besser vorausgesagt werden kann, welche CRCPatienten von einer Behandlung mit monoklonalen Antikörpern gegen EGFR (Epidermal Growth Factor Receptor) profitieren, z. B. Vectibix (Panitumumab) von Amgen und Erbitux (Cetuximab) von Imclome\ Bristol-Myers Squibb\ Merck. Eine solche Studie der belgischen Universität Leuven gibt Hinweise darauf, dass der KRAS-Mutationsstatus ein prognostischer Biomarker zur Beurteilung des Erfolgs von EGFR Therapien ist. In dieser Studie hatten etwa 40 % aller CRC-Patienten Mutationen im KRAS Gen. Die Untersuchungsdaten deuten darauf hin, dass diese Patienten nicht von EGFR Antikörpern profitieren und in manchen Fällen sogar negative Nebenwirkungen entwickeln, während Patienten ohne KRAS-Mutation sehr wahrscheinlich auf diese Behandlung ansprechen. In Anbetracht dieser Studien änderten die Europäischen Zulassungsbehörden die Indikation für Vectibix (Panitumumab), so dass nur Patienten, in deren Tumor die unmutierte KRAS-Genvariante vorliegt, das Medikament erhalten. Das U.S. National Comprehensive Cancer Network (NCCN) gab im November 2008 neue CRC-Leitlinien heraus, die empfehlen, lediglich Patienten mit Tumoren ohne mutiertes KRAS-Gen mit EGFR Medikamenten zu behandeln. Diese Organisation aus 21 Krebszentren schlug ebenfalls vor, dass Onkologen grundsätzlich den KRAS-Genstatus von CRC-Patienten vor jeglicher Behandlung feststellen sollten. Ein molekulares Testverfahren für den Krebs-Biomarker KRAS Abb. 2: EGFR Signalkette und KRAS KRAS ist der Haupteffektor der EGFR Signalkette und stellt einen wesentlichen Schalter in diesem Signalweg dar. Bestimmte KRAS-Mutationen können den Signalweg dauerhaft aktivieren, so dass er insensitiv gegen vorgeschaltete Inhibitoren, wie z. B. monoklonale Antikörper gegen EGFR, wird. Qiagen hat kürzlich den ersten molekularen Test basierend auf Pyrosequencing für KRAS eingeführt. Das PyroMark KRAS Kit ist CE-markiert für in-vitro diagnostische Zwecke und wird momen- tan bei metastasierten kolorektalen Krebserkrankungen zur Bestimmung des KRAS- Genstatus vor der Zweitlinientherapie mit Erbitux oder Vectibix angewendet. Das Kit ermöglicht es auf dem handlichen PyroMark Q24 MDx Instrument Mutationen in den Codons 12, 13 und 61 zu detektieren und quantifizieren. Die in Codon 12 und 13 oder 61 enthaltenen Genabschnitte werden durch PCR vervielfältigt und dann über den definierten Bereich sequenziert, um die relevanten Mutationen zu identifizieren und quantifizieren. Das Design des Testverfahrens erlaubt es sogar, zusätzliche seltenere Mutationen in den Codons zu erkennen. Der umgebende Sequenzkontext dient dabei als integrierte Qualitätskontrolle für die Ergebnisse. Wissenschaftlern gelingt mit Pyrosequencing eine schnellere, präzisere Entdeckung von Mutationen als mit herkömmlichen PCR-Methoden. Eine vielversprechende Zukunft für Pyrosequencing in der Krebsforschung Jedes Jahr werden mehrere Milliarden Dollar für die Verschreibung von Medi- kamenten und andere Therapien ausgegeben, die sich schließlich bei bestimmten Personen als ineffektiv oder sogar gefährlich herausstellen. Schätzungen zufolge gehen alleine in Deutschland etwa 58.000 Todesfälle jährlich auf die Verschreibung ungeeigneter Medikamente zurück [1]. In den Vereinigten Staaten belegen Studien, dass die am häufigsten verschriebenen Medikamente bei weniger als 60 % der Patienten wirken [2]. Ohne deutliche Verbesserung der Möglichkeit, die richtige Arznei den geeigneten Patienten zuordnen zu können, werden sich diese gegensätzlichen Effekte noch intensivieren und in einer alternden Gesellschaft häufiger werden. DieKonsequenz daraus sind steigende Behandlungskosten. Die Forschung rund um das KRASGen und seinen Einfluss auf die Krebsentwicklung sowie um die personalisierte Krebsbehandlung selbst steckt noch in den Kinderschuhen. Aber mehr und mehr Wissenschaftler nutzen diese Technologie und beginnen, die heutigen Grenzen medizinischer Therapien zu überwinden und die Effektivität und Sicherheit von Behandlungen zu maximieren. Quellen: [1] Institute of Clinical Pharmacology, University of Hannover, 7.3.2003 [2] Harvard Business Review, Oktober 2007 Kontakt Dr. Janina Schaper Qiagen GmbH Global Product Manager Hilden, Deutschland Tel.: 02103/29-16375 [email protected] www.pyrosequencing.com