entwicklung eines multiplex Pcr
Werbung
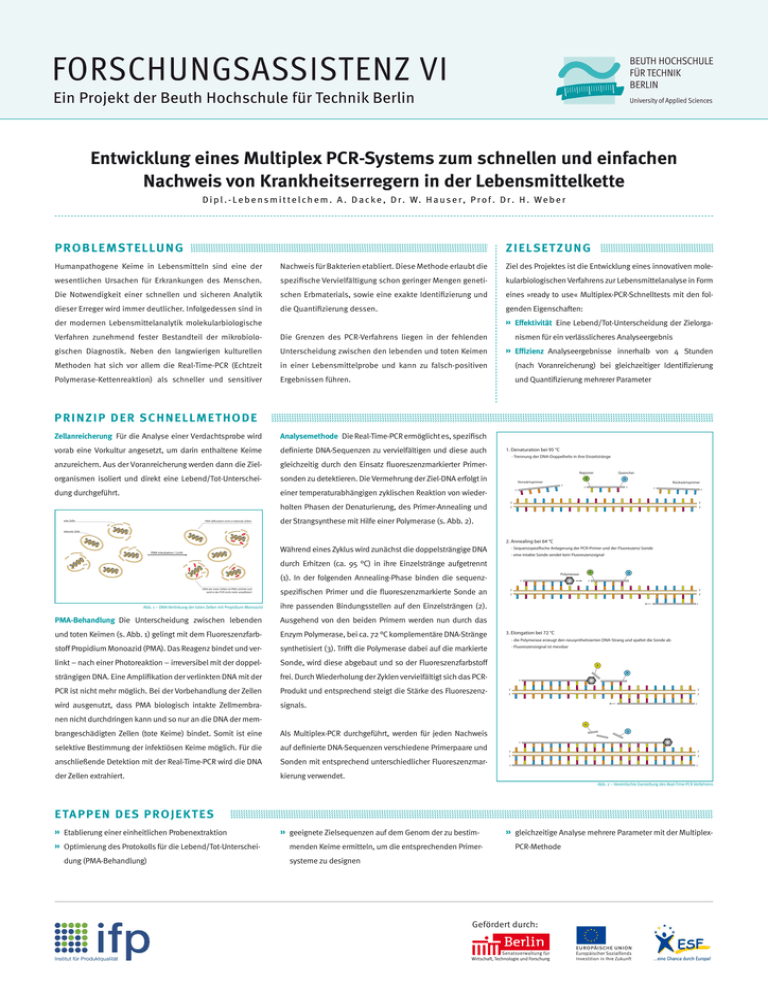
entwicklung eines multiplex Pcr-Systems zum schnellen und einfachen nachweis von Krankheitserregern in der lebensmittelkette D i p l . - L e b e n s m i t t e l c h e m . A . D a c k e , D r . W. H a u s e r , P r o f . D r . H . W e b e r ProBlemStellung zi el Set zung Humanpathogene Keime in Lebensmitteln sind eine der Nachweis für Bakterien etabliert. Diese Methode erlaubt die Ziel des Projektes ist die Entwicklung eines innovativen mole- wesentlichen Ursachen für Erkrankungen des Menschen. spezifische Vervielfältigung schon geringer Mengen geneti- kularbiologischen Verfahrens zur Lebensmittelanalyse in Form Die Notwendigkeit einer schnellen und sicheren Analytik schen Erbmaterials, sowie eine exakte Identifizierung und eines »ready to use« Multiplex-PCR-Schnelltests mit den fol- dieser Erreger wird immer deutlicher. Infolgedessen sind in die Quantifizierung dessen. genden Eigenschaften: » effektivität Eine Lebend/Tot-Unterscheidung der Zielorga- der modernen Lebensmittelanalytik molekularbiologische Verfahren zunehmend fester Bestandteil der mikrobiolo- Die Grenzen des PCR-Verfahrens liegen in der fehlenden gischen Diagnostik. Neben den langwierigen kulturellen Unterscheidung zwischen den lebenden und toten Keimen » effizienz Analyseergebnisse innerhalb von 4 Stunden Methoden hat sich vor allem die Real-Time-PCR (Echtzeit in einer Lebensmittelprobe und kann zu falsch-positiven (nach Voranreicherung) bei gleichzeitiger Identifizierung Polymerase-Kettenreaktion) als schneller und sensitiver Ergebnissen führen. und Quantifizierung mehrerer Parameter nismen für ein verlässlicheres Analyseergebnis Pri nziP d er Sc hne l l me thode zellanreicherung Für die Analyse einer Verdachtsprobe wird Analysemethode Die Real-Time-PCR ermöglicht es, spezifisch vorab eine Vorkultur angesetzt, um darin enthaltene Keime definierte DNA-Sequenzen zu vervielfältigen und diese auch anzureichern. Aus der Voranreicherung werden dann die Ziel- gleichzeitig durch den Einsatz fluoreszenzmarkierter Primer- organismen isoliert und direkt eine Lebend/Tot-Unterschei- 1. Denaturation bei 95 °C - Trennung der DNA-Doppelhelix in ihre Einzelstränge Reporter sonden zu detektieren. Die Vermehrung der Ziel-DNA erfolgt in R Vorwärtsprimer 3´ einer temperaturabhängigen zyklischen Reaktion von wieder- dung durchgeführt. holten Phasen der Denaturierung, des Primer-Annealing und tote Zelle PMA diffundiert nicht in lebende Zellen Quencher Q 5´ Rückwärtsprimer 3´ 5´ 5´ 3´ 3´ 5´ 5´ 3´ der Strangsynthese mit Hilfe einer Polymerase (s. Abb. 2). lebende Zelle 2. Annealing bei 64 °C Während eines Zyklus wird zunächst die doppelsträngige DNA PMA-Inkubation / Licht - Sequenzspezifische Anlagerung der PCR-Primer und der Fluoreszenz-Sonde - eine intakte Sonde sendet kein Fluoreszenzsignal durch Erhitzen (ca. 95 °C) in ihre Einzelstränge aufgetrennt (1). In der folgenden Annealing-Phase binden die sequenzDNA der toten Zellen ist PMA-verlinkt und wird in der PCR nicht mehr amplifiziert spezifischen Primer und die fluoreszenzmarkierte Sonde an Abb. 1 – DNA-Verlinkung der toten Zellen mit Propidium-Monoazid ihre passenden Bindungsstellen auf den Einzelsträngen (2). PmA-Behandlung Die Unterscheidung zwischen lebenden Ausgehend von den beiden Primern werden nun durch das und toten Keimen (s. Abb. 1) gelingt mit dem Fluoreszenzfarb- Enzym Polymerase, bei ca. 72 °C komplementäre DNA-Stränge stoff Propidium Monoazid (PMA). Das Reagenz bindet und ver- synthetisiert (3). Trifft die Polymerase dabei auf die markierte linkt – nach einer Photoreaktion – irreversibel mit der doppel- Sonde, wird diese abgebaut und so der Fluoreszenzfarbstoff strängigen DNA. Eine Amplifikation der verlinkten DNA mit der frei. Durch Wiederholung der Zyklen vervielfältigt sich das PCR- PCR ist nicht mehr möglich. Bei der Vorbehandlung der Zellen Produkt und entsprechend steigt die Stärke des Fluoreszenz- wird ausgenutzt, dass PMA biologisch intakte Zellmembra- signals. 5´ selektive Bestimmung der infektiösen Keime möglich. Für die Q 5´ 3´ 5´ 5´ 3´ 5´ 3. Elongation bei 72 °C - die Polymerase erzeugt den neusynthetisierten DNA-Strang und spaltet die Sonde ab - Fluoreszenzsignal ist messbar R Q 5´ 3´ 5´ 5´ 3´ 5´ nen nicht durchdringen kann und so nur an die DNA der membrangeschädigten Zellen (tote Keime) bindet. Somit ist eine R Polymerase R Q Als Multiplex-PCR durchgeführt, werden für jeden Nachweis auf definierte DNA-Sequenzen verschiedene Primerpaare und anschließende Detektion mit der Real-Time-PCR wird die DNA Sonden mit entsprechend unterschiedlicher Fluoreszenzmar- der Zellen extrahiert. kierung verwendet. 5´ 3´ 5´ 5´ 3´ 3´ 5´ Abb. 2 – Vereinfachte Darstellung des Real-Time-PCR Verfahrens e tA PPen d eS Proje KteS » Etablierung einer einheitlichen Probenextraktion » Optimierung des Protokolls für die Lebend/Tot-Unterscheidung (PMA-Behandlung) » geeignete Zielsequenzen auf dem Genom der zu bestimmenden Keime ermitteln, um die entsprechenden Primer- » gleichzeitige Analyse mehrere Parameter mit der MultiplexPCR-Methode systeme zu designen Gefördert durch:


