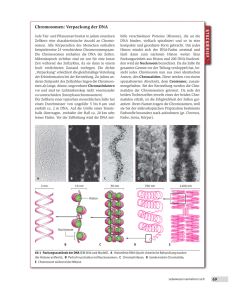
Allgemeine Molekulare Genetik 1
Werbung

Allgemeine Molekulare Genetik 1 (LvNr. 300132) SS10 Skriptum GNU General Public License 1 - Modellorganismen in der Genetik Genetische Studien werden in der Regel an Modellorganismen durchgeführt. Ein perfektes Modell gibt es auch hier nicht, alle haben Vor- und Nachteile. Vorteile Nachteile Fadenwurm ( Caenorhabditis elegans ) • • • • • • Kurze Generationszeit Kleines Genom Einfach zu halten (Agar) 100te Nachkommen Gute Genetik Tranformierbar • Wenig Phänotypen, biochem. Tests erforderlich • Genetisches Material schwer zu präparieren, da wenig Gewebe Acker-Schmalwand, Schotenkresse ( Arabidopsis thaliana ) • • • • Kleines Genom Kurze Generationszeit 100te Nachkommen Transformierbar • Dicotyle Pflanze, ökonom. Interesse allerdings bei Monocotylen • Generationszeit • Keine gezielten Knockouts Zebrafisch ( Danio rerio ) • Kleines Genom • Kurze Generationszeit • 100te Nachkommen Fruchtfliege ( Drosophila melanogaster ) • • • • • • Chromosomenkarte Polytän-Chromosomen 100te Nachkommen Leichte Haltung Transformierbar Klare Phänotypen • Schlechte Biochemie • Wenig homogenes Material Bäckerhefe ( Saccharomyces cerevisiae ) • • • • • Kleines Genom Leichte Haltung Gute Biochemie Transformierbar Homologe Rekombination möglich • Keine Entwicklungsvarianten (Analyse von Morphogenen nicht möglich) • Unizellulär Maus ( Mus musculus ) • Stellvertretend für Säuger • Transgenität • • • • • Schwer zu halten (Pflegeaufwand) Infektionsanfällig Keine gezielten Knockouts • Lange Generationszeit • Teuer • Haltungsaufwand 2 – Genetische Nomenklatur Gene und deren Mutanten werden häufig irreführend und kontraintuitiv benannt. Wildtypen (wildtypes) beschreiben funktionierende Organismen, Enzyme, etc., welche jedoch den Mutanten entsprechend bezeichnet werden. Zum Beispiel fehlt einer Mutante von Drosophila mit weißen -1- Augen der rote Farbstoff, daher wird das Gen, das für diesen kodiert, als „WHITE“-Gen bezeichnet. Neben phänotypischen Eigenschaften verwendet man auch biochemische Abweichungen (Stoffwechsel) oder immunologische Besonderheiten (Resistenzen). Weiters findet man unterschiedliche Schreibweisen mittels Groß- und Kleinbuchstaben sowie Plusund Minuszeichen, um Genotypen zu beschreiben. Dabei muss man teilweise Redundanzen in der Schreibweise beachten (bedingt durch von Anfang an nicht festgelegte Regeln, darum haben verschiedene Autoren verschiedene Schreibweisen verwendet), am Beispiel des Merkmals G: G G+ g g- ist äquivalent zu ist äquivalent zu ist äquivalent zu ist eindeutig. g+, zu G, zu G-, In genealogischen Stammbäumen verwendet man symbolische Darstellungsweisen der Phänotypen von Männchen und Weibchen, sowie „krank“ (besser wäre „phänotypisch betroffen“), „gesund“ (nicht betroffen) und Überträger. Betroffen Nicht betroffen Überträger Männchen Weibchen 3 – Mendel revisited Die Mendelschen Regeln gehören zum Standardrepertoire der klassischen Genetik und beschreiben die Vererbung von Merkmalen. Sie sind nur auf diploide Organismen mit haploiden Gameten anwendbar, es lassen sich ebenfalls Regeln für Organismen mit höherem Ploidiegrad ableiten. Die Schlüssel zu Mendels Erfolg sind damals wie heute Voraussetzung zum Erkenntnisgewinn des Genetikers: 1. 2. 3. 4. 5. 6. Wahl des Versuchsobjekts Nutzung reinerbiger Stämme Schutz vor Fremdbesamung Beschränkung auf wenige Merkmale Groß angelegte Versuchsreihen Statistische Auswertung Uniformitätsregel (Reziprozitätsregel) Werden 2 bezüglich eines Merkmals homozygote Individuen der Parentalgeneration (P) gekreuzt, so erhält man eine uniforme Filialgeneration (F 1), deren Individuen bezogen auf das untersuchte Merkmal untereinander phänotypisch gleich, aber heterozygot sind. Dies gilt für den Phänotyp (äußeres Erscheinungsbild) wie den Genotyp (Erbausstattung), welcher bei allen heterozygot (mischerbig) ist. Dabei ist es egal, welches der beiden Allele von der Mutter und welches vom Vater vererbt wird (reziproke Kreuzung). Die Ausprägung der Erbanlagen kann auf zweierlei Weise erfolgen, nämlich dominant-rezessiv oder intermediär (siehe auch Erbgänge). Dominante Merkmale überdecken dabei rezessive (Bsp. Blütenfarbe rot/weiß), intermediäre treten mit einer Wahrscheinlichkeit von 50% oder in einer Mischform in Erscheinung. -2- Zu einer Ausnahme kann es u.a. kommen, wenn ein Merkmal auf einem Geschlechtschromosom (Gonosom) liegt. Spaltungsregel (Segregationsregel) Kreuzt man 2 heterozygote Organismen der F1-Generation, so ergibt sich in der Filialgeneration 2 (F2) bei dominant-rezessivem Erbgang eine Aufspaltung in den Genotypen zu 1:2:1 und in den Phänotypen zu 3:1 in Bezug auf ein Merkmal (monohybrid), was aus der genetischem Ausstattung der Gameten resultiert (Gamet kann dominantes oder rezessives Allel tragen). Bei dihybrider Vererbung ergeben sich entsprechend mehr Gametenvarianten, nämlich 4 auf Grund der möglichen Allelkombinationen. Hier tritt eine Aufspaltung von 9:3:3:1 auf. Unabhängigkeitsregel (Neukombinationsregel) Die Unabhängigkeitsregel beschreibt Vererbung von Merkmalen bei der Kreuzung reinerbiger Individuen im dihybriden Erbgang. Die Allele werden unabhängig voneinander weiter gegeben. Kreuzt man Individuen der F1-Generation unter sich, so treten neue, reinerbige Individuen auf. Diese Regel gilt allerdings nur dann, wenn sich die für die Merkmale verantwortlichen Gene auf verschiedenen Chromosomen befinden oder wenn sie auf dem gleichen Chromosom so weit voneinander entfernt liegen, dass sie während der Meiose durch Crossing over regelmäßig getrennt voneinander vererbt werden (polygene Erbgänge). Befinden sich Gene auf dem gleichen Chromosom nahe beieinander, so werden sie in Kopplungsgruppen vererbt. Die 9:3:3:1 Aufspaltung ist jedoch nur zutreffend, wenn beide Individuen in Bezug auf die Allele homozygot sind. Ist dies nicht der Fall, erhält man Abweichungen von diesem Segregationsmuster (zB. 6:4:2:2:2:1). In jedem Fall treten genau 2 neue homozygote Varianten auf. Dominanz / Autosomal Dominante Vererbung Merkmale werden in jeder Generation exprimiert. Dominante Merkmale sind meistens die Wildtypen, Mutanten-Allele sind entsprechend rezessiv. Liegen in einer Pflanze beispielsweise ein Allel für einen weißen Farbstoff und ein Allel für einen roten Farbstoff vor, so wird die Expression eines Allels bevorzugt – hier sei es der rote Farbstoff. Rezessivität Merkmale können Generationen überspringen und treten phänotypisch nur bei Reinerbigkeit der Allele in Erscheinung. Die Eigenschaft Rezessivität verbindet der Autor mit der Vorstellung von Genen, welche im Vergleich zu dominanten Genen bzw. Allelen eine verringerte, ineffektive bis defekte oder weniger optimale Wirkung auf den Organismus haben. Da dominante Merkmale rezessive überdecken, praktisch eine Wirkung oder eine stärkere, effektivere Wirkung durch exprimierte Faktoren bzw. indirekt positiv regulierte Expression zeigen (vgl. Kompetitive Affinität von Transkriptionsfaktoren), liegt eine Analogie von rezessiv zu fehlend oder vermindert nahe. Ein Beispiel: Das Allel für Blutgruppenfaktor 0 verhält sich rezessiv gegenüber den Allelen für die Blutgruppenfaktoren A und B. Codominanz / Intermediäre Vererbung Codominante Vererbung tritt wie auch Dominanz und Rezessivität bei heterozygoten Organismen auf und beschreibt eine gemischte Merkmalsausprägung, somit liegt der Phänotyp in der Mitte der beiden homozygoten (intermediär). -3- Der Autor stellt sich das Phänomen so vor, dass sich im Laufe der Evolution zwei gleichwertige Pathways entwickelt haben, die parallel exprimiert werden können, ohne sich gegenseitig zu beeinflussen. Ein Beispiel für codominante Merkmale: Blutgruppen A und B – Trägt ein Individuum je ein Allel für eines der beiden Glykoproteine, werden beide exprimiert, man hat Blutgruppe AB. Dihybrider Erbgang / Polyhybride Erbgänge Betrachtet man mehrere Allele der Eltern und möchte die möglichen Geno- und Phänotypen des Offsprings auflisten, empfehlen sich Kreuzungstabellen. Bei dihybrider Vererbung mit homozygoten Eltern ergibt sich eine Aufspaltung im Phänotyp zu 9:3:3:1, die Anzahl möglicher haploiden Allelkombinationen in den Gameten beträgt 2 n. Kreuzungstabellen eines dihybriden und polyhybriden Erbgangs: AB Ab aB ab AB AABB AABb AaBB AaBb Ab AABb AAbb AbBb Aabb aB AaBB AaBb aaBB aaBb ab AaBb Aabb aaBb aabb Tabelle 1: Dihybrider Erbgang mit homozygoten Eltern (AAbb x aaBB) ABC ABC ABc AbC Abc aBC aBc abC abc AABBCC ABc AbC Abc aBC aBc abC abc Tabelle 2: Polyhybrider Erbgang mit homozygoten Eltern (AAbbCC x aaBBcc) zum selbst ausfüllen 4 – Ausnahmen Mendel'scher Genetik 4.1 Cytoplasmatische Vererbung (Mitochondrium / Plastid) 4.2 Maternaler Effekt 4.3 X-Inactivation / X-Linked 4.4 Homozygote Letalität 4.5 Imprinting 4.6 Su(var), E(var), pev -4- 4.1 Cytoplasmatische Vererbung – Endosymbiontenfortpflanzung Mitochondrien und Plastiden besitzen ein zirkuläres, reduziertes Genom, welches sie unabhängig vom nukleären Genom vererben. Die Vermehrung der Endosymbionten erfolgt unabhängig vom Zellzyklus, sie regeln quasi selbst ihre Population. Durch das permanente Nährstoffangebot ist ihre Genexpression dem Zellmetabolismus angepasst. Bei mitotischer Teilung der Wirtszellen erfolgt zufällige Aufteilung auf die Tochterzellen. Eine Zelle enthält aber stets maternale Mitochondrien, da diejenigen des Spermiums nicht in die Oocyte eindringen. Durch die asexuelle Vermehrung sammeln sich negative traits durch Mutationen an. Da man die Mutationsrate bestimmt durch die Fehlerfrequenz der Polymerasen in Abhängigkeit der Reparaturmechanismen kennt, kann man bei Vergleich zweier Organismen, die sich zB. an 23 Positionen unterscheiden, den Generationsunterschied für Stammbaumanalysen schätzen. So hält man die Abspaltung des Homo sapiens vor 100.000 – 150.000 Jahren als wahrscheinlich, da man auf diesen Zeitpunkt die mitochondriale „Eva“ datiert. Abb. 4.1 - Mutationsloci auf der menschlichen mtDNA und deren Krankheitsbilder (Jantsch Folienset 1/SS09). Befinden sich Mutationen im Mitochondrium, hat das z.T. erhebliche Auswirkungen auf den Organismus in Form von Metabolismusstörungen oder neurodegenerativen Effekten sowie Myopathien (siehe Bild 4.1). Bei Pflanzen verhält es sich anders, da Keimbahnzellen spontan aus Somazellen hervorgehen. Dabei kann es vorkommen, dass Gynander bezüglich der Plastiden vorliegen, sodass manche Nachkommen derselben Pflanze Plastiden besitzen, andere widerum nicht. In Neurospora crassa kann man die mitotischen Teilungsprodukte genau verfolgen, da die Gameten in den Octaden linear angeordnet sind. Mitotische Marker zeigen im Gegensatz zu nukleären Markern ein 1:0 oder 0:1 Segregationsmuster. In poky-Mutanten, eine mitochondriale Deletion, die die zu einer geringeren Anzahl an Ribosomen auf Grund verminderter rRNA-Synthese führt, kann man dieses Phänomen beobachten. -5- 4.2 Maternaler Effekt Der gravierende Unterschied zwischen dem maternalen Effekt und cytoplasmatischer Vererbung ist, dass bei cytoplasmatischer Vererbung vererbbare Information in Form von mtDNA der Plastiden vererbt wird, beim maternalen Effekt nur mRNA oder Proteine an ein Individuum weitergegeben werden, die nicht vererbbar sind. Der maternale Effekt überdeckt den Genotyp eines Individuums. Im Unterschied zur mitchondralen Vererbung, erbt ein Organismus die löslichen Faktoren des Cytoplasmas der Oocyte (versch. RNA-Typen, Enzyme, Peptide, … ), sodass zwischen nukleärem und plasmatischem Genotyp unterschieden werden kann bzw. muss, da Faktoren enthalten sein können, für die gar kein Allel vorliegt. Frühe Phänotypen werden also durch den maternalen Effekt festgelegt. Das Phänomen kann man an verschiedenen Organismen gut beobachten. Wanderschnecken (Lymnaea peregra) geben einen maternalen Faktor weiter, das Dextral-Protein, welches die Schlagrichtung von Cilien in der Morphogenese bestimmt, sodass die rechtgängige Drehrichtung des Gehäuses durch gerichtete Spülung von Wachstumsfaktoren zu Stande kommt. Das Gen ist dominant, allerdings kann auch bei homozygot rezessiven Allelen der Zygote die Schnecke dextral werden, da von der Mutter das Dextral-Protein in die Oocyte eingelagert wird. Nur wenn die Mutter homozygot rezessiv ist, kann eine sinistrale Schnecke entstehen. Die Diagnose situs inversus, eine seltene, aber nicht krankhafte anatomische Besonderheit (eine Heterotaxie) beim Menschen ist auch auf einen Ciliendefekt zurückzuführen, dabei liegen die inneren Organe spiegelverkehrt in der jeweils anderen Körperhälfte. Frösche können eine Mutation besitzen, welche die Synthese von Ribosomen verhindert. Laut Erwartung sollte eine Segregation von 3:0 eintreten, da der Organismus ohne Ribosomen nicht funktioniert. Das Oocytenplasma enthält jedoch Ribosomen, welche die Fehlfunktion einige Zeit kompensieren, dann stirbt der Organismus jedoch. Bei M. musculus gibt es die Fellfarben-Phänotypen gelb, braun und gelb-agouti (die Haare sind der Länge nach zweifarbig: braun und gelb). Wenn man agouti mit agouti kreuzt bekommt man 100% agouti. Wenn man gelb mit gelb kreuzt bekommt man 2/3 Yellow und 1/3 Agouti. Das vierte Individuum stirbt, da gelb homozygot letal ist, dadurch ergibt sich diese Verteilung. Gelb ist dominant. Warum führt aber Fellfarbe zum Tod? Das Gen, das für die Fellfarbe verantwortlich ist, wird immer zusammen mit dem MERC-Gen vererbt, welches in der Embryonalentwicklung von Bedeutung ist und eine Deletion haben kann, die rezessiv letal ist. Dominant letale Merkmale sind relativ selten, da sich tote Individuen in der Regel nicht fortpflanzen, bildet sich der Phänotyp also vor der Geschlechtsreife aus, werden Allele also effektiv aus dem Genpool entfernt. Manifestiert sich der Phänotyp allerdings erst nach der Fortpflanzung, hält sich eine Mutation in der Population (Chorea Huntington, Veitstanz). Drosophila besitzt das P-Element, welches in der Entwicklung die Stabilität des Genoms gefährdet, da es bei Integration in Exon-Sequenzen Defekte hervorrufen kann. Durch die maternale Vererbung spezieller piwi-interacting RNA (piRNA) wird in einem RNA-silencing Mechanismus die genomische Integrität aufrecht erhalten. Weiters kommt die Pigmentierung der Larven und Augen der Mehlmotte (Ephestia kuehnellia) durch Einlagerung von Proteinen aus dem Ovar in den Dotter der Oocyte zu Stande. -6- 4.3 X-Inactivation / X-Linked Traits Die Inaktivierung eines X-Chromosoms ist eine Art der Gendosisregulation und ist mit Imprinting verbunden. Weibchen sind Gynander, genetische Mosaike, bezüglich der X-Inaktivierung, da dies zufällig passiert bzw. von unbekannten Faktoren bestimmt wird. Äußerlich nicht feststellbar, äußert es sich indirekt durch zB Variationen in den Sekreten der Haut. Der molekulare Hintergrund basiert auf einem X-inactivation center (Xic) auf dem X-Gonosom, einem Chromosomenabschnitt mit den Regionen Xist, Tsix, Xite und anderen. Xist codiert eine ncRNA (non-coding RNA), welche das transkribierende X-Chromosom umhüllt, Tsix ist für die Paarung der X-Chromosomen verantwortlich. Wie verhalten sich die beiden Sequenzen in der Ontogenese? In undifferentiated ES cells, transcription of Tsix from both chromosomes maintains Xist expression at low levels and restricts Xist RNA to the transcription site (day 0). Induction of differentiation triggers X–X colocalization, counting and choice. Tsix is then downregulated on the presumptive X i , thereby allowing Xist RNAs (blue lines) to accumulate in cis (day 2). On Xa, the repression of Xist is maintained through the persistence of Tsix expression promoted by the Xite locus. Subsequently, the X i accumulates heterochromatic epigenetic marks to maintain the silent state (Nature Cell Biology vol8/issue3 03/2006 208). Das Phänomen kann X-chromosomal rezessive Phänotypen erscheinen lassen, wenn das gesunde Allel inaktiviert wird bzw. zwei rezessive Allele vorhanden sind. Männchen zeigen immer den kranken Phänotyp (zB. Rotgrünblindheit, Hämophilie). Cytologisch ist das inaktivierte X-Chromosom als Barr-Körperchen feststellbar. Die Xchromosomale Vererbung verursacht eine Art „Hemizygotie“ bei Männchen, da es kein paternales Allel für die x-linked genes gibt (bei Männchen findet natürlich keine X-Inaktivierung statt). 4.4 Homozygote Letalität Dieses Phänomen verursacht Abweichungen in den Segregationsmustern des Mendelschen Vererbungsmodells durch Verhinderung der Entwicklung eines Genotyps. Mögliche Ursachen sind pleiotrope Auswirkung (agouti), Defektmutation beider Allele oder Erbung beider defekter Allele eines Elternteils (uniparental disomy). Abweichung der Mendelschen Regeln durch Tod des Individuums. 4.5 Imprinting Evolutionärer Druck bezüglich variabler Expression postnataler Gene trat erstmalig bei den Monotremata auf. Die Marsupalier haben daraus Imprinting und X-Inaktivation „erfunden“, beide Phänomene traten vermutlich zeitgleich auf, da sie mechanistisch ähnlich sind. Später entwickelte sich Methylierung zur Stabilisierung der Imprints und die zufällige Inaktivierung eines XGonosoms. Auch bei Pflanzen findet man Imprinting, was allerdings schlecht untersucht ist. Diploide Organismen zeigen auf Grund des möglichen Vorhandenseins homozygoter Allele ein Phänomen, welches für Gendosiskompensation sorgt, also eine quantitative Regulation der Transkription. Dies ist notwendig, damit der Organismus nicht an Überexpression von Genen leidet. Das Imprinting ist eine Art Schaltersystem, welches Gene selektiv positiv oder negativ reguliert, sodass einmal ein maternales Allel, an einer anderen Stelle widerum der paternale Genotyp in Erscheinung tritt. Imprinting beeinflusst also die Expression eines Allels oder Genlocus in Abhängigkeit maternaler oder paternaler Vererbung. Beispielsweise wird in der Maus ausschließlich das paternal vererbte igf2-Allel sowie das maternale H19-Allel exprimiert -7- Zur Zeit kennt man etwa 80-100 Genloci, welche selektiv paternale oder maternale Gene aktivieren und mit Krankheiten, Morpho- oder Ontogenen assoziiert werden. Hat man ein aktives und ein inaktives Allel und das aktive ist mutiert, so kommt es zur Ausbildung von Krankheiten, da das gesunde inaktiv ist und nicht kompensieren kann. Welche biochemischen Mechanismen stecken dahinter, wie schafft es die Zelle selektiv zwischen Genregionen zu schalten? Die Antwort lautet Cytosinmethylierung. Systeme, die mit Methylierung arbeiten, findet man sogar in E. coli, das seine Erbinformation mit spezifischen Methylierungsmustern versieht. Sie besitzen darüber hinaus Restriktionsenzyme (EcoK12), welche spezifisch fremde DNA erkennen (beispielsweise von Phagen), die nicht methyliert ist, und spalten. Allerdings können Phagen auch Methylierung nutzen, um sich zu „tarnen“ – hier ist EcoK12 wirkungslos. Imprinting findet in CpG-reichen, upstream genreicher Regionen oder regulatorischer Sequenzen statt, in so genannten „CpG-islands“ oder „imprinting (control) centers (ICs)“. Ein IC kann sich über mehrere tausend Basenpaare erstrecken und die Verteilung von ICs folgt keiner (bisher bekannten) Logik. Ob eine Basensequenz als IC wirken kann, hängt außerdem vom Geschlecht des Individuums ab. Beim Imprinting wird eine Methylgruppe (CH3) kovalent an den Pyrimidinring von Cytosin gebunden. Dort wird in der Regel die DNA inaktiviert und kompaktiert, was aber nicht zwingend zu einer Inaktivierung der dadurch kontrollierten Gene führt. Man spricht dann von einem „imprinted gene“. Es wird nicht jedes Gen einzeln reguliert, sondern mehrere zu einem Cluster zusammengefasst. Man erklärt Imprinting mit zwei Mechanismen. Einerseits kennt man imprinting control elements (ICE), auch imprinting control regions (ICR) genannt, welche die Wirkung von Enhancern beeinflussen und von Proteinen wie CTCF gebunden werden. Das menschliche Genom enthält etwa 15.000 Bindungsstellen für CTCF (chromosomal transcription control factor). Man spricht vom -8- insulator binding. ICEs müssen nicht in unmittelbarer Nähe des Leserahmens eines Gens liegen. Wird ein ICE methyliert, kann ein Enhancer seine cis-Wirkung ausüben. Der zweite Mechanismus besteht im Vorhandensein von non-coding RNA (ncRNA), primärer RNA, die selbst kein Protein codiert, relativ lang und lausig gesplicet ist und im Kern verbleibt. Sie beeinflusst die quantitative Transkription sowie die räumliche Verteilung bestimmter Faktoren. Ein ICE kontrolliert die Expression einer bestimmten ncRNA, welche die Expression umliegender Gene verhindert. Wird das ICE methyliert entsteht keine ncRNA, umliegende Gene sind nicht mehr gesilenced. Die Menge der Transkription der ncRNA ist insofern von Bedeutung, da sie den Zustand des Chromatins, also die Packungsdichte bestimmt. Wie sie das fertig bringt und was die Selektivität bedingt ist jedoch unklar. Imprinting wirkt auf Wachstumsgene. Warum hat sich Imprinting entwickelt? Dazu hat man zwei Modelle, nämlich die Genetic Conflict Hypothesis und die Ovarian Time Bomb Hypothesis. Erstere besagt dass, „Because of multiple paternity, a mother's offspring are equally related to her but can be less related to each other. A mother's genetic interests are best served by keeping control over the distribution of her resources to these offspring, sharing it equally among them. Mothers can retain such control by inactivating fetal growth-enhancing genes that they pass offspring to obtain as much of this resource as maximizes their survival, even at the expense of half-sibs and the mother. Inactivating fetal growth inhibitors in his offspring serves this purpose.”, zweitere dass, “The spontaneous proliferation of an unfertilized egg in an ovary is a form of ovarian trophoblastic disease, essentially ovarian cancer. Inactivating the only (maternal) copy of early-acting growth enhancers lowers this risk, as does upregulating any growth inhibitors. This second change could leave the fetus with too much inhibitor, an imbalance that can be corrected by downregulating the paternal inhibitor’s copy. Thus, both the GCH and the OTH predict that fetal growth-affecting genes are likely to be targets of imprinting, and that growth enhancers should be maternally attenuated and growth inhibitors, paternally so.” (Folien Jantsch) Eine Krankheit, die durch Imprinting hervorgerufen werden kann, ist das Prader-Willi-Syndrom. Das betroffene Gen heißt SNRPN ( Small nuclear ribonucleoprotein-associated protein N), dessen Funktion im gewebsspezifischen Splicen liegt. Das maternale Allel ist durch Imprinting inaktiv, das paternale Allel ist aktiv. Ist das paternale Allel durch Mutation defekt, kommt es zur Krankheit. Ist der Vater Träger eines defekten Allels kommt es bei 50% der Nachkommenschaft zur Krankheitsbildung, da ein inaktives und ein defektes Allel zusammen kommen. Wird das defekte Allel über die Mutter vererbt, ist die Nachkommenschaft nicht betroffen, aber Träger. Phänotypen von imprinted genes können verschiedene zu Grunde liegende Ursachen haben. (a) Es kann eine Uniparentale Disomie (UPD) vorliegen. Dabei stammen beide Chromosomen eines homologen Chromosomenpaares von einem Elternteil. Disomie kann eine Überexpression oder den Ausfall einer genetischen Expression zur Folge haben. Da beide Allele aktiv oder oder inaktiv sind, verhindert eine Abberation in der Embryonalentwicklung die Entwicklung von Embryonen (b) Ein imprinted gene kann mutiert und dessen funktionierendes Allel inaktiviert sein und (c) eine ICR selbst kann eine Defektmutation erleiden. Der Imprint status verändert sich im Lebenzyklus eines Lebewesens. In der Keimbahn kommt es zur Löschung von Methylmarkern, vermutlich in den Urkeimzellen (primordial germ cells). In den reifen Gameten wird der Imprint status wieder hergestellt. Woher die beteiligten Enzyme wissen, welche Bereiche sie wieder methylieren sollen, ist nicht bekannt. DNMT1 – Desoxyribonucleinsäuremethyltransferase1 wirkt bei der somatischen Mitose, indem sie -9- hemimethylierte DNA nach der Replikation methyliert (maintenance methylation) DNMT3 – Desoxyribonucleinsäuremethyltransferase3 sorgt für Methylierung in den Gameten nach dem Entfernen der Methylmarker in den Urkeimzellen (de novo methylation) 4.6 Su(var), E(var), pev Durch Imprinting kommt es zu einem Phänomen namens position effect variegation (pev), welches sich auf die Expressivität eines Gens auswirkt. Kommt ein Gen durch Mutation (zB. Inversion eines Chromosomenabschnitts durch Bestrahlung in Drosophila) in die Nähe einer heterochromatinen Region, so kann die transkriptionelle Aktivität des Gens verringert bis ausgesetzt werden. Verschiedene Faktoren lassen suppressor variegation Su(var) und enhancer variegation E(var) entstehen, welche das gene silencing durch Ausbreitung oder Lockerung des Heterochromatins regulieren. Wird es enhanced, ist das Gen stärker gesilenced, wird es suppressed, wird die Region transkriptionell aktiver (vgl. Kapitel 6, Chromatinorganisation). 5 – Analyse genetischer Pathways Genetische Analyse hat zum Ziel, die Auswirkung der Ausprägung von Gen A auf die Ausprägung von Gen B zu untersuchen, also festzustellen, ob gleiche Phänotypen auch gleiche Ursachen haben. Meistens entspricht ein beobachteter Phänotyp nicht einem speziellen Genotyp, sondern es liegen spektrale Defekte zu Grunde. Die Funktion von Genen stellt man beim Vergleich von Mutanten fest. Hängen Enzyme oder andere Faktoren in einem molekularen Pathway bzw. einer enzymatischen Kaskade zusammen und fehlt einer der Faktoren, sodass die Reaktionskette unterbrochen wird, spricht man von Epistasie. Am Beispiel der Phenylketonurie: Sie hängt von zwei Schlüsselfaktoren ab – Phenylhydroxylase und Tetrahydropterin (Cofaktor). Nun kann einerseits das Enzym defekt oder ineffizient sein, andererseits auch der Cofaktor, dadurch ist der Pathway unterbrochen bzw. seine Leistung vermindert. Gene welche im gleichen Pathway aktiv sind bzw. einander in ihrer Funktion beeinflussen sind oftmals epistatisch zueinander. Das Fehlen von Genprodukt A verhindert die Wirkung von Genprodukt B. A ist daher epistatisch zu B. B ist hypostatisch zu A. Epistatische Gene müssen biochemisch nicht upstream von hypostatischen Genen agieren. Die Gelbfärbung von Labrador Retrievern ist epistatisch zu brauner und schwarzer Fellfärbung. Die Ablagerung des Farbstoffes im Fell wird verhindert, nicht aber in anderen Regionen wie der Nase. Pigment wird gebildet (B schwarz, oder b braun), aber in Abhängikeit des Gens E/e nur in der Nase oder auch im Fell abgelagert. Das Fucosyl-Transferase (Fut1) Gen (Bombay-Phänotyp) ist epistatisch zum AB0 System. Dies - 10 - führt zu einer 6:3:3:4 Segregation. Dieses Enzym stellt eine Vorstufe durch glycosidische Bindung von Fucose an die Substanz H her, welche nach Glykosylierung mit α-Galactose bzw. α-N-AcetylGalactosamin zum Glykoprotein der Blutgruppe B bzw. A wird. Fehlt das Enzym, kann nicht mit Galactose oder ihrem Derivat glykosyliert werden, man hat also Blutgruppe 0. Defekte Gene, welche im gleichen Pathway aktiv sind, können durch Komplementation analysiert werden. Mutationen in beiden Allelen eines Gens komplementieren einander nicht. Mutationen in unterschiedlichen Genen komplementieren einander. Voraussetzung für die Komplementation ist - 11 - Diploidie bzw. die Existenz von Wildtyp-Allelen. Ein Beispiel für Komplementation ist die Bildung von Blütenfarbstoffen, hier in Campanula (Glockenblume). Aus einem Precursor wird in zwei enzymatischen Reaktionen einer blauer Farbstoff gebildet. Die Parentalgeneration trägt in beiden Allelen von Enzym 1 oder Enzym 2 eine Mutation, sodass der Pathway unterbrochen ist (weiße Blüten, Precursor wird nicht vollständig umgesetzt). In der F1-Generation tauchen Individuen auf, die blaue Blüten haben. Warum? Diese Individuen haben ein mutiertes und ein gesundes Allel, letzteres kompensiert die Funktionslosigkeit des anderen, es ist wieder Enzym vorhanden, um den Precursor umzusetzen. Komplementation ist eingetreten. Selbstbefruchtung dieser Individuen führt in der F2-Generation zu einer phänotypischen 9:7 Segregation: 9 blau, 3:3:1 weiß. Nicht nur Gene, deren Produkte in gleichen Pathways aktiv sind, sondern auch Gene deren Produkte einander regulieren (zB. transkriptionell) zeigen eine atypische Segregation. Epistasis Analysen geben aufschluss darüber, ob zwei Prozesse funktionell miteinander gekoppelt sind. Für die Untersuchung von genetischen Pathways setzt man verschiedene Strategien ein. Supressor Mutationen revertieren den Phänotyp von Mutationen, indem Interaktionspartner des mutierten Genprodukts ebenfalls mutiert werden. Die primäre Mutation wird suprimiert. Dabei werden öfters Genloci identifiziert, deren Produkte vorher gar nicht mit dem Pathway assoziiert worden sind. Als Beispiel sei ein Gen m mutiert. Die Supressormutation s ermöglicht eine Interaktion mit dem mutierten Protein m. Genetische Screens für Supressoren ermöglichen die Identifikation von interagierenden Proteinen oder downstream targets eines Proteines m+. Supressoren funktionieren nach drei Wirkprinzipien: • • Interaction supressor. In einem Wildtyp binden Protein A und Protein B einander. Nun mutiert B, A kann nicht mehr binden. Eine Supressormutation in A stellt die Interaktion wieder her. Bypass supressor. In einem Wildtyp existieren ein funktionierender Pathway A und ein - 12 - • stummer Pathway B. Eine Mutation in A macht diesen funktionslos. Eine zweite Mutation in B aktiviert Pathway B, der Pathway A nun ersetzt. High copy supressor. Ein stabiles Genprodukt A tritt mit einem Produkt B in Wechselwirkung. Produkt A mutiert und entfaltet sich im ungebundenen Zustand. Produkt B wird vermehrt gebildet, um mehr A zu binden und durch die Wechselwirkung die Funktionalität von A aufrechtzuerhalten. Modifier oder synthetic lethals beeinflussen einander und verändern (verstärken) einander in deren Phänotyp. Genetische (Mutanten)-Screens für synthetic lethality und Suppressoren ermöglichen die Analyse komplexer Pathways. Einzelne Mutationen in einem Pathway stören meist nicht, mehrere Mutationen können aber zum Ausfall führen. Hier setzt synthetic lethality an, indem man Komplementationsverhältnisse stört, um Phänotypen zu erhalten. Als Beispiel zwei Proteine (A und B), die ein Heterodimer bilden und an DNA binden. Schwächt eine Mutation in A die Affinität zur DNA, so kann die B-Untereinheit die gemeinsame Funktion aufrechterhalten. Mutiert allerdings B auch noch (synthetic lethal), fällt das Heterodimer von der DNA ab und, falls A und B als Transkriptionsfaktor an einen Promoter gebunden haben, wird ein Gen nicht mehr exprimiert, man erhält einen Phänotyp, in solchen Fällen meistens den Tod. Penetranz und Expressivität sind weitere genetische bzw. molekulare Phänomene. Ersteres beschreibt, ob ein Merkmal sichtbar wird oder nicht, zweiteres, bestimmt das Maß, in dem ein Merkmal ausgeprägt wird. Variabilität in der Expressivität und Penetranz eines Allels können vom genetischen Hintergrund des Organismus, der Position des Gens, oder von Umweltfaktoren (Temperatur, Ernährung) abhängen und führt zu variablen Phänotypen. Ein Beispiel zur Penetranz ist die Fellfärbung von Schneehasen (Lepus timidus) und Siam-Katzen. Pigmentierung ist bei erhöhter Temperatur gestört. In den Extremitäten kann die Pigmentierung erfolgen, da die Körpertemperatur geringer ist und bestimmte Enzyme wirksam werden. Diese Tiere sind aber keinesfalls Chimären. Hat eine Familie genetische Prädisposition für Tumore, so heißt das nicht, dass diese auch in jedem Individuum entstehen, vielmehr bestimmen Umweltfaktoren, ob das Merkmal penetriert, also zum Vorschein kommt. 6 – Chromatinorganisation DNA ist im Zellkern mit Proteinen assoziiert. Das Chromatin ist aber keine homogene Masse, es existieren genreiche und genarme Regionen, andere sind widerum für Enzyme schwer zugänglich, vergleichbar mit einer schlechten Verkehrsanbindung. Sogenannte house keeping genes sind in jeder Zelle erreichbar, gewebsspezifische Promoter werden im Zuge der Zelldifferenzierung still gelegt. Die Totipotenz von Stammzellen geht durch Verpackung und Modifikation mit Inaktivierungsmarkern verloren, man spricht von epigenetic programming, das aber weit über das einfache Anund Ausschalten von Genen hinausgeht. Chromatin kann in zwei Zuständen, euchromatin und heterochromatin, vorliegen, die cytologisch durch Anfärben erkannt wurden. Euchromatin ist lockerer gepackt und erscheint heller als Heterochromatin. Zweiteres unterteilt man weiters in • konstitutives Heterochromatin - diese DNA-Regionen sind permanent unzugänglich. → Centromer: Hier sind keine Gene, diese Region hat andere Aufgaben. - 13 - • → Telomer: Chromosomenenden sind leicht anzugreifen, hier dürfen keine wichtigen Gene liegen → Genarme Regionen: verhindert Springen von Transposons und unnötige Aufblähung des Genoms fakultatives Heterochromatin – diese DNA-Regionen sind nur zeitweise inaktiv. → X-Inaktivierung Wie hat man erkannt, dass position effect variegation Auswirkung auf die Expressivität hat? In white+/+ Drosophila hat man mittels Röntgenstrahlung DSBs eingeführt. Die freien Enden wurden mit falscher Orientierung repariert, als Folge waren betreffende Ommatidien farblos, obwohl die genetische Information für die Färbung vorhanden, aber nicht mehr zugänglich war. 6.1 Nucleosomen Die molekulare Grundlage für die Zugänglichkeit von genetischer Information sind Nucleosomen. Sie sind aus verschiedenen Histonen aufgebaut, die zusammen ein Histonoktamer bilden. Die Histone H2A, H2B, H3 und H4 sind pro Nucleosom zweifach vorhanden. Die DNA-Doppelhelix wird in zwei Schlingen um ein Nucleosom gewunden, es entsteht die beads-on-a-string Struktur (10nm-Faser). Zwischen zwei Nucleosomen bleibt die DNA nackt (165 bp in Euchromatin, 146 bp in Heterochromatin). Wie ist man auf die Existenz von Nucleosomen gekommen? Eigentlich war es Unfall: Unbeabsichtigterweise ist es in einem Experiment zu Endonucleaseaktivität gekommen, im Gel hat man dann 146 bp und 165 bp Fragmente gefunden, dass diese Fragmente nicht weiter zersetzt wurden, hat man der protektiven Wirkung von Proteinen zugeschrieben. Das Histon H1 ist ein linker, der zu einem spiraligen Gerüst assembliert, an dem die Nucleosomen orientiert werden. So erreicht man eine höhere Packungsdichte (30nm-Faser). - 14 - Die N-Termini der Histone sind flexible Regionen, ragen aus der Oberfläche des Nucleosoms heraus und sind für Proteinmodifikationen zugänglich. Am häufigsten wird acetyliert, methyliert oder phosphoryliert, aber auch andere Modifikationen wie Ubiquitinierung oder SUMOylierung können stattfinden. Die DNA ist als Säure negativ geladen, die Histone als Base stark positiv. Proteinmodifikation verändert den Ladungscharakter der Histone und somit die Nettoladung des Nucleosoms. Das Anhängen saurer Reste vermindert zum Beispiel die Packungsdichte, da die eingebrachte negative Ladung die positive des Histons kompensiert und in Folge sich die DNA weitet, weil jetzt lokal nur noch negative Ladung vorherrscht (gleiche Ladung stößt sich ab!). Modifikation Aminosäure Enzymklasse Wirkung Acetylierung Lys Histonacetyltransferase (HAT) Negative Ladung Methylierung Lys / Arg Histonmethyltransferase (HMT) Hydrophobizität Phosphorylierung Ser / Thr Kinase Negative Ladung Die modifizierten Aminosäuren sind widerum Bindungsstellen für andere Proteine mit speziellen Domänen, die ihrerseits wieder Aminosäurereste modifizieren können. Acetylierte Lysine werden von Proteinen mit einer Bromodomäne erkannt, methylierte Lysine von Proteinen mit einer Chromodomäne und phosphorylierte Aminosäuren von Proteinen mit 14-3-3-Domänen. Die SETDomäne von einigen Methyltransferasen hat zum Beispiel hohe Spezifität für H3K9, dem Lysin an Position 9 von Histon 3. Bindet so eine Methyltransferase, werden manche umliegende Lysine methyliert. 6.2 Histoncode-Hypothese Heterochromatinbildung ist ein vielstufiger Prozess, man kann nicht einfach zwischen aktivem und inaktivem Chromatin unterscheiden, es gibt viele Abstufungen. Der Epigenetiker sucht in Histonmodifikationen versteckte Information in Form des Histoncodes. Seine Eigenschaften: • • • Modifikationen passieren an definierbaren Stellen Modifikationen passieren in enzymatischen Kaskaden abhängig von anderen Modifikationen Modifikationen führen zu Chromatinorganisationsstufen Dieser Code könnte kombinatorischen Charakter haben, ein Beispiel: H3S10 ist in aktivem und inaktivem Zustand phosphoryliert, im aktiven Zustand H3K9 und H3K14 zusätzlich acetyliert. - 15 - Methylierung findet sowohl in Eu- als auch in Heterochromatin, ein Acetylierungsmuster entscheidet dann über zugänglich oder unzugänglich. Acetylierung trägt in der Regel zur Aktivierung bei, methylierte Histone sind vermehrt in Heterochromatin zu finden. Lysin kann mono-, di- oder trimethyliert sein. H3K4me ist ein Aktivierungsmarker, H3K9 ein Inaktivierungsmarker. Der Histoncode ist (beinahe) universell, in jedem Organismus, dessen genetische Informationen mit Histonen strukturiert ist, findet man die Methylierung H3K9, die aber unterschiedliche Funktionen übernimmt. Man gliedert die Histon-modifizierenden Faktoren in zwei Gruppen: • • Su(var) → Erhöhen die Packungsdichte − Histondeacetylasen (HDAC) − Proteinphosphatasen (PPTasen) − S-Adenosylmethionin Synthetase − Heterochromatinprotein 1 (HP1) − Histonmethyltransferasen E(var) → Erniedrigen die Packungsdichte − Histonacetyltransferasen − SWI/SNF Um diese Gliederung zu verstehen, muss man um die Ecke denken. Man hat bei der Benennung Bezug auf die Expression einer Mutation genommen. Sie trat bei erhöhter Packungsdichte (Su(var)) seltener auf und bei E(var) konnte sie öfters beobachtet werden. 6.3 NuRD-Komplex Neben den genannten Enzymen gibt es andere Faktoren, die in differentiertem Gewebe exprimiert werden. Der NuRD-Komplex beinhaltet Histondeacetylasen (HDAC). Ist H3K4 methyliert, kann der NuRD-Komplex nicht binden, andernfalls wird H3 deacetyliert und in weiterer Folge methyliert. Wie gelangt der NuRD-Komplex an die richtigen Stellen? Im Zuge des imprintings kommt es zur Methylierung von CpG-islands der DNA, die von MBD2 erkannt wird. Der NuRD erkennt diese Marker mittels MBD3 (methyl-bindung domain) und übersetzt die Anweisung zur Stilllegung der Promoter auf die Proteinebene. Dieser Mechanismus ist bei der Differenzierung von Geweben sinnvoll, da manche Gene in manchen Geweben nutzlos sind. Beispielsweise wäre die Synthese von Alkoholdehydrogenase (ADH) im Auge alles andere als energieeffizient, daher sollen solche Gene nicht einmal von starken Transkriptionsaktivatoren wieder aktivierbar sein. Solche Erkenntnisse sind wertvoll, wenn man Stammzellen verstehen möchte. Gibt man LIF (leukemia inhibitory factor) zu einer ES-Kultur, erhält man so den undiffernzierten Zustand. Setzt man es ab, kommt es zur Differenzierung. Bei einer NuRD-Mutation bleiben viele Zellen undifferenziert, weil die Deacetylierung nicht stattfindet. Wie kommt es zur Reversion von Heterochromatin in Euchromatin? Dazu werden drei Strategien verfolgt: - 16 - • • • Marker entfernen: PPTasen (Phosphat), HDAC (Acetat) und Jumanji-Proteine (Methyl, erst vor wenigen Jahren entdeckt) Histon abbauen: Dabei gehen gleichzeitig alle Inaktivierungmarker verloren. Biochemisch läuft Proteolyse ab, Polyubiquitinierung markiert das Histon für den Abbau des N-Terminus oder des ganzen Proteins. Marker dazugeben: H3K9me, gebunden von HP1, ist total inaktiv. Wird aber H3S10 von Aurora B Kinase phosphoryliert, fällt HP1 ab. Eine mutierte Aurora B Kinase zeigt in Drosophila hyperkondensierte Polytänchromosomen. Wie werden Histonmodifikationen vererbt? Die spekulative gängige Meinung geht davon aus, dass die Nucleosomen in einem semikonservativen Prozess 50:50 auf die Tochterzellen weitergegeben werden. Neu synthetisierte Nucleosomen werden dabei mit den Markern versehen, die am Nucleosom am Doppelstrang gegenüber erkennbar sind. Um Transkription zu ermöglichen muss chromatin remodelling stattfinden, um eine Histon-freie Region zu schaffen. In der Hefe bindet GCN5 im Vorfeld der Transkription, welches HAT-Funktion ausübt. Das acetylierte Histon in der Umgebung des Promotors lockert sich. Nach Bindung der Polymerase wird SWI/SNF rekrutiert, das wie ein Schneepflug Nucleosomen disassembliert. Der experimentelle Beleg dafür ist, dass die Distanz zwischen den Nucleosomen beobachtbar größer wird. Nachdem die Polymerase weitergezogen ist reassemblieren die Nucleosomen wieder. Bei der X-inactivation kommt es auch zur Strukturveränderung des Chromatins. Sie passiert in somatischem Gewebe zufällig, in der Embryonalentwicklung allerdings nur zum Teil. Die Zelle muss feststellen können, wie viele X-Chromosomen sie hat. Woher weiß sie, wie viele vorhanden sind? Die molekulare Grundlage ist das XIC (X-chromosome inactivation center), fehlt es, kommt es nicht zur Inaktivierung. Es enthält verschiedene Sequenzen: Xist → Xist RNA (frei nach Hamlet: to eXist or not to eXist, that is the question) Tsix → Antisense Transkript zu Xist, down-regulation von Xist Xite → Regulator In YACs (yeast artificial chromosomes) wurden diese Sequenzen experimentell mittels Deletionen untersucht, bei Tsix-Deletion haben die X-Chromosomen nicht mehr gepaart. Liegt Tsix auf einem Autosom, so paaren X- und Autosom. Xist RNA wird konstitutiv exprimiert, wird aber schnell wieder degradiert. Irgendwann bleibt die RNA eines Chromosoms stabil und umhüllt das produktive X-Chromosom. Welches Signal dafür verantwortlich ist, ist unklar. Das von Xist RNA umhüllte Chromosom rekrutiert Proteinfaktoren, die über Histonmodifikation permanente Inaktivität festigen. In der Embryonalentwicklung gibt es diese Zufälligkeit nicht, es wird immer das paternale Allel exprimiert und somit das paternale X-Chromosom inaktiviert. Aber nur bis zum Blastocyste. Im Trophoblast (extraembryonales Gewebe, Placenta) bleibt weiterhin das paternale gesilencet, im ICM (inner cell mass) des Embryos beginnt der Kampf um Aktivierung von neuem. Phylogenetisch hat sich die Zufälligkeit der Inaktivierung erst bei den Placentariern entwickelt, bei den Marsupaliern, den „Erfindern“ der X-Inaktivierung, wird ausschließlich das paternale gesilencet. - 17 - 6.4 Chromatin in der Mitose In der Mitose wird Chromatin in Chromosomen verpackt. Mehrere Zentimeter lange Moleküle müssen dabei auf 1-5 µm (Faktor 2.000-10.000 für ein 10mm langes Chromosom) zusammen gepackt werden. Eine enorme Leistung, die in mehreren Kondensationsschritten vollbracht wird. Löst man ein Metaphasen-Chromosom in 3-5M NaCl, so werden ionische Wechselwirkungen gestört und die DNA liegt wieder als Faden vor, allerdings bleibt eine Proteinstruktur zurück, das protein scaffold. Dieses besteht aus Nicht-Histon-Proteinen wie Topoisomerasen und SMCProteinen (structural maintenance of chromosomes). Topoisomerasen, speziell Topoisomerase 2 (TopoII) kann mit Antikörpern nachgewiesen werden. Topoisomerasen lösen aufgestaute DNA-Schleifen auf und werden entsprechend ihrem biochemischen Mechanismus in zwei Gruppen geteilt: • • Typ1 Topoisomerasen: Führen Einzelstrangbrüche ein Typ2 Topoisomerasen: Führen Doppelstrangbrüche ein (TopoII) Im aktiven Zentrum sitzt immer ein Tyrosinrest, der in einem nukleophilen Angriff eine Umesterung eines Phosphodiesters der DNA vornimmt. Die zweite große Gruppe an scaffold-Proteinen sind SMC-Proteine. Die ATPasen SMC2 und SMC4 bilden einen durch den Zellzyklus kontrollierten Condensin-Komplex, zwei coiled-coils, die über ein hinge-Motiv verbunden sind. Kleisine verbinden die V- förmige Struktur zu einem Ring, der hilft, das Chromatin zu kompaktieren. Im (umstrittenen) loop model of chromosome organisation assoziieren matrix attachment regions (MARs) mit der hypothetischen nuclear matrix. Manche Forscher postulieren die nuclear matrix als eine Art chromosomales Cytoskelett. Das Argument, dass es keine Antikörper dagegen gibt/ geben kann, steht der high salt extraction gegenüber. Mit dieser Methode kann man unbekannte Proteine aus dem Zellkern extrahieren. Die Theorie klingt plausibel, experimentelle Evidenz ist allerdings noch ausständig. - 18 - 6.5 Karyotyping Karyotypisierung beruht auf der Extraktion und selektiven Färbung von Chromosomen. Im Gbanding setzt man Giemsa ein, das eine Unterscheidung von Eu- und Heterochromatin erlaubt. Chromosomen-Banding erlaubt die Erstellung eines Karyotyps und die Identifizierung chromosomaler Aberrationen (numerisch und strukturell). Jeder G-Bande wird eine Nummer zugewiesen, der kurze Arm wird als p-Arm, der lange als q-Arm bezeichnet. Eine eindeutige Bezeichnung einer Abberration könnte lauten: t(11;22)(p13;q11.2) – Translokation zwischen den Chromosomen 11 und 22, am p-Arm von Chromosom 22 schließt an Bereich 13 der zweite Bereich des q-Arms von Chromosom 11 an. Entsprechend der Lage des Centromers spricht man von metazentrischen, submetazentrischen, akrozentrischen und telozentrischen Chromosomen. Karyotypen sind Speziesgrenzen. Der chinesische und der indische Muntiac haben etwa die gleiche Menge an DNA, diese ist aber unterschiedlich auf Chromosomen verteilt (beim indischen Muntiac sind einige Chromsomen fusioniert). Die beiden Spezies können miteinander keine Nachkommen haben, da keine homologen Chromosomen paaren können. 6.6 Chromosomenabberationen Chromosomenabberationen können auf unterschiedliche Weise zu Stande kommen. Man versteht darunter Mutationen, bei denen ganze Chromosomenabschnitte deletiert, translociert, invertiert oder dupliziert werden sowie Ereignisse, die zum Verlust oder Gewinn zusätzlicher Erbinformation führen. Das Burkitt-Lymphom hat seine Ursache in der Translokation des c-myc-Gens von Chromosom 8 in die Nähe einer der Immunglobulin-Ketten Fc, λ, κ auf den Chromosomen 14, 22 oder 2. Beim Philadelphia-Chromosom ist es zum gegenseitigen Austausch zwischen Chromosom 9 und 22 gekommen. Dadurch entstehen neuartige Fusionsgene. Das ursprüngliche Gen c-ABL, eine Tyrosinkinase, fusioniert mit BCR, sodass in Folge ein BCR-ABL-Fusionsprotein gebildet wird, das eine konstitive Signaltransduktion nach sich zieht und Apoptose blockiert – eine Tumorzelle entsteht. Bei der Änderung in der Anzahl von Chromosomen unterscheidet man zwischen: • • abberrante Euploidie – Änderung der Zahl von ganzen Chromosomensätzen Aneuploidie – Änderung der Anzahl einzelner Chromosomen Monoploidien und Polyploidien sind abberrante Euploidien. Polyploidien unterscheidet man weiter in Autopolyploidie, mehrfacher Chromosomensatz der gleichen Spezies, und Allopolyploidie, die Chromosomensätze stammen von verschiedenen Spezies. Organismen, die parthenogenetisch reifen, sind oft monoploid. Staatenbildende Insekten umgehen die Meiose (Drohnen). Monoploide Organismen umgehen die Meiose bei der Gametogenese. Polyploidie ist ein häufiges Phänomen in Pflanzen. Indirekte Evidenz dafür sind die im Vergleich zu ungeradzahligen häufigen geradzahligen haploiden Chromosomensätze. Nematoden und Amphibien können abberrante Euploidien haben. Normalerweise geht mit dem Ploidiegrad die Körpergröße einher. Auf Grund von Segregationsfehlern in der Meiose sind Triploide und Alloploide Organismen in der Regel steril. Bei der Gametogenese können Trivalente oder andere Falschpaarungen auf Grund fehlender homologer Chromosomen auftreten, daher sind die Nachkommen aneuploid. - 19 - Wie erhält man wieder fertile Nachkommen? Die Lösung heißt komplette Verdopplung der Chromosomen durch Behandlung mit Colchizin, dem Gift der Herbstzeitlose. Es depolymerisiert Tubulin, somit zerfällt der Spindelapparat. Es findet keine Segregation statt, dafür stehen jetzt homologe Chromosomen zur Verfügung, um eine korrekte Segragation durchzuführen. Man behandelt zum Beispiel den Zweig einer Pflanze, damit die Blüten daran polyploide Gameten erzeugen. Aber es können immer noch Segregationsfehler auftreten: Quadrivalente (Chromosomen haben partielle Ähnlichkeit zueineander), Univalent und Trivalent. Sequenzierung von Genomen hat gezeigt, dass viele Pflanzen pseudo-polyploid sind, ihre Genome aus verschiedenen frühen Auto- und Allopolyploidisierungen hervorgegangen sind. Heute verhalten sie sich aber wie normale diploide Organismen. Fremdbestäubung in Pflanzen führt oft zur Entstehung steriler Hybride. Ein Beispiel ist die neue Spezies Raphanobrassica, die aus Hybridisierung von Vertretern der Gattungen Brassica und Raphanus von Karpetschenko (1928) erzeugt wurde. Die F1-Genertation war allerdings steril. Durch Behandlung mit Demecolcin (Colcemid, zweithäufigstes Alkaloid der Herbstzeitlose) wurde der Chromosomensatz dupliziert, es entstand ein amphidiploides, fertiles Individuum mit je einem diploiden Chromosomensatz der Eltern. Hybridisierung und Alloploidisierung sind häufige Ereignisse in der Entwicklung (Evolution) neuer Pflanzenspezies, speziell bei der Entstehung der heutigen Kulturpflanzen (Hybridisierung der Brassicaceaen B. nigra, B. oleracea und B. Campestris). Auch der moderne Weizen ist Resultat von Alloploidisierungen. Vor rund 10.000 Jahren sind Triticum monococcum und Triticum searsii zu einem tetraploiden Hybriden (Triticum turgidum) verschmolzen, später hat er mit dem wilden Weizen Triticum tauschii den hexaploiden Hybriden und heutige Grundnahrungsmittel Triticum aestivum gebildet. Wie geht man bei moderner Pflanzenzüchtung vor? Zuerst isoliert man die Protoplasten aus den Zellen der Eltern. Die Protoplasten werden mit chemischen Agentien zur Fusion angeregt. Nun hat man Hybride somatischer Zellen vorliegen. Die Stimulation zu Differenzierung und Teilung lässt ein kleines amphidiploides Pflänzchen entstehen, das aber steril ist. Endomitose kann zur Allopolyploidisierung führen, die den meiotischen Defekt kompensiert. Die Duplikation oder Deletion einzelner Chromosomen (Aneuploidie) entsteht hauptsächlich durch non-disjuntion in der ersten, selten auch in der zweiten, meiotischen Teilung. Aneuploidien sind beim Menschen sind meist nicht mit dem Leben vereinbar und führen zu - 20 - Spontanaborten, handelt es sich um kleinere Chromosomen werden Aneuploidien toleriert. Bei aneuploiden Chromosomensätzen unterscheidet man Trisomien, Monosomien und Nullosomien. • • • • X0 – Turner-Syndrom XXY – Klinefelter-Syndrom Trisomie 21 – Downs-Syndrom Trisomie 18 – Edwards-Syndrom Warum werden Aneuploidien kleiner Chromosomen toleriert? Dazu gibt es verschiedene Theorien, Tatsache ist, dass zum Beispiel Chromosom 21 sehr klein und genarm ist. Deswegen findet wenig Rekombination und physischer Kontakt statt, eine adäquate Segregation ist nicht gewährleistet. Die Gonosomen paaren über eine sehr kurze, pseudo-autosomale Region, wodurch auch hier Segregationsfehler passieren können. Das Risiko für eine Trisomie 21 korreliert exponentiell mit dem Alter der Mutter. PID könnte Aufschluss über einen vorliegenden Fall geben, ist in Österreich aber verboten, es widerspricht offenbar moralischen Wertvorstellungen. 6.7 Centromer Das Centromer beherbergt das Kinetochor, eine Proteinstruktur mit drei Komponenten. An die Corona (Motorproteine) binden Microtubuli. • • • inner kinetochor plate outer kinetochor plate corona - 21 - Was ist die Funktion, wie und warum bildet sich ein Kinetochor? Das Kinetochor ist ein Ankerpunkt für den Spindelapparat, Motorproteine üben Kraft aus, die die Chromosomen bzw. Schwesterchromatiden auseinanderzieht. Microtubuli haben (+)-Enden und (-)-Enden. Die (-)-Enden befinden sich an den Centrosomen (microtubuli organizing center), an den (+)-Enden wird Tubulin polymerisiert. Depolymerisation passiert an beiden Enden, am (+)-Ende viel schneller. Wie ist eine Centromersequenz definiert? Experimente in der Hefe zur Charakterisierung von CDEs (centromer DNA elements) beigetragen. Dazu wurden ade2– Hefestämme mit Plasmiden transformiert, die ade2 (Adeninbiosynthese) und verschiedene Fragmente des Hefegenoms enthalten haben. Normalerweise segregieren Plasmide nicht geordnet, sondern teilen sich irgendwie auf die Tochterzellen auf. Trägt ein Plasmid allerdings ein CDE-insert, so kann sich ein Kinetochor bilden und ade2 bleibt in beiden Zellen erhalten. Rote Kolonien signalisieren eine Unfähigkeit zur Adeninsynthese, hier sammelt sich ein farbiges Zwischenprodukt an. CDEs finden sich in fast allen HefeChromosomen. Bei S. pombe und höheren Eukaryoten bildet repetitive DNA die Centromere, auch die Centromergröße variiert. Gene, die in Centromerregionen inserieren, erfahren silencing, auch repetitive Elemente befinden sich an mehreren Positionen im Genom, daher vermutet man, dass ein Centromer epigenetisch definiert ist. In der Hefe binden verschiedene Proteinfaktoren an CDEs, die CBFs genannt werde (centromer bindung factors). CBF3 ist dabei ein Adapter für den Spindelapparat. Darüberhinaus befindet sich an den Centromeren eine Histon H3 Variante, das CSE4 (CenH3, CEN3 Nucleosom). Im Menschen sind Centromere durch α-Satelliten DNA (171 bp) definiert, die CENP-B Boxen enthält, das sind Bindungsstellen für das CENP-B Protein. Auf Chromosom 1 befindet sich eine zweite Region, die bei Verlust des ersten Centromers einspringen kann. Wie das passiert, ist unklar. Neben CENP-B sind CENP-A und CENP-C am Kinetochor beteiligt. CENP-A ist das Analogon zu CSE4, die Funktion von CENP-C ist unbekannt. 7 – Replikation Das Replizieren von Genomen ist eine erstaunliche Leistung, müssen doch Strukturen wie Telomere erhalten oder eine korrekte Segregation von Chromosomen gewährleistet werden. Die Replikation in prokaryotischen unterscheidet sich von Replikation in Eukaryoten. Der Zellzyklus wird durch checkpoints kontrolliert, besonders an heiklen Stellen wie Replikation oder Segregation der Chromosomen muss besonders reguliert werden. Fehlen die checkpoints, läuft der Zellzyklus weiter, aber unkontrolliert. Man unterscheidet zwischen unidirektionaler (Eukaryoten) und bidirektionaler (Prokaryoten) Replikation. Bakterielle Replikation ist bidirektional ausgehend von einem einzigen ORI. Sie wird initiiert, wenn ein bestimmter Grenzwert der Zellmasse überschritten wird. DnaA bindet an hochaffine Sequenzen am ORI, fördert das Schmelzen A-T-reicher Bereiche (DUE, DNA unwinding element) und das Beladen mit DnaB (Helicase). Vom Fis-Protein (factor of inversed stimulation) liegen etwa 50.000 Moleküle pro Zelle vor, die mit DnaA konkurrieren. Wächst eine Zelle verringert sich die relative Konzentration an Fis, die reprimierende Wirkung von Fis lässt nach, Replikation kann beginnen. Bakterielle DNA hat bestimmte Terminationssequenzen (terC, 23 bp), zwei ähnliche pro Nukleoid oder Plasmid. Diese sind beim Stoppen der Strangsynthese und der Dekatenierung mittels Topoisomerase II von Bedeutung. Für Replikation, speziell Strangsynthese, ist ein freies 3'-OH Ende erforderlich. Wie wird das zur Verfügung gestellt? Verschiedene Strategien sind entstanden, um das Problem zu lösen: - 22 - • • • DNA nick: Einzelstrangbruch RNA primer: DnaG (Primase) Einzelnes Cytosintriphosphat: Adenoviren Nachdem DnaA gebunden hat, wird DnaB und DnaC vom prepriming complex rekrutiert. Wie werden leading und lagging strand koordiniert? Die Synthese des lagging strands erfolgt diskontinuierlich, die Syntheserichtungen laufen genau genommen in entgegengesetzter Richtung. Es gibt zwei Modelle der Okazaki-Fragmentverlängerung: • • dissociation-association: Eine katalytische Untereinheit fällt ab, eine andere bindet recycling: Dieselbe katalytische Untereinheit wird wiederverwendet Das recycling-Modell ist das wahrscheinlichere, eine sliding clamp (β-Untereinheit der Polymerase) wird mittels ATP-abhängigen clamp loading factors an den DNA-Strang geladen, je einmal am leading und am lagging strand. Die clamp ist affin zur DNA Polymerase III, die τ-Untereinheiten sorgen für Dimerisierung des Komlexes. Das Äquivalent der clamp in Eukaryoten ist PCNA (proliferating cell nuclear antigen), ein Tumormarker. Das Voranschreiten der DNA Polymerisation stellt man sich so vor, dass die DNA durch den Replikationskomplex durchgezogen wird. Die diskontinuierliche Synthese des lagging strands verläuft unterschiedlich in Prokaryoten und Eukaryoten. In Bakterien hat DNA Pol III keine 5'-3' Exonuclease-Aktivität, DNA Pol I ersetzt die Okazaki-Fragmente mit DNA, Ligase verbindet die Lücke im Phosphatrückgrat. In Eukaryoten wird der Primer einfach im Zuge der Verlängerung des nächsten von FEN1 von der Matrize gelöst, die Strangsynthese wird fortgesetzt, bis er auf DNA trifft. Die 3'-5' Lücke wird ebenfalls von Ligase geschlossen. Viele DNA Polymerasen haben eine 3'-5' proofreading-Aktivität. Dabei wirkt eine Untereinheit der Polymerase als Exonuclease und entfernt Nukleotide, bis das mismatch gefunden wurde. Virale Polymerasen oder die Taq-Polymerase, das man für PCR einsetzt, haben keine ExonukleaseAktivität und daher hohe Fehlerraten bis 10-5. - 23 - Eukaryotische Chromosomen haben viele ARSs (autonomously replicating sequences, äquivalent zu ORIs). Sie liegen 40-100 kb auseinander und deshalb so zahlreich, weil sonst Jahre vergehen würden, bis zum Beispiel ein Mensch seine Embryonalphase beendet hätte. Welche Sequenzen das sind, hat man mit Hefescreens herausgefunden. Mutationen in den Bereichen A, B1, B2 und B3 einer ARS haben dessen Aktivität stark herabgesetzt. In Eukaryoten sind viele Polymerasen an der DNA Replikation beteiligt. Die Polymerasen α (Initiation), δ (leading strand Elongation, ε (lagging strand elongation, unsicher), γ (mitochondriale Replikation) haben replikative Funktionen. Man hat bisher keine Koordination zwsichen leading und lagging strand Synthese finden können. Die Initiation der Replikation hängt von ähnlichen Faktoren wie in Prokaroyten ab, allerdings gibt es kein Homolog zum τ-Protein, so liegen die Replikationskomplexe nicht dimer vor. Wie wird das gleichzeitige Auslösen der ARSs zur Replikation gewährleistet? Starke Kontrolle geht von Cyclinen und cyclin-dependant kinases (cdk) aus, dem Kontrollsystem des Zellzyklus. In der Hefe bleibt der ORC (origin recognition complex) permanent an ARS gebunden. Mit dem ORC sind in der G1-Phase Cdt1 und Cdc6 sowie MCM (maintenance of minichromosomes) assoziiert. In der S-Phase wird die Assemblierung durch Phosphorylierung dieser Faktoren verhindert, somit kann eine Initiation nur in der G1-Phase erfolgen. Kommt es zu einem Fehler, wird der Zellzyklus angehalten. Die weiteren wichtigen Schritte sind: (1) RNA Polymerase α (Primase) startet Replikation und (2) PCNA rekrutiert Polymerase δ, Elongation wird eingeleitet. 7.1 Spezialformen der Replikation – Organellen, Adenoviren, Retroviren Die Replikation von Mitochondrien und Plastiden sollte bidirektional sein, ist sie aber nicht. Sie replizieren asymmetrisch von zwei ARSs aus, beide Stränge separat. Ausgehend von einem ARS wird der H strand repliziert, es bildet sich ein displacement loop (D loop), der im EM sichtbar ist. Der Einzelstrang kann mit einer Nuclease, die für Einzelstränge spezifisch ist, degradiert und somit - 24 - nachgewiesen werden. Erreicht die Replikation die ARS des L strands, wird in die Gegenrichtung repliziert. Jeder Strang für sich repliziert unidirektional. Adenoviren sind lineare DNA Viren. Sie besitzen ein Protein mit Serin im aktiven Zentrum, das das 5'-terminale Cytosin bindet, die Doppelhelix geht auf und die DNA-abhängige Ad DNA Pol beginnt mit einem einzigen Nukleotid (freies 3'-OH) die Synthese. Der zweite DNA Strang bildet derweil eine Sekundärstruktur über Paarung der komplementären Enden. Die Serin-Brücke wird anschließend gespalten und das 5'-Ende mit einem Triphosphatrest geschützt, der vom Immunsystem benutzt wird (TLR-Interaktion), um die adenovirale DNA von eigener DNA zu unterscheiden (eigene hat cap-Struktur). Retroviren sind RNA Viren, die sich mittels Reverser Transkription über ein DNA-Intermediat replizieren. Die viral codierte Reverse Transkriptase übersetzt das RNA Genom bei Infektion eines Wirts in cDNA, die ins Genom der Wirts mittels Integrase integrieren und dort perisitieren kann. Aber auch diese Polymerase braucht einen Startpunkt, der Ablauf einer reversen Transkription ist folgender: 1. t-RNA des Wirts ist der Startpunkt 2. minus-Strang beginnt zu replizieren, bis Ende erreicht 3. Exonuclease legt die R-Region frei, somit wird der plus-Strang verkürzt 4. First jump ans andere Ende des plus-Strangs, Paarung mit der R-Region 5. Synthese bis Matrizenende erreicht ist 6. Entfernen der t-RNA, DNA-RNA-Hybrid liegt vor 7. Ribonuclease H degradiert RNA ineffizient, einzelne Nukleotide bleiben, dienen als Primer 8. Synthese des plus-Strangs 9. Second jump, Paarung an R-Region 10. U3 und U5 wurden jeweils dupliziert → LTRs zur Integration ins Genom - 25 - 7.2 Zusammenhalt von Metaphasenchromosomen Behandlung von Zellen mit Nocodazol hat gezeigt, dass die Schwesterchromatiden sich vorzeitig voneinander lösen, es also einen Faktor geben musste, der als Klebstoff wirkt. Cohesine verbinden Schwesterchromatiden in der Metaphase, dabei wird so lange gewartet, bis das Chromsom auf Spannung, also das Centromer an ein Mikrotubuli gebunden ist, sonst wirken die Motorproteine nicht. Nocodazol (Strukturformel) depolymerisiert Mikrotubuli, das Signal für die Motorproteine wird nie gegeben. Cohesine bestehen aus SMC1 und SMC3, Kleisine (Scc1, Scc3) schließen daraus einen Ring, der beide Chromatiden, oder zumindest einige Chromatinschleifen, umschließt. Die Tertiärstruktur und die Proteindomänen ähneln denen der Condensine. In der Anaphase bewirkt der APC (anaphase promoting complex), dass Securin, welches die Separase inhibiert, abgebaut wird. Dadurch kann Separase Scc1 abbauen, die Chromatiden können segregieren. In einem experimentellem Ansatz hat man die Cohesin-Gene unter induzierbare Promoter gestellt und in verschiedenen Stadien des Zellzyklus aktiviert. Die Fragestellung war, ob der Zusammenhalt mittels Cohesinen nach der DNA-Replikation etabliert wird. Das Ergebnis war aber, dass bereits vor der S-Phase Cohesin exprimiert werden muss, was ein Indiz dafür ist, dass die Polymerasen durch die Ringe hindurch synthetisieren. Nach welchem Mechanismus wird die Segregation reguliert? Mad2-Kinase inhibiert den APC, bis die Chromatiden auf Spannung sind (Mad-Pathway). Der APC ist eine Ubiquitin-abhängige Protease. Der dargestellte Prozess entspricht dem in der Hefe, in höheren Eukaryoten dürfte mehrere Ebenen der Regulation geben: (1) centromernahe Kohäsion (wie bisher dargestellt), (2) Polokinase, eine Aurorakinase, trennt Arme der Chromatiden. Es ist essentiell, dass sicher gestellt ist, dass die Spindel richtig verankert wird, sonst würde es zur asymmetrischen Verteilung und daher zu Segregationsdefekten und Aneuploidien kommen. Dazu gibt es eine Reihe regulatorischer Kinasen, die Aurorakinasen. Ein abweichendes Segregationsverhalten zeigen Polytänchromosomen in den Speicheldrüsen von Drosophila-Larven. Das sind Interphasechromosomen, die vergessen haben, nach der S-Phase zu segregieren (Endomitose). So entsteht ein Bündel aus 1024 Strängen mit einem Bandenmuster, das schon früh eine Genkartierung erlaubt hat. Die Folge der DNA-MAsse ist, dass viel mehr templates für die Transkription zur Verfügung stehen und enorm viel Protein synthetisiert wird. 7.3 Telomere Bei der Replikation linearer DNA ergibt sich ein Problem: Wenn das letzte Okazaki-Fragment vom lagging strand entfernt wird, kann die verbleibende Sequenz nicht 5'-3' synthetisiert werden, es bleibt ein 3'-Überhang am komlementären Strang. Im nächsten Replikationszyklus würde der nun leading strand als verkürztes template dienen, über mehrere Zyklen läuft man in eine sukzessive Verkürzung der DNA. Dies ist leider in somatischen Zellen der Fall. Warum erleidet man nicht sofort Schaden? Im Bereich der Telomere liegen hochrepetitive Elemente (TTAGGG) tausendfach hintereinander ohne codierende Funktion zu haben. Im subtelomeren Bereich befindet sich außerdem Heterochromatin. Irgendwann wären allerdings codierende Sequenzen betroffen, betroffene Zellen sterben dann. In Keimbahnzellen und leider auch Tumorzellen wirkt ein Mechanismus, der die Telomerlänge wiederherstellt. Darum ist es Therapieansatz, die Telomeraseaktivität in Tumoren zu inhibieren – allerdings befürchtet man Kollateralschäden, wenn auch epitheliale oder andere Stammzellen im adulten Menschen inhibiert werden. Die Maus kann bis zu 3 Generation vollkommen ohne Telomerase auskommen (knock out), dann treten aber - 26 - rasch Defekte und Tod ein. Die Telomerlängen sind von Spezies zu Spezies verschieden, die Maus hat zum Beispiel längere Telomere als der Mensch. Wie sieht der Mechanismus der Telomerverlängerung aus? Die humane Telomer Reverse Transkriptase (hTERT) verlängert mittels eines RNA-Templates immer den 3'-Überhang. Das RNATemplate ist recht unterschiedlich zwischen den Spezies. Für die Reparatur mittels hTERT ist eine noch vorhandene TTAGGG-Sequenz nötig, sonst ist der Schaden irreparabel. Auf Grund dersich wiederholenden Sequenz, kommt es zum slipping der hTERT, sodass mehrere Elongationszyklen den Überhang auf etwa 200 Basen erweitern. Die hTERT setzt sich aus den Unterheiten TP1 und p23 zusammen, die den Komplex stabilisieren, TERT ist der katalytische Teil. Man kennt mindestens zwei Cofaktoren, die zu TTAGGG hochaffin sind: • • TRF1 – Inhibiert die Telomeraseaktivität und reguliert somit die Telomerlänge TRF2 – Vermittelt t-loop Bildung; Verminderte Expression zeigt end-to-end Fusionen - 27 - Da auch nach Verlängerung noch ein 3'-Überhang bleibt, wurden von Organismen verschiedene Strategien entwickelt, dass durch den Einzelstrang eine ungewollte Reparatur durch einen Zellzyklus-Checkpoint ausgelöst wird. In der Hefe bindet Ku70/80 den 3'-Überhang und maskiert ihn dadurch. Fehlt Ku70/80, beobachtet man end-to-end Fusion, die jetzt dizentrischen Chromosomen werden von der Spindel gewaltsam auseinandergerissen. Telomere bilden im Menschen eine terminale schlaufenförmige Struktur, die verhindert, dass Chromosomenenden als Chromosomenbrüche erkannt werden. Man spricht vom loop model of telomer structure. Der Einzelstrang bildet einen d-loop, die Schlaufe bezeichnet man als t-loop. Die Struktur wurde allerdings nur selten beobachtet und gleicht eher einem Postulat, trotzdem hat es Eingang in die Lehrbücher gefunden. 8 – Genomorganisation Die Genomgröße korreliert nicht mit der Anzahl an Genen. Komplexere Organismen haben bei etwa gleicher Genzahl eklatant schwankende Genomgrößen, also Anzahlen an Basenpaaren (vgl. Fadenwurm ↔ Mensch ↔ Lilie). Speziell bei Pflanzen, deren Speziesgrenzen auf Grund von Polyploidien nicht klar definierbar sind, findet man deswegen extreme DNA-Mengen. Wenn die Genzahl etwa gleich ist, warum variiert dann die Genomgröße? Neben codierenden Sequenzen finden sich in Genomen eine Reihe von Elementen, die regulatorischer Natur, individueller Marker oder parasitäre Reminiszenzen sein können. 1. 2. 3. 4. 5. 6. 7. 8. Regulatorische Sequenzen Introns Non-coding RNAs: Ribosomale RNA, tRNAs, miRNAs Satelliten DNA Repetitive DNA DNA Transposons, Retrotransposons, Long interspersed elements (LINES) Short interspersed elements (SINES) Generell handelt es sich dabei um repetitive DNA, früher als junk DNA bezeichnet. Man unterscheidet zwischen hoch- und mittelrepetitiver DNA. Es gibt keine spezifischen Bereiche, in denen repititive DNA vorkommt, sie verteilt sich auf alle Chromosomen. - 28 - 8.1 Satellite DNA Unter diesem Begriff vereint man hochrepetitive DNA sowie Unterruppen der tandem repeats. Lange hat man weder zu Bedeutung noch zu Entstehung Theorien gehabt, heute weiß man, wie sie sich im Genom verbreiten: • • unequal crossing over – ändert die Zahl der Wiederholungen replication slippage – RNA Polymerase macht beim Auftreffen auf bestimmte Sequenzen vermehrt Fehler, der replizierte Strang kann seine Basenpaarung lösen, sodass die Polymerase zurückrutscht → und schon wurde ein kurzer Abschnitt dupliziert. Dadurch verändert sich das Verhältnis der G-C/A-T – Anteile im Genom, die ein spezifisches Gewicht aufweisen. In einer Dichtegradientenzentrifugation (Salzgradient: Cäsiumchlorid) kann nun der A-T-reiche Anteil (der fragmentierten Chromosomen, Anm.) als Satelliten DNA gewonnen werden (geringere Dichte, weniger Svedberg-Einheiten). Satelliten befinden sich meist an centromeren Regionen (CENP-8 boxes). Dieses Verfahren baut auf die Entdeckungen von Chargaff auf und wurde vor dem Siegeszug der Gelelektrophorese eingesetzt. VNTRs (variable number of tandem repeats) sind die Grundlage von Vaterschaftstests. VNTRs sind spezifisch für ein Individuum und ergeben einen DNA fingerprint, bestehend aus Kombinationen von Läng und Position von VNTRs im Genom (VNTRs haben 10-fach höhere Rekombinationswahrscheinlichkeit). Allerdings kann die Methode bei bottle neck Populationen, also Völkern, die sich aus wenigen Individuen entwickelt haben (nordamerikanische Ureinwohner), versagen, da die Variabilität zu gering ist (ein Merkmal kommt zB bei 2 Männern vor). 8.2 Tandem repeats Multiple copy genes haben regulatorische Funktion, da sie eine höhere Transkription erlauben, mehr RNA/Protein kann gebildet werden wegen mehr Ansatzstellen für die Polymerase. Histone und rRNA sind Beispiele, diese Gene liegen mehrfach hintereinander im Genom, da davon viel gebraucht wird. 8.3 Transposable elements Transposable Elemente machen einen Großteil des Genoms aus. Man unterscheidet zwischen DNAund RNA-Transposons: • • DNA-Transposon: Kann die Insertionsstelle wieder verlassen (non-replicative transposition) oder dupliziert sich während der Transposition (replicative transposition) RNA-(Retro-)Transposon: Verbleibt an der Insertionsstelle, integriert stets eine neue Kopie - 29 - DNA Transposons haben meist inverted repeats und führen bei Insertion zu target site duplication. Die target site wird dabei im ersten Schritt von Transposase gebunden, die einen sticky end Doppelstrangbruch einführt → die 3'-OH Reste der Host DNA werden dann mit 5'-Phosphat des Transposons ligiert, die verbleibenden Lücken vom Wirt geschlossen (braune Ellipsen). Die Verbreitung von DNA Transposons folgt meist einem excision-insertion Mechanismus, dabei verlässt das Element die Insertionsstelle und hinterlässt eine verdoppelte target site oder einen Doppelstrangbruch. Der Phage λ springt vollständig ohne target site duplication zu hinterlassen. Manchmal folgt die Verbreitung aber auch einem copy Mechanismus, bei dem nur eine Kopie an einer anderen Stelle integriert. Replikative sowie nicht-replikative Transposition benutzen das gleiche Intermediat. Es sei an Hand von Plasmiden (Mu-Phage) erklärt, da man es dabei am besten verstanden hat. Zuerst entstehen Einzelstrangbrüche (nicking), danach ligieren die Enden des Transposons ans target unter Ausbildung des strand transfer complex, eine dem Crossover ähnliche Struktur. Nun entscheidet es sich, ob der Donor herausbricht oder sich ein Cointegrat bildet, das zwei Kopien des Transposons beinhalten, bildet. Bricht der Donor, liegt das Plasmid linearisiert vor, das Transposons ist ohne Replikation gesprungen. Das Cointegrat kann mit einem weiteren Plasmid fusionieren und eine Kopie des Transposons weitergeben, das Transposon ist unter Replikation gesprungen. Transposition ist eine treibende Kraft der Evolution („parasitäre“ DNA, selfish gene), die für ein Individuum meist negative, für die Population aber in Summe positive Auswirkungen hat. Ist die Transposase auf Grund einer Mutation defekt, kann dies durch die Transposase eines anderen, mitunter nicht verwandten, Transposons kompensiert werden. Transposons können zu DNA rearrangement in Form von Verlust oder Inversion genomischer Information führen: → Verlust: Direkte repeats paaren, zirkuläre DNA verlässt das Genom → Inversion: Indirekte repeats paaren, eingeschlossene DNA wird invertiert - 30 - Die Eigenschaften von DNA-Transposons sind: • Codieren meistens eine eigene Transposase (vgl. Mais Ac-Element) • Können Deletionen in der Transposase haben (vgl. Mais Ds-Element) • Können andere Gene zwischen den LTRs tragen • Können durch eine in trans wirkende Transposase aktiviert werden • Migrieren lokal • Transposition kann präzise oder unpräzise sein • Transposition führt häufig zu target site duplication • Excision hinterlässt es einen toe-print, (Anlehnung an foot print, weil nur doppelte target site bleibt) Beispiele für DNA-Transposons sind IS1, IS2, IS4, IS5, IS10R, IS50R, IS903, alle mit Längen um die 1000 Basenpaare, inverted repeats um 9-41 bp und target sites um die 10 bp. Das Mais-Transposon kann in zwei Formen vorkommen, als activator-Element (AC) oder dissociator-Element (DS). Das Aktivatorelement ist ein typisches DNA Transposon mit 5 Exons im ORF. Dissoziatorelemente sind ähnlich, nur haben sie interne Deletionen oder sind mit Fremd-DNA kontaminiert, sodass die Transposase funktionslos ist. AC-Elemente können DS-Elemente aktivieren, da die terminalen Erkennungssquenzen erhalten bleiben. Die ursprüngliche Beobachtung von Barbara McClintock (1948) war, dass Chromosom 9 an einem DS-Locus gebrochen ist und zwar nur, wenn mit AC-Stämmen gekreuzt wurde. Nachkommen dieser Individuen zeigen verschiedene Färbung der Maiskörner: In unmittelbarer Nähe des DSLocus liegen verschiedene Gene, darunter colourless (c) bzw. pigmented (C), letzteres ist für die Färbung verantwortlich (Körner von wild wachsendem Mais sind braun). Auf Grund des Doppelstrangbruchs, hervorgerufen durch AC-aktiviertes Springen des DS-Locus, geht ein azentrisches Fragment verloren, die natürliche braune/violette Farbe wird nicht mehr exprimiert. Mitotische Expansion führt zum gefleckten Phänotyp. DS kann in pigmented inserieren und so seine Funktion stören. Körner dieses Genotyps sind dann gelb. Verlässt DS den C-Locus, bildet sich wieder die braune Farbe. Das ist eine weitere Variante, wie transposable Elemente zu einer Vielfalt an Phänotypen führen können ( → vgl. Farbmosaike bei Wildblumen, meist auf virale Infektion zurückzuführen). - 31 - Transposable Elemente eignen sich hervorragend, um Gene kaputt zu machen oder zu stören und so die Funktion in mutagenesis screens zu studieren. Der Genetiker wendet zwei approaches an: 1. loss of function: Transposon in den ORF eines Gens inserieren 2. gain of function: künstliches Transposon bringt ein neues/modifiziertes Gen mit einem starken Promoter (zB P-Element von Drosophila) Das P-Element von Drosophila ist erst vor etwa 200 Jahren in die Fruchtfliegenpopulation eingewandert, vermutlich über die Hämolymphe von Raubwespen. Eine Infektion äußert sich einem Phänomen, der hybrid dysgenesis. Man unterscheidet zwischen P- und M-Stämmen, erstere sind Wildtyp (praktisch alle Fruchtfliegen haben P-Elemente), M-Stämme sind Laborstämme und haben meist kein P-Element. P-Elemente springen nur in der Keimbahn, somatisch werden alle 4 Exons exprimiert, die Transposase funktioniert daher nicht. Kreuzt man ein M-Weibchen mit einem P-Männchen sind die Nachkommen steril (dysgene Kreuzung). Kreuzt man allerdings ein M-Weibchen mit einem P- oder M-Männchen erhält man fertile Nachkommen (reziproke Kreuzung). Warum? In der reziproken Kreuzung gibt das Weibchen über das Cytoplasma der Oocyte einen Repressor weiter, der die Transposition des P-Elements verhindert. In der dysgenen Kreuzung sammeln sich Mutationen auf Grund der ungezügelten Transposition und des fehlenden Repressors an, an Fortpflanzung können diese Fliegen nicht einmal mehr denken. Welcher molekulare Pathway liegt dem zu Grunde? Eine Sorte von siRNA, piRNA für PIWIinteracting RNA, etabliert Immunität gegen einwandernd P-Elemente im sogenannten ping-pong model for piRNA biogenesis. Endogene Kopien von inaktivierten P-Elementen sammeln sich an bestimmten Loci, die unter einem gemeinsamen Promoter stehen. Daraus entstehen antisense Transkripte, die prozessiert werden und mit PIWI, einem Argonaut-ähnlichem Protein, assoziieren. Der Komplex aus PIWI und piRNA kann nun Transposon-RNA spalten, die widerum die Spaltung der antisense Transkripte fördert – quasi ein Henne-Ei-Kreislauf (Anm. des Verfassers). Der Kreislauf produziert eine große Menge an piRNA, die mit dem Ooplasma vererbt wird – so sind Weibchen mit einem P-Element resistent gegen eindringende P-Elemente. Allerdings sind noch nicht alle Details geklärt. Der Pathway wirkt erst ab einer bestimmten Konzentration an Transposons, erst wenn 30-40 PElemente im Genom vorhanden sind wird piRNA aktiv. Wie verwendet man transposable Elemente zur Mutagenese? Ein gain of function approach ist, zwei verschiedene bakterielle Vektoren im syncytischen Stadium eines Drosphila-Embryos zu injizieren. Ein Vektor trägt ein Drosophila-Gen (ry+), der zweite, das Helferplasmid, den Selektionsmarker: In diesem Fall ein P-Element, um Transposition in späteren Generationen zu verhindern. Kosegregation des ry-Phänotyps mit einer bestimmten Mutation erlaubt Positionsbestimmung der Mutation. Flankierende Sequenzen des Inserts im Genom werden mittels inverser PCR bestimmt. 8.4 Retrotransposons Retrotransposons / RNA Transposons haben ein RNA-Intermediat im Gegensatz zu DNA-Transposons. Retrotransposons bleiben stets an der Insertionsstelle, sie inserieren Kopien, die revers aus dem RNA-Intermediat transkribiert werden. Retrotransposons und Retroviren sind strukturell und funktionell ähnlich, sie enthalten eine reverse Transkriptase, eine Integrase und LTRs, die Hüllproteine (Env) haben sie verloren. Die Replikation erfolgt wie bei Retroviren über zwei priming events ( vgl. adenovirale Replikation). - 32 - Wie hat man gefunden, dass es ein RNA-Intermediat gibt? Aus einem Plasmid mit Ty1 hat man ein Plasmid hergestellt, das einen mit Galactose induzierbaren Promoter vor Ty1 sowie ein Fragment eines anderen Gens innerhalb von Ty1 hat. Das primäre Transkript wurde in der Zelle gesplicet, danach hat man Kopien von Ty1 ohne Intron im Genom nachgewiesen. In eukaryotischen Genomen findet man drei Typen von Retrotransposons: Virale Superfamilie LINES Avirale Superfamilie Vertreter Ty1 (S. cerevisiae) Copia (D. melanogaster) L1 (Mensch) B1, B2, ID, B4 (Maus) SINES (Alu Sequenz) Pseudogene (7SL-RNA) Termini Long terminal repeats Keine repeats Keine repeats Target repeats 4-6 bp 7-21 bp 7-21 bp Enzymaktivität Reverse Transkriptase und/oder Integrase Reverse Transkriptase, Endonuklease Keine (zu wenig genetische Information) Organisation Kann Introns enthalten, werden gesplicet 1 oder 2 durchgehende ORFs Keine Introns Eigenschaften von Reverser Transkriptase: • • • • Korrekturlesefunktion 3' → 5' fehlt: Fehlerrate 10-5 Toleriert primer misalignment cDNA ist stets länger als RNA-template Trägt zur Rekombination bei Retrotransposons ohne LTRs (LINES, SINES) brauchen zur Insertion einen DNA nick. Das Retroelement codiert diese Funktion zusammen mit der reversen Transkriptase, die cis-Wirkung haben. Genprodukte von LINES fördern die Aktivierung von SINES. Das sind inaktive Transposons mit etwa 300 bp Länge, die in ihrer Funktion auf LINES angewiesen sind. LINES springen oft in aktive Gene – meist in Introns, andererseits hätte es fatale Folgen. LINES führen zu exon shuffling. Ein Gen X bestehe aus 4 Exons, zwischen Exon 3 und 4 liegt ein L1 Transposon. Die Exons 1 bis 3 werden normalerweise zu einem Protein gesplicet. Nun springt L1 zwischen Exon 2 und 3 und nimmt Exon 4 mit (read through). Das neue Protein besteht aus den Exons 1, 2 und 4, Exon 3 und 4 haben getauscht. Transposable Elemente im menschlichen Genom: LINES 1-5 kb 20.000 – 40.000 Kopien 21% des Genoms SINES 100-300 bp 1.500.000 Kopien 13% des Genoms DNA Transposons 80-3000 bp (inaktiv/aktiv) 300.000 Kopien 3% ds Genoms - 33 - 8.5 Introns Introns sind regulatorische Sequenzen, die nicht zur Codierung eines Proteins beitragen. Sie werden mittels splicing aus dem primären Transkript entfernt, sodass die prozessierte mRNA nur aus Exons besteht. Dieser Mechanismus ist eine evolutionäre Strategie, da Gene nicht ständig neu erfunden, sondern bewährte Bausteine (zB. funktionelle Proteindomänen zur Bindung von Ionen) nur als Blöcke verschoben werden müssen. Würden Organismen nur durch Punktmutationen evolvieren, wäre die Evolution bei nicht auf dem heutigen Stand. Der Trick besteht im alternative splicing. Dadurch erhält man proteomische Diversität, da eine genetische Basis gewebsspezifisch in verschiedene Proteine übersetzt wird ( → vgl. Myoglobine). Dies wird besonders in komplexen Organen (Hirn) und peipherem neuronalen Gewebe praktiziert. Die meisten Intronsequenzen folgen dem Consensus GU/AG und werden durch die snRNPs U1, U2, U4/U6 und U5 gesplicet. Eine kleine Fraktion ist durch einen AU/AC Consensus definiert (ATAC introns), diese werden durch die snRNPs U11, U12 zusammen mit U4/U6 und U5 gesplicet. U1 entspricht funktionell U11, U2 entspricht U12. Derzeit sind an die 40 U-snRNPs bekannt. Small nuclear ribonucleoproteins (snRNPs) sind Partikel aus RNA und Proteinkompnenten. Oft erfüllen RNPs katalytische Funktionen oder haben Signalaktivität mit der RNA als aktivem Teil. Beispiele für RNPs sind: • • • • snRNPs – Splicing 7SL RNPs – Teil des SRPs (signal recognition particle), verbindet Ribosomen mit rauem ER rRNA – Ribosom snoRNA- small nucleolar RNA, modifzierte RNA (Pseudouridinylierung, Methylierung) Die splice sites werden durch homologe Basenpaarung des primären Transkripts mit snRNPs definiert. Der branch point ist ein spezifisches Adenin, an dem Transesterfizierung stattfindet. Die Definition dieses speziellen Adenins ist eine enorme Leistung, das statistisch jedes vierte Nukleotid Adenin sein kann – so würde die RNA zerhackt anstatt sinnvoll prozessiert. snRNPs paaren auf 10 bp Länge, dadurch sinkt die Wahrscheinlichkeit einer AT-Mikrosequenz enorm → die splice site ist definiert. Der branch point bleibt ungepaart, das 2'-OH bleibt frei und kann das 5'-Phosphat der splice site angreifen. Biochemisch katalysiert das Spliceosom, der Komplex aus RNA und snRNPs, zwei aufeinander folgende Transesterfizierungen. Der Komplex hat 12,3 MDa und ist sogar im EM sichtbar. Das Produkt sind verbundene Exons sowie eine Lariat-Struktur. - 34 - Woher weiß man, dass die Basenpaarung essentiell ist? Man U1 mutiert, als Folge hat das splicing nicht mehr funktioniert. Dann hat man eine Supressormutation in der splice site eingebracht, dadurch wurde das Transkript wieder prozessiert. Wie werden Introns von Proteinen erkannt? Der E-Komplex bildet sich durch Interaktion beider splice sites. An der 5'-splice site bindet SF-2 (splicing factor 2) und U1. In der Nähe des branch points befindet sich eine pyrimidinreiche Region, wo U2AF bindet. SF-1/BBP verbindet U1 und U2AF und vervollständigt den E-Komplex. Die Definition von Introns kann mittels zwei verschiedene Strategien erfolgen. Je nachdem, was in größerer Menge vorliegt, wird das Kleine definiert, so wie man eher eine Oase in der Wüste definiert als das Gebiet wo Wüste ist. • intron definition – Bei großen Exons definiert SF-1 Introns durch Bindung • exon definition – Bei großen Introns legen SR-Proteine die Exons fest, der Rest ist dann Intron Wie erkennt die Zelle ordentlich gesplicete mRNA? An das Spliceosom bindet ein Protein, das nach dem Splicen als Markierung an der exon junction zurückbleibt. Hier assembliert der exon junction complex (EJC). Das Protein REF (Aly), eine Komponente des EJC, bindet an TAP/Mex, einen Exportfaktor, der fertige mRNA durch eine Kernpore transportiert. 8.6 Andere repetitive Elemente Manche zweiflügelige Insekten wie Drosophila besitzen keine Telomere, aber zwei retrotransposable Elemente ohne LTRs: HeT-A und TART. Daraus wird eine reverse Transkriptase exprimiert sowie eine non-coding RNA hergestellt. Das Primingsignal kommt vom Chromsomenende selbst, die Transposons hängen die Sequenz der RNA ans Ende an und verlängern so die Chromosomenenden. Multi copy genes sind die letzte Gruppe von repetitiven Elementen, die essentielle Transkripte liefern, wie zum Beispiel der Synthese von Ribosomen und Nucleoli. Information, die in großer Menge in der Zelle gebraucht wird, wie ribosomale RNA, ist in multi copy genes organisiert. Sie liegen in bis zu 1000 Kopien im Genom vor und sind in cluster gegliedert. Ribosomale Gene sind tandemartig organisiert, in Arabidopsis thaliana kann eine nucleolus organizing region (NOR) unterschieden werden, in der die Komponenten eines Ribosoms zu Blöcken sortiert in vielen Kopien hintereinander vorliegen. - 35 - Eine NOR kann entfernt mit einem Operon verglichen werden, da die Aktivierung des gemeinsamen Promoters einen polycistronischen 45S Precursor liefert. Daraus werden die 18S, 5.8S und 25S rRNA im Nucleolus prozessiert. Hier passiert auch der Zusammenbau der rRNA mit den in den Kern importierten Proteinkomponenten zum fertigen Ribozym, das ins Cytoplasma exportiert wird. Wie wird das sequenzspezifische Prozessieren durchgeführt? Die Prozessierung im Nucleolus ähnelt entfernt einem splicing-Mechanismus, mit der Ausnahme, dass mehrere Produkte statt einer reifen mRNA entstehen. Im Nucleolus befinden sich snoRNAs (small nucleolar RNAs), die mit rRNA sequenzspezifisch Basen paaren, daraufhin kommt es zur chemischen Modifikation der rRNA. • • C/D box snoRNA: Methylierung der rRNA H/ACA box snoRNA: Pseudouridinylierung – C5 statt N1 an die rRNA In einer Zelle können so viele Nucleoli wie Chromosomen entstehen, meistens fusionieren sie. Man hat experimentell die Transkription inhibiert, als Ergebnis haben sich die Nucleoli aufgelöst → der Nachweis, dass es sich dabei nicht um Zellorganellen handelt, sondern um Synthesekomplexe, die einen laufenden Betrieb repräsentieren. 8 – Meiose In der meiotischen Zellteilung kommt es zur Reduktion des diploiden Genoms auf einen haploiden Chromosomensatz und zur Durchmischung väterlicher und mütterlicher Allele durch Rekombination. Es läuft eine spezielle Form des mitotischen Programms ab. In einer verlängerten Prophase, die man in die verschiedenen Stadien Leptotän, Zygotän, Pachytän, Diplotän und Diakinese gliedert, kommt es zur Paarung der homologen Chromosomen und Rekombination zwischen Chromatiden. Jedes Stadium ist cytologisch und mittels molekularer Marker voneinander unterscheidbar. In der Diakinese kann theoretisch zwischen vier Chromatiden Rekombination stattfinden, zwischen - 36 - Schwesterchromatiden passiert das allerdings sehr selten (würde ja auch nichts bringen). Die grün markierten Chromatiden zeigen die Umwandlung eines Bivalents in die Kreuzform. Die Strukturen sind topologisch gleichwertig, Pachytän → Diplotän → Diakinese. Darunter sind mögliche Formen der Rekombination dargestellt: Stückaustausch zwischen (a) 2, (b) 4 und (c) 3 Chromatiden. Man unterscheidet zwei Arten von Rekombination: • • interchromosomal (intergenomisch) – zufällige Aufteilung der ♀/♂ Chromosomen intrachromosomal – Austausch zwischen homologen Chromosomen (crossing over) Liegen zwei Marker am gleichen Chromosom, sind sie zu einem gewissen Grad gekoppelt, da sie auf einer Segregationseinheit liegen (vgl. Haar- und Augenfarbe). Marker auf verschiedenen Chromosomen sind völlig unabhängig. In der Meiose erfolgen meist interchromosomale und intrachromosomale Rekombination gemeinsam. - 37 - Formen der Segregation sind an der linearen Abfolge der Ascosporen in Neurospora crassa ablesbar. Findet zwischen Centromer und Marker kein crossing over statt, kommt es zu einer 4:4 Aufteilung, man spricht von reduktionaler Segregation (links im Bild). Bei äquationaler Segregation (rechts im Bild) kommt es zu crossing over, man erhält ein 2:2:2:2 Muster. Im gesamten hat widerum 4 von jeder Sorte, nur die Anordnung ist anders (Perkins, Raju; 1995). Was geschieht auf molekularer Ebene? Spo11, ein TopoII-ähnliches Enzym, führt einen DSB ein. Die freien 5'-Enden werden von Nucleasen (Rad50, Mre11, Com1/Sae2) bearbeitet, sodass ein 3'Überhang entsteht. Dmc1/Rad51 fördern die Invasion des 3'-Überhangs in den Doppelstrang der Nachbarchromatide, ein d-loop entsteht. Jetzt können zwei Alternativen eintreten, mittels nicking entscheidet sich, ob es zur Bildung einer double holiday junction (DHJ) kommt oder nicht. Bildet sich eine DHJ, kann crossing over stattfinden. DHJs können zur Genkonversion führen, nämlich wenn durch die Nucleasen der Komplementärstrang eines Allels verloren geht und das Allel des anderen Elternteils als template zur Reparatur verwendet wird. Nach Abschluss des crossing over liegen dann nicht-komplementäre Sequenzen der Allele in einem Doppelstrang vor, mismatch repair entfernt die Information eines alten Allels und überschreibt sie mit dem neuen. In Neurospora crassa kann man eine 3:5 bzw. 2:6 Segregation beobachten. Im linken Teil des Bildes kann man einen Ascus mit einer 2:6 Aufteilung sehen. Wie wird eine holiday junction wieder aufgelöst? Die Proteine RuvA und RuvB assoziieren mit der junction, RuvC bindet an ein WTT*S Sequenzmotiv von RuvB und bildet so das Resolvasom. Der synaptonemale Komplex (SC) ist eine Proteinstruktur aus drei Komponenten (Rec8, Scp2/3, Scp1), der sich während dem Zygotän und Pachytän bildet. Er assembliert erst nach der Einführung von DSBs durch Spo11 und sorgt für Paarung der homologen Chromosomen. Man vermutet, dass er auch die Zahl möglicher crossing over reguliert (crossover interference). Chiasmata halten homologe Chromosomen ebenfalls verbunden, mitunter Jahre bis Jahrzehnte, denn in Säugern beginnt die Meiose bereits im Embryonalstadium und wird erst sukzessive beim Eisprung fortgesetzt. In der ersten meiotischen Teilung wird die Kohäsion distal von den Chiasmata gelöst. In der zweiten meiotischen Teilung wird Kohäsion in Centromernähe gelöst. Bis dahin sorgt Sgo1 - 38 - (Shugoshin, jap. für Schutzgeist) in der Hefe sowie MEI-S332 in der Fruchtfliege über Rekrutierung von Phosphatase für Zusammenhalt der Chromosomen. Hier liegt auch die Ursache für Trisomien: Der Zusammenhalt muss im Menschen 40 Jahre aufrecht erhalten werden. Im Laufe der Zeit kann die Kohäsion schwinden, auch bedingt durch geringe Rekombination und daher wenige Chiasmata, sodass es mit höherem Alter zu Missegregation kommen kann. Der Ansatzpunkt der Spindel am Kinetochor wird zwischen MeioseI und MeioseII umstrukturiert, Monopolin sorgt für die richtige Orientierung der Mikrotubuli. Das Produkt der männlichen Meiose sind 4 Spermien. Die Produkte der weiblichen Meiose sind eine Oocyte und drei Polkörperchen. PID ist in Österreich per se nicht erlaubt, daher zieht man Polkörperchen zu genetischen Untersuchungen heran, da aus ihnen kein Embryo entsteht. Oocyte und Polkörperchen enthalten unterschiedliches genetisches Material, so sind Resultate über eine monogenetische Eigenschaft stets indirekte Rückschlüsse. Polkörperchendiagnostik (PKD) setzt man besonders mit zunehmendem Alter der Mutter ein, um das Aneuploidierisiko abzuschätzen. 9 – Forward Genetics, Reverse Genetics Unter forward genetics versteht man Methoden der klassischen Genetik, man macht zuerst etwas kaputt und versucht dann zu bestimmen, was es war. Ein genetic screen folgt dem Schema: • • • Mutagenese Phänotypen isolieren Mutation/Gen im Genom finden Phänotypen, die relativ leicht erkennbar sind, können sein: asymmetrische Polarität; Polarität vertauscht, veränderte Augen oder Fühler, Blindheit, olfaktorisches Unvermögen (Drosophila). Daneben sind große Individuenzahlen erforderlich, da bei chemischer Mutagenese zumindest statistisch jedes Gen einmal betroffen sein muss, sonst kann man logischerweise nicht alle untersuchen. In der Praxis passiert das aber sowieso nicht, ein Gen wird in mehreren Individuen zerstört. Reverse genetics basiert auf bereits akkumuliertem molekularen Wissen. Angenommen man kennt ein Gen, dessen Produkt verschieden bekannte Domänen hat (Transmembrankinase). Dann sucht man im Genom nach ähnlichen Sequenzen, versucht diese Gene gezielt zu inhibieren und beobachtet anschließend die Auswirkung, beispielsweise Zellen verlieren Kontakt zu extrazellulärer Matrix. Humangenetik ist auf forward genetics angewiesen, selbtsverständlich führt man keine Mutagenese durch, aber es gibt ausreichend Individuen, sodass natürliche Mutationen auffindbar sind. 9.1 Chromosome mapping in Eukaryoten Meiotische Untersuchungen sind dafür unerlässlich, Merkmale können auf unterschiedliche Weise segregieren: • independant assortement – 2 Merkmale auf 2 verschiedenen Chromsomen • linkage – 2 Merkmale auf homologen Chromosomen, kein Austausch - 39 - • linkage – 2 Merkmale auf homologem Paar, Austausch zwischen 2 Nichtschwesterchromatiden Bei der Bestimmung der linkage ratio führt man eine Testkreuzung zwischen heterozygoten (F1 aus homozygoter Elterngeneration) und homozygot rezessiven oder X-chromosomal hemizygoten (männlichen) Individuen durch. Totale Kopplung findet in Abwesenheit von Rekombination statt, gekoppelte Gene verhalten sich wie ein einziger Locus in einer Rückkreuzung. Theoretisch gibt es so viele Kopplungsgruppen wie Chromosmen. Mit der Distanz zweier Genloci steigt die Wahrscheinlichkeit eines crossovers. Thomas Morgan und sein Schüler Sturtevant haben als erste diese Beziehung erkannt und haben bei Drosophila mit chromosome mapping begonnen. Ein Vorteil dabei war, dass in der männlichen Meiose keine Rekombination stattfindet. Sie erkannten außerdem, dass 2 Loci nur sehr selten komplett gekoppelt waren. Sie führten den Begriff des centi-Morgans (cM) ein: 1% Rekombinationswahrscheinlichkeit entspricht einer map unit und somit einem cM. Die theoretisch größte Frequenz ist 50 cM, Rekombinationsfrequenzen korrelieren nicht linear. Gene die doppelt so weit entfernt liegen, rekombinieren aber nicht doppelt so oft. Regionen mit vielen Rekombinationsereignissen nennt man hot spots. Man unterscheidet zwischen single crossovers (SCO) und double crossovers (DCO). Wozu betreibt man chromosome mapping? Den Genetiker interessieren die Position und Abfolge von Genen auf Chromosomen. Dazu wendet er three-point-mapping an. In einer Testkreuzung werden 3 Marker zueinander in Bezug gestellt. Im einfacheren Fall hat ein Elternteil nur rezessive, der andere nur dominante Merkmale. Man zählt beobachtete Phänotypen. Die häufigste Kombination stellt den parentalen Genotyp dar. SCOs geben die relative Nähe zueinander an, sagen aber nichts über die Reihenfolge aus. Dazu wertet man die DCOs aus und bestimmt, welches der drei Gene in der Mitte liegt. Zur Berechnung der cM werden die Individuenzahlen der SCOs und DCOs zweier Marker zusammengezählt und auf die Gesamtmenge der Individuen bezogen. Ein komplexeres Problem liegt vor, wenn die Verteilung der Allele (dominant/rezessiv) auf den elterlichen Chromosomen unbekannt ist. Man wählt folgende Herangehensweise: • • • NCO: Bestimmung der Allelverteilung in paternalem Gamet DCO: Bestimmung des mittigen Gens Rückschluss auf Allelkombinationen - 40 - Crossover interference beschreibt ein Phänomen, in dem ein Chiasma die Entstehung anderer Chiasmata in seiner Umgebung inhibiert. Dieser Effekt schwindet mit der Distanz. Folglich ist die Verteilung von Chiasmata nicht vollkommen zufällig. Liegen zwei Marker sehr weit entfernt, kann es passieren, dass dazwischen ein oder mehrere DCOs stattfinden, die aber nicht bemerkt werden, weil die Marker sich nicht verändert haben, also quasi parental vorliegen. Sowohl zu große als auch zu kleine Entfernung führen zu einer Diskrepanz zwischen hypothetischen und beobachteten map units. Moderne genetische Analysen benutzen SNPs (single nucleotide polymorphisms). Im Gegensatz zu phänotypischen Markern, die gut erkennbar und nicht letal sein dürfen, haben SNPs diese Nachteile nicht. Es sind individuelle Unterschiede in der Basensequenz der DNA, die meist in Introns liegen aber auch in Exons vorkommen, beispielsweise in den tRNA-Genen. Im Menschen kennt man bereits tausende SNPs. Wie weist man SNPs nach? Vorraussetzung ist die Amplifizierung von DNA Fragmenten mit PCR. 1. Restriktionsanalyse – Enzyme schneiden an spezifischen Sequenzen, befindet sich dort ein SNP, kann die restriction site nicht mehr erkannt werden, man erhält andere Banden im Gel 2. Sequenzieren – Teuer, wenn es nicht anders geht Was nützen SNPs? SNPs verwendet man zur Stammbaumanalyse generell und zum Zurückverfolgen von Erbkrankheiten, aber auch zur Lokalisierung von Mutationen. Dazu vergleicht man SNPs verschiedener Populationen (Kaukasier, Asiaten, … ), was zur Eingrenzung einer Genregion führt, die bei allen Populationen vorkommt. Haben alle untersuchten Individuen zum Beispiel ein Colon-Karzinom, so könnte in dieser Genregion eine verantwortliche Mutation liegen. Eine Sequenzierung des Bereichs gibt dann Gewissheit. Voraussetzungen für forward genetic screens sind: • • • Mutagenesefähigkeit mittels Chemikalien oder Transposons Rascher Lebenszyklus und leichte Kultivierbarkeit im Labor Kleines Genom - 41 - • • Analyse phänotypischer Marker ( vgl. Morphogene ↔ Hefe ) Transformierbarkeit ( Reversion von Mutationen ) Eigenschaften, Ziele und Methoden von reverse genetic screens sind: • • • • • Erfordert akkumuliertes molekulares Wissen Die Funktion Y von Gen X will man herausfinden Versuchsziel (das zu untersuchende Gen) sucht man sich meist in bioinformatic screens Genfunktion inhibieren → Phänotyp studieren Methoden: knock-out und knock-down (RNA-Interferenz) In Hefe führt man einen knock-out mittels homologer Rekombination durch , bei C. elegans und Drosophila wird diese Methode wenig angewandt. Ein Gen wird durch vollständiges Ersetzen oder Insertion einer Resistenzkassette inhibiert. Da das Genom bekannt ist, können Resistenzkassetten designed werden, die Homologiearme zur homologen Rekombination besitzen. Zur Herstellung der Kassetten verdaut man klonierte Gene mit Restriktionsenzymen und ligiert ein insert hinein, in diesem Fall eine Kanamycin-Resistenz. Die Transformation erfolgt direkt durch die Membran (Bakterien haben im Gegensatz spezielle Kanäle). Die freien DNA-Enden werden von der Hefe als kaputt erkannt und leiten Reparatur ein. Stolpert der Mechanismus über die Homologien im Genom, kommt es zur Rekombination, das alte Gen wird degradiert. An haploiden strains kann der Phänotyp direkt bestimmt werden. - 42 - Die Maus als diploider Organismus erfordert mehr Aufwand, weil das zweite Allel den knock-out überdeckt. Daneben funktioniert ein knock-out bei der Maus nur in embryonalen Stammzellen (ES, aus der inner cell mass der Blastocyste). Eine weitere Schwierigkeit sind die Introns und repetitiven Elemente – möchte man ein ganzes Gen ausknocken, so erreichen die Längen der Rekombinationskassetten leicht 20-22 kb Länge, was in Plasmiden nicht kloniert werden kann. Daher muss man sich Genregionen aussuchen, meist der Promoter mit 2-3 Exons. Problematisch sind auch LINES und SINES (22% des Genoms) – sie verursachen den Effekt, dass die Rekombinationskassette an vielen verschiedenen Stellen im Genom inserieren könnte. Die Rekombinationskassette enthält eine Neomycin-Resistenz sowie eine Thymidinkinase, beide zur Selektion. Thymidinkinase erzeugt bei Behandlung der Zellen mit Ganciclovir ein toxisches Produkt, als Folge sterben Zellen, bei denen die Kassette falsch inseriert hat. Neomycin im Medium lässt Zellen leben, die überhaupt rekombiniert haben. Damit ist es leider nicht getan. Die selektierten Zellen müssen nun in eine Blastocyste implantiert werden, so erhält man eine Chimäre. Tritt der Fall ein, dass die transgenen Zellen an der Keimbahn teilnehmen, kann man sie mit dominanten Individuen rückkreuzen, man erhält transgene Mäuse, die homozygot sind und endlich einen Phänotyp zeigen. Bei anderen Organismen, wo homologe Rekombination nicht oder weniger gut funktioniert, setzt man RNA interference (RNAi) ein. Es handelt sich dabei um Ausnutzung eines zelleigenen antiviralen Mechanismus, der zur Unterdrückung von Transposons konstitutiv läuft. - 43 - Doppelsträngige RNA wird dabei vom DICER in 21bp Fragmente (siRNA) gespalten. Die konstante Länge kommt durch zwei RNAse-Domänen am DICER zu Stande. Der RISC (RNA induced silencing complex) bindt siRNA und begibt sich in der Zelle auf die Suche nach homologen Sequenzen. Wird eine gefunden, degradiert das Argonaut-Protein die gefundene mRNA. Man verfolgt zwei Strategien bei der Methode: Bei niederen Eukaryoten (C. elegans) wird dsRNA unter einen induzierbaren Promoter gestellt oder über die Nahrungsaufnahme (Bakterien, die dsRNA synthetisieren) eingebracht. Bei höheren Eukaryoten muss man gleich die 21bp Fragmente einsetzen, da dsRNA viele andere zelluläre Abwehrmechanismen in Gang setzt. Verschiedene Gewebe sind unterschiedlich zugänglich für die siRNA, die Leber von Mäusen erhält über die Pfortader reichlich siRNA, das Hirn ist auf Grund der Blut-Hirn-Schranke gänzlich unzugänglich. Mit RNAi hat man in Mäusen erfolgreich Hepatitis C behandelt. Neben siRNA setzt man auch microRNA (miRNA) ein. Diese sind endogen als hairpin precursor vorhanden. Er wird von einem DICER-ähnlichen Molekül gespalten, die Fragmente sind zum target aber nur partiell homolog, sodass es nicht zur Degradierung sondern nur zur translationellen Inhibition kommt. Im Menschen sind derzeit an die 700 miRNAs bekannt, von vielen kennt man noch keine targets (orphans). Die Funktion ist fine tuning der Genexpression, da die Menge translatierbarer mRNA reguliert wird. Interessant ist der systemische Effekt, der durch einen zellulären Amplifikationsmechanismus erzeugt wird. RNA-abhängige RNA Polymerasen (RDRP) amplifizieren siRNA und miRNA, beladener RISC kann zwischen Zellen über spezielle Kanäle diffundieren. Das Phänomen wurde bei Pflanzen entdeckt, deren Blätter von einem Virus befallen waren. Behandlung eines Blattes führte zur Heilung der anderen Blätter Genome wide reverse genetic screens wurden durchgeführt oder laufen in: • • • S. cerevisiae – mutant collection, Datenbank von 15.000 Mutanten, bei manchen kennt man die Auswirkungen des knock-outs noch gar nicht C. elegans – RNAi, Identifikation augenscheinlicher Phänotypen, zB keine Neuropeptide Drosophila – läuft - 44 - Probleme, die sich Stellen: (1) Phänotypen sind nicht immer offensichtlich, (2) RNAi funktioniert nicht quantitativ, (3) knock-out essentieller Gene tötet das Individuum, bevor es studiert werden kann. 10 – Populationsgenetik Das Hardy-Weinberg-Gleichgewicht beschreibt den Zusammenhang zwischen Allelfrequenzen und Genotypfrequenzen in einer idealen Population. Kennzeichen einer idealen Population sind: • • • • • Sehr große Individuenzahl: Der zufällige Verlust eines Individuums oder Gendrift verändert praktisch nicht die Häufigkeit der Allele, was bei einer kleinen Population relativ große Auswirkungen hätte. Panmixie: Alle Paarungen, auch von Trägern verschiedener Genotypen, sind gleich wahrscheinlich und gleich erfolgreich. Es gibt keine Selektion, somit also weder Selektionsvorteile noch -nachteile für die Träger bestimmter Gene (Genotyp), die sich phänotypisch auswirken. Es finden keine Mutationen statt. Es findet keine Migration statt, die die Allelfrequenz (Genpool) verändern. Die ideale Population ist ein theoretisches Konstrukt, da in der Realität mindestens eine der Bedingungen, welche mit Ausnahme der Individuenzahlen alles Evolutionsfaktoren sind, nicht erfüllt wird. Evolution findet also stets dann statt, wenn die obigen Voraussetzungen nicht gelten. Eine Anwendung ist, ob alle möglichen Allele in einer Population adäquat auftreten oder ob positive oder negative Selektion auftritt. Das Punett-Quadrat stellt die Genotypverteilung dar, die Gleichgwichtsbeziehung berechnet sich aus: p² 2∗ p∗qq² =1 Weiblich Männlich A(p) a(q) A(p) AA(p²) Aa(pq) a(q) Aa(pq) aa(q²) Es liegen beim Menschen zwei Serotypen bezüglich des CCR5-Rezeptors vor. Dieses Oberflächenprotein ist Co-Rezeptor für das HI-Virus. Korrespondierende Genotypen zu den Phänotypen: Genotyp wt/wt wt/Δ32 Δ32/Δ32 Phänotyp Anfällig für HIV-1 Verlauf von AIDS verlangsamt Resistent gegen viele HIV-1 Stämme Obwohl Δ32 einen Selektionsvorteil bringt, kommt es nicht zu Abweichungen des Gleichgewichts, da in diesem Fall die Durchseuchung mit HIV (noch) zu gering ist, um deutliche Veränderungen zu sehen. In der obigen Tabelle ist eine exemplarische Berechnung der Allel- und Genotypfrequenzen für zwei Allele dargestellt. - 45 - Das gleiche Prinzip kann bei drei Allelen angewandt werden (p² + q² + r² + 2pq + 2pr + 2qr) , zum Beispiel bei den Blutgruppen. Phänotypisch eindeutig manifestiert sich die Blutgruppe 0 (r²). Damit kann man nun die Genotypfrequenz des zusammengesetzten Phänotyps Blutgruppe A berechnen, da Blutgruppe A durch die Allelkombinationen AA oder A0 entstehen kann. Kennt man die Frequenz rezessiver Allele, kann man die Frequenzen der anderen berechnen. 0.0004% (q²) der Bevölkerung leiden an cystischer Fibrose. Wie hoch ist der Anteil der Heterozygoten? 1. pq ²= p² 2∗p∗qq² 2. q= q² =0.02 3. pq=1 ⇒ p=1−q ⇒ p=0.98 4. 2pq =0.04⇒ 1/25 der Bevölkerung heterozygot Dieses Diagramm zeigt, dass selbst bei geringem Anteil an Betroffenen ( zB. 10%), 40% der Population Überträger sind. Homozygot rezessive Individuen werden meist negativ selektiert, Merkmale persistieren aber in den heterozygoten über viele Generationen. Je stärker selektiert wird, desto schneller nimmt die Frequenz ab, erreicht aber nie null. - 46 - Quellen Audio-Aufnahmen von ephrodite Jantsch Folien SS10 #Replicon http://www.nature.com/nrm/journal/v3/n11/images/nrm951-i1.jpg - 47 -