4 Nukleinsäuren Tag 2
Werbung

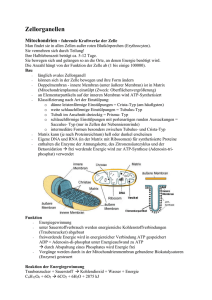
Biochemieseminar 5.4 Proteinsortierung 5.4 erstellt von H. Wolfes und J. Alves Proteinsortierung Die Proteinbiosynthese findet im eukaryontischen Cytosol statt. Dabei können die Ribosomen entweder frei vorliegen oder an das endoplasmatische Retikulum (ER) gebunden sein. Proteine, die an der Membran des ER synthetisiert werden, treten durch die Membran hindurch oder werden in sie eingebaut. Sie werden im Lumen glycosyliert oder modifiziert. Durch Vesikeltransport können sie über den Golgiapparat, in dem sie weiter modifiziert werden, zur Zellmembran oder in die Lysosomen gebracht werden. Ferner müssen Proteine aus dem Cytosol in den Kern, in die Mitochondrien und die Peroxisomen transportiert werden. Signalpeptide SRP und SRP-Rezeptor Die freien und am ER gebundenen Ribosomen sind Proteinsortierungswege in der Zelle strukturell identisch, die Lokalisation der Ribosomen hängt von den Proteinen ab, die am Ribosom synthetisiert werden. Sekretorische Proteine enthalten eine kurze Peptidsequenz aus hydrophoben Aminosäuren am N-Terminus des Proteins, die man Leadersequenz nennt. Diese kann vom Signalerkennungspartikel (SRP) erkannt und gebunden werden. Dabei wird die Translation angehalten. Das SRP ist ein Komplex aus einem RNA-Molekül, sechs Proteinuntereinheiten und einem GTP. Es wird vermutet, Kristallstruktur einer Domäne des E.coli SRP: Science 2000, 287, 1232-1239. Blau dargestellt ist die SRPRNA, rot zeigt die Phosphatreste der RNA. Das Protein ist magenta hervorgehoben mit gelben Bereichen der hydrophoben Aminosäuren in der Bindungsspalte. Rechts im Vergleich die freie Form der SRP-RNA. Die RNA verändert bei Bindung an das Protein allosterisch ihre Struktur. dass die im SRP enthaltene RNA den Zugang zur A-Stelle versperrt, indem sie an die ribosomale RNA bindet. Der Komplex aus SRP und Ribosomen diffundiert im Cytosol bis er an einen in der ER- 1 Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves Membran verankerten SRP-Rezeptor bindet. Der SRP-Rezeptor besteht aus zwei verschiedenen Polypeptidketten. Nach der Bindung wird das GTP zu GDP hydrolysiert und das SRP dissoziiert ab. Ein aus mehreren Proteinen bestehendes Translokon bindet die Leadersequenz und ermöglicht die Synthese des gesamten Proteins durch die Membran hindurch. Andocken eines Ribosoms an die ER-Membran und Weiterführen der Proteinsynthese Das Protein-Translokon kann eine inaktive und eine aktive Konformation einnehmen. Nach der Bindung des Signalpeptids geht das Translokon in die aktive Form über und zieht die wachsende Proteinkette schlaufenförmig durch die Lipid-Doppelschicht. Das Translokon ist also eine wässrige, in der Membran verankerte Pore. Nach der vollständigen Translokation wird wieder die inaktive Konformation eingenommen, der zur LipidDoppelschicht offen und damit zu den wässrigen Kompartimenten Inaktives und aktives Translokon. Die Ribosomen sind in dieser Zeichnung nicht wiedergegeben. geschlossen ist. Dies hat zur Folge, dass das hydrophobe Signalpeptid in die Doppelschicht diffundiert, wo es vom Protein 2 Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves abgespalten und später abgebaut wird. Es gibt einen verwandten, essentiellen Proteinsekretionsapparat in Bakterien, der zur Toxizität der Antibiotika Chloramphenicol und Tetracyclin beiträgt, da er vermehrt abgebaut wird, wenn ihn Ribosomen „verstopfen“, die während der Translation von zu sekretierenden Proteinen gehemmt werden. ER-Retentions-Signal Im Lumen des endoplasmatischen Retikulums assoziieren die Untereinheiten neu synthetisierter Proteine, ferner werden sie gefaltet. Proteine, die permanent im ER verweilen enthalten am C-Terminus eine kurze Aminosäuresequenz, die als Retentions-Signal dient. Viele von ihnen unterstützen die Faltung von Proteinen (Chaperone), die in das Lumen des ER synthetisiert wurden. Im Gegensatz zum Cytosol befindet sich im ER ein höher oxidiertes Milieu, sodass hier Disulfidbrücken ausgebildet werden können. Die im ER lokalisierte Protein-Disulfid-Isomerase (PDI) katalysiert die Umbildung von S-S-Brücken, sodass die stabilste Proteinkonformation gefunden werden kann. Synthese membranständiger Proteine. Bei der Synthese membranständiger Proteine läuft der Transport zum Translokon genauso ab wie bei der Synthese luminaler oder sekretorischer Proteine. Auch hier wird die Signalsequenz im Translokon erkannt und gebunden. Ist sie endständig, wird sie später abgeschnitten. Zusätzlich enthalten membranständige Proteine eine Transmembransequenz. Sie besteht aus hydrophoben Aminosäuren, die von der lipophilen Membran gut aufgenommen werden können. Das Translokon öffnet sich und gibt dann die Transmembranregion frei. Das Ribosom löst sich ab und translatiert im Zytosol weiter. Ablauf der Synthese eines membranständigen Proteins mit einer Transmembrandomäne. Kernständige Proteine In eukaryontischen Zellkernen wird die Kernmembran durch etwa 4000 Kernporen perforiert. Diese Poren besitzen eine achtfache Symmetrie, bestehen aus über 100 Proteinen und haben ein Molekulargewicht von etwa 125x106 g/mol. Die Kernpore ist ein ringförmiges Gebilde, 3 Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves das in die Kernhülle eingebettet ist und in der Mitte den „zentralen Transporter“ enthält. Die Grundstruktur bilden speichenartige Proteinuntereinheiten, Nucleoporine (NUP), die in der zentralen Ebene symmetrisch und zu beiden Seiten teils asymmetrisch aufgebaut sind. Entsprechend werden sie als die symmetrischen bzw. nucleound cytoplasmatischen NUPs bezeichnet. Im Nucleoplasma laufen die NUP Fibrillen aufeinander zu und bilden käfigförmige (körbchenförmige) Strukturen. Der Durchmesser des offenen Kanals kann zwischen 9 und 26 nm variieren. Es wird vermutet, dass es sich um einen blendenartigen Verschluss handelt. Kleine Proteine werden per Struktur der Kernpore Diffusion durch die Kernporen in den Kern transportiert. Größere Proteine, die vom Cytoplasma in den Kern gelangen sollen, enthalten ein Signal zur nukleären Lokalisation. Das „nuclear localization signal (NLS) ist eine Sequenz von 4-8 meist positiv geladenen (Lys, Arg) Aminosäuren, die sich auf der Oberfläche des Proteins befinden und auch doppelt im Abstand von 10 Aminosäuren vorliegen können. Die NLS des Proteins wird von Importin α erkannt und gebunden. Der Komplex wird durch die Kernpore transportiert. Im Zellkern assoziiert das G-Protein Ran in seiner GTP-Form, die hier durch ein GDP-Austauschprotein eingestellt wird, an Importin β, sodass Importin α und das eingeschleuste Protein freigesetzt werden. Importin α und der Importin β-Ran(GTP)-Komplex werden Import von Proteinen in den Zellkern mit der Kontrolle durch das G-Protein Ran 4 aus dem Kern geschleust. Hier Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves wird das GTP hydrolysiert, und Ran(GDP) lässt Importin α und β für die Bindung eines neuen NLS-Proteins frei. Die NLS ist hinreichend für den Transport in den Kern. Wird ein Protein, das üblicherweise im Cytosol lokalisiert ist, mit einer NLS exprimiert, wird dieses Protein in den Kern importiert. NLS können auch durch ein gebundenes Protein maskiert sein. Dies verhindert den Kernimport. Beispielsweise liegt der GlucocorticoidTranslokation des Glucocorticoidrezeptors in den Zellkern nach Stimulation der Zelle mit Glucocorticoid Rezeptor in Abwesenheit des Hormons gebunden an das Chaperon Hsp90 im Cytoplasma vor. In diesem Komplex ist die NLS nicht zugänglich. In die Zelle diffundiertes Glucocorticoid bindet an den Rezeptor. Dadurch wird der Komplex mit Hsp90 aufgelöst. Die dann zugängliche NLS befähigt den Rezeptor durch eine Kernpore geschleust zu werden. Er bindet an spezifische DNA-Sequenzen und reguliert die Expression Glucocorticoid-abhängiger Gene. Proteintransport in die Mitochondrien Mitochondrien enthalten 1000 – 2000 Proteine, von denen die allermeisten im Kern kodiert sind und im Cytosol synthetisiert werden. In den Membranen der Mitochondrien gibt es keine Poren, die ein nativ gefaltetes Protein passieren lassen könnten, da gerade die innere mitochondrielle Membran sogar für kleine Ionen sehr undurchlässig sein muss. Es sind bisher 5 Wege charakterisiert worden, wie die Proteine je nach ihren speziellen Anforderungen in die verschiedenen Kompartimente und Membranen der Mitochondrien gebracht werden. Für den Hauptweg (Weg 1 in der Abbildung), der vor allem die vielen Matrixproteine transportiert, haben diese eine N-terminale Signalsequenz, die eine amphiphile α-Helix mit einer stark positiv geladenen und einer hydrophoben Seite bilden kann. Dieses Signal bindet an den TOM40 (transport through outer membrane)-Komplex und leitet sie durch die Membran an den TIM23 (transport through inner membrane)-Komplex weiter. Auch durch diesen Komplex wird das Signalpeptid unter Ausnutzung des Membranpotentials bis auf die Innenseite der inneren Membran (negativ) gebracht, wo es durch Chaperone gebunden wird. Der ganze Prozess erfordert auch auf der cytosolischen Seite die Hilfe von Chaperonen, da das Protein entfaltet werden muss, um durch die Poren von TOM und TIM hindurch zu passen. Unter 5 Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves Import von Proteinen in die Mitochondrien ATP-Verbrauch wird in der mitochondriellen Matrix das Protein mit Hilfe von Chaperonen gefaltet und das Signalpeptid wird abgespalten. Die schnelle Bindung an Chaperone im Mitochondrium verhindert ein „Zurückrutschen“ des Proteins ins Cytosol. Einige der Proteine haben auch eine zweite interne, hydrophobe Signalsequenz. Sie werden von TIM nicht vollständig in die Matrix entlassen, sondern in die innere mitochondrielle Membran sortiert. Eine ganze Reihe wichtiger Membranproteine der Mitochondrien sind so hydrophob, dass sie sich nicht im Cytosol falten können. Sie werden deshalb von Chaperonen im ungefalteten Zustand gehalten und zum Mitochondrium gebracht. Sie haben dafür Signalsequenzen an verschiedenen Stellen der Primärsequenz, die bisher nur schlecht charakterisiert sind. Nachdem sie TOM passiert haben, werden sie im Intermembranraum von Chaperonen gebunden und zum TIM22-Komplex der inneren Membran (Weg 2) oder TOB/SAMKomplex in der äußeren Membran (Weg 3) geleitet, von denen sie eingebaut werden. Proteine für den Intermembranraum werden von TOM an den TIM40-Komplex weiter geleitet (Weg 4), der sie oxidiert indem er ihre Faltung durch Disulfidbrücken stabilisiert. Die dabei abgezogenen Elektronen werden über Cytochrom c in die Atmungskette eingeschleust. Die dreizehn in den Mitochondrien codierten Proteine sind Untereinheiten der Komplexe der Atmungskette und werden direkt von den mitochondriellen Ribosomen an OXA in der inneren Membran abgegeben und gefaltet (Weg 5). 6 Biochemieseminar 5.4 Proteinsortierung erstellt von H. Wolfes und J. Alves In die Peroxisomen werden Proteine und sogar Proteinkomplexe in fertig gefaltetem Zustand hineintransportiert. Zwar sind die Membranen relativ durchlässig für die Substrate der peroxisomalen Enzyme, der Mechanismus des Transports über die Membran ist aber noch weitgehend unverstanden. Klar ist nur, dass zwei Rezeptorproteine die peroxisomalen Proteine an spezifischen, kurzen Signalsequenzen im Cytosol erkennen und zu den Peroxisomen bringen. Zusammen mit weiteren membranständigen Proteinen findet dann die Translokation statt. Grundlegende Literatur: Löffler, Basiswissen Biochemie, 7. Auflage S. 277-279 Löffler Petrides Heinrich, Biochemie & Pathobiochemie, 8. Auflage S. 187-190, 301307 Rassow Hauser Netzker Deutzmann, Biochemie, 3. Auflage S. 359-360, 365-372, 470 Themen, die im Vortrag angesprochen werden sollten: Synthese von Proteinen am rauen endoplasmatischen Retikulum • SRP • SRP-Rezeptor • Translokator • Protein-Disulfid-Isomerase • Membranständige Proteine Kerntransport • NLS • Importin • Ran Proteintransport in die Mitochondrien • TOM-Komplex • TIM-Komplexe 7