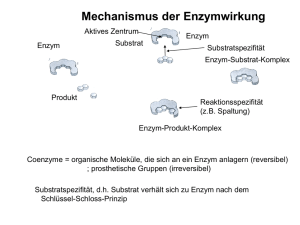
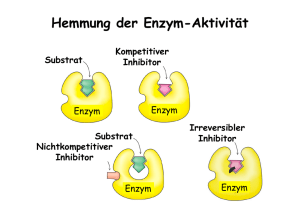
1. Eigenschaften von Enzymen 2. Aktivationsenergie 3. Katalytische
Werbung

Enzyme 1. Eigenschaften von Enzymen 2. Aktivationsenergie 3. Katalytische Mechanismen 4. Lysozym 5. Serin Proteasen 6. Reaktionskinetik 7. Enzym Inhibition 8. Kontrolle der Enzymaktivität Enzyme agieren schnell und präzise Einführung 1. Enzyme katalisieren eine grosse Bandbreite von chemischen Reaktionen, welche in der Zelle ablaufen 2. Enzymologie, Studium der Enzyme (eingeführt 1878; Greek: en, in; zyme, Hefe), Fermentation: Glucose -> Ethanol 12 enzym-katalysierte Schritte 3. James Summer, 1926, Kristallierung der Urease aus Jackbohne; gezeigt, dass es sich um ein Protein handelt 4. Es gibt auch andere Katalysten, zB. Ribozyme (Peptidbinung; “RNA-world”), nur aus 4 Basen 5. Proteine sind vielfältiger, 20 funktionelle Bausteine Einführung (2) Enzyme beschleunigen die Rate von chemischen Reaktionen in dem sie die freie Energiebarriere, welche Reaktanden und Produkte trennt, erniedrigen 1) Generelle Eigenschaften von Enzymen Lernziele: 1) Verstehen, dass sich Enzyme von normalen chemischen Katalysatoren unterscheiden in der Reaktions-Rate, -Bedingungen, -Spezifität und -Kontrolle 2) Verstehen der molekularen Grundlage der Stereound Geometrischen-Spezifität von Enzymen 3) Verstehen der Funktion und Arten von enzymatischen Kofaktoren Eigenschaften von Enzymen Enzyme unterscheiden sich von chemischen Katalysatoren: - Höhere Reaktionsraten, 106-1012 Reaktionen/sec - Mildere Reaktionsbedingungen (Temp, pH, …) - Höhere Reaktionsspezifität (keine Seitenprodukte) - Können reguliert werden Definition Katalysators: beschleunigt die chemische Reaktion ohne dabei selber verbraucht zu werden Klassifizierung von Enzymen - Namensgebung: -ase, Urease, Alkohol Dehydrogenase, aber keine festen Regeln - Systematisch: 6 Klassen, je nach Art der chemischen Reaktion, die katalysiert wird (http://expasy.org/enzyme/) Enzyme binden Substrate spezifische - Enzyme binden Ihr Substrat durch nicht-kovalente Kräfte: van der Waals-, elektrostatische-, hydrophobe-Interaktionen, Wasserstoffbrücken - Geometrische Komplementarität - Elektrostatische Komplementarität - “Induced fit” bei Substratbindung - “Schloss-Schlüssel” Modell (Emil Fischer) Enzyme sind stereospezifisch - Enzyme sind sehr spezifisch sowohl bei der Bindung von chiralen Substraten als auch bei der Katalyse von stereospezifischen Reaktionen - Enzyme sind selbst chiral, L-Aminosäuren -> Aktives Zentrum = Aktive Stelle/Site ist asymmetrisch / stereo selektiv Die Stereospezifität der Substratbindung Einige Enzyme benötigen Kofaktoren - Kofaktoren sind kleinmolekulare Verbindungen, welche als “chemische Zähne” agieren können um Reaktionen zu übernehmen, welche von Seitenketten der Aminosäuren nicht erbracht werden können - Müssen mit der Nahrung aufgenommen werden, zB. alle Vitamine - Metallionen: Cu2+, Fe3+, Zn2+ toxisch: Cd2+ und Hg2+ können Zn2+ ersetzen und das Enzym inaktivieren - Organische Moleküle, Koenzyme, können temporär als Kosubstrat mit dem Enzym assoziieren, zB. NicotinamidAdenin-Dinucleotide (NAD+) (B3) Klassen von enzymatischen Kofaktoren zB. Cu2+, Fe3+, Zn2+ zB. NAD+ zB. Häm, Biotin (B7) Struktur und Reaktion von (Vit B3) + NAD NAD+ ist ein obligater Kofaktor der Alkohol Dehydrogenase (ADH) Reaktion NADH dissoziiert nach der Reaktion vom Enzym und muss in einer unabhängigen Reaktion re-oxidiert werden. Erst dann ist der Reaktionszyklus abgeschlossen Prosthetische Gruppen Sind permanent mit dem Enzym assoziiert (≠ Kosubstrate), zB. Häm Gruppe in Hämo- oder Myoglobin Holoenzyme = Enzym + Kofaktor Komplex, aktiv Apoenzyme = Enzym ohne Kofaktor, inaktiv 2) Aktivierungsenergie und die Reaktionskoordinate Lernziele: 1) Verstehen, dass das Enzym die freie Energie entlang des Reaktionsweges ändert, nicht jedoch die Energiebilanz der Gesamtreaktion Der Übergangszustand Theorie des Übergangszustandes: ~1930s HA-HB + HC -> HA + HB-HC Übergangszustand: HA--HB--HC Übergangszustand = Zustand der höchsten freien Energie = unstabilster Zwischenzustand Die Reaktanden nähern sich entlang eines Weges tiefster freier Energie = Reaktionskoordinate Diagramm des Übergangszustandes / Reaktionskoordinate: Blot der freien Energie versus der Reaktionskoordinate Diagramm des Übergangszustandes (Symmetrisch) Übergangszustand Substrat Produkt Diagramm des Übergangszustandes (Asymmetrisch, exotherm) Freie Energie der Aktivierung Freie Energie der Reaktion Aktivierungsenergie und die Reaktionskoordinate Je grösser die Energie, welche benötigt wird um den Übergangszustand zu erreichen, desto langsamer, unwahrscheinlicher die Reaktion Wenn die freie Energie der Reaktion negativ ist, ∆G < 0, so ist die Reaktion spontan und setzt Energie frei (exotherm). Die Geschwindgkeit/Wahrscheinlichkeit der Reaktion hängt aber weiterhin von der Grösse der Aktivierungenergie ab Diagramm der Übergangszustände einer zwei-Schritt Reaktion Schrittlimitierend Geschwindigkeitsbestimmend Erster Schritt ist limitierend Zweiter Schritt ist limitierend “Flaschenhals” Katalysatoren reduzieren die freie Energie der Aktivierung, ∆G‡ Der Katalyst ermöglicht einen Reaktionsweg, dessen freie Energie des Übergangszustandes gegenüber der nicht katalysierten Reaktion reduziert ist Katalysatoren reduzieren die freie Energie der Aktivierung, ∆G‡ Die Reaktionsrate ist proportional zu e-∆G‡/RT ∆∆G‡ von 5.7 kJ/mol (1/2 Wasserstoffbrücke) erhöht die Reaktionsrate 10-fach ∆∆G‡ von 34 kJ/mol (Bruchteil einer kovalenten Bindung) erhöht die Reaktionsrate 106-fach Beachte: der Katalysator beschleunigt sowohl die Vorwärts- als auch die Rückreaktion, Nur das ∆GReaktion bestimmt, ob die Reaktion vorwärts oder rückwärts abläuft 3) Katalytische Mechanismen Lernziele: 1) Verständnis der chemischen Grundlage von SäureBase Katalyse, kovalenter Katalyse und Metallionen Katalyse 2) Verstehen, wie Enzyme Reaktionen beschleunigen durch Proximitäts- und Orientierungseffekte sowie durch Bindung des Übergangszustandes Katalytische Mechanismen Wir lernen enzymatische Reaktionsmechanismen zu verstehen, indem wir die entsprechenden nicht-enzymatischen Reaktionen von Modellverbindungen anschauen. Gebogene Pfeile repräsentieren Elektronenpaare Die chemischen Regeln müssen dabei allzeit respektiert werden, dh. nie 5 Substituenten auf einem C, oder 2 auf H etc. Typen von katalytischen Mechanismen a) Säure-Base Katalyse b) Kovalente Katalyse c) Metallionen Katalyse d) Proximitäts- und Orientierungs Effekte e) Präferenzielles Binden des Übergangszustandes In Enzymen werden oft mehrere dieser Mechanismen zur tatsächlichen Katalyse kombiniert 3A) Säure-Base Katalyse geschieht mittels Protonentransfer Generelle Säurekatalyse: Protonentransfer von einer Säure erniedrigt die freie Energie des Übergangszustandes einer Reaktion Bsp. Keto-Enol Tautomerisierung (a), beschleunigt durch Protonentransfer (b) oder Protonenabstraktion (c) (dh. generelle Basenkatalyse) Wenn sowohl Säuren wie auch Basen vorhanden wären, würde diese Beispielreaktion noch weiter beschleunigt: Koordinierte Säure-Base Katalyse Base-catal. Acid-catal. Non-catalyzed Koordinierte Säure-Basen Katalyse In einem Enzym können Asp, Glu, His, Cys, Tyr, Lys als Protonendonor oder Akzeptoren agieren und damit Säure-Base Katalyse durchführen Die 3D-Struktur eines Enzymes erlaubt ihm mehrere katalytische Gruppen strategisch um das Substrat zu positionieren, um damit koordinierte Säure-Base Reaktionen zu katalysieren. Dies ist daher ein häufig anzutreffender katalytischer Mechanismus Der pH beeinflusst die Enzym-Aktivität Die meisten Enzyme sind nur in einem engen pH-Bereich aktiv ca. 5-9 Reaktionsrate zeigt Normalverteilung in Abhängigkeit des pH-Wertes, widerspiegelt Ionisationsgrad von katalytisch wichtigen Resten Das pH-Optimum gibt Informationen über katalytisch wichtige Reste: wenn bei 4/5 -> Glu, Asp; 6 -> His; 10 -> Lys pK Wert dieser Reste im Protein variiert je nach chemischer Mikroumgebung um +/- 2 Einheiten RNase A ist ein Säure-Base Katalyst Rinder Pankreas RNase A: Verdauungsenzym, welches vom Pankreas in den Dünndarm sekretiert wird Ein 2’,3’ zyklisches Nukleotid wurde als Reaktionsintermediat isoliert Die pH-Abhängigkeit der Reaktion deutet auf 2 wichtige Histidine hin (Position 12 und 119), welche als koordinierte Säure-Base Katalysten agieren und eine 2-Schritt Reaktion beuschleunigen Der RNase A Mechanismus 2-Schritt Reaktion: a) His 12 agiert als generelle Base, und löst damit den nukeophilen Angriff des 2’ O auf P aus. b) His 119 agiert als generelle Säure und protoniert die Abgangsgruppe Danach kommt Wasser in das aktive Zentrum, das 2’,3’-zyklische Intermediat wird hydrolisiert durch eine Umkehrung des ersten Schrittes, wobei His 12 jetzt als generelle Säure und His 119 als generelle Base agieren 3B) Kovalente Katalyse wird meist durch einen nukleophilen Angriff ausgelöst Kovalente Katalysten beschleunigen die Reaktion, indem sie temporär eine Bindung zwischen Katalyst und Substrat eingehen Normalerweise greift dabei eine nukleophile Gruppe des Enzyms eine elektrophile Gruppe des Substrates an = nucleophile Katalyse Bsp: Decarboxylation von Acetoacetat Decarboxylation von Acetoacetat Katalysiert durch primäre Amine, RNH2 Wichtige nukleophile und elektrophile Gruppen Gruppen in Enzymen Substrate Wichtige Aspekte der kovalenten Katalyse Je stabiler die gebildete kovalente Bindung zwischen Enzym und Substrat, desto schwieriger ist es, sie wieder aufzulösen (im letzten Schritt der Reaktion) Daher müssen gute kovalente Katalysten: (i) starke Nucleophile sein und gleichzeitig (ii) eine gute Abgangsgruppe bilden. Die ist der Fall für Imidazol und Thiol-Gruppen in His and Cys. Daneben können auch Asp, Ser, und einige Coenzyme (Thiamin Pyrophosphate, Pyridoxal Phosphate) als nukleophile in der kovalenten Katalyse wirken 3C) Metall Ionen als Katalysten 1/3 aller bekannten Enzyme brauchen Metallionen für die Katalyse Metalloenzyme enthalten stark gebundene Metallionen: Fe2+, Fe3+, Cu2+, Mn2+, Co2+ Na+, K+, und Ca2+ spielen strukturelle Funktionen und nehmen nicht an der Katalyse teil Mg2+, Zn2+ können sowohl strukturelle als auch katalytische Funktionen haben Beteilligung von Metallionen an der Katalyse 1. Metallionen können Substrate binden und sie damit für die Reaktion optimal orientieren 2. Metallionen können Oxidations-Reduktion Reaktionen durchführen, indem sie ihren Oxidationsstatus ändern, zB Fe2+, Fe3+ 3. Metallionen können elektrostatisch stabilisieren und negative Ladungen abschirmen Metallionen wirken oftmals wie ein Proton und können Wasser polarisieren und damit Hydroxylionen (OH-) produzieren Die Funktion von Zn2+ der Karbonischen Anhydrase CO2 + H2O <-> HCO3- + H+ Zn2+ polarisiert Wasser, welches dann CO2 angreift 3D) Katalyse kann auf Proximitäts- und Orientierungs-Effekten beruhen Enzyme sind meist sehr viel grösser als ihre Substrate Durch eine orientierte Bindung und Immobilisierung des Substrates können Enzyme Reaktionen auf 4 Arten beschleunigen: 1. Substrat wird in der Nähe der katalytischen Gruppen gebunden 2. Substrat wird in der richtigen Orientierung gebunden (bis zu 100-fache Beschleunigung) 3. Stabilisierung des Übergangszustandes durch elektrostatische Interaktionen 4. Einfrieren von Translations- und Rotations-Bewegungen des Substrates (up to 107-fold) Inter- versus intramolekulare Reaktion gelbe Farbe 24-fach schneller durch räumliche Nähe Beispiel für das sterische Einfrieren 315-fach schneller wenn R = CH3 gegenüber R = H 3E) Enzyme katalysieren Reaktionen durch präferenzielles Binden des Übergangszustandes Ein Enzym kann den Übergangszustand einer Reaktion stärker binden als das Substrat oder das Produkt der Reaktion. Diese Stabilisierung des Übergangszustandes, zusammen mit der produktiven räumlichen Orientierung der aktiven Gruppen, kann die hohe Effizienz der enzymatischen Reaktionen erklären. Bsp. wenn ein Enzym den Übergangszustand mit 34.2 kJ/mol (= 2 Wasserstoffbrücken) stabilisiert, wird dadurch die Reaktion 106-fach beschleunigt. Präferentielles Binden des Übergangszustandes nicht-katalysiert katalysiert Analoge des Übergangszustandes sind effiziente Enzym-Inhibitoren Bsp. Prolin Racemase Inhibitoren 160-fach grössere Affinität 4) Lysozym Lernziele: 1) Verständnis der Substrat-Bindung an Lysozym 2) Verstehen des katalytischen Mechanismus von Lysozym Lysozym Lysozym ist ein Ezym, welches die bakterielle Zellwand abbaut. Lysozym ist weitverbreitet und wirkt bakterizid (zB. Tränen-, Nasenflüssigkeit. Das Hühner-Eiweiss Enzym ist das am besten charakterisierte (HEW), 14.3 kD, einzelnes 129 AS Polypeptid, 4 Disulfidbrücken, Katalyserate 108-fach Hydrolisiert β(1->4) Glykosidische Bindung zwischen NAcetylmuraminsäure (NAM) und N-Acetylglukosamin (NAG) in Peptidoglycanen der Zellwand Hydrolysiert auch Chitin: β(1->4) NAG Die Schnittstelle von Lysozym β(1->4) • Die Röntgenstruktur von Lysozym als erstem Enyzm wurde 1965 gelöst • Ellipsoidale Form mit prominentem Spalt über der gesamten Oberfläche • Bindet 6 Saccharid-Einheit, A-F, binden in dem Spalt, • Spaltung zwischen D/E Einheit (4/5) Die katalytische Stelle von Lysozym wurde durch Modellbau identifiziert Lysozym geht eine kovalente Bindung mit dem Substrat ein Transition state analog inhibition of lysozyme NAG lactone binds to the D subside with about 9.2 kJ/mol greater affinity than does NAG 5) Serin-Proteasen Lernziele: 1) Verstehen, wie Serin-Proteasen die Peptidbindung spalten und wie sie dabei den Übergangszustand stabilisieren 2) Verstehen, dass viele Proteasen als inaktive Vorläufer synthetisiert werden (Zymogene, ProEnzyme) Serin-Proteasen • Grosse Klasse von proteolytischen Enzymen • Sehr reaktives Ser in aktiver Stelle ( ≠ cut after Ser ! ) - Verdauungsenzyme - Wichtige Enzyme in der Embryonalentwicklung - Blutgerinnung - Entzündung - Viele weitere zelluläre Prozesse • Konzentration auf Chymotrypsin, Trypsin, Elastase Alle drei sind Verdauungsenzyme, welche vom Pankreas gebildet und in den Dünndarm sekretiert werden. Trypsin, Chymotrypsin und Elastase haben Substrat-Spezifizität Alle drei Ser-Proteasen spalten Peptide, aber mit unterschiedlicher SubstratSpezifität: Chymotrypsin spaltet nach grossen, hydrophoben AS Trypsin nach positiv-geladenen AS (Lys, Arg) Elastase nach kleinen neutralen AS => haben unterschiedliche Substrat-Bindetaschen Diisopropylphosphofluoridat (DIPF) inaktiviert Ser-Proteasen DIPF reagiert nur mit Ser 195 von Chymotrypsin, sehr toxisch. Markiert aber keine anderen Serine im Enzym - Weshalb ? 5B) Die Röntgenstruktur enthält Information über die Katalyse, Substratspezifität und Evolution dieser Ser-Proteasen • Chymotrypsin, Trypsin, haben ca. 240 AS, 40% Sequenz-Identität • Alle haben ein reaktives Ser und ein wichtiges His • Sehr ähnliche 3D Struktur, Chymotrypsin gelöst 1967 • Aktive Stelle: His 57, Ser 195, Asp 102 bilden eine katalytische Triade, sind unter sich vernetzt mittels Wasserstoffbrücken Die AS in der aktiven Seite von Chymotrypsin His 57, Ser 195, Asp 102 bilden eine katalytische Triade, sind unter sich vernetzt mittels Wasserstoffbrücken 5C) Der katalytische Mechanismus von Serin-Proteasen • Aufklärung des katalytischen Mechanismus basierend auf der Struktur von Chymotrypsin • Mechanimus gilt für alle Ser-Proteasen sowie für viele andere hydrolytische Enzyme wie zB. Lipasen, Hydrolasen (generell Esterasen) • Da Enzyme sowohl die Vorwärts- wie die Rückreaktion katalysieren, können sie auch zur Synthese, dh. Verknüpfung von Esterbindungen und nicht nur für deren Spaltung eingesetzt werden Formation of the tetrahedral intermediate 1. After chymotrypsin has bound substrate: Ser 195 nucleophilic attack on peptide’s carbonyl group to form tetrahedral intermediate, resembles transition state of this covalent catalysis, - Proton on Ser is absorbed by His 57 to form imidazolium ion (general base catalysis), aided by Asp 102 2. Decomposition of the tetrahedral intermediate • Decomposition to the acylenzyme intermediate and scission of the peptide bond • Driven by donation of proton from N3 of His 57 (general acid catalysis) • Helped by polarizing effect of Asp 102 on His 57 (electrostatic catalysis) 3. Amine leaving group is replaced by water • The amine leaving group (the new N-terminus of the cleaved peptide) is released from the enzyme and replaced by water from the solvent 4. Hydrolysis of the acylenzyme intermediate By the addition of water, formation of a second tetrahedral intermediate 5. Reversal of step 1 Yields the carboxylate product, that is the new Cterminus of the peptide, and regenerates the active enzyme Der tetrahedrale Übergangszustand wird vom Enzym durch die Bereitstellung eines Oxyanion Loches vorweggenommen und stabilisiert mit Substratbindung tetrahedraler Üz Diese präferentielle Stabilisierung des tetrahedralen Üz ist verantwortlich für die katalytische Effizienz 5D) Proteasen werden als inaktive Vorläufer, den Zymogenen, synthetisiert • Proteolytische Enzyme werden oft in Form von inaktiven Vorläufern, den Proenzymen bzw Zymogenen, synthetisiert (haben N-termiale Extensionen). • Akute Pankreatitis ist durch die prämature Aktivierung von Verdauungsenzymen charakterisiert. • Enteropeptidase konvertiert Trypsinogen zu Trypsin, ebenfalls eine Ser-Protease, welche aber hormonell kontrolliert wird. Die Aktivierung von Trypsinogen zu Trypsin Die Blut Koagulationskaskade • Bei Verletzung der Blutgefässe, werden diese verschlossen durch Aggregation von Platelet-Zellen (Thrombozyten, kleine, kernlose Zellen) und Ausbildung eines unlöslichen Netzwerkes von Fibrin, welches weiteres Austreten von Blutzellen verhindert. • Fibrin wird vom löslichen und zirkulierenden Fibrinogen gebildet durch Aktivierung der SerProtease Thrombin. • Thrombin ist das letzte Enzym in der KoagulationsKaskade, Aktivierung geschieht auf Platelets. Die Blutkoagulations Kaskade Nervengifte DIPF und andere ähnliche Organophosphat-Verbindungen agieren als potente Nervengifte Inaktivieren die Acetylcholinesterase (Ser in aktiver Seite) => keine Inaktivierung des Nervenimpluses, Spasma tetrahedrales Phosphat = Übergangszustand Analog Tokyo Subway Terror Attack, 1995 Eigenschaften von Enzymen 6. Reaktions-Kinetik 7. Enzym Inhibition 8. Kontrolle der Enzym-Aktivität 6) Reaktions Kinetik Lernziele: 1) Verstehen, dass Reaktionsgleichungen den Verlauf von Reaktionen erster Ordnung und solcher zweiter Ordnung beschreiben 2) Verstehen, was die Michaelis-Menten Konstante beschreibt und was die Gleichung über die Initialgeschwindigkeit und die maximale Geschwindigkeit aussagt 3) Verstehen, wie der Lineweaver-Burk Blot verwendet wird, um KM und Vmax zu berechnen Reaktions-Kinetik Der Verlauf chemischer Reaktionen kann durch Reaktionsgleichungen beschrieben werden Untersuchung der Rate von enzymatischen Reaktionen = Enzym-Kinetik kann Aufschlüsse über den Mechanismus der Reaktion liefern A -> P Sequenz von Elementarreaktionen, mit Intermediaten A -> I1 -> I2 -> P Die Beschreibung jeder einzelnen Elementarreaktion führt zu einer mechanistischen Beschreibung der Gesamtreaktion Die Ordnung einer Reaktion gibt an wieviele Moleküle an einer Elementarreaktion teilnehmen Für eine Unimolekulare Reaktion: A -> P, ist die Bildung von P gleich der Abnahme von A und dies entspricht der Geschwindigkeit (v) der Reaktion Die Proportionalitäts-Konstante ist die Raten-Konstante, k Die Geschwindigkeit zu einer gegebenen Zeit ist also proportional zur Konzentration von A = Reaktion erster Ordnung; Einheit k (sec-1), v (Mol/sec) Bsp. Radioaktiver Zerfall, k entspricht der Halbwertszeit Bimolekulare Reaktion: A + B -> P ist eine Reaktion zweiter Ordnung Einheiten von k (Mol-1 sec-1), v (Mol/sec) B) Enzym Kinetik wird durch die MichaelisMenten Beziehung beschrieben • Die Reaktionsrate und Effizienz von Enyzmen kann gemessen werden • 1902 β-Fructofuranosidase: Sucrose + H2O -> Glukose + Fructose • Falls [Sucrose] >> [Enzyme] • k1 k2 E + S <-> ES -> P + E k -1 K2 ist schrittbestimmend; wir nehmen an, die Reaktion sei irreversibel Der Verlauf einer Enzym-katalysierten Reaktion Steady state Die Michaelis-Menten Beziehung setzt voraus, dass ES konstant ist = steady-state Die Michaelis-Menten Gleichung beschreibt die Rate einer enzymatischen Reaktion als Funktion der Substartkonzentration Bildung des Produktes ausgehend von ES ist eine Reaktion erster Ordnung Steady-State Annahme: [S] >> [E] -> [S], [E] und [ES] bleiben konstant Blot der Geschwindigkeit (vo) einer enzymatischen Reaktion versus Substratkonzentration [S] Die Michaelis Konstante beschreibt die Substratkonzentration, bei der das Enzym mit halb-maximaler Geschwindigkeit arbeitet Michelis Konstante KM is defined as: Die Maximale Geschwindigkeit: Vmax = k2 [E]T Die Initial-Geschwindigkeit: Michelis-Menten Gleichung: (bevor 10% des Substrates umgesetzt wurde) Die Michaelis Konstante ist ein Mass für die Affinität des Enzyms zum Substrat Bei der Substrat Konzentration bei der [S] = KM ; vo = Vmax/2 KM entspricht daher der Substratkonzentration bei der das Enzym mit halb-maximaler Geschwindigkeit arbeitet Falls ein Enzym ein kleines KM hat, erreicht es eine hohe Geschwindigkeit bereits bei tiefer Substratkonzentration KM hängt ab vom pH, der Temperatur und entspricht der Affinität des Enzymes zum Substrat Die katalytische Konstante Kcat entspricht der Umsatzzahl Die katalytische Konstante eines Enzyme ist definiert als: auch bekannt als Umsatzzahl (turnover number), entspricht der Anzahl Reaktionen, die ein Enzym pro Sekunde katalysiert Für einfache Reaktionen: kcat = k2 Beachte: kcat ist eine Konstante, aber Vmax hängt ab von der Menge an Enzym in der Reaktion Kcat/KM beschreibt die Effizienz eines Enzyms Umsatzzahl dividiert durch Affinität zum Substrat (gross/sehr klein -> sehr gross) Die effizientesten Enzyme haben Kcat/KM Werte im Bereich 108-109 M-1 sec-1 Dh. diese Enzyme katalysieren die Umsetzung vom Substrat jedesmal, wenn sie ein Substrat bindet und werden daher durch die Diffusion des Substrates zum Enzym oder des Produktes weg vom Enzym limitiert. Sind also katalytisch perfekt ! C) Kinetische Messungen erbringen Werte für Vmax und KM • Vmax ist aus einer hyperbolen Kurve von vo gegen [S] nur schwierig zu bestimmen, da der Wert nur asymptotisch erreicht wird • In der Praxis werden die Werte von Vmax und KM daher aus Doppel-Reziprok Blots (Lineweaver-Burk) herausgelesen • Doppel-Reziprok der Michaelis-Menten Gleichung • Lineare Gleichung Die Doppel-Reziprok Darstellung: Lineweaver-Burk 7) Enzym Inhibition Lernziele: 1) Verstehen, dass ein kompetitiver, nichtkompetitiver und ein gemischter Inhibitor mit dem Enzym oder dem Enzym-Substratkomplex interagiert und dadurch die KM und Vmax Werte des Enzyms ändert Enzym-Hemmung • Viele kleinmolekulare Substanzen verändern die Aktivität eines Enzyms indem sie das Enzym binden und dadurch die Substratbindung oder den katalytischen Umsatz hemmen • Substanzen, welche die Aktivität eines Enzyms reduzieren, werden als Inhibitoren/Hemmstoffe bezeichnet • Pharmazeutisch wichtige Hemmstoffe, zB. gegen AIDS, hemmen virale Enzyme (DNA/RNA Polymerasen) Klassen von Inhibition Irreversible Inhibitoren: Inaktivieren das Enzym, Bsp Reagentien, welche AS in der aktiven Stelle kovalent modifizieren (DIPF, Aspirin) Reversible Inhibitoren: Können strukturell dem Substrat ähnlich sein, aber nicht umgesetzt werden: (i) kompetitiv (ii) unkompetitiv (iii) gemischte Inhibitoren/nicht-kompetitiv A) Kompetitive Inhibitoren binden an die Substrat-Bindestelle des Enzyms Der kompetitive Inhibitor kompetitiert mit dem normalen Substrat um die Bindung an das Enzym. Bindet an die Substrat-Bindestelle Der kompetitive Inhibitor ähnelt daher dem Substratoder Produkt, kann aber vom Enzym nicht umgesetzt werden Bsp. Hemmung der Succinat Dehydrogenase durch Malonat Produkt-Inhibition • Da enzymatische Reaktionen in beide Richtungen ablaufen können, kann natürlich auch das Produkt an die “SubstratBindestelle” binden und dadurch das Enzym inhibieren. • Viele metabolische Enzyme sind Produktinhibiert (feed-back inhibition) Analoge des Übergangszustandes sind speziell effiziente Inhibitoren, da sie mit höherer Affinität an das Enzym binden als das Substrat: Bsp. Adenosin Deaminase KM is 3 10-5 M Analog des Übergangszustandes KI of 1.5 10-13 M KI 3 10-4 M Die Stärke der kompetitiven Inhibition hängt davon ab, welcher Anteil des Enzyms den Inhibitor gebunden hat Generelles Modell der kompetitiven Inhibition Der kompetitive Inhibitor reduziert daher die Konzentration an aktivem Enzym: [E]T = [E] + [ES] + [EI] Michaelis-Menten Die Beziehung wird durch einen Faktor α ergänzt α ist eine Funktion der Konzentration des Inhibitors und seiner Affinität für das Enzym Beziehung von vo versus [S] in Gegenwart verschiedener Konzentrationen eines kompetitiven Inhibitors Lineweaver-Burk Blot eines kompetitiv gehemmten Enzymes Note intersection at 1/Vmax -> comp Inh Ein kompetitiver Inhibitor kann durch Erhöhung der Substratkonzentration neutralisiert werden In Gegenwart von [I] scheint [S] kleiner zu sein als es wirklich ist α kann daher als Faktor verstanden werden, durch den [S] erhöht werden muss, um die Inhibition zu neutralisieren Wenn [S] unendlich gross wird, erreicht vo das Vmax (unabhängig von [I]) Ein kompetitiver Inhibitor ändert daher die Umsatzzahl des Enzyms nicht ! Bsp. Methanol-Vergiftung • Wird durch Verabreichung von Ethanol behandelt – Weshalb ? • Methanol wird in der Leber durch die Alkoholdehydrogenase zu Formaldehyd umgewandelt. Formaldehyd ist sehr toxisch (führt zu Blindheit, Tod) • Ethanol kompetitiert mit Methanol um die Bindung zur Alkoholdehydrogenase, dies reduziert die Bildung von Formaldehyd • Der Körper hat daher mehr Zeit, um Methanol auszuscheiden Alkohol Dehydrogenase B) Ein unkompetitiver Inhibitior bindet den Enzym-Substrate-Komplex und nicht die Substratbindestelle des Enzyms, wie dies der kompetitive Inhibitor macht Lineweaver-Burk Blot eines unkompetitiven Inhibitors Note that all lines have identical slope of KM/Vmax Unkompetitive Inhibition kann daher nicht einfach durch höhere Substratkonzentration neutralisiert werden Nichtkompetitive Inhibitoren brauchen auch nicht dem Substrat zu gleichen. Aber seine Bindung an den ES-Komplex verändert die aktive Stelle des Enzyms so, dass es katalytisch unwirksam wird C) Gemischte Inhibitoren binden sowohl das freie Enzym wie auch den ES-Komplex Mixed Inhibition = Nicht-Kompetitive Inhibition, Spezialfall: Rein Nicht-kompetitiv, KI = KI’ Lineweaver-Burk Blot eines gemischten Inhibitors Im Spezialfall des rein nicht-kompetitiven Inhibitors schneiden sich die Geraden bei –1/KM KM ändert daher in dem Fall nicht HIV Enzym-Inhibitoren • Das humane HIV Virus führt zum acquired immunodeficiency syndrome (AIDS), in dem es das Immunsystem des Wirtes zerstört • Das virale RNA Genom dieses Retrovirus wird durch die Reverse Transkriptase in DNA umgeschrieben und dann in das Wirtsgenom integriert • Virale Proteine werden als lange Vorläufer synthetisiert und diese Polyproteine werden dann durch die HIV Protease in aktive Proteine geschnitten • Keine effiziente Impfung ! => Inhibitoren Inhibitoren der ReversenTranskriptase Archetyp ist AZT, welches in DNA eingebaut wird, aber dann die Kettenverlängerung verhindert, da es keine 3’ OH Gruppe besitzt. Zelleigene Polymerasen haben tiefere Affinitäten für die Nukleosid Analoge als die Reverse-Transkriptase Inhibitoren der HIV Protease Die HIV Protease ist ein Homodimer von 99 Resten und gehört mechanistisch zu den Aspartat Proteasen (wie Pepsin, gastrische Proteasen, welche bei tiefem pH aktiv sind) Spaltet Phe-Pro oder Tyr-Pro, peptidomimetische Inhibitoren: Ritonavir, Saquinavir enthalten grosse Gruppen, welche die aktive Stelle binden und dem tetrahedralen Übergangszustand ähnlich sind. Inhibitoren der HIV Protease HIV Inhibitoren • Beide Klassen von Inhibitoren haben Nebeneffekte und müssen mehrmals täglich während vielen Jahren, eingenommen werden. • Nucleoside Analoge zB. beeinträchtigen die Replikation des Genoms von sich schnell teilenden Zellen, zB blutbildende Stammzellen im Knochenmark • Ausbildung von Resistenz gegen diese Inhibitoren limitiert deren Effektivität, schnelles Auftreten von mutierten Formen des Virus • Keine “magic bullet drugs”, kombinatorische Therapie 3) Kontrolle der Enzym-Aktivität Metabolische Prozesse müssen koordiniert werden indem die entsprechenden Enzyme reguliert werden. 2 Arten der Kontrolle: 1. Kontrolle der Menge an Enzym: abhängig von Synthese und Abbau, kann innerhalb von Minuten (Bakterien) bzw. Stunden (Eukaryonten) reguliert werden. 2. Kontrolle der Enzym-Aktivität: kann über allosterische Effektoren (Bsp. Hämoglobin, ACTase) und kovalente Modifikationen (Phosphorylation) reguliert werden. A) Allosterische Kontrolle Allosterische Kontrolle der Aspartat Transcarbamoylase (ATCase) Erster und ratenlimitierender Schritt der Pyrimidin Synthese Beide Substrate binden Kooperativ an das Enzym Feedback Inhibition der ATCase reguliert die Pyrimidin Synthese Allosterische Hemmung der ATCase durch Cytidin Triphosphat (CTP), ein Pyrimidin (Feedback Hemmung) Allosterische Aktivation durch Adenosin Triphosphat (ATP), ein Purin Nucleotid => Purin/Pyrimid Verhältnis Kinetik der ACTase Reaktion Sigmoidale anstelle der hyperbolischen Kurve -> kooperative Bindung der beiden Substrate Die allosterischen Effektoren, ATP und CTP, verschieben die Kurve nach links bzw. rechts Regulation der ACTase Aktivität Durch die beschriebene Feedback Hemmung und die allosterische Aktivierung kann die ACTase die Synthese der Purine und Pyrimidin Nukleotide koordinieren. Ein ausgewogenes Verhältnis von Purin und Pyrmidinen wird für die Replikation und Transkription verwendet und ist wichtig, um die Fehlerrate der DNA Polymerase bei der Replikation tief zu halten. Allosterische Bindungen ändern die Substratbindungsstelle der ACTase • ACTase (300 kD), zusammengesetzt c6r6 Untereinheiten, katalytisch (c) und regulatorisch (r) • Katalytische Untereinheit als set von 2 Trimeren angeordnet 2(c3), komplexiert mit 3 Sets regulatorischen Dimeren 3(r2) • Jedes regulatorische Dimer kontaktiert zwei katalytische Untereinheiten, die jeweils in unterschiedlichen C3 Trimeren liegen • Isolierte katalytische Trimere sind aktiv und nicht allosterisch reguliert durch ATP oder CTP • Die regulatorischen Untereinheiten reduzieren die Aktivität der katalytischen Untereinheit Struktur der ACTase 2x katalytische c3 Trimere 3x regulatorische r2 Dimere CTP Bindung zu r2 Dimer Änderungen in der tertiär und quarternär Struktur der ACTase kein Substrat tiefe Affinität T Zustand Substrat Bindung induziert T -> R Transition Allosterische Effektoren verändern die Substrat-Bindung der ACTase • ATP bindet präferentiell den R-Zustand der ATCase (R, hohe Substrat-Affinität) -> friert damit die hohe Substrataffinität des Enzyms ein • CTP als Hemmer bindet dagegen den T-Zustand des Enzyms (T, tiefe Substrat-Affinität) -> stabilisiert den tief affinen Zustand • T -> R Transition durch konformationelle Änderungen, hauptsächlich des Quarternärzustandes • Substrat-Bindung an eine der Untereinheiten erhöht daher die Substrataffinität der benachbarten Untereinheiten Allosterische Änderungen in anderen Enzymen gleichen denen in Hämoglobin und der ACTase • Allosterische Enzyme kommen oftmals vor und katalysieren strategisch wichtige Reaktionen • sind meistens symmetrisch aufgebaut, mit mindestens zwei Untereinheiten, über welche Änderungen der Quarternärstruktur kommuniziert werden und damit die Substratbindung der benachbarten Untereinheit verändert wird • Quarternäre Änderungen sind meistens Rotationsbewegungen der Untereinheiten gegeneinander • Die Sekundärstruktur ist bei T -> R Transitionen meist nicht betroffen B) Kontrolle der Enzymaktivität durch kovalente Modifikationen, Phosphorylation • Zusätzlich zur allosterischen Regulation werden viele Enzyme durch kovalente Modifikationen reguliert • Die häufigste dieser kovalenten Modifikationen ist die Phosphorylierung/Dephosphorylierung von bestimmten Aminosäuren im Protein:Thr, Ser, Tyr (etwa 30% aller humanen Proteine werden phosphoryliert) • Durch Protein Kinasen, bzw. Protein Phosphatasen Ein Phosphorylierungs- / Dephosphorylierungs-Zyklus Thr, Ser, Tyr Thr-P, Ser-P, Tyr-P Beispiel: Glykogen Phosphorylase • Katalysiert Phosphorolyse (Bindungsspaltung durch Substitution mit einer Phosphatgruppe) von Glykogen α(1->4)-verbundene Glukose zu Glukose-1-Phosphat (G1P) Glykogen + Pi <-> Glykogen + G1P (n residues) (n-1 residues) • Schrittmacher Enzym der Glykolyse Struktur der Muskel Phosphorylase Homo-Dimer von 842 AS Phosphorylierte Form, aktiv, Phosphorylase a (Ser14-P) Dephospho Form, Phosphorylase b - Aktiviert durch AMP (=wenig Energie) - Inhibiert durch ATP, G1P Phosphorylierung und Dephosphorylierung verändert die enzymatische Aktivität und gleicht einer allosterischen Kontrolle • Die Glykogen Phosphorylase hat zwei konformationelle Zustände R (Aktiv), T (Inaktiv, Substrat Zugang blockiert) • Phosphorylation von Ser 14 induziert T -> R Übergang und die R Form, kann nun durch allosterische Effektoren wie zB. AMP reguliert werden • ATP und Glucose-6-Phosphat (G6P) binden den T Zustand der Phosphorylase b und inaktivieren das Enzym Conformationelle Wechsel der Glykogen Phosphorylase Inaktiv Aktiv Ser 14, AMP Phosphorylase b Phosphorylase a Kontrolle der Glykogen Phosphorylase