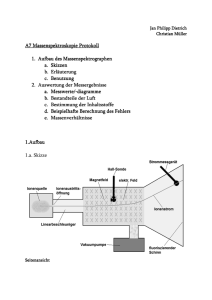
Massenspektrometrische Ermittlung der Proteintargets von
Werbung

Massenspektrometrische Ermittlung der
Proteintargets von zytotoxischen Metallkomplexen
in Blutserum und E. coli-Zellen
Dissertation
Zur Erlangung des Grades eines
Doktors der Naturwissenschaften
der Fakultät für Chemie
an der Ruhr-Universität Bochum
vorgelegt von
Joanna Will
Bochum 2008
Moim rodzicom
Die vorliegende Arbeit wurde in der Zeit von Juli 2005 bis Mai 2008 am Lehrstuhl für
Analytische Chemie der Ruhr-Universität Bochum erstellt.
An dieser Stelle danke ich ganz besonders Herrn Prof. W. S. Sheldrick, PhD, für die
interessante Themenstellung, ständige, gewinnbringende Diskussionsbereitschaft, seine
Begeisterungsfähigkeit und den mir gewährten Freiraum bei der Durchführung dieser Arbeit.
I
Inhaltsverzeichnis
II Abkürzungen/Aminosäuren
1
Einleitung
1
2
Problemstellung
5
3
Kenntnisstand
7
3.1
Platinverbindungen
7
3.2
Rutheniumverbindungen
18
3.3
Methoden
21
4
5
6
Reaktionen von metallhaltigen Zytostatika mit Peptiden
26
4.1
Chromatographische Modellstudien
26
4.2
Massenspektrometrische Studien
35
4.2.1 Modellstudien mit [(η6-p-Cymol)RuCl2(DMSO)]
37
4.2.2 Modelluntersuchung mit [Pt(dien)(H2O)]2+
40
4.2.3 Modellstudien mit cis-[PtCl2(NH3)2]
42
4.2.4 Schlussfolgerungen für LC2/ESI-MS2-Analysen
46
Proteintargets in Escherichia coli
48
5.1
Proteintargets von [(η6-p-Cymol)RuCl2(DMSO)]
49
5.2
Proteintargets von [Pt(dien)(H2O)](NO3)2
61
5.3
Proteintargets von cis-[PtCl2(NH3)2]
70
5.4
Diskussion
99
5.4.1 Wechselwirkung von [(η6-p-Cymol)RuCl2(DMSO)] mit E. coli-Zellen
100
5.4.2 Wechselwirkungen von Platinkomplexen mit E. coli-Zellen
105
Serumproteine
114
6.1.
117
Cisplatinaddukte des Serumalbumins
6.1.1 Blutserumproben
117
6.1.2 Serumalbumin:Cisplatin Proben
126
6.2
136
Cisplatinaddukte des Serotransferrins
6.2.1 Blutserumproben
136
6.2.2 Serotransferrin:Cisplatin Proben
143
6.3
154
Cisplatinaddukte des Apolipoproteins A1
6.3.1 Blutserumproben
154
7
6.3.2 Apolipoprotein A1:Cisplatin Proben
158
6.4
Cisplatinaddukte des Alpha-1-Antitrypsins
161
6.5
Cisplatinsddukte des Alpha-2-Macroglobulins
165
6.6
Cisplatinaddukte des Apolipoproteins A2
169
6.7
Cisplatinaddukte weiterer abundanter Proteine
172
6.8
Diskussion
173
Proteintargets in humanem und bakteriellem Thioredoxin
183
7.1
Proteintargets in humanem Thioredoxin
185
7.2
Proteintargets in Thioredoxin E. coli
188
7.3
Vergleich von humanem und E. coli Thioredoxin
194
8
Proteintargets in Cyclooxygenase-2
195
9
Zusammenfassung
200
10 Experimenteller Teil
206
10.1
Ausgangssubstanzen
206
10.2
Biologische Proben
207
10.3
Massenspektrometrische Untersuchungen
208
10.4
Chromatographische Untersuchungen
212
10.5
Kernresonanzspektroskopie
212
11 Anhang
214
12 Literatur
220
II. Abkürzungen
(k)Da
(kilo)Dalton
2DE
zweidimensionale Gelelektrophorese
5´-GMP2-
5´-Guanosinmonophosphat
Ac
Acetyl
bpy
2,2-Bipyridin
Cisplatin
cis-Diammindichloroplatin(II)
Cymol
4-Methylisopropylbenzol
dien
2,2'-Diaminodiethylamin
DMSO
Dimethylsulfoxid
DNA
Desoxyribonukleinsäure
en
Ethylendiamin
ESI
Electrospray Ionization
HPLC
High pressure/performance liquid chromatography
HSAB
Hard Soft Acid Base
HSQC
Heteronuclear Single Quantum Correlation
HT-29
Humane Kolonadenkarzinomzelllinie
IC
Inhibitory concentration
ICAT
Isotope-Coded Affinity Tags
ICP
Inductively Coupled Plasma
IP
Ionenpaar
LC
Flüssigkeitschromatographie
MALDI
Matrix Unterstützte Laser Desorption/Ionisation
MCF-7
Humane Mammakarzinomzelllinie
MS/MS
Tandem-Massenspektrometrie
MudPIT
Multidimensionale-Protein-Identifizierungs-Technologie
NAMI-A
ImH[trans-RuCl4(DMSO)Im]), (New Antitumor Metastasis Inhibitor)
NMR
Kernresonanz
PAGE
Polyacrylamid-Gelelektrophorese
RAPTA
Ruthenium(II)-Aren-PTA (PTA = 1.3.5-Triaza-7-phosphaadamantan)
RP
reversed-phase
SDS
Natriumdodecylsulfat
TOF
Flugzeit, Time-of-flight
Allgemeine Erläuterungen
Angaben zu den stöchiometrischen Verhältnissen (z. B. 1:1-Ansatz) beziehen sich immer auf
das Stoffmengenverhältnis von Metallkomplex zu Ligand.
Die in dieser Arbeit angegebenen Prozente zur Eluentenzusammensetzung sind als
Volumenprozente zu verstehen.
Aminosäuren
Einleitung
1
1. Einleitung
Die im normalen Sprachgebrauch als Krebs bezeichnete Krankheit umfasst eine Vielzahl von
Formen und verwandten Erkrankungen, bei denen Körperzellen unkontrolliert wachsen, sich
teilen und gesundes Gewebe zerstören können. Aus dem unkontrolliert wachsenden Gewebe
kann sich ein Tumor entwickeln. Dabei gibt es zwei Formen: benigne und maligne Tumoren.
Benigne Tumore sind durch lokal begrenztes Wachstum gekennzeichnet, Zellen maligner
Tumore hingegen können sich aus dem Zellverband des Tumors lösen und mit dem Blutstrom
oder durch das lymphatische System in andere Körperteile gelangen. So können dort dann
Metastasen entstehen. Krebs ist mit 10,9 Millionen Neuerkrankungen jährlich [1] nach wie
vor die zweithäufigste Todesursache weltweit, nach Herz-Kreislauferkrankungen. Die
folgende Grafik stellt die prozentuale Verteilung der krebsbedingten Sterbefälle ausgewählter
Tumorarten nach Geschlecht unterteilt dar.
Abb.1.1: Prozentualer Anteil ausgewählter Tumorlokalisationen aus allen Krebssterbefällen in
Deutschland 2004 [2]
Einleitung
2
Zur Behandlung von Krebs stehen den Medizinern immer leistungsfähigere diagnostische
Systeme und ein umfangreiches Arsenal an Methoden zur Verfügung. Ein Beispiel stellt die
Hyperthermie dar, die auf gezielter lokaler oder einheitlicher Überwärmung beruht und die
Temperaturempfindlichkeit von Tumorzellen ausnutzt. Bei der Strahlentherapie wird der
Tumor Röntgen- oder Gammastrahlung ausgesetzt, wodurch die erkrankten Zellen geschädigt
werden und absterben. Des Weiteren wird sehr häufig ein operativer Eingriff zur Entfernung
der Tumoren praktiziert, der jedoch nicht selten nur eingeschränkt möglich ist, da entartetes
Gewebe von gesundem nicht exakt abgegrenzt werden kann. Eine wichtige Rolle spielt die
medikamentöse Behandlungsweise, die Chemotherapie.
Mit der zufälligen Beobachtung ROSENBERGS und seiner Mitarbeiter vor über 40 Jahren, dass
Hydrolyseprodukte einer Platinelektrode die Zellteilung von Escherichia coli hemmten [3],
entwickelte sich daraus folgend, nach einer Reihe von Untersuchungen Cisplatin mit der
Zulassung in den USA 1979 zum bedeutendsten Zytostatikum [4-6]. Weitere intensive
Forschungsarbeit, die seit über vier Jahrzehnten andauert, beschäftigt sich mit der
Entwicklung neuer für die gesunden Zellen weniger schädlicher [7] Verbindungen.
Andererseits sind aber auch die Resistenzentwicklung und nicht auf alle Krebsarten
erweiternde Verwendung Gegenstand vieler Arbeiten. Trotz des hohen Aufwandes sind
bislang nur zwei Folgeverbindungen, Carbo- und Oxaliplatin, weltweit als Zytostatika
zugelassen worden, während weitere wie Nedaplatin regional beschränkt eingesetzt werden.
Cisplatin besitzt eine sehr gute Wirksamkeit bei Hodenkarzinomen, Ovarialtumoren, Tumoren
im Kopf-Hals-Bereich und Blasentumoren. Weniger wirksam bis unwirksam ist Cisplatin bei
Tumoren des Magen-Darm-Traktes, vor allem bei Kolonkarzinomen, sowie Tumoren der
Lunge [8-14].
Nicht zuletzt aus diesem Grund richten sich immer mehr Arbeiten auf die Entwicklung
nichtplatinhaltiger Metallkomplexe. Die bekanntesten Beispiele sind das von KEPPLER et al.
synthetisierte KP1019 (Indazolium trans-[tetrachlorobis(1H-indazol)ruthenat(III)]) [15-19]
und das von SAVA et al. synthetisierte, gegen Metastasen aktive NAMI-A (New Antitumor
Metastasis Inhibitor) (Imidazolium [trans-Imidazoldimethylsulfoxidtetrachlororuthenat(III)],
ImH[trans-RuCl4(DMSO)Im]) [20-23]. Ebenso zu erwähnen sind die von DYSON
beschriebenen Komplexe der RAPTA-Klasse, wobei RAPTA für Ruthenium(II)-Aren-PTA
(mit PTA = 1.3.5-Triaza-7-phosphaadamantan) steht [24-27].
Neben den genannten Platin- und Rutheniumverbindungen erwiesen sich weitere
Übergangsmetallkomplexe, wie z. B. das Titanocendichlorid im Zellexperiment ebenfalls als
wirksam gegen Brust-, Lungen- und Darmkarzinome [28]. Außer dem oben erwähnten
Einleitung
3
Metallocenkomplex zeigten diverse Ferroceniumsalze positive Resultate an verschiedenen
Tumorzelllinien.
Daneben
wurden
als
weitere
Substanzklasse auch
Metall-Alkin-
Verbindungen [29, 30] mehrfach untersucht. Cobalt-Alkin-Komplexe, z.B. Co-ASS
(Hexacarbonyl[2-propyn-1-ylacetylsalicylat]dicobalt), weisen hohe zytotoxische Wirksamkeit
bei Tests an humanen Brustkrebs- und Leukämiezelllinien (z. B. für die humane MCF-7Brustkrebszelllinie gilt für Co-ASS ein IC50-Wert von 1,4 µM) auf [31-33].
Studien, die sich mit der Aufklärung der Wirkungsmechanismen der Antitumormittel
beschäftigen, zeigten für Cisplatin, dass eine Koordination der Hydrolyseprodukte des
Cisplatins an die DNA für den zytotoxischen Effekt in den Zellen verantwortlich ist. Ferner
ist bekannt, dass die Koordination an die DNA eine Hinderung der Replikation und
Transkription bewirkt. Cisplatin kann verschiedene Arten von cross-links innerhalb der DNA
ausbilden, am häufigsten 1,2-intrastrand cross-links, die zu einer Verkrümmung der DNA
führen. Diese wird wiederum durch verschiedene Proteine, unter anderem die HMG-Proteine
(high mobility group) erkannt, die ihrerseits eine Reparatur der veränderten DNA-Regionen
ausführen, andererseits eine solche Reparatur aber auch verhindern können. In der Literatur
wird gar die Verursachung der Apoptose durch diese Proteinreaktionen diskutiert [34, 35].
Eine schematische Darstellung der möglichen DNA-Cisplatin-Addukte findet sich in der
folgenden Abbildung.
Abb.1.2.: Hauptaddukte aus DNA und Cisplatin: (a) interstrandcross-link, (b)
1,2-intrastrandcross-link, (c) 1,3-intrastrandcrosslink, (d) Protein-DNA-cross-link [35]
Einleitung
4
Die Wechselwirkung des Metallkomplexes mit der DNA ist zwar für den therapeutischen
Effekt Cisplatins verantwortlich, jedoch reagiert nur ein geringer Teil des eingesetzten
Zytostatikums tatsächlich mit dieser. Der Großteil (65-98% [36, 37]) des intravenös
verabreichten Krebstherapeutikums bindet hauptsächlich an Proteine. Diese eigentlichen
Hauptreaktionen des Cisplatins, die für einen Teil der Toxizität verantwortlich gemacht
werden, sind bislang deutlich weniger erforscht als die Reaktionen mit der DNA und sind
deswegen Gegenstand vieler aktueller Studien.
Problemstellung
5
2. Problemstellung
Die Zielsetzung der Arbeit ist es, einen Beitrag zur Aufklärung der Reaktionen von
Einzelproteinen
bzw.
Proteinen
in
lebenden
Zellsystemen
und
Blutserum
mit
antitumorwirksamen Metallkomplexen zu leisten. Hierbei soll untersucht werden, ob durch
die Kopplung einer ein- oder zweidimensionalen flüssigchromatographischen Trennung mit
der ESI-Tandem-Massenspektrometrie grundsätzlich die Möglichkeit besteht Proteintargets
bzw. Protein-Bindungsstellen von Metallkomplexen in komplexen Proteingemischen zu
bestimmen.
Hierfür
werden
die
Modellverbindungen
[Pt(dien)(H2O)](NO3)2
und
[(η6-p-
Cymol)RuCl2(DMSO)] sowie die zytotoxischen Komplexe Hexacarbonyl[2-propyn-1-ylacetylsalicylat]dicobalt (Co-ASS) und Cisplatin untersucht.
Zu Beginn der Arbeit soll gezeigt werden, welche Voraussetzungen für eine erfolgreiche
„bottom-up“ LC2/MS2 –Analyse von Gemischen von Metall-Protein-Komplexen erfüllt
werden müssen. Hierbei muss zunächst geprüft werden, ob bestimmte Metallfragmente an
Peptidsequenzen koordiniert bleiben:
a) während des enzymatischen Verdaus der Proteinaddukte sowie
b) während der chromatographischen Trennung der entstandenen tryptischen Peptide
unter Verwendung von reversed phase- und SCX-Säulen.
Dies gilt insbesondere für die kinetisch bevorzugten Bindungsstellen an O- und SDonoratome, die möglicherweise beim Verdau bzw. bei der chromatographischen Trennung
zu Gunsten von thermodynamisch begünstigten His- und Cys-Resten aufgegeben werden
könnten. Gelingt diese Trennung, muss außerdem anschließend festgestellt werden, ob die
metallierten Peptidionen:
a) massenspektrometrisch detektiert werden können und ob
b) die so erhaltenen gasförmigen Ionen anschließend durch CID-MS zu metallierten
Ionen der b+- und y+-Reihen fragmentiert werden können.
Schließlich muss die Eignung der bisher zur Identifikation von posttranslationalen
Modifikationen verwendeten Software der massenspektrometrischen Proteomics für die
zuverlässige Bestimmung von Metallierungstellen in Proteintargets überprüft werden.
In Voruntersuchungen werden deswegen chromatographische und vor allem ESI-MS2 –
Studien von wässrigen Lösungen aus Modellpeptiden mit den jeweiligen Metallkomplexen
durchgeführt.
Problemstellung
6
Die Proteintargets des Cisplatins und des Komplexes [(η6-p-Cymol)RuCl2(DMSO)] in dem
Bakterium E. coli sollen aufgeklärt werden. Das Darmbakterium wird als einfaches Modell
für andere Zellsysteme eingesetzt, vor allem um die Vorgänge in komplexen humanen Zellen
besser zu verstehen. Es gilt sowohl die Proteintargets als auch die spezifischen
Bindungsstellen der Metallkomplexe in dem Bakterium durch Tandem-Massenspektrometrie
zu ermitteln. Außerdem sollen die Cisplatin-Bindungsstellen in den wichtigsten
Serumproteinen bestimmt werden.
Zwecks Validierung und Überprüfung der Ergebnisse für Blutserumproben werden auch die
abundanten Proteine Albumin, Serotransferrin, Alpha-2-Macroglobulin, Apolipoprotein A1
und Alpha-1-Antitrypsin einzeln untersucht. Ein Vergleich der Bindungsstellen des Cisplatins
in zwei Proteinen aus unterschiedlichen Organismen (hier bakteriell und human), den
Thioredoxinen wird angestrebt. Zusätzlich sollen Bindungsstellen des potentiellen
Zytostatikums Co-ASS in der Cocylooxygenase-2 ermittelt werden.
Die Messungen werden sowohl mit der LC-ESI-MS/MS-Methode als auch mit Hilfe der
MudPIT- Technologie (Multidimensional Protein Identification Technology) erfolgen.
Hierbei sollen die Möglichkeiten und Einschränkungen der Methode zur Bestimmung von
Proteintargets allgemein durchleuchtet werden.
Es soll außerdem geklärt werden, ob die Metallkomplexe bestimmte Aminosäurenreste als
Bindungsstellen bevorzugen.
Kenntnisstand
7
3. Kenntnisstand
3.1 Platinverbindungen
Wie bereits in Kapitel 1 erwähnt, ist das Ziel aller aktuellen platinhaltigen Chemotherapeutika
die DNA. Sowohl Cis- als auch Carboplatin werden ausschließlich intravenös verabreicht. Ein
erheblicher Teil des Cisplatins wird an Serumproteine gebunden [36] bevor es zur DNA
gelangen kann. Geschwindigkeitsbestimmend bei der Platinierung der Oligonukleotide [3840] ist die Hydrolyse des Cisplatins, welche auf Grund der hohen Chloridkonzentration im
Blut (100mM) erst in der Zelle signifikant stattfindet, wie das folgende Schema zeigt.
Abb.3.1: Schematische Darstellung der Aufnahme von Cisplatin in die Zelle nach
intravenöser Injektion [41]
Eine vollständige Hydrolyse des Cisplatins bei [Cl]- = 4mM, was der Chloridkonzentration in
der Zelle entspricht, und einem pH-Wert von 7,4 (T = 37°C) wurde von MILLER und HOUSE
bereits nach zwei Stunden beobachtet [42]. Neben der Hydrolyse des Cisplatins ist auch die
Ringöffnung des Dicarboxylatringes des Carboplatins Gegenstand vieler Arbeiten. Die
Stabilität des Carboplatins ist im Vergleich zu der des Cisplatins deutlich höher. Mit Hilfe der
15
N/ 1H-NMR-Spektroskopie wurde die Ringöffnung in wässrigen Medien durch Nitrat,
Phosphat und Chlorid von SADLER untersucht [43] und dabei die Langlebigkeit des
Carboplatins in Wasser bewiesen.
Das Platin stellt nach der HSAB-Theorie eine leicht polarisierbare Lewis-Säure dar, die
bevorzugt eine Bindung mit einem „weichen“ Liganden eingeht. Als potentielle
Koordinationsmöglichkeiten kommen demnach für das (NH3)2Pt(II)-Fragment besonders die
Schwefeldonoratome des Methionins bzw. des Cysteins sowie die aromatischen
Stickstoffdonoratome des Histidin-Imidazolrings in Frage.
Kenntnisstand
8
Die Gruppe um SHELDRICK führte zahlreiche Untersuchungen der Reaktionen von Cisplatin
mit methionin- bzw. histidinhaltigen Oligopeptiden durch. Daraus resultiert [44-48] das
folgende Reaktionsschema.
Abb.3.2: Allgemeiner Reaktionsweg methioninhaltiger Peptide mit Cisplatin nach
SHELDRICK
Es bildet sich zunächst ein monodentater κS-Komplex aus, dem ein mehrfacher Ringschluss
zu bi-, tri- und tetradentaten Komplexen folgen kann. Unter bestimmten Voraussetzungen tritt
bei dieser Reaktion eine Peptidspaltung auf. Der trans-Effekt des Schwefels unterstützt hier
die Bildung der tridentaten Bischelate und die auftretende Spaltreaktion.
Die Thiolfunktion in nicht methyliertem Cystein kann zur Ausbildung von Dimeren mit einer
Disulfidbindung führen, die eine wichtige Rolle bei der Strukturbildung von Proteinen spielt.
Bei Reaktionen cysteinhaltiger Peptide mit Platinverbindungen, insbesondere Glutathion,
konnte eine Spaltung dieser Bindung durch Cisplatin beobachtet werden. Dieser Effekt wurde
bei Albuminen beobachtet [49-52] und könnte für einen Teil der Toxizität des Zytostatikums
verantwortlich sein.
Bei Reaktionen von Histidin mit Pt(II) –Verbindungen stellen die Aminogruppe, die beiden
Imidazolstickstoffe (N1 und N3) aber auch die Carboxylat-Sauerstoffatome potentielle
Koordinationsstellen dar. APPELTON stellte für die Reaktionen von Histidin und
histidinhaltigen Dipeptiden mit Cisplatin mehrere Reaktionsschemata [53] auf. Seinen
Untersuchungen zufolge wird zunächst eine kinetisch bevorzugte Sauerstoffkoordination
eingegangen. Über mehrere Stufen lagert sich der Komplex zu dem stabilen Endprodukt mit
κ2N3,NH-Koordination [53] um. Eine κ2N1,NH Koordination wird aus sterischen Gründen
nicht gebildet. Bei monofunktionellen Komplexen wie [Pt(dien)(H2O)]2+ wurde eine
Koordination an einem der beiden Stickstoffatome des Imidazolringes als thermodynamisch
Kenntnisstand
9
stabiles Endprodukt der Reaktion beobachtet [54, 55]. Zwischen diesen beiden Modi existiert
ein Gleichgewicht, das je nach Reaktionsbedingungen, insbesondere dem pH-Wert, N1-oder
N3-Koordination bevorzugen kann.
Bei den Umsetzungen mit konkurrierenden methionin- und histidinhaltigen Peptiden bilden
sich nach dem Reaktionsschema unter Abb.3.2 Makrochelate, die auf Grund der freien
Drehbarkeit und fehlender Ringspannung besonders stabilisiert werden. Werden Peptide mit
Sequenzen His-(Aa)n–Met (n = 0-3) mit monofunktionalem [Pt(dien)(H2O)]2+ umgesetzt, so
findet zunächst die kinetisch bevorzugte Koordination des Schwefels statt. Anschließend setzt
eine langsame Migration zum thermodynamisch bevorzugten N1-Stickstoff ein. Eine
Koordination an N3 konnte nur in deutlich geringerem Maße beobachtet werden [56, 57].
Mit Cisplatin und Modellpeptiden, Proteinen sowie Oligonukleotiden wurden zahlreiche
Studien durchgeführt, um den Metabolismus des Zytostatikums im Organismus weiter
aufzuklären. Dabei kann das Bindungsverhalten unter Verwendung diverser Methoden
untersucht werden.
Aus [1H,
15
N] und [1H,
13
C]-HSQC-NMR Studien mit dem Serumprotein Serotransferrin
konnte SADLER die Methioninkoordinationen an M256 und M499 als bevorzugte Stellen für
[Pt(en)Cl2] und cis-[PtCl2(NH3)2] beobachten. Die Platinierungsgeschwindigkeit des Met256
ist vermutlich höher als die des Met499. Des Weiteren stelle SADLER fest, dass M313 keine
bevorzugte Bindungsstelle im Gesamtprotein darstellt. Dieses Methionin liegt beim intakten
Protein innen. Der S,N-Chelat des M313 konnte nur bei der Umsetzung mit dem
rekombinierten N-Bereich und nicht beim intakten Protein beobachtet werden [58].
Auch die Gruppe um DYSON hat Untersuchungen der Wechselwirkungen Cisplatins mit dem
Serumprotein Transferrin durchgeführt. Aus 1:10 Transferrin:Cisplatin-Ansätzen nach
15/30/180 min Inkubation bei 20°C konnte mittels ESI-MS/MS an tryptischen Peptiden sowie
UV/Vis-Analysen eine Koordination des Cisplatinfragmentes [(NH3)2PtCl]+ an die
Aminosäure Threonin 457 nachgewiesen werden. Dieses Beispiel stellt die einzige bisher
veröffentlichte Arbeit dar, in der es gelungen ist eine Bindungsstelle in einem MetallkomplexProteinaddukt durch „bottom-up“ Tandem-Massenspektrometrie zu bestimmen [59]. Die
deconvulierten Massenspektren des freien (a) und des koordinierten Transferrins (b) aus
dieser Studie sind in der unteren Abbildung dargestellt.
Kenntnisstand
10
a)
b)
∆m/z = 264
Abb.3.3: Massenspektren (a) Serotransferrin sowie (b) eines Reaktionsgemisches
Serotransferrin/Cisplatin [59]
Aus der Massendifferenz von 264 zwischen den Peaks in den Spektren m/z = 79519 in a) und
m/z = 79786 in b), die dem Fragment [(NH3)2PtCl]+ entspricht, schloss die Gruppe auf eine
kovalente Bindung zwischen dem Metallkomplex und dem Protein. Bei theoretischen
Modelluntersuchungen konnten sie die Zugänglichkeit der Aminosäure T 457 für eine
Cisplatinkoordination bestätigen.
Da Serumalbumin mit einer Häufigkeit von etwa 50 % im Organismus vorliegt, ist eine
Koordination
eines
verabreichten
Arzneimittels
bzw.
Metallkomplexes
an
dieses
Transportprotein vorauszusehen. KEPPLER führte Untersuchungen durch, die Bindungsrate des
Cisplatins an Albumin zu bestimmen. Zur Veranschaulichung dient die von ihm erstellte
Verteilung in Abbildung 3.4.
Kenntnisstand
11
Abb.3.4: Bindungsrate von Cisplatin an Serumalbumin [60]
In der Grafik ist die Anzahl gebundener Pt-Atome pro Protein bei den unterschiedlichen
Metallkomplex-Protein-Ansätzen gegen die Inkubationszeit aufgetragen. Dabei kennzeichnen
a, b, c, b, d die folgenden [Cisplatin]/[HSA]-Verhältnisse: (a) 1:1; (b) 4:1, (c) 10:1 und (d)
20:1. Nach etwa sechs Tagen liegt (wie aus Kurve (a) erkennbar) Cisplatin zu weniger als
10% ungebunden vor.
[1H,
15
N]-HSQC-NMR-spektroskopische Daten von SADLER zeigen nach 2h Inkubation bei
37°C in Anwesenheit von Chloridionen einige Pt-O-Kreuzpeaks (δ15N im Bereich von -65 bis
-75), transständig zu Amminliganden, die nach 9h nicht mehr beobachtet werden [61] (siehe
Abbildung 3.5).
2h
9h
17h
Abb.3.5: [1H, 15N]-HSQC-NMR der 1:1 rHSA-Cisplatingemische als Funktion der Zeit [61]
Kenntnisstand
12
SADLER vermutete wegen beobachteter Pt-S-Kreuzpeaks nach 17 h Inkubation die bevorzugte
Bindung einer an der Oberfläche lokalisierten Methioninseitenkette M298, die einer κ2SM,NMKoordination zugeschrieben wurde. Zusätzlich konnten monofunktionale Addukte an die freie
Thiolgruppe C34 und einer oder zwei weiteren Methioninseitenketten im Serumalbumin
festgestellt werden.
Im Folgenden findet sich das Reaktionsschema, welches den oben genannten Befunden
entsprechen soll. Dabei stehen die Kennzeichnungen (a) bis (f) und (a´), (d´) für NMRspektroskopisch beobachtete Spezies.
Abb.3.6: Reaktionsschema Albumin/Cisplatin nach SADLER [61]
Bei Reaktionen von Carboplatin mit Cytochrom c, einem in den Mitochondrien
vorkommenden Protein, konnte die Gruppe um ZHU mittel ESI-MS wiederum Methionin als
Bindungsstelle identifizieren. Hierbei konnten M65 und M80 auf Grund der benachbarten
Spaltung der M-X-Bindung charakterisiert werden [62].
Kenntnisstand
13
Über ESI-MS konnte GIBSON ebenfalls Methionin als bevorzugte Koordinationsstelle und die
Bildung von vier verschiedenen Adduktypen bei der Rektion von Ubiquitin mit Cisplatin
nachweisen [63] (siehe Abbildung 3.7).
Abb.3.7: Deconvuliertes ESI-MS der Ubiquitin-Cisplatin-Addukte [63]
Aus seinen Studien stellte GIBSON einen möglichen Reaktionsweg auf, der von mono- über
bidentate zu tridentaten S,N,O-Koordinationen von Cisplatin im Protein führt. Dabei folgt
dem Verlust des Aqualiganden (m/z = 8812 zu m/z = 8792) die vermutete Chelatbildung
zwischen dem Thioether und dem α-Amin des N-terminalen Methionins (M1). Ein NH3Verlust, durch den trans-Effekt des Thioether-Schwefels ausgelöst, führt zur Reaktion mit
einer weiteren Aminosäurenseitenkette und somit zum tridentaten Ubiquitin-CisplatinAddukt.
Weitere massenspektrometrische Studien zu Wechselwirkungen des Ubiquitins mit
Platinverbindungen führten DYSON und HARTINGER durch. Dabei konnten sie beim Vergleich
von MALDI- und ESI-MS die Bildung von diversen Metallkomplex-Protein-Addukten
beobachten [64, 65].
Kenntnisstand
14
Abb.3.8: Deconvulierte Massenspektren der 2:1-Ansätze dreier Platinverbindungen mit
Ubiquitin [65]
Aus der Abbildung 3.8 ist erkennbar, dass sowohl Cisplatin (CDDP) als auch Transplatin
(TDDP) sowie das Oxaliplatin Addukte mit dem Protein ausbilden. Mit Hilfe der „top-down“Methode konnte M1 als Bindungsstelle von Cis- und Oxaliplatin, wie bereits GIBSON zeigen
konnte [63, 67], bestätigt werden. Diese Strategie hatten MANDAL et al. 2006 erstmals
verwendet, um Hinweise auf die Pt(II)-Koordination der Cysteine C5 und C7 des
Metallothioneins zu erhalten [66]. Für die Ubiquitin:Cisplatin-Reaktion stellte DYSON das
folgende Schema auf.
Abb.3.9:Hydrolyse und Reaktionsschema für Cisplatin und Ubiquitin [64]
Kenntnisstand
15
Beim Vergleich der von den untersuchten Pt(II)-Komplexen gebildeten Produkte fällt auf,
dass Cisplatin eine höhere Tendenz zur Bildung von Addukten des Typs Ub-[Pt(NH3)] und
Ub-[Pt(NH3)2] aufweist, was auf stabilere bi- oder tridentate Chelate hindeuten könnte. Die
mittels nESI-QToF-MS bestimmten Addukte aus Cis-, Trans-, Oxaliplatin und Ubiquitin nach
3 und 8 Tagen sind in der Tabelle 3.1 dargestellt.
Tab.3.1: Addukte des Ubiquitins mit Cis- (CDDP), Trans- (TDDP) und Oxaliplatins mit den
mittels nESI-QToF-MS bestimmten relativen Intensitäten nach 3 oder 8d
Inkubation bei 37°C [64]
Die Gruppe um MESSORI beschäftigte sich mit den Reaktionen von mehreren
Platinverbindungen und Cytochrom c. Dabei wurden die jeweiligen Platinkomplexe (Cis-,
Trans-, Carbo- und Oxaliplatin) mit dem Protein bei unterschiedlichen Metall:ProteinVerhältnissen zwischen 24-168h bei 37°C inkubiert [68]. Deconvulierte ESI-MS-Spektren der
Arbeitsgruppe des reinen Proteins (A) und 3:1 Cisplatin:Cytochrom c-Ansatzes nach 72h
Inkubation (B) sind in der folgenden Abbildung zu sehen.
Kenntnisstand
16
Abb.3.10: Deconvulierte ESI-MS-Spektren des reinen Proteins (A) und des
Cisplatin:Cytochrom c-Gemisches, 3:1, 72h/37°C (B) [68]
Aus dem Spektrum B) konnten MESSORI und PIERACCINI eine 1:1-Koordination (m/z =
12589) des [Pt(NH3)2]2+-Fragmentes an das Cytochrom c (∆m = 227) und eine 2:1Koordination desselben Platinrestes (m/z = 12816) beobachten, wobei noch Protein in der
Lösung enthalten ist (m/z = 12361). Die Vermutung, dass M65 des Proteins die bevorzugte
Bindungsstelle für die Platinkomplexe darstellen könnte, bestätigten sie in der nachfolgenden
NMR-spektroskopischen Studie. Dabei wurden aus der Umsetzung der unten dargestellten
Platin-Iminoether-Verbindung trans-[PtCl2{(E)-HN=C(OCH3)CH3}2] (trans-EE) und der
äquimolaren Menge an Protein die in der Abbildung 3.12 dargestellten Kreuzpeaks erhalten.
Abb.3.11: Eingesetzter trans-[PtCl2{(E)-HN=C(OCH3)CH3}2]-Komplex
Kenntnisstand
17
Abb.3.12: Überlagerte [1H, 13C]-HSQC-NMR-Spektren des reinen Proteins (blau) und des 1:1
trans-EE:Protein-Ansatzes nach 168h/37°C [69]
Aus der Beobachtung des neuen Kreuzpeaks und den bereits beschriebenen Verschiebungen
[Ref. 50 und 51 in 69] konnte M65 als bevorzugte Bindungsstelle bestätigt werden.
Dieselbe Gruppe konnte für das Protein Lysozym durch eine Röntgenstrukturanalyse den
Imidazolring des Histidins H15 als Bindungsstelle für das Cisplatin bestimmen [70]. Dabei
tränkten sie die nach etwa fünf Tagen bei 4°C entstandenen Lysozym Kristalle mit einer 10fach höher konzentrierten Cisplatinlösung und konnten nach 72h Inkubation bei wiederum
4°C die in der Abbildung 3.13 gezeigte Koordination des Cisplatins bei dem Protein
röntgenographisch bestimmen. Aus den begleitenden ESI-MS-Studien mit 1:3 ProteinCisplatin-Gemischen konnten sie nach 72h Inkubation bei 37°C eine Koordination des
[(NH3)2PtCl]+-Fragmentes an das Lysozym feststellen [70].
Abb.3.13: Schematische Darstellung der Cisplatin:H15-Wechselwirkung im Lysozym [70]
Kenntnisstand
18
3.2 Rutheniumverbindungen
Im Bereich der Platinverbindungen sind aus dem Bestreben, die bei Cisplatinbehandlung
auftretenden Nebenwirkungen zu senken und dessen Resistenzen zu vermeiden, einige
weitere Verbindungen erfolgreich in der Therapie aufgenommen worden (z. B. Carboplatin).
Neben Platin als Metallzentrum werden jedoch auch eine Reihe weiterer Metalle getestet. Ein
besonderes Augenmerk liegt dabei auf Ruthenium und seinen Verbindungen. Die eingangs
erwähnten bekanntesten Beispiele sind das KP1019, NAMI-A und RAPTA-Verbindungen.
Aus den Arbeiten mit (η6-Aren)Ru(II)-Verbindungen konnte bei Reaktionen mit Aminosäuren
die Bildung von Chelatkomplexen beobachtet werden [71]. Weitere Studien zum
Reaktionsverhalten mit Aminosäuren führte EXNER durch [72]. Er konnte zeigen, dass aus
polymeren Dichloro-η4-dien-Ruthenium(II)-Komplexen und Aminosäuren in wässrigen
Lösungen Verbindungen des Typs [Ru(Aminosäure)2(η4-dien)] mit einer N,O(Carboxylat)Koordination entstehen.
Eine Besonderheit bei der Verwendung von Rutheniumverbindungen als Antitumorwirkstoffe
stellen die Transportmöglichkeiten durch das Blut dar. Analog zum Transport von
Eisenverbindungen im Blutkreislauf über Transferproteine wie Albumin oder Transferrin,
sind die gleichen Proteine in der Lage Ruthenium(III)-Verbindungen zu befördern [73, 74].
Tumore im Darm etwa haben einen hohen Bedarf an Eisen [75], so dass die Proteine das
Eisen bzw. das Ruthenium dorthin transportieren. Aus Zellaufnahmetests konnten KEPPLER et
al. einen erhöhten Eintritt der KP1019-Verbindung in die Zelle beobachten, wenn diese mit
dem Fe(III)-haltigen Transferrin gebunden vorlag.
Abb.3.14: Zellaufnahme von KP1019 in humane Darmkrebszellen SW480 [75]
Bei diesen Untersuchungen stellen sie mittels ESI-MS fest, dass nach einer Inkubation des
KP1019-Komplexes mit Transferrin (3:1) von 10 min bei 37°C zwei Äquivalente des
Kenntnisstand
19
Komplexes an Transferrin gebunden vorliegen. Beim Vergleich der Reaktivität des
Komplexes KP1019 konnte jedoch die eindeutige Bevorzugung der Bildung von Albumingegenüber Transferrin-Addukten ermittelt werden [76].
Die Arbeitsgruppe von KOZLOWSKI et al. untersuchte unter anderem mittels UV/Vis- und
Fluoreszenzspektroskopie
die
Wechselwirkungen
tetrachlorobis(imidazol)ruthenat(III)]-/
zwischen
dem
Imidazol[trans-
trans-Indazol(bisindazole)tetrachlororuthenat(III)
-
Komplex und dem humanen Serumalbumin. Auf Grund erniedrigter Aktivität in der IIADomäne (in der Umgebung des Trp 214) des Proteins vermuteten sie die Bindung der
Komplexe an ein Histidin in der genannten Region, das H242 oder weitere in dieser Domäne
lokalisierte Histidinstellen [77, 78].
In seinen Studien der Wechselwirkungen der RAPTA-Komplexe mit Proteinen beobachteten
DYSON et al. Bindungen der Ru(II)-Komplexe an recombiniertem Serumalbumin (rHSA) und
Ubiquitin [79, 80], wie am Beispiel des rHSA im MALDI-TOF-Spektrum gezeigt (Abbildung
3.15).
Abb.3.15: MALDI-TOF-Spektren des rHSA und des rHSA-RAPTA-Konjugates aus [79]
Aus der Differenz von m/z = 1901 zwischen den erhaltenen Hauptmassenpeaks schlossen sie
auf eine drei- bis vierfache Koordination des Metallkomplexes an das Protein.
SADLER konnte mittels [1H,
15
N]-HSQC-NMR aus Reaktionen des antitumoraktiven [(η6-
Biphenyl)RuCl(en)](PF6)-Komplexes
mit
Cytochrom
c
Informationen
zu
dessen
Bindungsstellen in dem Protein bekommen. Dabei erhielt er nach einer 2h Inkubation des
Kenntnisstand
20
Metallkomplexes mit dem Protein bei 37°C keine Hinweise auf eine Histidinkoordination,
sondern auf α-Amino- oder Carboxylatgruppenkoordinationen [81]. Die kinetische
Bevorzugung der (η6-Aren)Ru(II)-Komplexe für anionische Sauerstoffatome wurde von
SADLER bereits für Mononucleotide, wie 5´-GMP beobachtet [82], wo einer schnellen
Bindung an die 5´-Monophosphatgruppe die lamgsame Migration zum thermodynamisch
bevorzugten N7-Atom der Nucleobase des 5´-GMP2- folgt, wie das von ihm aufgestellte
Schema in Abbildung 3.16 zeigt.
Abb.3.16: Reaktion des [(η6-Bipy)Ru(en)Cl]+-Komplexes mit 5´-GMP2-, 1:1, pH = 7,15, 25°C
(K = Speziesanteil in % nach 55 min Reaktion) [82]
Das Vorliegen einer Histidinkoordination des [(η6-p-Cymol)Ru(Aceton)3](CF3SO3)2Komplexes bei Lysozym, einem aus 129 Aminosäuren aufgebauten Protein, konnte SADLER
kristallographisch bestätigen [83]. Die Abbildung zeigt die von ihm bestimmte Bindungsstelle
H15.
Kenntnisstand
21
Abb.3.17: Links: Proteinstruktur des Lysozyms mit gekennzeichneter Bindungsposition des
Ru(II)-Komplexes; rechts: Bindungstasche vergrößert im space-filling Modell [83]
ESI-MS-Studien von MESSORI et al. konnten die Bildung von Ruthenium-Protein-Addukten
durch RAPTA-Komplexe sowohl mit Lysozym als auch mit dem oben erwähnten Cytochrom
c bestätigen [84]. Auch konnten sie H15, wie unter 3.1 bereits dargestellt, als
Cisplatinbindungsstelle im Lysozym zuordnen.
3.3 Methoden
Proteine können in ihrer intakten Form ohne vorherige Spaltung nach einer Trennung (z. B.
2D-Gelelektrophorese) durch Tandem-Massenspektrometrie analysiert werden („top-down“Methode) [85]. Proteinmischungen können auch vor der Überführung in die Gasphase
enzymatisch in sehr kleine Peptide verdaut und mittels Tandem-Massenspektrometrie aus den
erhaltenen Peptidfragmentspektren [86, 87] sequenziert werden („bottom-up“-Methode). Die
Komplexität der Protein- bzw. Peptidmischung kann eine Auftrennung der Proteine bzw. der
Peptide vor der massenspektrometrischen Analyse erfordern. Eine in der Literatur oft
beschriebene Methode ist die Trennung der Proteinmischung auf einem eindimensionalen
SDS-Polyacrylamidgel. Nach erfolgter Gelelektrophorese wird der Gelstreifen in einzelne
Stücke zerlegt und ein „In-Gel-Verdau“ durchgeführt. Die so entstandenen Peptide werden in
der zweiten Dimension über eine reversed phase-Chromatographie getrennt und im
angeschlossenen Massenspektrometer durch MS/MS sequenziert [88]. Eine Kombination der
ICP- Massenspektromterie mit der 1D- Gelelektrophorese wurde zur Bestimmung der
Wechselwirkung des Cisplatins mit dem Protein ompA aus E. coli verwendet [89]. Dabei
betrug die Inkubationszeit 30 min bei 37°C mit 10mM Cisplatin in 500 µl wässriger Lösung.
Aus der ESI-MS/MS- Analyse der Proteinbande des Gels mit dem höchsten Platinanteil
Kenntnisstand
22
wurde das Membranprotein ompA durch die Übereinstimmung seiner Peptidsequenzen mit
dem tatsächlichen Spektrum identifiziert. Dabei wurden Peptidionen von etwa 23% des
Proteins erhalten (= 23% coverage).
Nach der Inkubation (24h bei 37°C) von humanem Blutplasma mit Cisplatin und NAMI-A
konnten DYSON et al. nach der Trennung mittels PAGE, SDS-PAGE und 2DGelelektrophorese und der Identifizierung durch laser ablation inductively coupled plasmaMassenspektrometrie (LA-ICP-MS) Adduktbildung mit drei abundanten Serumproteinen:
Albumin, α2-Macroglobulin und Serotransferrrin bestätigen [90]. Beim Vergleich der
Peakintensitäten der einzelnen Profile konnten sie feststellen, dass das Cisplatin eine höhere
Affinität zum Abumin und NAMI-A hingegen zum Transferrin aufweist. Zur Verdeutlichung
dienen beide SDS-Profile des mit dem jeweiligen Metallkomplex inkubierten Blutplasmas.
A
B
Abb.3.18: LA-ICP Massenspektren/ SDS-PAGE des mit Cisplatin (A) oder NAMI-A(B)
inkubierten Blutplasmas [90]
Aufgrund hoher Abundanz mancher Proteine (cytosolische oder lösliche) in den Zellen bzw.
der resultierenden Peptide werden weniger abundante Membranproteine bzw. Peptide oftmals
überlagert und somit nicht detektiert. Diese Tatsache macht es beim Umgang mit gesamten
Zellsystemen (wie z. B. dem Bakterium Escherichia coli) wichtig die Zellbestandteile
voneinander zu trennen. Der schematische Aufbau einer Bakterienzelle in Abbildung 3.19 soll
dies verdeutlichen.
Kenntnisstand
23
Abb.3.19: Schematischer Aufbau einer Bakterienzelle [91]
Bakterienzellen sind normalerweise von einer festen, schützenden Zellwand umgeben. Die
darunter liegende Zellmembran regelt die Stoffpassage in das Zytoplasma (das halbflüssige
Zellinnere) und aus diesem heraus. Die DNA, das Erbmaterial, liegt im Nucleoid, dem
Kernäquivalent der Bakterien (Bakterien haben keinen von einer Membran umhüllten
Zellkern). An den Ribosomen findet die Proteinsynthese statt. Viele Bakterien verfügen über
Pili, Strukturen, die aus der Zelle herausragen, um DNA zu einem anderen Bakterium
übertragen zu können. Eine Liste der zur Proteinbestimmung in E. coli verwendeten
Methoden und der identifizierten Proteine (1627 Proteine identifiziert) findet sich in einem
Übersichtsartikel von LEE et al. [92].
Heutzutage finden auch häufig nicht 2D-PAGE basierte Proteomanalysen Anwendung (sog.
Shotgun-Verfahren). Bei den Technologien wie ICAT (Isotope Coded Affinity Tags) [93] und
MudPIT (Multidimensional Protein Identification Technology) [94] werden die Proteine vor
ihrer
Proteolyse
nicht
getrennt,
sondern
nur
die
entstehenden
Peptide
mittels
Flüssigkeitschromatographie in einer bzw. mehreren Dimensionen. Die Peptide werden dann
mittels ESI-MS/MS analysiert.
In der folgenden Darstellung ist der schematische Aufbau von MudPIT dargestellt.
Kenntnisstand
Denaturated Proteins
Proteingemisch
24
Peptides
2D-LC
Peptidgemisch
Datenbanksuche
Database search
2D-LC
Tandem MS
Abb.3.20: Schematischer Aufbau eines MudPIT-Experimentes [94]
Diese Methode erlaubt die zweidimensionale Trennung von Peptiden auf einer Kapillarsäule
und ist direkt (online) mit einem ESI-Massenspektrometer gekoppelt. Beim Beladen der
Kapillarsäule binden die Peptide zunächst an ein Kationen-Austauschmaterial, von dem sie
sequenziell durch die Verwendung eines Salzstufengradienten auf das folgende reversed
phase-Material eluiert werden. Die an das reversed phase-Material gebundenen Peptide
werden ihrer Hydrophobizität entsprechend mittels organischer Modifier eluiert und direkt im
nachfolgenden ESI-Massenspektrometer sequenziert. Die Verwendung moderner ESIMassenspektrometer, welche Scangeschwindigkeiten von bis zu drei MS/MS-Spektren pro
Sekunde erlauben, ermöglicht die Identifizierung von bis zu 20.000 Peptiden bzw. 1.500
Proteinen pro Tag unter Verwendung des SEQUEST-Algorithmus. Dieses Verfahren zeichnet
sich vor allem für die Identifizierung löslicher bzw. cytosolischer Proteine als sehr erfolgreich
aus [95, 96].
Die Auswertung erfolgt bei SEQUEST auf Grund statistischer Berechnungen. Zu der Masse
eines gemessenen Peptides werden in einem ersten Schritt 500 Peptide, deren Massen am
besten mir der gemessenen Masse übereinstimmen, aus der Datenbank extrahiert. Von diesen
500 Peptiden werden theoretische Sequenzspektren (MS/MS-Spektren) berechnet und ein
theoretisches Spektrum wird dazu erstellt. Durch einen bildlichen Vergleich zwischen
theoretischen und experimentellen Spektren werden für jedes Peptid die statistischen
Parameter
berechnet
[85].
Abschließend
werden
die
Peptide
nach
steigendem
Korrelationsfaktor aufgelistet, wobei das Peptid mit der höchsten Übereinstimmung in den
Kenntnisstand
25
Spektren den größten XCorr-Wert erhält. Ein weiterer Parameter ist der Sp-Wert (preliminary
score), der auf dem Vergleich der Übereinstimmung der Anzahl und m/z-Werte der Ionen im
theoretischen MS/MS-Spektrum mit dem experimentellen Spektrum basiert. Der RSp-Wert
(rank preliminary score) gibt den Rang des zugeordneten Sp-Wertes an. Die Anzahl der
Ionen, die im aufgenommenen MS/MS-Spektrum zugeordnet werden können, wird im
Verhältnis zu der theoretisch möglichen Anzahl prozentual angegeben. Der ∆Cn-Wert
(DeltaCn correlation value) gibt den Unterschied zwischen der erst- und der zweitbesten
Zuordnung der Peptidsequenz an. Bei den Angaben in der vorliegenden Arbeit handelt es sich
bei den ∆Cn-Angaben der identifizierten Peptidsequenzen stets um die ∆Cn- Werte zur
nächsten, unterschiedlichen Peptidsequenz. Die Massendifferenz ∆m berücksichtigt maximale
Massendifferenzen zwischen der Masse des experimentellen Vorläuferions und der des
theoretischen Ions. Die für die einzelnen Analysen gewählten Einschränkungen für die
genannten Parameter finden sich in den dazugehörigen Kapiteln.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
26
4. Reaktionen von metallhaltigen Zytostatika mit Peptiden
In Anlehnung an die vorhandenen Ergebnisse mehrerer Arbeitsgruppen [45, 55, 148, 149]
bezüglich des Reaktionsverhaltens von Aminosäuren mit Metallkomplexen wurden im
Rahmen dieser Arbeit Modellpeptide mit unterschiedlichen Aminosäurensequenzen
ausgewählt, Umsetzungen mit Platin- bzw. Rutheniumkomplexen durchgeführt und die dabei
entstandenen Produkte chromatographisch bzw. massenspektrometrisch untersucht. Mit Hilfe
dieser Voruntersuchungen sollte die prinzipielle Eignung von ausgewählten Metallkomplexen
für die vorgesehenen MudPIT-Messungen überprüft werden.
4.1. Chromatographische Modellstudien
Mit dem Reaktionssystem AcSerGlyOH/Cisplatin wurden chromatographische Messungen im
analytischen Maßstab durchgeführt, um die Speziesentwicklung und deren Stabilität zu
analysieren. Dieses Dipeptid enthält zwei potentielle O-Donoratome und wurde ausgewählt,
um zu überprüfen, (a) ob κO-koordinierte Platinkomplexe bei einem niedrigen pH-Wert
(~2,1)
unter
den
Bedingungen
der
IPRP-HPLC
getrennt
und
(b)
anschließend
massenspektrometrisch detektiert werden können. Die entstandenen Produkte konnten offline
massenspektrometrisch charakterisiert werden. Bei der offline-Kopplung werden die Produkte
am
Auslass
des
UV-Detektors
aufgefangen
und
manuell
in
den
Einlass
des
Massenspektrometers injiziert.
Ein Konzentrationsvergleich ist dabei immer nur für dieselbe Substanz zu unterschiedlichen
Zeitpunkten möglich, da mit dem UV-Detektor Absorptionswerte von Substanzen gemessen
werden, deren molare Extinktionskoeffizienten unbekannt sind.
Das Dipeptid AcSerGlyOH wurde mit der äquimolaren Menge Cisplatin inkubiert. Dabei
erfolgte eine kontinuierliche chromatographische Untersuchung der Reaktionslösung während
der Inkubation bei 37°C über etwa 500 Stunden. Die Reaktion verlief bei einem pH-Wert von
7,8.
In
der
folgenden
Abbildung
Reaktionsgemisches dargestellt.
ist
die
zeitabhängige
Produktverteilung
des
Reaktionen von metallhaltigen Zytostatika mit Peptiden
27
Abb.4.1: Zeitabhängige Produktverteilung des Systems Cisplatin:AcSerGlyOH, 1:1, pH = 7,8
Die Cisplatinkonzentration nimmt in den ersten 10 Stunden ab und stabilisiert sich dann über
die nächsten 300 Stunden. Unter dem Signal des Cisplatins können sich ebenfalls
chromatographisch gleichwertige Hydrolyseprodukte, wie z.B. [(NH3)2Pt(H2O)]2+ verbergen.
Bereits nach einer Reaktionszeit von 0,1 Stunden kann das erste Produkt (κOG in der
Abbildung 4.1) detektiert werden, welches nach etwa 100 Stunden sein Konzentrationsmaximum erreicht und danach an Peakfläche abnimmt. Aus dem kinetischen Verlauf der
beiden gebildeten Spezies kann vermutet werden, dass das κOG-Produkt als Vorläufer des
Endproduktes in monodentater Form vorliegt. Es handelt sich dabei um den neutralen
Komplex [PtCl(AcSGO-κOG)(NH3)2]. Die Bildung des κ2OG,NG-Folgeproduktes kann erst
nach fünf Stunden Reaktionszeit beobachtet werden. Es zeigt nach etwa 500 Stunden seine
höchste Peakfläche und verliert danach wieder stetig an Konzentration.
Abb.4.2: Produkte des Systems AcSerGlyOH:Cisplatin
Reaktionen von metallhaltigen Zytostatika mit Peptiden
28
Für die κOG-Spezies konnten zwei Massenpeaks bei m/z = 467 (theoretisch m/z = 467,5) und
bei m/z = 425 (theoretisch m/z = 425,5) für die Abspaltung der Acetylfunktion beobachtet
werden. Die Trennung bei pH = 2,1 (IPRP-HPLC) sowie die erfolgreiche Detektion des κOGProduktes bestätigen prinzipiell die Eignung der LC/MS-Kopplungsmethoden zur
Identifizierung solcher Pt-Bindungsstellen in tryptisch gespaltenen Peptiden bei MudPITAnalysen.
Das Massenspektrum des κ2NG,OG-Produktes ist in der folgenden Abbildung zu sehen.
Abb.4.3: ESI-Massenspektrum des Molekülpeaks [Pt(AcSGOH)(NH3)2-H] +
In dem obigen Spektrum ist für den Molekülpeak m/z = 432 des κ2NG,OG-Produktes das für
Platin charakteristische Isotopenpattern in der eingefügten Vergrößerung zu erkennen. Eine
Abspaltung eines Amminliganden kann neben dem Peak des Hauptproduktes bei m/z = 415
beobachtet werden. Die untere Tabelle 4.1 zeigt eine Auflistung zugeordneter Massenpeaks
mit relativen Intensitäten über 25% aus dem ESI-Massenspektrum des κ2NG,OG-Produktes.
Die restlichen Massenpeaks enthalten kein Platin, wie aus deren Verteilung erkennbar ist.
Eine Zuordnung dieser Peaks war nicht möglich.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
29
Tab.4.1: ESI-Massenspektrum des Molekülpeaks [Pt(AcSGOH)(NH3)2-H] +
Komplex
[Pt(AcSGOH)(NH3)2-H] +
[Pt(AcSGOH)(NH3)-H] +
[Pt(AcSGOH)(NH3)2- CH2OH]+
m/z (beob.)
432
415
402
m/z (theor.)
432
415
401
Die vermutliche Struktur des κ2NG,OG-Produktes ist in Abbildung 4.2 dargestellt. Hierbei
handelt es sich um einen bidentaten Komplex, der zwei Rotamere bilden kann (Rotation um
die C-N-Bindung im Glycin). Um die vorgeschlagene Koordination zu bestätigen, wurden 1HNMR-Spektren des Reaktionsansatzes pH =7,5 in Abhängigkeit von der Zeit aufgenommen.
Die Aufspaltung der βS-Protonen (in Abbildung 4.4 (a) Dublett bei δ = 3,6 ppm) zu zwei
benachbarten Dubletts (in Abbildung 4.4 (b) bei δ = 3,58-3,6 ppm) beweist ihre chemische
Nichtäquivalenz in den Rotameren.
Abb.4.4: 1H-NMR-Spektren des Systems AcSerGlyOH:Cisplatin nach 0,1h (a) und 24h (b)
Inkubation bei 37°C
Der Vergleich der αG-Signale im oberen Spektrum des Peptid-Cisplatin-Gemisches nach 0,1 h
und 24 h Inkubation zeigt eine Hochfeld-Verschiebung des Signals von 3,7 nach 3,5 ppm. Die
Reaktionen von metallhaltigen Zytostatika mit Peptiden
30
Aufspaltung der Protonen der Glycingruppe zum Dublett bei δ ≈ 3,5 ppm kann durch die
gleichzeitige Koordination des Platins am Sauerstoff der Carboxylgruppe und am
Amidstickstoff dieses Glycins erklärt werden. Da die αG-Protonen in einem fünfgliedrigen
Chelatring vorliegen, sind sie chemisch nicht äquivalent und weisen unterschiedliche
Verschiebungswerte auf. Dieses Verhalten bestätigt die obige Vermutung, dass es sich bei
dem gebildeten Komplex um eine Koordination am C-terminalen Sauerstoff handelt.
Um die Stabilität der gebildeten Produkte zu prüfen, wurden den Reaktionsgemischen nach
längerer Inkubationszeit (mindestens 500h) weitere Aminosäuren als Konkurrenz angeboten.
Dazu wurde dem System AcSerGlyOH:Cisplatin die äquimolare Menge an AcMetOH oder
AcHisOH hinzugefügt und der Reaktionsansatz wiederum bei 37°C und pH = 4,5 inkubiert.
Abhängig
von
der
Zeit
erfolgte
darauf
hin
eine
chromatographische
und
massenspektrometrische Untersuchung dieser Systeme. Hierbei sollte festgestellt werden, ob
und wie schnell die mögliche Migration des Cisplatin-Fragmentes von AcSerGlyOH zu den
thermodynamisch
bevorzugten
weicheren
S-
bzw.
N(Imidazol)-Donoratomen
der
konkurrierenden Aminosäuren AcMetOH und AcHisOH abläuft. Es sollte untersucht werden,
ob ein Koordinationswechsel unter MudPIT-Bedingungen zu erwarten ist.
Eine Produktverteilung des Systems nach Reaktion mit AcMetOH in Abhängigkeit von der
Zeit ist in Abbildung 4.5 dargestellt.
Abb.4.5: Zeitabhängige Produktverteilung des Systems AcSerGlyOH:Cisplatin mit AcMet,
1:1:1, pH = 4,5
Reaktionen von metallhaltigen Zytostatika mit Peptiden
31
Aus der Abbildung ist erkennbar, dass bereits nach 0,1h Reaktionszeit die Konzentration der
zugesetzten Aminosäure abnimmt. Gleichzeitig entsteht ein Produkt mit einer Retentionszeit
von etwa 30min, welches nach etwa 100h sein Konzentrationsmaximum erreicht. Dabei
handelt es sich um die κS:κ2OG,NG-Spezies. Ebenso bildet sich ein Produkt mit einer
Retentionszeit von etwa 11min, dessen Intensität in den 100h Reaktionszeit zunimmt,
vermutlich das κS:κ2NG,NS(SG)-Produkt. Die κO-Spezies wird in der Abbildung 4.5 nicht
gezeigt, da sie nach 500h nur noch in geringer Konzentration in Lösung vorliegt.
Die parallel beobachtete Abnahme der Intensität des κ2NG,OG-Produktes deutet auf eine
Beteiligung dieses Komplexes bzw. der im Gleichgewicht vorliegenden κOG- und κNGSpezies bei der Neubildung der methioninhaltigen Produkte, die vermutlich bei der Bildung
der κS:κ2OG,NG-Spezies dem Verlauf in der unteren Formulierung folgt:
Abb.4.6: mögliche Reaktionsfolge des κ2NG,OG-Produktes zum κS:κ2OG,NG-Produkt
Von beiden Produkten aus dem Ansatz AcSerGlyOH:Cisplatin mit AcMetOH, 1:1:1 bei 37°C
konnten ESI-Massenspektren aufgenommen werden, diese sind in den Abbildungen 4.7 und
4.8 dargestellt. Nachfolgend ist das Massenspektrum des chromatographisch bei etwa 30min
retardiernden Produktes aus dem System AcSerGlyOH:Cisplatin und AcMetOH zu finden.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
32
Abb.4.7: ESI-Massenspektrum des Molekülpeaks [Pt(κS:κ2OG,NG)(NH3)+H]+
Es finden sich mehrere Peaks mit dem Platinpattern im Spektrum, z. B. m/z = 644, 606, 589
und 415. Die Tabelle 4.2 listet die Zuordnung der wichtigsten Peaks aus dem ESIMassenspektrum der chromatographisch getrennten Spezies zu.
Tab.4.2: ESI-Massenspektrum des Molekülpeaks [Pt(κS:κ2OG,NG)(NH3)+H]+
Komplex
[Pt(AcSGOH)(AcM)(NH3)2+Na]+
[Pt(AcSGOH)(AcM)(NH3)+H]+
[Pt(AcSGOH)(AcM)+H]+
[Pt(AcSGOH)(NH3)+H]+
m/z (beob.)
644
606
589
415
m/z (theor.)
645
606
589
415
Die untere Abbildung zeigt das ESI-Massenspektrum des bei etwa 11min chromatographisch
retardiernden Produktes aus dem System AcSerGlyOH: Cisplatin und AcMetOH.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
33
Abb.4.8: ESI-Massenspektrum des Molekülpeaks [Pt(κS:κ2NG,NS)(NH3)]+
In dieser Darstellung weisen die zwei Hauptpeaks bei m/z = 373 und m/z = 548, der
Massenpeak bei m/z = 563 und auch weitere Peaks das charakteristische Platinpattern auf. Die
Peakzuordnung findet sich in der folgenden Tabelle. Die beobachteten m/z-Werte bestätigen
das Vorliegen von koordinierten HSerGlyOH und AcMetOH Liganden in dem Komplex, d. h.
bei der Bildung des Komplexes muss eine Acetylspaltung stattgefunden haben.
Tab.4.3: ESI-Massenspektrum des Molekülpeaks [Pt(κS:κ2NG,NS)(NH3)]+
Komplex
[Pt(HSGOH)(AcM)(NH3)]+
[Pt(HSGOH)(AcM)]+
[Pt(HSGOH)(NH3)]+
m/z (beob.)
563
548
373
m/z (theor.)
564
547
373
Aus den Massenspektren der gebildeten Produkte nach Zugabe der N-terminal geschützten
Aminosäure Methionin (AcMet) kann die Beibehaltung der Adduktbildung zu AcSerGlyOH
bzw. HSerGlyOH bestätigt werden. Beide Produkte weisen entsprechend ihren Massenpeaks
Reaktionen von metallhaltigen Zytostatika mit Peptiden
34
vermutlich die monodentate κS-Koordination des Methionins auf, da AcMet während der
Fragmentierung abgespalten wird. Eine massenspektrometrische Spaltung eines κ2S,Ngebundenen AcMetOH wird in der Regel nicht beobachtet [149, 150]. Diese Studie zeigt
außerdem,
dass
die
Pt-O-Bindung
im
κS:κ2OG,NG-Komplex
nicht
von
einer
Schwefelkoordination innerhalb von etwa 100h bei 37°C und pH = 4,5 verdrängt wird.
Zu den vorliegenden Beobachtungen lässt sich der folgende mögliche Reaktionsweg
aufstellen.
Abb.4.9: Mögliche Reaktionswege von AcSerGlyOH:Cisplatin nach Zugabe von AcMetOH
bei pH = 4,5
Reaktionen von metallhaltigen Zytostatika mit Peptiden
35
Aus der Darstellung lässt sich erkennen, dass beide Endprodukte aus der κO-Spezies
entstehen können. Aus dem Reaktionsweg wird deutlich, dass bei dem κS:κ2NG,OG-Komplex
die Pt-O-Bindung erhalten bleibt. Wird statt des κ2OG,S-Zwischenproduktes die κ2NG,SSpezies gebildet, dann kann im weiteren Verlauf eine Acetylspaltung auftreten, die durch den
trans-Effekt des Schwefels unterstützt wird. Eine analoge Spaltung konnten MANKA et al. für
serinhaltige Peptide mit der Sequenz –SerMet- beobachten [44]. In den Studien wurde eine
Acetylspaltung für AcSerMetOH ab pH < 4,5 nachgewiesen. Die Endprodukte der Reaktion
des Systems AcSerGlyOH:AcMetOH:Cisplatin sind in Abbildung 4.10 gezeigt.
Abb.4.10: Reaktionsprodukte des Systems AcSerGlyOH:Cisplatin nach Zugabe von
AcMetOH
Bei der Umsetzung des Systems AcSerGlyOH:Cisplatin mit AcHisOH konnte keine Reaktion
mit der Aminosäure bei pH = 4,5 beobachtet werden. Die Anfangskonzentration blieb auch
nach etwa 300h Inkubation beinahe unverändert. Ebenso gleich bleibend konnte die
Konzentration des κ2NG,OG-Produktes im Verlauf der Reaktionszeit detektiert werden. Dieses
Verhalten zeigt, dass die Bindungen im κ2NG,OG-Produkt gegenüber einer möglichen
Imidazol-Bindung stabil bleiben.
4.2. Massenspektrometrische Modellstudien
Studien zu Stabilitäten der Pt-Peptid-Bindungen unter massenspektrometrischen Bedingungen
(MS bzw. MS/MS) wurden mit ausgewählten Metallkomplex-Peptid-Reaktionsansätzen
durchgeführt.
Es
wurden
die
folgenden
Metallkomplexe
eingesetzt:
[(η6-p-
Cymol)RuCl2(DMSO)], [Pt(dien)(H2O)](NO3)2 und cis-[PtCl2(NH3)2]. Die Strukturformeln
der Verbindungen sind in der nachstehenden Abbildung gezeigt.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
M = 384 g.mol-1
M = 316 g.mol-1
36
M = 300 g.mol-1
Abb.4.11: Strukturen und molare Massen der eingesetzten Metallkomplexe
Für die massenspektrometrischen Untersuchungen sollten die gebildeten Peptid-Komplexe
kinetisch relativ stabil sein. Eine umfangreiche Fragmentierung zu b+- und y+-Ionen ist für
MudPIT-Charakterisierungen essentiell und die zusätzliche Bestätigung der Zuordnung
einzelner metallierter Fragmentionen durch die typische Isotopenverteilung eines Metalls
hilfreich.
Die
Fragmentierungsionen
werden
unter
Verwendung
der
Tandem-
Massenspektrometrie erhalten. Im Fall von Peptiden wird die CID-Energie meist so gewählt,
dass hauptsächlich die Peptidbindungen fragmentieren. Die so erhaltenen Fragmente werden
als b+- und y+-Serie bezeichnet [151]. Zur Veranschaulichung dient die folgende Abbildung.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
N-Terminus
37
C-Terminus
CID
Vorläuferion
m/z
Fragmentionen-Spektrum
Fragmentionen
Abb.4.12: Die Bildung von MS/MS-Fragmentionenserien und ihre MS/MS-Spektren
[nach 151]
Beide Serien entstehen durch Spaltung der bevorzugt gespaltenen Amidbindung zwischen
dem Carbonyl-Kohlenstoff und dem Amid-Stickstoff der Peptidkette.
4.2.1. Modellstudien mit [(η6-p-Cymol)RuCl2(DMSO)]
In seinen Untersuchungen mit [(η6–Aromat)Ru]2+-Verbindungen konnte SIEBERT bidentate
κ2- und tridentate κ3-Komplexe der Metallfragmente mit unterschiedlichen Aminosäuren und
Peptiden mittels IPRP-HPLC erfolgreich trennen [148], womit die Stabilität von RuAddukten unter chromatographischen Bedingungen bei pH ~ 2,1 eindeutig bewiesen wurde.
Für die Untersuchung der Möglichkeit Ru-O-Bindungen massenspektrometrisch detektieren
und deren Fragmentierungsverhalten prüfen zu können, wurde eine Peptidsequenz mit
mehreren O-Donoratomen als potentielle Koordinationsstellen ausgewählt. Aus dem Ansatz
des Peptids der Sequenz Ile-Val-Asp-Ala-Gln-Gly-Asn-Asp-Val-Leu-Ile-Pro-Gly (HI-V-D-AQ-G-N-D-V-L-I-P-GOH) mit der äquimolaren Menge des Rutheniumkomplexes wurde das
Reaktionen von metallhaltigen Zytostatika mit Peptiden
38
unten dargestellte Massenspektrum aufgenommen (Abb. 4.13). Die Inkubation betrug
ebenfalls 24h bei 37°C und wurde in einer wässrigen Lösung bei pH = 4,1 durchgeführt.
Abb.4.13: Massenspektrum des Reaktionsgemisches IVDAQGNDVLIPG und [(η6-p-Cymol)
RuCl2(DMSO)] nach 24h bei 37 °C (M = [(η6-p-Cymol)Ru(Peptid)H-1]+)
Der bei m/z = 1545 mit höchster Intensität beobachtete Massenpeak zeigt das für Ruthenium
charakteristische Isotopenmuster auf und entspricht dem berechneten Fragment für
[(η6-p-Cymol)Ru(Peptid)H-1]+. Das Spektrum enthält die Ionen b8+ bis b11+ und y10+ und y11+.
Daneben sind sowohl mehrere zweifach geladene (b92+ bis b112+) als auch modifizierte Ionen
(η6-p-Cymol-Abspaltung) zugeordnet. Diese Befunde lassen vermuten, dass in der
Peptidsequenz HI-V-D-A-Q-G-N-D-V-L-I-P-GOH die Asparaginsäure D8 die bevorzugte
Bindungsstelle für den eingesetzten Rutheniumkomplex darstellt. Dennoch kann aus dem
Vorhandensein des ruthenierten Ions y10+ eine Beteiligung der Asparaginsäure D3 an der
Koordination des Rutheniumfragmentes nicht ausgeschlossen werden.
In der folgenden Abbildung ist ein MS/MS-Spektrum des Hauptpeaks aus dem
Reaktionsansatz des Komplexes [(η6-p-Cymol)RuCl2(DMSO)] mit dem zweiten Peptid PheLys-Val-Gly-His-Phe-Leu-Gly (HF-K-V-G-H-F-L-GOH) nach 24h Inkubation bei 37 °C und
Reaktionen von metallhaltigen Zytostatika mit Peptiden
39
pH = 2,5 dargestellt. In dieser Peptidsequenz stellt der Histidinrest die thermodynamisch
begünstigte Koordinationsstelle dar. Die Masse des Peaks mit m/z = 1138 entspricht der
Masse des Peptids mit dem [(p-Cymol)Ru]2+-Fragment; beide Chloride und der DMSOLigand sind bereits abgespalten worden.
Abb.4.14: MS/MS-Spektrum des Molekülpeaks [(η6-p-Cymol)Ru(FKVGHFLG)H-1]+ bei m/z
=1138
In dem Massenspektrum lassen sich Zuordnungen für mehrere Fragmente finden. So ist ein
Massenpeak für die Koordination des [(η6-p-Cymol)Ru]2+-Fragmentes an das Peptid nach
Abspaltung der Carboxy-Gruppe (CO2H) zu sehen (m/z = 1093). Aus dem Massenpeak
[Ru(FKVGHFLG)H-1]+ sind die Ionen b5´+, b6´+ und y6´+ durch CID-Fragmentierung erhalten
worden. Es kann ein Massenpeak für die erfolgte Abspaltung der letzten drei C-terminalen
Aminosäuren [F-L-GOH]+ des [(η6-p-Cymol)Ru(FKVGHFLG)H-1]+-Fragmentes beobachtet
werden. Da in dem Massenspektrum der Peak mit höchster relativer Intensität (100%) durch
Reaktionen von metallhaltigen Zytostatika mit Peptiden
40
die Abspaltung des (p-Cymol)-Liganden entstanden ist (m/z = 1004), wurde diese
Beobachtung für weitere Experimente genutzt. Der Verlust des aromatischen Liganden wird
als „neutral loss“ bei allen weiteren massenspektrometrischen Untersuchungen mit diesem
Komplex einbezogen. In den MudPIT-Untersuchungen wurden CID-MS-Spektren von
neutral-loss-Ionen ([M-(p-Cymol)]n+) aus den drei höchsten Massenpeaks [M]n+ angefertigt,
falls solche Ionen detektiert wurden.
Die Untersuchungen mit den Modellpeptiden weisen darauf hin, dass das Rutheniumatom bei
der CID-MS-Fragmentierung koordiniert bleibt. Somit sind die Voraussetzungen für MudPITAnalysen für den Rutheniumkomplex erfüllt. Für [(η6-p-Cymol)RuCl(en)]+ wurde dagegen
der Verlust des gesamten Rutheniumfragmentes bei der CID-MS-Fragmentierung beobachtet,
mit der Folge, dass eine LC2/MS2-Analyse der Proteintargets dieses Komplexes nicht möglich
ist.
4.2.2. Modelluntersuchung mit [Pt(dien)(H2O)]2+
Auch mit dem Metallkomplex [Pt(dien)(H2O)]2+ wurden Reaktionen mit unterschiedlich
sequenzierten Peptiden als Modellsysteme untersucht. Der Platinkomplex kann auf Grund
seines tridentaten Liganden (dien) nur monodentate Bindungen ausbilden. Deswegen musste
zuerst geprüft werden, ob MS/MS-Analysen möglich sein würden. Aus den Arbeiten von
WOLTERS [55] sind potentielle Aminosäuren, die von dem Komplex koordiniert werden
können bereits bekannt. Darin wurden Reaktionen unter anderem mit methionin- und
histidinhaltigen Peptiden chromatographisch analysiert. Die entstandenen Spezies konnten bei
pH = 2,1 durch IPRP-HPLC getrennt werden. Im Rahmen dieser Arbeit wurde das
Dekapeptid Gly-Ala-Leu-Thr-Ans-Val-Ser-Met-Ala-Lys (HG-A-L-T-N-V-S-M-A-KOH) mit
der Thioetherseitenkette des Methionins als Reaktionspartner für das [Pt(dien)]2+-Fragment
ausgewählt. Das MS/MS-Spektrum des äquimolaren Reaktionsgemisches nach 24h
Inkubation bei 37 °C bei pH = 4,4 mit dem Ausschnitt aus dem Massenspektrum der
Reaktionslösung ist in der folgenden Abbildung dargestellt.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
41
Abb.4.15: MS/MS-Spektrum des Molekülipeaks [Pt(dien)(GALTNVSMAK)H-1]+ bei m/z =
1287
Das Massenspektrum zeigt in der Vergrößerung bei m/z = 1287 den Peak des Peptid[Pt(dien)]2+-Adduktes nach der Inkubation von 24h bei 37°C. Es sind zwei platinierte Ionen
der b+-Serie zugeordnet, b8+ und b9+ und die nicht platinierten Ionen [y9+-CO2H]+, y8+ und y6+
aus der y+-Serie zu erkennen. Diese Beobachtungen lassen auf eine Koordination an der
Aminosäure Methionin M8 schließen. Die Untersuchung mit dem Modellpeptid bestätigt die
prinzipielle Eignung des [Pt(dien)]2+-Fragmentes für MudPIT-Analysen. Allerdings wäre die
Zahl der b+- und y+-Ionen (4 von 18 möglichen) zu gering, um eine Identifizierung der
Sequenz zu ermöglichen.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
42
4.2.3. Modellstudien mit Cisplatin
Ein äquimolarer Ansatz wurde für Cisplatin mit dem Peptid der Sequenz Phe-Lys-Val-SerGly-Thr-Leu-Val-Gly (HF-K-V-S-G-T-L-V-GOH) für 24h in wässriger Lösung (pH = 4,5)
bei 37 °C inkubiert. Diese Peptidsequenz enthält sowohl am Threonin- als auch am Serinrest
Hydroxygruppen, welche als potentielle Koordinationsstellen für das Cisplatin in Frage
kommen können. Diese Lösung wurde direkt ins Thermo Finnigan LCQ Massenspektrometer
mit Hilfe einer 100 µl Spritze injiziert und im Bereich von m/z = 300-2000 detektiert. Das
Nonapeptid hat eine molare Masse von 907 Da. In der folgenden Abbildung sind das MS/MSSpektrum des intensivsten Massenpeaks (m/z = 1100) und ein Ausschnitt des
Gesamtspektrums dargestellt.
Abb.4.16: MS/MS-Spektrum des Molekülpeakss [(Pt(FKVSGTLVG)H-1)]+ bei m/z = 1100
Reaktionen von metallhaltigen Zytostatika mit Peptiden
43
In dem vergrößerten Ausschnitt des Spektrums sind Massenpeaks für mehrere Peptidaddukte
der einzelnen Cisplatinfragmente zu erkennen. Auch das charakteristische Isotopenpattern des
Platins ist in der Vergrößerung eindeutig zu sehen. Der intensivste Massenpeak mit der Masse
m/z = 1100 entspricht dem Ion [Pt(Peptid)H-1)]+. Es gibt außerdem einen Peak mit der Masse
m/z = 1117, welcher dem Peptidkomplex des Fragmentes [(NH3)Pt]2+ zugeordnet werden
kann.
Ein
weiterer
Peak
mit
der
Masse
m/z
=
1135
entspricht
dem
Ion
[{(NH3)2Pt]}(Peptid)H-1]+. Außerdem ist der zweitintensivste Massenpeak mit m/z = 1171 für
die Koordination des [(NH3)2PtCl]+-Restes an das Peptid zu sehen. Das Massenspektrum
bestätigt wieder, dass Platinaddukte ohne Verlust des Pt-Fragmentes unter ESI-Bedingungen
ionisiert werden können. Da die Molekülionen mit den [Pt]2+-, [(NH3)Pt]2+- und [(NH3)2Pt]2+Fragmenten
durch
neutral
loss
von
Ammin-
bzw.
Aqualiganden
bei
der
massenspektrometrischen Ionisierung entstehen können, ist eine Bestimmung des in Lösung
vorhandenen Platinfragmentes in diesem wie in vielen anderen Fällen nicht möglich. Die
Tatsache, dass der [Pt]2+-Komplex das Molekülion mit der höchsten relativen Intensität
liefert, spricht jedoch dafür, dass zumindest ein bidentater Chelatkomplex in Lösung
vorliegen muss.
Um über die Möglichkeit der Peptidspaltung unter Beibehaltung des Platinfragmentes und die
über genauere Bindungsstelle des Metallfragmentes Aussagen treffen zu können, wurden von
den einzelnen Massenpeaks der Peptid-Cisplatin-Addukte MS/MS-Spektren aufgenommen.
Dabei wird das ausgewählte Mutterion mit der zur Spaltung der Peptidbindungen
erforderlichen CID-Energie beschossen und somit weiter fragmentiert. Anhand des
Abspaltungsmusters der enthaltenen Massendifferenzen, die den Aminosäurenmassen
entsprechen, kann die Sequenz des Peptids identifiziert werden. In dem MS/MS-Spektrum des
Ions [(Pt(FKVSGTLVGH-1)]+ in Abb. 4.15 sind von 16 möglichen b+- und y+-Ionen (jeweils
8) 10 Ionen zugeordnet, davon drei y+- und sieben b+-Ionen. Die Beobachtung von 62,5% der
möglichen
Fragmentionen
würde
unter
MudPIT-Bedingungen
eine
eindeutige
Charakterisierung der Peptidsequenz ermöglichen. Aus der Fragmentierung der Peptidsequenz
HF-K-V-S-G-T-L-V-GOH wurden die platinierten Ionen b2+ bis b8+ und die nicht platinierten
Ionen y5+ bis y7+ erhalten. Dieser Befund lässt eine Zuordnung der Koordination des
[(NH3)2Pt]2+-Fragmentes an der Aminosäure Lysin (K2 in der Sequenz) zu.
Ein weiterer äquimolarer Ansatz wurde mit Cisplatin und dem Heptapeptid der Sequenz
Val-Ser-Glu-His-Glu-Ala-Thr (HV-S-E-H-E-A-TOH) nach 24h Inkubation bei 37°C und pH
= 4,1 massenspektrometrisch untersucht. In diesem Fall bietet der Histidinrest die
thermodynamisch günstige Bindungsstelle an.
Reaktionen von metallhaltigen Zytostatika mit Peptiden
44
Das entsprechende Tandem-Massenspektrum des Hauptpeaks von [(NH3)2Pt(VSEHEAT)H-1]+
findet sich in Abbildung 4.16 Darin ist zur Verdeutlichung des Platinmusters ein Ausschnitt
des Massenspektrums des Peptid-Cisplatin-Gemisches eingefügt. Im Gegensatz zum letzten
Beispiel weisen die [(NH3)2Ptl]2+- und [(NH3)2PtCl]+-Addukte die höchste relative Intensität
auf, was für eine mögliche monodentate [(NH3)2PtCl]+-Koordination spricht.
Abb.4.17: MS/MS-Spektrum des Molekülpeaks [(NH3)2Pt(VSEHEAT)H-1]+ bei m/z = 999
Das MS/MS-Spektrum enthält Abspaltungen der Ammingruppen des Cisplatinrestes (m/z =
983, m/z = 965). Daneben konnten drei b+-Ionen detektiert werden, welche aus den
modifizierten Massenpeaks resultieren. Dabei stammen b4´+, b5´+ und b6´+ vom Massenpeak
m/z = 965 (Abspaltung beider Ammingruppen) und b4´´+ aus dem Massenpeak bei m/z = 948
(Abspaltung der C-terminalen Hydroxygruppe). Aus dem Vorkommen der drei platinierten
Ionen der b+-Serie (b4´+, b5´+ und b6´+) kann vermutet werden, dass das Cisplatinfragment die
Aminosäure H4 aus der Peptidsequenz HV-S-E-H-E-A-TOH koordiniert. Das MS/MSSpektrum verdeutlicht, dass neutral loss Vorgänge die Identifizierung einer Peptidsequenz
Reaktionen von metallhaltigen Zytostatika mit Peptiden
45
erschweren oder verhindern können, wenn nicht genügend b+- und y+-Ionen aus dem
ursprünglichen Molekülion zugeordnet werden können. Die relativ geringe Zahl von b+- und
y+-Ionen ist auch typsich für kurze Peptidsequenzen (n < 10), was häufig die Charaktrisierung
solcher Sequenzen erschweren kann.
Ein äquimolarer Ansatz aus Cisplatin und dem längeren Peptid (n = 13) Ile-Val-Asp-Ala-GlnGly-Asn-Asp-Val-Leu-Ile-Pro-Gly (HI-V-D-A-Q-G-N-D-V-L-I-P-GOH) wurde für 24h bei
37°C und pH = 4,2 inkubiert und massenspektrometrisch analysiert. Dieses Modellpeptid
wurde ausgewählt, um das Fragmentierungsverhalten von κO-koordinierten Addukten zu
überprüfen. Das MS/MS-Spektrum des entstandenen Adduktes
[(NH3)2Pt(IVDAQGNDVLIPG)H-1]+ ist in der folgenden Abbildung dargestellt. Wiederum ist
ein Ausschnitt des Massenspektrums eingefügt.
Abb.4.18: MS/MS des Molekülpeaks [(NH3)2Pt(IVDAQGNDVLIPG)H-1]+ bei m/z = 1537
Für dieses Peptid dominiert das Molekülion des [(NH3)2Pt]2+-Adduktes, was für eine monobzw. bidentate Koordination spricht. In dem Spektrum sind Abspaltungen der einzelnen
Reaktionen von metallhaltigen Zytostatika mit Peptiden
46
Aminosäuren des Peptids erkennbar. Aus den Massendifferenzen lässt sich nun der so
genannte Fingerabdruck ablesen. Abhängig von der Lage der Ladung sollten in dieser
Sequenz bis zu zwölf b+- und zwölf y+-Ionen zugeordnet werden. Im vorliegenden Spektrum
konnten tatsächlich sieben b+-Ionen (b5+ bis b11+) und drei y+-Ionen (y6+, y9+ und y10+)
zugeordnet werden, d. h. 42% der möglichen Fragmentionen. Eine Zuordnung von mehr als
30-35% reicht in der Regel aus, um eine Peptidsequenz eindeutig zu bestimmen. Das MS/MSSpektrum verdeutlicht, dass im Vergleich zu den Modellpeptiden mit nur sieben oder neun
Aminosäureresten anteilig mehr Fragmentionen für das längere Peptid (mit 13 Resten)
erhalten wurden. Im Allgemeinen ergeben Peptidsequenzen mit etwa 10-21 Resten die besten
MS/MS-Spektren. Bei kürzeren und längeren Peptiden ist häufig der Anteil der möglichen b+und y+-Fragmentionen zu gering, um eine eindeutige Zuordnung zuzulassen.
Auch in dieser Darstellung konnten modifizierte Ionen der b+-Serie nach Abspaltung der
Amminliganden des Cisplatins zugeordnet werden (b5´+ bis b11´+). Die eingesetzte
Peptidsequenz HI-V-D-A-Q-G-N-D-V-L-I-P-GOH enthält zwei Asparaginsäuren (D3 und
D8), welche als potentielle Koordinationsstellen für das Cisplatin in Frage kommen. Da
platinierte Ionen ab b5+ sowie bis y10+-Ionen zu beobachten sind, kann die Bindungsstelle zu
Asparaginsäure D3 zugeordnet werden. Die Abwesenheit der Ionen b1+ bis b3+ sowie der
Ionen yk+ für k > 10 steht im Einklang mit dieser Zuordnung. Die MS- und MS/MSUntersuchungen bestätigen, dass eine Fragmentierung zu einer adäquaten Zahl von
platinierten b+- und y+-Ionen auch bei gleichzeitigen neutral loss Reaktionen möglich ist.
4.2.4. Schlussfolgerungen für LC2/ESI-MS2-Analysen
Die beschriebenen Voruntersuchungen ergeben eine Reihe von Voraussetzungen an die
mittels MudPIT-Analyse zu behandelnden Systeme. Um die Flüssigchromatographie und die
Massenspektrometrie miteinander kombiniert einsetzen zu können, muss eine zufriedenstellende Trennung des Reaktionsgemisches möglich sein. Dabei müssen signifikante Anteile
der zu trennenden Komplexe bei den relativ niedrigen pH-Werten der IPPR- sowie SCXChromatographie (pH ≈ 2) stabil bleiben. Aus den vorangegangenen Studien wurde deutlich,
dass auch die erhaltenen κO-Spezies des Cisplatins diesen Anforderungen genügen.
Andererseits werden bei der verwendeten „bottom-up“-Methode, bei welcher die zu
untersuchenden Proteine enzymatisch in kleine Peptide gespalten werden, pH-Werte um 7,8
benötigt. Die chromatographischen Untersuchungen bei einem pH- Wert von 7,8 bestätigen,
dass auch κO-Komplexe über einen langen Zeitraum (bis 500h) in schwach alkalischer
Reaktionen von metallhaltigen Zytostatika mit Peptiden
47
Lösung stabil sind. Zahlreiche Untersuchungen von Peptid/Cisplatin-Reaktionssystemen
haben gezeigt, dass κN- und κS-Addukte unter alkalischen Bedigungen (7 < pH < 9,5) recht
stabil sind [45, 149]. Die Metallfragment-Peptid-Bindungen müssen auch während der
MS/MS-Fragmentierung aufrecht erhalten bleiben. Gute Übereinstimmungen mit den
theoretischen, in Datenbanken vorhandenen Spektren lassen sich nur dann erzielen, wenn die
zu untersuchenden Verbindungen/Systeme eine Fragmentierung in möglichst hoher Anzahl an
b+- und y+-Ionen (mindestens 30-35%) auch bei neutral loss ergeben.
Die
Voruntersuchungen
bestätigen
außerdem,
dass
die
Komplexe
[(η6-p-
Cymol)RuCl2(DMSO)], [Pt(dien)(H2O)](NO3)2 und cis-[PtCl2(NH3)2] alle diese Voraussetzungen erfüllen und deswegen für MudPIT-Analysen prinzipiell geeignet sind. Für die
zytotoxische Verbindung [(η6-p-Cymol)RuCl(en)]+ werden sowohl die chromatographischen
als auch die massenspektrometrischen Bedingungen nicht erfüllt.
Vergleichsuntersuchungen
an
den
vorhandenen
Finnigan
LCQ
und
LTQ
Massenspektrometern belegen, dass die wesentlich höhere Scanzahl des LTQs (bis zu drei
MS/MS-Spektren pro Sekunde bei LTQ, ein MS/MS-Spektrum alle zwei Sekunden bei LCQ)
für die effektive Adduktcharakterisierung essentiell ist. Bei LCQ-Untersuchungen wurden in
der Regel nur etwa 10-20% der bei den LTQ-Messungen gefundenen Proteinaddukte
identifiziert.
Proteintargets in Escherichia coli
48
5. Proteintargets in Escherichia coli
Wie die Modellsysteme gezeigt haben, können die untersuchten Metallkomplexe kinetisch
stabile Bindungen mit den Seitenketten unterschiedlicher Aminosäuren bilden und diese unter
den massenspektrometrischen und chromatographischen Messbedingungen bestehen bleiben.
Mit den Erkenntnissen aus den Voruntersuchungen konnten Umsetzungen mit Zellsystemen
vorgenommen werden. Dabei wurden Metallkomplexe ausgewählt, die cytotoxisch wirksam
sind oder als potentielle Zytostatika mit ihren Metallzentren diskutiert werden. Des Weiteren
ist es für die massenspektrometrischen Methoden von Vorteil Komplexe zu verwenden,
welche unter Abspaltung der Liganden kinetisch bzw. thermodynamisch stabile Bindungen
eingehen oder Chelate mit den Peptiden bilden können.
Wie in Kapitel 3 bereits beschrieben, wurde als Modell für Reaktionen im zellulären
Gesamtsystem das Darmbakterium Escherichia coli in der Forschung mehrfach verwendet.
Vor den Umsetzungen mit Metallkomplexen erfolgte die Züchtung des Bakteriums wie im
(Kap. 10) beschrieben unter 37°C mit Zugabe des LB-Mediums. Nach der Inkubation einer
Suspension der E. coli-Zellen mit dem Metallkomplex wird durch Zentrifungieren ein Pellet
erhalten. Nach dem nachfolgenden enzymatischen Verdau werden die (halb-)tryptischen
Peptide chromatographisch getrennt und direkt über ESI-MS/MS-Messungen auf die
Bindungsstellen der Metallkomplexfragmente in den Proteinen des Bakteriums analysiert. In
der Literatur wurde, wie in Kapitel 3 schon erwähnt, ein Protein-Cisplatin-Komplex aus E.
coli Zelle bereits von DYSON [89] beschrieben. Hierbei wurde mittels 1D-PAGE, ICP-MS und
ESI-MS/MS-Analysen
die
Koordination
des
[(NH3)2PtCl]+-Fragmentes
an
ein
Membranprotein, das ompA festgestellt. Allerdings gelang es den Autoren nicht die
Bindungsstelle des Cisplatinfragmentes zu ermitteln.
Bei einer MudPIT-Analyse werden die auf SCX-Material geladenen Peptide mittels eines
Salzstufen-/RP-Gradienten auf die RP-Phase gewaschen und so aus den einzelnen Zyklen ins
ESI-Massenspektrometer eluiert. So können bis zu 1500 Proteine pro Tag identifiziert
werden, wobei die Identifizierung von Proteinen durch Zuweisung theoretischer
Peptidsequenzen unter Verwendung geeigneter Suchalgorithmen erfolgt. Die Auswertung
basiert bei SEQUEST auf statistischen Berechnungen. Dabei werden der Masse eines
gemessenen Peptides entsprechend in einem ersten Schritt 500 Peptide, deren Massen am
besten mit der gemessenen Masse übereinstimmen, aus der Datenbank extrahiert. Von diesen
500 Peptiden werden theoretische Sequenzspektren (MS/MS-Spektren) berechnet und in
einem theoretischen Spektrum abgebildet. Durch einen bildlichen Vergleich zwischen dem
Proteintargets in Escherichia coli
49
theoretischen und dem gemessenen Spektrum wird für jedes Peptid ein so genannter KreuzKorrelationsfaktor (XCorr) berechnet. Abschließend werden die Peptide nach fallendem
Korrelationsfaktor aufgelistet, wobei das Peptid mit der höchsten Übereinstimmung den
größten XCorr erhält und somit zur Proteinidentifizierung herangezogen wird.
5.1. Proteintargets von [(η6-p-Cymol)RuCl2(DMSO)]
Für die nachstehend disktutierte MudPIT-Analyse wurde ein Metallzentrum gewählt, welches
ein deutlich erkennbares Isotopenpattern aufweist. Auch auf Grund der aktuellen Studien
bezüglich
neuer
Zytostatika
eignet
sich
eine
Rutheniumverbindung
als
Testsystem/Reaktionspartner für das Zellsystem. Es wurde der Komplex [(η6-pCymol)RuCl2(DMSO)] mit einem Arenliganden, DMSO und zwei Chloriden am
Rutheniumzentrum gewählt. Wie oben beschrieben (Kapitel 4.2) wurde jedoch in den
Vorstudien
das
Abspalten
des
Arenliganden
unter
den
massenspektrometrischen
Ionisierungsbedingungen bei dem Rutheniumkomplex festgestellt und musste deshalb bei der
Analyse beachtet werden. Diese Einstellung wurde als „neutral-loss“ beim Messvorgang
vorgegeben, so dass die drei höchsten Molekülpeaks nur nach dem Verlust der Liganden als
[M-(p-Cymol)]n+-Ionen zur Fragmentierung herangezogen wurden.
Die massenspektrometrische Analyse der chromatographisch getrennten Peptide liefert neben
nicht koordinierten Proteinstücken auch die mit einem Rutheniumfragment modifizierten
Sequenzen. In die Suchoptionen wurden die potentiellen Aminosäuren einbezogen: C, D, E,
H, K, Y, W, S, T und M. Für die mit dem Rutheniumfragment koordinierten Peptidsequenzen
wurde eine ursprüngliche Liste mit etwa 80 Proteintargets erhalten und prinzipiell nach den
im Folgenden beschriebenen Punkten analysiert, um so genannte „false-positive“ Ergebnisse
zu vermeiden. Die XCorr-Werte aller Peptidsequenzen betragen mindestens 2,5 für zweifach
und 3,5 für dreifach geladene Moleküle, die Sp-Werte sind größer als 500 und die
Massenspektren weisen mindestens 30 % Ionenzuordnung bei Di- bzw. Trikationen auf. Alle
Sequenzen werden bezüglich ihrer ∆Cn-Werte geprüft, um sicherzustellen, dass nicht auch
nicht koordinierte Peptidsequenzen in Frage kommen. Um falsche Zuordnungen von
Koordinationen zu vermeiden, werden nicht nur die SEQUEST-Werte sondern auch die
einzelnen MS/MS-Spektren der Proteintargets auf ihre Qualität und das Vorhandensein des
typischen Ruthenium-Isotopenmusters bei ausgewählten Molekülionen beurteilt. Dabei wird
auch darauf geachtet, dass die zugeordneten Ionen nicht im Untergrundrauschen eingeteilt
Proteintargets in Escherichia coli
50
sind (S/N-Verhältnis). Nach der Anwendung dieser Prüfungen/Vergleiche zur Vermeidung
von falsch positiven Befunden wird eine Liste mit modifizierten Peptidsequenzen aufgestellt.
In der Tabelle ist eine Liste der nach der SEQUEST-Datenbanksuche identifizierten Proteine
aus E. coli, die mit dem [(η6-p-Cymol)RuCl2(DMSO)]-Komplex modifiziert wurden,
dargestellt.
Tab.5.1: Proteintargets von [(η6-p-Cymol)RuCl2(DMSO)] in E. coli
Protein/Peptid
Bindungs- z XCorr ∆Cn
stelle
1 PPID_ECOLI
V.SD*AASNDTESLAGAEQAAGVK.A
D393
2 OSMY_ECOLI
V.VT*LSGFVESQAQAEEAVK.V
T88
V.VT*LSGFVESQAQAEEAVK.V
T88
3 CSPC_ECOLI
S.KD*VFVHFSAIQGNGFK.T
D27
S.KD*VFVHFSAIQGNGFK.T
D27
4 SUCC_ECOLI
A.K*GLTDAAQQVVAAVEGK.
K372
5 DING_ECOLI
K.IAFPPID*SPVVI.T
D619
6 TRAI1_ECOLI
K.S*VSMMAMLGGDK.R
S89
7 DBPA_ECOLI
L.T*LCGGQPFGMoxQR.D
T105
8 HRPA_ECOLI
A.QGAVMAT*EK.V
T719
* = Ru2+
Sp
RSp Ionen
2
4,91
0,15 1718,0
1
23/40
2
2
4,28
3,57
0,21 1199,7
0,16 1093,8
1
1
18/34
18/34
2
2
3,05
3,18
0,2 910,5
0,25 732,4
1
1
16/30
16/30
2
3,11
0,19 829,4
1
18/32
2
3,49
0,21 883,7
1
15/22
2
2,70
0,00 999,2
2
14/22
2
2,58
0,01 863,9
1
14/22
2
2,50
0,17 517,9
14
10/16
Die Proteine aus der Tabelle 5.1 können nach ihren Funktionen in der Zelle eingeteilt werden.
Es sind vier stressregulierende Proteine (ppiD, osmY, cspC und sucC) und vier Helikasen,
eine damage-inducible DNA-Helikase dinG sowie traI, dbpA und hrpA. Die Helikasen traI,
dbpA und hrpA erfüllen nicht alle Kriterien bezüglich ihrer SEQUEST-Werte, werden jedoch
an dieser Stelle wegen ihrer wichtigen Funktionen in der Zelle aufgelistet. Beispielhaft beträgt
der ∆Cn-Wert der Sequenz aus der Helikase traI 0,00 im Vergleich zu einer nicht ruthenierten
Sequenz der Proteins purA und sollte danach als koordinierte Sequenz ausgeschlossen
werden. Dennoch wurde es als Protein, an welches der Rutheniumkomplex bindet, aufgelistet,
da das zugehörige MS/MS-Spektrum eindeutig das Rutheniumpattern bei den entsprechenden
Ionen aufweist. Aus dem 192 kDa Protein wurde die Sequenz [89S*VSMMAMLGGDK100]2+
Proteintargets in Escherichia coli
51
an der Aminosäure Serin koordiniert. Die Helikase traI wurde als DNA-Helikase I
identifiziert und ist eine DNA-abhängige ATPase. Die folgende Abbildung zeigt das
Massenspektrum aus der MudPIT-Analyse.
Abb.5.1: MudPIT-MS/MS-Spektrum von [S*VSMMAMLGGDK]2+ aus traI
In dem Massenspektrum sind 12 der 22 möglichen b+- und y+-Ionen zugeordnet. Davon sind
sechs b+- und sechs y+-Ionen sowie zusätzlich ein b2+ und zwei y2+-Ionen in der Abb. 5.1
beschriftet. Koordiniert wurde in der Peptidsequenz anhand der SEQUEST-Parameter die
erste Aminosäure der Sequenz Serin 89. Somit enthalten alle b+- jedoch keines der y+-Ionen
ein Rutheniumatom, was für ausgewählte Ionen bestätigt werden konnte. In dem
Massenspektrum (Abb.5.1) ist die charakteristische Isotopenverteilung des Metalls bei
mehreren b+-Ionen erkennbar und wird für b10+ exemplarisch vergrößert eingefügt. Die
folgende Tabelle enthält die SEQUEST-Werte des Peptids aus der Helikase traI sowie weitere
acht Sequenzen, welche der Masse von m/z = 1328 (mit ∆m ± 2,5) entsprechen.
Proteintargets in Escherichia coli
52
Tab.5.2: SEQUEST-Parameter von [S*VSMMAMLGGDK]2+ aus traI im Vergleich zu
anderen potentiellen Peptidsequenzen
Sequenz
K.S*VSMMAMLGGDK.R
D.LFDKET*FAEK.L
K.SVS*MMAMLGGDK.R
D.NFDEMT*DINK.V
R.MoxE*NFTTNLNQ.L
I.SWMVC*DMVEK.P
K.SVSM*MAMLGGDK.R
-.MoxQTQLTEE*M*.R
R.IFNDRGEVHIE.A
Q.FQK*S*HH*LK.T
-.FNIS*GSVFVEK.-
XCorr
2,703
2,7
2,599
2,426
2,421
2,383
2,347
2,338
2,338
2,327
2,174
∆Cn
Sp RSp Ionen
Protein
0
999,2
2 14/22 TRAI1_ECOLI
0,001 784,8 11 12/18 PURA_ECOLI
0,038 863,7
8 13/22 TRAI1_ECOLI
0,103 910,5
6 12/18 YFGB_ECOLI
0,104
681
22 11/18 CLPA_ECOLI
0,118 960,6
5 13/18 MTFA_ECOLI
0,132 651,8 27 12/22 TRAI1_ECOLI
0,135 559,7 65 10/16 GLCF_ECOLI
0,135 455,7 207 10/20 DMSA_ECOLI
0,139 819,7 10 11/14 GSPA_ECOLI
0,196 1081,5 1 14/20 NUPG_ECOLI
Aus der oberen Tabelle wird deutlich, dass für das Massenspektrum der Sequenz
[LFDKET*FAEK]2+ des Proteins pura ebenfalls gute Übereinstimmungen mit dem
aufgenommenen Spektrum vorhanden sind. Die Helikase traI weist neben der Aminosäure
S89 ein weiteres Serin (S 91) und ein Methionin (M92) als potentielle Angriffsstellen für
Ruthenium auf. Aus den bewertenden Werten in Tabelle 5.2 wird jedoch offensichtlich, dass
die zweite Bindungsstelle M 92 (mit ∆Cn = 0,132) auf Grund der deutlich schlechteren
Übereinstimmung der beobachteten und theoretischen Spektren wenig wahrscheinlich ist.
Zwischen S89 und S91 kann dagegen nicht eindeutig entschieden werden, da bj+-Ionen mit j <
3 und yk+ mit k > 9 im MS/MS-Spektrum fehlen. Das Vorhandensein eines potentiellen y112+Ions ist für die SEQUEST-Entscheidung zu Gunsten von S89 ausschlaggebend.
Wie aus den zugehörigen SEQUEST-Werten aus der Tabelle 5.2 deutlich wird, würden die
traI und die restlichen zwei Helikasen dbpA und hrpA wegen ihrer niedrigen ∆Cn- Werte als
unsichere Targets eingestuft werden. Dieselbe potentiell ruthenierte Sequenz der traI Helikase
wurde allerdings auch in der zweiten unabhängigen MudPIT-Analyse detektiert, so dass die
Bindungsstellen S89 bzw. S91 hierdurch bekräftigt werden.
In der folgenden Abbildung findet sich ein Beispiel für ein eindeutig bestimmtes modifiziertes
Peptid aus einem Protein des E. coli Bakteriums, die Helikase dinG (probable ATPdependent helicase dinG). Die Helikase dinG besteht aus 716 Aminosäuren mit etwa 81 kDa
Molekularmasse. Sie wird als DNA damage-inducible Helikase des Bakteriums E. coli
bezeichnet und ist für die folgenden Funktionen verantwortlich: Beteiligung an DNAReparatur und eventuell auch Replikation. Das Massenspektrum des Peptids der Sequenz
Proteintargets in Escherichia coli
53
[613IAFPPID*SPVVI624]2+, mit den Aminosäuren zwischen 613 und 624 ist in der Abbildung
5.2 dargestellt. Modifiziert wurde mit Ruthenium die Aminosäure Asparaginsäure D619.
Abb.5.2: MudPIT-MS/MS-Spektrum von [IAFPPID*SPVVI]2+ aus dinG
In den Vergrößerungen in Abbildung 5.2 wurden zwei ausgewählte Ionen dargestellt, das b7+
(IAFPPID*) und das y6+ (D*SPVVI), um das Rutheniumisotopenpattern bei diesen Ionen zu
verdeutlichen. Aus dem Spektrum ist ersichtlich, dass y6+ ein Rutheniumatom enthält, so dass
D619 eindeutig als Koordinationsstelle zugeordnet werden kann. Für die MudPITMassenspektren des Rutheniumkomplex/E. coli-Gemisches werden sowohl die Masse des
Fragmentes [(p-Cymol)102Ru]2+ als auch mit Berücksichtigung des „neutral loss“ nur die
Masse des [102Ru]2+-Restes bei der Modifikation der Peptidionen beachtet. Wenn neutral loss
Peaks von den jeweils drei intensivsten Massenpeaks identifiziert werden konnten, wurden
MS/MS Spektren dieser Ionen aufgenommen und dem Spektrum mit möglichen theoretischen
Proteintargets in Escherichia coli
54
Peptidsequenzen angeglichen. Für die angegebene Masse in der Suchoption wurde der
Ladungsausgleich mit Abgabe von zwei Protonen berücksichtigt, so dass sich eine Differenz
von 100 Masseneinheiten für das [102Ru]2+-Fragment ergab. Bei dem in Abbildung 5.2
dargestelltem Spektrum wurden insgesamt 15 b+- und y+-Ionen von 22 möglichen zugeordnet.
Aus der „Markierung“ dieser Sequenz mit dem Metallfragment wird deutlich, dass alle Ionen
der b+-Serie ab b7+ das charakteristische Isotopenmuster des Rutheniums aufzeigen müssen,
da diese Fragmente alle das Metall enthalten. Im Gegensatz dazu zeigen in dieser Sequenz die
Ionen der y+-Serie bis y5+ dieses Pattern nicht. Die Werte des SEQUEST Algorithmus für
dieses Peptid sind in der Tabelle 5.3 dargestellt.
Tab.5.3: SEQUEST-Parameter von [IAFPPID*SPVVI]2+ aus dinG im Vergleich zu anderen
potentiellen Peptidsequenzen
Sequenz
XCorr ∆Cn
Sp RSp Ionen
Protein
K.IAFPPID*SPVVI.T
3,494
0 883,70 1 15/22 DING_ECOLI
K.IAFPPIDS*PVVI.T
3,211 0,08 591,10 6 13/22 DING_ECOLI
K.T*PEVIT*PPEQG.G 2,773 0,21 537,80 10 12/20 PTV3B_ECOLI
K.TPE*VIT*PPEQG.G 2,737 0,22 445,60 24 11/20 PTV3B_ECOLI
H.E*IIGD*IVPLAK.R 2,667 0,24 524,30 12 12/20 HIS6_ECOLI
F.ETVDPVTQAPLAK.I 2,65 0,24 642,60 3 14/24 PUUC_ECOLI
A.D*LVVNDTTYK*.F 2,644 0,24 517,40 13 12/18 STFR_ECOLI
A.D*LVVNDTT*YK.F 2,635 0,25 435,00 28 11/18 STFR_ECOLI
K.IAFPPIDSPVVIT.E 2,593 0,26 652,50 2 14/24 DING_ECOLI
A.D*LVVND*TTYK.F 2,592 0,26 455,50 19 11/18 STFR_ECOLI
In der Tabelle 5.3 sind zehn mögliche Peptidsequenzen eingetragen. Drei der zehn
Peptidsequenzen stammen aus dem Protein dinG, die restlichen stammen aus vier weiteren
Proteinen, ptv3B, his6, puuC und stfR. Zwei der Sequenzen sind nicht mit einem
Rutheniumfragment koordiniert, sechs weisen eine potentielle zweifache Koordination auf.
Wie aus den Werten erkennbar ist, zeigt die Koordination der Asparaginsäure D619 die am
besten übereinstimmende Zuordnung. Die Modifikation des Serins 620 ist nach den XCorr-,
Sp- und RSp-Werten weniger wahrscheinlich als die der Asparaginsäure D619. Da das
zugehörige b7+-Ion das Rutheniumpattern aufweist, kann die Koordinationsstelle S620
eindeutig ausgeschlossen werden.
Ein weiteres Proteintarget aus dem Bakterium E. coli, für welches ein von Ruthenium
koordiniertes Peptid gefunden wurde, ist das cspC (cold shock-like protein cspC). Dabei
handelt es sich um ein 7 kDa großes Protein, in welchem an der Aminosäure D27 der
Peptidsequenz [26KD*VFVHFSAIQGNGFK41]2+ Ruthenium koordiniert wird. Das Protein ist
Proteintargets in Escherichia coli
55
für die Regulierung der Expression zweier wichtiger Stressproteine (rpoS und uspA)
verantwortlich [92]. In der Abbildung 5.3 ist das zugehörige Massenspektrum zu dem
detektierten Proteinausschnitt gezeigt.
Abb.5.3: MudPIT-MS/MS-Spektrum von [KD*VFVHFSAIQGNGFK]2+ aus cspC
Das Massenspektrum enthält 18 Peaks der möglichen 30 b+- und y+- Ionen. In der
Vergrößerung ist ein Beispiel aus der b+-Ionenserie eingefügt. Auch in diesem Fall ist bei dem
b8+-Ion das Rutheniumpattern klar erkennbar. Diese Beobachtung entspricht der schrittweisen
Fragmentierung der Peptidsequenz, die dazu führt, dass alle b+-Ionen ab b2+ sowie das y15+Ion rutheniert vorliegen. Die Sequenz des Proteins mit der Koordination des
Rutheniumfragmentes an die Asparaginsäure D27 konnte in zwei unabhängigen MudPITAnalysen beobachtet werden.
Die SEQUEST-Parameter des Peptides aus cspC und weiterer der Masse von m/z = 1896 (∆m
± 2,5) entsprechender Sequenzen sind in Tabelle 5.4 dargestellt.
Proteintargets in Escherichia coli
56
Tab.5.4: SEQUEST-Parameter von [KD*VFVHFSAIQGNGFK]2+ aus cspC im Vergleich zu
anderen potentiellen Peptidsequenzen
Sequenz
S.KD*VFVHFSAIQGNGFK.T
S.K*DVFVHFSAIQGNGFK.T
A.TPLIPQPVTDAIGLQGMoxK.T
L.VTDMT*DPDW*EPIMoxK.K
R.YRT*QLENTE*IQWL.E
R.YRT*QLENT*EIQWL.E
S.KDVFVH*FSAIQGNGFK.T
R.YRT*QLE*NTEIQWL.E
R.HGHNEADEPSATQPLMY.Q
R.TLVTASTPGAD*MT*PGKMox.D
XCorr
3,051
2,959
2,45
2,324
2,22
2,203
2,188
2,181
2,06
2,049
∆Cn
0
0,03
0,2
0,24
0,27
0,28
0,28
0,29
0,33
0,33
Sp RSp Ionen
Protein
910,5 1 16/30 CSPC_ECOLI
910,5 1 16/30 CSPC_ECOLI
530,4 3 15/34 CYSX_ECOLI
348,1 51 10/26 PPSA_ECOLI
455,2 8 11/24 CYDC_ECOLI
455,2 8 11/24 CYDC_ECOLI
528,3 4 13/30 CSPC_ECOLI
363,3 37 10/24 CYDC_ECOLI
285 150 10/32 ODO1_ECOLI
340,7 55 11/32 FLGF_ECOLI
Die Tabelle enthält zehn Sequenzen aus sechs unterschiedlichen Proteinen des Bakteriums
(cspC, cysX, ppsA, cycD, odo1 und flgF), wobei nur die Zuordnungen im cspc Protein die
gewählten Kriterien erfüllen (z. B. Sp-Wert > 500). Das Massenspektrum enthält keinen
eindeutigen Hinweis darauf, dass die Koordination des Lysin 26 ausgeschlossen werden kann.
Das für das y15+-Ion beobachtete Isotopenmuster lässt eine eindeutige positive Identifizierung
der Isotopenverteilung des Rutheniums nicht zu. Somit wird keine der beiden benachbarten
Koordinationsmöglichkeiten (K26 und D27) abgelehnt.
Eine zusätzliche Möglichkeit der Bestätigung/Bekräftigung oder Ablehnung der angegebenen
Koordination des Metalls bzw. des Komplexfragmentes an das Peptid besteht darin, sich die
räumliche „Zugänglichkeit“ der Seitenkette einer Aminosäure bei Vorhandensein einer
Kristallstruktur des Proteins anzuschauen. Liegt die Seitenkette sterisch günstig, ist eine
Koordination an diese Aminosäure wahrscheinlich. Dieses Kriterium kann dann auch
herangezogen werden, wenn aus den Parametern und den Ionenzuordnungen im
Massenspektrum nicht eindeutig geklärt werden kann, welche Aminosäure koordiniert wurde.
In der folgenden Abbildung ist die Struktur des Proteins cspA (Struktur bekannt und
vergleichbar mit cspC) mit der Sequenzfolge Serin 2 bis Leucin 70 dargestellt.
Proteintargets in Escherichia coli
57
Abb.5.4: Die Proteinstruktur des cspA Proteins mit den gekennzeichneten Seitenketten von
K14, T20 und D27 (PDB ID: 1mjc, [97])
In der Abbildung sind die Seitenketten möglicher Koordinationsstellen dargestellt. Es sind
auch Aminosäuren berücksichtigt, deren Seitenketten in der Tertiärstruktur des Proteins
räumlich in der Umgebung liegen. In diesem Fall sind drei Aminosäuren als potentielle
Bindungsstellen in der Illustration hervorgehoben, die sterisch für eine tridentate faciale
Koordination bevorzugt sein könnten. Die Anordnung spricht für eine D27-Koordination.
Das Massenspektrum des Peptids eines ebenfalls stressregulierenden Proteins sucC (succinylCoA synthetase beta chain) ist in der folgenden Abbildung dargestellt. Es handelt sich hierbei
um die Sequenz [372K*GDLTDAAQQVVAAVEGK388]2+, in welcher an der Aminosäure
Lysin K372 Ruthenium koordiniert.
Proteintargets in Escherichia coli
58
Abb.5.5: MudPIT-MS/MS von [K*GDLTDAAQQVVAAVEGK]2+ aus sucC
Das Spektrum enthält insgesamt 19 der 32 möglichen b+- und y+-Ionen, wovon elf aus der b+und acht aus der y+-Serie stammen. Nur 18 dieser Ionen wurden für die SEQUESTAuswertung eingesetzt. Die Vergrößerung in dem Massenspektrum zeigt das b9+-Ion, welches
das Pattern des koordinierten Rutheniums aufweist. Bei einer K372-Koordination sind alle b+und keine der y+-Ionen rutheniert. In der angegebenen Sequenz kommen außer der
Aminosäure K372 auch die Seitenketten von T375 und D376 als potentielle
Koordinationsstellen für Ruthenium vor (siehe Tabelle 5.5).
Proteintargets in Escherichia coli
59
Tab.5.5: SEQUEST-Parameter von [K*GDLTDAAQQVVAAVEGK]2+ aus sucC im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
A.K*GLTDAAQQVVAAVEGK.
A.KGLT*DAAQQVVAAVEGK.
A.KGLTD*AAQQVVAAVEGK.
V.MoxGKAVETED*EDWNF.
K.GMLTTDVESYDKLSLD.Q
V.MoxGKAVE*TEDEDWNF.
T.MAADSS*S*TVM*ELLK.L
T.T*MAADSSSTVM*ELLK.L
K.TTMAADSSSTVM*ELLK.L
R.GMoxSLPM*FGKLM*ATNA.A
XCorr
3,105
2,776
2,747
2,525
2,489
2,469
2,444
2,435
2,43
2,386
∆Cn
0
0,106
0,115
0,187
0,198
0,205
0,213
0,216
0,218
0,232
Sp RSp Ionen
Protein
829,4 1 18/32 SUCC_ECOLI
581,6 2 16/32 SUCC_ECOLI
554,3 3 16/32 SUCC_ECOLI
273,2 82 11/26 RIR4_ECOLI
457,6 4 15/30 PITA_ECOLI
232,8 179 10/26 RIR4_ECOLI
257,3 120 12/26 YDJF_ECOLI
363,8 21 14/28 YDJF_ECOLI
339,5 28 14/30 YDJF_ECOLI
305 50 12/28 ALN_ECOLI
Die Tabelle listet zehn Sequenzen mit der Masse m/z = 1787 (∆m ± 2,5) auf, dabei stammen
die drei Peptide, deren Parameter die Kriterien erfüllen, alle aus dem sucC Protein. Im Falle
der Koordination des Threonins 375 sollte das b3+-Ion nicht das typische Rutheniumpattern
aufweisen, bei der Asparaginsäure 376 auch das b4+-Ion nicht. Da die charakteristische
Verteilung jedoch im MS/MS-Spektrum (Abb.5.5) für beide Ionen klar erkennbar ist, sind
diese Bindungsstellen als unwahrscheinlich einzustufen. Auch nach der ∆Cn-Begrenzung
(∆Cn < 0,10) können diese Stellen vernachlässigt werden.
Aus der Betrachtung der Seitenketten der oben genannten Aminosäuren in der Proteinstruktur
kann jedoch festgestellt werden, dass neben K372 die Seitenketten der Aminosäuren T375
und D376 auch räumlich ebenfalls potentielle Bindungsstellen für den Rutheniumkomplex
darstellen können. Eine gleichzeitige Beteiligung aller drei Stellen (Abb.5.6) an der Bindung
(z. B. unter Bildung eines Chelatkomplexes) wäre ebenfalls möglich. Die Abbildung 5.6 stellt
die Struktur des etwa 41 kDa Proteins sucC aus E. coli von Aminosäure Methionin 1 bis
Valin 385 dar.
Proteintargets in Escherichia coli
60
Abb.5.6: Die Gesamtstruktur des Proteins sucC (PDB ID: 2scu, [98])
In der Gesamtstruktur wird deutlich, dass die Seitenketten der denkbaren Bindungsstellen
sterisch gut zugänglich an der Oberfläche des Proteins lokalisiert sind. Die Succinyl-CoASynthetase besteht aus zwei Untereinheiten, einer α- und einer β-Untereinheit. Die β-chainUntereinheit ist als Enzym aktiv, wenn alle Substratbindungsseiten belegt sind und katalytisch
kooperieren, sofern unterschiedliche Substrate wechselwirken und katalysiert werden sollen.
In der Vergrößerung (Abbildung 5.7) wird die räumliche Nähe der potentiellen Seitenketten
offensichtlicher. Es sind neben den drei oben genannten Aminosäuren (K372, T375 und D
376) auch die Seitenketten der Aminosäuren Q379 und Q380 gekennzeichnet.
Proteintargets in Escherichia coli
61
Abb.5.7: Mögliche Koordinationsstellen des [(η6-p-Cymol)Ru]2+-Fragmentes in dem Protein
sucC
Aus der Vergrößerung des koordinierten Bereiches wird erkennbar, dass auf Grund
vergleichbarer Zugänglichkeit der Seitenketten z. T. mehrere Bindungsstellen für ein
Metallfragment nicht ausgeschlossen sind.
5.2. Proteintargets von [Pt(dien)(H2O)](NO3)2
Die im Folgenden beschriebene Analyse erfolgte nach der Umsetzung des E. coli Bakteriums
mit dem Komplexkation [Pt(dien)(H2O)]2+. Der Komplex [PtCl(dien)Cl] ist als nicht
zytotoxische Verbindung [99] in der Literatur diskutiert/verwendet worden und eignet sich
besonders für Modelluntersuchungen von Platin(II)-Komplexen wegen seines tridentaten
Liganden dien (Diethylentriamin). Da für die weiteren Reaktionen mit Biomolekülen die
Hydrolyse des Komplexes [Pt(dien)Cl]Cl der geschwindigkeitsbestimmende Schritt ist [57],
wurde im Rahmen dieser Arbeit die Aqua-Spezies [Pt(dien)H2O]2+ für die Reaktionen
gewählt. Bei höheren pH-Werten (pH > 4) dominieren das [Pt(dien)(OH)]+ und
hydroxoverbrückte Spezies [100]. Da bei beiden Verbindungen die Reaktionsgeschwindigkeit
erhöht ist, wurde für die Inkubation mit E. coli eine kurze Zeit von 30 min gewählt.
Nach der SEQUEST-Analyse der in dieser Messung detektierten Peptide des Ansatzes
[Pt(dien)]2+/E. coli konnte nach Überprüfung der einzelnen Spektren um falsch positive
Zuordnungen zu vermeiden eine Tabelle von sieben platinierten Peptidsequenzen, die unter
Proteintargets in Escherichia coli
62
5.6 zu finden ist, aufgestellt werden. Berücksichtigt wurden Modifikationen mit der Masse
296 des [Pt(dien)]2+-Fragmentes, mit Abzug von zwei Protonen für den Ladungsausgleich.
Tab.5.6: Proteintargets von [Pt(dien)]2+ in E. coli
Protein/Peptid
Bindungs- z XCorr ∆Cn
stelle
1 RL7_ECOLI
[email protected]
M15
2 ALDB_ECOLI
[email protected]
E427
3 SSB_ECOLI
R.PQQSAPAAPSNEPPMDFD@DDIPF.
D172
4 YICC_ECOLI
[email protected]
M97
5 EFTU_ECOLI
[email protected]
E343
6 RBSA_ECOLI
[email protected]
E2
7 DNAK_ECOLI
[email protected]
E429
@ = [Pt(dien)]2+
Sp
RSp Ionen
3
7,20
0,69 2648,3
1
43/96
3
5,94
0,54 2236,7
1
41/88
3
3,86
0,43 824,4
1
33/88
2
2,85
0,25 771,1
1
17/28
3
6,91
0,39 1761,5
1
38/92
2
3,16
0,10 534,3
6
13/30
3
5,40
0,50 1544,6
2
33/92
Die Tabelle enthält sieben Proteine, welche die eingestellten SEQUEST-Bedingungen
erfüllen. Dabei muss der XCorr- Wert mindestens 1,8 für einfach, 2,5 für zweifach und 3,5 für
dreifach geladene Moleküle, der Sp-Wert mindestens 500, der RSp-Wert weniger als 10 und
die Zuordnung der Ionen im Massenspektrum aus der Peptidequenz mindestens 30%
betragen. Die MS/MS-Spektren der von SEQUEST aufgelisteten Proteine wurden gesondert
betrachtet und auf das Vorliegen von Platinmuster in, als platiniert zugeordneten
Fragmentionen sowie auf Unterschiede zu anderen möglichen platinierten oder nicht
platinierten Peptiden, welche derselben Masse entsprechen, geprüft. Es wurden als potentielle
Koordinationsstellen die Seitenketten der Aminosäuren M, H, C, D, E, S, T, K, W und Y
berücksichtigt.
Das Beispiel aus der Proteinliste (Tab.5.6), welches diese Kriterien am besten erfüllt, ist das
13 kDa Protein rl7 (50S ribosomal protein L7/L12). Das Protein bietet in der Zelle die
Bindungsstelle für Faktoren, die an der Proteinsynthese beteiligt sind. Bei der
massenspektrometrischen
Analyse
wurde
das
Fragment
der
Sequenz
[6DQIIEAVAAM@SVMDVVELISAMEEK30]3+ identifiziert. Das Peptid ist an der
Proteintargets in Escherichia coli
63
Aminosäure Methionin 15 von dem [Pt(dien)]2+-Fragment koordiniert. Das zugehörige
MS/MS-Spektrum ist in der folgenden Abbildung dargestellt.
Abb.5.8: MudPIT-MS/MS-Spektrum von [DQIIEAVAAM@SVMDVVELISAMEEK]3+ aus
rl7
Von 96 möglichen Fragmentionen (für einfach und zweifach geladene Ionen) des Peptids sind
17 (nur einfach geladene in Abbildung 5.8 dargestellt, SEQUEST teilt insgesamt 43 Ionen zu)
in dem Massenspektrum zugeordnet worden. Da der eingesetzte Komplex als Metallzentrum
Platin enthält, welches eine charakteristische Isotopenverteilung seiner drei Hauptisotope
besitzt, können die entsprechenden Fragmente des Peptids auf das Vorhandensein des Musters
geprüft werden. Dabei sollten bei der Koordination dieser Aminosäure M15 die b+-Ionen ab
b10+ und die y+-Ionen ab y16+ das Platinmuster aufweisen. In dem oberen Spektrum (Abb.5.8)
ist exemplarisch die Verteilung des b10+-Ions vergrößert eingefügt. Das Isotopenmuster für
b10+, b12+ und b15+ steht im Einklang mit einer M15-Koordination. Die y+-Ionen ab y16+
werden nicht beobachtet, was ebenso auf die Bindung von M15 hindeutet.
In der unten dargestellten Tabelle sind die SEQUEST-Werte und die anderen möglichen
Sequenzen zu dieser Fragmentmasse von m/z = 3019 (∆m ± 2,5) aufgelistet.
Proteintargets in Escherichia coli
64
Tab.5.7: SEQUEST-Parameter von [DQIIEAVAAM@SVMDVVELISAMEEK]3+ aus rl7 im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
XCorr ∆Cn
Sp RSp Ionen
Protein
[email protected]
7,198
0
2648,3 1
43/96 RL7_ECOLI
[email protected]
7,115 0,012 2282,7 3
41/96 RL7_ECOLI
[email protected]
6,601 0,083 2356,3 2
40/96 RL7_ECOLI
[email protected]
4,296 0,403 1128,3 4
32/96 RL7_ECOLI
[email protected]
3,767 0,477 798,1
5
28/96 RL7_ECOLI
[email protected]
2,241 0,689 195,8 52 18/92 RFAK_ECOLI
[email protected]
2,204 0,694 138,8 263 17/96 RNFC_ECOLI
R.QMLMoxSDSCKIAIAAGIDDPQNPIGTDAVK.V 2,193 0,695 263,1 15 22/112 DHAM_ECOLI
[email protected]
2,172 0,698 205,6 40 18/88 EUTR_ECOLI
R.QMoxLMSDSCKIAIAAGIDDPQNPIGTDAVK.V 2,166 0,699 298,4 10 23/112 DHAM_ECOLI
Es sind Fragmente aus fünf unterschiedlichen Proteinen mit den jeweiligen Peptiden in der
Tabelle eingetragen. Dabei enthält das Proteinfragment des rl7 zwei weitere Aminosäuren
S16 und M18, die als potentielle Bindungsstellen für den Metallkomplex nach der SEQUESTAnalyse in Betracht kommen. Beim Prüfen der Zuordnungen, der SEQUEST-Werte und der
einzelnen Ionen des Peptids wird jedoch deutlich, dass die Aminosäure M15 des rl7Fragmentes als Bindungsstelle zugeordnet werden kann. Durch das Vorliegen des nicht
platinierten y15+-Ions für die Sequenz von S16-K30 kann zu Gunsten von M15 statt S16
entschieden werden, obwohl S16 einen XCorr-Wert aufweist, der nur geringfügig kleiner ist
als der für die M15-Koordination. Einen zusätzlichen wichtigen Hinweis darauf gibt der
Abbruch der Fragmentierung in der y+-Ionen-Reihe (ab y15+), welcher häufig durch das
koordinierte Metall verursacht wird. Dessen positive Ladung kann zusätzliche Spaltungen in
der Peptidsequenz nach sich ziehen. Die Peptide der restlichen vier Proteine (rfaK, rnfC,
dhaM und eutR) aus der Tabelle 5.7 können auf Grund der geringen Übereinstimmung mit
dem theoretischen MS/MS-Spektrum und der daraus resultierenden niedrigen SEQUESTParameter (∆Cn ≥ 0,69, Sp < 300, RSp ≥ 10) eindeutig vernachlässigt werden.
Die Bindungsstelle des Metallfragmentes an die Seitenkette des Methionins M15 kann in der
bekannten Struktur des Proteins dargestellt werden. Aus der Sequenz des Proteins ist der
Bereich Serin 2 bis Lysin 121 einbezogen. Abbildung 5.9 zeigt die Proteinstruktur unter
besonderer Kennzeichnung der Aminosäure M15.
Proteintargets in Escherichia coli
65
M15
Abb.5.9: Die Proteinstruktur von rl7 (PDB ID: 1ctf [101])
Die Seitenkette der Aminosäure ist wie in der Darstellung erkennbar sterisch gut zugänglich,
da sie in einer Schleife zwischen zwei Helices liegt und koordiniert werden kann.
Ein weiteres mit [Pt(dien)]2+ modifiziertes Protein ist das 43 kDa große Protein eftU
(elongation factor Tu). Es ist in der Zelle für die GTP -abhängige Bindung der AminoacyltRNA an die Ribosome während der Proteinbiosynthese zuständig. Eine Beteiligung des
Proteins an der Zellwachstumsregulierung wird vermutet. Es besteht aus 394 Aminosäuren.
Detektiert wurde bei der MudPIT- Analyse des Pt(dien)2+-E coli- Ansatzes das Fragment der
Sequenz [335TTDVTGTIE@LPEGVEMVMPGDNIK358]3+, welches die Modifikation an der
Aminosäure Glutaminsäure E343 enthält. Das MS/MS-Spektrum des 24 Aminosäuren langen
Peptids ist in der Abbildung 5.10 dargestellt.
Proteintargets in Escherichia coli
66
Abb.5.10: MudPIT-MS/MS-Spektrum von [TTDVTGTIE@LPEGVEMVMPGDNIK]3+ aus
eftU
Das Peptid enthält 92 Ionenfragmente (einfach und zweifach geladene Ionen), davon sind 17
einfach geladene Ionen im Massenspektrum in Abb.5.10 (insgesamt 38 Ionen von SEQUEST)
zugeordnet worden. Es sind sieben b+- und zehn y+-Ionen einbezogen. Die Serie der b+-Ionen
ab dem b9+ sollte bei einer Koordination an der Glutaminsäure 343 das charakteristische
Platinpattern des Metallfragmentes enthalten, aus der y+-Ionen-Serie y16+ bis y23+, wobei nur
die Ionen b10+ und b15+ beobachtet werden. Im oberen Spektrum ist das Isotopenmuster für das
b10+-Ion in der Vergrößerung gezeigt, welches eindeutig für eine Platinierung an E343 oder an
einer N-terminal zu diesem liegenden Aminosäure spricht.
In der nachfolgenden Tabelle ist eine Liste der zehn Peptide aufgestellt, welche nach den
SEQUEST-Parametern in Frage kommen.
Proteintargets in Escherichia coli
67
Tab.5.8: SEQUEST-Parameter von [TTDVTGTIE@LPEGVEMVMPGDNIK]3+ aus eftU im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
[email protected]
XCorr
6,912
6,336
5,878
5,618
5,15
5,13
4,859
4,791
4,724
4,223
∆Cn
0
0,083
0,15
0,187
0,255
0,258
0,297
0,307
0,317
0,389
Sp RSp Ionen
Protein
1761,5 1 38/92 EFTU_ECOLI
1550,2 2 37/92 EFTU_ECOLI
1255,7 4 33/92 EFTU_ECOLI
1291,7 3 36/92 EFTU_ECOLI
715,3
9 27/92 EFTU_ECOLI
657,2 10 26/92 EFTU_ECOLI
909
5 33/92 EFTU_ECOLI
843,7
7 32/92 EFTU_ECOLI
881,3
6 32/92 EFTU_ECOLI
396,3 55 22/92 EFTU_ECOLI
In der Peptidsequenz des eftU Proteins sind neun weitere Aminosäuren als potentielle
Bindungsstellen vorhanden. Die SEQUEST-Werte zeigen, dass nur zwei Sequenzen aus der
Tabelle abgelehnt werden würden, wenn nur die Parameter XCorr, Sp und RSp (XCorr > 3,5;
Sp ≥ 500; RSp ≤ 10) betrachtet werden. Jedoch weisen die platinierten Sequenzen 3-10 aus
Tabelle 5.8 alle ∆Cn > 0.1 gegenüber Sequenz 1 auf. Es verbleibt somit nur T341 als weitere
potentielle Bindungsstelle, wenn die Spektrenübereinstimmung berücksichtigt wird. Die
Ionen b7+ und b8+ im Massenspektrum in Abb. 5.10 sollten bei der Koordination von T341 mit
[Pt(dien)]2+ platiniert vorliegen, was nicht eindeutig bestätigt werden konnte. Einen Hinweis
auf die Koordination von E343 liefert jedoch das Fehlen der Ionen yk+ für k > 15.
In der nachstehenden Abbildung ist die Struktur des Proteins (Aminosäuren Threonin 8 bis
Serin 394) dargestellt.
Proteintargets in Escherichia coli
68
E343
T341
Abb.5.11:Die Proteinstruktur von eftU aus E. coli mit der Kennzeichnung von T341 und E343
(PDB ID: 1efc [102])
Die gekennzeichneten Seitenketten von T341 und E343 liegen, wie in der Illustration
erkennbar ist, für eine Koordination sterisch günstig. Sowohl die Glutaminsäure 343 als auch
das Threonin 341 sind in einem β-Faltblatt lokalisiert.
Ein Peptid des Proteins ssb (single- stranded DNA- binding protein) wurde aus dem Ansatz
[Pt(dien)]2+/E. coli Bakterium ebenfalls mit einer koordinierten Aminosäure detektiert. Es
handelt sich hierbei um ein 19 kDa Protein mit 178 Aminosäuren, welches in der Zelle
essentiell für die Chromosomenreplikation ist. Es ist somit in die DNA-Rekombination und Reparatur einbezogen. Aus der Gesamtsequenz des ssb Proteins wurde das Peptid
[155PQQSAPAAPSNEPPMDFD@DDIPF177]3+ mit der Modifikation der Asparaginsäure
D172 identifiziert. Das zugehörige Massenspektrum ist in Abbildung 5.12 dargestellt.
Proteintargets in Escherichia coli
69
Abb.5.12: MudPIT-MS/MS-Spektrum von [PQQSAPAAPSNEPPMDFD@DDIPF]3+ aus ssb
Im MS/MS-Spektrum zugeordnet sind 15 Ionen (nur einfach geladene dargestellt, 33
insgesamt zugeordnet) von 88 (einfach und zweifach geladene Moleküle) möglichen Ionen.
Die Ionen b18+ bis b22+ sollten die typische Verteilung des koordinierten Metalls enthalten,
werden im Spektrum jedoch nicht beobachtet. Aus der y+-Serie ist das Muster bei den Ionen
y6+ bis y22+ zu erwarten, beobachtet werden die y2+, y6+, y8+ sowie y10+- y12+-Ionen. Im
MS/MS-Spektrum des Peptids (Abbildung 5.12) sind zwei Ausschnitte der Ionen mit
unterschiedlichen Ionenverteilungen dargestellt. Das b8+-Ion trägt, wie erwartet keine
Isotopenverteilung des Platins, wobei die vergrößerte Darstellung des y11+-Ions das erwartete
Pattern aufweist.
Die folgende Tabelle listet die mit der Massengenauigkeit von 2,5 vorkommenden Peptide
unterschiedlicher Proteine auf.
Proteintargets in Escherichia coli
70
Tab.5.9: SEQUEST-Parameter von [PQQSAPAAPSNEPPMDFD@DDIPF]3+ aus ssb im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
R.PQQSAPAAPSNEPPMDFD@DDIPF.
R.PQQSAPAAPSNEPPMDFDDD@IPF.
R.PQQSAPAAPSNEPPMDFDD@DIPF.
R.PQQSAPAAPSNEPPM@DFDDDIPF.
R.PQQSAPAAPSNEPPMD@FDDDIPF.
R.PQQSAPAAPSNE@PPMDFDDDIPF.
R.PQQSAPAAPS@NEPPMDFDDDIPF.
[email protected]
[email protected]
[email protected]
XCorr
3,863
3,804
3,777
3,715
3,628
3,174
3,099
2,368
2,272
2,209
∆Cn
0
0,015
0,022
0,038
0,061
0,179
0,198
0,387
0,412
0,428
Sp RSp Ionen
Protein
824,4 1 33/88 SSB_ECOLI
690,4 4 31/88 SSB_ECOLI
755,9 2 32/88 SSB_ECOLI
658,7 5 30/88 SSB_ECOLI
725
3 31/88 SSB_ECOLI
535,5 6 28/88 SSB_ECOLI
476,1 7 26/88 SSB_ECOLI
207,8 57 22/84 YNEI_ECOLI
191,7 94 21/84 YNEI_ECOLI
193,6 86 21/84 YNEI_ECOLI
Daraus lässt sich erkennen, dass aus dem Protein ssb außer D172 eine Reihe weiterer
potentiellen Koordinationsstellen (D174, D173, M169, D170 mit ∆Cn < 0,1) in Frage
kommen. Neben den Asparaginsäuren-Seitenketten können Methionin, Glutaminsäure oder
Serin aus ssb prinzipiell gebunden werden. Aus der Verteilung im Massenspektrum und den
SEQUEST-Werten aus der Tabelle 5.9 wird deutlich, dass neben D172 auch eine
Koordination von D173 oder D174 aus ssb möglich wäre. Nach dem Parameter ∆Cn ≤ 0,1
kommen außerdem auch D170 und M169 in Betracht. Da bj+-Ionen mit j > 13 und yk+-Ionen
mit 6 > k > 2 fehlen, kann nicht eindeutig zwischen den drei D-Resten D172-D174
entschieden werden.
5.3. Proteintargets von cis-[PtCl2(NH3)2]
Das in der medizinischen Praxis am häufigsten eingesetzte Krebstherapeutikum aus der
Klasse der Metallkomplexe ist Cisplatin [41]. Zu dem Reaktionsverhalten von Cis- und
Carboplatin mit Biomolekülen sind, wie im Kapitel 3 erwähnt, zahlreiche Studien
durchgeführt worden, jedoch ist die Rolle ihrer Wechselwirkungen mit den Proteinen bisher
kaum geklärt. Die MudPIT-Analysen mit diesem Metallkomplex sollen einen Beitrag zum
besseren Verständnis leisten. Dazu wurde wiederum das Darmbakterium E. coli als Modell
für eine lebende Zelle mit dem Zytostatikum umgesetzt und auf Modifikationen der Proteine
untersucht.
Welche Fragmente aus dem Metallkomplex gebildet werden und koordinieren können, wurde
in den Modellstudien (Kapitel 4) untersucht. Es sind Fragmente mit den folgenden Massen
Proteintargets in Escherichia coli
71
möglich: [195Pt]2+ mit 193, [(NH3)195Pt]2+ mit 210, [(NH3)2195Pt]2+ mit 227 und
[(NH3)2195PtCl]+ mit 263. Bei den jeweiligen Massen wurde der Ladungsausgleich mit der
Abgabe von Protonen berücksichtigt.
Zu den einzelnen Massen erscheinen in den SEQUEST-Listen mehrere Proteine, die
mindestens ein modifiziertes Peptid enthalten. Aus diesen Listen wurden zusätzlich zu den
SEQUEST-Werten, wie im Experiment mit dem Ruthenium- und [Pt(dien)]2+-Komplex, die
jeweiligen Massenspektren zur Beurteilung herangezogen, um eine Endliste mit den
modifizierten Proteinen zu erhalten. Die so erhaltenen Proteintargets können in fünf Gruppen
(DNA- und RNA- bindende Proteine, stressregulierende Proteine, katalytisch funktionierende
Proteine, Effluxproteine und Proteine mit anderen Funktionen) gegliedert werden, die nach
ihren Funktionen eingeteilt wurden. Diese Aufteilung ist in der unteren Tabelle gezeigt.
Tab.5.10: Proteintargets von Cisplatin in E. coli (Teil I, Teil II auf Seite 72)
Protein
arti
eftU
Peptidsequenz
DNA- und RNA-bindende Proteine
R.HYEIVFM§VHPDQSEQVPGMIER.Y
K.MoxECID~NEVVVTVNEKLK.D
T.C~DGYEIEEGEFR.I
R.FMELIDALAQE~TADMPL.H
S.FISDILYADIE~SK.A
V.PNMS~HSEIMoxEDIKKA.
K.MoxRPGD#VLGALTGDIGLDGADIG.K
K.MoxRPGD~VLGALTGDIGL.D
R.RM#LIITGPNMGGKSTYM.R
Stressregulierende Proteine
.M§QGSVTEFLKPR.L
E.MoxGYDWLGRMoxPY§K.G
A.M#LQDIATLTGGTVISEEIGMELEK.A
I.VMoxDDADLELAVK#AIVDSR.V
Effluxproteine
R.QE§LDAQQALVSETEGTIKADEASV.A
R.KAQALGVAID#DINDTLQ.T
Proteine mit anderen Funktionen
K.FIM§DKHPEITTVPYDSYQNAK.L
K.MVVTLIHPIAM§DDGLR.F
pthP
mukE
fimA1
ceaN
.M§FQQEVTITAPNGLHTR.P
R.STT#LIPRSVLSELDMMVG.K
A.LSLS#STAALAAATTVNGGTVHFK.G
K.LIDAESIK#GTEVYTF.H
rs6
finQ
top1
uvrD
rep5
intD
dbpA
muts
rpoA
ompA
ch60
aldA
mdtA
acrD
Bindungsstelle
z
XCorr ∆Cn Ionen
M9 (E5, Y4, H3)
D193 (C191, E190)
C689
E471 (T472)
E15(S16, K17)
S376
D399
D399
M609
3
2
2
2
2
2
3
2
2
6,50
4,01
2,81
2,56
2,74
2,54
4,17
2,88
2,97
0,38
0,13
0,47
0,13
0,14
0,13
0,19
0,1
0,1
35/84
16/32
12/22
16/32
13/24
15/28
26/84
15/30
14/32
M1 (S4)
Y84 (K85)
M288(D291)
K271
2
2
2
2
2,89
2,54
5,20
3,09
0,24
0,12
0,51
0,14
13/22
12/22
24/46
15/34
E164 (D166)
D742(D743)
2
2
3,48
3,06
0,40 18/46
0,28 15/32
M142
M369 (M359, H365,
T362)
M1 (T9)
T90(T89,S88,S96)
S17 (S15, S18, T19)
K129
3
2
4,41
3,59
0,37 33/80
0,32 16/30
2
2
2
2
3,58
3,75
2,98
2,60
0,38
0,14
0,13
0,14
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
20/32
18/34
19/44
15/28
Proteintargets in Escherichia coli
72
Tab.5.10: Proteintargets von Cisplatin in E. coli (Fortsetzung von Seite 71)
Protein
enO
g3p1
cisY
ebgC
tdcE
his2
speA
yciA
mdH
thiO
dceA
Peptidsequenz
Bindungsstelle
Katalytisch funktionierende Proteine
R.GNPT§VEAEVHLEGGFVGMAAAPSGASTGSR.E T20 (E22, E24, H26)
K.RSD§IEIVAINDLLDADYMAYMLK.Y
D26 (S25, E28)
R.HTM§IHEQITR.L
M113
V.HE§GQIVICDIHEAYR.F
E112 (H111)
K.T§APLMoxDDVLDYDKVMDSL.D
T467
H.AVSGEVLMLGYM$NPEALDK.T
M37 (Y36)
R.MoxADKMYVNFS$LFQSMPD.A
S490
L.MoxSQMDIGGAILAKEIAH~GR.V
H51
K.SIGTLSAFEQNALEGMLDT~.L
T298 (D297)
I.H~LTDDSFDTDVLK.A
H7
F.EMDFAELLLEDY#K.A
Y441(K442,D440)
z
XCorr ∆Cn Ionen
3
3
2
2
6,88
6,06
3,62
3,07
3,09
3,84
3,11
3,63
3,56
3,56
3,09
2
2
2
2
2
2
2
0,52 41/116
0,53 37/88
0,39 15/18
0,13 15/28
0,23
0,13
0,22
0,18
0,28
0,22
0,25
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
Es werden einzelne Proteine mit relevanten Funktionen und/oder bekannten Kristallstrukturen
aus den Funktionsklassen beschrieben.
Stressregulierende Proteine
Aus der Gruppe der stressregulierenden Proteine konnten vier koordinierte Peptidsequenzen
aus vier Proteinen ch60, aldA, ompA und rpoA identifiziert werden.
In der unteren Abbildung ist das Massenspektrum für das Peptid des Proteins aldA (aldehyde
dehydrogenase A) aus dem Bakterium E. coli dargestellt. Das aus 479 Aminosäuren
bestehende, etwa 52 kDa große Protein katalysiert Oxidationen der Aldehydsubstrate zu den
korrespondierenden
Carboxysäuren
[260VMoxDDADLELAVK#AIVDSR277]2+
[103].
wurde
Das
mit
Peptid
dem
mit
Fragment
der
Sequenz
[(NH3)2PtCl]+
modifiziert. Mox steht für den Mthionin-Rest, der häufig unter MS-Bedingungen gebildet
wird und eine Masse [Methionin]+16 aufweist.
17/34
16/36
14/32
17/36
18/36
22/24
14/24
Proteintargets in Escherichia coli
73
Abb.5.13: MudPIT-MS/MS-Spektrum von [VMoxDDADLELAVK#AIVDSR]2+ aus aldA
Das Massenspektrum enthält 16 zugeordnete b+- und y+- Ionen, wobei insgesamt aus der
Sequenz 34 vorkommen könnten. Bei der SEQUEST-Auswertung wurden 15 dieser Ionen
berücksichtigt. Von den neun einbezogenen b+- Ionen sind für eine K271-Koordination b12+b17+ platiniert. Bei den sieben einbegriffenen y+- Ionen lässt sich dieses Verhalten ebenfalls
für y7+ bis y17+ beobachten. In der oberen Abbildung ist die charakteristische
Isotopenverteilung am Beispiel des y11+-Ions in der Vergrößerung verdeutlicht.
In einem zusätzlichen Ausschnitt aus dem Massenspektrum (Abb. 5.14) wird auf das y11+-Ion
besonders eingegangen. Am Beispiel dieses Ions kann sowohl das Fragmentierungsmuster des
Cisplatins als auch die Abspaltung der einzelnen Liganden bekräftigt werden.
Proteintargets in Escherichia coli
74
Abb.5.14: y11+-Ion aus [VMoxDDADLELAVK#AIVDSR]2+ aus aldA
Das an die Aminosäure K271 koordinierte Cisplatinfragment ist [(NH3)2PtCl]+. Wie aus der
Abbildung ersichtlich, ist die neutral loss-Abspaltung von beiden Ammoniakliganden
eindeutig erkennbar. Die Beobachtung dieses Verhaltens kann bei der Zuordnung und
Bewertung weiterer Massenspektren als hilfreiches Kriterium hinzugezogen werden. Die
erfolgte SEQUEST-Zuordnung bestätigt die für die Modellpeptide festgestellte Möglichkeit
einer erfolgreichen Analyse von b+- und y+-Ionen trotz begleitender neutral loss.
Zu dem Peptid aus dem Protein aldA ist in der folgenden Tabelle eine Darstellung der
SEQUEST-Werte der Peptide zu der Masse von m/z = 2240 (∆m ± 2,5) zu sehen.
Tab.5.11: SEQUEST-Parameter von [VMoxDDADLELAVK#AIVDSR]2+ aus aldA im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
I.VMoxDDADLELAVK#AIVDSR.V
D.KMDTLAGIFGIGQH#PK#.G
I.VMoxDDADLELAVKAIVD#SR.V
K.D#TQPVTGQSPGQFRDVPF.G
K.DT#QPVTGQSPGQFRDVPF.G
-.MLRNGNKYLLMoxLVSIIMLT.A
-.MoxLRNGNKYLLMLVSIIMLT.A
XCorr
3,088
2,643
2,559
2,455
2,449
2,435
2,395
∆Cn
0
0,144
0,171
0,205
0,207
0,212
0,225
Sp RSp Ionen
Protein
595,6 1 15/34 ALDA_ECOLI
439,5 12 13/30 SYGB_ECOLI
407,3 23 13/34 ALDA_ECOLI
354,1 34 12/34 SGBU_ECOLI
354,1 34 12/34 SGBU_ECOLI
321,2 53 12/36 PGAB_ECOLI
389,8 26 14/36 PGAB_ECOLI
Wie aus der Tabelle erkennbar ist, sind zwei Aminosäuren in dem Peptid aus dem Protein
aldA als unterschiedliche Modifikationen für eine Zuordnung der Massenpeaks möglich.
Neben dem Lysin K271 könnte die Asparaginsäure D275 auch koordiniert werden. Das
Platinpattern bei den Ionen b13+ und b14+ in dem Massenspektrum weist jedoch darauf hin,
dass eine Bindung des Metallfragmentes am Lysin vorliegen muss. Auch der hohe ∆Cn-Wert
Proteintargets in Escherichia coli
75
von 0,171 für eine D275-Koordination spricht gegen diese Bindungsstelle. In der Tabelle
kommen außerdem Peptide der Proteine sygb, apc, sgbu, pgab und apc mit den
entsprechenden Molekülmassen vor. Nach Betrachtung der SEQUEST-Werte wird jedoch
deutlich, dass diese Zuordnung der Peaks im Massenspektrum unzureichend ist.
Ein weiteres stressregulierendes Protein, welches nach der Umsetzung des Bakteriums mit
Cisplatin ein Platinfragment bindet, ist das Protein ompA (outer membrane protein A). Es
wurde bereits bei einer Umsetzung des Bakteriums mit Cisplatin als bindendes Protein
identifiziert [89]. In der Studie von DYSON wurde das 37 kDa große Protein mit dem
Platinfragment [(NH3)2PtCl]+ modifiziert gefunden. Jedoch gelang es seiner Arbeitsgruppe
nicht die Bindungsstelle zu ermitteln. Nach der Umsetzung im Rahmen dieser Arbeit wurde
ein Peptidfragment aus dem Protein mit Platin nach Abspaltung aller Liganden koordiniert
identifiziert. Wie zuvor erläutert kann das [Pt]2+-Fragment auch bei der MS-Ionisierung durch
Verlust des Chloridions bzw. durch neutral loss eines oder beider Amminliganden entstehen.
Das zugehörende Massenspektrum ist in Abbildung 5.15 dargestellt.
Abb.5.15: MudPIT-MS/MS-Spektrum von [MoxGYDWLGRMoxPY§K]2+ aus ompA
Proteintargets in Escherichia coli
76
Das Fragment des Proteins, welches mit dem Platinrest [Pt]2+ modifiziert wurde, besteht aus
der Sequenz [74MoxGYDWLGRMoxPY§K85]2+. In dem Massenspektrum sind 12 von 22
Ionen des Peptids zugeordnet. Die eingefügte Vergrößerung zeigt das Platinpattern des Ions
y9+, welches als Fragment des Peptids das Metall enthält. Die folgende Tabelle zeigt neun
weitere mögliche Zuordnungen anderer Peptide zu dem Massenspektrum.
Tab.5.12: SEQUEST-Parameter von [MoxGYDWLGRMoxPY§K]2+ aus ompA im Vergleich
zu anderen potentiellen Peptidsequenzen
Sequenz
E.MoxGYDWLGRMoxPY§K.G
E.MoxGYDWLGRMoxPYK§.G
R.ALLAY§RDGARIH§.A
-.M§GPVMoxLD§VEGYE.L
P.KLQIDFMoxTIHAS§K.G
-.M§GPVMoxLDVE§GYE.L
-.MoxGPVM§LD§VEGYE.L
P.KLQIDFMoxTIH§ASK.G
F.YE§LFY§D§DAK.R
K.RLPE§GSTLPWHRV.V
-.SLYCATSRDMoxIK§F.-
XCorr
2,713
2,477
2,407
2,394
2,257
2,245
2,185
2,164
2,155
2,139
1,521
∆Cn
0
0,087
0,113
0,118
0,168
0,173
0,195
0,203
0,206
0,212
0,44
Sp
535,4
463,5
468,2
421,5
409,2
287,1
423,4
409,2
338,4
286,7
734,8
RSp
12
28
27
52
56
302
50
56
147
307
1
Ionen
12/22
11/22
11/22
11/22
11/24
10/22
11/22
11/24
8/16
9/24
13/24
Protein
OMPA_ECOLI
OMPA_ECOLI
TETR3_ECOLI
NAGZ_ECOLI
HELD_ECOLI
NAGZ_ECOLI
NAGZ_ECOLI
HELD_ECOLI
MUTS_ECOLI
YBAZ_ECOLI
IUCA_ECOLI
Es sind in der Tabelle zwei Peptidfragmente aus dem Protein ompA vorhanden, welche
jeweils eine Modifikation aufzeigen (Tyrosin 84 und Lysin 85). Eine genaue Zuordnung der
Bindungsstelle kann in diesem Fall nicht erfolgen, da yk+-Ionen mit k < 3 nicht im MS/MSSpektrum (Abb. 5.15) dargestellt und die m/z-Werte für b11+ (M74-Y84) und y11+ (G75-K85)
identisch sind. Somit ist die Zuordnung von b11+unzuverlässig. Die Sequenzen der Peptide der
sechs weiteren Proteine können auf Grund ihrer niedrigen SEQUEST-Werte (∆Cn > 0,11) alle
ausgeschlossen werden. Das ompA Protein gehört mit seinen 346 Aminosäuren zu den
Hauptmembranproteinen von E. coli bzw. anderen gramnegativen Bakterien [104]. Es wird
diskutiert, dass die Aufgabe des Proteins die Bildung von Kanälen und Transport durch die
Membran sein könnten. In der folgenden Illustration ist die Struktur des Proteins mit der
eingekreisten Seitenkette der potentiell koordinierten Aminosäure Tyrosin 84 dargestellt.
Zusätzlich ist die Seitenkette von K85 hervorgehoben.
Proteintargets in Escherichia coli
77
K85
Y84
Abb.5.16: Proteinstruktur von ompA mit gekennzeichneten Seitenketten Y84 und K85
(PDB ID:1gjp [105])
In der Struktur des Proteins ist die Zugänglichkeit der Seitenkette des Tyrosins 84 in einer
Schleife klar zu erkennen und bestätigt somit die Bevorzugung dieser Position (bzw. K85).
Fehlende Stücke in den Schleifen der Proteinstruktur in der oberen Abbildung sind durch
Fehlstellen in der Kristallstruktur zu erklären.
DNA- und RNA-bindende Proteine
In der Gesamtliste der Proteintargets aus E. coli findet sich das Protein mutS, ein Protein aus
der mismatch repair -Gruppe (DNA mismatch repair protein mutS). Dabei handelt es sich um
ein etwa 95 kDa großes Protein, welches in der Region 614 bis 621 das ATP-Zentrum enthält.
ATP ist dabei ein Nucleotid, Adenosintriphosphat, ein energiereicher Baustein der DNA und
RNA. Außerdem ist ATP die Energiequelle einer Zelle und reguliert die energieliefernden
Prozesse darin. Bei der Hydrolyse von ATP entsteht das ADP (Adenosindiphosphat), ein
energiereiches Molekül in den Zellen. Aus dem Protein wurde massenspektrometrisch aus der
MudPIT-Analyse die halbtryptisch gespaltene Peptidsequenz
[608RM#LIITGPNMGGKSTYM624]2+ mit der [(NH3)2PtCl]+ Modifikation an der Stelle
Methionin M609 gefunden. Das zugehörige Massenspektrum ist nachfolgend dargestellt.
Proteintargets in Escherichia coli
78
Abb.5.17: MudPIT-MS/MS-Spektrum von [RM#LIITGPNMGGKSTYM]2+ aus mutS
Wie aus der Abbildung erkennbar, sind 10 b+- und 11 y+- Ionen von möglichen 32 Ionen im
Massenspektrum zugeordnet. Für die SEQUEST-Analyse wurden 14 Ionen verwendet. Die
Ionen b2+ bis b16+ und y16+ sollten das charakteristische Pattern des Metallzentrums des
koordinierten Fragmentes aufzeigen. In dem Massenspektrum ist eine Vergrößerung des Ions
b14+
eingefügt,
welche
die
charakteristische
Isotopenverteilung
des
koordinierten
Metallfragmentes verdeutlicht.
In der unteren Tabelle sind weitere Peptide aufgelistet, deren Masse mit m/z = 2133 der des
koordinierten Fragmentes aus mutS entspricht (∆m ± 2,5).
Proteintargets in Escherichia coli
79
Tab.5.13: SEQUEST-Parameter von [RM#LIITGPNMGGKSTYM]2+ aus mutS im Vergleich
zu anderen potentiellen Peptidsequenzen
Sequenz
R.RM#LIITGPNMGGKSTYM.R
D.RM#LDMoxGFSDAIDDVIR.F
R.H#TKMoxNVGDVQRLSVAVV.V
R.HT#KMoxNVGDVQRLSVAVV.V
P.TGLLNEMoxIAYLNS#EER.N
R.DGVGGNSSFAT#MoxIDREPT.Q
K.TASIPINSNLIVHVGNNVRI.D
R.ATLCILD#PRLLLDEKSA.S
D.WTFDLLDVYLAE#IDR.V
P.TGLLNE#MoxIAYLNSEER.N
-.TMRIID#T#NINVMDA.-
XCorr
2,791
2,504
2,477
2,456
2,283
2,245
2,24
2,227
2,216
2,146
2,051
∆Cn
0
0,103
0,113
0,12
0,182
0,196
0,198
0,202
0,206
0,231
0,265
Sp RSp Ionen
Protein
564,7 3 14/32 MUTS_ECOLI
467,3 20 13/30 DBPA_ECOLI
370,4 63 11/32 FLIF_ECOLI
370,4 63 11/32 FLIF_ECOLI
354,5 83 12/30 YDEO_ECOLI
362,9 73 12/34 YMFK_ECOLI
287,8 213 11/38 YTFN_ECOLI
432 27 12/32 YEIR_ECOLI
513,2 7 12/28 YCGB_ECOLI
327 130 12/30 YDEO_ECOLI
713,8 1 13/26 CDAR_ECOLI
Die Tabelle enthält neben dem Protein mutS zehn Sequenzen anderer Proteine, welche
ebenfalls von dem Cisplatinfragment [(NH3)2PtCl]+ koordiniert sein könnten. Darin kommt
ein Peptid vor, welches an zwei Aminosäuren jeweils das Metallfragment gebunden haben
sollte. Da diese mehrfache Modifizierung im Metallmuster der Ionen nicht feststellbar ist,
wird sie als Koordinationsmuster ausgeschlossen. Aus der detektierten Sequenz von mutS ist
außer Methionin 609 keine der potentiellen Aminosäuren (z. B. Threonin 613) als
Koordinationsstelle in der SEQUEST-Liste zu finden. Auf Grund des ∆Cn-Kriteriums (∆Cn >
0,10) können die restlichen Peptidsequenzen ausgeschlossen werden. Die Lokalisierung der
Aminosäure und ihrer Seitenkette kann in der Tertiärstruktur des Proteins mutS betrachtet
werden. Die Koordination ist räumlich in der folgenden Abbildung in der Gesamtstruktur des
Proteins dargestellt.
Proteintargets in Escherichia coli
80
ADP-Zentrum
Abb.5.18: Struktur des DNA mismatch repair Proteins mutS (PDB ID: 1e3m [106])
Das 95 kDa Protein liegt als Tetramer vor und enthält in der eingekreisten Region das ADPZentrum. In der folgenden Abbildung ist zur Verdeutlichung ein Ausschnitt aus der
Gesamtstruktur dargestellt, welcher die mit dem Cisplatinfragment modifizierte Stelle
(Methionin 609) und die ADP-Region zeigt.
Abb.5.19: Vergrößerung der Struktur des DNA mismatch repair Proteins mutS
Proteintargets in Escherichia coli
81
Die Modifikation der Aminosäure M609 liegt räumlich in der Nähe des Energiezentrums des
DNA-bindenden Proteins mutS, so dass bei der Koordination des Metallkomplexrestes eine
Beeinflussung dessen vermutet werden kann.
Im Zusammenhang mit mutS wird in der Literatur die DNA-Helikase uvrD (DNA helicase II)
diskutiert [107]. Dabei handelt es sich um ein Protein, welches an dem NER-Mechanismus
beteiligt ist. Der uvrD Helikase werden mehrere Funktionen zugeschrieben. Einerseits
kontrolliert sie die Rekombinase recA [108], andererseits entwindet sie die DNA und
vermittelt die Bindung der Polymerase I [109].
Das Massenspektrum des modifizierten Peptids [461FMELIDALAQE~TADMPL477]2+ der 720
Aminosäuren langen Helikase ist in der Abbildung 5.20 dargestellt. Wie an der
Kennzeichnung in der Sequenz erkennbar, wurde die Glutaminsäure E 471 koordiniert.
Abb.5.20: MudPIT-MS/MS-Spektrum von [FMELIDALAQE~TADMPL]2+ aus uvrD
Aus der Fragmentierung des Peptids ist ersichtlich, dass einer Koordination der
Glutaminsäure 471 bei den Ionen b11+ bis b16+ das Platinpattern bei den Massenpeaks
vorhanden sein muss. Bei den y+-Ionen ist das für die Ionen y7+ bis y16+ der Fall. Die
Proteintargets in Escherichia coli
82
Vergrößerung in der oberen Abbildung 5.20 zeigt das Ion y7+ mit der diskutierten
Isotopenverteilung. Von den 32 möglichen Ionen, werden in dem oberen Spektrum 18
Fragmentionen beobachtet, wobei SEQUEST 16 für die Analyse einbezieht.
Die SEQUEST-Parameter des Peptids aus dem Protein uvrD sind in der unteren Tabelle
dargestellt. Dabei sind zehn weitere Peptide aufgeführt, welche die Masse m/z = 2136 (mit
der Massengenauigkeit ∆m ± 2,5) tragen.
Tab.5.14: SEQUEST-Parameter von [FMELIDALAQE~TADMPL]2+ aus uvrD im Vergleich
zu anderen potentiellen Peptidsequenzen
Sequenz
R.FMELIDALAQE~TADMPL.H
R.FMELIDALAQET~ADMPL.H
R.FMELIDALAQETAD~MPL.H
D.FT~PGNSTANAELLC~DK.F
G.WY~IS~AEIDDLNWR.S
E.CGDS~LDSINATSS~AIVK.Y
E.CGD~SLDSINATSS~AIVK.Y
G.TKEGT~EEPWY~VVDK.D
E.C~GDSLDSINATSS~AIVK.Y
K.S~Y~WILMTVLLVTQN.G
-.VGCTS~KQSVSQC~VK~.-
XCorr
2,562
2,513
2,286
2,235
2,206
2,195
2,195
2,177
2,141
2,134
1,532
∆Cn
0
0,019
0,108
0,128
0,139
0,143
0,143
0,15
0,164
0,167
0,402
Sp RSp Ionen
Protein
671,3 3 16/32 UVRD_ECOLI
671,3 3 16/32 UVRD_ECOLI
666,7 4 16/32 UVRD_ECOLI
532,5 11 15/30 YZBC_ECOLI
498,7 13 14/24 YBCH_ECOLI
452,4 17 14/32 RIR1_ECOLI
452,4 17 14/32 RIR1_ECOLI
254,8 199 11/26 TRMU_ECOLI
452,4 17 14/32 RIR1_ECOLI
242,1 248 10/26 YHFK_ECOLI
825,4 1 15/26 RZOD_ECOLI
In der Tabelle sind die Zuordnungen der Massenspektren nach fallenden XCorr-Werten für
die einzelnen Peptidsequenzen aufgelistet. Für das uvrD Protein sind Modifikationen mit dem
Platinfragment an mehreren Aminosäuren (E471, T472 und D474) prinzipiell möglich. Die
Sp- und XCorr-Werte dieser Modifikationen unterscheiden sich nur unwesentlich
voneinander, wobei der ∆Cn-Wert für letztere (D474) das ∆Cn-Kriterium knapp nicht erfüllt
(∆Cn > 0,1). Auch anhand der zugeordneten b+-Ionen (bj+ ≤ b9+, b13+, b14+) und der y+-Ionen
(yk+ ≤ y5+, yk+ ≥ y7+) kann eine genaue Feststellung der modifizierten Aminosäure in dem
Protein nicht erfolgen, es kommen somit zwei Bindungsstellen in Frage: E471 und T472. Die
restlichen Peptide der sechs Proteine (uvrD, yzbC, rir1, trmU, yhfK und rzoD) aus der Tabelle
weisen alle mehrfache Koordinationen auf, für diese geben die Isotopenverteilungen der
Ionen im MS/MS-Spektrum jedoch keinen Hinweis. Zur Verdeutlichung der möglichen
Bindungsstelle E471 in der Gesamtstruktur des Proteins dient die folgende Abbildung. Das
Protein uvrD weist sowohl die Aktivität einer ATPase als auch die einer Helikase auf.
Proteintargets in Escherichia coli
83
E471
Abb.5.21: Die Proteinstruktur der DNA-Helikase (PDB ID: 1pjr [110])
Es ist die Struktur einer vergleichbaren DNA-Helikase (DExx box) gezeigt. In der Illustration
ist der Bereich der bekannten Kristallstruktur des Proteins zwischen Aminosäure M1 und
K680 zu sehen. Aus der Darstellung ist ersichtlich, dass die Seitenkette der Glutaminsäure
471 für eine Koordination sterisch gut zugänglich ist.
Die unten stehende Vergrößerung des koordinierten Bereiches dient zur Verdeutlichung der
Zugänglichkeit beider möglicherweise koordinierter Seitenketten der Aminosäuren E471 und
T472.
T472
E471
Abb.5.22: Vergrößerung der Strukutr von uvrD im Bereich der möglichen
Koordinationsstellen E471/T472
Proteintargets in Escherichia coli
84
Beide Aminosäuren sind in einer Helix lokalisiert und ihre Seitenketten sind, wie aus der
Abbildung erkennbar, leicht für ein koordinierendes Platinfragment zugänglich.
Ein weiteres DNA-bindendes Protein des Darmbakteriums, welches von Cisplatin koordiniert
wurde, ist eine DNA-Topoisomerase top1. Topoisomerase ist als Enzym an der Replikation
der DNA einer Zelle beteiligt und spielt dementsprechend im Zusammenhang mit Krebs eine
bedeutende Rolle. Die Topoisomerase I in E. coli ist zusammen mit zwei weiteren
Topoisomerasen an der Regulation des Überspiralisierungsgrades und der DNA-Topologie
beteiligt [111]. Mit einer Blockierung der Topoisomerase würde das unkontrollierte
Wachsen/Fortpflanzen der Zelle beeinflusst werden. Bei dem durchgeführten Experiment
bindet das Fragment [(NH3)2Pt]2+ des Cisplatins an die Aminosäure C689 des 97 kDa
Proteins. Das Massenspektrum des Peptids der Sequenz [689C~DGYEIEEGEFR700]2+ ist in der
Abbildung 5.23 dargestellt.
Abb.5.23: MudPIT-MS/MS-Spektrum von [C~DGYEIEEGEFR]2+ aus top1
Das Massenspektrum enthält von 22 möglichen Fragmentionen zugeordnete 12 Ionen. Alle
b+-Ionen des Peptids enthalten Platin als Fragment und weisen somit das charakteristische
Pattern auf. Dagegen fehlt dieses bei den y+-Ionen des Peptids. Der vergrößerte Ausschnitt
Proteintargets in Escherichia coli
85
des Ions y4+ weist der Koordinationsstelle des [(NH3)2Pt]2+-Fragmentes (C689) entsprechend
keine Platinverteilung auf. Im obigen Spektrum werden zwei der genannten b+-Ionen und alle
yk+-Ionen für k ≥ 2 beobachtet. Die folgende Tabelle zeigt die möglichen Sequenzen, welche
mit der gefundenen Masse von m/z = 1674 (∆m ± 2,5) aus dem Bakterium detektiert werden
könnten.
Tab.5.15: SEQUEST-Parameter von [C~DGYEIEEGEFR]2+ aus top1 im Vergleich zu
anderen potentiellen Peptidsequenzen
Sequenz
T.C~DGYEIEEGEFR.I
T.CD~GYEIEEGEFR.I
T.CDGY~EIEEGEFR.I
Y.LPKT~QQLQMGFR.K
V.Y~LFSGIVSALYGR.E
R.C~FMLCTSHAMMoxR.D
Y.LPK~TQQLQMGFR.K
T.CDGYE~IEEGEFR.I
K.ALGAPVELIKVHNTPD.G
E.HAIGM~MMoxTLNRR.I
XCorr
2,807
2,407
1,771
1,497
1,462
1,444
1,407
1,394
1,364
1,341
∆Cn
0
0,143
0,369
0,467
0,479
0,486
0,499
0,503
0,514
0,522
Sp RSp Ionen
Protein
526
1 12/22 TOP1_ECOLI
435,2 2 11/22 TOP1_ECOLI
275,8 11 9/22 TOP1_ECOLI
379,6 4 11/22 RUMA_ECOLI
261,9 19 9/24 YFDE_ECOLI
162,7 147 9/22 YOAA_ECOLI
315,4 6 10/22 RUMA_ECOLI
192,4 65 8/22 TOP1_ECOLI
236,9 25 11/30 MANB_ECOLI
228,8 26 10/22 LDHD_ECOLI
Es ist ersichtlich, dass fünf Fragmente anderer Proteine nach der Gesamtmasse des Peptids
zugeordnet werden könnten. Aus den SEQUEST-Werten der Sequenzen von rumA, yfdE,
yoaA, amnB und ldhD, die in der Tabelle dargestellt sind, wird jedoch deutlich, dass eine
passende Zuordnung und Übereinstimmung des aufgenommenen Massenspektrums nur mit
dem der Peptidsequenz C689-R700 aus der Topoisomerase erreicht wurde. Auf die ∆Cn- und
Sp-Werte der neun Sequenzen, die neben der Koordination des Cysteins 689 in der
Topoisomerase aufgelistet sind, bezogen, erfüllt keine dieser Modifikationen die
erforderlichen Kriterien. Somit wird deutlich, dass C689 der Topoisomerase I vom
Cisplatinfragment koordiniert wird.
Katalytisch funktionierende Proteine
Eine weitere Proteingruppe, aus welcher modifizierte Peptidfragmente identifiziert werden
konnten, enthält katalytisch funktionierende Proteine. Ein Beispiel davon stellt das Protein
yciA (acyl-CoA thioester hydrolase) dar. Es handelt sich hierbei um ein 14 kDa Protein,
welches die Hydrolyse der Thioesterbindung zweier Coenzyme A katalysiert. Aus dem
Protein wurde die Sequenz [35MoxSQMDIGGAILAKEIAH~GR53]2+ mit dem Platinfragment
Proteintargets in Escherichia coli
86
[(NH3)2Pt]2+ an der Aminosäure H51 modifiziert. Das zugehörige Massenspektrum aus der
MudPIT-Analyse ist in der unteren Abbildung dargestellt.
Abb.5.24: MudPIT-MS/MS-Spektrum von [MoxSQMDIGGAILAKEIAH~GR]2+ aus yciA
In dem Spektrum sind von 36 Ionen 17 zugeordnet worden. Die Ionen y3+ bis y18+ sollen das
charakteristische Platinpattern aufweisen. Bei den b+-Ionen kommen aus der Modifizierung
resultierend nur b17+ und b18+ mit dem Isotopenmuster vor, werden im obigen Spektrum
jedoch nicht beobachtet. Die Vergrößerung in dem Spektrum in Abbildung 5.24 zeigt das Ion
y15+, welches platiniert vorliegt. Das vorliegende Peptid wurde an der Aminosäure Histidin 51
koordiniert. Eine Auflistung weiterer möglicher Peptide aus ycia und aus sonstigen Proteinen,
welche die Masse m/z = 2242 (mit ∆m ± 2,5) tragen, ist in der Tabelle 5.16 dargestellt.
Proteintargets in Escherichia coli
87
Tab.5.16:SEQUEST-Parameter von [MoxSQMDIGGAILAKEIAH~GR]2+ aus yciA im
Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
L.MoxSQMDIGGAILAKEIAH~GR.V
L.MoxSQMDIGGAILAKE~IAHGR.V
L.MSQMoxDIGGAILAKEIAH~GR.V
F.NYMoxSHEQDWQEFRDAIR.I
L.MoxSQMDIGGAILAK~EIAHGR.V
K.K~NPD~EVAAWLADDSQE.L
L.MSQMoxDIGGAILAKE~IAHGR.V
K.RQGLKVPEDLSIIGFDNIDL.T
-.M~HINIAWQDVDTVLLDM.D
R.Y~SQT~GTLKFSDPTVDE.T
XCorr
3,632
3,246
3,138
2,98
2,84
2,781
2,752
2,639
2,576
2,478
∆Cn
0
0,106
0,136
0,179
0,218
0,234
0,242
0,273
0,291
0,318
Sp RSp Ionen
Protein
888,2 2 17/36 YCIA_ECOLI
524
5 14/36 YCIA_ECOLI
762,9 3 16/36 YCIA_ECOLI
999,3 1 15/32 BETA_ECOLI
338,2 47 12/36 YCIA_ECOLI
404,5 15 12/30 KORB1_ECOLI
436,4 10 13/36 YCIA_ECOLI
246 220 10/38 CYTR_ECOLI
476,7 8 13/32 YRFG_ECOLI
279,5 133 11/30 MDTE_ECOLI
In der modifizierten Peptidsequenz M35-R53 des Proteins yciA gibt es weitere potentielle
Aminosäuren, die durch das [(NH3)2Pt]2+-Fragment möglicherweise koordiniert sein könnten.
Dabei gibt es neben Histidin 51 die Glutaminsäure 48 und das Lysin 47, an die [(NH3)2Pt]2+
binden könnte. An den ∆Cn- Werten (∆Cn > 0,10) lässt sich jedoch erkennen, dass die
Koordination des Histidins 51 (mit Oxidation des Methionins 35) eine wesentlich bessere
spektrale Übereinstimmung liefert. Außerdem werden modifizierte Peptidsequenzen weiterer
drei Proteine in der Tabelle aufgelistet (betA mit oxidiertem Methionin, korbI, yrfG und
mdtE). Die Sp- und die XCorr-Werte für diese Sequenzen erfüllen jedoch, mit Ausnahme des
betA-Proteins, nicht die gewählten Kriterien (Sp ≥ 500, XCorr ≥ 2,5). Die Zuordnung des
Proteins betA kann ebenfalls ausgeschlossen werden, da wie unter Abb 5.24 am Beispiel des
y15+-Ions dargestellt eine Platinierung in der zum MS/MS-Spektrum gehörenden
Peptidsequenz vorliegt.
In der folgenden Abbildung ist die Struktur des Proteins dargestellt.
Proteintargets in Escherichia coli
88
H51
Abb.5.25: Proteinstruktur des Proteins yciA (PDB ID: 2g6s [112])
In der Darstellung ist der Imidazolring des Histidins als sterisch leicht zugängliche Seitenkette
für eine Koordination erkennbar. Die modifizierte Aminosäure des Proteins ist in einer
Schleife lokalisiert. Es ist die Struktur eines vergleichbaren Thioesterase-Proteins dargestellt.
Ein weiteres Protein mit katalytischer Funktion, welches von einem Platinfragment
koordiniert wurde, ist das etwa 46 kDa große Protein enO (enolase). Das Enzym überführt
unter Abspaltung von Wasser 2-Phosphoglycerat in Phosphoenolpyruvat. Das Peptid aus
diesem Protein mit der Sequenz [17GNPT§VEAEHLEGGFVGMAAAPSGASTGSR46]3+
wurde an der Aminosäure Threonin 20 mit dem Platinfragment [Pt]2+ modifiziert. Das
MS/MS-Massenspektrum des Peptids ist in der Abbildung 5.26 dargestellt.
Proteintargets in Escherichia coli
89
Abb.5.26: MudPIT-MS/MS-Spektrum von
[GNPT§VEAEHLEGGFVGMAAAPSGASTGSR]3+ aus enO
Aus den gekennzeichneten Zuordnungen ist ersichtlich, dass von den insgesamt 116 Ionen
(für ein- und zweifach geladene) 19 einfach geladene Peaks im Spektrum (von 41 von
SEQUEST gefundenen Ionen) zugeordnet werden konnten. Die b+-Ionen, welche das Pattern
des Metalls aufzeigen sollen, sind alle b+-Ionen ab b4+. Bei den y+-Ionen sind es die Ionen y27+
bis y29+. Viele der aufgezählten Ionen können im MS/MS-Spektrum unter Abb 5.26
beobachtet werden. In der oberen Abbildung ist das b15+-Ion als Vergrößerung dargestellt. Es
weist erwartungsgemäß das Platinpattern auf, wenn die Koordination am Threonin 20
vorliegt.
Auch in dieser Sequenz sind mehrere Aminosäuren enthalten, welche als potentielle
Bindungsstellen für ein Platinfragment in Frage kommen würden. Zum Vergleich dient die
Auflistung in Tabelle 5.17, welche auch Peptide aus anderen Proteinen berücksichtigt, die
eine Masse von m/z = 3051 (mit ∆m ± 2,5) tragen.
Proteintargets in Escherichia coli
90
Tab.5.17:SEQUEST-Parameter von [GNPT§VEAEHLEGGFVGMAAAPSGASTGSR]3+ aus
enO im Vergleich zu anderen potentiellen Peptidsequenzen
Sequenz
R.GNPT§VEAEVHLEGGFVGMAAAPSGASTGSR.E
R.GNPTVEAE§VHLEGGFVGMAAAPSGASTGSR.E
R.GNPTVE§AEVHLEGGFVGMAAAPSGASTGSR.E
R.GNPTVEAEVH§LEGGFVGMAAAPSGASTGSR.E
R.GNPTVEAEVHLE§GGFVGMAAAPSGASTGSR.E
R.ILE§LRD§VQLIGHNSYEQIRAT§.L
K.NAGVMoxTNNVD§VSGGILNNAGEMTAQITMox.N
R.QVE§GVYGAQIVAGGGELHLNGAMoxPERW.R
R.LNVTLT§RDDVMoxGE§SLYNPMLPGIV.A
R.LNVTLTRD§DVMoxGE§SLYNPMLPGIV.A
XCorr
6,884
6,748
6,636
6,59
5,823
3,43
3,396
3,339
3,326
3,277
∆Cn
0
0,02
0,036
0,043
0,154
0,502
0,507
0,515
0,517
0,524
Sp RSp Ionen
Protein
1451,5 1 41/116 ENO_ECOLI
1149,6 3 38/116 ENO_ECOLI
1318,2 2 40/116 ENO_ECOLI
973
4 36/116 ENO_ECOLI
730
5 32/116 ENO_ECOLI
453,2 22 23/80 YAIV_ECOLI
328,3 164 24/108 YDBA_ECOLI
455
21 27/104 FAEI_ECOLI
440,9 25 26/92 SYR_ECOLI
440,9 25 26/92 SYR_ECOLI
Den Massen entsprechend könnten auch Peptide aus den Proteinen yaiv, ydby, faei oder syr
mit ihrem Massenspektrum zugeordnet werden. Beim Betrachten der SEQUEST-Werte zu
diesen Peptidsequenzen fällt jedoch auf, dass die Zuordnungen für eine wesentlich geringere
Ionenzahl erfolgen konnten, so dass diese Proteinmodifikationen ausgeschlossen werden
können. In dem Peptid aus eno finden sich neben Threonin 20 drei Glutaminsäuren (E22, E24
und E28) sowie Histidin 26, die mit ihren Seitenketten ebenfalls nach der Tabelle als
Angriffsstellen für ein Cisplatinfragment dienen. Auf Grund der fehlenden bj+-Ionen mit j ≤5
kann keine sichere Zuordnung zu T20 bzw. E22 erfolgen. In der folgenden Abbildung ist ein
Ausschnitt aus der Struktur des Proteins mit den potentiellen Bindungsstellen dargestellt.
T 20
Abb5.27: Die Struktur des Proteins enO (PDB ID: 2fym [113])
Proteintargets in Escherichia coli
91
Die Seitenkette des Threonins, die in der oberen Abbildung eingekreist ist, liegt in einem βFaltblatt. Die Zugänglichkeit der Aminosäure für eine Koordination wird beim Betrachten der
Illustration deutlich. Auch die in Tabelle 5.17 aufgelisteten potentiellen Angriffsstellen sind
auch in dem Faltblatt lokalisiert und liegen ebenfalls sterisch zugänglich. Somit kann bei dem
Peptid [17GNPT§VEAEHLEGGFVGMAAAPSGASTGSR46]3+ aus dem Protein enO keine
genaue Bindungsstelle angegeben werden. Aus den SEQUEST-Werten wird deutlich, dass
außer Threonin 20 und Glutaminsäure 22 auch E24 sowie H26 von dem Cisplatinfragment
koordiniert werden könnten. Jedoch deutet die Platinierung der Ionen bj+ für j ≥ 6, wie oben
bereits erwähnt, auf T20 und E22 als die wahrscheinlichen Bindungsstellen hin.
Ein ebenfalls mit katalytischer Funktion von einem Platinfragment koordiniertes Protein ist
das 48 kDa Protein cisY (citrate synthase). Das Enzym aus dem Citratzyklus katalysiert die
Kondensation von Oxalacetat mit Acetyl-CoA zu Citrat. Aus diesem Protein wurde das
tryptisch gespaltene Peptid [111HTM§IHEQITR120]2+ an der Aminosäure M113 mit dem
Metallfragment [Pt]2+ modifiziert. Das MS/MS Spektrum des Peptids ist in der folgenden
Abbildung dargestellt.
Abb.5.28: MudPIT-MS/MS-Spektrum von [HTM§IHEQITR]2+ aus cisY
Proteintargets in Escherichia coli
92
Eine Zuordnung der Peptidfragmente erfolgte bei der SEQUEST-Analyse für 15 von 18
möglichen Ionen. In Abb. 5.28 sind 14 dieser Ionen beschriftet. Die b+-Ionen ab b3+ sind
platiniert, dagegen sind es bei den y+-Ionen nur die Ionen y8+ und y9+. Der vergrößerte
Ausschnitt in der Abbildung zeigt das y8+-Ion, welches die Platinverteilung aufweist. Weitere
Möglichkeiten von Peptiden auch aus anderen Proteinen mit der Masse m/z = 1459 (∆m ±
2,5), welche von dem Platinfragment koordiniert sein könnten, sind in der Tabelle 5.18 zu
sehen.
Tab.5.18: SEQUEST-Parameter von [HTM§IHEQITR]2+ aus cisY im Vergleich zu anderen
potentiellen Peptidsequenzen
Sequenz
R.HTM§IHEQITR.L
R.HTMIH§EQITR.L
R.HT§MIHEQITR.L
R.H§TMIHEQITR.L
R.HTMIHE§QITR.L
Y.T§QLFEEQITR.P
D.FQGTES§LNTLR.D
I.QLE§S§PKITR.N
T.LNWPMIGEQLTR.R
P.VGFK§NGTDGNTR.I
XCorr
3,619
3,195
2,95
2,794
2,756
2,214
2,211
2,158
2,086
2,084
∆Cn
0
0,117
0,185
0,228
0,238
0,388
0,389
0,404
0,424
0,424
Sp RSp Ionen
Protein
580,3 1 15/18 CISY_ECOLI
357,1 4 13/18 CISY_ECOLI
393,2 3 13/18 CISY_ECOLI
354,6 5 12/18 CISY_ECOLI
188,9 67 11/18 CISY_ECOLI
273,7 12 11/18 AMPH_ECOLI
164,3 130 11/20 FTSX_ECOLI
271,2 13 11/16 YAIW_ECOLI
219,2 33 11/22 HIS7_ECOLI
198,8 54 11/22 AROH_ECOLI
Es sind vier andere Proteine (ampH, ftsX, yaiW und aroH) mit einer Modifikation durch ein
Cisplatinfragment in der Tabelle aufgelistet. Ein Peptid aus dem Protein his7 erscheint in der
Aufstellung ohne eine Koordination. Die SEQUEST-Parameter zeigen jedoch, dass die
Übereinstimmung mit dessen theoretischem Massenspektrum in diesem Fall gering ist. Die
Isotopenverteilung des Metalls bei den Ionen im Massenspektrum zeigt außerdem, dass eine
Platinkoordination vorliegen muss. Mögliche Bindungsstellen des Proteins cisy sind die
Aminosäuren Methionin 113, Histidin 115 und 111, Threonin 112 und Glutaminsäure 116.
Beim Betrachten der Sp- und der XCorr-Werte der aufgezählten Varianten fällt jedoch auf,
dass nur die Übereinstimmung mit den Datenbankdaten für die Koordination an Methionin
113 zufrieden stellend (Sp > 500, ∆Cn < 0,10) ist und die weiteren Aminosäurereste somit als
Bindungsstelle ausgeschlossen werden können.
In der Abbildung 5.29 ist die Struktur des Proteins dargestellt, welche die Möglichkeit bietet,
sich die Koordination des Proteins mit dem Platinfragment in räumlicher Verteilung
anzuschauen.
Proteintargets in Escherichia coli
93
M113
Abb.5.29: Die Struktur des Proteins cisY (PDB ID: 1k3p [114])
Die von Cisplatin koordinierte Aminosäure ist in der Darstellung eingekreist. Die
Zugänglichkeit der in einer Schleife zwischen zwei Helices lokalisierten Seitenkette wird so
verdeutlicht.
Proteine mit anderen Funktionen
Neben den katalytisch funktionierenden Proteinen wurden auch Proteintargets mit anderen
Funktionen aus dem Bakterium identifiziert.
So wurde beispielhaft das Protein eftU, ein Elongationsfaktor mit einer koordinierten
Aminosäure aus dem Bakterium-Cisplatin- Gemisch gefunden. Es handelt sich hierbei um das
43 kDa große abundante Protein, welches bereits bei der Umsetzung mit [Pt(dien)]2+
koordiniert wurde. Das Protein ist in der Zelle unter anderem an der Proteinsynthese beteiligt.
Aus dem Protein wurde das Peptid der Sequenz [359MVVTLIHPIAM§DDGLR374]2+ mit der
Molekülmasse m/z = 1974 (∆m ± 2,5) massenspektrometrisch detektiert. Das Peptid ist von
dem Platinrest [Pt]2+ an der Aminosäure M369 koordiniert. Das MS/MS-Spektrum des
Peptids ist in der folgenden Abbildung dargestellt.
Proteintargets in Escherichia coli
94
Abb.5.30: MudPIT-MS/MS-Spektrum von [MVVTLIHPIAM§DDGLR]2+ aus eftU
Das MS/MS-Spektrum enthält Zuordnungen von 19 Ionen des Proteinfragments, wobei
insgesamt 30 b+- und y+-Ionen von der Sequenz entstehen können. Die b+-Ionen ab dem Ion
b11+ sind platiniert, bei den y+-Ionen ist das ab dem Ion y6+ der Fall. Dieses Verhalten ist an
einem Beispiel, dem y8+ in dem vergrößerten Ausschnitt verdeutlicht. Die Isotopenverteilung
des eingesetzten Metalls ist bei y8+ und vielen der restlichen Ionen eindeutig zu erkennen. Die
folgende Tabelle zeigt zehn der Massengenauigkeit von ∆m ± 2,5 für m/z = 1974
entsprechenden Peptide.
Proteintargets in Escherichia coli
95
Tab.5.19: SEQUEST-Parameter von [MVVTLIHPIAM§DDGLR]2+ aus eftU im Vergleich zu
anderen potentiellen Peptidsequenzen
Sequenz
K.MVVTLIHPIAM§DDGLR.F
K.M§VVTLIHPIAMDDGLR.F
K.MVVTLIH§PIAMDDGLR.F
K.MVVT§LIHPIAMDDGLR.F
K.MVVTLIHPIAMD§DGLR.F
I.LTMLAAGES§NK§EIGR.A
I.LTMLAAGE§SNK§EIGR.A
I.LTMLAAGES§NKE§IGR.A
K.MVVTLIHPIAMDD§GLR.F
I.LTMLAAGE§SNKE§IGR.A
XCorr
3,589
3,556
3,529
3,43
2,886
2,451
2,409
2,372
2,361
2,329
∆Cn
0
0,009
0,017
0,044
0,196
0,317
0,329
0,339
0,342
0,351
Sp RSp Ionen
Protein
664
1 16/30 EFTU_ECOLI
612
2 17/30 EFTU_ECOLI
437,3 5 14/30 EFTU_ECOLI
463,3 4 15/30 EFTU_ECOLI
412,7 7 14/30 EFTU_ECOLI
350 13 13/28 YHJB_ECOLI
350 13 13/28 YHJB_ECOLI
350 13 13/28 YHJB_ECOLI
289,3 32 13/30 EFTU_ECOLI
350 13 13/28 YHJB_ECOLI
Neben sechs Koordinationsstellen an unterschiedlichen Aminosäuren des Proteins eftu sind in
der Tabelle 5.19 vier Bindungsmöglichkeiten im Protein yhjB aufgelistet. Alle vier Peptide
erfüllen jedoch die Kriterien bezüglich ihrer SEQUEST-Werte nicht (∆Cn > 0,1 und
Sp < 500) und werden nicht näher betrachtet. In eftU sind insbesondere zwei Methioninstellen
als potentielle Bindungsstellen zu erwähnen, M369 und 359. Die restlichen Modifikationen
dieser Sequenz (z. B. H365, T362) werden auf Grund ihrer niedrigen Sp-Werte
ausgeschlossen. Einen Hinweis auf die Bevorzugung des Methionins 369 gibt das in Abb.
5.30 dargestellte y8+-Ion, welches das Platinpattern aufweist. Bei einer Koordination an
Methionin 359 sollte dieses Ion sich nicht im MS/MS-Spektrum befinden. Außerdem ist das
Fehlen von bis zu zwei nicht platinierten bj+-Ionen (b6+, b7+) mit j < 11 und das
Vorhandensein aller bj+-Ionen mit j ≥ 11 im Einklang mit einer M369-Koordination. In der
unteren Abbildung ist die Struktur des Proteins eftU zu finden.
Proteintargets in Escherichia coli
96
T341
E343
M 359
M 369
Abb.5.31: Die Struktur des Proteins eftU (PDB ID: 1efc [102])
In der abgebildeten Struktur sind die Seitenketten beider möglichen Methioninstellen (M359
und M369) gekennzeichnet. Zusätzlich sind die möglichen Bindungsstellen des [Pt(dien)]2+Fragmentes T341/E343 in der Struktur hervorgehoben. Alle vier Seitenketten sind im
vergleichbaren Bereich des Proteins eftU lokalisiert. Die räumliche Darstellung deutet
ebenfalls darauf hin, dass eine Koordination an M369 wahrscheinlich ist, da dessen
Seitenkette sterisch leicht zugänglich ist.
Das Protein Thioredoxin-1 thiO, ein etwa 12 kDa großes Protein wird im Zusammenhang mit
der Krebstherapie diskutiert, es wird zum Beispiel in Tumorzellen überexprimiert und führt
zur Resistenz von humanen Zellen gegen Cisplatin [155, 161]. Es ist in der Zelle für die
redoxchemischen Abläufe zuständig, speziell bei der Umwandlung von Ribose zu
Desoxyribose bei der DNA-Synthese. Eine Inaktivierung des Proteins würde Einfluss auf das
Wachstum der Zelle haben. Von dem 109 Aminosäuren enthaltenden Protein wurde
massenspektrometrisch das Peptid der Sequenz [7H~LTDDSFDTDVLK20]2+ mit der
Modifikation mit dem Platinfragment [(NH3)2Pt]2+ identifiziert. Es wurde von dem
Metallfragment an der Aminosäure H7 koordiniert. Das aufgenommene Massenspektrum des
Peptids ist in Abbildung 5.32 dargestellt.
Proteintargets in Escherichia coli
97
Abb.5.32: MudPIT-MS/MS-Spektrum von [H~LTDDSFDTDVLK]2+ aus thiO
Eine Zuordnung von 20 b+- und y+- Ionen ist im MS/MS-Spektrum zu sehen, für die
SEQUEST-Analyse wurden 22 Ionen berücksichtigt. Aus dem Proteinfragment können
maximal 24 b+- und y+- Ionen entstehen, was auf eine Zuordnung von etwa 92% bedeutet.
Wie aus der Peptidsequenz erkennbar, sollen alle b+-Ionen das Platinpattern aufweisen. Bei
den y+-Ionen soll das Muster des Metall nicht vorkommen. In der Abbildung ist zur
Verdeutlichung die Isotopenverteilung des b11+-Ions vergrößert zu sehen. Bei allen y+-Ionen
fehlt das typische Platinisotopenmuster.
Da in der Sequenz des Peptids weitere potentielle Bindungsstellen für das Platinfragment
vorhanden sind, müssen diese auch mit Hilfe anderer Kriterien geprüft werden. Die untere
Tabelle listet die in Frage kommenden Peptide mit der jeweiligen Kennzeichnung der
koordinierten Aminosäure auf. Es werden ebenfalls der Masse m/z = 1734 (∆m ± 2,5)
entsprechende Peptide anderer Proteine aufgelistet.
Proteintargets in Escherichia coli
98
Tab.5.20: SEQUEST-Parameter von [H~LTDDSFDTDVLK]2+ aus thiO im Vergleich zu
anderen potentiellen Peptidsequenzen
Sequenz
I.H~LTDDSFDTDVLK.A
K.H~LPEPFRIRVIE.P
I.HLT~DDSFDTDVLK.A
K.MoxICH~EGMTVILDSGS.T
K.MoxIHE~GMTVILDSGS.T
D.GT~LVDS~EVICSR.A
K.MoxIHEGMT~VILDSGS.T
D.GT~LVDSE~VICSR.A
D.GTLVD~S~EVICSR.A
K.MoxIHEGM~TVILDSGS.T
XCorr
3,558
2,781
2,563
2,51
2,431
2,33
2,283
2,253
2,245
2,219
∆Cn
0
0,218
0,28
0,295
0,317
0,345
0,358
0,367
0,369
0,377
Sp
1931,2
419,9
1175,7
367,9
368
204,2
211,9
204,2
204,2
211,9
RSp
1
17
2
29
28
397
345
397
397
345
Ionen
22/24
13/22
19/24
13/26
13/26
9/22
11/26
9/22
9/22
11/26
Protein
THIO_ECOLI
TNAA_ECOLI
THIO_ECOLI
GATR_ECOLI
GATR_ECOLI
YIEH_ECOLI
GATR_ECOLI
YIEH_ECOLI
YIEH_ECOLI
GATR_ECOLI
Wie in der Tabelle zu sehen ist, sind aus weiteren Proteinen drei Peptide mit der oben
angegebenen Massengenauigkeit eingetragen. Aus den Sp- und XCorr- Werten dieser Peptide
geht jedoch hervor, dass diese Spektrenzuordnungen wenig Übereinstimmung einbringen.
Somit ist die Koordination an eine Aminosäure im Protein Thioredoxin bestätigt. Die
SEQUEST-Parameter zeigen, dass nur die Modifikation des Histidins 7 in Frage kommt, für
T9 erfüllt der ∆Cn-Wert mit ∆Cn = 0,28 nicht die Kriterien. Aus der Abbildung kann die
Proteinstruktur zur Veranschaulichung herangezogen werden.
C34
H7
C32
Abb.5.33: Die Struktur des Proteins thiO (PDB ID: 2trx [115])
Proteintargets in Escherichia coli
99
In der räumlichen Darstellung wird die gute Zugänglichkeit der Seitenkette des koordinierten
Histidins erkennbar. Die Seitenkette des Methionins 1 liegt in unmittelbarer Nähe des
Histidins und könnte gegebenenfalls von einer Migration zu H7 kinetisch bevorzugt gewesen
sein. Gekennzeichnet sind in der Gesamtstruktur des Thioredoxins auch die Cysteinstellen
C32 und C34. Diese stellen das aktive Zentrum des Proteins dar.
Verglichen mit der Tabelle 5.6, die Proteintargets aus dem Ansatz [Pt(dien)]2+ und E. coliBakterium auflistet, sind von den mit einem der Cisplatinfragment koordinierten Peptide
wesentlich mehr Proteintargets (31 Proteine, siehe Tab. 5.10) identifiziert worden. Dies kann
vor allem an der Möglichkeit Cisplatins liegen, stabile Chelate und Bischelate auszubilden,
was die Pt(dien)2+-Verbindung nicht kann. Die Funktionen, Bedeutungen und Aufgaben der
einzelnen Proteine werden im nachfolgenden Kapitel diskutiert.
5.4. Diskussion
Voraussetzung für eine erfolgreiche MudPIT-Analyse mit Metallkomplexen und ganzen
Zellsystemen ist eine kinetisch stabile Metall-Protein-Bindung, welche sowohl gegenüber der
biphasischen chromatographischen Trennung als auch den massenspektrometrischen
Bedingungen bestehen bleibt. Eine große Anzahl an b+- und y+- Ionen, die sich unter den
MS/MS-Ionisierungen bilden konnten, wirkt sich positiv auf eine genauere Zuordnung der
Peptidsequenz
des
theoretischen
Massenspektrums
aus.
Eine
charakteristische
Isotopenverteilung des reagierenden Metalls ist bei der Identifizierung der metallierten Ionen
hilfreich. Beide für die Analysen mit dem Bakterium Escherichia coli verwendeten Metalle,
Platin und Ruthenium zeigen ein charakteristisches Isotopenpattern auf.
E. coli wird in der Forschung als Modellsystem häufig verwendet. Mit der gekoppelten HPLC
und Tandem-Massenspektrometrie wurden 1147 Proteine einer Gesamtzahl von 4288 aus dem
Bakterium identifiziert [120]. Durch seine unkomplizierte Handhabung hat E. coli seine hohe
Bedeutung in den Laboren erlangt. Untersuchungen haben gezeigt, dass der Ablauf vieler
grundlegender Vorgänge in menschlichen Zellen im Wesentlichen dem der E. coli-Zellen
gleicht [116]. Deswegen wird das Bakterium als Zellsystem häufig dafür verwendet, um die
Vorgänge in menschlichen Zellen besser verständlich zu machen.
Proteintargets in Escherichia coli
100
5.4.1. Wechselwirkung von [(η6-p-Cymol)RuCl2(DMSO)] mit E. coli-Zellen
Aus früheren Studien ist bekannt, dass eine Migration von [(η6-Aren)Ru]-Fragmenten zu
anderen Bindungsstellen oder deren Verlust unter den sauren Bedingungen der SCXChromatographie bei monodentaten Komplexen wahrscheinlich ist [82, 128]. Für bidentate
Ru-Aminosäure Komplexe, wie [(η6C6H6)Ru(L-Ala-κ2N,O)Cl], wurde aber eine hohe
Stabilität bei RP-Trennungen unter Einsatz von Trifluoressigsäure (TFA, pH = 2) beobachtet
[128]. Für die massenspektrometrischen Untersuchungen bzw. MudPIT-Analysen eignen sich
damit bi- oder tridentate Chelate aus Peptiden und Metallkomplexen besser. Der hier gewählte
Komplex [(p-Cymol)RuCl2(DMSO)] kann nach Abspaltung der drei labilen Liganden, wie die
Chloride und das DMSO, bi- oder tridentate Komplexe ausbilden. Für die in Kapitel 5.1
diskutierten Möglichkeiten in einer Peptidsequenz mehrere Bindungsstellen aufzulisten
(Tabelle 5.1) sollte beachtet werden, dass der Rutheniumkomplex unter der niedrigen
Chloridkonzentration in den E. coli Zellen schnell folgende Komplexe bilden kann: [{(pCymol)Ru}2(OH)3]+, [(p-Cymol)Ru(DMSO)(H2O)2]2+ oder [(p-Cymol)Ru(H2O)3]2+. Alle drei
Komplexe haben ebenfalls drei labile Liganden, deren Abspaltungen eine tridentate Bindung
begünstigen. Bei dem verwendeten pH-Wert von 7 könnte der Komplex κ2N,O-Chelate mit
thermodynamisch stabilen Fünf- oder Sechsringen unter Einfügen der Aminosäuren S, T oder
D ausbilden [129]. Aus Tabelle 5.1 lässt sich für die ersten vier koordinierten Sequenzen
feststellen, dass jeweils die erste oder zweite Aminosäure als Bindungsstelle fungiert. Alle NTermini dieser Peptide sind nicht tryptisch gespalten. Diese Beobachtung könnte vermuten
lassen, dass eine Hydrolyse der benachbarten Amidgruppe von dem koordinierten RuFragment gefördert wird, wie es bereits von KLEINE für Platin bestimmt wurde [46]. Eine Nterminale Chelatisierung ist für eine gute Fragmentierung der b+-Ionen mit enthaltenem
Rutheniumkomplexrest und y+-Ionen ohne das Metall unter massenspektrometrischen
Bedingungen von Vorteil. Bei einer Koordination des Metallkomplexes an einer weiter zum
C-Terminus lokalisierten Aminosäure in der Sequenz fehlt diese Möglichkeit der
Stabilisierung.
Der Tabelle 5.1 kann entnommen werden, dass das [(p-Cymol)Ru]2+-Fragment bevorzugt die
harten Sauerstoffatome der Aminosäuren D, T und S koordiniert. Auch ist die Aminogruppe
des Lysins ein Target des Rutheniumfragmentes. Diese zum Teil zweifach bestätigten
Koordinationen in zwei unabhängigen Proben lassen vermuten, dass diese Aminosäuren mit
ihren Seitenketten spezifische Targets für den Komplex darstellen. Diese Ergebnisse scheinen
unerwartet zu sein, da bisher hauptsächlich die Imidazolatome aus Histidylresten oder die
Proteintargets in Escherichia coli
101
weichen Schwefelatome aus Cysteinyl- oder Methionylresten als potentielle Targets für
Rutheniumkomplexe diskutiert wurden [52]. Zudem wurde in der Arbeitsgruppe SADLER ein
[(p-Cymol)RuCl2(Lysozym)]-Komplex
kristallographisch
mit
einer
koordinierten
Histidylseitenkette in der H15 Aminosäure charakterisiert [83]. Für den zytotoxischen
Komplex [(Biphenyl)RuCl(en)](PF6) nach Umsetzung mit dem Protein Cytochrom c wurden
allerdings NMR-spektroskopisch die Koordinationen von Carboxylat- und Aminogruppen
identifiziert [81]. Die kinetische Bevorzugung von [(η6-Aren)Ru]-Verbindungen anionische
Sauerstoffatome zu koordinieren, wurde bei einer 2h-Reaktion mit 5´-MonophosphatNucleotiden festgestellt [82]. Hierbei wurde auch nach einer schnellen Bindung an die 5´Monophosphatgruppe eine langsame Migration des Rutheniumfragmentes zum N7-Atom der
Nucleobase
im
5´-GMP
beobachtet.
Mit
diesen
bisherigen
Erkenntnissen
zum
Koordinationsverhalten von Rutheniumkomplexen wird deutlich, dass die unter den im
Rahmen der vorliegenden Arbeit verwendeten Bedingungen (3 Stunden Inkubation bei 37°C
und pH = 7) identifizierten Bindungsstellen für den Rutheniumkomplex (D, T, S und K) im
Einklang mit bisherigen Studien stehen.
In den beiden MudPIT-Analysen mit dem Rutheniumkomplex sind zweimal identische
Proteine ermittelt worden. Es handelt sich hierbei um die Proteine cspC und osmY, in denen
die Aminosäuren D/K und S/T modifiziert wurden. Zur Veranschaulichung sind in Abbildung
5.34 zwei MS/MS Spektren des Proteins cspC dargestellt, die aus den unterschiedlichen
Analysen stammen.
Proteintargets in Escherichia coli
102
Abb.5.34: MudPIT-MS/MS-Spektren des Proteins cspC aus zwei unabhängigen Analysen des
E. coli Bakteriums
Proteintargets in Escherichia coli
103
Die Vergleichbarkeit der beiden Spektren wird beim Betrachten dieser schnell deutlich. Bis
auf geringe Intensitätsunterschiede und leicht abweichende SEQUEST-Werte sind beide
Spektren beinahe identisch.
Das cspC Protein gehört der cspA-Familie an, welche neun Homologe enthält. Nur zwei
davon, cspC und cspE werden bei 37°C gebildet. Beide Proteine sind in der
Expressionsregulierung einbezogen, wobei sie für die Encodierung der Gene für die Proteine
ppiD, osmY und sucC verantwortlich sind.
Im
Einklang
mit
durchgeführten
Studien
zur
Reaktionseigenschaften
der
[(η6-
C6H6)Ru(H2O)3]2+-Spezies mit 5´-ATP2- werden stabile Produkte/Makrochelate aus dem
eingesetzten Rutheniumkomplex bzw. seiner Aquaspezies mit Biomolekülen aus der Zelle
erwartet. Bei der oben genannten Reaktion mit 5´-ATP2- entstanden nämlich stabile
κ3N7,O(Pβ),O(Pγ)-Makrochelate [130], wobei die Bildung das energieliefernde Zentrum des
Moleküls zerstört.
Bei der Koordination des [(η6-p-Cymol)RuCl2(DMSO)]-Komplexes an das Protein cspC ist
zu beobachten, dass die benachbarten Seitenketten der Aminosäuren Threonin 20,
Asparaginsäure 27 und Lysin 14 für eine tridentate κ3O,O´,N Anordnung des
Metallfragmentes beinahe optimal liegen. Zur Veranschaulichung dient die Abbildung 5.35.
H31 mögliche
Cisplatinbindungsstelle
K26
D27
K14
T20
Abb.5.35: Struktur des Proteins cspC mit gekennzeichneten möglichen Koordinationsstellen
Proteintargets in Escherichia coli
104
Aus der Darstellung der Struktur wird deutlich, dass eine Alternative κ3O,N´,N-Bindung mit
dem Amidstickstoff der Aminosäure D27 und Lysin K26 zu einem Sechschelatring möglich
wäre. Diese Formation kann als Schritt vor der tryptischen Spaltung angenommen werden und
würde das Vorkommen der durchgehenden b+-Ionenreihe von b8+ bis b15+ erklären (siehe
Abbildung 5.3). Eingekreist wurde in der Abbildung 5.40 die Seitenkette des H31, welche
möglicherweise
ein
Cisplatinfragment
koordiniert.
Die
modifizierte
Peptidsequenz
[FVH~FSAIQGNGFK]2+ erscheint nicht in Tab 5.10, da der ∆Cn-Wert mit ∆Cn = 0,06 nicht
das Mindestkriterium erfüllt.
Die Struktur (Abb. 5.6) und das MS/MS Spektrum (Abb. 5.5) für das sucC Protein wurden in
Kapitel 5.1 bereits diskutiert. Die für cspC vorgestellte Erläuterung der Stabilität der Bindung
gilt auch für das sucC Protein. In der koordinierten Peptidsequenz kommen nämlich in der
Nachbarschaft die Seitenketten anderer Aminosäuren ebenfalls für die Formation von stabilen
Makrochelaten in Frage. Dies ist in Abbildung 5.41 dargestellt.
K372
T375
D376
Abb.5.36: Vergrößerung der Struktur des Proteins sucC mit möglichen Koordinationsstellen
Eine mögliche Wechselwirkung des Metallfragmentes mit den Aminosäuren K372, T375 und
D376 wird aus der Abbildung ersichtlich. Die Einbindung der Aminosäure K372 in den
Metallproteinkomplex wird durch das Vorhandensein des Rutheniumisotopenpatterns für das
Ion b3+ bestätigt (siehe Abb. 5.5).
Proteintargets in Escherichia coli
105
Weitere koordinierte Proteine (siehe Tabelle 5.1) aus dem Rutheniumkomplex E. coli Ansatz
sind die Helikasen. Das sind Enzyme, welche die Doppelstränge der DNA oder RNA trennen.
Sie sind für viele zelluläre Prozesse notwendig, wie z. B. Replikation, Reparatur oder
Transkription. Eine schematische Darstellung der Funktionsweise ist in Abbildung 5.37
gezeigt. In der Abbildung sind die allgemeinen Vorgänge bei der Replikation gezeigt.
An der Reparatur von Schäden der DNA beteiligt sind aus E. coli nur zwei Helikasen: dinG
und uvrD. Von dem eingesetzten Rutheniumkomplex koordiniert wurde aus dieser Analyse
die Helikase dinG massenspektrometrisch identifiziert. Eine der identifizierten Helikasen ist
die RNA-Helikase hrpA, sie gehört wie die ermittelte Helikase dbpA der DEAD
Helikasenfamilie an, wobei ihre Funktionen nicht vollständig geklärt sind. Es wird diskutiert,
dass die hrpA Helikase an der Stressregulierung teilnimmt. Die DNA-Helikase traI1 wurde
ebenfalls als mögliches von dem Rutheniumkomplex koordiniertes Protein identifiziert.
Aus den koordinierten Aminosäuren der in Tabelle 5.1 aufgelisteten Proteine wird deutlich,
dass die Carboxygruppe aus der Asparaginsäure, die Hydroxygruppen aus Serin und Threonin
und die Stickstoffe der Aminogruppen aus Lysin häufige Targets für den [(η6-pCymol)RuCl2(DMSO)]-Komplex im neutralen pH-Bereich darstellen. Die fehlende
Koordination der weichen Aminosäuren Methionin oder Histidin kann einerseits durch die
kinetische Benachteiligung bedingt sein, andererseits aber schlechtere Zugänglichkeit im
Protein bedeuten. Eine zusätzliche Erklärung kann der Ladungsausgleich liefern. Die
Koordination der Aminosäuren D, S oder T reduziert die Anfangsladung des [(η6-pCymol)Ru]2+-Fragmentes zu 1+. Bei der Bindung der Aminosäuren M oder H wird die
Ladung von 2+ nicht verändert. Diese Tatsache könnte eine erfolgreiche SEQUEST- Analyse
wegen zusätzlicher MS/MS Fragmentierung und einer daraus resultierenden zu kleinen Zahl
an b+- und y+-Ionen einschränken.
5.4.2. Wechselwirkungen von Platinkomplexen mit E. coli-Zellen
Die Tabelle 5.6 gibt alle vom [Pt(dien)]2+-Fragment koordinierten Proteine des Bakteriums
an. Alle MS/MS-Spektren der sieben Proteine wurden auf das Vorhandensein des
Platinpatterns geprüft. Die SEQUEST-Parameter der modifizierten Proteine erfüllen alle die
Auswahlkriterien bezüglich der XCorr-, der Sp-, der ∆Cn-Werte und auch der
Ionenübereinstimmung.
Der für die Analysen gewählte pH-Wert von etwa 7 verursacht eine Deprotonierung der
Carboxylatgruppen in Asparagin- und Glutaminsäuren. Die Seitenketten dieser Aminosäuren
Proteintargets in Escherichia coli
106
und die Thioether-Schwefelatome aus Methionin stellen kinetisch bevorzugte Bindungsstellen
für Cisplatin dar [99, 54]. Ebenso sind die Hydroxysauerstoffe der Serine, Threonine und
Tyrosine kinetisch attraktive Koordinationsstellen für die Metallfragmente. Obwohl der
Metallkomplex [Pt(dien)Cl]Cl keine zytotoxische Wirkung aufweist, eignet er sich als
Modellsystem für die Untersuchungen des Bindungsverhaltens von Platinkomplexen [99].
Das [(dien)Pt]2+-Fragment mit dem tridentaten Liganden (dien) kann nur monodentat
koordiniert werden, so dass die erste Bindungsstufe an Proteine und DNA für mehrfach
chelatisierte Komplexe wie Cisplatin mit Hilfe von [Pt(dien)(H2O)]2+ untersucht werden kann.
[Pt(dien)(H2O)]2+ mit einem pKa von 5,87 und das bei höheren pH-Werten überwiegende
[Pt(dien)(OH)]+ reagieren schnell mit Peptiden, wie FRÖHLING und SHELDRICK zeigen
konnten [46, 56]. Dabei bildete sich der κS-Komplex aus dem [Pt(dien)]2+-Fragment und HHisMet-OH bei 37°C und pH = 6,5 bereits zu etwa 50% nach 30 min Reaktionszeit. Wie
ebenfalls
in
früheren
Studien
gezeigt
wurde,
wandert
das
[Pt(dien)]2+-Fragment
intramolekular vom Thioether-Schwefel in [Pt(dien)(H-Met-OH-κS)]2+ zum terminalen
Stickstoff in [Pt(dien)(H-Met-OH-κN)]2+ [117]. Eine langsame intermolekulare κS zu κN
Isomerisierung wurde auch bei weiteren Migrationsuntersuchungen für M-koordinierte
[Pt(dien)]2+-Komplexe beobachtet [118]. Diese zeigten, dass Pt(II)-Fragmente intramolekular
vom Schwefelatom einer Aminosäure zum N7-Atom einer Nukleobase [119] bzw. zum
Imidazol-N-Atom in H-His-Met-OH [46] wandern können. So kann die Vermutung
aufgestellt werden, dass die identifizierten K- und H-Bindungsstellen des [Pt(dien)]2+Fragmentes in E. coli möglicherweise von einer derartigen intramolekularen Migration von
einer Schwefelbindung in den Peptiden stammen könnten, wenn dies wie bei M1 in
Thioredoxin in der unmittelbaren Nachbarschaft zu einem K- oder H-Rest liegt.
Aus der Analyse nach der Umsetzung des [Pt(dien)]2+-Komplexes mit dem Darmbakterium
wurden sieben koordinierte Proteintargets identifiziert (siehe Tabelle 5.6). Davon können drei
(rl7, eftU und dnaK) der Proteine in hohen Konzentrationen während des Wachstums in einer
Zelle vorgefunden werden [120]. Die Proteine rl7 und eftU sind an der Biosynthese beteiligt.
Das 69 kDa Protein dnaK (chaperone protein) ist am Ausgleich des osmotischen Schocks
beteiligt [92]. Eine für die Zellteilung und das Wachstum wichtige Aufgabe des Proteins ist,
dass es in die DNA-Replikation eingebunden ist. Bei den restlichen in Tabelle 5.6
aufgelisteten Proteinen finden sich ebenfalls die kinetisch bevorzugten Aminosäuren
Methionin, Serin und Glutaminsäure als Bindungsstellen. Diese Proteine (aldB, ssb und rbsA)
werden von der Zelle unter Stressbesingungen überproduziert [92], was ersichtlich bei einer
Behandlung des Bakteriums mit dem Metallkomplex der Fall ist. Das Protein yicC, welches
Proteintargets in Escherichia coli
107
auch mit dem Platinfragment koordiniert detektiert wurde, ist für das Überleben der E. coliZellen in stationären Kulturen und Wachstum unter speziellen Bedingungen erforderlich
[121]. Besonderer Aufmerksamkeit bedarf aus der Liste in Tabelle 5.6 das Protein ssb. Es ist
wie oben erwähnt an der DNA- Replikation beteiligt. Es hat die Aufgabe die während der
Replikation entstandenen Gabeln auseinander zu halten. Die folgende schematische
Darstellung soll dies verdeutlichen.
Abb.5.37: Schematische Darstellung der DNA-Replikation [122]
Die DNA-Helikase bewegt sich in 5'-3'-Richtung entlang des Stranges, an den sie gebunden
ist und entwindet die Doppelhelix unter Verbrauch von ATP. Die entstehenden EinzelstrangBereiche werden durch Einzelstrang-bindende-Proteine (ssb-Proteine) abgedeckt. Diese
binden die DNA im Bereich der Aminosäuren W55-F61.
Eine Koordination des Platinfragmentes an dieses Protein würde eine strukturelle
Veränderung und damit Funktionsbeeinflussung bis zur Inaktivierung mit sich führen. Auch
im menschlichen Organismus trägt das ssb-Protein dieselbe Rolle, so dass seine Koordination
eine Störung sowohl im Replikations- als auch im Reparaturmechanismus einer Zelle
bedeuten würde.
Für die Analyse des Bindungsverhaltens von Cisplatin an die Proteine des Bakteriums wurde
eine Reaktionszeit von drei Stunden gewählt. Wie aus der Tabelle 5.10 ersichtlich, wurde für
diesen Ansatz eine Gesamtzahl von 31 mit einem Cisplatinfragment koordinierter Proteine
identifiziert. Dabei sind sieben Proteine DNA-und RNA-bindend (uvrD, top1, finQ, mutS,
dbpA, rep5, intD und rs6) und vier stressregulierend (ch60, aldA, rpoA und ompA) sowie
weitere 20 Proteine mit anderen Funktionen. In drei Proteinen wurden die Koordinationen an
Proteintargets in Escherichia coli
108
die thermodynamisch bevorzugten Aminosäuren Histidin und Cystein (H51 in yciA, H7 in
thiO und C689 in top1) begründet in der längeren Inkubationszeit beobachtet.
Im Vergleich zu den bisherigen Studien, in denen Cisplatin mit einzelnen Proteinen inkubiert
und auf Bindungsstellen analysiert wurde, kann man eine Bestätigung finden, dass die
Koordination an die thermodynamisch bevorzugten Seitenketten der Aminosäuren und H mit
längerer Inkubationszeit an Bedeutung gewinnen. So wurden im Protein Ubiquitin von zwei
Gruppen das Methionin M1 und Histidin H68 als Pt(II)-Koordinationsstelle unter
Verwendung der NMR-Spektroskopie und massenspektrometrisch mit der „top-down“Methode charakterisiert [65, 67]. Dabei wurden von den Arbeitsgruppen Reaktionszeiten
zwischen einem und sechs Tagen bei 37°C gewählt.
In Abbildung 5.38 ist die kristallographische Darstellung des Proteins mit den von Cisplatin
koordinierten Aminosäuren zu sehen [67].
Abb.5.38: Kristallstruktur des Proteins Ubiquitin mit M1 und H68 [67]
Die Bildung eines Chelatringes zwischen der Aminosäure Methionin M1 des Proteins und der
terminalen Aminogruppe würde die Koordination stabilisieren.
Im Zusammenhang mit Cisplatin oder anderen Metallkomplexen werden in der Krebstherapie
unterschiedliche
Proteine
diskutiert.
Eine
graphische
Darstellung
des
/Wirkmechanismus von Cisplatin nach LIPPERT ist in Abbildung 5.39 zu sehen.
Bindungs-
Proteintargets in Escherichia coli
109
Abb.5.39: Auswirkungen der Cisplatin-DNA-Addukte auf einige Proteine [123]
Wie aus der Abbildung 5.39 erkennbar sind mehrere Proteingruppen an den Mechanismen
beteiligt. So sind die high-mobility-group Proteine (HMG) bei der Reparatur der beispielhaft
durch die Koordination eines Metallfragmentes veränderten DNA wirksam, können aber
ebenso eine Reparatur verhindern. In der Diskussion steht auch die Verursachung der
Apoptose durch diese Proteinreaktionen [124]. Bevorzugt binden die HMG-Proteine an
gestörte oder geknickte DNA, wie z. B. durch eine Bindung an Cisplatin verursacht. Die
Struktur eines Cisplatin-DNA-Adduktes mit einem High-mobility-group-Protein nach
OHNDORF ist in Abb. 5.40 dargestellt.
Proteintargets in Escherichia coli
110
Abb.5.40: Struktur der an die DNA gebundenen HMG-Proteine [124]
Durch die Bindung der Proteine wird eine Reparatur der betroffenen DNA verhindert. So
wirkt Cisplatin besonders effektiv bei Tumoren, in denen eine Überexpression der Proteine
vorliegt, wie z. B. Hoden- oder Brustkrebs [125]
Eine Inhibition der Replikation beschädigter DNA wird durch einen Kontrollmechanismus
gewährleistet, in welchem das Genregulatorprotein p53 eine Rolle spielt. Es aktiviert die
Transkription eines Gens, welches letztendlich der beschädigten DNA bei ihrer Reparatur
verhilft. Bei Fehlen oder einem Defekt von p53 führt die ungehemmte Replikation der DNA
zu einer hohen Mutationsrate und zur Bildung von Zellen, die zu Krebs neigen. Bei etwa der
Hälfte aller Krebserkrankungen beim Menschen finden sich Mutationen im p53-Gen [116].
Eine zentrale Rolle in der Cisplatindiskussion stellt die Gruppe der MMR-Proteine
(Mismatch-Reparatur) dar. Die Bedeutung dieser Proteine beim Menschen wurde erkannt, als
entdeckt wurde, dass eine vererbte Veranlagung für bestimmte Krebsarten, z. B. bestimmte
Dickdarmkrebsformen, von einer Mutation im Gen für eines der Mismatch-ReparaturProteine verursacht wird. Da die meisten Krebsarten aus Zellen entstehen, in denen sich viele
Mutationen anhäufen, besteht für eine Zelle mit defekter Mismatch-Reparatur-Funktion ein
großes Risiko zu einer Krebszelle zu entarten. Die Reparaturproteine korrigieren Fehler, die
bei
der
DNA-Replikation
entstehen.
Die
durch
die
Fehlpaarung
erzeugte
Geometrieverzerrung der Doppelhelix wird von den Proteinen erkannt und behoben. Eine
DNA-Polymerase kann die entstandene Lücke in der Helix auffüllen und eine DNA-Ligase
schließt diese (siehe Abbildung 5.37).
Proteintargets in Escherichia coli
111
In E. coli ist das Protein mutS eines der Proteine aus der MMR-Gruppe, welche für eine
schnelle Fehlpaarungskorrektur verantwortlich sind. Die Struktur des Proteins mit einem
DNA-Fragment ist in der Abb.5.41 dargestellt.
Abb.5.41 Struktur des mutS-Proteins mit DNA und ADP-Region (rot) [106]
Das asymmetrisch erscheinende Dimer des Proteins „umfasst“ die DNA (in der Abbildung
5.41 oben, rot dargestellt). Unten ist in der Struktur (Abb.5.41, ebenfalls rot) des mutSProteins das gebundene ADP-Molekül zu sehen.
Bei der Umsetzung des Darmbakteriums mit Cisplatin wurde das Protein mutS, mit dem
Fragment [(NH3)2PtCl]+ modifiziert. Die koordinierte Aminosäure Methionin 609 befindet
sich in der ADP-Region. Durch diese Bindung wird einerseits die Konformation des Proteins
geändert und andererseits die Hydrolyse von ATP beeinflusst. Eine indirekte Deaktivierung
dieses Proteins würde eine Unterbrechung oder Verhinderung der Reparatur von
fehlerhafter/geschädigter DNA bedeuten. Könnte ein zytotoxischer Metallkomplex das mutS
Protein spezifisch koordinieren, würde die mutierte DNA nicht repariert werden und die Zelle
nicht überleben. Auch würde das Protein nicht agieren um den Fehler der DNA zu
korrigieren, welcher durch die Cisplatinbindung und damit Verkrümmung entstanden ist.
Ein weiteres Protein, welches in den Replikationsmechanismus der Zellen involviert ist,
wurde ebenfalls mit der Cisplatinmodifikation beschrieben. Die DNA-Helikase uvrD gehört
zu einer der in Abbildung 5.34 dargestellten Gruppe der NER-Proteine (nucleotide excision
repair). Eine Helikase nutzt in der Zelle die Energie aus der Hydrolyse von ATP, um sich auf
Proteintargets in Escherichia coli
112
der DNA zu bewegen und sie zu entwinden. Die uvrD-Helikase aus E. coli wird in der
Literatur im Zusammenhang mit dem MMR-Mechanismus diskutiert. Dabei entwindet uvrD
die DNA, nachdem mutS, mutL und mutH den beschädigten oder fehlerhaften Strang
repariert haben.
Weitere Proteine, welche z. B. in die Replikation, Transkription und Transkription der DNA
eingebunden sind, sind die Topoisomerasen. Es gibt drei Topoisomerasen, mit
unterschiedlichen Funktionen. Die Topoisomerase 1, die wie die Topoisomerase 3
energieunabhängig Einzelstrangbrüche verurscht, wurde bei der MudPIT-Analyse des
Bakteriums E. coli von einem Cisplatinfragment an der thermodynamisch bevorzugten
Aminosäure Cystein koordiniert.
Das Protein Thioredoxin 1 wurde in der MudPIT-Analyse an der Aminosäure H7 mit
Cisplatin koordiniert identifiziert. Die Thioredoxin-Reduktase, welche das Thioredoxin
oxidieren
und
reduzieren
kann,
reguliert
eine
Vielzahl
von
Rezeptoren
und
Transkriptionsfaktoren. In einer Studie mit dem [(bpy)Pt]2+-Komplex wurden mittels
MALDI-TOF-MS diese Bindungsstellen aus einem 1:10 Ansatz identifiziert [126]. Die
Massenspektren des reinen Proteins und des Gemisches mit dem [(bpy)Pt(en)]Cl2-Komplex
sind in der Abbildung 5.42 dargestellt.
Abb.5.42: MALDI-TOF MS-Spektrum des T. thermophilus HB8 Thioredoxins (A) und nach
der Umsetzung mit Pt(bpy)(en)2+ (B) [126]
Eine Zuordnung der Bindungsstelle erfolgte in dieser Studie nach weiterer Fragmentierung
und Hinzuziehen der UV/Vis Spektroskopie. Im Vergleich zum humanen Thioredoxinsystem
Proteintargets in Escherichia coli
113
wird die Aktivität des TRX-TRXR-Systems in E. coli nur unwesentlich von Cisplatin
beeinflusst [127].
Aus den ermittelten Proteintargets für Cisplatin in E. coli lässt sich über die Spektrenzahl (ni)
des jeweiligen Proteins feststellen, dass von den niedrig abundanten Proteinen in den Zellen
mindestens 10% koordiniert vorliegen müssen. Besonders die Modifikation der Proteine
mutS, uvrD oder top 1 mit Cisplatin würde somit die DNA-Reparatur-Funktion gestört
werden. In der folgenden Tabelle ist eine grobe Übersicht der Cisplatin Proteintargets
gegenüber der erwarteten Zahl an Peptiden in nicht koordinierter Form Oi dargestellt.
Tab.5.21:Konzentrationsbereiche für Cisplatin Proteintargets
Konzentrationsbereich
Hohe Abundanz
4
(~10 -105) 50 ≥ ni/Oi > 5.0
Mittlere Abundanz
(~103-104) 5.0 ≥ ni/Oi > 0,5
Niedrige Abundanz
(~102-10+) ni/Oi < 0,5
Nicht beobachtet
(~102-103)
Protein (ni/Oi)
g3p1 (485/29,7)
eftU (324/34,4)
mdH (145/22,5)
cisY (123/29,0)
enO (91/ 34,7)
rs6 (23/11,4)
artI (8/22,6)
top1 (4/80,7)
mutS, aldA, acrD, ceaN,
finQ, dceA, mdtA, ebgC,
rpoA (167/27,5)
pthp (38/6,68)
ch60 (224/45,3)
ompA (39/25,8)
speA (23/36,4)
thiO (17)
uvrD (2/58,3)
intD, yciA, mukE, rep5
his2, fimA1, dbpA, tdcE,
Da für Proteine höherer oder mittlerer Abundanz Addukte mit 1% oder weniger Kopien
massenspektrometrisch detektierbar sind, resultieren sicherlich diese Proteintargets aus
Wechselwirkungen von O- und S-Donoratomen an den Oberflächen der Proteine [131].
Serumproteine
114
6. Serumproteine
Proteine stellen für Cisplatin nach der Infusion den primären Reaktionspartner dar. Studien zu
Interaktionen von Cisplatin mit Proteinen ergaben folgende prozentuale Bindungsanteile für
Cisplatin an Serumproteine: 72,3% ± 6,5% für Albumin, 39,5% ± 2,5% für α1-acidGlycoprotein und 49,2% ± 1,9% für γ-Globulin [52, 132]. In der Abbildung 6.1 ist ein
silbergefärbtes Gel-Elektropherogram der Serumproteine dargestellt.
Abb.6.1: Silbergefärbtes Gel-Elektropherogram der Serumproteine [133]
Es ist eindeutig aus der 2D-PAGE-Untersuchung erkennbar, dass das Serumalbumin unter
den Serumproteinen in beträchtlicher Häufigkeit vorkommt. Daneben sind unter anderem die
Ig-Faktoren,
Fibrinogene,
Haptoglobine,
Serotransferrin,
Apolipoproteine
und
Serumproteine
115
Macroglobuline vertreten. Diese Proteine stellen insgesamt 90 % der Serumproteine dar, wie
ANDERSON gezeigt hatte (siehe Abbildung).
Abb.6.2: Verteilung der abundanten Serumproteine [134]
Im Rahmen dieser Arbeit sollen mit Hilfe der mit Flüssigchromatographie kombinierten
Tandem-Massenspektrometrie die Adduktbildung zwischen Serumproteinen und Cisplatin
analysiert werden. Dabei werden Proteintargets des Metallkomplexes aus zwei unabhängigen
Proben von Blutserum sowie den wichtigsten Serumproteinen gesucht bzw. aus NMRUntersuchungen bekannte Bindungsstellen bestätigt.
Das Antitumormittel Cisplatin wird in der Krebstherapie intravenös verabreicht. Der größte
Teil des Metallkomplexes erreicht (wie in Kapitel 1 und 3 erwähnt) nicht die DNA, sondern
bindet vor dem Erreichen der DNA an Serumproteine. Bisher konnte die Bildung von
Cisplatin-Addukten durch die drei abundanten Serumproteine Albumin, Serotransferrin und
Alpha-2-Macroglobulin massenspektrometrisch bestätigt werden [90], jedoch konnten bei
dieser Studie für die Proteine keine Angaben über die Koordinationsstellen getroffen werden.
Aus ESI-MS/MS Untersuchungen gelang es allerdings DYSON et al. Threonin T457 als
Cisplatinbindungsstelle in Serotransferrin aus einem 1:10 (Cisplatin:Serotransferrin)-Gemisch
nach 15 oder 30 min Inkubation bei 20°C und nach vorangegangenem Trypsinverdau zu
identifizieren.
Für die MudPIT-Analysen wurden die tryptisch gespaltenen Proteinmischungen aus den
Blutserumproben bzw. aus den einzelnen Serumproteinen Albumin, Serotransferrin,
Apolipoprotein A1, Alpha-1-Antitrypsin und Alpha-2-Macroglobulin auf die SCX-Säule
geladen und unter Verwendung von 12 chromatographischen Salzgradientenstufen
massenspektrometrisch analysiert. Die Gemische der jeweiligen Proteinproben sind nach der
Serumproteine
116
Vorbereitung auf eine eindimensionale RP-C18- Säule geladen und mit einem linearen
Acetonitril/Wasser/TFA-Gradienten über 180 min chromatographisch getrennt und ins
Finnigan LTQ Massenspektrometer eingeführt worden. Nach der Messung werden die
aufgenommenen ESI-MS/MS-Spektren unter Verwendung des SEQUEST- Algorithmus mit
den in Datenbanken eingetragenen Peptidspektren aus humanen Proteinen verglichen.
Die SEQUEST- Datenbanksuche beinhaltete die folgenden potentiellen platinierten
Aminosäurereste: C, D, E, H, K, M, S, T und Y. Die Massen dieser Reste wurden nach den
vier Massen 193, 210, 227 und 263 für die Cisplatinfragmente [195Pt]2+, [(NH3)195Pt]2+,
[(NH3)2195Pt]2+ und [(NH3)2195PtCl]+ entsprechend modifiziert. Für den Ladungsausgleich
wurden Protonenverluste bei den einzelnen Fragmenten berücksichtigt (ein Proton bei
[(NH3)2195PtCl]+, bei den drei restlichen oben genannten Cisplatinfragmenten zwei Protonen).
Es wurden bei der Datenbanksuche folgende Kriterien bezüglich der SEQUEST- Parameter
verwendet: XCorr-Wert für einfach geladene XCorr ≥ 1,8, für zweifach XCorr ≥ 2,5 und für
dreifach geladene Moleküle XCorr ≥ 3,5; Sp-Werte ≥ 500; Ionenzuordnung ≥ 30%; ∆CnWerte ≥ 0,1; RSp-Werte ≤ 6 und ∆m ± 2,5. Nach dem Erstellen einer ersten Liste der
Peptidsequenzen, die diese Bedingungen erfüllen, wurden die einzelnen MS/MS-Spektren
nach den folgenden Kriterien kritisch überprüft, um false-positive Zuordnungen zu
vermeiden:
(a) Vorhandensein einer adäquaten Zahl von platinierten b+- und y+-Ionen
(b) Vorliegen vom charakteristischen Isotopenmuster in einer signifikanten Zahl der als
platiniert zugeordneten b+- und y+-Ionen
(c) Vorliegen einer ausreichenden Zahl von b+- und y+-Ionen im Messbereich m/z > 1000,
welche relative Intensitäten aufweisen, die sich klar vom Untergrundrauschen
unterscheiden.
Nach dem Entfernen von Einträgen, die diese Kriterien nicht erfüllen, wurden die in den
nachfolgenden Unterkapiteln erläuterten Listen von Proteintargets erhalten. Es ist davon
auszugehen, dass in mehreren Fällen tatsächliche Proteintargets auf Grund von false-negative
Bewertungen aus der ursprünglichen Liste entfernt wurden.
Aus den Analysen der einzelnen Proteine mit Cisplatin werden ausschließlich Sequenzen
aufgeführt, deren XCorr-Werte ≥ 2,4 für zweifach geladene und XCorr ≥ 3,3 für dreifach
geladene Moleküle betragen. Außerdem müssen die Sp-Werte ≥ 500, ∆Cn-Werte ≥ 0,1, RSpWerte ≤ 5, ∆m ± 2,5 betragen und die Ionenzuordnung mindestens 30 % ergeben. Die
Analysen der Ansätze aus Cisplatin und Serumproteinen wurden nach unterschiedlichen
Serumproteine
117
Inkubationszeiten (3h, 168h) und z. T. mit drei- oder fünffachen Überschüssen an
Metallkomplex durchgeführt.
6.1. Cisplatinaddukte des Serumalbumins
6.1.1. Blutserumproben
Aus der MudPIT-Analyse der Blutserumproben konnten zahlreiche Cisplatin-Protein-Addukte
detektiert werden. Im Folgenden wird zunächst das Serumalbumin behandelt und mit den
Messungen des einzelnen Proteins mit Cisplatin verglichen. Mit diesem abundanten Protein
wurden mehrere Reaktionen mit Cisplatin angesetzt. Um die Ausbildung unterschiedlicher
Addukte zu ermöglichen, wurden die Reaktionszeiten und Verhältnisse der Ansätze variiert.
Mit 3h Inkubationszeit bei 37 °C wurden Reaktionen im Verhältnis 1:1, 1:3 und 1:5
(Albumin:Cisplatin) untersucht. Eine Inkubationszeit von 168h bei 37 °C wurde für das
Verhältnis 1:5 gewählt.
Die Blutserumanalyse für die erste unabhängige Probe ergab eine Liste von modifizierten
Peptiden aus Serumalbumin, die in der untenstehenden Tabelle aufgeführt sind.
Tab.6.1: Proteintargets von Cisplatin in Serumalbumin aus der MudPIT-Analyse der ersten
Blutserumprobe
Peptid
Bindungsstelle
z XCorr ∆Cn
K.ALVLIAFAQYLQQCPFEDH$V.K C34/E37/D38/H39 2
K.DVFLGM$FLYEYA.R
2
D324/M329
Sp
RSp Ionen
4,20
3,03
0,25
0,34
1165,1
585,2
3
1
18/38
14/22
K.DVFLGM$FLYEYAR.R
K.VFD$EFKPLVEEPQNLIK.Q
K.AVM$DDFAAFVE.K
1
D324/M329
D375/E376/K378 2
M548/D549/D550 2
4,10
4,06
2,97
0,24
0,12
0,18
1018,4
817,7
674,2
6
1
1
21/48
19/32
14/20
F.DE~FKPLVEEPQNLIK.Q
D375/E376/K378 2
4,66
0,01
771,5
1
19/28
K.AVM~DDFAAFVE.K
M548/D549/D550 2
2,85
0.12
542,3
6
12/20
K.ALVLIAFAQYLQQC#PFED.H C34/E37/D38/H39 3 3,95
0.17 1059,5
R.H#PYFYAPELLFF.A
2 3,02
0,35 685,7
H146/Y148
R.H#PYFYAPELLFFA.K
0,40 1102,1
H146/Y148/Y150 2 4,13
R.RH#PYFYAPELLFFA.K
0,29 606,6
H146/Y148/Y150 2 3,20
P.Y#FYAPELLFFAKR.Y
2 2,82
0,17 963,2
Y148
2+
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt] , # = [(NH3)2PtCl]+
3
1
1
1
1
23/68
15/22
19/24
16/26
18/24
Serumproteine
118
Unter den Bindungsstellen sind, falls mehrere potentielle Aminosäurereste vorhanden sind,
die wahrscheinlich bevorzugten Bindungsstellen aus der angegebenen Peptidsequenz fett
gedruckt. Die angegebenen ∆Cn-Werte beziehen sich auf die nächste, sich von der ersten
unterscheidende Peptidsequenz. Hierbei werden Eintragungen für gleiche Peptidsequenz aber
mit anderen Bindungsstellen (z. B. C34, E37 oder D38 statt H39 in der ersten Sequenz der
Tabelle) nicht berücksichtigt.
Es wurden insgesamt 12 Peptidsequenzen aus Serumalbumin identifiziert, wobei fünf mit dem
[(NH3)Pt]2+-, zwei von dem [(NH3)2Pt]2+- und fünf von [(NH3)2PtCl]+-Fragment koordiniert
wurden. Der Tabelle kann außerdem entnommen werden, dass zweimal C34/E37/D38/H39,
viermal der Bereich/H146/Y148/Y150, zweimal M329/D324, zweimal D375/E376/K378 und
zweimal M548/D549/D550 platiniert wurden.
Nachstehend ist die Liste der modifizierten Proteintargets aus Serumalbumin mit Cisplatin aus
der MudPIT-Analyse mit der zweiten unabhängigen Blutserumprobe dargestellt.
Tab.6.2: Proteintargets von Cisplatin in Serumalbumin aus der MudPIT-Analyse der zweiten
Blutserumprobe
Peptid
Bindungsstelle
z XCorr ∆Cn
K.VFD$EFKPLVEEPQNLIK.Q
D375/E376
2
K.ALVLIAFAQYLQQCPFED$HV.K C34/E37/D38/H39 2
Sp
RSp Ionen
3,88
4,31
0,12 525,1
0,18 970,6
1
1
16/32
16/38
3,64
0,26 556,3
1
16/28
-.DE#FKPLVEEPQNLIK.D375/E376
3 3,77 0,11 712,3
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
4
26/56
F.DE~FKPLVEEPQNLIK.Q
D375/E376
2
Insgesamt sind vier Peptidsequenzen in der zweiten Tabelle aufgelistet. Davon wurden nach
den besten XCorr-Werten aus den jeweiligen Sequenzen dreimal D375/E376 und einmal
C34/E37/D38/H39 koordiniert.
Aus dem Vergleich der Tabellen 6.1 und 6.2 lässt sich erkennen, dass in beiden
Blutserumproben die Bindungsstellen C34/E37/D38/H39 und D375/E376 identifiziert
wurden.
Die
restlichen
Koordinationsstellen
D324/M329,
M548/D549/D550
und
H146/Y148/Y150 sind aus der ersten Blutserumprobe identifiziert worden und müssen
deswegen in den Einzelproben validiert werden.
Die in den Tabellen 6.1 und 6.2 aufgelisteten Peptidsequenzen wurden bezüglich ihrer
MS/MS-Spektren geprüft und bei gleichen Koordinationsstellen miteinander verglichen, um
Serumproteine
119
falsche Zuordnungen zu vermeiden bzw. um im Falle von benachbarten möglichen
Bindungsstellen, falls möglich, die wahrscheinlichere Position zu ermitteln. Dazu wird im
Folgenden eine Auswahl an Beispielen mit den unterschiedlichen Platinfragmenten erläutert.
Zu der mit [(NH3)Pt]2+ in der ersten Tabelle aufgeführten koordinierten Sequenz
[373VFD$EFKPLVEEPQNLIK389]2+ ist in der folgenden Abbildung 6.3 das zugehörige
MS/MS-Spektrum dargestellt. Die gleiche Sequenz konnte mit einem XCorr-Wert von 3,88
(statt 4,06 hier) auch in der zweiten Blutserumprobe detektiert werden.
Abb.6.3: MudPIT-MS/MS-Spektrum von [VFD$EFKPLVEEPQNLIK]2+ in Serumalbumin
aus der ersten Blutserumprobe
In dem MS/MS-Spektrum sind von 32 möglichen Peptidionen 19 zugeordnet. Alle acht b+Ionen liegen platiniert vor, bei den y+-Ionen ist keines der 11 dargestellten Ionen platiniert.
Das an Asparaginsäure 375 gebundene [(NH3)Pt]2+-Fragment ruft diese Verteilung bei den
einzelnen Ionen hervor. In der Abbildung 6.3 wird das charakteristische Platinmuster für das
Ion b10+ gezeigt. In der Tabelle 6.3 sind weitere potentielle Sequenzen, welche der Masse m/z
= 2256 (mit ∆m ± 2,5) entsprechen mit ihren SEQUEST-Werten dargestellt. Es sind zehn
Serumproteine
120
Peptidsequenzen nach abnehmenden XCorr-Werten aufgelistet, deren theoretische MS/MSSpektren teilweise Übereinstimmung mit dem experimentellen Spektrum aufweisen.
Tab.6.3: SEQUEST-Parameter von [VFD$EFKPLVEEPQNLIK]2+ im Vergleich zu den
anderen potentiellen Peptidsequenzen in Serumalbumin aus der ersten Blutserumprobe
Sequenz
K.VFD$EFKPLVEEPQNLIK.Q
K.VFDEFK$PLVEEPQNLIK.Q
K.LPEFAPGEEESGC$SGVVK$.P
K.VFDE$FKPLVEEPQNLIK.Q
K.LPEFAPGEEE$SGCSGVVK$.P
K.VFDEFKPLVE$EPQNLIK.Q
M.YVYWTQLNM$FQTLK$.Y
K.LPEFAPGEEESGCS$GVVK$.P
Q.EKLPMTFQS$C$VITK$.E
K.TKQNQILDE$EFQNSPPAS.V
XCorr
4,065
3,592
3,589
3,514
3,494
3,327
3,112
3,083
3,056
3,004
∆Cn
0
0,116
0,117
0,136
0,14
0,182
0,234
0,241
0,248
0,261
Sp
817,7
508,7
316
634
235,3
357,9
252,3
223,5
216,4
268,2
RSp
1
3
21
2
126
7
89
169
196
63
Ionen
19/32
16/32
15/34
17/32
13/34
15/32
12/26
13/34
11/26
13/34
Protein
ALBU_HUMAN
ALBU_HUMAN
DDR2_HUMAN
ALBU_HUMAN
DDR2_HUMAN
ALBU_HUMAN
RIB2_HUMAN
DDR2_HUMAN
THS7A_HUMAN
NEIL3_HUMAN
Aus der Auflistung lässt sich erkennen, dass von den 16 Aminosäuren der Peptidsequenz
neben D375 weitere zwei Reste (Lysin 378 und Glutaminsäure 376) als Koordinationsstellen
für das [(NH3)Pt]2+-Fragment in Frage kommen könnten. Die Modifikationen D375, E376
und K378 des Albumins erfüllen wie aus der Tabelle erkennbar die SEQUEST-Kriterien
bezüglich ihrer Sp- und XCorr-Werte. Allerdings weisen beide Sequenzen ∆Cn-Werte größer
als 0,1 sowie deutlich niedrigere Sp-Werte auf und können deshalb als weniger
wahrscheinlich eingeordnet werden Die Abwesenheit der Ionen yk+ für k ≥ 15 und das
Vorhandensein des charakteristischen Platinpatterns bei b3+ stellen klare Hinweise für die
Bevorzugung der Koordination der Asparaginsäureseitenkette (D375) dar. Jedoch ist eine
Unterscheidung zwischen zwei benachbarten Bindungsstellen anhand eines einzelnen Ions im
Bereich 300 < m/z < 800 wegen der Vielzahl mit unterschiedlichen Fragmentionen immer mit
Vorsicht zu bewerten.
Aus
der
MudPIT-Analyse
der
zweiten
Blutserumprobe
konnten
fünf
platinierte
Peptidsequenzen detektiert werden. Ein Beispiel eines mit dem [(NH3)2Pt]2+-Fragment
koordinierten Peptids stellt die Sequenz [375DE~FKPLVEEPQNLIK389]2+ dar, welche auch
bei der ersten Blutserumprobe detektiert werden konnte. In diesem Fall wird allerdings E376
statt D375 als die wahrscheinlichere Bindungsstelle aufgelistet. Das MS/MS-Spektrum ist in
der folgenden Abbildung zu sehen.
Serumproteine
121
Abb.6.4: MudPIT-MS/MS-Spektrum von [DE~FKPLVEEPQNLIK]2+ in Serumalbumin aus
der zweiten Blutserumprobe
Die Sequenz könnte insgesamt 28 b+- und y+-Ionen bei einer vollständigen Fragmentierung
bilden, davon sind 13 im oberen Spektrum und 16 in der Tabelle 6.4 von SEQUEST
zugeordnet. Bei den dargestellten b+-Ionen sind alle (ab b2+) platiniert, für die y+-Ionen würde
das nur für das Ion y14+ gelten, welches jedoch nicht detektiert wurde. Es sind die y+-Ionen
y3+, y6+ bis y9+, y11+ und y12+ im oberen Spektrum zugeordnet.
Die nachstehende Tabelle zeigt drei weitere Sequenzen aus der SEQUEST-Liste, welche der
Masse m/z = 2027 (mit ∆m ± 2,5) entsprechen.
Tab.6.4: SEQUEST-Parameter von [DE~FKPLVEEPQNLIK]2+ im Vergleich zu den anderen
potentiellen Peptidsequenzen in Serumalbumin aus der zweiten Blutserumprobe
Sequenz
F.DE~FKPLVEEPQNLIK.Q
F.D~EFKPLVEEPQNLIK.Q
F.DEFK~PLVEEPQNLIK.Q
K.E~K~CKNSHLYPLIE.T
XCorr
3,636
3,588
3,13
2,695
∆Cn
0
0,013
0,139
0,259
Sp RSp Ionen
Protein
556,3 1 16/28 ALBU_HUMAN
556,3 1 16/28 ALBU_HUMAN
403,2 8 14/28 ALBU_HUMAN
243,2 294 11/24 THS7A_HUMAN
Serumproteine
122
Potentiell können Modifizierungen der Aminosäuren D375 oder K378 in der Peptidsequenz
mit dem Cisplatinfragment [(NH3)2Pt]2+ ebenfalls vorliegen. Die vierte Sequenz mit einer
zweifachen Koordination weist, wie aus den SEQUEST-Werten erkennbar, nur wenig
Übereinstimmung auf und wird nicht weiter diskutiert. Auch K378 kann mit Sp < 500 und
∆Cn > 0,1 als Koordinationsstelle ausgeschlossen werden. Aus den Sp- und ∆Cn-Werten der
beiden ersten möglichen Bindungsstellen E376 und D375 wird deutlich, dass eine
Entscheidung zu Gunsten einer dieser Stellen anhand der SEQUEST-Parameter nicht möglich
ist. Für beide Sequenzen wird die gleiche Zahl von b+- und y+-Ionen zugeordnet. Die zur
Unterscheidung zwischen D375 und E376 benötigten y13+- und y14+-Ionen werden nicht
beobachtet (s. Abb. 6.4). Ein Vergleich der SEQUEST-Parameter aus Tabelle 6.3
verdeutlicht, dass dort für die Koordination von E376 der ∆Cn-Wert mit 0,116 auf eine
wesentlich schlechtere Übereinstimmung hindeutet, als die Zuordnung von D375. In der
folgenden Abbildung sind die Seitenketten beider Aminosäuren in dem Ausschnitt aus der
Proteinstruktur gezeigt.
D375
E376
Abb.6.5: Vergrößerter Ausschnitt aus der Proteinstruktur des Serumalbumins [PBD ID:
1n5uA [135])
Beide Seitenketten sind in einer Helix lokalisiert. Es ist dabei auch erkennbar, dass D375 an
der Oberfläche des Proteins wesentlich besser zugänglich ist. Beim Betrachten der in den
Tabellen 6.1 und 6.2 aufgelisteten Peptidsequenzen mit D375/E376 Koordinationen aus den
Analysen beider Blutserumproben und deren zugehöriger MS/MS-Spektren sowie
Serumproteine
123
SEQUEST-Tabellen konnte festgestellt werden, dass D375 die bevorzugte Bindungsstelle für
Cisplatin darstellt.
Ein weiteres Beispiel aus der MudPIT-Analyse der ersten Blutserumprobe bietet das Peptid
der Sequenz [546AVM$DDFAAFVE556]2+. Die Koordination dieser Peptidsequenz wurde aus
der
ersten
Blutserumprobe
zweimal
beobachtet,
wobei
auch
die
benachbarten
Asparaginsäuren potentielle Bindungsstellen bieten. Das MS/MS-Spektrum der am Methionin
548 mit dem [(NH3)Pt]2+-Rest koordinierten Peptidsequenz ist in Abbildung 6.6 dargestellt.
Abb.6.6: MudPIT-MS/MS-Spektrum von [AVM$DDFAAFVE]2+ in Serumalbumin aus der
ersten Blutserumprobe
Das oben dargestellte Spektrum enthält 14 zugeordnete b+- und y+-Ionen, wobei 20 durch eine
schrittweise Fragmentierung der Sequenz erhalten werden können. Daneben finden sich zwei
b2+- und zwei [bj-NH3]+-Ionen im Spektrum. Entsprechend der Modifizierung von M548 sind
die dargestellten Ionen b3+-b10+ platiniert. Die Tabelle 6.5 listet die SEQUEST-Werte aus der
Serumproteine
124
Übereinstimmung mit den theoretischen MS/MS-Spektren für weitere neun Sequenzen mit
der Masse m/z = 1425 (∆m ± 2,5) mit abnehmenden XCorr-Werten auf
Tab.6.5: SEQUEST-Parameter von [AVM$DDFAAFVE]2+ im Vergleich zu den anderen
potentiellen Peptidsequenzen in Serumalbumin aus der ersten Blutserumprobe
Sequenz
K.AVM$DDFAAFVE.K
R.MoxVFD$RGTKVF.V
L.YCVFFGC$FTK.K
R.MoxVFDRGT$KVF.V
H.YTKTSDGLC$VK.L
K.AVMD$DFAAFVE.K
M.YTEVWNNLGTTK.S
K.AVMDD$FAAFVE.K
H.ELAEAFQKDFTK.S
R.MoxDTD$LETMoxDL.D
XCorr
2,967
2,436
2,423
2,301
2,298
2,268
2,25
2,2
2,195
2,19
∆Cn
0
0,179
0,183
0,224
0,225
0,236
0,241
0,259
0,26
0,262
Sp
674,2
363,7
283,4
322,6
245,8
388,9
320,6
271,9
415,3
331,1
RSp Ionen
Protein
1
14/20 ALBU_HUMAN
35 10/18 FANCB_HUMAN
187 9/18 UBP22_HUMAN
85 10/18 FANCB_HUMAN
431 9/20
FRK_HUMAN
23 12/20 ALBU_HUMAN
89 11/22 S35B4_HUMAN
247 10/20 ALBU_HUMAN
12 11/22 CH3L2_HUMAN
72 10/18
U520_HUMAN
Die Peptidsequenz [AVM$DDFAAFVE]2+ des Serumalbumins stellt mit der Bindungsstelle
M548 die eindeutig beste Zuordnung in der Tabelle dar, was die deutlich höheren SEQUESTParameter im Vergleich zu den restlichen Sequenzen beweisen. Zwei weitere potentielle
Bindungsstellen in dem Proteinausschnitt wären die Aminosäuren D549 und D550, jedoch ist
die Übereinstimmung mit dessen theoretischem Spektrum wesentlich schlechter, was in den
Werten Sp < 500, RSp > 5 und ∆Cn > 0,1 deutlich wird. Bei der Koordination von D449 oder
D550 sollte im MS/MS-Spektrum in Abb. 6.6 das intensive nicht platinierte y8+-Ion nicht zu
beobachten sein. Da das Metallpattern nicht für y8+ beobachtet wird und außerdem keine yk+Ionen mit k > 8 vorhanden sind, ist die Koordination der Aminosäure M548 am
wahrscheinlichsten.
In der ersten Blutserumprobe konnten vier Peptidsequenzen mit den möglichen
Bindungsstellen H146, Y148 und/oder Y150 identifiziert werden. Da es sich bei diesen
potentiellen Koordinationsstellen um benachbarte Aminosäuren in der Proteinsequenz
handelt, wird als Beispiel das MS/MS-Spektrum der vom [(NH3)2PtCl]+-Fragment
koordinierten
Peptidsequenz
[148Y#FYAPELLFFAKR160]2+
gezeigt,
das
eine
H146
Koordination ausschließt. Das Proteinstück bindet an der Aminosäure Tyrosin 148 das
Cisplatinfragment. Das MS/MS-Spektrum ist in der folgenden Abbildung dargestellt.
Serumproteine
125
Abb.6.7: MudPIT-MS/MS-Spektrum von [Y#FYAPELLFFAKR]2+ in Serumalbumin aus der
ersten Blutserumprobe
Das oben dargestellte MS/MS-Spektrum enthält 15 (18 von SEQUEST zugeordnet) von 24
möglichen Ionen. Davon sind sieben b+- und acht y+-Ionen zugeordnet. Wie aus der
koordinierten Sequenz erkennbar, sind alle b+- (b1+-b4+, b8+, b10+ und b12+ im Spektrum
dargestellt) aber keine y+-Ionen (y4+, y5+ und y7+ bis y12+) platiniert. Eine Auflistung sonstiger
möglicher Peptidsequenzen, welche die Masse m/z = 1929 (mit ∆m ± 2,5) tragen, ist in der
Tabelle 6.6 aufgeführt.
Serumproteine
126
Tab.6.6: SEQUEST-Parameter von [Y#FYAPELLFFAKR]2+ im Vergleich zu den anderen
potentiellen Peptidsequenzen in Serumalbumin aus der ersten Blutserumprobe
Sequenz
P.Y#FYAPELLFFAKR.Y
L.E#T#T#AYFFMK.P
E.VYYFGE#VHM#KK.G
E.VYYFGE#VHMK#K.G
E.VYYFGE#VHMKK#.G
Y.PMPVMoxAYIQD#AQKR.L
G.S#RDGGASS#T#GSR.D
G.S#RDGGASST#GS#R.D
Y.VY#LS#MAYYFSR.D
L.ILELKLPPTDAYY#K.L
XCorr
2,824
2,353
2,238
2,217
2,139
2,048
2,024
2
1,985
1,968
∆Cn
0
0,167
0,207
0,215
0,242
0,275
0,283
0,292
0,297
0,303
Sp
963,2
331,4
357,5
357,5
357,5
275,1
272,5
289,7
361,7
320,2
RSp
1
67
45
45
45
197
207
154
42
86
Ionen
18/24
8/16
11/20
11/20
11/20
11/26
11/22
11/22
10/20
10/26
Protein
ALBU_HUMAN
FMN2_HUMAN
KCNF1_HUMAN
KCNF1_HUMAN
KCNF1_HUMAN
MA2A2_HUMAN
MINP1_HUMAN
MINP1_HUMAN
FTMT_HUMAN
U5S1_HUMAN
In der Tabelle sind neben der an Y148 koordinierten Peptidsequenz aus Serumalbumin
weitere Sequenzen aus sechs verschiedenen Proteinen (fmn2, kcnf1, ma2a2, minp1, ftmT und
u5s1) aufgelistet. Beim Betrachten deren SEQUEST-Werte wird jedoch deutlich, dass diese
Sequenzen die erforderlichen Kriterien eindeutig nicht erfüllen (für alle Sequenzen gilt Sp <
500, RSp >> 6, ∆Cn > 0,1). Da Übereinstimmungen der theoretischen Spektren mit den
beobachteten massenspektrometrischen Fragmentierungen alle nicht zufrieden stellend sind,
kann Y148 als Koordinationsstelle zugeordnet werden. Interessant ist hier die Tatsache, dass
Y150 nicht als Bindungsstelle in der SEQUEST-Liste vorkommt, d.h. ∆Cn für Y150 muss
größer als 0,303 sein. Hierfür ist die eindeutige Zuordnung von b1+, y11+ und y12+
entscheidend.
6.1.2. Serumalbumin:Cisplatin Proben
Für eine Validierung der bei den Blutserumproben erhaltenen Bindungsstellen wurden
Ansätze mit den einzelnen Proteinen und dem Zytostatikum nach unterschiedlichen
Inkubationszeiten analysiert. Die Untersuchungen dienen auch der Überprüfung der
Reproduzierbarkeit der LC/ESI-MS2- bzw. MudPIT-Analysen. Dazu sind die jeweiligen
Reaktionslösungen über eine RP-C18-Säule ohne vorherige SCX-Extraktion getrennt und
direkt ins gekoppelte Finnigan LTQ Massenspektrometer injiziert worden, um ESI-MS/MSMessungen durchzuführen. Im Folgenden werden die Tabellen der einzelnen Analysen
aufgeführt.
Serumproteine
127
Tab.6.7: Koordinierte Peptidsequenzen aus dem 1:1 Ansatz Albumin:Cisplatin nach 3h
Inkubation bei 37°C
Peptid
P.Y§FYAPELLFFAK.R
Y.FY§APELLFFAK.R
Bindungsstelle
Y148/Y150
D375
z XCorr ∆Cn Sp RSp Ionen
2 3,39 0,82 1453,2 1 17/22
2 3,11 0,71 866,9
1 17/20
K.VFD$EFKPLVEEPQNLIK.Q
D375
2
V.FD$EFKPLVEEPQNLIK.Q
D375
3
Y.FY$APELLFFAK.R
Y150
2
R.H$PYFYAPELLFFAK.R
H146/Y148/Y150 2
P.Y$FYAPELLFFAK.R
Y148/Y150
2
K.DVFLGM$FLYEY.A
M329
2
A.VM$DDFAAFVEK.C
M548
2
5,35
4,25
3,15
2,88
2,79
2,54
2,47
0,54
0,69
0,53
0,69
0,81
0,65
0,77
924,6
1010,6
1111,5
760,9
791,7
513,2
615,5
1
1
1
1
1
1
1
20/32
29/60
16/20
15/26
16/22
11/20
16/20
F.DE~FKPLVEEPQNLIK.Q
E.FAEVSK~LVTDLTK.V
K.VFD~EFKPLVEEPQNLIK.Q
V.FD~EFKPLVEEPQNLIK.Q
3,85
3,81
3,71
3,59
0,67
0,81
0,65
0,81
949,0
1540,1
1442,7
864,7
1
2
1
1
25/56
18/24
32/64
26/60
F.DE#FKPLVEEPQNLIK.Q
D375/E376
2 3,66 0,51 508,0
F.DEFK#PLVEEPQNLIK.Q K378/E376/D375 3 3,48 0,46 910,0
F.Y#APELLFFAK.R
Y150
2 2,60 0,77 781,9
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
1
1
16/28
24/56
14/18
D375/E376
K233
D375
D375
3
2
3
3
Die Tabelle 6.7 enthält die koordinierten Sequenzen des äquimolaren Systems
Cisplatin:Serumalbumin mit den vier möglichen Platinfragmenten. Es wurde sechsmal die
Seitenkette des Bereiches H146/Y148/Y150, einmal K233, einmal M329, siebenmal
D375/E376/K378 und einmal M548 koordiniert.
Von den in den Blutserumproben identifizierten Bindungsstellen (siehe Tabelle 6.1 und 6.2)
konnten in dieser Analyse H146/Y148/Y150, M329, D375/E376/K378 und M548 wiederholt
identifiziert werden und können somit als charakteristische Koordinationspositionen für
Cisplatin eingeordnet werden. Die mögliche Bindungsstelle K233 ist in der äquimolaren
Albumin:Cisplatin-Analyse zum ersten Mal bestimmt und muss deswegen bei weiteren
Ansätzen validiert werden.
Aus dem 1:3 Ansatz Serumalbumin:Cisplatin konnten nach 3h Inkubation bei 37°C die in der
Tabelle 6.8 aufgelisteten koordinierten Peptidsequenzen identifiziert werden. In dieser
Untersuchung konnten aus dem Bereich C34-H39 des Proteins E37 und C34 als potentielle
Serumproteine
128
Bindungsstellen identifiziert werden. Diese und die weiteren Sequenzen sind in der folgenden
Tabelle zu sehen.
Tab.6.8: Koordinierte Peptidsequenzen aus dem 1:3 Ansatz Albumin:Cisplatin nach 3h
Inkubation bei 37°C
Peptid
K.VFD$EFKPLVEEPQNLIK.Q
Bindungsstelle
D375
z XCorr ∆Cn
Sp RSp Ionen
3 3,52 0,58 940,6
1 28/64
K.ALVLIAFAQYLQQCPFE~DHV.K C34/E37/D38/H39 3
K.VFD~EFKPLVEEPQNLIK.Q
D375
3
E.FAE~VSKLVTDLTK.V
E230
2
4,34
3,36
2,48
0,66 1715,1
0,72 1207,8
0,73 1460,5
2
1
1
34/76
30/64
16/24
K.ALVLIAFAQYLQQC#PFEDHV.K
C34/E37/D38
3 3,35 0,65 715,8
P.Y#FYAPELLFFAK.R
Y148/Y150
2 3,13 0,65 1931,5
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
1
24/76
19/22
Bei diesem Verhältnis sind die in den Blutserumproben gefundenen Bindungsstellen
C34/E37/D38/H39, Y148/Y150 und D375 wieder identifiziert worden. Zum ersten Mal wurde
die Glutaminsäure E230 als Bindungsstelle zugeordnet und bedarf noch einer Bestätigung.
Allerdings wurde die benachbarte Aminosäure K233 als potentielle Koordinationsstelle bei
dem 1:1 Ansatz (Tab. 6.7) identifiziert. Die Seitenkette aus dem Bereich C34/E37/D38/H39
ist zweimal, Y148/Y150 einmal, E230 einmal und D375 zweimal als Bindungsstelle
zugeordnet worden.
Eine weitere Liste (Tabelle 6.9) koordinierter Peptide des Serumproteins wurde aus dem
Reaktionsansatz Albumin-Cisplatin im Verhältnis 1:5 nach 3h Inkubation bei 37°C erhalten.
Es konnten erneut Bindungsstellen (Y148/Y150, M329, D375) aus den Analysen der
Blutserumproben und der Protein:Cisplatin-Gemische bestätigt werden. Diese sind in der
untenstehenden Tabelle gezeigt. Zusätzlich wurde ein Ansatz aus Serumalbumin und
Cisplatin im Verhältnis 1:5 nach einer Inkubation von 168h bei 37 °C untersucht. Mit der
längeren Reaktionszeit sollte sowohl die kinetische Langzeitstabilität der gefundenen
Bindungsstellen als auch die mögliche langsame Ausbildung von thermodynamisch
bevorzugten Koordinationen (z. B. Histidinreste) geprüft werden. Eine Liste der von einem
Cisplatinfragment koordinierten Peptide des Serumalbumins aus diesem Ansatz findet sich
ebenfalls in der nachstehenden Tabelle.
Serumproteine
129
Tab.6.9: Koordinierte Peptidsequenzen aus den 1:5 Ansätzen Albumin:Cisplatin nach 3h und
nach 168h Inkubation bei 37°C
Peptid
1:5 nach 3h bei 37°C
K.VFD$EFKPLVEEPQNLIK.Q
K.DVFLGM$FLYEYAR.R
P.Y$FYAPELLFFAK.R
Bindungsstelle
z
XCorr
D375
M329
Y148/Y150
3
3
2
4,15
4,07
2,64
0,60 1051,4
0,74 1079,7
0,77 1310,7
1
2
1
30/64
23/48
18/22
F.DE~FKPLVEEPQNLIK.Q
K.VFD~EFKPLVEEPQNLIK.Q
E376/D375
D375
3
3
3,69
3,57
0,69 1034,7
0,69 994,7
3
1
27/56
28/64
C34/E37/D38/H39 3
C34/E37/D38/H39 2
2
Y150
4,64
4,53
3,74
0,82 1193,9
0,75 523,5
0,87 1497,8
2
1
1
30/72
15/36
17/20
K.ALVLIAFAQYLQQCPFED$H.V C34/E37/D38/H39 2
K.ALVLIAFAQYLQQCPFEDH$VK.L C34/E37/D38/H39 0
K.VFD$EFKPLVEEPQNLIK.Q
2
D375
F.Y$APELLFFAK.R
2
Y150
Y.FY$APELLFFAK.R
2
Y150
4,19
3,80
4,38
3,07
2,82
0,47 722,4
0,63 676,4
0,48 624,7
0,81 1210,4
0,39 601,2
1
1
1
1
1
23/72
26/80
17/32
17/18
14/20
P.Y#FYAPELLFFAK.R
2
3,40
0,75 1797,8
Y148/Y150
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
19/22
1:5 nach 168h bei 37°C
K.ALVLIAFAQYLQQCPFE§DH.V
K.ALVLIAFAQYLQQC§PFEDH.V
Y.FY§APELLFFAK.R
∆Cn
Sp
RSp Ionen
Aus diesen Analysen wurden insgesamt 14 Proteinfragmente erhalten. Die Bindungsstellen
Y148/Y150, M329 und D375/E376 (in zwei unterschiedlichen Sequenzen) konnten wieder als
bevorzugte Stellen nach 3h Reaktion bei 37°C bestätigt werden und können als spezifische
Koordinationsstellen für Cisplatin betrachtet werden. Alle nach 168h Inkubation
identifizierten Peptidsequenzen weisen die bekannten Bindungsstellen C34/E37/D38/H39 (bei
zwei unterschiedlichen Sequenzen), Y148/Y150 (bei drei unterschiedlichen Sequenzen) und
D375 auf. Interessant ist die Tatsache, dass die O-Donorreste Y148/Y150 und D375 auch
nach 168h Inkubation detektierbar sind. Dieser Befund bestätigt die hohe kinetische Stabilität
solcher
Bindungsstellen.
Außerdem
weist
die
Analyse
darauf
hin,
dass
Histidinbindungsstellen für eine Cisplatinkoordination in Serumalbumin offensichtlich
weniger begünstigt sind als bisher angenommen.
In der unten stehenden Abbildung ist aus der Analyse nach 3h Inkubation das MS/MSSpektrum des Peptids [324DVFLGM$FLYEYAR336]3+, welches mit [(NH3)Pt]2+ koordiniert
Serumproteine
130
wurde, dargestellt. Es muss hier noch geklärt werden, ob die benachbarten Aminosäuren
D324 oder Y332 auch als Cisplatinbindungsstellen in Frage kommen.
Abb.6.8: MS/MS-Spektrum von [DVFLGM$FLYEYAR]3+ aus dem 1:5 Ansatz
Albumin:Cisplatin nach 3h Inkubation bei 37°C
In dem Spektrum sind von den 48 möglichen (für einfach- und zweifach geladene, von
SEQUEST 23 zugeordnet) Ionen des Proteinstückes [324DVFLGM$FLYEYAR336]3+ 10
einfach geladene Ionen zugeordnet worden. Wie aus der Bindungsstelle des [(NH3)Pt]2+Fragmentes in der Sequenz erkennbar, sind die Ionen b6+ bis b12+ und y8+ bis y12+ platiniert. Es
sind im oberen Spektrum die Ionen b2+, b7+ und b8+ sowie y1+ bis y7+ dargestellt. Das
Nichtvorhandensein von yk+-Ionen mit k > 7 bei der gleichzeitig hohen relativen Intensität des
y7+-Ions spricht eindeutig für M329 als Bindungsstelle.
Für die Molekülmasse von m/z = 1835 (∆m ± 2,5) sind weitere koordinierte Peptidsequenzen
grundsätzlich möglich. Diese Modifikationen sind in der Tabelle 6.10 dargestellt.
Serumproteine
131
Tab.6.10: SEQUEST-Parameter von [DVFLGM$FLYEYAR]3+ im Vergleich zu anderen
potentiellen Peptidsequenzen aus dem 1:5 Ansatz Albumin:Cisplatin nach 3h Inkubation bei
37°C
Sequenz
K.DVFLGM$FLYEYAR.R
K.D$VFLGMFLYEYAR.R
K.DVFLGMFLY$EYAR.R
K.DVFLGMFLYE$YAR.R
K.PLVEEPQNLIK$QNC.E
V.PQVST$PT$LVEVSR.N
V.PQVS$TPT$LVEVSR.N
V.PQVST$PTLVE$VSR.N
V.PQVST$PTLVEVS$R.N
F.QNALLVRY$T$K$.K
XCorr
4,07
3,441
1,647
1,122
1,056
1,054
0,955
0,946
0,934
0,876
∆Cn
0
0,154
0,595
0,724
0,741
0,741
0,765
0,768
0,771
0,785
Sp
RSp Ionen
1079,7
2 23/48
1103,5
1 23/48
258,3
3 15/48
157,5
15 13/48
239,4
4 14/52
217,1
5 13/48
172,7
10 1248
183,4
8
1248
171,1
11 1248
203,6
6 11/36
Wie aus den SEQUEST-Parametern zu entnehmen ist, kommen nur die beiden ersten
Proteinstücke als koordinierte Sequenzen in Betracht. Für die restlichen Peptidsequenzen sind
die Sp-Werte alle Sp < 500 und ∆Cn > 0,59. Aus der Sequenz [DVFLGM$FLYEYAR]3+
stellen die Aminosäuren D324, M329, Y332 und E333 potentielle Bindungspartner für ein
Cisplatinfragment dar. Die einzigen zufriedenstellenden Übereinstimmungen zu dem
aufgenommenen MS/MS-Spektrum werden für die theoretischen MS/MS-Spektren zu dieser
Peptidsequenz mit der Koordinaiton an der Aminosäure M329 oder D324 erreicht (48%
Ionenzuordnung). Anhand der wesentlich besseren XCorr- und ∆Cn-Werte im Vergleich zu
D324 kann M329 als Bindungsstelle zugeordnet werden.
Die Lokalisierung der Seitenketten der beiden denkbaren Bindungsstellen D324 und M329 im
Protein ist in der folgenden Abbildung zu sehen.
Serumproteine
132
M329
D324
Abb.6.9: Ausschnitt aus der Proteinstruktur des Serumalbumins
In der Vergrößerung ist der Ausschnitt der gesamten Proteinstruktur mit den zwei
Seitenketten dargestellt, welche für das [(NH3)Pt]2+-Fragment als Koordinationsstellen in
Frage kommen: D324 und M329. Es ist aus der Darstellung deutlich ersichtlich, dass das
Methionin sterisch am besten zugänglich für eine Koordination ist.
Wie schon erläutert, lassen die SEQUEST-Parameter häufig bei benachbarten Resten, wie
zum Beispiel zwischen C34 und E37, keine eindeutige Zuordnung zu. Da auf Grund der
SEQUEST-Parameter die Zuordnung eines bestimmten Restes in dem Bereich C34-H39 nicht
bestätigt möglich ist, wird an dieser Stelle als Beispiel das MS/MS-Spektrum der mit [Pt]2+
koordinierten Sequenz [ALVLIAFAQYLQQC§PFEDH]2+ aus dem 1:5 Ansatz nach 168h
(Tab. 6.9) näher analysiert.
Serumproteine
133
Abb.6.10: MS/MS-Spektrum von [ALVLIAFAQYLQQC§PFEDH]2+ aus dem 1:5 Ansatz
Serumalbumin:Cisplatin nach 168h Inkubation bei 37°C
Im Spektrum wurden 15 Massenpeaks den Fragmentionen des Peptids zugeordnet. Aus der
Sequenz ist erkennbar, dass je 18 b+- und y+-Ionen gebildet werden können. Fünf dieser b+Ionen (b14+ bis b18+) und 13 der y+-Ionen (y6+ bis y18+) sollen platiniert sein. Im Spektrum sind
aus der b+-Serie nur die Ionen b5+ - b8+, b10+, b12+ und b13+ sowie asu der y+-Serie die Ionen
y6+, y7+, y9+ -y14+ einbezogen. Die Beobachtung von nur platinierten yk+-Ionen mit k ≥ 6 sowie
das Fehlen der platinierten Ionen bj+ für j > 13 steht eindeutig im Einklang mit der Zuordnung
von C34 als Bindungsstelle. In allen weiteren Peptidsequenzen mit den möglichen
Koordinationsstellen C34, E37, D38, H39 fehlen auch die analogen b+-Ionen mit
Aminosäure-Resten n ≥ 34.
Die folgende Tabelle enthält weitere potentielle Peptidsequenzen aus Serumalbumin, deren
Masse ebenfalls m/z = 2400,5 (∆m ± 2,5) beträgt.
Serumproteine
134
Tab.6.11: SEQUEST-Parameter von von [ALVLIAFAQYLQQC§PFEDH]2+ im Vergleich zu
den anderen potentiellen Peptidsequenzen aus dem 1:5 Ansatz Serumalbumin:Cisplatin nach
168h Inkubation bei 37°C
Sequenz
XCorr
K.ALVLIAFAQYLQQC§PFEDH.V
4,533
K.ALVLIAFAQYLQQCPFE§DH.V
4,168
K.ALVLIAFAQYLQQCPFED§H.V
4,161
K.ALVLIAFAQYLQQCPFEDH§.V
4,132
K.ALVLIAFAQY§LQQCPFEDH.V
2,578
K.D§VFLGM§FLYEYARRHP.D
1,124
K.ATKEQLKAVM§DDFAAFVE§.K 1,065
K.ATKEQLKAVMD§DFAAFVE§.K 0,951
K.ATKEQLKAVMDD§FAAFVE§.K 0,911
K.ATKEQLKAVM§D§DFAAFVE.K 0,89
∆Cn
0
0,081
0,082
0,089
0,431
0,752
0,765
0,79
0,799
0,804
Sp
523,5
523,5
523,5
523,5
163,5
81,3
55,4
55,4
55,4
53,6
RSp
1
1
1
1
2
3
6
6
6
7
Ionen
15/36
15/36
15/36
15/36
10/36
7/30
6/34
6/34
6/34
6/34
Anhand der SEQUEST-Parameter kann jedoch wiederum die bevorzugte Koordination einer
der vier Aminosäuren des Proteinstückes [ALVLIAFAQYLQQC§PFEDH]2+ nicht eindeutig
bestätigt werden. Neben dem Cystein 34 stehen E37, D38 und H39 mit ihren Seitenketten als
potentielle Bindungsstellen für das Metallfragment zur Verfügung. Verglichen mit der
Bindungsstelle C34 weisen die drei Reste E37/D38/H39 relativ hohe ∆Cn-Werte von 0,0810,089 auf. Allerdings können für alle vier potentiellen Bindungsstellen 15 b+- und y+-Ionen
zugeordnet werden. Die restlichen Sequenzen aus der Tabelle 6.11 können auf Grund sehr
niedriger SEQUEST Werte (z. B. Sp-Werte < 200) eindeutig als Bindungsstellen
ausgeschlossen. Die Zugänglichkeit der oben genannten Aminosäuren ist in dem Ausschnitt
der Proteinstruktur in Abb.6.11 gezeigt.
Serumproteine
135
C34
D38
E37
H39
Abb.6.11:Ausschnitt aus der Proteinstruktur des Serumalbumins
Die Abbildung zeigt eine Vergrößerung des von Cisplatin koordinierten Bereiches im Protein,
wobei die Seitenketten der Aminosäuren C34, E37, D38 und H39 auf ihre Zugänglichkeit für
eine Bindung betrachtet werden. Wie aus der Darstellung erkennbar, ist eine Koordination der
Aminosäuren C34, E37 oder D38 begünstigt. Der Imidazolring von H39 liegt nicht an der
Oberfläche der Helix, wie das bei den restlichen gekennzeichneten Bindungsstellen der Fall
ist und wird somit als weniger wahrscheinlich als Koordinationsstelle vermutet. Dabei ist
auch zu bemerken, dass die freie Thiolatgruppe des Cysteins 34 schon von mehreren Autoren
als Bindungsstelle des Cisplatins identifiziert wurde, z. B. durch Blockierung der
Thiolatfunktion [12-15].
Zusammenfassend konnten aus der Untersuchung zweier unabhängiger Blutserumproben und
der Serumalbumin:Cisplatin-Ansätze die Seitenketten der Aminosäuren C34 (E37, D38),
Y150 (Y148), M329, D375 und M548 als Cisplatinbindungsstellen ermittelt werden. Somit
können diese Bindungsstellen als spezifisch für das Zytostatikum angesehen werden. Die
Lokalisierung der wahrscheinlich koordinierten Seitenketten C34, Y150, M329, D375 und
M548 wird in der folgenden Abbildung illustriert.
Serumproteine
136
Abb.6.12: Serumalbuminstruktur mit bevorzugten Cisplatinbindungsstellen
Die Seitenketten von drei bevorzugten Bindungsstellen für Cisplatin liegen auf der Oberfläche
des Proteins verteilt. Die Aminosäuren M329, D375 und M548 sind dabei in α-Helices des
Transportproteins lokalisiert, dagegen sind die Aminosäuren C34 und Y150 in zu
Bindungstaschen gehörenden flexiblen Schleifen (L55-P59 und R144-Y150) positioniert.
6.2. Cisplatinaddukte des Serotransferrins
6.2.1. Blutserumproben
Ein
weiteres
abundantes
Serumprotein,
dessen
Bindungsverhalten
sowohl
aus
Reaktionsansätzen des Proteins mit Cisplatin als auch aus der Blutserumanalyse
massenspektrometrisch untersucht wurde, ist das Serotransferrin. Für die Untersuchungen mit
dem Einzelprotein wurden wieder Ansätze aus Protein:Cisplatin 1:1 und 1:5 nach 3h
Inkubation bei 37 °C und 1:5 nach 168h Inkubation bei 37 °C verwendet.
Aus den MudPIT-Analysen der beiden Blutserumproben konnte eine Liste aus koordinierten
Peptiden des Serotransferrins erhalten werden, die in Tabelle 6.12 gezeigt wird.
Serumproteine
137
Tab.6.12: Proteintargets von Cisplatin in Serotransferrin aus den MudPIT-Analysen der
Blutserumproben
Peptid
erste Blutserumprobe
K.IM§NGEADAMSLDGGFVYIAGK.C
Bindungsstelle
z XCorr ∆Cn
RSp Ionen
4,49
0,48 560,3
1
22/40
2
2,86
0,10 890,1
1
15/22
D.NCED~TPEAGYFAVAVVK.K
C418/E419/D420 2
K.IM~NGEADAMoxSLDGGFVYIAGK.C M382/E385/D387 2
3,94
5,14
0,40 837,8
0,50 870,5
1
1
21/32
19/40
M.NGE#ADAMoxSLDGGFVYIAGK.C
E385/D387
2 4,57 0,23 1503,6
2+
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt] , # = [(NH3)2PtCl]+
1
20/36
zweite Blutserumprobe
K.MoxY$LGYEYVTAIR.N
M382/E385/D387 2
Sp
Y314
Bei den Analysen wurden insgesamt fünf Peptidsequenzen des Serotransferrins mit einer
möglichen Cisplatinkoordination identifiziert. Bei den in Tabelle 6.12 angegebenen
Peptidsequenzen handelt es sich um drei mögliche Bindungsbereiche/-stellen: Y314,
C418/E419/D420 und M382/E385/D387. In beiden Blutserumproben ist somit der Bereich
M382/E385/D387 als Bindungsstelle identifiziert worden. Die restlichen Positionen für eine
Cisplatinkoordination müssen in den Einzelanalysen bestätigt werden.
Die Peptidsequenzen aus der Tabelle wurden auf die Qualität der MS/MS-Spektren geprüft,
um wiederum falsche Zuordnungen auszuschließen und bei denkbaren benachbarten
Aminosäuren zu Gunsten einer Bindungsstelle entscheiden zu können.
In der folgenden Abbildung ist das MS/MS-Spektrum der mit [(NH3)2Pt]2+ koordinierten
Sequenz
[381IM~NGEADAMoxSLDGGFVYIAGK401]2+
dargestellt.
Hierbei
soll
die
Bevorzugung einer der drei möglichen Koordinationsstellen M382/E385/D387 festgestellt
werden.
Serumproteine
138
Abb.6.13: MudPIT-MS/MS-Spektrum von [IM~NGEADAMoxSLDGGFVYIAGK]2+ in
Serotransferrin aus der zweiten Blutserumprobe
In dem oberen Spektrum konnten von 40 möglichen Ionen 19 b+- und y+-Ionen (17 im
Spektrum dargestellt) zugeordnet werden. Aus den Abspaltungen der einzelnen Aminosäuren
sollten der Bindungsstelle Methionin 382 entsprechend die Ionen b2+ bis b20+ und aus der ySerie das y20+ platiniert sein. Den Massenspeaks in Abbildung 6.13 wurden Ionen b8+ und b12+
sowie y4+, y5+ und y7+ bis y19+ zugeordnet. Für eine Entscheidung zu Gunsten einer der drei
potentiellen Bindungsstellen sind vor allem die Ionen yk+ mit k ≥ 15 bei D387 und k ≥ 17 bei
E395 als Koordinationsstellen ausschlaggebend.
Die folgende Tabelle listet die Peptidsequenzen auf, welche der Masse von m/z = 2403 (mit
∆m ± 2,5) entsprechen.
Serumproteine
139
Tab.6.13: SEQUEST-Parameter von [IM~NGEADAMoxSLDGGFVYIAGK]2+ im Vergleich
zu anderen potentiellen Peptidsequenzen in Serotransferrin aus der zweiten Blutserumprobe
Sequenz
K.IM~NGEADAMoxSLDGGFVYIAGK.C
K.IMNGE~ADAMoxSLDGGFVYIAGK.C
K.IMNGEAD~AMoxSLDGGFVYIAGK.C
K.IMoxNGE~ADAMSLDGGFVYIAGK.C
K.IMoxNGEAD~AMSLDGGFVYIAGK.C
V.DLIGGKCELLSDDSLAVSS~PR.L
XCorr
5,144
4,245
3,423
2,718
2,712
2,546
∆Cn
0
0,175
0,335
0,471
0,473
0,505
Sp RSp Ionen
Protein
870,5 1 19/40 TRFE_HUMAN
533,5 4 16/40 TRFE_HUMAN
368,3 29 14/40 TRFE_HUMAN
259,4 197 12/40 TRFE_HUMAN
259,4 197 12/40 TRFE_HUMAN
412 19 14/40 ACPH_HUMAN
Die Peptidsequenz [IM~NGEADAMoxSLDGGFVYIAGK]2+ enthält 20 der insgesamt 698
Aminosäuren des Transferrins. Wie aus der Tabelle 6.13 erkennbar, weist das Peptid weitere
potentielle Angriffsstellen für ein Metallfragment auf: E385 und D387. Aus den zugehörigen
SEQUEST-Werten dieser Koordinationsstellen wird deutlich, dass nur M382 und E285 in
Betracht gezogen werden. Die Zuordnung der nicht platinierten yk+-Ionen für k = 17-19 in
hoher relativer Intensität spricht in diesem Fall für die M382.Koordination. Aus der Tabelle
6.12 wird ersichtlich, dass die Aminosäurefolge [IMNGEADAMSLDGGFVYIAGK]2+
(einmal mit der Oxidation des M389) mit M382 als Platinierungsstelle in beiden
Blutserumproben jeweils einmal identifiziert werden konnte. Daneben konnte für die zweite
Blutserumprobe zusätzlich die Peptidsequenz [NGEADAMoxSLDGGFVYIAGK]2+ mit einer
Platinierung der Aminosäure E385 detektiert werden. Da aus den experimentellen MS/MSSpektren die Platinverteilung bei den Ionen bj+ für j ≤ 5 nicht eindeutig hervorgeht, kann eine
Koordination des des Methionin M382 in den beiden ersten Sequenzen nicht eindeutig
bestätigt werden.Das Vorhandensein der nicht platinierten y17+-y19+-Ionen kann auch durch
Verlust des [(NH3)2Pt]2+-Fragmentes verursacht sein. Eine Betrachtung der Proteinstruktur ist
hier hilfreich. Der vergrößerte Bereich ist in der untenstehenden Abbildung zu sehen.
Serumproteine
140
E385
M382
Abb.6.14: Ausschnitt aus der Gesamtstruktur des Serotransferrins (PDB ID: 2hau
[136])
In der Illustration sind die Seitenketten des Methionins und der Asparaginsäure
hervorgehoben. M382 ist in einer Helix, E385 in einer Schleife lokalisiert. Bezogen auf die
Zugänglichkeit der Seitenketten für eine Koordination wäre die Asparaginsäure 385 eindeutig
als bevorzugte Stelle einzustufen. Bei den beiden Peptidsequenzen mit M382 als zugeordnete
Bindungsstelle liegt ein [Pt]2+- bzw. [(NH3)2Pt]2+-Fragment vor. Die Bildung eines
κ2SM382,OE385-Makrochelates wäre deshalb jeweils möglich. Bei der dritten Sequenz sowie bei
zwei Sequenzen aus den Serotransferrin:Cisplatin-Ansätzen (Tab. 6.15 und 6.17) mit E385Koordination wird dagegen das monodentate [(NH3)2PtCl]+-Fragment gefunden, was andere
Koordinationsstellen ausschließen würde. Außerdem weisen diese Sequenzen alle N383 als
N-treminalen Rest auf.
In der Tabelle 6.12 findet sich eine weitere Peptidsequenz mit einer möglichen
Cisplatinkoordination an C418/E419/D420: [417NCED~TPEAGYFAVAVVK433]2+. Um eine
dieser Aminosäuren möglicherweise als bevorzugte Bindungsstellen einteilen zu können, wird
das MS/MS-Spektrum der koordinierten Peptidsequenz aus Serotransferrin in der folgenden
Abbildung dargestellt.
Serumproteine
141
Abb.6.15: MudPIT-MS/MS-Spektrum von [NCED~TPEAGYFAVAVVK]2+ in
Serotransferrin aus der zweiten Blutserumprobe
Aus der Sequenz [417NCED~TPEAGYFAVAVVK433]2+ wird deutlich, dass bei der
Koordination des [(NH3)2Pt]2+ -Fragmentes an D420 alle b+-Ionen ab b4+ und alle y+-Ionen ab
y14+ platiniert sind. Von den 32 möglichen Ionen sind 18 im Spektrum zugeordnet (SEQUEST
konnte insgesamt 21 Ionen zuordnen). Zu erkennen sind die platinierten Ionen b5+, b6+, b8+,
b10+, b11+ und b13+ bis b16+ sowie die nicht platinierten Ionen y3+ - y9+, y12+, y13+, wie am
Beispiel des platinierten b6+-Ions in der Vergrößerung im Spektrum gezeigt. Das Fehlen der
Ionen bj+ für j < 5 und yk+ für k > 13 bietet einen wichtigen Hinweis darauf, dass D420 die
bevorzugte Bindungsstelle sein könnte.
Zu der Fragmentmasse m/z = 2041 (∆m ± 2,5) sind in der folgenden Tabelle die theoretisch
möglichen Peptidsequenzen aufgelistet, welche mit den zehn besten XCorr-Werten von
SEQUEST gefunden wurden.
Serumproteine
142
Tab.6.14: SEQUEST-Parameter von [NCED~TPEAGYFAVAVVK]2+ im Vergleich zu
anderen potentiellen Peptidsequenzen in Serotransferrin aus der zweiten Blutserumprobe
Sequenz
D.NCED~TPEAGYFAVAVVK.K
D.NC~EDTPEAGYFAVAVVK.K
D.NCE~DTPEAGYFAVAVVK.K
D.NCEDT~PEAGYFAVAVVK.K
D.NCEDTPE~AGYFAVAVVK.K
D.NCEDTPEAGY~FAVAVVK.K
T.YTPSQE~GPYM~VSVK.Y
R.SMPTAEATH~PMoxPT~AGA.T
T.YTPS~QEGPYM~VSVK.Y
R.TELSM~PIQSHVIMTPQ.S
XCorr
3,937
3,855
3,802
3,661
2,922
2,799
2,38
2,341
2,32
2,259
∆Cn
0
0,021
0,034
0,07
0,258
0,289
0,395
0,405
0,411
0,426
Sp RSp Ionen
Protein
837,8 1 21/32 TRFE_HUMAN
825,5 2 21/32 TRFE_HUMAN
825,5 2 21/32 TRFE_HUMAN
697,4 3 20/32 TRFE_HUMAN
349,2 8 16/32 TRFE_HUMAN
345
10 15/32 TRFE_HUMAN
283,3 45 12/26 FLNB_HUMAN
331,7 12 13/30 CX055_HUMAN
283,3 45 12/26 FLNB_HUMAN
220,1 212 13/30 ATF7_HUMAN
Aus Tabelle wird deutlich, dass in der oben angegebenen Peptidsequenz mehrere potentielle
Bindungsstellen
für
ein
Cisplatinfragment
enthalten
sind.
Dazu
gehören
neben
Asparaginsäure 420, Cystein 418, Glutaminsäure 419 und Threonin 421. Außerdem wird aus
den zugehörigen ∆Cn-Werten erkennbar, dass die theoretischen MS/MS-Spektren aller vier
möglichen Koordinationsstellen D420, C418, E419 und T421 vergleichbar gute
Übereinstimmungen mit dem aufgenommenen Spektrum liefern (∆Cn ≤ 0,07 und 20 bzw. 21
von 32 Ionen). Alle weiteren Peptidsequenzen in der Tabelle erfüllen die Sp-, ∆Cn- und RSpKriterien nicht (Sp < 500, ∆Cn > 0,1 und RSp > 6) und können als mögliche Bindungsstellen
eindeutig ausgeschlossen werden. Die Vergrößerung des Bereiches mit den vier potentiellen
Bindungsstellen ist in der folgenden Abbildung gezeigt.
Serumproteine
143
C418
E419
D420
T421
Abb.6.16: Ausschnitt aus der Struktur von Serotransferrin
In der Abbildung 6.16 ist der oberhalb diskutierte Bereich im Ausschnitt aus der
Proteinstruktur dargestellt. Alle vier Aminosäuren sind in einer Schleife zwischen zwei βFaltblättern lokalisiert. Aus der Positionierung der Seitenketten kann nicht eindeutig zu
Gunsten einer der vier möglichen Bindungsstellen entschieden werden.
Für Cisplatin konnte aus beiden Blutserumanalysen somit E385 (M382) eindeutig als
Bindungsstelle bestätigt werden. Die möglichen Y314 und D420 Koordinationen müssen
dagegen bei den Einzeluntersuchungen validiert werden.
6.2.2. Cisplatin:Serotransferrin Proben
Aus dem äquimolaren Ansatz von Transferrin mit Cisplatin konnten die in der Tabelle 6.15
aufgelisteten koordinierten Peptidsequenzen detektiert werden.
Serumproteine
144
Tab.6.15: Koordinierte Peptidsequenzen aus dem 1:1 Ansatz Serotransferrin:Cisplatin nach 3h
Inkubation bei 37°C
Peptid
K.M§YLGYEYVTAIR.N
Bindungsstelle z XCorr ∆Cn Sp RSp Ionen
M313/Y314 2 3,80 0,65 1641,6 1 20/22
M.NGE$ADAMoxSLDGGFVYIAGK.C
K.IM$NGEADAMSLDGGFVYIAGK.C
E385
M382/E385
2
2
4,25
2,96
0,73 828,3
0,74 500,2
1
1
18/36
17/40
M.NGE~ADAMSLDGGFVYIAGK.C
D.LIWE~LLNQAQEHFGK.D
E385
E265
2
2
6,25
4,04
0,79 2299,8
0,43 1637,9
1
1
27/36
19/28
M.NGE#ADAMSLDGGFVYIAGK.C
E385
2 4,65 0,73 1065,0
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
19/36
Es sind insgesamt sechs Proteinabschnitte von einem der Cisplatinfragmente koordiniert.
Dabei wurde jeweils einmal E265, M313/Y314 und M382/E385 und dreimal E385 platiniert.
Von den in den Blutserumproben gefundenen Bindungsstellen konnten M313/Y314 und
M382/E385 erneut identifiziert werden. Die Stelle C418/D420 wurde dagegen in dieser
Analyse bzw. in den weiteren Untersuchungen nicht wieder beobachtet und kann deswegen
nicht als spezifische Bindungsstelle gelten.
Da Y314 als Bindungsstelle neben der oxidierten Methionin 313 Stelle in der ersten
Blutserumprobe (siehe Tabelle 6.12) identifiziert wurde, ist es interessant die erste
Peptidsequenz des äquimolaren Ansatzes nach 3h Inkubation zu überprüfen.
Die Abbildung 6.17 zeigt ein MS/MS-Spektrum dieser Sequenz, welche mit dem [Pt]2+Fragment
modifiziert
wurde.
Es
handelt
sich
dabei
um
[313M§YLGYEYVTAIR324]2+ des Transferrins mit der Masse m/z = 1673.
das
Peptid
Serumproteine
145
Abb.6.17: MS/MS-Spektrum von [M§YLGYEYVTAIR]2+ aus dem 1:1 Ansatz
Serotransferrin:Cisplatin nach 3h Inkubation bei 37°C
In dem Spektrum sind 17 von 22 möglichen Ionen zugeordnet, davon sind sieben aus der b+und zehn aus der y+-Serie. Von den 22 möglichen Ionen sind 20 bei der SEQUEST-Analyse
berücksichtigt worden. Alle 11 b+-Ionen und keines der y+-Ionen sind für eine M313Koordination platiniert.
Die folgende Tabelle enthält die für eine Masse von m/z = 1673 (∆m ± 2,5) möglichen
weiteren Peptidsequenzen aus dem Serumprotein Transferrin. Diese können einerseits
Koordinationen des Cisplatinfragmentes an einer anderen Aminosäure der oben angegebenen
Sequenz aufweisen (d. h. Y314, Y317, E 318 oder Y319) oder weitere koordinierte Peptide
des Proteins mit der entsprechenden Masse sein.
Serumproteine
146
Tab.6.16: SEQUEST-Parameter von [M§YLGYEYVTAIR]2+ im Vergleich zu anderen
potentiellen Peptidsequenzen aus dem 1:1 Ansatz Serotransferrin:Cisplatin nach 3h
Inkubation bei 37°C
Sequenz
K.M§YLGYEYVTAIR.N
K.MY§LGYEYVTAIR.N
K.MYLGY§EYVTAIR.N
K.MYLGYE§YVTAIR.N
K.MYLGYEY§VTAIR.N
H.S§TIFENLANKADR.D
H.ST§IFENLANKADR.D
H.STIFE§NLANKADR.D
K.MYLGYEYVT§AIR.N
K.C§D§EWS§VNSVG.K
XCorr
3,801
3,558
2,376
1,941
1,579
1,325
1,293
1,265
0,888
0,698
∆Cn
0
0,064
0,375
0,489
0,585
0,651
0,66
0,667
0,766
0,816
Sp
1641,6
1623,5
519,1
371,1
308,3
185,1
161
161
90,7
57,8
RSp
1
2
3
4
5
6
7
7
8
12
Ionen
20/22
20/22
15/22
13/22
13/22
9/24
8/24
8/24
10/22
5/18
Aus den SEQUEST-Werten und dem MS/MS-Spektrum (Abb 6.17) der Sequenz M313-R324
wird deutlich, dass sowohl die Zuordnung des Methionins 313 als auch die des Tyrosins 314
als Bindungsstelle für [Pt]2+ eine akzeptable Übereinstimmung mit dem theoretischen
Spektrum liefern. Da im obigen MS/MS-Spektrum das b1+-Ion nicht beobachtet wird und das
zugeordnete y11+ von sehr geringer Intensität ist, kann hier keine Entscheidung zu Gunsten
von M313 bzw. Y314 resultieren.
Die folgende Abbildung zeigt den Ausschnitt aus der Proteinstruktur des Serotransferrins,
wobei die Seitenketten des M313 und Y314 von besonderem Interesse sind.
M313
Y314
Abb.6.18: Ausschnitt aus der Proteinstruktur des Serotransferrins
Serumproteine
147
Beide Aminosäuren sind in einer Helix lokalisiert, jedoch ist aus der Position der
Methioninseitenkette erkennbar, dass diese durch mögliche Wechselwirkungen mit den
Aminosäuren der benachbarten Helices für eine Koordination weniger bevorzugt ist. Diese
Beobachtung steht mit den Befunden von SADLER in Einklang, der mittels NMRSpektroskopie keine Anhaltspunkte für eine M313-Koordination im gesamten Protein
feststellen konnte [58]. Es ist interessant, dass die von SADLER bzw. DYSON identifizierten
Stellen M256, M499 und T457 bei den 1:1 Ansätzen nach 3h Inkubation nicht beobachtet
wurden. Die Produkte der Reaktion zwischen Serotransferrin und Cisplatin nach einer
Inkubation von 3h bei 37 °C im Verhältnis 1:5 wurden ebenfalls massenspektrometrisch
untersucht. Die Liste der koordinierten Peptide daraus findet sich in Tabelle 6.17 Neben den
schon beim 1:1 Ansatz identifizierten Bindungsstellen M313/Y314 und M382/E385 wird bei
dieser höheren Konzentration erstmals auch die schon von DYSON ermittelte T457Koordination bestätigt. Offensichtlich sind hierfür höhere Konzentrationen an Metallkomplex
notwendig, wobei von DYSON sogar ein zehnfacher Überschuss an Cisplatin verwendet wurde
[137].
Tab.6.17: Koordinierte Peptidsequenzen aus dem 1:5 Ansatz Serotransferrin:Cisplatin nach 3h
Inkubation bei 37°C
Peptid
Bindungsstelle z Xcorr ∆Cn
Sp
RSp Ionen
K.IM§NGEADAMSLDGGFVYIAGK.C M382/E385 2 4,21 0,72 1412,6 1 26/40
K.MY§LGYEYVTAIR.N
Y314
2 3,59 0,65 732,7
1 14/22
A.KM§YLGYEYVTAIR.N
M313/Y314 2 2,82 0,58 1225,6 1 17/24
R.T§AGWNIPMoxGLLYNK.I
T457
2 2,72 0,62 628,2
1 15/26
M.NGE$ADAMoxSLDGGFVYIAGK.C
R.T$AGWNIPMoxGLLYNK.I
K.M$YLGYEYVTAIR.N
E385
T457
M313/Y314
2
2
2
4,34
3,73
2,65
0,75 1047,9
0,43 711,7
0,83 760,7
1
1
1
20/36
15/26
14/22
M.NGE~ADAMSLDGGFVYIAGK.C
E385
2
6,07
0,80 1829,4
1
24/36
M.NGE#ADAMSLDGGFVYIAGK.C
E385
2 5,49 0,83 1783,2
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
23/36
Wie der Tabelle zu entnehmen ist, wurden im Serotransferrin nach der 1:5 Umsetzung mit
Cisplatin insgesamt neun koordinierte Peptidsequenzen detektiert. Es konnte dreimal die
Koordination des Bereiches M313/Y314, viermal M382/E385 und zweimal T457 identifiziert
werden.
Serumproteine
148
Das nachstehende Massenspektrum zeigt die ESI-MS2-Fragmentierungen der koordinierten
Sequenz [457T$AGWNIPMoxGLLYNK470]2+, welche an der Aminosäure Threonin 457
[(NH3)Pt]2+ bindet.
Abb.6.19: MS/MS-Spektrum von [T$AGWNIPMoxGLLYNK]2+ aus dem 1:5 Ansatz
Serotransferrin:Cisplatin nach 3h Inkubation bei 37°C
Es konnten 15 Ionen von 26 möglichen den Peaks im oberen Spektrum zugeordnet werden.
Die Ionen b1+ bis b13+ aber keine der beobachteten y+-Ionen sind platiniert. Es wurden im aus
der b+-Serie die Ionen b4+-b6+, b8+, b11+ und b12+ sowie aus der y+-Serie y2+-y4+, y6+, y8+, y9+y11+ und y102+ beobachtet.
Die folgende Tabelle listet Peptidsequenzen des Serumproteins auf, welche für den Vergleich
des beobachteten MS/MS-Spektrums mit dem theoretischen Spektrum die besten SEQUESTWerte lieferten.
Serumproteine
149
Tab.6.18: SEQUEST-Parameter von [T$AGWNIPMoxGLLYNK]2+ im Vergleich zu anderen
potentiellen Peptidsequenzen aus dem 1:5 Ansatz Serotransferrin:Cisplatin nach 3h
Inkubation bei 37°C
Sequenz
R.T$AGWNIPMoxGLLYNK.I
V.GRTAGWNIPMoxGLLYNK.I
R.TAGWNIPMoxGLLY$NK.I
R.TAGWNIPMoxGLLYNK$.I
R.TAGWNIPMGLLYNKIN.H
R.DD$T$VCLAKLHDR.N
R.M$D$AKMYLGYEY.V
R.MD$AKM$YLGYEY.V
R.D$DT$VCLAKLHDR.N
R.MD$AKMY$LGYEY.V
XCorr
3,734
2,138
2,113
1,784
1,767
1,173
1,159
1,158
1,081
1,081
∆Cn
0
0,427
0,434
0,522
0,527
0,686
0,69
0,69
0,711
0,711
Sp
711,7
334,2
161,8
136,1
144,1
162
74
31,2
162
31,2
RSp Ionen
1
15/26
2
12/30
4
10/26
6
9/26
5
10/30
3
7/22
10
7/20
38
5/20
3
7/22
38
5/20
In der Tabelle 6.18 finden sich zehn Peptidsequenzen, welche der Masse m/z = 1805 (mit ∆m
± 2,5) entsprechen. Aus den Sp- und ∆Cn-Werten dieser Proteinstücke wird deutlich, dass
keine andere Sequenz in Frage kommt. In der unteren Darstellung ist ein Ausschnitt aus der
Proteinstruktur des Transferrins präsentiert, welche die koordinierte Aminosäure T457 zeigt.
T457
Abb.6.20: Ausschnitt aus der Gesamtstruktur des Serotransferrins
Die Zugänglichkeit der Seitenkette des Threonins aus Tabelle 6.17 lässt sich in der
Darstellung gut erkennen. Die modifizierte Aminosäure ist in einer Schleife lokalisiert. Sie
Serumproteine
150
dient im Protein als Bicarbonat-Bindungsstelle, was bei einer Koordination dieser
Threoninstelle eine Blockierung mit sich bringt.
Von den in der Literatur diskutierten Bindungsstellen im Serumalbumin konnte somit bisher
die von SADLER beobachtete M256 und M499 Koordination [58] nicht identifiziert werden.
Mit einem fünffachen Überschuss an Cisplatin wurden deswegen die Reaktionsprodukte auch
nach 168h Inkubation bei 37°C mit Serotransferrin untersucht. Eine Tabelle der mit einem
Cisplatinfragment modifizierten Peptidsequenzen aus dem Serumprotein findet sich unter
6.19.
Tab.6.19: Koordinierte Peptidsequenzen aus dem 1:5 Ansatz Serotransferrin:Cisplatin nach
168h Inkubation bei 37°C
Peptid
Bindungsstelle z XCorr ∆Cn Sp RSp Ionen
R.SM§GGKEDLIWELLNQAQEHFGK.D
S255/M256
3 5,19 0,76 1874,2 1 32/84
K.IM§NGEADAMSLDGGFVYIAGK.C M382/E385/D387 2 4,39 0,76 1373,1 1 25/40
K.M§YLGYEYVTAIR.N
M313/Y314
2 4,13 0,72 1944,8 1 21/22
R.T§AGWNIPMGLLYNK.I
T457
2 3,25 0,62 970,1
1 17/26
M.Y§LGYEYVTAIR.N
Y314
2 2,67 0,68 1926,1 1 17/20
R.SM$GGKEDLIWELLNQAQEHFGK.D
R.T$AGWNIPMoxGLLYNK.I
K.IM$NGEADAMSLDGGFVYIAGK.C
M.Y$LGYEYVTAIR.N
K.MoxY$LGYEYVTAIR.N
S255/M256
T457
M382/E385
Y314
Y314
3
2
2
2
2
4,84
3,89
3,59
3,11
2,93
0,48 1881,3
0,57 996,7
0,82 882,8
0,70 888,8
0,75 750,8
1
1
1
1
1
33/84
19/26
18/40
13/20
13/22
M.NGE~ADAMSLDGGFVYIAGK.C
E385
2 6,33 0,73 2187,1
D.LIWE~LLNQAQEHFGK.D
E265
2 3,27 0,42 1079,8
2+
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt] , # = [(NH3)2PtCl]+
1
1
25/36
17/28
Die oben dargestellte Tabelle listet insgesamt 12 modifizierte Peptidsequenzen des
Serotransferrins auf. Es konnten die in den Blutserum- und/oder den Einzelanalysen
identifizierten Bindungsstellen M382/E385, M313/Y314 und T457 bestätigt werden. Bei
dieser Umsetzung nach 168h Inkubation konnte auch die von SADLER beobachtete M256Koordination zum ersten Mal ermittelt werden. Ebenfalls zum ersten Mal wurde die
Koordination der Glutaminsäure 265 beobachtet. Neben der Methioninseitenkette weist die
Peptidsequenz mehrere potentielle Koordinationsstellen (S255 oder E260) auf.
Das MS/MS-Spektrum der Peptidsequenz [255SM§GGKEDLIWELLNQAQEHFGK276]3+ des
Transferrins ist in der folgenden Abbildung dargestellt.
Serumproteine
151
Abb.6.21: MS/MS-Spektrum von [SM§GGKEDLIWELLNQAQEHFGK]3+ aus dem 1:5
Ansatz Serotransferrin:Cisplatin, nach 168h Inkubation bei 37°C
Das Spektrum beinhaltet 25 zugeordnete Ionen (einfach und zweifach geladene), wobei aus
der Sequenz 84 Fragmentionen möglich sind und von SEQUEST 36 zugeordnet wurden. Von
der Bindungsstelle des Cisplatinrestes resultierend weisen alle bj+-Ionen für j ≥ 2 und nur y21+
die Platinierung auf. Mit der Molekülmasse m/z = 2741 (∆m ± 2,5) wurden in Tab. 6.20
dargestellte weitere mögliche Peptidsequenzen durch SEQUEST ermittelt. Es handelt sich bei
den ersten acht Sequenzen um Koordinationen des Cisplatinfragmentes an unterschiedlichen
Aminosäuren der gleichen Sequenz, die als potentielle Bindungsstellen möglich sind. Um
genau festzustellen, welche der gefundenen Koordinationen zutreffend ist, wird zunächst die
Liste aus der untenstehenden Tabelle 6.20 betrachtet.
Serumproteine
152
Tab.6.20: SEQUEST-Parameter von [SM§GGKEDLIWELLNQAQEHFGK]3+ im Vergleich
zu anderen potentiellen Peptidsequenzen aus dem 1:5 Ansatz Serotransferrin:Cisplatin, nach
168h Inkubation bei 37°C
Sequenz
R.SM§GGKEDLIWELLNQAQEHFGK.D
R.S§MGGKEDLIWELLNQAQEHFGK.D
R.SMGGK§EDLIWELLNQAQEHFGK.D
R.SMGGKED§LIWELLNQAQEHFGK.D
R.SMGGKE§DLIWELLNQAQEHFGK.D
R.SMGGKEDLIWE§LLNQAQEHFGK.D
R.SMGGKEDLIWELLNQAQE§HFGK.D
R.SMGGKEDLIWELLNQAQEH§FGK.D
Q.QH§LFGS§NVTD§CSGNFCLFR.S
K.DCH§LAQVPSHT§VVARSMGGKED.L
XCorr
5,189
5,18
5,177
5,15
5,127
4,425
1,566
1,515
1,252
1,179
∆Cn
0
0,002
0,002
0,008
0,012
0,147
0,698
0,708
0,759
0,773
Sp
RSp Ionen
1874,2
1
32/84
1874,2
1
32/84
1826,7
2
32/84
1776,2
3
32/84
1689,4
4
31/84
1179,1
5
28/84
257,7
6
17/84
216,7
7
16/84
194,9
8
14/72
81,5
32 10/84
In der Auflistung sind insgesamt zehn Proteinfragmente mit den besten theoretischen
Übereinstimmungen aufgeführt. Aus den SEQUEST-Parametern in der Tabelle 6.20 wird
deutlich, dass die Bindungen des Platinrestes an S255, K259, E260 und D261 auch in Frage
kommen (∆Ch < 0,1). Entscheidend für eine Zuordnung zu Gunsten einer der möglichen
Koordinationsstellen wären beispielsweise die Ionen b1+ für S255 oder y18+-y21+ für K259.
Diese Ionen werden allerdings im Spektrum in Abbildung 6.21 nicht beobachtet. Die folgende
Abbildung zeigt die Lokalisierung der fünf möglichen Bindungsstellen in der Proteinstruktur.
D261
S255
M256
E260
K259
Abb.6.22 Ausschnitt aus der Proteinstruktur des Serotransferrins
Serumproteine
153
In der Abbildung 6.22 ist der Bereich der Gesamtstruktur des Proteins vergrößert dargestellt,
um
die
räumliche
Verteilung
der
möglicherweise
koordinierten
Seitenketten
zu
veranschaulichen. Aus der Darstellung wird deutlich, dass keine der Aminosäuren als
Bindungsstelle für ein Cisplatinfragment eindeutig ausgeschlossen werden kann. Jedoch wird
besonders für M256 die gute Zugänglichkeit auch in der unteren Abbildung bestätigt.
M256
Abb.6.23 Ausschnitt aus der Proteinstruktur des Serotransferrins
In der Illustration wird die Lokalisierung der genannten Aminosäure an der Oberfläche der
Proteinstruktur (in einer Schleife) deutlich. Für das Vorliegen von M256 als
Cisplatinbindungsstelle sprechen die NMR-spektroskopischen Beobachtungen von SADLER,
die eine Koordination dieser Aminosäure bestätigen [58]. Auch bei dem 1:5 Ansatz
Serotransferrin:Cisplatin nach 168h Inkubation konnte das von SADLER als Bindungsstelle
ermittelte Methionin 499 nicht bestätigt werden.
Die aus den unabhängigen Blutserumanalysen und/oder den Einzeluntersuchungen ermittelten
Aminosäuren M256, E265, Y314, E385 und T457 können als spezifische Bindungsstellen für
Cisplatin eingestuft werden. Die von SADLER in seinen NMR-spektroskopischen
Untersuchungen beobachtete Koordination von M499 konnte in den vorliegenden Analysen
nicht bestätigt werden.
Bei den oben genannten Bindungsstellen des Cisplatins wird die Bevorzugung der
Koordination der kinetisch begünstigten O-Donoratome bestätigt, wie sie bereits bei den
Serumalbumin-Studien beobachtet werden konnten. Die Lokalisierung und Verteilung der
Serumproteine
154
fünf Bindungsstellen im Protein ist in der folgenden Abbildung dargestellt. D420 wurde nur
in der zweiten Blutserumprobe gefunden und konnte nicht durch die Serotransferrin:Cisplatin
Ansätze bestätigt werden.
E385
M256
T457
Y314
E265
Abb.6.24: Struktur des Serotransferrins mit den bevorzugten Cisplatinbindungsstellen
Aus der Abbildung 6.24 wird deutlich, dass die Bindungsstellen auf der Oberfläche des
Proteins bzw. in den Bindungstaschen lokalisiert sind.
6.3. Cisplatinaddukte des Apolipoproteins A1
6.3.1. Blutserumproben
In den beiden Blutserumproben konnte neben den beiden Proteinen Serumalbumin und
Serotransferrin eine Reihe weiterer abundanter Proteine als Cisplatin-Targets detektiert
werden. Eines davon ist das abundante Serumprotein Apolipoprotein A1 (ApoA1), das ein
Molekülgewicht von etwa 30 kDa aufweist. Es besteht aus 267 Aminosäuren, wird
hauptsächlich in der Leber und im Darm synthetisiert und transportiert wasserunlösliche
Lipide im Blut. Eine Auflistung der mit einem Metallkomplexfragment modifizierten
Peptidsequenzen findet sich in der folgenden Tabelle.
Serumproteine
155
Tab.6.21: Koordinierte Peptidsequenzen aus Apolipoprotein A1 aus den MudPIT-Analysen
der beiden Blutserumproben
Peptid
erste Bluserumprobe
K.WQE§EMELYR.Q
Bindungsstelle z XCorr ∆Cn
Sp
RSp Ionen
E110
2
3,77
0,11
774,5
5
14/16
K.DLATVYVDVLK~.D
K.VSFLSALEEYT~.K
K.LREQLGPVTQEFWD~N.L
K23
T237
D73
2
2
2
3,02
4,33
3,34
0,28 710,5
0,31 1021,8
0,22 1104,1
1
1
1
15/20
18/20
16/28
K.VS#FLSALEEYT.K
S228
2
3,08
0,23
1
15/20
1
16/26
998,7
zweite Bluserumprobe
K.LLD$NWDSVTSTFSK.L
D48
2 3,50 0,15 885,2
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
In der Tabelle sind sechs Sequenzen aufgezählt, welche aus beiden Untersuchungen erhalten
wurden. Dabei sind alle vier möglichen Cisplatinfragmente als Koordinationspartner zu
beachten. Der Aminosäure Serin 228 wurde das [(NH3)2PtCl]+-Fragment zugeordnet. Aus
beiden Blutserumproben wurden die möglichen Koordinationen von K23, D48, D73, E110,
S228 und T237 mit einem der Cisplatinfragmente beobachtet.
Ein Beispiel einer koordinierten Peptidsequenz des Apolipoprotein A1 aus der ersten
Blutserumanalyse
60
ist
die
mit
[(NH3)2Pt]2+
markierte
Aminosäurefolge
74 2+
[ LREQLGPVTQEFWD~N ] . Das zugehörige MS/MS-Spektrum ist nachfolgend
dargestellt. Die Peptidsequenz weist neben Asparaginsäure 73 weitere potentielle
Koordinationsstellen wie Glutaminsäure 70 für ein Cisplatinfragment auf. Diese
Möglichkeiten werden durch Überprüfung der Zuordnungen im MS/MS-Spektrum und der
SEQUEST-Werte für die weiteren Aminosäuren betrachtet.
Serumproteine
156
Abb.6.25: MudPIT-MS/MS-Spektrum von [LREQLGPVTQEFWD~N]2+ in Apolipoprotein
A1 aus der ersten Blutserumprobe
Das obere Spektrum enthält 14 b+- und y+-Ionen, wobei bis zu 28 aus der Fragmentierung
entstehen können und SEQUEST 16 zuordnen konnte. Aus der b+-Serie konnten die Ionen b4+
bis b12+ und aus der y+-Reihe y4+, y7+, y10+, y11+ sowie y13+ zugeordnet werden. Aus der
Peptidsequenz und der koordinierten Position D73 resultiert, dass die Ionen b14+ und y2+-y14+
platiniert sind, wobei im Spektrum unter Abb. 6.25 fünf der genannten y+-Ionen beobachtet
werden können.
Die nachfolgende Tabelle listet fünf weitere Proteinbruchstücke auf, welche der Masse m/z =
2060 (∆m ± 2,5) entsprechen und deren theoretische MS/MS-Spektren mit dem
aufgenommenen Spektrum teilweise übereinstimmend sind.
Serumproteine
157
Tab.6.22: SEQUEST-Paramter von [LREQLGPVTQEFWD~N]2+ im Vergleich zu anderen
potentiellen Peptidsequenzen in Apolipoprotein A1 aus der ersten Blutserumprobe
Sequenz
K.LREQLGPVTQEFWD~N.L
K.VS~LLGPVTT~PEH~Q.L
K.VS~LLGPVTTPE~H~Q.L
K.VS~LLGPVTT~PE~HQ.L
R.S~T~RPTQQFYQPPR.A
K.VS~LLGPVT~TPEH~Q.L
XCorr
3,344
2,614
2,559
2,515
2,421
2,402
∆Cn
Sp
RSp Ionen
Protein
0
1104,1
1
16/28 APOA1_HUMAN
0,218 615,5
13 12/24 RGAP1_HUMAN
0,235 468,6
84 11/24 RGAP1_HUMAN
0,248 615,5
13 12/24 RGAP1_HUMAN
0,276 591,3
17 12/24 TDRD3_HUMAN
0,282 493,3
63 11/24 RGAP1_HUMAN
Außer der oben genannten Peptidsequenz des ApoliproteinA1 können auf Grund der
SEQUEST-Parameter alle aufgeführten Proteinstücke eindeutig als koordinierte Sequenzen
ausgeschlossen werden. Diese Aminosäurenfolgen wurden zwei- oder dreifach von einem
Cisplatinfragment koordiniert zugeordnet. Das MS/MS-Spektrum (Abb. 6.25) weist jedoch
bei keinem der dargestellten Ionen ein Isotopenpattern auf, welches auf Mehrfachkoordination
hindeuten würde.
Eine Darstellung der Bindungsstelle D73 für ein Cisplatinfragment in dem Serumprotein
Apolipoprotein A1 ist in der nachstehenden Illustration zu sehen.
D73
Abb.6.26: Ausschnitt aus der Proteinstruktur des Apolipoproteins A1 (PBD ID: 2a01A [138])
Der Ausschnitt aus der Struktur in Abb. 6.26 zeigt die gute Zugänglichkeit von D73 für eine
Koordination. Die Asparaginsäure ist auf der Oberfläche des Proteins in einer Helix
lokalisiert.
Serumproteine
158
6.3.2. Apolipoprotein A1:Cisplatin Proben
Für eine Validierung der identifizierten möglichen Bindungsstellen K23, D48, D73, E110,
S228 und T237 wurde das Serumprotein mit Cisplatin inkubiert und auf entstandene Addukte
untersucht. Es wurden zwei Analysen mit dem Apolipoprotein A1 und Cisplatin durchgeführt.
Bei diesen Reaktionen wurden die Protein-Metallkomplex-Verhältnisse von 1:1 bei einer
Inkubation von 3h und 1:5 bei 168h und jeweils 37 °C gewählt.
In der Tabelle 6.23 ist die Liste der koordinierten Peptide aus dem äquimolaren
Reaktionsansatz nach 3h Inkubation und aus dem 1:5 Ansatz nach 168h Inkubation bei 37°C
dargestellt.
Tab.6.23: Koordinierte Peptidsequenzen aus den 1:1 und 1:5 Ansätzen Apolipoprotein
A1:Cisplatin nach 3h und nach 168h Inkubation bei 37°C
Peptid
Bindungsstelle z XCorr ∆Cn
1:1 Ansatz nach 3h/37°C
R.VKDLATVYVDVLKD§.S
D24
2 2,60 0,64
K.DLATVYVDVLK§D.S
K23
2 2,53 0,67
Sp
RSp Ionen
642,2
605,6
1
1
15/26
13/22
K.VS$FLSALEEYTK.K
S228
2
3,58
0,89 1321,1
1
19/22
V.K#DLATVYVDVLK.D
R.VKDLATVYVD#V.L
R.VK#DLATVYVDVLK.D
V.S#FLSALEEYTK.K
K12
D20
K12
S228
2
2
2
2
3,69
2,85
2,68
2,64
0,81 1783,2
0,74 746,6
0,65 655,9
0,76 1123,9
1
1
1
1
19/22
14/20
14/24
15/20
1:5 Ansatz nach 168h/37°C
K.LLD$NWDSVTSTFSK.L
D48
2
3,68
0,75 1274,2
1
18/26
K.LLDNWDSVTSTFSK~L.R
K59
2 3,09 0,81 608,3
1
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
16/28
Die Tabelle enthält neun mit einem der vier Cisplatinfragmente modifizierte Peptide des
Apolipoproteins A1. Von den bei dem 1:1 bzw. 1:5 Ansatz ermittelten Bindungsstellen
wurden K23 und S228 in der ersten und D48 in der zweiten Blutserumprobe identifiziert.
Für die sowohl aus der ersten Blutserumprobe als auch aus dem äquimolaren Protein:Cisplatin
Ansatz identifizierte mögliche Bindungsstelle aus der Sequenz [227VS$FLSALEEYTK338]2+,
in der das Serin 228 mit [(NH3)Pt]2+ koordiniert wird, ist nachfolgend das experimentell
bestimmte MS/MS-Spektrum abgebildet.
Serumproteine
159
Abb.6.27: MS/MS-Spektrum von [VS$FLSALEEYTK]2+ aus dem 1:1 Ansatz
ApoA1:Cisplatin nach 3h Inkubation bei 37°C
Von 22 möglichen Ionen der genannten Sequenz wurden von SEQUEST 19 b+- und y+- Ionen
(im Spektrum 14 Ionen) zugeordnet. Die Ionen b5+ bis b11+, aber keines der y+-Ionen (y11+ im
Spektrum nicht dargestellt) sind platiniert. In benachbarter Position zu S228 finden sich ein
weiteres Serin (S231), Glutaminsäuren (E234 und E235) sowie Tyrosin (Y236), welche als
potentielle Bindungsstelle für Cisplatin in Frage kommen könnten. Wie aus den SEQUESTParametern der Tabelle 6.24 und der fehlenden Platinverteilung der Ionen y8+- y10+ ersichtlich,
können diese Koordinationspositionen alle ausgeschlossen werden.
Serumproteine
160
Tab.6.24: SEQUEST-Parameter von [VS$FLSALEEYTK]2+ im Vergleich zu anderen
potentiellen Peptidsequenzen aus dem 1:1 Ansatz ApoA1:Cisplatin nach 3h Inkubation bei
37°C
Sequenz
K.VS$FLSALEEYTK.K
K.VSFLS$ALEEYTK.K
K.VSFLSALE$EYTK.K
K.VSFLSALEE$YTK.K
K.VSFLSALEEY$TK.K
-.MKAAVLTLAVLFL.T
Q.E$E$MELY$R.Q
Q.EE$M$ELY$R.Q
-.M$KAAVLTLAVLFL.T
Q.E$EM$ELY$R.Q
XCorr
3,578
2,371
1,458
1,018
0,551
0,41
0,391
0,364
0,36
0,351
∆Cn
0
0,337
0,593
0,715
0,846
0,885
0,891
0,898
0,899
0,902
Sp
1321,1
410,3
133,3
88,1
32,9
18,6
63,8
38,1
18,6
38,1
RSp
1
2
3
4
8
17
5
7
17
7
Ionen
19/22
13/22
8/22
7/22
5/22
4/24
4/12
3/12
4/24
3/12
Weitere mögliche Peptidsequenzen mit der Masse m/ = 1597 (innerhalb der Genauigkeit von
∆m ± 2,5) sind in der Tabelle aufgelistet. Da jedoch bei allen diesen Sequenzen die
SEQUEST-Werte unzureichend sind (alle Sp-Werte < 500), wird deutlich, dass die
Zuordnung der Bindungsstelle in [VS$FLSALEEYTK]2+ für die Aminosäure S228 eindeutig
ist.
Werden nun die Bindungsstellen aus allen Analysen verglichen (sowohl Blutserum als auch
einzelne Protein:Cisplatin-Ansätze), lässt sich erkennen, dass drei Bindungsstellen bestätigt
werden konnten: K23, D48 und S228.
In der folgenden Abbildung ist die Gesamtstruktur des Serumproteins mit den drei
Seitenketten zu sehen, die als bevorzugte Bindungsstellen für Cisplatin identifiziert werden
konnten.
D48
K23
S228
Abb.6.28: Struktur des Proteins Apolipoprotein A1
Serumproteine
161
Aus der Abbildung wird deutlich, dass die gekennzeichneten Seitenketten leicht zugänglich
für eine Cisplatinkoordination lokalisiert sind. Aus der Struktur wird jedoch auch ersichtlich,
dass der parallele Aufbau der α-Helices des Proteins mehrere nicht spezifische Angriffsstellen
für Metallkomplexe bietet. So ist es auch zu erklären, dass weitere Bindungsstellen (D20,
K59, D73, E110 und T237) bei den einzelnen Analysen gefunden wurden. Es fällt auf, dass
nur N(Amino)- bzw. O-Donoratome ermittelt wurden. Die geringe Zahl der identifizierten
Bindungsstellen (zwei Peptidsequenzen, s. Tab. 6.23) bei dem 1:5 Ansatz spricht auch dafür,
dass Apolipoprotein A1 keine spezifischen Koordinationspositionen aufweist. Nach 168h
Inkubation ist deswegen eine gleichmäßige Verteilung der Cisplatinfragmente über viele
Positionen zu vermuten.
6.4. Cisplatinaddukte des Alpha-1-Antitrypsins
Blutserumproben
In den MudPIT-Analysen der beiden Blutserumproben konnten platinierte Peptidsequenzen
des abundanten Serumproteins Alpha-1-Antitrypsin detektiert werden. Davon sind sechs
Proteinabschnitte mit einem Metallkomplexrest modifiziert. Die Liste der koordinierten
Proteinstücke ist in Tabelle 6.25 zu sehen.
Tab.6.25: Koordinierte Peptidsequenzen aus Alpha-1-Antitrypsin aus den MudPIT-Analysen
der ersten und zweiten Blutserumprobe
Peptid
erste Blutserumprobe
K.FNK§PFVFLMIEQNTK.S
Bindungsstelle z XCorr ∆Cn
Sp
RSp Ionen
K368
3
4,67
0,16 1018,7
3
24/56
L.NQPD$SQLQLTTGNGLFLSEGLK.L
D107
2
3,12
0,28 651,1
2
15/42
zweite Blutserumprobe
K.FNK§PFVFLMoxIEQNTK.S
K368
3
4,69
0,24 1157,1
1
25/56
Q.HLE$NELTHDIITK.F
L.NQPD$SQLQLTTGNGLFLSEGLK.L
E264
D107
2
2
3,66
3,44
0,21 1365,7
0,34 781,4
1
1
18/24
16/42
N.K#PFVFLMoxIEQNTK.S
K368
2 4,84 0,31 1617,9
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
21/24
Serumproteine
162
Das Alpha-1-Antitrypsin ist ein etwa 47 kDa großes Protein, welches aus 418 Aminosäuren
besteht. Das Antitrypsin hemmt Serinproteasen und schützt das Körpergewebe vor an
Entzündungsprozessen beteiligten Enzymen [139]. Von den insgesamt sechs Peptidsequenzen
aus Tabelle 6.25 sind die Koordinationsstellen D107 und K368 aus beiden und E264 aus der
zweiten Blutserumprobe identifiziert worden. Das MS/MS-Spektrum des fragmentierten
Molekülpeaks der an der Aminosäure Lysin 368 mit [Pt]2+ koordinierten Sequenz
[366FNK§PFVFLMIEQNTK380]3+ ist exemplarisch für die koordinierten Peptidsequenzen aus
diesem Protein in der Abbildung 6.29 dargestellt.
Abb.6.29: MudPIT-MS/MS-Spektrum von [FNK§PFVFLMIEQNTK]3+ in A1AT aus der
ersten Blutserumprobe
Das Spektrum enthält 14 der möglichen 56 (einfach und zweifach geladene, so dass
SEQUEST 24 zuordnet) b+- und y+-Ionen der Sequenz [FNK§PFVFLMIEQNTK]3+.
Entsprechend der Koordination an K368 sind die Ionen b3+ bis b14+ und y13+ und y14+
platiniert. Bei vielen der im Spektrum zugeordneten b+-Ionen lässt sich diese Verteilung
erkennen.
Serumproteine
163
Die nachfolgende Tabelle listet weitere Peptidsequenzen auf, deren theoretische MS/MSSpektren mit dem aufgenommenen Spektrum Übereinstimmungen ergeben. Dabei sind neun
Proteinstücke mit absteigenden XCorr-Werten dargestellt.
Tab.6.26: SEQUEST-Parameter von [FNK§PFVFLMIEQNTK]3+ im Vergleich zu anderen
potentiellen Peptidsequenzen in A1AT aus der ersten Blutserumprobe
Sequenz
K.FNK§PFVFLMIEQNTK.S
K.NLY§KDVMoxLENFRNLA.S
T.VC§H§HIENVLKEDAR.G
K.NLYK§DVMoxLENFRNLA.S
K.NLYKD§VMoxLENFRNLA.S
T.VC§HH§IENVLKEDAR.G
T.VCH§H§IENVLKEDAR.G
N.K§AVQLENELENFT§K.Q
L.TVRC§E§ALTGELETLK.E
-.APRT§LLQVLQDPALK§.-
XCorr
4,666
3,919
3,597
3,562
3,344
3,341
3,307
3,042
3,041
2,635
∆Cn
0
0,16
0,229
0,236
0,283
0,284
0,291
0,348
0,348
0,435
Sp
1018,7
938,6
1056
772,2
561,6
824,8
848
604,3
527,1
1148,8
RSp
3
5
2
23
234
17
10
140
373
1
Ionen
24/56
23/56
24/52
22/56
19/56
22/52
22/52
18/52
19/56
25/56
Protein
A1AT_HUMAN
ZN177_HUMAN
DTX1_HUMAN
ZN177_HUMAN
ZN177_HUMAN
DTX1_HUMAN
DTX1_HUMAN
MFN1_HUMAN
CCD89_HUMAN
ICB1_HUMAN
Die SEQUEST-Werte der Proteine zn177, dtx1, mfn1, ccd89 und icb1 sind im Vergleich zu
der oben diskutierten Sequenz deutlich schlechter, obgleich ihre Sp-Werte die gewählten
Kriterien (Sp-Wert ≥ 500) erfüllen. Die schlechtere Zuordnung zeigt sich jedoch in den ∆CnWerten deutlich (∆Cn ≥ 0,16). Somit kann bei diesem Proteinstück auf Grund des Vorliegens
von platinierten Ionen im MS/MS-Spektrum und der SEQUEST-Parameter die Aminosäure
K368 als Bindungsstelle eindeutig bestätigt werden. Diese Bindungsstelle konnte, wie aus
Tabelle 6.25 hervorgeht, zweimal bei der zweiten Blutserumprobe ebenfalls identifiziert
werden.
Die Zugänglichkeit der Seitenkette des Lysins lässt sich in dem in der Abbildung 6.30
dargestellten Ausschnitt aus der Proteinstruktur zeigen.
Serumproteine
164
K368
Abb.6.30: Ausschnitt aus der Proteinstruktur von Alpha-1-Antitrypsin (PDB ID: 1hp7 [139])
Die Seitenkette ist, wie aus der Abbildung erkennbar, in einer Schleife lokalisiert, die zu einer
Bindungstasche gehört. Somit kann K368 von dem Cisplatinfragment leicht koordiniert
werden.
Mit dem reinen Serumprotein Alpha-1-Antitrypsin wurden ebenfalls Reaktionsansätze mit
Cisplatin untersucht. Dabei sind wiederum Produkte des äquimolaren Protein-CisplatinGemisches nach 3h und des 1:5 -Gemisches nach 168h Inkubation bei 37 °C analysiert
worden. Beide Analysen ergaben jedoch keine mit einem Cisplatinfragment koordinierten
Peptidsequenzen, deren SEQUEST-Parameter die gewählten Kriterien erfüllen (XCorr ≥ 3,3
für dreifach, XCorr ≥ 2,4 für zweifach geladene Molekülionen; ∆Cn ≥ 0,1; Sp-Wert ≥ 500;
RSp-Wert ≤ 5; ∆m ± 2,5 und 30% Ionenzuordnung).
Aus den beiden Blutserumproben konnten somit D107 und K368 als Cisplatinbindungsstellen
ermittelt werden. Die Koordination von E264 konnte nicht bei A1AT:Cisplatin Ansätzen
bestätigt werden. Die zweifach beobachtete Koordination des O- und die des N(Amino)Donoratoms weist darauf hin, dass es sich bei D107 und K368 um spezifische
Bindungsstellen des Cisplatins handelt. Beide Seitenketten können in der folgenden
Darstellung in der Gesamtstruktur des Alpha-1-Antitrypsins betrachtet werden.
Serumproteine
165
D107
K368
Abb.6.31: Proteinstruktur von Alpha-1-Antitrypsin
Die Seitenketten der koordinierten Aminosäuren D107 und K368 sind in flexiblen Schleifen
lokalisiert, wobei D107 an der Oberfläche und K368 in einer Bindungstasche gelegen sind.
6.5. Cisplatinaddukte des Alpha-2-Macroglobulins
In der Literatur [90] wurde aus Reaktionen von Cisplatin und NAMI-A mit Blutserum das
Alpha-2-Macroglobulin (A2MG) als Proteintarget massenspektrometrisch charakterisiert. In
dieser Studie konnte jedoch keine Bindungsstelle bestimmt werden, lediglich eine
Adduktbildung zwischen dem Protein und Cisplatin wurde bestätigt. Aus der MudPITAnalyse der zweiten Blutserumprobe konnte das koordinierte Protein ebenfalls detektiert
werden (siehe Tabelle 6.27).
Tab.6.27: Koordinierte Peptidsequenzen von A2MG aus der zweiten Blutserumprobe
Sp RSp Ionen
Peptid
Bindungsstelle z XCorr ∆Cn
R.LLIYAVLPTGDVIGDS~.A
S532
2 2,88 0,33 744,3
1 22/30
S.NE#EVMoxFLTVQVK.G
E75
2 4,32 0,12 1511,5 1
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
17/22
Serumproteine
166
Die Tabelle enthält zwei Peptidsequenzen aus dem etwa 168 kDa großen Protein. Es wurden
die Seitenketten eines Serin und einer Glutaminsäure des Proteins platiniert.
Die Peptidsequenz [74NE#EVMoxFLTVQVK85]2+ wurde von dem Metallrest [(NH3)2PtCl]+
an der Glutaminsäure 75 koordiniert. In direkt benachbarter Position steht für eine mögliche
Cisplatinkoordination eine weitere Glutaminsäure zur Verfügung. Das zugehörige MS/MSSpektrum in der nachstehenden Abbildung soll klären, ob eine Entscheidung zu Gunsten einer
dieser Aminosäuren als Koordinationsstelle gefällt werden kann.
Abb.6.32: MudPIT-MS/MS-Spektrum von [NE#EVMoxFLTVQVK]2+ in A2MG aus der
zweiten Blutserumprobe
In dem Spektrum konnte eine Zuordnung von 17 Ionen von insgesamt 22 möglichen erfolgen.
Die Ionen b2+, b5+- b11+ und y2+- y4+ und y6+- y10+ sind dargestellt. Bei einer E75-Koordination
sollen alle beobachteten b+-Ionen platiniert sein. In der folgenden Tabelle sind andere
mögliche Peptidsequenzen mit der Masse m/z = 1717 (∆m ± 2,5) aufgelistet.
Serumproteine
167
Tab.6.28: SEQUEST-Parameter von [NE#EVMoxFLTVQVK]2+ im Vergleich zu anderen
potentiellen Peptidsequenzen in A2MG aus der zweiten Blutserumprobe
Sequenz
S.NE#EVMoxFLTVQVK.G
-.NEE#VMoxFLTVQVK.-.K#NEM#LLSKVK.-.FVKSLNK#QMNPF.-.IYDGGARTE#DEVQ.-.PYLTVDQMoxMDFINL.-.IYDGGARTED#EVQ.-.FVKSLNKQM#NPF.-.K#NE#MLLSKVK.-.PYLTVDQMMoxDFINL.-
XCorr
4,324
4,022
3,795
3,65
3,498
3,469
3,434
3,408
3,404
3,342
∆Cn
0
0,07
0,122
0,156
0,191
0,198
0,206
0,212
0,213
0,227
Sp
RSp Ionen
Protein
1511,5
1
17/22 A2MG_HUMAN
1121,6
2
15/22 A2MG_HUMAN
919,4
9
13/18 CCD93_HUMAN
1031,3
3
15/22 XPOT_HUMAN
733,3
38 13/24 CLD18_HUMAN
815,1
23 15/26 PLCB1_HUMAN
725,5
44 13/24 CLD18_HUMAN
780,6
29 14/22 XPOT_HUMAN
715
49 12/18 CCD93_HUMAN
811,5
24 15/26 PLCB1_HUMAN
Aus der Tabelle wird deutlich, dass neben E75 auch die benachbarte Glutaminsäure E76 als
potentielle Bindungsstelle in Frage kommt. Anhand des beobachteten b2+-Ions in dem
MS/MS-Spektrum (Abb. 6.32), welches bei der Bindung an E76 nicht vorkommen sollte und
kein Platinpattern aufweisen dürfte, kann E75 als die wahrscheinlich bevorzugte Stelle
angesehen werden. Die vergleichbaren ∆Cn-Werte (und weitere Parameter, wie Sp-Werte) der
Peptidsequenzen beider Koordinationsstellen lassen jedoch keine eindeutige Entscheidung zu
Gunsten einer dieser Bindungsstellen zu.
Das Serumprotein Alpha-2-Macroglobulin wurde außerdem mit Cisplatin nach 3h und nach
168h Inkubation bei 37 °C bei einem Protein:Metallkomplex-Verhältnis von 1:5 sowie beim
1:1 Verhältnis nach 3h Inkubation auf entstandene Produkte untersucht, die in der Tabelle
6.29 aufgelistet sind.
Serumproteine
168
Tab.6.29: Koordinierte Peptidsequenzen aus den Ansätzen A2MG:Cisplatin nach 3h (1:1 und
1:5) und nach 168h (1:5) Inkubation bei 37°C
Peptid
1:1 Ansatz nach 3h/37°C
L.FFT§VLQDVPVR.D
Bindungsstelle z XCorr ∆Cn
Sp
RSp Ionen
T1409
2
2,85
0,31
532,4
1
11/20
1:5 Ansatz nach 3h/37°C
L.FFT§VLQDVPVR.D
T1409
2
2,66
0,14
580,4
2
11/20
L.IY~AVLPTGDVIGDSAK.Y
Y520
2
3,34
0,44
659,3
1
20/30
S.NE#EVMoxFLTVQVK.G
S.NE#EVMFLTVQVK.G
E75
E75
2
2
4,33
4,06
0,31 1286,7
0,33 1473,1
1
1
16/22
17/22
1:5 Ansatz nach 168h/37°C
K.M§VSGFIPLKPTVK.M
F.FT§VLQDVPVR.D
M1362
T1409
2
2
2,87
2,86
0,57 1906,9
0,55 1366,5
1
1
22/24
14/18
K.DMYSFLED~MoxG.L
D649
2
2,78
0,58
1
12/18
1
1
28/60
15/22
801,0
L.LQQVS#LPELPGEYSMK.V
S1281
3 4,04 0,52 1037,1
S.NE#EVMFLTVQVK.G
E75
2 3,04 0,29 1087,2
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
In der Tabelle findet sich beim 1:1 Ansatz die Peptidsequenz [1430FFT§VLQDVPVR1440]2+,
welche an der Seitenkette des Threonins 1409 von [Pt]2+ koordiniert wird. Dieses Ergebnis,
dass nur eine metallierte Peptidsequenz in ausreichender Konzentration vorliegt um
identifiziert werden zu können, erscheint nachvollziehbar, wenn die Größe des Proteins (1474
Aminosäuren und etwa 163 kDa) gegenüber der äquimolaren Umsetzung mit Cisplatin in
Betracht gezogen wird.
Die Reaktionen mit einem 5:1 Metallkomplexüberschuss liefern dagegen mehrere
koordinierte Peptidsequenzen. Nach 3h Inkubation sind vier Peptide aus dem Serumprotein
von einem der Cisplatinfragmente koordiniert und identifiziert worden. Die Aminosäuren
T1409 und Y250 sowie zweimal E75 wurden mit einem Metallfragment modifiziert. Bei dem
1:5 Ansatz mit längerer Inkubationszeit von 168h bei 37°C konnten fünf von einem
Cisplatinfragment koordinierte Peptidsequenzen identifiziert werden. Dabei wurden jeweils
einmal M1362, T1409, S1282, D649 und E75 modifiziert.
Eine Betrachtung der Cisplatin-Bindungsstellen in der Gesamtproteinstruktur entfällt an
dieser Stelle, da von der Struktur nur kurze Fragmente bekannt sind und die entsprechenden
Serumproteine
169
Bindungsstellen nicht dargestellt werden können. Es lässt sich feststellen, dass eine
spezifische Bindung des Cisplatins an die Aminosäuren E75 und T1409 vermutet werden
könnte, da diese jeweils aus drei unterschiedlichen Analysen als Koordinationsstellen
identifiziert werden konnten.
6.6. Proteintargets in Apolipoprotein A2
Aus beiden Blutserumproben konnten Peptidsequenzen eines weiteren abundanten Proteins
detektiert werden, des Apolipoproteins A2 (ApoA2). Es handelt sich hierbei um ein etwa 11
kDa großes Protein mit 100 Aminosären, welches die HDL-Struktur (high density lipoprotein)
stabilisieren kann. Die Liste der identifizierten Peptide, welche mit einem Cisplatinfragment
modifiziert wurden, ist in der Tabelle 6.30 dargestellt.
Tab.6.30: Koordinierte Peptidsequenzen des Apolipoproteins A2 aus der ersten und zweiten
Blutserumprobe
Sp
RSp Ionen
Peptid
Bindungsstelle z XCorr ∆Cn
erste Blutserumprobe
K.KAGTELVNFLSYFVELGT§QP.A
T72
2 4,19 0,38 1288,4 1 21/38
E.PC$VESLVSQYFQTVTDYGK.D
C6
2 3,94
0,26
542,3
1
17/36
K.KAGTELVNFLSYFVELGT~QPA.T
V.E~SLVSQYFQTVTDYGK.D
T72
E8
2 3,65
2 5,46
0,18 1252,1
0,52 1886,5
1
1
21/40
22/30
P.C#VESLVSQYFQTVTDYGK.D
C6
2 3,65
0,25 1186,7
1
21/34
zweite Blutserumprobe
P.C§VESLVSQYFQTVTDYGK.D
C6
2 4,54 0,49 1193,5
§ = [Pt]2+, $ = [(NH3)Pt]2+, ~ = [(NH3)2Pt]2+, # = [(NH3)2PtCl]+
1
22/34
In der Tabelle finden sich sechs von einem der Cisplatinfragmente koordinierte Peptide. Es
lässt sich erkennen, dass in beiden Blutserumproben Cystein 6 bzw. Glutaminsäure 8
koordiniert vorliegt. Es wurde aus der ersten Blutserumprobe zweimal die Modifizierung von
T72 identifiziert, wobei sich beide Sequenzen um eine Aminosäure (Alanin am C-Terminus)
unterscheiden.
Serumproteine
Das
MS/MS-Spektrum
170
des
halbtryptisch
gespaltenen
Peptids
der
Sequenz
[6C#VESLVSQYFQTVTDYGK23]2+, welches in beiden Serumproben mit dem Fragment
[(NH3)2PtCl]+ platiniert wurde, ist in der Abbildung 6.33 zu finden.
Abb.6.33: MudPIT-MS/MS-Spektrum von [C#VESLVSQYFQTVTDYGK]2+ in ApoA2 aus
der ersten Blutserumprobe
Aus der an der Aminosäure C6 koordinierten Sequenz sind insgesamt 18 Fragmentionen (21
Ionen von SEQUEST zugeordnet) mit sechs b+- und 12 y+-Ionen zugeordnet worden. Die
Ionen b1+ bis b17+ und keine aus der y+-Reihe sollten für eine C6-Koordination platiniert sein.
Jedoch kann diese Tatsache nicht für die bj+-Ionen mit j ≤ 4 eindeutig bestimmt werden. Das
Peptid enthält neben C6 mit E8 und S9 weitere potentielle Bindungsstellen für das Cisplatin.
Die potentiellen Sequenzen mit der Masse von m/z = 2331 (Massenbereich ∆m ± 2,5) sind in
der Tabelle 6.31 aufgelistet.
Serumproteine
171
Tab.6.31: SEQUEST-Parameter von [C#VESLVSQYFQTVTDYGK]2+ im Vergleich zu
anderen potentiellen Peptidsequenzen in ApoA2 aus der ersten Blutserumprobe
Sequenz
XCorr
P.C#VESLVSQYFQTVTDYGK.D 3,653
P.CVE#SLVSQYFQTVTDYGK.D 3,614
P.CVES#LVSQYFQTVTDYGK.D 3,432
I.C#STAQRMoxYQSLEHELQK.H 2,735
P.CVESLVS#QYFQTVTDYGK.D 2,696
R.C#IDSGLWVPNSKASEAK#.E
2,596
R.C#IDSGLWVPNSK#ASEAK.E
2,582
R.T#FAAQSLVQLLSKLPCGEY.A 2,574
R.C#IDSGLWVPNSKAS#EAK.E
2,518
R.C#IDSGLWVPNSKASE#AK.E
2,438
∆Cn
0
0,011
0,06
0,251
0,262
0,289
0,293
0,295
0,311
0,333
Sp
RSp Ionen
Protein
1186,7 1 21/34 APOA2_HUMAN
945,2
2 19/34 APOA2_HUMAN
812,5
3 18/34 APOA2_HUMAN
274,2 154 11/32 SYNE1_HUMAN
355,6 30 13/34 APOA2_HUMAN
437
7 14/32 CDC37_HUMAN
382,6 18 13/32 CDC37_HUMAN
256,6 223 12/36 CNDD3_HUMAN
382,6 18 13/32 CDC37_HUMAN
382,6 18 13/32 CDC37_HUMAN
Aus dem Protein Apolipoprotein A2 ist die oben genannte Sequenz mit den drei
unterschiedlichen Bindungsstellen in der Tabelle aufgeführt. Die Bindungsstellen C6, E8 und
S9 ergeben mit dem theoretischen Spektrum des Peptids die besten Übereinstimmungen mit
dem aufgenommenen Spektrum, so dass nur diese Modifikationen die SEQUEST-Kriterien
erfüllen. Bei den unter Abb. 6.33 zugeordneten Ionen b1+ und b2+ lässt sich keine
Platinverteilung erkennen, die im Falle einer Koordination an Cystein zu beobachten sein
müsste. Die yk+-Ionen werden außerdem nur für k ≤ 15 beobachtet, was möglicherweise für
die E8-Koordinaiton sprechen würde. Ein weiterer Hinweis auf eine Platinierung von E8 ist
die Identifizierung einer Peptidsequenz aus der ersten Blutserumprobe, welche an Valin 7
gespalten wurde (siehe Tab. 6.30). Die vermutlich spezifische Koordinationsstelle E8 (oder
C6, S9) für Cisplatin ist in der folgenden Abbildung 6.34 dargestellt.
S9
E8
C6
Abb.6.34: Ausschnitt aus der Proteinstruktur von Apolipoprotein A2 (PDB ID: 2ou1 [140])
Serumproteine
172
Die Vergrößerung aus der Struktur in der oberen Abbildung enthält den vermutlich von
Cisplatin koordinierten Bereich des Proteins. Die Seitenketten der drei möglichen
Koordinationsstellen C6, E8 und S9 sind alle sterisch für eine Cisplatinkoordination leicht
zugänglich, wie in der Abbildung zu erkennen ist.
6.7. Cisplatinaddukte weiterer abundanter Proteine
Neben den bisher diskutierten abundanten Serumproteinen konnten aus den MudPITAnalysen der beiden Blutserumproben weitere Proteintargets detektiert werden. Im Folgenden
ist eine Auswahl von sieben Proteinen dargestellt, welche mit entsprechenden
Peptidsequenzen aus beiden Messreihen identifiziert werden konnten.
Tab.6.32: Weitere koordinierte Serumproteine aus beiden Blutserumproben
Protein
A1AG1_HUMAN
APOC2_HUMAN
CERU_HUMAN
HV305_HUMAN
IGHG2_HUMAN
IGHG3_HUMAN
TTHY_HUMAN
BindungsPeptid
stelle
z XCorr ∆Cn
K.T~YMLAFDVNDE.K
T109
2 3,07 0,19
A.AM#STYTGIFTDQVLSVLK.G
M60
2 3,77 0,21
I.S~VDTEHSNIYLQNGPDR.I
S27
2 6,01 0,46
-.EVQLVESGGGLVQPGGS~L.R
S17
2 3,46 0,19
V.S~VLTVVHQDWLNGK.E
S183
2 5,05 0,43
V.S~VLTVLHQNWLDGK.E
S147
2 5,62 0,38
R.YTIAALLSPYSYSTTAVVT~N.P
T123
2 4,29 0,36
2+
2+
2+
§ = [Pt] , $ = [(NH3)Pt] , ~ = [(NH3)2Pt] , # = [(NH3)2PtCl]+
Sp RSp Ionen
1181,1 1 15/20
640,8
2 15/34
1560,6 1 21/32
1194,6 1 19/34
2351,1 1 23/26
2830,0 1 23/26
1254,0 1 25/38
Es handelt sich dabei um die Proteine: Alpha-1-Glycoprotein, Apolipoprotein C2,
Ceruloplasmin, Ig heavy chain V-III-Protein, Ig gamma-2 und -3 chain C-Proteine und das
Prealbumin. Auch bei diesen Proteinen wurden im Wesentlichen die kinetisch bevorzugten
Bindungsstellen für Cisplatin identifiziert (Methionin, Serin und Threonin). Aus beiden
Blutserumanalysen konnten auch einige nicht abundante Proteine identifiziert werden, deren
experimentelle MS/MS-Spektren mit den theoretischen Spektren gute Übereinstimmungen
ergeben. Bei seiner Studie mit S. cerevisiae konnte YATES [141] zeigen, dass 71,6% aller
abundanten Proteine mit etwa 104-105 Kopien pro Zelle aus einem MudPIT-Lauf identifiziert
werden können, wogegen es nur noch 6,5% sind bei 102-103 Kopien pro Zelle sind. Wird die
Tatsache berücksichtigt, dass durchschnittlich nur etwa 30-50% der Peptidsequenzen bei
abundanten Proteinen durch MudPIT detektiert werden, bedeutet dies, dass sicherlich nur ein
kleiner Anteil der nicht abundanten Proteintargets durch MudPIT identifiziert werden können.
Serumproteine
173
Es konnten nur wenige Proteine als Targets für Cisplatin in beiden Analysen identifiziert
werden, vor allem auch wenige Proteine mit für die DNA-Reparatur oder Apoptose-Induktion
relevanten Funktionen. Um falsche Schlussfolgerungen zu vermeiden, wird auf eine
detaillierte Auflistung dieser Proteine verzichtet. Bemerkenswert ist die CisplatinKoordination von Proteinen wie MSH6 (DNA mismatch repair protein), Helikasen,
unterschiedliche Zinkfinger-Formen, Topoisomerasen oder HMGA2 (high mobility group
protein), die allerdings nur jeweils in einer der beiden Blutserumproben identifiziert wurden.
In beiden Blutserumproben konnte allerdings eine Koordination an ein ssbP3-Protein (singlestranded DNA-binding protein 3) detektiert werden, jedoch ist diese an zwei
unterschiedlichen Serinseitenketten (S142 und S306) erfolgt, so dass daraus nicht auf eine
spezifische Wechselwirkung des Cisplatins mit dem Protein geschlossen werden kann.
6.8. Diskussion
Für die einzelnen Serumproteine wurden in den zugehörigen Unterkapiteln Tabellen mit den
koordinierten Peptidsequenzen des jeweiligen Proteins zu allen durchgeführten Analysen
aufgelistet. An dieser Stelle werden die Bindungsstellen des Cisplatins in den Proteinen erneut
betrachtet, um mögliche charakteristische Wechselwirkungen zu erkennen.
Bei den Untersuchungen der Interaktionen zwischen Metallkomplexen wie Cisplatin und
Serumproteinen ist bisher nur wenig über genaue Bindungsstellen bekannt. Beispielweise
konnte aus der Reaktion zwischen Serotransferrin und Cisplatin eine Koordinationsstelle des
[(NH3)2PtCl]+-Fragmentes an Threonin 457 durch „bottom-up“ ESI-MS/MS identifiziert
werden (siehe Kapitel 3) [137]. Das Cystein 34 im Serumalbumin wurde von den
Arbeitsgruppen GONIAS und PIZZO [142] und MOMBURG et al [143] durch biochemische
Techniken als Bindungsstelle des Cisplatins identifiziert.
Wie aus den Tabellen 6.1, 6.2, 6.8 und 6.9 ersichtlich wird, konnten in den unterschiedlichen
Analysen Cystein 34 bzw. die benachbarten Seitenketten E37, D38 und H39 des
Serumalbumins als eine Cisplatinbindungsstelle identifiziert werden. Wie jedoch bereits beim
Beispiel aus Tabelle 6.11/Abb. 6.10-6.13 deutlich wird, stellen diese durch SEQUEST
bevorzugten Koordinationsstellen meist nicht die einzig möglichen Bindungsstellen dar. In
allen MS/MS-Spektren der betroffenen Sequenz konnten keine bj+-Ionen für Fragmente mit
Aminosäureresten ab 34 beobachtet werden. Das gilt für die nicht dargestellten MS/MSSpektren
dieser
Sequenz
und
deutet
auf
eine
tatsächliche
Koordination
des
Cisplatinfragmentes an Cystein. Der in Tabelle 6.1 angegebene XCorr-Wert für die Histidin-
Serumproteine
174
Koordination als beste Zuordnung ist durch eine SEQUEST-Zuordnung von y+-Ionen im
niedrigen Massenbereich entstanden. Die Peaks solcher Ionen weisen allerdings häufig
vernachlässigbare Intensitäten auf und befinden sich in einem Bereich (m/z ≤ 800) mit hohem
Untergrundrauschen. Die Zuordnung ist deswegen vielfach unzuverlässig. Im Einklang mit
den Beobachtungen anderer Gruppen [142-144] deuten diese darauf hin, dass die freie
Thiolgruppe des C34 in der Tat die bevorzugte Bindungsstelle für das Cisplatin aufweist.
Die Gruppe um NEAULT [144] konnte mit Hilfe von IR-Studien zeigen, dass
Tyrosinseitenketten aus dem Serumalbumin an einer Cisplatinbindung beteiligt sind.
Sowohl bei den einzelnen Analysen mit Blutserum als auch mit dem reinen Protein konnten
Peptidsequenzen mit den koordinierten Tyrosinresten Y148 oder Y150 identifiziert werden.
Dabei wurden Sequenzen mit unterschiedlichen Spaltungen beobachtet, die keine klare
Zuordnung von Y148 bzw. Y150 ermöglichen. SADLERS NMR-Studien fanden kein Indiz
dafür, dass Histidin in die Cisplatinbindung involviert sein könnte, so dass die benachbarte
Aminosäure H146 als Angriffsstelle ausgeschlossen wird. Wie aus den Auflistungen
erkennbar, scheinen die Tyrosinseitenketten des Y148 und Y150 als Bindungsstellen zu
konkurrieren.
Aus
der
äquimolaren
Cisplatin-Albumin-Analyse
konnte
auch
eine
2+
Modifizierung mit dem [Pt] -Rest identifiziert werden, so dass bei dieser Koordination eine
Beteiligung beider Seitenketten nicht ausgeschlossen wird. Die Möglichkeit einer
gleichzeitigen Koordination lässt sich in der Vergrößerung aus der Proteinstruktur gut zeigen.
Y150
Y148
Abb.6.35: Ausschnitt aus der Proteinstruktur des Serumalbumins
Serumproteine
175
Aus Abbildung 6.35 wird deutlich, dass beide Seitenketten in einer Schleife liegen und in
direkter Nachbarschaft zueinander stehen. Zu der mit [(NH3)2PtCl]+ koordinierten
Peptidsequenz [145RH#PYFYAPELLFFA158]2+ aus Tabelle 6.1 wird an dieser Stelle das
MS/MS-Spektrum dargestellt (Abb. 6.36).
Abb.6.36: MudPIT-MS/MS-Spektrum von [RH#PYFYAPELLFFA]2+ aus Serumalbumin der
ersten Blutserumprobe
Das Ausschließen der Histidin-Koordination wurde oben diskutiert. In dem dargestellten
Spektrum der oben genannten Peptidsequenz wird deswegen nach Hinweisen für Y148 oder
Y150 als Bindungsstelle gesucht. Ein Anhaltspunkt für die Bevorzugung der Koordination an
Y148 bietet die fragliche Zuordnung des Ions y12+ in Abbildung 6.36, die als Einzellinie zu
beobachten ist. Die sehr geringe Intensität dieses Peaks stellt die Zuordnung in Frage, so dass
sie Anzeichen auf die bevorzugte Bindung an Aminosäure Y148 darstellt. Die Tatsache, dass
die y9+- und y10+-Ionen ebenfalls im Vergleich zu y7+ und y8+ sehr wenig intensiv sind, lässt
jedoch auch eine Y150-Bindungsstelle zu.
Mittels NMR-Spektroskopie (1H,
15
N HSQC) konnte aus dem Gemisch aus Serumalbumin
und Cisplatin nach einer Inkubation von 24h/37°C ein κ2SM,NM-Komplex charakterisiert
Serumproteine
176
werden [61]. Wegen seiner Lokalisierung auf der Oberfläche des Proteins wurde von den
Autoren das Methionin 298 als Bindungsstelle vermutet. Bei dieser Studie wurden auch
Addukte mit der Thiolgruppe des Cysteins 34 und an weiteren Methioninstellen beobachtet.
Die Lage der M298 in der Proteinstruktur ist in der folgenden Abbildung gezeigt.
M298
Abb
Abb.6.37: Proteinstruktur des Serumalbumins
Die 2D-NMR-Analysen weisen somit daruf hin, dass die Aminosäure Cystein C34 nicht die
einzige bevorzugte Angriffsstelle für Cisplatin darstellt [61]. Außer M298 wurden auch die
Methioninpositionen 87 und 446 auf Grund ihrer Zugänglichkeit an der Oberfläche auch als
mögliche Koordinationsstellen diskutiert. Das von SADLER aufgestellte Reaktionsschema zur
Reaktion von Serumalbumin mit Cisplatin ist in Kapitel 3 in der Abildung 3.6 gezeigt.
Das
Serumprotein
Albumin
weist
in
seiner
Proteinsequenz
insgesamt
sechs
Methioninpositionen auf: M87, M123, M298, M329, M446 und M548.
Aus Tabellen 6.1, 6.2, 6.7-6.9 ist erkennbar, dass von den sechs Methioninseitenketten zwei
(M329 und M548) sowohl aus den MudPIT- als auch aus den LC-MS/MS-Analysen für die
einzelnen Protein-Cisplatin-Analysen als Bindungsstellen für Cisplatin bestätigt werden
konnten. Die tryptisch gespaltenen Fragmente mit diesen Methioninstellen würden
Peptidsequenzen mit den folgenden Aminosäurenanzahlen ergeben: um M87 12
Aminosäuren, für M123 19 Aminosäuren, für M298 27 Aminosäuren, für M329 13
Aminosäuren, für M446 21 Aminosäuren und für M548 12 Aminosäurenreste. Da das
tryptische Fragment um M298 ein Proteinteilstück aus 27 Aminosäuren (S287-K313) liefert,
Serumproteine
177
könnte die Länge des Peptids der Grund dafür sein, dass der Rest massenspektrometrisch
nicht detektiert werden kann und keine ausreichende Fragmentierung ergeben würde.
Neben den oben diskutierten Bindungsstellen (C34, Y148, Y150, M329 und M548) kann
durch die Identifizierung mehrerer koordinierter Peptidsequenzen aus Serumalbumin auch
eine Carboxylatgruppe (D375/E376) als Koordinationsstelle charakterisiert werden.
Die untere Abbildung zeigt die Proteinstruktur des Serumalbumins mit den CisplatinBindungsstellen.
III B
III A
II B
II A
IB
IA
Abb.6.38: Struktur des Serumalbumins mit den identifizierten Koordinationsstellen
Die Verteilung der identifizierten Bindungsstellen für Cisplatin im Protein wird aus der
Darstellung deutlich. Die Lokalisierung der Aminosäurenseitenketten von M329, D375 und
M548 an der Oberfläche einzelner α-Helices [145] indiziert ihre Bereitschaft zur Bindung.
Dagegen liegen die Seitenketten von C34 und Y150 in flexiblen Schleifen von
Bindungstaschen der Subdomäne IA (für C34) und IA/IIA (für Y150).
Die folgende Tabelle gibt die gesamte Sequenz für Serumalbumin mit allen 609
Aminosäuren. Dabei ist zu beachten, dass die Nummerierung der Aminosäuren entsprechend
der tatsächlichen Proteinkette erfolgt. Für Serumalbumin sind es die Aminosäuren D25-L609.
Die mittels MudPIT-Technologie identifizierten Bereiche im Serumalbumin aus der ersten
Blutserumprobe-Analyse werden grün markiert. Dabei wurden die folgenden Kriterien
Serumproteine
178
verwendet: XCorr-Werte für einfach XCorr ≥ 1,8, für zweifach XCorr ≥ 2,5 und für dreifach
geladene Moleküle XCorr ≥ 3,5; Sp-Werte ≥ 500; Ionenzuordnung ≥ 30% und die erste (mit
dem höchsten XCorr-Wert) einbezogene Peptidsequenz zu einer bestimmten Masse. Diese
Einstellungen gehören zu den häufig verwendeten in der Proteomik [z. B. 96,146].
Tab.6.33: Die gesamte Proteinsequenz des Serumalbumins mit den massenspektrometrisch
detektierten Sequenzen in grün
MKWVTFISLLFLFSSAYSRGVFRRDAHKSEVAHRFKDLGEENFKALVLIAFAQYLQQ
CPFEDHVKLVNEVTEFAKTCVADESAENCDKSLHTLFGDKLCTVATLRETYGEMAD
CCAKQEPERNECFLQHKDDNPNLPRLVRPEVDVMCTAFHDNEETFLKKYLYEIARR
HPYFYAPELLFFAKRYKAAFTECCQAADKAACLLPKLDELRDEGKASSAKQRLKCA
SLQKFGERAFKAWAVARLSQRFPKAEFAEVSKLVTDLTKVHTECCHGDLLECADDR
ADLAKYICENQDSISSKLKECCEKPLLEKSHCIAEVENDEMPADLPSLAADFVESKDV
CKNYAEAKDVFLGMFLYEYARRHPDYSVVLLLRLAKTYETTLEKCCAAADPHECY
AKVFDEFKPLVEEPQNLIKQNCELFEQLGEYKFQNALLVRYTKKVPQVSTPTLVEVS
RNLGKVGSKCCKHPEAKRMPCAEDYLSVVLNQLCVLHEKTPVSDRVTKCCTESLVN
RRPCFSALEVDETYVPKEFNAETFTFHADICTLSEKERQIKKQTALVELVKHKPKATK
EQLKAVMDDFAAFVEKCCKADDKETCFAEEGKKLVAASQAALGL
In der Sequenz wurden alle detektierten Peptidsequenzen grün gekennzeichnet. Daraus ist
erkennbar, dass etwa 54% der Aminosäuren aus einem der detektierten Fragmente
identifiziert werden konnten. Die oben genannten sechs Methioninstellen sind eingerahmt und
es wird deutlich, dass drei Stellen (rot unterlegt) M87, M298 und M446 in keiner der
Analysen gefunden wurde. Die fünf detektierten Cisplatinbindungsstellen sind eingerahmt
und gelb gefärbt. Ähnliche „coverage“-Werte wurden für die zweite Blutserumprobe sowie
für die Einzelanalysen von Serumalbumin erhalten.
Den Tabellen 6.12, 6.15, 6.17 und 6.19 können die aus allen Analysen identifizierten
Bindungsstellen des Cisplatins im Serotransferrin entnommen werden. Unter den
Koordinationsstellen findet sich die Seitenkette des Tyrosins 314. Bei den Untersuchungen
des Serotranferrin/Cisplatin-Systems konnten etwas höhere SEQUEST-Parameter für die
alternative Koordination an M313 beobachtet werden (Tab. 6.16). Auf Grund einer fraglichen
Zuordnung des b+-Ions (z. B. Abb. 6.17), kann jedoch nicht zwischen M313 und Y314 als
Bindungsstelle für Cisplatin entschieden werden. Eine Zuordnung zu Y314 wird durch die
Beobachtung begründet, dass zusätzlich zu dieser Peptidsequenz [MY§LGYEYVTAIR]2+ mit
Serumproteine
179
der Koordination des [Pt]2+-Fragments auch koordinierte Sequenzen beobachtet wurden, in
denen das Methionin fehlt (siehe Tab. 6.19). Aus [1H,
13
C] und [1H,
15
N]-HSQC-NMR
Studien von SADLER konnten außerdem keine Hinweise darauf erhalten, dass M313 an der
Koordination des Cisplatins beteiligt ist [58].
In Serotransferrin sind neun Methioninstellen enthalten: M26, M109, M256, M309, M313,
M382, M389, M464 und M499 mit den jeweiligen tryptisch gespaltenen Peptidsequenzen der
Längen von 9, 10, 5, 4, 12, 21, 21, 14 und 15 Aminosäureresten. Auch hier erfolgt die
Nummerierung entsprechend der tatsächlichen Proteinkette (V20-P698). Zur Verdeutlichung
der
Lokalisierung
dieser
Aminosäuren
dient
die
folgende
Auflistung
der
Serotransferrinsequenz.
Tab.6.34: Die gesamte Proteinsequenz des Serotransferrins mit den massenspektrometrisch
detektierten Sequenzen in grün
MRLAVGALLVCAVLGLCLAVPDKTVRWCAVSEHEATKCQSFRDHMKSVIPSDGPSV
ACVKKASYLDCIRAIAANEADAVTLDAGLVYDAYLAPNNLKPVVAEFYGSKEDPQT
FYYAVAVVKKDSGFQMNQLRGKKSCHTGLGRSAGWNIPIGLLYCDLPEPRKPLEKA
VANFFSGSCAPCADGTDFPQLCQLCPGCGCSTLNQYFGYSGAFKCLKDGAGDVAFV
KHSTIFENLANKADRDQYELLCLDNTRKPVDEYKDCHLAQVPSHTVVARSMGGKED
LIWELLNQAQEHFGKDKSKEFQLFSSPHGKDLLFKDSAHGFLKVPPRMDAKMYLGY
EYVTAIRNLREGTCPEAPTDECKPVKWCALSHHERLKCDEWSVNSVGKIECVSAETT
EDCIAKIMNGEADAMSLDGGFVYIAGKCGLVPVLAENYNKSDNCEDTPEAGYFAVA
VVKKSASDLTWDNLKGKKSCHTAVGRTAGWNIPMGLLYNKINHCRFDEFFSEGCAP
GSKKDSSLCKLCMGSGLNLCEPNNKEGYYGYTGAFRCLVEKGDVAFVKHQTVPQN
TGGKNPDPWAKNLNEKDYELLCLDGTRKPVEEYANCHLARAPNHAVVTRKDKEAC
VHKILRQQQHLFGSNVTDCSGNFCLFRSETKDLLFRDDTVCLAKLHDRNTYEKYLGE
EYVKAVGNLRKCSTSSLLEACTFRRP
In der oberhalb dargestellten Proteinsequenz des Serotransferrins sind wiederum die in einer
der Blutserumproben identifizierten Abschnitte grün markiert. Zur Verdeutlichung der
Verteilung der Bindungsstellen für Cisplatin aus den Analysen sind auch die modifizierten
Bereiche der reinen Protein:Metallkomplex-Gemische eingeteilt. Unter Berücksichtigung der
oben genannten Kriterien (XCorr-Werte für einfach XCorr ≥ 1,8, für zweifach XCorr ≥ 2,5
und für dreifach geladene Moleküle XCorr ≥ 3,5; Sp-Werte ≥ 500; Ionenzuordnung ≥ 30%
Serumproteine
180
und die erste (mit dem höchsten XCorr-Wert) einbezogene Peptidsequenz zu einer
bestimmten Masse) konnten aus Serotransferrin etwa 51% der Aminosäuren identifiziert
werden. Von den oben genannten neun Methioninen konnten drei in keiner der
durchgeführten Analysen detektiert werden: M26, M309 und M499. Wie bereits erwähnt
untersuchte SADLER die Cisplatinbindungsstellen in humanem Transferrin mittels NMRSpektroskopie [58]. Wie in der folgenden Abbildung aus diesen Studien gezeigt, konnte eine
Koordination an M256 und M499 beobachtet werden.
Abb.6.39: 2D [ 1H, 13C] HSQC : A Transferrin; B Transferrin nach Inkubation 24h/37°C aus
[58]
In der Darstellung sind E und E´ Pt-S13CH3-Gruppen und auf Grund abnehmender
Intensitäten der Resonanzen während der Reaktion den Seitenketten von M256 und M499
zugeordnet. Wie aus Tabelle 6.19 ersichtlich, konnte im Rahmen der eigenen Studien die
Modifizierung des M256 bei einem fünffachen Cisplatinüberschuss nach einer Inkubation von
168h/37°C beobachtet werden. Da M499 nicht zu den massenspektrometrisch beobachteten
Aminosäuren gehört, kann keine Aussage getroffen werden, ob dieser Methioninrest eine
Bindungsstelle darstellt.
Aus beiden Analysen aus dem Protein und dem ebenfalls fünffachen Überschuss an Cisplatin
(3h und 168h Inkubation bei 37°C) konnte T457 als Bindungsstelle des Metallkomplexes
identifiziert werden (s. Tab. 6.17, 6.19). Diese Koordination konnte wie bereits erwähnt
DYSON in seinen LC/MS/MS-Studien feststellen [137]. Dabei wurde ein Protein:CisplatinVerhältnis von 1:10 und Inkubationszeiten von 15 und 30 min bei 37°C verwendet.
Serumproteine
181
Zur Veranschaulichung der Lokalisierung der von einem Cisplatinfragment koordinierten
Seitenketten im Serotransferrin findet sich in der folgenden Darstellung die Gesamtstruktur.
E385
M256
T457
Y314
E265
Abb.6.40: Proteinstruktur des Serotransferrins mit bevorzugten Cisplatinbindungsstellen
In der Abbildung sind die aus den einzelnen Analysen erhaltenen Bindungsstellen des
Cisplatins in Serotransferrin hervorgehoben. Daraus ersichtlich ist Verteilung der
Aminosäuren in den beiden Bereichen des Serumproteins. Aus dem N-lobe (V1-T336)
wurden M256, E265 und Y314 und die restlichen zwei Seitenketten (E385, T457) aus dem Clobe (N337-P679) identifiziert. Von den fünf Koordinationen sind vier kinetisch bevorzugte
Stellen (E265, Y314, E385 und T457), was mit den Befunden aus den Analysen mit dem
Serumalbumin (z. B. Y150, D375) übereinstimmt. Wie schon DYSON [59] mittels UV/Visspektroskopischer Arbeiten zeigen konnte, konkurriert Cisplatin mit Fe(III) um Bindungen im
Serotransferrin. Die hier ermittelten Bindungsstellen bestätigen diese Annahmen, wie die
nachstehende Abbildung zeigt.
Serumproteine
182
E385
Y314
Y95
H249
Y188
D63
Abb.6.41: Ausschnitt aus der Proteinstruktur des Serotransferrins mit vier Fe(III)Bindungsstellen
Aus der Abbildung wird die räumliche Nähe der Seitenketten besonders deutlich. Zur
besseren Unterscheidung der Aminosäuren wurden die Fe(III)-Bindungsstellen lilafarben und
die zwei Cisplatinbindungsstellen gelb dargestellt.
Vergleichbare Nähe liegt bei den Fe(III) Bindungsstellen des C-lobes und einer weiteren
Koordinationsstelle für Cisplatin T457. Diese Aminosäure ist als Bicarbonat-Bindungsstelle
bekannt [147].
Proteintargets in humanem und bakteriellem Thioredoxin
183
7. Proteintargets in humanem und bakteriellem Thioredoxin
In einer weiteren Teilstudie wurden im Rahmen dieser Arbeit die Bindungseigenschaften des
Zytostatikums Cisplatin mit Thioredoxin untersucht. Thioredoxine gehören allgemein zur
Familie kleiner Enzymproteine und wirken als Redoxintermediate bei der Umwandlung von
Ribose zur Desoxyribose zur DNA-Synthese [152]. Das aktive Zentrum des Proteins besteht
häufig aus der Sequenz Cys-Gly-Pro-Cys. Die zum Thioredoxin-System (siehe Abbildung
7.1) gehörende Reduktase ist in vielen physiologischen Prozessen im Organismus und
dementsprechend auch im Zellwachstum (auch von Tumorzellen) involviert. Zu dem Enzym
wurden aus mehreren Studien Hinweise darauf erhalten, dass antitumorwirksame Gold (I)und Gold (III)-Komplexe die Reduktase hemmen [153, 154].
TR
Trx-(SH)2 + XS2
TR
Trx-S2 + NADPH
Trx-S2 + X(SH)2
Trx-(SH)2 + NADP+
Abb.7.1: Intrazelluläres Disulfid-Reduktionssystem [152]
Thioredoxin wird in vielen menschlichen Tumorzellen überexprimiert. Die Redoxaktivität des
Proteins ist entscheidend für die proliferationssteigernde Wirkung. Des Weiteren werden die
apoptosehemmende Wirkung und die mögliche Beteiligung an einer Resistenzentwicklung
der Zellen gegenüber einigen Antitumormittel [155, 161] im Zusammenhang mit Thioredoxin
diskutiert. Dabei konnte eine Cisplatin-Resistenz bei humanen Thioredoxin nicht jedoch bei
Thioredoxin aus E. coli beobachtet werden [126, 127].
Proteintargets in humanem und bakteriellem Thioredoxin
184
Bei der Analyse mit dem Darmbakterium E. coli konnte die Koordination eines
Cisplatinfragmentes an eine Peptidsequenz des Thioredoxins identifiziert werden. Es wurde
hierbei eindeutig die Seitenkette des Histidins 7 modifiziert. Wie in Kapitel 3 diskutiert,
diente das Bakterium bei den Umsetzungen als Modellsystem für Vorgänge in anderen
Organismen.
So stellte sich bei dieser Beobachtung die Frage, ob das humane Thioredoxin Ähnlichkeiten
im Reaktionsverhalten gegenüber Cisplatin aufweist. Für den direkten Vergleich sind im
Folgenden die Proteinsequenzen des humanen und des aus E. coli stammenden Thioredoxins
dargestellt.
Tab.7.1: Aminosäure-Sequenzen der Thioredoxine /E. coli und human
Thioredoxin aus E. coli (109 Aminosäuren)
MSDKIIHLTDDSFDTDVLKADGAILVDFWAEWCGPCKMIAPILDEIADEYQGKLTVA
KLNIDQNPGTAPKYGIRGIPTLLLFKNGEVAATKVGALSKGQLKEFLDANLA
Humanes Thioredoxin (105 Aminosäuren)
MVKQIESKTAFQEALDAAGDKLVVVDFSATWCGPCKMIKPFFHSLSEKYSNVIFLEV
DVDDCQDVASECEVKCMPTFQFFKKGQKVGEFSGANKEKLEATINELV
Die Tabelle 7.1 enthält die Proteinsequenzen der beiden Thioredoxine. Das oben erwähnte
aktive Zentrum des jeweiligen Proteins ist rot unterlegt. Beim Thioredoxin aus E. coli sind es
die Aminosäuren C33-C36 und beim humanen Thioredoxin C32-C35. Des Weiteren wurden
in den beiden Sequenzen die einzigen Histidinstellen eingerahmt bzw. bei E. coli, da das H7
die Bindungsstelle für Cisplatin darstellt, grün gekennzeichnet. Es fällt auch, dass humanes
Thioredoxin kein Histidin in der Nähe des N-terminalen Methionins enthält. Aus dem
Vergleich der oben dargestellten Proteinsequenzen ist ersichtlich, dass in dem humanen
Thioredoxin neben den Cysteinen im katalytischen Zentrum (C32 und C35) noch drei weitere
Cysteine enthalten sind (C62, C69, C73), die im bakteriellen Thioredoxin fehlen.
Für weitere Vergleiche wurden mit beiden Proteinen jeweils folgende Umsetzungen
durchgeführt: äquimolare Ansätze aus Thioredoxin und Cisplatin mit 3h Inkubation bei 37°C,
sowie Ansätze im 1:5-Verhältnis jeweils nach 3h und nach 168h bei 37°C. Die bei pH = 7,8
(NH4HCO3-Puffer)
inkubierten
Protein-Metallkomplex-Gemische
wurden
nach
dem
Entfernen nicht umgesetzter Cisplatinreste (3kDa cut-off-Filter) mit Trypsin in Peptide
Proteintargets in humanem und bakteriellem Thioredoxin
185
gespalten. Die eingeengte Lösung wurde auf eine RP-C18-Säule geladen und über LC/ESIMS/MS analysiert.
7.1. Proteintargets in humanem Thioredoxin
Eine Auflistung der koordinierten Peptidsequenzen im humanen Thioredoxin ist in der
nachstehenden Tabelle gezeigt. Für die Bindungsstellen wurden in allen folgenden Tabellen
die zum Teil zusätzlich zur bevorzugten Koordinationsstelle Aminosäuren angegeben, deren
theoretische MS/MS-Spektren ebenfalls gute Übereinstimmungen mit den experimentellen
Spektren ergeben (Differenzen bei den ∆Cn-Werten < 0,1, Sp > 500, RSp <6).
Tab.7.2: Koordinierte Peptidsequenzen aus humanem Thioredoxin mit Cisplatin
Peptid
Bindungsstelle z XCorr ∆Cn Sp RSp Ionen
Thioredoxin:Cisplatin, 1:1, 3h/37°C
E.K§LEATINELV.K96
2 3,38 0,48 1506,5 1 17/18
A.FQE§ALDAAGDK.L
E13
2 3,29 0,57 1181,8 1 16/20
K.LVVVDFSATWC~GP.C
C32
2 3,37 0,60 1250,0 1 16/24
K.TAFQEALDAAGDKLVVVDFSATWC~GP.C
C32/T30
2 3,15 0,37 1223,0 1 23/50
K.LVVVDFSATWC#GP.C
C32
2 2,87 0,29 720,0
1 13/24
Thioredoxin:Cisplatin, 1:5, 3h/37°C
K.LVVVDFSATWC~GP.C
C32
2
2,48
0,50 1300,2
1
16/24
Thioredoxin:Cisplatin, 1:5, 168h/37°C
A.FQE§ALDAAGDK.L
E13
2 3,22 0,49 1081,6
K.LVVVDFSATWC~GP.C
C32
2 2,47 0,35 1614,2
2+
2+
2+
§ = [Pt] -, $ = [(NH3)Pt] -, ~ = [(NH3)2Pt] -, # = [(NH3)2PtCl]+-Fragment
1
1
16/20
17/24
Die Tabelle 7.2 führt insgesamt acht mit einem Cisplatinfragment modifizierte
Peptidsequenzen aus drei Analysen auf, die aus unterschiedlichen Protein:MetallkomplexVerhältnissen (1:1, 1:5) und mit variierten Inkubationszeiten (3h, 168h) entstanden sind. Aus
der Liste lässt sich vermuten, dass die Seitenketten des Cystein 32 eine spezifische
Bindungsstelle für Cisplatin bietet, da sie aus allen drei Analysen identifiziert werden konnte.
Daneben wurde in zwei Analysen (1:1, 3h/37°C und 1:5, 168h/37°C) die Seitenkette der
Glutaminsäure E13 und einmal bei dem 1:1-Ansatz, 3h/37°C des Lysins 96 modifiziert. Zu
der Peptidsequenz [22LVVVDFSATWC#GP34]2+ aus der Tabelle 7.2 findet sich nachfolgend
das aufgenommene MS/MS-Spektrum.
Proteintargets in humanem und bakteriellem Thioredoxin
186
Abb.7.2: MS/MS-Spketrum von [LVVVDFSATWC#GP]2+ aus dem 1:1-Ansatz von
humanem Thioredoxin mit Cisplatin nach 3h Inkubation bei 37°C
In dem oberen Spektrum sind von 24 möglichen b+- und y+-Ionen 11 zugeordnet worden,
wobei SEQUEST 13 einfach geladene Ionen einteilt. Wie aus der Bindungsposition C32 in
der Peptidsequenz zu erkennen ist, sind die zugeordneten Ionen y6+-y11+ alle platiniert. Die
folgende Tabelle listet die SEQUEST-Werte für mögliche Koordinationen der Aminosäuren
T30 und S28 aus der Peptidsequenz LVVVDFSATWCGP.
Tab.7.3: SEQUEST-Parameter von [LVVVDFSATWC#GP]2+ aus dem 1:1-Ansatz von
humanem Thioredoxin mit Cisplatin nach 3h Inkubation bei 37°C
Sequenz
XCorr
K.LVVVDFSATWC#GP.C 2,87
K.LVVVDFSAT#WCGP.C 2,398
K.LVVVDFS#ATWCGP.C 2,063
∆Cn
0
0,165
0,287
Sp
720
498,7
297
RSp
1
2
6
Ionen
Protein
13/24 THIO_HUMAN
11/24 THIO_HUMAN
9/24 THIO_HUMAN
Sowohl die Sp- als auch die ∆Cn-Werte sprechen für das Fehlen einer guten
Übereinstimmung der theoretischen Spektren für eine Koordination des T30 oder des S28.
Auch die Ionenzuordnung und die Zuordnung der Ionen b9+ und y6+ in dem MS/MS-Spektrum
Proteintargets in humanem und bakteriellem Thioredoxin
187
in Abb. 7.2 sprechen für eine eindeutige Zuordnung von C32 als Bindungsstelle für Cisplatin.
Diese Vermutung lässt sich durch die Tatsache bekräftigen, dass in allen drei Analysen der
Thioredoxin-Cisplatin-Gemische dieser Cysteinrest als Cisplatinbindungsstelle identifiziert
wird (siehe Tabelle 7.1). Aus zwei der Untersuchungen (1:1 nach 3h und 1:5 nach 168h
Inkubation bei 37°C) konnte außerdem E13 als mögliche Koordinationsstelle für Cisplatin
bestimmt werden. Diese beiden Bindungsstellen sind in der Gesamtstruktur des Proteins mit
ihren Seitenketten in der Abbildung 7.3 gezeigt.
C32
C35
M38
E13
H43
Abb.7.3: Gesamtstruktur des humanen Thioredoxins mit E13, C32, C35 und H43 (PDB ID
1ert [166])
In der Struktur wurden neben den Cysteinen C32 und C35 sowie des E13 die Seitenketten des
des Methionins 38 und des Histidins H43 in dem humanen Protein gekennzeichnet. In keiner
der drei Untersuchungen konnte das S-Atom des Methionins bzw. der Imidazolring dieser
Histidinseitenkette als Bindungsstelle für Cisplatin ermittelt werden.
Proteintargets in humanem und bakteriellem Thioredoxin
188
7.2. Proteintargets in Thioredoxin E. Coli
Für einen Vergleich der Bindungsstellen im bakteriellen Thioredoxin und die Validierung der
erhaltenen Modifikation des H7 aus der MudPIT-Analyse des E. coli-Bakteriums mit
Cisplatin (Kapitel 5.3) wurden Untersuchungen von Ansätzen aus dem reinen Protein und
Cisplatin aufgenommen. Es wurden auch in diesem Fall vor der jeweiligen LC/ESI-MS/MSAnalyse die Protein:Metallkomplex-Verhältnisse (1:1, 1:5) und Inkubationszeiten (3h, 168h)
der Gemische variiert.
In der folgenden Tabelle sind die koordinierten Peptidsequenzen aus dem äquimolaren Ansatz
Thioredoxin (E. coli):Cisplatin dargestellt.
Tab.7.4: Koordinierte Peptidsequenzen aus dem 1:1-Ansatz Thioredoxin E. coli:Cisplatin
nach 3h Inkubation bei 37°C
Bindungsstelle MH+
M38
2000,06
z XCorr ∆Cn
Sp
RSp Ionen
2 5,75 0,74 2263,9 1 21/30
K.M$IAPILDEIADEYQGK.L
K.IIH$LTDDSFDTDVLK.A
P.CK$MIAPILDEIADEYQGK.L
I.APILD$EIADEYQGK.L
M38
H7
K37/C36/M38
D44/E45
2017,06
1942,91
2248,37
1772,7
2
2
2
2
6,09
3,85
3,09
2,56
0,68 1988,2
0,58 989,8
0,51 850,4
0,51 507,6
1
1
1
1
25/30
17/28
17/34
11/26
I.H~LTDDSFDTDVLK.A
K.LNIDQNPGT~A.P
K.M~IAPILDEIADEYQGK.L
H7
T67
M38
1733,60
1270,11
2034,06
2
2
2
5,40
3,77
3,76
0,73 2250,0
0,30 1206,7
0,82 1285,3
1
1
1
23/24
15/18
21/30
S.D#KIIHLTDDSFDTDVLK.A
I.H#LTDDSFDTDVLK.A
K.IIHLTDDSFDTD#V.L
D3/K4
H7/T9
D16/T15
2239,17
1769,60
1754,58
2
2
2
3,86
3,53
2,52
0,71 1563,4
0,49 690,5
0,23 758,8
1
1
1
21/32
14/24
14/24
Peptid
K.M§IAPILDEIADEYQGK.L
§ = [Pt]2+-, $ = [(NH3)Pt]2+-, ~ = [(NH3)2Pt]2+-, # = [(NH3)2PtCl]+-Fragment
Die oberhalb gezeigt Tabelle listet die Peptidsequenzen des Proteins mit den jeweiligen
Cisplatinkoordinationen auf. Mit einem der Metallkomplexfragmente wurden D3/K4/H7,
T15/D16, C36/K37/M38, D44/E45 und T67 modifiziert. Neben den kinetisch bevorzugten
Bindungsstellen nach der kurzen Inkubationszeit für Cisplatin (D, T und M) konnte auch das
thermodynamisch begünstigte H7 als Bindungsstelle in Übereinstimmung mit der MudPIT-E.
coli-Analyse bestätigt werden.
Eine weitere Untersuchung erfolgte mit dem Gemisch aus einem Protein:Cisplatin-Ansatz, in
dem ein fünffacher Metallkomplex-Überschuss eingesetzt wurde.
Proteintargets in humanem und bakteriellem Thioredoxin
189
Tab.7.5: Koordinierte Peptidsequenzen aus dem 1:5-Ansatz Thioredoxin E. coli:Cisplatin
nach 3h Inkubation bei 37°C
Peptid
K.M§IAPILDEIADEYQGK.L
D.KIIH§LTDDSFDTDVLK.A
K.M$IAPILDEIADEYQGK.L
Bindungsstelle z XCorr ∆Cn
Sp
RSp
M38
2 4,58
0,92 2448,90 1
H7/K4
2 3,7
0,82 1274,10 1
M38
I.H~LTDDSFDTDVLK.A
H7
P.CK~MIAPILDEIADEYQGK.L C36/K37/ M38
K.LNIDQNPGT~A.P
T67
M.IAPILDE~IADEYQGK.L
E45/D44
Ionen
23/30
21/30
2
6,07
0,7
2226,30
1
25/30
2
2
2
2
5,45
4,65
3,29
2,87
0,83 2277,70
0,83 1708,00
0,59 868,40
0,73 509,30
1
1
1
1
23/24
21/34
13/18
16/28
S.D#KIIHLTDDSFDTDVLK.A
D3/K4/H7
3 3,86
0,89 1393,90 1
S.DKIIH#LTDDSFDTDVLK.A
H7
3 3,32
0,78 736,70 3
K.IIHLTDDSFDTD#V.L
D16/T15
2 2,5
0,41 789,30 1
§ = [Pt]2+-, $ = [(NH3)Pt]2+-, ~ = [(NH3)2Pt]2+-, # = [(NH3)2PtCl]+-Fragment
28/64
22/64
15/24
Die Tabelle 7.5 listet die Peptidsequenzen des Thioredoxins (E. coli) aus der Analyse bei
einem Protein:Metallkomplex-Verhältnis von 1:5 nach 3h Inkubation bei 37°C auf. Die
Bindungsstellen D3/K4/H7, T15/D16, C36/K37/M38, D44/E45 und T67 aus Tabelle 7.4
können alle bestätigt werden. Nur D103 aus dem äquimolaren Ansatz kommt in der Liste des
1:5 Ansatzes nicht mehr vor. Wiederum ist H7 neben M38 und den O-Donor-Seitenketten
(D44/E45, D16, T67) als Bindungsstelle eindeutig identifiziert worden. Für eine weitere
Validierung der möglichen spezifischen Koordinationsstellen wurde die Inkubation des 1:5Ansatzes auf 168h verlängert.
Aus der Untersuchung des Protein-Metallkomplex-Gemisches (1:5) nach 168h Inkubation
sind die in Tabelle 7.6 aufgeführten Peptidsequenzen bestimmt worden.
Proteintargets in humanem und bakteriellem Thioredoxin
190
Tab.7.6: Koordinierte Peptidsequenzen aus dem 1:5-Ansatz Thioredoxin E. coli:Cisplatin
nach 168h Inkubation bei 37°C
Peptid
K.M§IAPILDEIADEYQGK.L
I.IH§LTDDSFDTDVLK.A
K.LNID§QNPGTAPK.Y
Bindungsstelle
M38
H7
D62
z XCorr ∆Cn
2 5,43 0,77
2 3,19 0,80
2 2,91 0,69
Sp
1976,0
825,6
524,5
RSp Ionen
1
26/30
1
17/26
1
12/22
K.M$IAPILDEIADEYQGK.L
P.CK$MIAPILDEIADEYQGK.L
L.LLFK$NGEVAATK.V
D.K$IIHLTDDSFDTDVLK.A
M38/D44/E45
C36/K37/ M38
K83
K4
3
2
2
2
5,71
3,56
3,30
2,86
0,74
0,72
0,73
0,59
2736,6
534,0
933,8
736,3
1
2
2
1
35/60
16/34
17/22
15/30
K.MIAPILD~EIADEYQGK.L
K.MoxIAPILDEIADEYQGK~LTV.A
D44/E45
K53/T55
3
2
4,50
3,69
0,78
0,70
1116,7
697,6
2
1
25/60
16/36
I.H#LTDDSFDTDVLK.A
H7
2 4,38 0,61 2859,4
1
M.IAPILD#EIADEYQGK.L
D44/E45
2 3,95 0,71 1237,0
2
K.IIHLTDDSFDTD#V.L
D16
2 2,79 0,43 854,5
1
§ = [Pt]2+-, $ = [(NH3)Pt]2+-, ~ = [(NH3)2Pt]2+-, # = [(NH3)2PtCl]+-Fragment
22/24
17/28
14/24
Auch die Ergebnisse aus dieser Tabelle bestätigen beinahe alle bisher identifizierten
Bindungsstellen des Cisplatins in dem Thioredoxin. Allerdings konnte T67 als Bindungsstelle
nicht zum dritten Mal bestätigt werden. Mit dem [(NH3)2Pt]2+-Fragment wurde zum ersten
Mal die Koordination der Seitenketten K53 (T55), K83 und mit dem [Pt]2+-Fragment die der
Aminosäure D62 nach der langen Inkubationszeit detektiert.
Zu der aus allen drei zuvor beschriebenen Analysen identifizierten Peptidsequenz
[38MIAPILD$EIADEYQGK53]3+ mit der Cisplatinbindungsstelle D44 ist in der unteren
Abbildung das MS/MS-Spektrum aus der Analyse Thioredoxin:Cisplatin, 1:5 nach 3h
Inkubation gezeigt.
Proteintargets in humanem und bakteriellem Thioredoxin
191
Abb.7.4: MS/MS-Spektrum von [MIAPILD$EIADEYQGK]3+ aus dem 1:5-Ansatz von
Thioredoxin E. coli: Cisplatin nach 3h Inkubation bei 37°C
In dem MS/MS-Spektrum oberhalb konnten 24 Ionen zugeordnet werden, wobei SEQUEST
32 einfach- und zweifach geladene Ionen einbezieht. Die Ionen b7+-b10+ sind der
Bindungsstelle D44 entsprechend platiniert. Diese Verteilung ist für keines der dargestellten
y+-Ionen zu sehen, da keine yk+-Ionen für k > 9 im Spektrum vorhanden sind.
Die folgende Tabelle zeigt weitere mögliche Peptidsequenzen des E. coli Thioredoxins mit
möglichen Cisplatinkoordinationen und der Masse m/z = 2017 (mit ∆m ± 2,5).
Proteintargets in humanem und bakteriellem Thioredoxin
192
Tab.7.7: SEQUEST-Parameter von [MIAPILD$EIADEYQGK]3+ und einer weiteren Peptidsequenz aus dem 1:5-Ansatz von Thioredoxin E. coli:Cisplatin nach 3h Inkuabtion bei 37°C
Sequenz
XCorr
K.MIAPILD$EIADEYQGK.L 5,745
K.M$IAPILDEIADEYQGK.L 5,605
K.MIAPILDE$IADEYQGK.L 5,165
K.MIAPILDEIAD$EYQGK.L 2,656
K.MIAPILDEIADE$YQGK.L 2,473
K.MIAPILDEIADEY$QGK.L 1,902
K.AD$GAILVDFWAEWC$.G 1,798
K.AD$GAILVDFWAE$WC.G 1,516
K.ADGAILVD$FWAEWC$.G 1,508
K.MIAPILDEIADEYQGK$.L 1,452
∆Cn
0
0,024
0,101
0,538
0,57
0,669
0,687
0,736
0,738
0,747
Sp
2020,6
2750,3
1540,7
403,1
341,7
264
141,5
127,3
79,6
175,1
RSp
2
1
3
4
5
6
8
9
11
7
Ionen
32/60
35/60
29/60
20/60
19/60
18/60
13/52
12/52
10/52
15/60
Protein
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
THIO_ECOLI
Aus der Auflistung werden die ersten drei Peptidsequenzen mit den potentiellen
Bindungsstellen D44, M38 und E45 betrachtet. Die Sp- und ∆Cn- Werte aller restlichen
Sequenzen erfüllen die Kriterien nicht (Sp < 500, ∆Cn > 0,1) und können als koordinierte
Sequenzen ausgeschlossen werden. Für die Bindung des Cisplatinfragmentes an D44 oder
M38 kann allein aus den SEQUEST-Parametern keine Entscheidung zu Gunsten einer dieser
Stellen gefällt werden (für beide gilt ∆Cn < 0,1). Die im MS/MS-Spektrum in Abbildung 7.4
dargestellten Massenpeaks der Ionen b3+ und b5+ wurden mit geringen Intensitäten detektiert
und können deswegen nur unzureichend auf eine mögliche Platinverteilung geprüft werden.
Bei einer M38-Koordination wären diese b+-Ionen nicht zu beobachten.
Die vergrößerte Darstellung des Ausschnittes aus der Proteinstruktur ist in Abbildung 7.5 zu
sehen.
M38
D44
Abb.7.5: Ausschnitt aus der Proteinstruktur von Thioredoxin E. coli mit M38 und D44
Proteintargets in humanem und bakteriellem Thioredoxin
193
Aus der oberen Darstellung kann kein eindeutiger Hinweis dafür erhalten werden, welche
Seitenkette für eine Koordination begünstigt wäre, so dass beide Stellen M38 und D44 als
mögliche Bindungsstellen für Cisplatin in Frage kommen. Allerdings wird M38 in allen
Analysen als Koordinationsstelle mehrfach identifiziert.
Aus den drei Analysen des einzelnen Proteins mit Cisplatin und der MudPIT-Analyse der E.
coli-Zellen mit dem Zytostatikum konnten somit einige möglicherweise spezifische
Bindungsstellen ermittelt werden. Dabei handelt es sich um H7, D16 (T15), M38 (C36, K37),
D44 und E45.
Die Lokalisierung der Aminosäuren im Protein ist in der folgenden Abbildung
gekennzeichnet.
C33
C36
M38
H7
D16
D44
Abb.7.6: Gesamtstruktur des Thioredoxins (E. coli) mit H7, D16, K37, M38 und D44
In der oberen Darstellung wurden neben den genannten Cisplatinbindungsstellen die
Seitenketten der Cysteine des aktiven Zentrums des Proteins C33 und C36 gekennzeichnet.
Wie aus den Tabellen 7.4-7.6 zu entnehmen, ist eine Koordination des Cisplatins an C33 nicht
beobachtet
und
ist
an
C36
unwahrscheinlich.
Insgesamt
konnte
bei
den
massenspektrometrischen Analysen etwa 50% der Peptidsequenzen detektiert werden und
C36 in nicht koordinierten Proteintargets beobachtet werden.
Proteintargets in humanem und bakteriellem Thioredoxin
194
7.3. Vergleich von humanem und E. coli Thioredoxin
Wie oben bereits erwähnt, wird das Thioredoxin durch einige Metallkomplexe in seiner
Wirkung gehemmt. Eine Bindung an das C32 und das benachbarte D26 wird für die
hemmende Wirkung einer Cadmiumverbindung verantwortlich gemacht [156]. MALDI-ToF
MS-Studien („top-down“-Methode) mit Thioredoxin aus Thermus thermophilus HB8 haben
gezeigt, dass das [Pt(bpy)]2+-Fragment mit den Cysteinseitenketten C31 und C34 des aktiven
Zentrums in dem Protein eine Chelatbildung eingeht. Aus dieser Blockierung des aktiven
Zentrums wird die Cisplatinresistenz in den Zellen, die Thioredoxin überexprimieren (z. B.
Kolonkarzinome [157]), vermutet.
Ein Vergleich der oben beschriebenen Analysen (Tab 7.2 und 7.4-7.6) des humanen
Thioredoxins und aus E. coli mit Cisplatin zeigt, dass obwohl die Proteinsequenz des
humanen Thioredoxins Histidin enthält, keine Hinweise auf eine Cisplatinbindung an H43
erhalten werden konnten. Obwohl in beiden Proteinen das Cystein C32 (bei E. coli)/C33
(human) in der Schleife vor der Helix enthalten, wurde es nur im humanen Thioredoxin als
mögliche Cisplatinbindungsstelle ermittelt. Des Weiteren konnte im Gegensatz zum E. coli
Thioredoxin keine M38-Koordination im humanen Thioredoxin beobachtet werden. Zu
erwähnen ist in dieser Hinsicht, dass diese Aminosäure trotz einer coverage-Rate von etwa
93% nur nicht koordiniert detektiert werden konnte. Diese Beobachtungen könnten die
Befunde zum Reaktionsverhalten des humanen bzw. E. coli Thioredoxins mit Cisplatin
bestätigen, welche von einer Inaktivierung des humanen Proteins durch eine Platinbindung an
eines oder durch Chelate an beide Cysteine ausgehen.
Proteintargets in Cyclooxygenase-2
195
8. Proteintargets in Cyclooxygenase-2
Cyclooxygenasen (COX) sind die wesentlichen Enzyme in der Prostaglandinsynthese und im
Inneren des endoplasmatischen Retikulums, innerhalb der Kernhülle und im Golgiapparat
lokalisiert. COX-2 kommt wird einer Reihe von Tumorzellen überexprimiert vor. Das durch
die COX-2 gebildete Prostaglandin-H2 stimuliert Entzündungsreaktionen und regt die Bildung
der Wachstumsfaktoren an, weswegen vermutet wird, dass COX-2 eine Rolle für das
Tumorwachstum spielen könnte [158]. Daher wird derzeit die präventive oder therapeutische
Wirkung von COX-2 Inhibitoren in der Krebsbehandlung insbesondere von Tumoren des
Magen-Darm-Traktes untersucht.
Zum Wirkmechanismus der nicht modifizierten Acetylsalicylsäure (ASS oder Aspirin®) ist
bekannt, dass die von ihr ausgehende Hemmung der Cyclooxygenasen (COX-1 und COX-2)
über eine Acetylierung des Serins 529 (bei COX-1) und des Serins 516 (bei COX-2)
stattfindet
[159].
In
einer
separaten
Umsetzung
des
reinen
Proteins
mit
dem
schmerzhemmenden Wirkstoff konnte die Acetylierung dieser Stelle nachgewiesen werden.
Diese Modifikation der COX-2 nach der Reaktion mit dem in der folgenden Abbildung
gezeigten Komplex ist in Tabelle 8.1 aufgeführt.
O
O
Co2(CO)6
O
O
Abb.8.1: Struktur des Komplexes Co-ASS
Wie in der Einleitung bereits erwähnt, zeigt die Modifikation der ASS zu einem
metallorganischen Derivat zusätzlich zu den bekannten Eigenschaften der ASS (wie
Reduktion der zellulären PGH2-Bildung durch die irreversible COX-1 und COX-2 Hemmung)
zytotoxische Wirkung. Bisher ist jedoch noch weitestgehend ungeklärt geblieben, wie dieser
Effekt des Co-ASS-Komplexes begründet ist. In der vorliegenden Arbeit wurden mittels LCESI
MS/MS
mögliche
Bindungsstellen
des
(Hexacarbonyl[2-propyn-1-ylacetylsalicylat]dicobalt)
potentiellen
(Co-ASS)
Antitumorkomplexes
untersucht,
um daraus
mögliche Hinweise auf eine Wechselwirkung zu erhalten. Dazu wurden die tryptisch
verdauten Reaktionslösungen ohne vorherige SCX-Extraktion über eine RP-C18-Säule
Proteintargets in Cyclooxygenase-2
196
getrennt und direkt ins gekoppelte Massenspektrometer injiziert. Die Datenbanksuche
beinhaltete die mögliche Acetylierung (Ac entspricht -CO-CH3) der Aminosäuren C, D, E, H,
K, M, R, S, T und Y mit der Masse m/z = 42, nach Abzug eines Protons für den
Ladungsausgleich. Außerdem wurde die mögliche Metallierung mit Cobaltionen sowie eine
ASS-Kopplung an den entsprechenden Seitenketten überprüft.
Die folgende Tabelle listet die Peptidsequenzen des Proteins auf, welche mit der Acetylierung
einer Aminosäurenseitenkette aus beiden Analysen (1:1 Ansatz mit ASS und mit Co-ASS)
identifiziert werden konnten.
Tab.8.1: Acetylierte Peptidsequenzen der COX-2 nach 3h Inkubation bei 37°C jeweils mit
ASS und mit Co-ASS
Peptid
Bindungsstelle MH+ z XCorr ∆Cn
Sp RSp Ionen
Acetylsalicylsäure
K.PRPDAIFGETMVEVGAPFS^LK.G
S516
2304,61 3 4,75 0,73 1275,9 1 32/80
Co-ASS-Komplex
K.QLPDSNEIVEK^LLLR.R
K166
1810,05 2 4,80 0,79 1284,7 1 21/28
L.PDSNEIVEK^LLLR.R
K166
1568,76 2 3,13 0.75 1253.7 1 17/24
K.LK^FDPELLFNK.Q
K346
1406,61 2 3,80 0,65 658,2
1 15/20
V.PPAVQK^VSQASIDQSR.Q
K432
1753,9 2 3,13 0,72 771,6
1 17/30
R.SGLDDINPTVLLK^ER.S
K598
1712,89 2 4,27 0,70 822,7
2 18/28
^ = Ac
COX-2 wurde für jeweils 3h bei 37°c mit ASS und Co-ASS inkubiert. Aus der Umsetzung
mit ASS konnte die Acetylierung der Serinseitenkette von S516 eindeutig bestätigt werden.
Daneben konnte keine weitere Acetylierungsstelle der ASS in der Cyclooxygenase-2 mit ∆m
± 2,5, XCorr ≥ 3,75 für dreifach, XCorr ≥ 2,5 für zweifach und einfach geladene Moleküle,
Sp ≥ 500, RSp ≤ 5, mindestens 30% Ionenzuordnung und ∆Cn < 0,1 beobachtet werden.
Die Analyse der Reaktion der COX-2 mit dem Metallkomplex Co-ASS ergab das Resultat,
dass Acetylierungen mehrerer Lysinseitenketten stattfinden. Wie in der Tabelle 8.1
dargestellt, wurden K166 (aus zwei unterschiedlichen Peptidsequenzen), K346, K432 und
K598 mit dem Acetylrest modifiziert. Eine Acetylierung von S516 wurde nach den obigen
SEQUEST-Kriterien nicht beobachtet, ebenso eine Metallierung oder Modifikation mit ASS.
Als Beispiel ist in der folgenden Abbildung das MS/MS-Spektrum der acetylierten
Peptidsequenz [156QLPDSNEIVEK^LLLR170]2+ gezeigt.
Proteintargets in Cyclooxygenase-2
197
Abb.8.2: MS/MS-Spektrum von [156QLPDSNEIVEK^LLLR170]2+ aus dem 1:1-Ansatz COX-2
mit Co-ASS nach 3h Inkubation bei 37°C
In dem obigen MS/MS-Spektrum konnten 18 von 28 möglichen b+- und y+-Ionen (21 von
SEQUEST eingeteilt) zugeordnet werden. Die Tabelle 8.2 zeigt Peptidsequenzen des Proteins,
welche ebenfalls acetyliert mit der Masse m/z = 1810 (∆m ± 2,5) vorkommen könnten.
Tab.8.2: SEQUEST-Parameter von [156QLPDSNEIVEK^LLLR170]2+ und einer weiteren
Peptidsequenz aus dem 1:1-Ansatz COX-2 mit Co-ASS nach 3h Inkubation bei 37°C
Sequenz
K.QLPDSNEIVEK^LLLR.R
K.QLPDSNEIVE^KLLLR.R
K.QLPDSNE^IVEKLLLR.R
K.QLPDSNEIVEKLLLR^.R
K.QLPDS^NEIVEKLLLR.R
K.QLPD^SNEIVEKLLLR.R
K.QLP^DSNEIVEKLLLR.R
K.EMSAE^LEALY^GDIDAV.E
K.EMSAELE^ALY^GDIDAV.E
XCorr
4,797
4,612
3,591
3,006
2,623
2,262
1,999
0,995
0,901
∆Cn
0
0,038
0,251
0,373
0,453
0,528
0,583
0,793
0,812
Sp
1284,7
1093,4
371
546
213
166,4
135,9
37,3
51,2
RSp
1
2
4
3
5
6
7
23
14
Ionen
21/28
20/28
15/28
14/28
12/28
11/28
10/28
6/30
7/30
Proteintargets in Cyclooxygenase-2
198
Von den neun in Tabelle 8.2 aufgeführten Peptidsequenzen würden nur die ersten beiden
möglichen Modifikationen die oben genannten Kriterien erfüllen. Für die restlichen Peptide
gilt Sp < 500 und/oder ∆Cn > 0,1. Die Zuordnung von y5+ spricht eindeutig für K166 las
Acetylierungsstelle.
Nach einer Inspektion der in Tabelle aufgeführten Acetylierungen des Proteins COX-2
können die vier Lysinseitenketten bezüglich ihrer Lokalisierung im Protein betrachtet werden.
Die Proteinstruktur mit den möglichen acetylierten Lysinstellen ist in der nachstehenden
Abbildung gezeigt.
K346
K598
S516
K166
Y371
K432
Abb.8.3: Proteinstruktur der Cyclooxygenase-2 mit K166, K346, K432 und K598 sowie Y371
und S516 (PDBID:1VOX [160])
In der Struktur sind die acetylierten Seitenketten von K166, K346, K432 und K598 sowie das
Aktive Zentrum (Y371) und die von ASS acetylierte Aminosäure S516 hervorgehoben. Die
Vermutung,
dass
der
Co-ASS-Komplex
Acetylierungen
der
Seitenketten
in
der
Cyclooxygenase-2 verursacht, konnte bekräftigt werden, indem eine LC-ESI-MS/MS-Analyse
Proteintargets in Cyclooxygenase-2
199
des reinen Proteins durchgeführt wurde. Die Prüfung der gespaltenen Peptidsequenzen auf
eine mögliche Acetylierung ergab keine Ergebnisse, welche die Sp-, ∆Cn-, XCorr, RSp-,
Ionenzuordnung oder ∆m-Kriterien erfüllen würde.
Aus den erhaltenen möglichen Acetylierungen der Cyclooxygenase-2 nimmt K346 eine
besonders interessante Rolle ein. Diese Aminosäure ist nämlich in der Umgebung des aktiven
Zentrums des Enzyms, welches bei Y371 liegt, positioniert. Aus den bekannten Befunden
bezüglich der Wirkwiese der Acetylsalicylsäure, dass die Acetylierung der Seitenkette S516
für die Wirkung verantwortlich ist, kann vermutet werden, dass die von Co-ASS ausgehende
Acetylierung der Lysinseitenketten ebenfalls einen Effekt auf die Enzymfunktionen auslösen
kann. Die beobachten zytotoxischen Eigenschaften des Co-ASS-Komplexes gegenüber den
Zelllinien MCF-7 und HCT-29 [31-33], die nicht von ASS ausgelöst werden, lassen
vermuten, dass die Modifikation der Acetylsalicylsäure mit dem organometallischen Rest eine
wichtige Rolle beim Wirkmechanismus spielt.
Zusammenfassung
200
9. Zusammenfassung
In der vorliegenden Arbeit wurden komplexe Proteingemische mittels kombinierter
flüssigchromatographischer und ESI-MS/MS Methoden auf mögliche Bindungsstellen von
Übergangsmetallkomplexen untersucht. Für die Bestimmung von Proteintargets in lebenden
Escherichia coli-Zellen und in humanen Blutserumproben wurde die MudPIT-Technologie
(Multidimensional Protein Identification Technology) eingesetzt. Dabei wurden folgende
Verbindungen
untersucht:
[(η6-p-Cymol)RuCl2(DMSO)],
[Pt(dien)(H2O)](NO3)2
und
Cisplatin (cis-[(NH3)2PtCl2]).
Aus den durchgeführten Voruntersuchungen mit Modellpeptiden und dem jeweiligen
Metallkomplex konnten sowohl an die chromatographisch als auch massenspektrometrisch zu
untersuchenden Systeme eine Reihe von Anforderungen gestellt werden, damit eine MudPITAnalyse des Gemisches erfolgreich verlaufen kann. Die gebildeten Protein- und
Peptidaddukte müssen eine hohe kinetische Stabilität über einen weiten pH-Bereich
aufweisen. Dabei liegt während des enzymatischen Verdaus ein leicht alkalisches Milieu bei
pH ≈ 7,8 vor, während der SCX-/RP-Trennung, welche bei pH ≈ 2,3 durchgeführt wird, ein
relativ niedriger pH-Wert vorkommt. Die Peptid-Metall-Bindung muss während des
Fragmentierungsvorgangs soweit intakt bleiben, dass eine zufrieden stellende Anzahl an
Fragmentionen (z. B. b+- und y+-Ionen) entstehen kann, die eine Identifizierung der
Peptidsequenz durch den Vergleich mit theoretischen MS/MS-Spektren ermöglicht. Dies
sollten im Allgemeinen mindestens 30% Ionenzuordung sein, auch im Beisein von neutral
loss Fragmentierungen der weiteren Liganden, z. B. Amminliganden bei cis-[(NH3)2PtCl2].
Ein charakteristisches Pattern des eingesetzten Metallzentrums kann bei der Zuordnung der
Ionen eine Hilfestellung bieten, was bei Ruthenium und Platin mit deren Isotopenverteilungen
gewährleistet ist. Vergleichsuntersuchungen an den vorhandenen Finnigan LCQ und LTQ
Massenspektrometern belegten, dass die höhere Scanzahl (ein MS/MS-Spektrum in zwei
Sekunden vs. drei MS/MS-Spektren pro Sekunde) des LTQs für eine effiziente
Adduktcharakterisierung essentiell ist.
Die Analyse der Produkte nach 3h Inkubation des Komplexes [(η6-p-Cymol)RuCl2(DMSO)]
mit dem Bakterium Escherichia coli (E. coli) bei 37°C ergab eine Liste von acht Proteinen,
welche rutheniert identifiziert werden konnten. Dabei handelt es sich um vier
stressregulierende Proteine ppiD, osmY, cspC und sucC, eine damage inducible Helikase
dinG und weitere drei Helikasen traI1, dbpA und hrpA. Die bevorzugten Bindungspositionen
Zusammenfassung
201
stellen dabei die Carboxylatgruppen der Aspartate, die Hydroxygruppen der Serin- und
Threoninreste sowie Aminostickstoffe aus Lysinseitenketten.
Abb.9.1: Proteinstruktur des cold shock Proteins cspA
Die beobachtete K26/D27-Koordination des cspC-Proteins ist besonders interessant, da durch
dieses cold shock Protein das gesamte SOS-System der Zelle beeinflusst wird. Zusätzlich
könnte die beobachtete Koordination der DNA-damage-inducible Helikase dinG einen
Eingriff in das Replikations- und Reparatursystem der Zelle bedeuten. Die Koordination
weiterer drei Helikasen traI1, dbpA und hrpA lässt die Vermutung zu, dass die Interaktion
von (η6-p-Cymol)Ru(II)-Komplexen mit den Helikasen für die antiproliferative Aktivität der
Verbindungen von Bedeutung ist.
Aus der Umsetzung der Modellverbindung [Pt(dien)(H2O)](NO3)2 mit dem Darmbakterium
E. coli nach 30 min Inkubation bei 37°C konnten sieben platinierte Peptidsequenzen mittels
MudPIT identifiziert werden. Es wurden die Proteine rl7, eftU, aldB, dnaK, ssb, rbsA und
yicC mit jeweils einer koordinierten Peptidsequenz gefunden. Die Bevorzugung der
Koordination von kinetisch begünstigten Seitenketten konnte auch hier beobachtet werden.
Die Carboxylatseitenketten der Glutamin- und Asparaginsäure stellen den Hauptteil der
koordinierten Aminosäuren dar. Es konnten allerdings auch zwei Koordinationen an das
weichere Schwefelatom in Methionin beobachtet werden.
Zusammenfassung
202
In einer weiteren Untersuchung wurde E. coli mit Cisplatin für 3h bei 37°C inkubiert und
durch eine MudPIT-Analyse auf koordinierte Peptidsequenzen geprüft. Es konnten insgesamt
31 Proteintargets ermittelt werden. Auch in dieser Analyse überwiegen die Koordinationen
der kinetisch attraktiven Carboxylatsauerstoffatome oder Hydroxygruppen. Außerdem
konnten neun Proteintargets mit Methionin-Bindungsstellen sowie drei thermodynamisch
bevorzugte Koordinationen an C- und H- Reste beobachtet werden.
Die aus dieser Analyse hervorgehende Identifizierung der DNA-bindenden Proteine mutS
(mismatch repair protein), uvrD (DNA-Helikase II) und top1 (Topoisomerase 1) weist auf
eine mögliche Beeinträchtigung des DNA-Reparatur-Systems unter Einfluss von Pt(II)Koordinationen hin. Die Ermittlung einer Vielzahl an diesen und anderen, wenig abundanten
Proteine in den Zellen (~ 102-103 Kopien pro Zelle) als spezifische Proteintargets für Cisplatin
in E. coli bedeutet bei einer Nachweisgrenze von etwa 10 fmol, dass etwa 10% oder mehr der
Kopien dieser Proteine von Cisplatin koordiniert vorliegen müssen. Im abundanten
Redoxprotein Thioredoxin (103-104 Kopien pro Zelle), einem in vielen Krebszellen
überexprimierten Protein, konnte die Koordinationsstelle H7 aus der Analyse des Bakteriums
sowie in Einzeluntersuchungen des Proteins mit Cisplatin als spezifische Bindungsstelle
eindeutig bestätigt werden. Im Gegensatz zum humanen Thioredoxin wird auch der
Methioninrest M38 in E. coli Thioredoxin, jedoch nicht wie bei der humanen Version eine der
benachbarten Cysteinstellen (C33/C36) des aktiven Zentrums als Koordinationsstelle
identifiziert.
C33
C32
M38
H7
M38
E13
Abb.9.2: Proteinstrukturen des humanen (links) und des E. coli (rechts) Thioredoxins mit
bevorzugten Cisplatinbindungsstellen
Zusammenfassung
203
Auch aus den Analysen der Blutserumproben und der Untersuchungen mit einzelnen
Serumproteinen und Cisplatin wird die Bevorzugung der Koordination der häufiger
vorkommenden Sauerstoff-Donoratome bei dem verwendeten pH ≈ 7 bekräftigt. Diese
Befunde decken sich mit aus den E. coli-Analysen erhaltenen Ergebnissen. Aus früheren
Studien waren für Serumalbumin C34, sowie 2-4 Methioninreste als Cisplatinbindungsstellen
ermittelt worden. C34 sowie M329 und M548 konnten in den hier beschriebenen Analysen
der Blutserumproben und des Serumalbumins neben Y148/Y150 und D375/E376 ermittelt
werden. Die kinetisch bevorzugte Koordination durch O-Donorseitenketten könnte wegen
ihrer hohen Bildungsgeschwindigkeit für die Transportfunktion von pharmakologisch
relevanten Metallkomplexen des Proteins zu Tumorzellen eine bedeutende Rolle spielen. κOkoordinierte Cisplatinfragmente können anschließend wegen der schwächeren Pt-OBindungen zu thermodynamisch bevorzugten weicheren Bindungsstellen, wie den
Nukleobasen der DNA, wandern. Im Gegenteil dazu verhindert die langsame Bildung von
κ2S,N-Chelaten (wie bei der C34 bzw. M329 oder M548 Koordination) die Migration der
Metallkomplexe.
Abb.9.3: Proteinstruktur des Serumalbumins und die ermittelten Cisplatinbindungsstellen
Zusammenfassung
204
Auch die charakteristischen κO-Bindungsstellen des Cisplatins im Serotransferrin (Y314,
E385 und T457) können neben M256 beim Transport des Antitumormittels direkt nach der
Verabreichung eine wichtige Rolle spielen. Zur Veranschaulichung ist untenstehend die
Proteinstruktur
des
abundanten
Serumproteins
Serotransferrin
mit
den
Cisplatinbindungsstellen gezeigt.
E385
M256
T457
Y314
E265
Abb.9.4: Struktur des Serotransferrins mit den ermittelten Cisplatinbindungsstellen
Der Befund, dass nur wenige Histidinreste von Cisplatin sowohl im E. coli-Bakterium als
auch im Blutserum koordiniert wurden, bestätigt einerseits die von SADLER durchgeführten
NMR-Studien [15, 17], in denen Histidin-Addukte als Bindungsstelle nicht bei
Serumproteinen Albumin und Serotransferrin nachgewiesen werden konnte. Andererseits ist
es jedoch auch möglich, dass Histidin keine adäquate Anzahl an b+- und y+-Fragmentionen
liefern und die koordinierten Peptidsequenzen entsprechend nur niedrige SEQUESTParameter zugeteilt bekommen. Wie jedoch die Identifizierung der H7-Koordinationsstelle im
Thioredoxin des E. coli Baktriums zeigt, ist im Allgemeinen eine Histidinzuordnung auch aus
einer MudPIT-Analyse möglich.
Anhand der vorliegenden Untersuchungen lässt sich feststellen, dass die in dieser Arbeit
entwickelte LC2/ESI-MS2-Methode erstmals die Möglichkeit eröffnet, sowohl Proteintargets
Zusammenfassung
205
als auch die Bindungsstellen in diesen Targets komplexer Proteingemische zu bestimmen.
Hierbei muss allerdings immer der Anteil der durch ESI-MS/MS zu detektierenden
Sequenzen (coverage) berücksichtigt werden. Typische Werte liegen bei etwa 30-55%. In
Einzelfällen sind wesentlich höhere Werte möglich (z. B. bei humanem Thioredoxin 93%).
Die Identifizierung der gleichen Bindungsstellen aus unabhängigen Blutserumproben bzw. bei
Messwiederholungen für Protein:Cisplatin-Reaktionsgemische belegt die Reproduzierbarkeit
und somit die Zuverlässigkeit dieser Technik.
Experimenteller Teil
206
10. Experimenteller Teil
10.1. Ausgangssubstanzen
Peptide
Die Peptide FKVSGFLG, FKVGHFLG, GALTNVSMAK und IVDAQGNDVLIPG wurden
von der Firma PSL (Heidelberg), VSEHEAT von Genscript Corporation (USA) und AcSG,
AcM sowie AcH von BACHEM (Weil am Rhein) erworben.
Cisplatin wurde bei ChemPUR (Karlsruhe) erworben und wie erhalten eingesetzt.
Proteine/Protease
Die
Proteine
Alpha-1-Antitrypsin,
Alpha-2-Macroglobulin,
Cyclooxygenase-2,
Apolipoprotein A1, Serumalbumin, Thioredoxin (human), Thioredoxin (E. coli) und
Transferrin wurden bei Sigma (Heidelberg) erworben.
Trypsin wurde bei Promega (Mannheim) gekauft.
Metallkomplexe
1. [PtI(dien)]I wurde wie in der Literatur beschrieben [162] hergestellt.
1) Darstellung von PtI2·H2O: Es werden 2,5 g (6,02 mmol) K2[PtCl4] in 40 ml H2O unter
Rühren gelöst und bei 90°C werden 3,0g (18mmol) KI zugegeben. Nach 3,5h wird
über eine D3-Glasfritte der gebildete schwarze Niderschlag abfiltriert, mehrmals mit
H2O gewaschen und an der Luft getrocknet. Ausbeute: 2,2 g = 4,7 mmol (78%).
2) Darstellung von [PtI(dien)]I: 2,0 g (4,3 mmol) PtI2·H2O werden mit etwa 1 ml H2O
zu einer Suspension verrieben und dazu werden innerhalb von 1,5h 0,5 ml (4 mmol)
Diethylentriamin in 1 ml H2O hinzugetropft. Die Suspension wird zur Trockene
eingedampft und der Rückstand mit 90°C heißem H2O extrahiert. Nach dem
Abfiltrieren wird die gelbe Lösung im Eisbad gekühlt und die entstandenen gelben
Kristalle abgetrennt. Die mit eiskaltem H2O gewaschenen Kristalle können an der Luft
getrocknet werden. Ausbeute: 1,6 g = 2,9 mmol (67%). Die Reinheit wurde mittels IRSpektroskopie überprüft.
3) [Pt(dien)(H2O)](NO3)2 wurde aus [PtI(dien)]I durch Zugabe von zwei Äquivalenten
AgNO3 und 24 h im Dunkeln Rühren nach anschließender Zentrifugation (gebildetes
AgI bei 6°C) erhalten.
2. [(η6-p-Cymol)RuCl2(DMSO)] wurde nach den Vorschriften [163, 164] dargestellt.
Experimenteller Teil
207
1) Darstellung von [(η6-p-Cymol)RuCl2]2: 2 g (7,3 mmol) RuCl3·H2O (bei Chempur
erworben) wurden in 100 ml Ethanol gelöst und mit 13,6 g (0,1 mol) α-Phellandren
versetzt und 4h bei 80°C refluxiert. Nach dem Abfiltrieren und Waschen mit wenig
kalten Ethanol wurde [(η6-p-Cymol)RuCl2]2 erhalten. Ausbeute: 1,7 g = 2,8 mmol
(76%)
2) Darstellung von [(η6-p-Cymol)RuCl2(DMSO)]: 80 mg (0,13 mmol) [(η6-pCymol)RuCl2]2 wurden mit zwei Äquivalenten (20,5 mg; 0,2 mmol) DMSO in einer
1:1 Methanol/Methylendichlorid-Lösung über 4h bei 60°C refluxiert. Nach dem
Einengen und Abfiltrieren der rotbraun gefärbten Lösung wurden bräunliche Kristalle
erhalten.
Ausbeute:
70
mg
=
0,2
mmol
(70%).
Die
Reinheit
wurde
röntgenkristallographisch bestimmt [165].
3. Cisplatin wurde bei Chempur erworben und wie erhalten eingesetzt.
4. Hexacarbonyl[2-propyn-1-ylacetylsalicylat]dicobalt (Co-ASS) wurde von Dr. Ingo Ott (FU
Berlin) zur Verfügung gestellt und wie erhalten eingesetzt.
Lösungsmittel/Chemikalien
HPLC-grade
Methanol
wurde
bei
Baker,
Acetonitril,
Ammoniumacetat
und
Ammoniuhydrogencarbonat wurden bei Merck gekauft. Pentafluorpropionsäure (PFP) wurde
von ACROS erworben. Das bidestillierte Wasser wurde aus dem VE-Wasser der RuhrUniversität Bochum mit einer Anlage der Firma Büchi hergestellt.
10.2. Biologische Proben
Escherichia coli
50 µl von der E. coli BL21-Stamm Suspension (vom Lehrstuhl für Biochemie, Ruhr
Universität Bochum zur Verfügung gestellt) wurden mit 2,5 ml LB-Medium (lysogeny broth)
und 50 µl Chloramphenicol für 2-3h bei 37°C geschüttelt. Danach wurden weitere 48 ml des
LB-Mediums hinzugefügt und für weitere 12h bei 37°C geschüttelt. Nach dem
Zentrifungieren wurde ein Zellpellet erhalten, welches für die Umsetzungen mit den
Metallkomplexen verwendet wurde. Dabei besteht das LB-Medium aus bacto-Trypton, bactoHefeextrakt, NaCl und H2O (pH ≈ 7).
Experimenteller Teil
208
Blutserum
Humanes Blutserum wurde von zwei unterschiedlichen Personen nach einer Blutabnahme
durch Abtrennen (Zentrufugieren) von den festen Bestandteilen des Blutplasmas erhalten
(vom Berufsgenossenschaftlichen Universitätsklinikum Bergmansheil GmbH Bochum mit
Einwilligung der Personen zur Verfügung gestellt).
10.3. Massenspektrometrische Untersuchungen
10.3.1. ESI-MS und ESI-MS/MS Messungen an Modellpeptiden/Metallkomplex
Reaktionssystemen
Alle Modellpeptid:Metallkomplexansätze wurden in wässrigen Lösungen (50µM) im
Verhältnis 1:1 für 24h bei 37°C inkubiert und direkt massenspektrometrisch analysiert.
Die
ESI-MS
und
MS/MS
Untersuchungen
wurden
an
einem
Finnigan
LCQ
Massenspektrometer (Thermo Electron Corp., San Jose, CA, USA) durchgeführt. Dieses
wurde stets im positiven Modus bei einer Kapillartemperatur von 200°C, bei einer CIDEnergie zwischen 35-40% und einer Sprayspannung von 1,8 kV bedient. Die Flussraten
wurden zwischen 0,5-1,0 µl·min-1 variiert.
10.3.2. LC-ESI-MS/MS Messungen an Protein/Cisplatin Reaktionslösungen
Alle Protein:Cisplatin Ansätze wurden in NH4HCO3-Puffer (10mM, pH = 7,8) in 1:1, 1:3 oder
1:5 Verhältnissen (1-5 µM) für 3h (1:1, 1:3, 1:5) oder 168h (1:5) bei 37°C inkubiert.
Metallkomplexüberschüsse wurden durch Verwendung eines 3 kDa Ausschlussfilters
(Microcon, YM-3, Millipore) nach Herstellerangaben entfernt. Durch Zugabe von Trypsin
(1:50 w/w) erfolgte der enzymatische Verdau der jeweiligen Proben für 12h bei 37°C. Durch
Zugabe von 98% Ameisensäure wurde der Verdau abgestoppt (pH ≈ 2,3). Unverdaute
Proteine wurden durch Verwendung eines 10 kDa (Microcon, YM-10, Millipore)
Ausschlussfilters entfernt.
Eindimensionale LC-ESI
Für alle LC-ESI-Analysen wurden zwei Pufferlösungen verwendet: Puffer A (2% Acetonitril,
0,1% Ameisensäure) und Puffer B (80% Acetonitril, 0,1% Ameisensäure). Die nanoLC-ESIAnalysen
der
Peptid:Metallkomplex-Gemische
wurden
an
einem
Finnigan
LTQ
Massenspektrometer (Thermo Electron Corp., San Jose, CA, USA) durchgeführt. Zur
eindimensionalen Peptidtrennung wurde eine Kapillarsäule mit einem Innendurchmesser von
75 µm, gefüllt mit 10 cm RP-C18 Material (Eclipse XDB, Hewlett Packard), verwendet. Die
Experimenteller Teil
209
Peptidmischungen wurden unter Hochdruck auf die Kapillarsäule geladen und 5 min mit
Puffer A gespült. Die Elution erfolgte durch einen linearen Gradienten von 0 bis 55 % Puffer
B über 120 min und einen folgenden linearen Gradienten bis 100% Puffer B über 20 min. Die
angelegte Ionisierungsspannung betrug 1,8 kV bei einer Flussrate von 0,12 µl / min. Die
Kapillartemperatur betrug 200°C und die CID-Energie wurde zwischen 35-40% variiert. Die
MS/MS Spektren wurden unter Verwendung der Xcalibur-Software aufgenommen.
10.3.3. Multidimensionale-Protein-Identifizierungs-Technologie (MudPIT)
10.3.3.1 MudPIT E. coli
Das nach dem Zentrifugieren erhaltene Zellpellet konnte für eine Umsetzung mit dem
jeweiligen Metallkomplex verwendet werden. 500 µg des Zellpellets wurden mit 2mM cis[PtCl2(NH3)2] bzw. 10mM [Pt(dien)(H2O)](NO3)2 oder [(η6-p-Cymol)RuCl2(DMSO)] in 500
µl destilliertem Wasser 3h (30 min bei [Pt(dien)(H2O)](NO3)2) bei 37°C inkubiert. Nach der
Inkubation wurde mehrmals mit 10 mM NH4HCO3-Puffer gewaschen, um ungelöste Reste
abzutrennen. Mit Hilfe eines Douncers wurden die Zellen durch das Zerstören der Zellwände
geöffnet. Vor dem Verdau mit der Protease Trypsin wurde die Proteinkonzentration mit Hilfe
des Bradford-Assays bestimmt und die Lösung filtriert. Dafür wurde ein 3kDa
Ausschlussfilter (cut-off, Microcon, YM-3, Millipore) zum Abtrennen nicht gebundener
Metallkomplexe verwendet. Durch Zugabe von Trypsin (1:50 w/w, 2,4 µg) erfolgte der
enzymatische Verdau der jeweiligen Proben für 12h bei 37°C. Das Abstoppen des Verdaus
erfolgte durch Zugabe von 98% Ameisensäure (2-4µl, pH ≈ 2,3). Anschließend wurden mit
einem 10 kDa (Microcon, YM-10, Millipore) Ausschlussfilter nicht verdaute Proteine von der
Messlösung abgetrennt und die Lösung auf etwa 20 µl eingeengt. Die auf die Säule geladene
Protein-Metallkomplex-Mischung wurde unter Verwendung von 12 chromatographischen
Salzgradientenstufen massenspektrometrisch detektiert.
10.3.3.2. MudPIT Blutserum
Die Proteinkonzentration wurde unter Verwendung des Bardford-Assays bestimmt und betrug
jeweils etwa 70µg/µl Serum. Jeweils 1µl des Blutserums wurde mit einem zehnfachen
Überschuss an Cisplatin (20µM) in 500 µl NH4HCO3-Puffer (10mM, pH = 7,8) für 3h bei
37°C inkubiert. Nicht umgesetztes Cisplatin wurde vor dem Verdau aus der Mischung mit
einem
3kDa
(Microcon,
YM-3,
Millipore)
Ausschlussfilterfilter
abgetrennt.
Das
Protein:Cisplatin-Gemisch wurde für 12h bei 37°C mit der Protease Trypsin (1,4µg) verdaut.
Mit der Zugabe von 3µl 98%-iger Ameisensäure wurde der pH-Wert auf etwa 2,3 eingestellt.
Experimenteller Teil
210
Es folgte ein Abfiltrieren der nicht verdauten Proteine (10 kDa cut off Filter, Microcon, YM10, Millipore) und Einengen der Mischung auf etwa 20 µl. Für die MudPIT-Analyse wurden
die Mischungen auf die Säule geladen und unter Verwendung von 12 chromatographischen
Salzgradientenstufen massenspektrometrisch detektiert.
10.3.3.3. LC2/ESI-MS2 Messungen
Vier Pufferlösungen wurden bei allen Analysen verwendet: Puffer A (2% Acetonitril, 0,1%
Ameisensäure), Puffer B (80% Acetonitril, 0,1% Ameisensäure), Puffer C (250 mM
Ammoniumacetat, 0.1% Ameisensäure, Acetonitril) und Puffer D (1,5 M Ammoniumacetat,
0.1% Ameisensäure, Acetonitril).
Die Analysen der Proteingemische erfolgten durch Verwendung eines HPLC-Systems
bestehend aus einer Pumpe (Dual Gradient System, Dionex), einem automatischen
Probenapplikator (Famos) und einer Säulenschalteinheit (Switchos), die direkt mit einem
Ionenfallen-Massenspektrometer Finnigan LTQ (Thermo Electron Corp., San Jose, CA, USA)
verbunden war. Die Peptidmischung wurde zunächst auf eine dreiphasige Kapillarsäule (100
µm C18-RP-Materials (Eclipse XDB, Innendurchmesser) geladen, bestehend aus 12 cm eines
C18 Hewlett Packard), 4 cm eines starken Kationen-Austauschmaterials (SCX, Whatman) und
abschließend 4 cm des C18-RP-Materials. Die Äquilibrierung der Säule erfolgte durch Spülen
mit Puffer A. Einer 30 min Waschstufe mit Puffer A folgte zur Entfernung nicht an SCX
gebundener Peptide der lineare Gradient über 180 min bis 100% Puffer B. Die schrittweise
Elution der Peptide von der SCX-Säule erfolgte durch Erhöhen des Puffer C-Gehaltes in 10%Schritten und Puffer D mit 33, 50 und 75% für 2 min zwecks Ionenaustausch. Jeder Salzstufe
folgte eine 7 min Waschstufe mit Puffer A und über 70 min ein Gradient zuerst bis 50%
Puffer B, danach bis 100% über weitere 20 min. Nach einer 5min Stufe mit 100% Puffer B
schloss sich eine 20 min Stufe mit 100% Puffer D an. Die angelegte Ionisierungsspannung für
die ESI-MS/MS Messungen betrug 1,8 kV, die CID-Energie lag bei 35-40% und die
Flussraten zwischen 0,15-0,25 µl·min-1. Das LTQ wurde mittels der Xcalibur-Software
gesteuert, wobei MS-Spektren in einem Massenfenster von 350 – 2000 m/z aufgenommen
wurden und die drei intensivsten Peptid-Ionen sequenziell isoliert und in der Ionenfalle
fragmentiert wurden.
Experimenteller Teil
211
10.3.3.4. Auswertung der MS/MS Daten mit dem SEQUEST Algorithmus
Die Interpretation der MS/MS Spektren erfolgte mit Hilfe des SEQUEST Algorithmus. Die
generierten MS/MS Daten wurden zunächst in einem spezifischen Datenformat (BioworksProgramm) abgelesen und in Excel-Format überführt.
Als mögliche Modifikationen wurden die Oxidation von Methioninen (+16 Da) und die
jeweiligen Metallierungen verwendet. Es wurde die Protease Trypsin in der SEQUEST
Suchmaske mit der zusätzlichen Option auch halbtryptisch oder nicht tryptisch gespaltene
Peptide zu berücksichtigen angegeben.
Als Filterkriterien wurden folgende Faktoren verwendet:
1) XCorr (cross correlation) ist eine Funktion für die Kalkulation der Korrelation
zwischen zwei Signalserien mit Cτ = Σnj=1 IRj ITj+τ (τ ist die relative Verschiebung
zwischen dem theoretischen und dem experimentellen MS/MS-Spektrum im Bereich
-k < τ < k; R und T sind Fourier transformiert; IRj ist die gesamte Intensität im
Intervall j des experimentellen Spektrums; n ist die Anzahl der entsprechenden Peaks).
Die Verwendung eines Bereiches -k < τ < k ermöglicht eine bessere Diskriminierung
zwischen vergleichbaren Spektren. Die resultierenden XCorr-Werte werden weniger
durch Verunreinigungen beeinflusst.
2) Sp (preliminary scoring) verwendet die Stetigkeit der b+- und y+-Ionen mit
Sp = (Σnj=1)n (1+Cβ)IM/ητ (C ist die Anzahl der Peaks, die zusätzlich zum passenden
Peak auch den vorhergehenden Peak zugeordnet bekommen haben, wobei der Faktor
1+ Cβ das mit β = 0,075 berücksichtigt; ητ ist die Gesamtzahl der zu erwartenden
Peaks). IM berücksichtigt die Aminosäurereste, die Immonium-Ionen liefern können.
3) RSp (rank preliminary scoring) gibt den Rang der Sp-Werte an.
4) ∆Cn (delta correlation value) bezieht sich auf normalisierte XCorr-Werte, so dass der
höchste XCorr-Wert gleich 1 gesetzt wird. Bei ∆Cn wird der normalisierte XCorrWert für eine Sequenz von dem höchsten normalisierten XCorr-Wert abgezogen.
5) Ionen bezeichnen die Anzahl der zugeordneten Fragmentionen im Verhältnis zur
Anzahl der möglichen Fragmentionen.
6) ∆m ist die Differenz zwischen der experimentellen Vorläuferion-Masse und der
theoretischen Masse für eine Peptidsequenz. Für die eingesetzten Metallkomplexe
wurde jeweils ein ∆m ± 2,5 verwendet, um auch die Isotopenverteilung der
Metallatome zu berücksichtigen.
Experimenteller Teil
212
10.4. Chromatographische Untersuchungen
Für die chromatographischen Untersuchungen wurde die Umkehrphasen-IonenpaarHochdruck-Flüssigkeitschromatographie (IPRP-HPLC) eingesetzt. Als stationäre Phase kam
eine silicagelbasierte C18-Phase von der Firma Macherey-Nagel mit einer durchschnittlichen
Porengröße von 10nm und einer Korngröße von 5µm für die analytischen Trennungen zum
Einsatz. Die mit der NUCLEODUR-Pyramid-Phase gefüllte Säule weist, bezogen auf die
aggressiven Trennbedingungen (z. B. pH-Wert ≈ 2) eine gute Langzeitstabilität auf. Die
chromatographischen Säulen waren 25 cm lang bei einem Innendurchmesser von 4mm.
Für das untersuchte chromatographische System wurde ein Stufengradient verwendet, bei
dem einem Gemisch aus 4% Methanol und 96% Wasser nach 25 min ein Eluentengemisch
mit 12% Methanol und 88% Wasser mit jeweils 0,1% Pentafluorpropionsäure (PFP) als
Ionenpaarreagenz folgte. Die Wellenlänge betrug bei allen Messungen 220nm. Die
Temperatur war bei 25°C eingestellt und die Flussrate bei 0,8 ml·min-1.
Der Chromatograph für die analytischen Lösungen bestand aus einer LaChrom Anlage der
Firma Merck und setzte sich aus einer HPLC Gradientenpumpe L-6200A, einem UVDetektor L-4250 und einem Säulenthermostat L-7360 zusammen. Zur Injektion diente ein 6Wege-Ventil 7125 von Rheodyne mit einer 20µl Probenschleife. Die Aufnahme der
chromatographischen Daten wurde mit einem 20 Bit A/D-Wandler der Firma Knaur bei einer
Datensammelrate von 1Hz mit dem Eurochrom Integration Package V.1.5 durchgeführt.
Für die kinetische Untersuchung wurden je 0,8 µmol Cisplatin und Peptid eingewogen und in
10ml Wasser gelöst. Die Einstellung des pH-Wertes erfolgte über die Zugabe von 0,1 M
NaOH oder HNO3. Die Lösung wurde über die gesamte Reaktionsdauer bei 37°C inkubiert.
Bei den durchgeführten Konkurrenzuntersuchungen wurde wiederum je 0,8 µmol Peptid
hinzugefügt und die Lösung weiter bei 37°C inkubiert.
10.5. Kernresonanzspektroskopie
Die Aufnahme der NMR-Spektren erfolgte in der SC-Abteilung der Ruhr-Universität Bochum
an dem FT-NMR-Spektrometer DRX200 der Firma Bruker bei ν = 200,13 MHz. Als
Lösungsmittel diente in allen Fällen D2O und als Frequenzsstandard in D2O gelöstes Natrium3-(Trimethylsilylpropionat)-D4. Es wurden 5mm Probenröhrchen verwendet. Die Auswertung
der Spektren erfolgte mit dem Programm MestRe-C2.3a der Universidade de Santiago de
Compostela.
Experimenteller Teil
213
Die Reaktionslösungen für die zeitabhängigen 1H-NMR-Untersuchungen wurden durch
Einwaage von je 0,8 mmol Cisplatin und Peptid hergestellt, die in 1ml D2O gelöst werden.
Die Einstellung des pH*-Wertes erfolgt über Zugabe verdünnter NaOD und DNO3. Die
Angaben der pH*-Werte sind für den Isotopeneffekt des Deuteriums unkorrigiert (pH = pH*).
Anhang
214
11. Anhang
MudPIT-MS/MS-Spektren der Proteintargets aus E. coli (nicht in Kapitel 5 diskutiert)
1.Proteintargets von [(η6-p-Cymol)RuCl2(DMSO)] in E. coli (Tab. 5.1)
PPID_ECOLI
V.SD*AASNDTESLAGAEQAAGVK.A
DBPA_ECOLI
L.T*LCGGQPFGMoxQR.D
OSMY_ECOLI (a)
V.VT*LSGFVESQAQAEEAVK.V
OSMY_ECOLI (b)
V.VT*LSGFVESQAQAEEAVK.V
HRPA_ECOLI
A.QGAVMAT*EK.V
Anhang
215
2. Proteintargets von Pt(dien)]2+ in E. coli (Tab. 5.6)
ALDB_ECOLI
[email protected]
RBSA_ECOLI
[email protected]
YICC_ECOLI
[email protected]
DNAK_ECOLI
[email protected]
Anhang
216
3. Proteintargets von cis-[PtCl2(NH3)2] in E. coli (Tab. 5.10)
FINQ_ECOLI
K.MoxECID~NEVVVTVNEKLK.D
REP5_ECOLI
S.FISDILYADIE~SK.A
INTD_ECOLI
V.PNMS~HSEIMoxEDIKKA
DBPA_ECOLI
K.MoxRPGD~VLGALTGDIGL.D
DBPA_ECOLI
K.MoxRPGD#VLGALTGDIGLDGADIG.K
RS6_ECOLI
R.HYEIVFM§VHPDQSEQVPGMIER.Y
Anhang
CH60_ECOLI
A.M#LQDIATLTGGTVISEEIGMELEK.A
ACRD_ECOLI
R.KAQALGVAID#DINDTLQ.T
FIMA1_ECOLI
A.LSLS#STAALAAATTVNGGTVHFK.G
217
RPOA_ECOLI
.M§QGSVTEFLKPR.L
MDTA_ECOLI
R.QE§LDAQQALVSETEGTIKADEASV.A
CEAN_ECOLI
K.LIDAESIK#GTEVYTF.H
Anhang
218
ARTI_ECOLI
K.FIM§DKHPEITTVPYDSYQNAK.L
PTHP_ECOLI
.M§FQQEVTITAPNGLHTR.P
DCEA_ECOLI
F.EMDFAELLLEDY#K.A
MDH_ECOLI
K.SIGTLSAFEQNALEGMLDT~.L
G3P1_ECOLI
K.RSD§IEIVAINDLLDADYMAYMLK.Y
EBGC_ECOLI
V.HE§GQIVICDIHEAYR.F
Anhang
TDCE_ECOLI
K.T§APLMoxDDVLDYDKVMDSL.D
SPEA_ECOLI
R.MoxADKMYVNFS$LFQSMPD.A
219
HIS2_ECOLI
H.AVSGEVLMLGYM$NPEALDK.T
MUKE_ECOLI
R.STT#LIPRSVLSELDMMVG.K
Literaturverzeichnis
220
12. Literaturverzeichnis
[1] Parkin DM, Bray F, Ferlay J, Pisani P, CA:A Cancer Jour. for Clinic., 2005, 55, 2, 74
[2] Amtliche Todesursachenstatistik, Statistisches Bundesamt, Wiesbaden, 6. überarbeitete
Auflage, 2008, Krebs in Deutschland 2003 – 2004
[3] Rosenberg B, vanCamp L, Krigas T, Nature, 1965,698
[4] Rosenberg B, van Camp L, Trosko JE, Mansour VH, Nature, 1969, 222, 385
[5] McAuliffe CA in: Hartley FR (Ed.), Chemistry of Platin Group Metals, Recent
Developments, 1991, Elsvier, Amsterdam
[6] Rosenberg B in: Lippert B (Ed.), Chemistry and Biochemistry of a Leading Anticancer
Drug, 1999, Wiley VCH, Weinheim, 3
[7] Dwyer PJO, Stevenson JP, Johnson SW in: Lippert B (Ed.), Chemistry and Biochemistry
of a Leading Anticancer Drug, 1999, Wiley VCH, Weinheim, 31
[8] Einhorn LH, Donahue JP, Ann. Intern. Med., 1977, 87, 293
[9] Young RC, von Hoff DD, Gormley P, Makuch P, Cassidy J, Howser D, Bull JM, Cancer
Treat. Rep., 1979, 63, 1539
[10] Wittes RE, Cvitkovic E, Shah J, Gerold FP, String EW, Cancer Treat. Rep. 1977, 61,
359
[11] Scerocki JS, Hilaris BS, Hopfan S, Martini N, Barton D, Golbey RB, Wittes RE, Cancer
Treat. Rep., 1979, 63, 1593
[12] Panettiere F, Leichman LP, Tilchen EJ, Chen TT, Cancer Treat. Rep., 1984, 68, 1023
[13] Salem PA, Habborbi N, Anaissie E, Briki E, Issa P, Abbas J, Khalyl M, Cancer Treat.
Rep., 1985, 69, 891
[14] Higby DJ, Wallace HJ, Albert DJ, Holland JF, Cancer, 1974, 33, 1219
[15] Galanski M, Arion VB, Jakupec MA, Keppler BK, Curr. Pharm. Dess., 2003, 9, 2078
[16] Keppler BK, Lipponer KG, Stenzel B, Kratz F, Metal Complexes in Cancer Chemistry,
1993, VCH, Weinheim
[17] Pieper T, Borsky K, Keppler BK, Top. Biol. Inorg. Chem., 1999, 1, 171
[18] Dittich C, Scheulen ME, Jaehde U, Kynast B, Gneist M, Richly H, Schaad S, Arion VB,
Keppler BK, Proc. Am. Assoc. Cancer Res., 2005, 46, 472
[19] Lipponer KG, Vogel E, Keppler BK, Metal-Based Drugs, 1996, 3, 243
[20] Sava G, Pacor S, Bregant F, Anti-Cancer Drugs, 1990, 1, 99
[21] Pacor S, Sava G, Ceschia V, Bregant F, Mestroni G, Alessio E, Biochemistry, 1991, 34,
2369
Literaturverzeichnis
221
[22] Sava G, Pacor S, Bergamo A, Cocchieto M, Mestroni G, Alessio , Chem.Biol. Interact.,
1995, 95, 109
[23] Bergamo A, Gagliardi R, Furlani A, Alessio E, Mestroni G, Sava G, JPET, 1999, 289,
559
[24] Allardyce CS, Dyson PJ, Ellis DJ, Heath SL, Chem. Comm., 2001, 15, 1396
[25] Scolaro C, Bergamo A, Brescacin L, Delfino R, Cocchieto M, Laurenczy G, Geldbach
TJ, Sava G, Dyson PJ, J. Med. Chem., 2005, 48, 4161
[26] Ang WH, Daldini E, Scolaro C, Scopelliti R, Juillerat-Jeannerat L, Dyson PJ, Inorg.
Chem., 2006, 45, 9006
[27] Ang WH, Dyson PJ, Eur. J. Inorg. Chem., 2006, 20, 4003
[28] Kröder N, Kleeberg UR, Mross K, Edler L, Hossfeld DK, Onkologie, 2000, 23 (1), 60
[29] Keppler BK, New Journal of Chemistry, 1990, 14 (6-7), 389
[30] Haiduc I, Silvestru C, Coord. Chem. Rev., 1990, 99, 253
[31] Ott I, Dissertation, FU Berlin, 2004
[32] Ott I, Kircher B, Gust R, Jour. Inorg. Chem., 2004, 98 (3), 485
[33] Ott I, Schmidt K, Kircher B, Schumacher P, Wiglenda T, Gust R, Jour. Med. Chem.,
2005, 48 (2), 622
[34] Eastman A in: Lippert B (Ed.), Chemistry and Biochemistry of a Leading Anticancer
Drug, 1999, Wiley VCH, Weinheim, 111
[35] Gonzalez VM, Fuertes MA, Alonso C, Perez JM, Mol. Pharm., 2001, 59 (4), 657
[36] DeConti RC, Toftness BR, Lange RC, Creasey WA, Cancer Res., 1973, 33, 1310
[37] Kratz F, Metal Complexes in Cancer Chemotherapy, 1993, Wiley VCH
[38] Bodenner DL, Dedon PC, Keng PC, Borch RF, Cancer Res., 1986, 46, 2754
[39] Horacek P, Drobnik J, Biochim. Biophys. Acta, 1971, 254, 341
[40] Bancroft DP, Lepre CA, Lippard SJ, J. Am. Chem. Soc., 1990, 112, 6860
[41] Ott I, Gust R, Pharm unserer Zeit, 2006, 35, 124
[42] Miller SE, House DA, Inorg. Chim. Acta, 1990, 173, 53
[43] Frey U, Ranford JD, Sadler PJ, Inorg. Chem., 1993, 32, 1333
[44] Manka S, Becker F, Hohage O, Sheldrick WS, J.Inorg.Biochem., 2004, 98, 1947
[45] Hahn M, Dissertation, Ruhr-Universität Bochum, 2000
[46] Hahn M, Kleine M, Sheldrick WS, J.Biol.Inorg.Chem., 2001, 6, 556
[47] Hohage O, Sheldrick WS, J.Inorg.Biochem., 2006, 100, 1506
[48] Manka S, Dissertation, Ruhr-Universität Bochum, 2004
[49] Appleton T, Inorg.Chem., 1988, 27, 130
Literaturverzeichnis
222
[50] Gibson D, Kasherman Y, Kowarski D, Freikmann I, J.Biol.Inorg.Chem., 2006, 11, 179
[51] Wei H, Wang X, Liu Q, Mei Y, Lu A, Guo Z, Inorg.Chem., 2005, 44, 6077
[52] Timerbaev AR, Hartinger CG, Aleksenka SS, Keppler BK, Chem.Rev., 2006, 1006, 2224
[53] Appleton TG, Ross FB, Inorg.Chim.Acta, 1996, 252, 79
[54] Appleton TG, Pesch FJ, Wienken M, Menzer S, Lippert B, Inorg.Chem., 1992, 31, 4410
[55] Wolters D, Dissertation, Ruhr-Universität Bochum, 1998
[56] Fröhling CDW, Sheldrick WS, Chem.Comm., 1997, 1737
[57] Hahn M, Wolters D, Sheldrick WS, Hulsbergen FB, Reedijk J, J.Biol.Inorg.Chem., 1999,
4, 412
[58] Cox MC, Barnham KJ, Frenkiel TA, Hoeschele JD, Mason AB, He Q-Y, Woodworth
RC, Sadler PJ, J. Biol. Inorg. Chem., 1999, 4, 621
[59] Khalaila I, Allardyce CS, Verma SS, Dyson PJ, ChemBioChem., 2005, 6, 1788
[60] Timerbaev AR, Aleksenko KS, Polec-Pawlak K, Ruzik R, Hartinger CG, Keppler BK,
Electrophoresis, 2004, 25, 13, 1988
[61] Ivanov AI, Christodoulou J, Parkinson JA, Barnham KJ, Tucker A, Woodrow J, Sadler
PJ, J. Biol. Chem., 1998, 273, 14721
[62] Yang G, Miao R, Jin C, Mei Y, Tang H, Hong J, Guo Z, Zhu L, J. Mass Spectrom., 2005,
40, 1005
[63] Gibson D, Costello CE, Eur. Mass Spectrom., 1999, 5, 501
[64] Hartinger CG, Ang WH, Casini A, Messori L, Keppler BK, Dyson PJ, J.Anal. At.
Spectr., 2007, 22, 960
[65] Hartinger CG, Tsybin YO, Fuchser J, Dyson PJ, InorganicChemistry, 2008, 47, 1, 17
[66] Mandal R, Li XF, Rapid Comm. Mass Spectrom., 2006, 20, 48
[67] Peleg-Shulman T, Najajreh Y, Gibson D, J. Inorg. Biochem., 2002, 91, 306
[68] Casini A, Gabbiani C, Messori L, Pieraccini G, ChemMedChem, 2006, 1, 413
[69] Casini A, Gabbiani C, Arnesano F, Messori L, Biochemistry, 2007, 46, 12220
[70] Casini A, Francese S, Moneti G, Messori L, Chem.Commun., 2007, 156
[71] Heeb S, Dissertation, Universität Kaiserslautern, 1990
[72] Exner R, Dissertation, Universität Kaiserslautern, 1990
[73] Hartinger CG, Hann S, Koellensperger G, Sulyok M, Groessl M, Timberbaev AR,
Rudnev AV, Stingeder G, Keppler BK, Int. J. Clin. Pharmacol. Ther., 2005, 43, 583
[74] Sulyok M, Hann S, Hartinger CG, Keppler BK, Stingeder G, Koellensperger G, J. Anal.
At. Spectrom., 2005, 20, 856
Literaturverzeichnis
223
[75] Pongratz M, Schluga P, Jakupec MA, Arion VB, Hartinger CG, Allmaier G, Keppler BK,
J. Anal. At. Spectrom., 2004, 19, 46
[76] Polec-Pawlak K, Abramski JK, Keppler BK, Jarosz M, Electrophoresis, 2006, 27, 1128
[77] Trynda-Lemiesz L, Keppler BK, Kozlowski H, Jour. Inorg. Biochem., 1999, 73, 3, 123
[78] Trynda-Lemiesz L, Keppler BK, Kozlowski H, Jour. Inorg. Biochem., 2000, 78, 4, 341
[79] Ang WH, Daldini E, Dyson PJ, Inorganic Chemistry, 2007, 46, 22, 9048
[80] Scolaro C, Hartinger CG, Bergamo A, Dyson PJ, Dalton Trans., 2007, 5065
[81] Wang FY, Bella J, Parkinson JA, Sadler PJ, J. Biol. Inorg. Chem., 2005, 10, 2, 147
[82] Chen HM, Parkinson JA, Morris RE, Sadler PJ, J. Am. Chem. Soc., 2003, 125, 1, 173
[83] McNae IW, Fishburne K, Wang FY, Sadler PJ, Chem. Commun., 2004, 16, 1786
[84] Casini A, Gabbiani C, Dyson PJ, Messori L, ChemMedChem, 2007, 2, 631
[85] Eidhammer I, Flikka K, Martens L, Mikalsen S-O, Computational Methods for Mass
Spectrometry Proteomics, 2007, Wiley VCH
[86] Hunt DF, Yates JR, Shabanowitz J, Winston S, Hauer CR, Proc. Nat. Acad. Sci. USA,
1986, 83, 17, 6233
[87] Hunt DF, Buko AM, Ballard JM, Shabanowitz J, Giordani AB, Biomed. Mass Spectrom.,
1981, 8, 397
[88] Taylor SW, Fahy E, Zhang B, Glenn GM, Warnock DE, Wiley S, Murphy AN, Gaucher
SP, Ghosh SS, Nat. Biotechnol., 2003, 21, 281
[89] Allardyce CS, Dyson PJ, Birtwistle H, Coffey J, Chem. Commun., 2001, 2708
[90] Khalaila I, Bergamo A, Bussy F, Sava G, Dyson PJ, Int. Jour. Oncol., 2006, 29, 261
[91] Encarta, Enzyklopädie, online Version
[92] Han MJ, Lee SY, Microbiol. Mol. Biol. Rev., 2006, 70, 2, 362
[93] Gygi SP, Rist B, Gerber SA, Turecek F, Gelb MH, Aebersold R, Nat. Biotechnol., 1999,
17, 994
[94] Wolters DA, Washburn MP, Yates JR 3rd, Anal. Chem., 2001, 73, 23, 5683
[95] Washburn MP, Ulaszek R, Deciu C, Yates JR 3rd, Anal. Chem., 2002, 74, 7, 1650
[96] Washburn MP, Wolters DA, Yates JR 3rd, Nat. Biotechnol., 2001, 19, 242
[97] Schindelin H, Jiang W, Inouye M, Heinemann U, Proc. Natl. Acad. Sci. USA, 1994, 91,
5119
[98] Fraser ME, James MN, Wolodko WT, J. Mol. Biol., 1999, 285, 1633
[99] Reedijk J, Chem. Rev., 1999, 99, 2499
[100] Fröhling CDW, Dissertation, Ruhr-Universität Bochum, 1997
[101] Leijonmarck M, Liljas A, J. Mol. Biol., 1987, 195, 555
Literaturverzeichnis
224
[102] Song H, Parsons MR, Leonard G, Phillips SEV, J. Mol. Biol., 1999, 285, 1245
[103] Di Constanzo L, J. Mol. Biol, 2007, 366, 2, 482
[104] Wang Y, Biochem. Biophys. Res. Comm., 2002, 292, 2, 396
[105] Pautsch A, Schulz GE, J. Mol. Biol., 2000, 298, 273
[106] Lamers MH, Perrakis A, Enzlin J, Winterwerp HHK, De Wind N, Sixma TK, Nature,
2000, 407, 711
[107] Kunkel TA, Erie DA, Annu. Rev. Biochem., 2005, 74, 681
[108] Petranovic M, Nagy B, Petranovic D, Biochimie, 2001, 83, 11-12, 1041
[109] Van Houten B, Microbiol. Mol. Biol. Rev., 1990, 54, 1, 18
[110] Subramanya HS, Bird Lem Brannigan JA, Wigley DB, Nature, 1996, 384, 379
[111] Zechiedrich EL, Chen D, Lilley DM, Cozzarelli NR, J. Biol. Chem., 2000, 275, 8103
[112] Sugandan S, Kumar S, Brainerd JA, nicht publiziert
[113] Chandran V, Luisi BF, J. Mol. Biol., 2006, 358, 8
[114] Nguyen NT, Maurus R, Stokell DJ, Ayed A, Duckworth HW, Brayer GD,
Biochemistry, 2001, 40, 13177
[115] Katti SK, LeMaster DM, Eklund H, J. Mol. Biol., 1990, 212, 167
[116] Alberts, Bray, Hopkin, Johnson, Lewis, Raff, Roberts, Walter, Lehrbuch der
molekularen Zellbiologie, 3. Auflage, 2005, Wiley VCH
[117] Teuben JM, Zubiri MR, Reedijk J, J. Chem. Soc., Dalton Trans., 2000, 369
[118] Fichtinger-Schepman AMJ, van der Veer JL, den Hartog JHJ, Lohman PHM, Reedijk J,
Biochemistry, 1985, 22, 707
[119] Reedijk J, Chem. Com., 1996, 7, 801
[120] Corbin RW, Paliy O, Jordan MI, Kustu S, Hunt DF, Proc. Natl. Acad. Sci. USA, 2003,
100, 9232
[121] Poulsen P, Jensen KF, Res. Microbiol., 1991, 142, 283
[122] Biologie für Mediziner, online Version
[123] Zamble DB, Lippard SJ in: Lippert B (Ed.), Chemistry and Biochemistry of a Leading
Anticancer Drug, 1999, Wiley VCH, Weinheim, 73
[124] Murphy FV IV, Sweet RM, Churchill MEA, The EMBO Journal, 1999, 18, 6610
[125] He Q, Liang CH, Lippard SJ, Proc. Natl. Acad. Sci. USA, 2000, 97, 5768
[126] Kato M, Yamamoto H, Okamura T, Nobuko M, Ueyama N, Dalton Trans., 2005, 1023
[127] Arner ESJ, Nakamura H, Sasada T, Yodoi J, Spyrou G, Free Radi. Biol. Med., 2001, 31,
1170
[128] Götze HJ, Sheldrick WS, Siebert AFM, Fresenius J. Anal. Chem., 1993. 346, 634
Literaturverzeichnis
225
[129] Sovago I,Osz K, Dalton Trans., 2006, 32:, 3841
[130] Phadtare S, Tadigotla V, Shin WH, Severinov K, J. Bacteriol., 2006, 188, 2521
[131] WillJ, Sheldrick WS, Wolters D, J. Biol. Inorg. Chem., 2008, 13, 421
[132] Takada K, Kawamura T, Mauda S, Oka T, Yoshikawa H, Ike O, Hitomi S, Biopharm.
Drug Dispos., 1999, 20, 421
[133] Hochstrasser DF, Clin. Chemistry and Laboratory Medicine, 1993, 36, 825
[134] Anderson NL, Anderson NG, Mol. Cell. Proteomics, 2002, 1, 845
[135] Wardell M, Wang Z, Ho JX, Robert J, Ruker F, Ruble J, Carter DC, Biochem. Biophys.
Res. Comm., 2002, 291, 813
[136] Wally J, Halbrooks PJ, Vonrhein C, Rould MA, Everse SJ, Mason AB, Buchanan SK,
J. Biol. Chem., 2006, 281, 24934
[137] Allardyce CS, Dyson PJ, Coffey J, Johnson N, Rap. Comm. Mass Spectr., 2002, 16, 933
[138] Ajees AA, Anantharamaiah GM, Mishra VK, Hussain MM, Murthy KHM, Proc. Natl.
Acad. Sci.USA, 2006, 103, 2126
[139] Kim S, Woo J, Seo EJ, Yu M, Ryu S, J. Mol. Biol., 2001, 306, 109
[140] Kumar MS, Carson M, Hussain MM, Murthy HMK, Biochemistry, 2002, 41, 11681
[141] Liu H, Sadygov RG, Yates JR 3rd, Anal. Chem., 2004, 76, 4193
[142] Gonias SL, Pizzo S, J. Biol. Chem., 1983, 258, 5764
[143] Momburg R, Bordeaux M, Sarrazin M, Chauvet M, Briand C, J. Pharm. Pharmacol.,
1987, 39, 3003
[144] Neault JF, Tajmir-Riahi HA, Biochim. Biophy. Acta, 1998, 1384, 153
[145] Wardell M, Wang Z, Ho JX, CarterDC, Biochem. Bioohys. Res. Comm., 2002, 291, 813
[146].Wolters DA, Washburn MP,Yates JR 3rd, Analytical Chemistry, 2001, 73, 23, 5685
[147] swiss-prot online-Datenbank
[148] Siebert AFM, Dissertation, Ruhr-Universität Bochum, 1995
[149] Hohage O, Dissertation, Ruhr-Universität Bochum, 2007
[150] Kleine M, Dissertation, Ruhr-Universität Bochum, 2003
[151] Roepstorff P, Fohlmann J, Biomed. Mass Spectrom., 1984, 11, 601
[152] Becker K, Gromer S, Schirmer RH, Müller S, Eur. J. Biochem., 2000, 267, 6118
[153] Hill KE, McCollum GW, Boeglin ME, Burk RF, Biochem. Biophys. Res. Commun.,
1997, 234, 293
[154] Gromer S, Arscott LD, Williams CH, Schirmer RH, Becker K, J. Biol. Chem., 1998,
273, 20096
[155] Powis G, Mustacich D, Coon A, Free Radical Biology Medicine, 2000, 29, 312
Literaturverzeichnis
226
[156] Rollin-Genetet F, Berthomieu C,.Davin AH, Quemeneur E, Eur. J. Biochem., 2004,
271, 1299
[157] Hütter G, Dissertation, FU Berlin, 2000
[158] Simmons DL, Botting RM, Hla T, Pharmacol. Rev., 2004, 56, 387
[159] Chandrasekharan NV, Simmons DL, Genome Biology, 2004, 5, 9, 241
[160] Mahanta S, Sarma PK, Buragohain AK, nicht publiziert,
[161] Ott I, Gust R, Arch. Pharm., 2007, 340, 117
[162] Watt GW, Cude WA, Inorg. Chem., 1968, 7, 335
[163] Vashisht Gopal YN, Jayaraju D, Kondapi AK, Biochemistry, 1999, 38, 4382
[164] Frodl A, Dissertation, Ruhr-Universität Bochum, 2001
[165] Chandra M, Pandey DS, Puerta MC, Valerga P, Acta Cryst., 2002, E58, m28 - m29
[166] Weichsel A, Gasdaska JR, Powis G, Montfort WR, Structure, 1996, 4, 735
Danksagung
Meinem Mann danke ich einfach für alles.
Meiner Familie danke ich ebenfalls für jegliche mir gewährte Unterstützung.
Bei allen aktiven und ehemaligen Mitarbeitern der Arbeitskreise Sheldrick und Iris Oppel
möchte ich mich sowohl für die fachliche, als auch für die freundschaftliche Hilfe und ihre
aufbauenden Worte bedanken.
Dr. Oliver Hohage danke ich für seine beruhigende und immer wieder aufmunternde Art und
natürlich für die fachlichen Tipps.
Dr. Kirsten Föcker, Me Harlos, Anna Kromm, Yvonne Geldmacher, Ruth Bieda und Dr.
Michael Scharwitz danke ich für die netten Stunden vor allem außerhalb des MS-Bunkers.
Den aktiven und ehemaligen Mitgliedern der Arbeitsgruppe von Dr. Dirk Wolters (besonders
Dr. Dirk Wolters, Sabine Bendix und Dr. Andreas Kyas) danke ich für die Möglichkeit der
Mitbenutzung der Geräte und die vielen praktischen Tipps.
Jan-Amadé Bangert, Gerd Kock und Dr. Carsten Berghaus danke ich nicht nur für die
computer-technische Unterstützung, sondern auch für die unterhaltsame Zeit.
Der Arbeitsgruppe von Prof. Gust (FU Berlin), besonders Dr. Ingo Ott sowie den weiteren
Mitgliedern der Forscherguppe, danke ich für die gute Zusammenarbeit und die nette
Atmosphäre bei allen FOR 630-Treffen.
Bei allen, die zum Gelingen der Arbeit in irgendeiner Weise beigetragen haben, aber nicht
namentlich genannt sind, möchte ich mich dafür entschuldigen. Mein Dank gilt natürlich auch
Euch.
Lebenslauf
Name:
Geburtsdatum:
Geburtsort:
Familienstand:
Nationalität:
Joanna Maria Will, geb. Sajonz
23.08.1977
Oppeln
verheiratet
deutsch
Schulbildung
Sept. 1984 – Dez. 1990
Jan. 1991 – Juni 1997
Juni 1997
Volksschule in Oderwinkel, Kreis Oppeln
Ursulinengymnasium Werl
Allgemeine Hochschulreife, Juni 1997
Berufsausbildung
Aug. 1997 – Juli 1999
Aug. 1999 – Febr. 2000
März 2000
Pharmazeutisch-technische Lehranstalt in Castrop-Rauxel
Berufspraktikum im Rahmen der Ausbildung zur
pharmazeutisch-technischen Assistentin
Abschluss als staatlich geprüfte pharmazeutisch-technische
Assistentin
Studium
April 2000 – Mai 2005
Oktober 2002
Nov. 2004 – Mai 2005
Studiengang Diplom-Chemie an der Ruhr-Universität Bochum
Vordiplom der Chemie
Anfertigung der Diplomarbeit am Lehrstuhl für Analytische
Chemie bei Prof. W. S. Sheldrick, Thema der Arbeit:
„Wechselwirkungen zytostatisch wirksamer Platinkomplexe mit
5´-GMP2- in Gegenwart schwefelhaltiger Substanzen“
Mai 2005
Diplom der Chemie
Juli 2005 – Mai 2008
Promotion am Lehrstuhl für Analytische Chemie bei
Prof. W. S. Sheldrick, Thema der Arbeit:
„Massenspektrometrische Ermittlung der Proteintargets von
zytotoxischen Metallkomplexen in Blutserum und E. coli-Zellen“
Veröffentlichungen
1. Will J, Kyas A, Sheldrick WS, Wolters DA,
Identification of (η6-arene)ruthenium(II) protein binding sites in E.coli cells by combined
multidimensional liquid chromatography and ESI tandem mass spectrometry: specific binding
of [(η6-p-cymene)RuCl2(DMSO)] to stress-regulated proteins and to helicases
J. Biol. Inorg. Chem., 2007, 12, 883
DOI 10.1007/s00775-007-0242-x
2. Will J, Wolters DA, Sheldrick WS
Multidimensional LC-ESI MS/MS analysis of protein targets for cisplatin and
[(η6-p-cymene)RuCl2(DMSO)] in human serum and E. coli cells
J. Biol. Inorg. Chem., 2007, 12 (Suppl 1), 50, P093
3. Will J, Sheldrick WS, Wolters DA
Characterisation of cisplatin coordination sites in cellular E. coli DNA binding proteins by
combined biphasic liquid chromatography and ESI tandem mass spectrometry
J. Biol. Inorg. Chem., 2008, 13, 421
DOI 10.1007/s00775-007-0333-8
4. Will J, Wolters DA, Sheldrick WS
Characterisation of Cisplatin Binding Sites in Human Serum Proteins using Hyphenated
Multidimensional Liquid Chromatography and ESI Tandem Mass Spectrometry
ChemMedChem, 2008, eingereicht
5. Ott I, Kircher B, Bagowski C, Vlecken DHW, Ott E, Will J, Sheldrick WS, Gust R
Bioorganometallic Aspirin: A Hexacarbonyldicobalt Complex as NSAID like Anticancer
Agent and Angiogenesis-Inhibitor
Angewandte Chemie, 2008, eingereicht
Poster/Präsentation
1. Will J, Kyas A, Sheldrick WS, Wolters DA
Shotgun identification of metal-protein complexes in whole cell systems by combined
Multidimensional Liquid Chromatography ans ESI tandem mass spectrometry
3rd International Symposium on Bioorganometallic Chemistry, Mailand, 5.-8. Juli 2006,
Poster
2. Will J, Wolters D, Sheldrick WS
Identification of protein targets for cisplatin and [(η6-p-cymene)RuCl2 (DMSO)] in E.coli
cells and human blood serum using multidimensional LC-ESI MS/MS analysis
FOR 630-Treffen in Berlin, Eggersdorf, 30.Mai- 1.Juni 2007, Präsentation
3. Will J, Wolters D, Sheldrick WS
Identification of protein targets in human serum using the multidimensional LC-ESI MS/MS
Symposium on Medicinal Organometallic Chemistry, St. Martin in der Pfalz, 2.-5. April
2008, Poster