ausgangsdatei f.rs netz - an der Universität Duisburg
Werbung

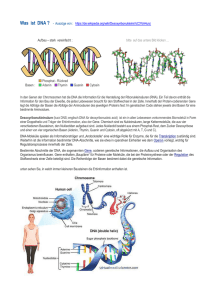
Z ukunft Biochips Mikroarrays – Zukunftstechnik für genetische Grundlagenforschung Die Entschlüsselung des menschlichen Genoms im Rahmen des Human Genome Project erschließt ein enormes Potenzial für die Erforschung von Genstrukturen und Genfunktionen im menschlichen Erbgut. Ziele solcher Interpretationen des genetischen Bauplanes sind insbesondere die Frühdiagnose von Krankheitsveranlagungen sowie die Effizienzsteigerung hochwirksamer Medikamente. Eine wesentliche Rolle bei der Analyse dieser Genfunktionen spielen Biochips oder – wie es eigentlich korrekt heißen müsste – DNAMicroarrays. Solche Biochips stellen heute unverzichtbare Werkzeuge in der molekularbiologischen Grundlagenforschung dar, um etwa das Zusammenspiel der Gene in den aktiven Stoffwechselwegen in unterschiedlichen Zellstadien aufzuklären oder Genveränderungen bei Krebserkrankungen zu untersuchen. Die gleichzeitige Erfassung der Aktivität tausender charakteristischer Gene erlaubt außerdem in vielfältiger Art und Weise die Präzisierung medizinischer Befunde. Je mehr genetische Informationen in den kommenden Jahren und Jahrzehnten aus den Genomprojekten gewonnen werden, desto mehr spezielle BiochipAnwendungen werden denkbar sein. Fast täglich werden neue Belege gefunden, dass bereits kleine Abweichungen in einzelnen DNA-Basenpaaren eines Gens für das Zustandekommen von Krankheitsbildern verantwortlich sind. Für die schnelle Diagnose individueller Krankheitsvarianten bieten sich Biochips geradezu an. Die Entwicklung von Verfahren zur Genfunktionsanalyse ist ein weltweit enorm wachsender Forschungs- und Wirtschaftsmarkt, an dem deutsche Forschungsinstitute – und hier auch Wissenschaftler der UDE – maßgeblich beteiligt sind. C hancen für die Krebsforschung DNA-Mikroarrays bieten die Möglichkeit, ein Expressionsprofil – eine Art Fingerabdruck – aller Gene eines Organismus für ein bestimmtes Gewebe oder einen bestimmten Zelltyp zu erstellen, wenn die Sequenz dieser Gene bekannt ist. Ein Expressionsprofil ist ein Analyseverfahren, das anzeigt, welche Gene in einer bestimmten Situation tatsächlich in Proteine übersetzt werden. Um ein Expressionsprofil gewinnen zu können, muss zunächst aus dem Untersuchungsmaterial die so genannte messenger-RNA (mRNA) isoliert werden. Diese Boten-RNS ist die Arbeitskopie eines Gens, die im normalen Zellstoffwechsel aus dem Zellkern wandert, um in den Ribosomen der Zelle in Protein übersetzt zu werden. Die isolierte mRNAPopulation, die je nach Zelltyp aus 30.00050.000 verschiedenen mRNA-Spezies besteht, wird enzymatisch in eine doppelsträngige cDNA, also eine Kopie der ursprünglichen DNA, umgeschrieben, aus der wieder eine cRNA durch in vitro-Transkription gewonnen wird. Hierbei wird ein spezielles Nukleotid eingebaut, das eine spätere Markierung der cRNA erlaubt. Nukleotide sind die Grundbausteine der DNA. Sie bestehen aus einer der vier basischen Stickstoffverbindungen Adenin, Cytosin, Guanin und Thymin, einem Zuckeranteil sowie einer Phosphatgruppe. Die so modifizierte cRNA des Untersuchungsmaterials wird auf den Mikroarray aufgetragen (Abb. 2). Unter geeigneten Bedingungen binden nun die modifizierten cRNAMoleküle spezifisch an die auf dem Array vorhandenen komplementären Genfragmente. Abbildung 1: Schematischer Aufbau eines DNA-Mikroarrays, der aus einer großen Zahl von DNA-Molekülen besteht, die in kleinen Sektoren auf dem Glasträger immobilisiert sind (weiße Quadrate). Herausvergrößert ist ein sog. Probeset, das aus jeweils ca. 20 verschiedenen Oligonukleotiden mit perfekter Sequenz (obere Reihe) und den dazugehörigen 20 Kontrolloligonukleotiden (untere Reihe) besteht. Kontrolloligokuleotide enthalten einen Basenaustausch (Mismatch) gegenüber der perfekten Sequenz und sollten eine deutlich schwächere oder keine Hybridisierung zeigen. 70 FORUM Forschung 2003/2004 Abbildung 2: Darstellung des Verfahrens zur Messung von Genaktivität mittels DNA-Mikroarray-Hybridisierung. Die isolierte mRNA wird enzymatisch modifiziert und auf den Mikroarray aufgetragen. Nach dem Waschen bleiben nur perfekt komplementäre mRNA-Moleküle an den immobilisierten DNA-Fragmenten gebunden und ergeben nach Färbung mit Streptavidin-Phycoerythrin (hier gelb dargestellt) ein spezifisches Signal im Laser-Scanner. Ungebundene cRNA-Moleküle, die keine komplementären DNA-Fragmente auf dem Array finden, werden durch Waschen entfernt, und die gebundenen cRNA-Moleküle werden mit einem Farbstoff markiert. Schließlich wird der Mikroarray in einem Scanner ausgemessen, indem der Farbstoff durch Laserlicht angeregt und die Fluoreszenz in den verschiedenen Sektoren, die den bekannten Genfragmenten entsprechen, gemessen wird (Abb. 3, 4). Die Stärke der Fluoreszenz in den verschiedenen Genfragmenten ist ein sehr präzises Maß für die Menge der im Ausgangsmaterial vorhandenen mRNA-Moleküle und damit in erster Näherung auch ein Maß für die Menge der entsprechenden Proteine. Da mehrere Mikroarrays parallel beschickt und nacheinander ausgewertet werden können, kann zum Beispiel das Expressionsprofil des derzeit bekannten Anteils des menschlichen Genoms (> 90 %) in einem bestimmten Gewebe in einem einzigen Arbeitsgang analysiert werden. Jede Zellart kann so anhand ihres charakteristischen Expressionsprofils identifiziert werden. Das Verfahren ermöglicht eine quantitative Aussage über das Expressionsniveau der Gene eines gesamten Genoms in einem Arbeitsgang und in sehr kurzer Zeit. Weiterentwicklungen dieser einfachsten Form des Mikroarrays erlauben darüber hinaus auch, bestimmte Mutationen wie etwa Polymorphismen, die auf einzelnen Basenaustauschen im DNA-Strang beruhen (so genannte single nucleotide polymorphisms oder SNPs), für eine Reihe von Genen zu erkennen. Diese neue Technologie und vor allem die Messung der Expressionsprofile verspricht einen rascheren Zugang zur Identifikation von Signalübertragungswegen, ein besseres Verständnis der Wirkung von Medikamenten und auch einen differenzierteren Zugang zu einer molekular definierten Prognose von Erkrankungen als herkömmliche Verfahren. Insbesondere für die Krebsforschung könnte diese neue Technologie von großer Bedeutung sein. Clusteranalysen von Expressionprofilen, also die Auswertung zahlreicher Einzelresultate aus einer Vielzahl von Tumorgeweben, könnten Auskunft über den Verlauf der Erkrankung und über die Mechanismen der Therapieresistenz geben. Wie bei jeder neuen Technologie wird allerdings abzuwarten sein, welche der erhofften Anwendungen unser Verständnis molekularbiologischer und biochemischer Zusammenhänge tatsächlich deutlich vertiefen wird. 71 Universität Duisburg–Essen A uf 1,5 cm2 12000 Gene Ein DNA-Mikroarray besteht aus einem Glasträger, auf dem Fragmente bekannter Gene oder noch wenig charakterisierter, einzelner exprimierter Sequenzen (EST s) in einem dicht gepackten, geordneten Muster aufgebracht sind. Die Anordnung der Genfragmente ist durch ein Koordinatensystem definiert. Schon heute sind die Voraussetzungen für eine sehr umfassende MikroarrayAnalyse des menschlichen Genoms und anderer Genome, deren Sequenz vollständig bekannt ist, gegeben. Im Falle der von der Firma Affymetrix hergestellten DNA -Mikroarrays werden statt cDNA -Fragmenten von mehreren hundert Abbildung 3: Ergebnis der Messung eines DNABasenpaaren kurze Oligonukleotide in einem Mikroarrays im Laser-Scanner (Übersicht). Gezeigt ist ein photolithographischen Verfahren direkt auf Pseudocolor-Bild. Die Floureszenzintensität nimmt von dem Glasträger synthetisiert. Hierbei han- schwarz (inaktiv, keine Expression) über blau, grün, gelb, delt es sich um kurze Nukleinsäurefäden, die rot und weiß (höchste Intensität) zu. aus nur wenigen Bausteinen bestehen. Da sowohl das verwendete Material als auch das Hefe abdecken. Die Arrays, die Gensequenzen Herstellungsverfahren starke Parallelen zur der Ratte und der Maus enthalten, werden insComputerchip-Herstellung aufweisen, hat sich besondere bei der Analyse der Wirkungsweise die alternative Bezeichnung Biochip oder auch von therapeutisch interessanten neuen WirkGen-Chip für Mikroarray eingebürgert. In dem stoffen an Zellkulturen und Versuchstieren und angesprochenen Array-Herstellungsverfahren in der Grundlagenforschung zunehmend an Bewerden pro Gen bis zu 20 verschiedene 25erdeutung gewinnen. Oligonukleotide in winzigen Sektoren auf den Träger aufgebracht. Heute lassen sich so auf eiIm Vergleich zu herkömmlichen RNA-Bener ca. 1,5 cm2 großen Fläche Fragmente von stimmungsmethoden, mit denen bei ähnlichem bis zu 12.000 verschiedenen Genen mit dazugeZeitaufwand allenfalls bis zu 10 Gene parallel gemessen werden können, stellt dies eine für hörigen Kontrollen – in insgesamt mehr als 400.000 Sektoren – in einem geordneten MusBiologen und Mediziner hoch interessante Innovation dar, die letztlich die Kosten für ter unterbringen (Abb. 1). Die Firma Affymetrix bietet für verschiedene Fragestellungen Arrays die Durchführung einer solchen Analyse rechtmit einer unterschiedlichen Auswahl und Anzahl fertigt. Ein weiterer, großer Vorteil der Methode ist, dass vergleichsweise wenig Gewebe von Genen an und hat Genchips entwickelt, die annäherungsweise die gesamten Genome verbzw. mRNA für die Untersuchungen benötigt schiedener Spezies wie Mensch, Maus, Ratte oder wird. DNA (engl.: Desoxyribonucleic-acid, deutsch: DNS) Träger der genetischen Information in der Zelle. DNA besteht aus den Basen Adenin, Thymin, Guanin und Cytosin, dem Zucker Desoxyribose sowie Phosphorsäure. Jeweils zwei Molekülstränge sind durch Wasserstoffbrücken parallel miteinander verbunden und in einem gewundenen Strang, der so genannten Doppel-Helix, miteinander verdrillt. Bestimmte Untereinheiten der DNA bewirken jeweils die Produktion einer Aminosäure. Mehrere solcher Untereinheiten bilden ein Gen. RNA (engl.: Ribonucleic-acid, deutsch: RNS ) Einfach-strängiges Polynucleotid, besteht im Unterschied zur DNA aus den Basen Adenin, Guanin, Cytosin und Uracil, dem Zucker Ribose und einem Phos- phorsäurerest. Es gibt unterschiedliche Formen von RNA , zum Beispiel die Transfer- oder tRNA als Aminosäureüberträger, die messenger- oder mRNA als Informationsüberträger bei der Proteinsynthese in der Zelle sowie die ribosomale oder rRNA als Bestandteil der Ribosomen. cDNA (copy- oder complementary-DNA ), Kopie eines RNA -Teils. Basenpaare Verbindung von je zwei der vier Basenpaare in der DNA über Wasserstoffbrücken, die die doppelsträngige Struktur der DNA ergibt. Adenin bindet nur mit Thymin, Cytosin ausschließlich mit Guanin. 72 FORUM Forschung 2003/2004 „P harmacogenomics“ Einen sehr hohen Stellenwert wird die Mikroarray-Analyse in Zukunft in der Grundlagenforschung einnehmen, da sie erlaubt, auf molekularer Ebene in die verschiedensten Prozesse der biologischen Steuerung Einblick zu nehmen. Erste Erfahrungen mit Zellen in Kultur zeigen, dass etwa die Stimulierung von Membranrezeptoren mit Liganden oder die Expression konditioneller Allele von Transkriptionsfaktoren eindeutige und reproduzierbare Messergebnisse liefern. Membranrezeptoren sind bestimmte Molekülgruppierungen der Zellmembran, die aus integrierten Proteinen, Lipiden und einem Enzym bestehen. Liganden sind Signalstoffe, die auf die Membranrezeptoren einwirken und so chemische Reaktionen der Zelle auslösen. Ein Allel ist die Ausprägung eines bestimmten Merkmals aufgrund eines Gens; unter Transkriptionsfaktoren versteht man Proteine, die die Übersetzung der Gene in RNA steuern. Dies wird die Aufklärung von Signalübertragungswegen und die Identifizierung von Zielgenen vieler bekannter Transkriptionsregulatoren (Gene, die die Bildung von Transkriptionsfaktoren bewirken) enorm erleichtern und beschleunigen. Neben kultivierten Zellen können auch verschiedene Gewebe aus Modellorganismen analysiert werden, wenn sie vergleichbare Zellpopulationen enthalten. Modellorganismen sind Tier- oder Pflanzenarten, die wissenschaftlich gut untersucht werden können und stellvertretend für eine größere Gruppe von Lebewesen stehen. Bekannte Beispiele sind Mäuse oder die Taufliege Drosophila. Eine schier unerschöpfliche Quelle für Mikroarray-Analysen bieten die Vielzahl von Mausmutanten, die mittels Gene Targeting, also durch das gezielte Abschalten bestimmter Gene, hergestellt wurden und die spezifisch eingeführte Gendefekte aufweisen. Eine weitere Anwendung von DNA-Mikroarrays kann mit dem Wort Pharmacogenomics beschrieben werden. Hierbei handelt es sich um die Analyse von Effekten, die Pharmaka oder andere niedermolekulare Wirkstoffe auf das Transkriptionsprofil von Zellen haben. Ein Ziel ist hier, toxische Wirkungen von Pharmaka mit spezifischen Transkriptionsprofilen zu verbinden. Dies kann die Auswahl von Wirkstoffen erheblich vereinfachen, da man ohne Tierversuche solche Kandidaten aussortiert, die bekannte Toxizitätsmuster aufweisen. Eine Vielzahl der heute üblichen Therapeutika greift nämlich direkt in Signalwege ein. Man kann davon ausgehen, dass etwa 10% der wichtigsten Pharmaka dadurch wirken, dass sie mittelbar oder unmittelbar Transkription Abbildung 4: Ausschnittsvergrößerung eines DNA-Mikroarrays nach Hybridisierung. Beispielhaft sind die Ergebnisse für die Probesets für das Gen adipogenesis inhibitory factor, das in diesem Experiment keine Expression zeigt, und für das Fibromodulin-Gen gezeigt. Das Fibromodulin-Gen wird exprimiert, da in der oberen Reihe des Probesets, die die perfekt hybridisierenden Oligonukleotide enthält, stärkere Signale zu sehen sind als in der unteren Reihe, die die mismatchOligonukleotide enthält. kontrollieren. Ein Beispiel sind Cortison, Östrogen oder Thyroxin, die an Kernrezeptoren, speziellen Proteinen im Zellkern, binden, welche selbst als Transkriptionsfaktoren agieren. Das zur Immununterdrückung eingesetzte Cyclosporin wirkt etwa dadurch, dass es das Enzym Calcineurin daran hindert, den Transkriptionsfaktor NF-AT (Nuclear Factor of Activated T-cells) zu aktivieren. Das weitverbreitete Salicylat verhindert unter anderem die Aktivierung des Transkriptionsfaktors NF kappa B . Diese und weitere Beispiele unterstreichen die große Bedeutung der Transkriptionskontrolle bei der Wirkung von Pharmaka. Es ist offensichtlich, dass der Erstellung von Transkriptionsprofilen für klinische Wirkstoffe in sehr naher Zukunft eine enorme Bedeutung zukommen wird. Ko n t a k t Univ.-Prof. Dr. Tarik Möröy Priv.-Doz. Dr. Ludger Klein-Hitpass Institut für Zellbiologie und Tumorforschung Universitätsklinikum Essen Tel. 02 01/7 23-33 80 Tel. 02 01/7 23-59 04 [email protected] [email protected] http://www.ifz.uni-essen.de/ 73 Universität Duisburg–Essen