Lösungen Aminosäuren und Proteine
Werbung
Lösungen zum Thema „Aminosäuren und Proteine“ der LK-Chemie-Abiturjahrgängen
von 1982 – 1998
Kursleiter Klaus Bentz/ Kollegiatin Maria Dickert
Abiturjahrgang 2005
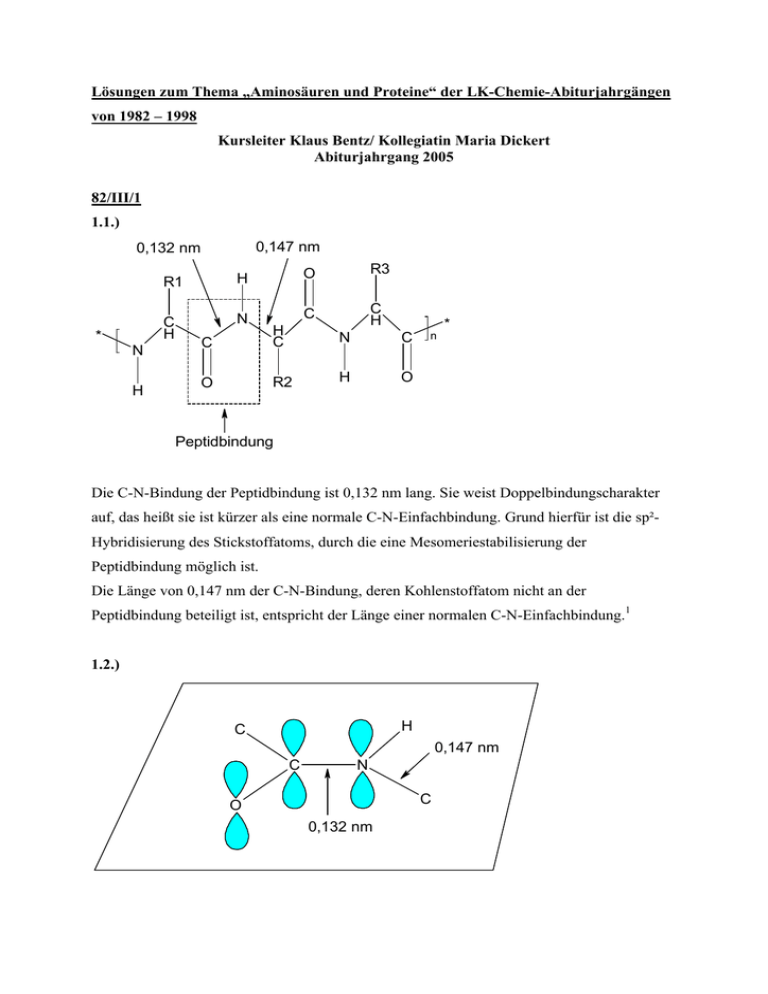
82/III/1
1.1.)
0,147 nm
0,132 nm
*
N
H
H
O
R3
R1
C
H
N
C
C
H
*
C
H
C
N
C
O
R2
H
O
n
Peptidbindung
Die C-N-Bindung der Peptidbindung ist 0,132 nm lang. Sie weist Doppelbindungscharakter
auf, das heißt sie ist kürzer als eine normale C-N-Einfachbindung. Grund hierfür ist die sp²Hybridisierung des Stickstoffatoms, durch die eine Mesomeriestabilisierung der
Peptidbindung möglich ist.
Die Länge von 0,147 nm der C-N-Bindung, deren Kohlenstoffatom nicht an der
Peptidbindung beteiligt ist, entspricht der Länge einer normalen C-N-Einfachbindung.1
1.2.)
H
C
0,147 nm
C
N
C
O
0,132 nm
Die Bindungswinkel betragen 120°. Die Reste sind in Transform angeordnet. Diese
Konfiguration wird bevorzugt, da die Reste einen großen Raumbedarf haben, und so
möglichst weit voneinander entfernt sind. Die Peptidbindung ist planar und starr, die Bindung
ist also um die eigene Achse nicht frei drehbar. Grund hierfür ist, dass das Kohlenstoffatom,
das Stickstoffatom und das Sauerstoffatom der Peptidbindung sp2-hybridisiert sind. Die
Valenzstriche in der Skizze kennzeichnen die Sigmabindungen zwischen den Atomen. Die
freien py-Orbitale überlappen sich zu einer delokalisierten ð-Elektronenwolke. Die
Delokalisation der Elektronen führt zur Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
R2
C
+
N
R1
O
R2
C
}
N
R1
O
O
R2
C
H
Enolform
Ketoform
1.3.) Verantwortlich für den räumlichen Bau eines Polypeptids ist die Tertiärstruktur.
Stabilisiert wird die Tertiärstruktur durch:
o Esterbindungen zwischen freien Carboxylgruppen in Seitenketten und freien
Hydroxylgruppen.
O
R1
C
O
R2
o Peptidbindungen zwischen freien Carboxylgruppen in den Seitenketten und freien
Aminogruppen in den Seitenketten.
O
R1
C
N
R2
H
o Disulfidbrücken zwischen Cysteinresten.
R1
S
S
R2
o Ionale Bindungen zwischen COO- -Gruppen und NH3+-Gruppen.
H
O
R1
C
O
H
N
+
R2
H
o Wasserstoffbrückenbindungen zwischen stark positiv polarisierten
Wasserstoffatomen und stark elektronegativen Sauerstoffatomen.
R1
H
O
O
H
R2
o Van-der-Waal´sche Anziehungskräfte zwischen unpolaren Seitengruppen.
H
H
R1
C
H
H
C
R2
H
H
82/III/2
2.1.) Serin hat einen IEP von 5,68. Da Serin eine saure Hydroxy-Gruppe im Rest aufweist, die
polarisiert werden kann.16 Valin hat einen IEP von 5,96. Es handelt sich um eine Aminosäure
mit unpolarem Rest.3 Lysin hat einen IEP von 9,74. Sie enthält eine zweite Amino-Gruppe
im Rest. Diese NH2-Gruppe sorgt für den stark basischen IEP. Bei der Reaktion mit einer
Säure nimmt zuerst diese Amino-Gruppe im Rest ein Proton auf, bevor die COO--Gruppe ein
Proton aufnimmt. Deshalb liegt Lysin bei einem stark sauerem pH-Wert als doppeltes Kation
vor.
Die Struktur der Aminosäuren bei einem pH = 9,74:
o Der IEP von Serin ist niedriger als der gegebene pH-Wert. Serin liegt als Anion vor:
O
H2N
H
C
C
O
CH2
OH
o Der IEP von Valin ist niedriger als der gegebene pH-Wert. Valin liegt als Anion vor:
O
H2N
H
C
HC
CH3
C
CH3
O
o Der IEP von Lysin ist gleich dem gegebenem pH-Wert. Lysin liegt als Zwitterion vor:
O
+
H3N
H
C
C
O
CH2
CH2
CH2
CH2
NH2
2.2.) Skizze 1 (von der Seite):
Minus-Pol
Plus-Pol
Gleichstrom
Trägerstreifen
(aus Papier)
Elektrolytlösung
(Pufferlösung)
Elektrode
Skizze 2 (von oben):
Anion
Minus-Pol
Plus-Pol
Zwitterion
Kation
Startlinie
Bei der Elektrophorese dient die Pufferlösung zum Einstellen und Aufrechterhalten eines
konstanten pH-Wertes. Ein Gemisch aus verschiedenen Aminosäuren wird auf die Startlinie
des Trägerstreifens aufgebracht. Je nach dem spezifischen IEP der Aminosäuren, liegen diese
beim eingestellten pH-Wert entweder als Anion, Kation oder Zwitterion vor. Beim
Einschalten des Gleichstroms wandern die Aminosäuren entsprechend ihrer Ladung nun zum
Plus-Pol, Minus Pol oder bleiben auf der Startlinie stehen. Wanderungsrichtung und geschwindigkeit der Aminosäuren hängen von ihrer Ladung, ihrer Größe und der Stärke der
angelegten Spannung ab.4
Bei einem pH = 9,74 bleibt Lysin als Zwitterion auf der Startlinie stehen. Serin und Valin
liegen als Anionen vor und wandern Richtung Plus-Pol. Da die Molekülmasse von Serin
kleiner ist als die von Valin, wandert Serin schneller und weiter als Valin.
83/I/4
4.) KS (Glycin): 1,6 · 10-10
KB (Glycin): 2,5 · 10-12
KS (Essigsäure): 1,75 · 10-5
KS (Monochlorethansäure): 1,36 · 10-3
4.1.) In wässriger Lösung liegt Glycin als Zwitterion vor. Es weist einen ampholyten
Charakter auf.
{
O
H2N
C
H2
C
O
+
OH
H3N
C
H2
C
O
Zwitterion
}
Bei Zugabe von Base reagiert die NH3+-Gruppe als Säuregruppe, auf sie bezieht sich der KSWert.
O
+
H3N
C
H2
C
O
O
+
Zwitterion
OH
H2N
C
H2
C
O
+
Anion
Base
H2O
Wasser
Bei Zugabe von Säure reagiert die COO--Gruppe als basische Gruppe, auf sie bezieht sich der
KB-Wert.
O
+
H3N
C
H2
C
O
O
Zwitterion
+
H3O
+
Säure
+
H3N
C
H2
C
OH
Kation
+
H2O
Wasser
Rechnung:
Nach Definition liegt fest, dass KS · KB = 10-14 beträgt.
Da KS (Glycin) = 1,6 · 10-10 gilt: 1,6 · 10-10 · KB = 10-14 : (1,6 · 10-10)
KB =
10 −14
= 6,25 ⋅ 10 −5
−10
1,6 ⋅ 10
Da KB (Glycin) = 2,5 · 10-12 gilt: 2,5 · 10-12 · KS = 10-14 : (2,5 · 10-12)
KS =
10 −14
= 4 ⋅ 10 −3
2,5 ⋅ 10 −12
4.2.) Glycin: KS (COOH) = 4 · 10-3
Essigsäure: KS (COOH) = 1,75 · 10-5
Der KS-Wert von Glycin ist größer als der KS-Wert von Essigsäure. Die COOH-Gruppe von
Glycin ist demnach stärker sauer als die COOH-Gruppe von Essigsäure. Grund dafür ist, dass
die NH2-Gruppe des Glycins einen starken –I-Effekt auf die COOH-Gruppe ausübt. Dadurch
wird das Anion leicht gebildet, und zusätzlich stabilisiert. Bei der Essigsäure hat der +-IEffekt der Methylgruppe einen gegenteiligen Effekt. Die Abgabe des Protons wird erschwert,
und das Anion kann nur unter hohem Energieaufwand gebildet werden.6
84/III/3
3.1.) Merkmal eines ampholyten Moleküls ist, dass es sowohl als Säure, als auch als Base
reagieren kann. Das heißt es kann sowohl Protonen abgeben, als auch aufnehmen. In
wässriger Lösung liegt Glycin überwiegend als Zwitterion vor.
{
O
H2N
C
H2
C
O
+
H3N
OH
C
H2
C
O
}
Zwitterion
Beim Zwitterion des Glycins dient die NH3+- Gruppe als Säuregruppe, sie kann bei der
Reaktion mit einer Base Protonen abgeben.
O
+
H3N
C
H2
C
O
O
+
Zwitterion
OH
H2N
C
H2
C
O
+
Anion
Base
H2O
Wasser
Die COO--Gruppe des Glycins fungiert als basische Gruppe, sie kann bei der Reaktion mit
einer Säure Protonen aufnehmen.3
O
+
H3N
C
H2
C
O
O
+
Zwitterion
H3O
+
Säure
+
H3N
C
H2
C
OH
Kation
+
H2O
Wasser
3.2.) Isoelektrischer Punkt (IEP) = Der pH-Wert an dem eine Aminosäure überwiegend als
Zwitterion vorliegt.
IEP (Glycin): 6,0
IEP (Glutaminsäure): 3,22
IEP (Lysin): 9,74
Glu
Minus-Pol
Plus-Pol
Gly
Lys
Startlinie
(Pufferlösung eingestellt auf den pH = 6)
Wanderungsrichtung: Glycin:
Der IEP von Glycin entspricht exakt dem eingestellten pH-Wert der Pufferlösung, d.h.
Glycin liegt als Zwitterion vor und bleibt auf der Startlinie stehen.
Glutaminsäure:
Der IEP von Glutaminsäure liegt unterhalb des eingestellten pH-Wertes der Pufferlösung. Es
wird Base zum Zwitterion hinzugegeben. Glutaminsäure gibt zwei Protonen ab, und liegt
somit als doppeltes Anion vor. Die Glutaminsäure wandert zum Plus-Pol.
1. Schritt:
O
O
HO
C
C
CH2
O
+
OH
O
CH2
O
H2O
H2C H C
C
O
H2C H C
C
O
+
NH3
+
NH3
Wasser
Anion
Base
Zwitterion
2.Schritt:
O
O
C
O
C
CH2
O
H2C H C
O
C
+
OH
Lysin:
CH2
O
H2C H C
O
C
+
H2O
NH2
+
NH3
Anion
O
Base
doppeltes Anion
Wasser
Der IEP von Lysin liegt über dem eingestellten pH-Wert der Pufferlösung. Das Zwitterion
wird angesäuert und nimmt zwei Protonen auf. Lysin liegt somit als doppeltes Kation vor, und
wandert zum Minus-Pol.
1. Schritt:
+
H2N
H3N
CH2
H2C
CH2
+
+
O
CH2
H2C
H3O
CH2
+
O
H2O
H2C H C
C
O
H2C H C
C
O
+
+
NH3
NH3
Zwitterion
Kation
Säure
Wasser
2. Schritt:
+
+
H3N
H3N
CH2
H2C
CH2
+
O
+
H3O
CH2
H2C
CH2
+
O
H2C H C
C
OH
H2C H C
C
O
+
+
NH3
NH3
Wasser
doppeltes Kation
Säure
Kation
84/III/4
4.1.)
O
H2
C
C
*
C
H2
H2
C
C
H2
H2
C
H
N
C
C
H2
O
Peptidbindung
4.2.)
H2O
H2
C
C
H2
H2
C
C
H2
N
H
*
n
H
R
C
N
R
O
Die Bindungswinkel betragen 120°. Die Reste sind in Transform angeordnet. Diese
Konfiguration wird bevorzugt, da die Reste einen großen Raumbedarf haben, und so
möglichst weit voneinander entfernt sind. Die Peptidbindung ist planar und starr, die Bindung
ist also um die eigene Achse nicht frei drehbar. Grund hierfür ist, dass das Kohlenstoffatom,
das Stickstoffatom und das Sauerstoffatom der Peptidbindung sp2-hybridisiert sind. Die
Valenzstriche in der Skizze kennzeichnen die Sigmabindungen zwischen den Atomen. Die
freien py-Orbitale überlappen sich zu einer delokalisierten ð-Elektronenwolke. Die
Delokalisation der Elektronen führt zur Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
C
O
+
N
R1
R2
C
R2
}
O
Ketoform
N
R1
R2
C
O
H
Enolform
Die Bindungsverhältnisse in der Peptidbindung sind somit nicht klar definiert. Als Folge
weist die C-O-Doppelbindung (Länge: 123 pm) der Ketoform einen
Einfachbindungscharakter auf, das bedeutet sie ist länger als eine normale Doppelbindung
(Länge: 122 pm). Die C-N-Einfachbindung (Länge: 132 pm) der Ketoform hingegen weist
einen Doppelbindungscharakter auf, sie ist kürzer als eine normale Einfachbindung (Länge:
147 pm).1
4.3.1.)
O
*
H2
C
H2
C
C
O
C
H2
C
H2
C
H2
C
O
C
H2
O
n
*
Esterbindung
4.3.2.) Der Reaktionsmechanismus der dieser Reaktion zugrunde liegt ist die
säurekatalytische Esterbildung:
Ausgangsprodukte:
O H
2
HO C C
R1
C
H2
H2
C
H2
HO C
O
C C OH
H2
R2
C OH
H2
Ethandiol
Hexandisäure
1. Ausbildung der mesomeren Grenzformel:
{
O
R1
O
C OH
OH
+
R1
C OH
}
+
H
+
R1
+
C OH
Katalysator
2. Addition von Alkohol:
OH
R1
+
C OH
OH
+
HO R2
R1
H
C OH
O
+
R2
3. Intermolekulare Protonenwanderung und Wasserabspaltung:
OH
R1
H
C OH
O
OH
R1
+
OH
+
C O H
R1
+
C O R2
+
H2O
O H
R2
R2
4. Rückführung des Katalysators und Mesomeriestabilisierung:
OH
R1
86/II/2
+
C O R2
{
O
O
R1
+
C O R2
R1
C O R2
}
+
H
+
Katalysator
2.1.) Das Protein wird durch die Salzsäure säurehydrolytisch in die einzelnen Aminosäuren
gespalten.1
R
R
*
C
N H
C
H
O
n
*
+
n H3O
+
n
Cl
C
H
+
Cl
H3N
C
OH
O
2.2.) Definition: Isoelektrischer Punkt = Der pH-Wert an dem eine Aminosäure in wässriger
Lösung überwiegend als Zwitterion vorliegt.
Aminosäuren liegen in wässriger Lösung als Zwitterion vor.
H
H
{
O
H2N
C
O
+
H3N
C
C
C
O
OH
}
R
R
Die COO--Gruppe fungiert als basische Gruppe, und fängt bei Zugabe von Säure H+-Ionen ab.
O
O
+
H3N C C O
H2
+
H3O
+
+
H3N C C OH
H2
Säure
Zwitterion
+
Kation
H2O
Wasser
Die NH3+-Gruppe fungiert als Säuregruppe, und gibt bei Zugabe von Base H+-Ionen ab. Diese
H+-Ionen reagieren mit den OH--Ionen zu Wasser.3
O
O
+
H3N C C O
H2
Zwitterion
+
OH
H2N C C O
H2
Base
Anion
+
H2O
Wasser
So ändert sich der pH-Wert einer Aminosäurelösung durch Zugabe von Base und Säure nur
wenig. Aminosäuren haben eine Pufferwirkung.8
2.3.)
O
*
C
H2
C
H2
N
H
H2
C
H2
C
H2
C
H2
C
H
N
C
H2
C
C
H2
C
H2
C
C
H2
n
*
O
Peptidbindung
In beiden Fällen handelt es sich um Makromoleküle deren Bausteine durch Peptidbindungen
verknüpft sind. Diese Peptidbindungen sind starr und planar. Die Bindungswinkel betragen
120°. Die C-, N- und O-Atome der Peptidbindung sind sp2-hybridisiert. Die Atome sind durch
Sigmabindungen verknüpft. Die freien ð-Orbitale überlappen sich zu einer delokalisierten
Elektronenwolke, was zur Ausbildung von mesomeren Grenzformeln führt.1 Durch
Ausbildung von intermolekularen Wasserstoffbrückenbindungen können sich mehrere
Makromoleküle zu größeren Strukturen aneinanderlagern.9
*
N
R1
H
C
H
N
H
C
H
C
O
R2
O
R3
C
C
H
N
*
C
n
O
H
Wasserstoffbrückenbindung
H
N
*
87/I/3
O
R2
H
O
C
H
C
N
C
C
H
N
C
C
H
R1
H
O
R3
3.1.) Skizze 1 (von der Seite):
Minus-Pol
Plus-Pol
Elektrode
Gleichstrom
Trägerstreifen
(aus Papier)
Elektrolytlösung
(Pufferlösung)
Skizze 2 (von oben):
Anion
Minus-Pol
Zwitterion
Plus-Pol
Kation
Startlinie
Bei der Elektrophorese dient die Pufferlösung zum Einstellen und Aufrechterhalten eines
konstanten pH-Wertes. Ein Gemisch aus verschiedenen Aminosäuren wird auf die Startlinie
des Trägerstreifens aufgebracht. Je nach dem spezifischen IEP der Aminosäuren, liegen diese
beim eingestellten pH-Wert entweder als Anion, Kation oder Zwitterion vor. Beim
Einschalten des Gleichstroms wandern die Aminosäuren entsprechend ihrer Ladung nun zum
Plus-Pol, Minus Pol oder bleiben auf der Startlinie stehen. Wanderungsrichtung und -
geschwindigkeit der Aminosäuren hängen von ihrer Ladung, ihrer Größe und der Stärke der
angelegten Spannung ab.4
3.2.) Alanin:
Der IEP von Alanin muss dem eingestellten pH-Wert der Pufferlösung entsprechen, damit
liegt Alanin als Zwitterion vor und bleibt auf der Startlinie stehen.
Glutaminsäure:
Glutaminsäure weist zwei Carboxylgruppen auf. Der IEP von Glutaminsäure liegt unterhalb
des eingestellten pH-Wertes der Pufferlösung. Es wird Base zum Zwitterion hinzugegeben.
Dadurch gibt Glutaminsäure zwei Protonen ab, und liegt als doppeltes Anion vor. Die
Glutaminsäure wandert zum Plus-Pol.
1. Schritt:
O
O
HO
C
C
CH2
O
+
OH
O
CH2
O
H2O
H2C H C
C
O
H2C H C
C
O
+
NH3
+
NH3
Wasser
Anion
Base
Zwitterion
2.Schritt:
O
O
C
O
C
CH2
O
H2C H C
C
O
+
OH
O
CH2
H2C H C
C
O
+
H2O
NH2
+
NH3
Anion
O
Base
doppeltes Anion
Wasser
Lysin:
Lysin weist zwei Aminogruppen auf. Der IEP von Lysin liegt somit über dem eingestellten
pH-Wert der Pufferlösung. Das Zwitterion wird angesäuert und nimmt zwei Protonen auf.
Lysin liegt als doppeltes Kation vor, und wandert zum Minus-Pol.
1. Schritt:
+
H2N
H3N
CH2
H2C
CH2
O
+
+
H3O
CH2
H2C
CH2
+
O
H2O
H2C H C
C
O
H2C H C
C
O
+
+
NH3
NH3
Zwitterion
Kation
Säure
Wasser
2. Schritt:
+
+
H3N
H3N
CH2
H2C
CH2
O
+
+
H3O
H2C
CH2
O
+
H2O
H2C H C
C
OH
H2C H C
C
O
+
+
NH3
NH3
Kation
CH2
Säure
doppeltes Kation
Wasser
3.3.) Es gibt sechs mögliche Aminosäuresequenzen:
Ala-Glu-Lys
Ala-Lys-Glu
Glu-Ala-Lys
Glu-Lys-Ala
Lys-Ala-Glu
Lys-Glu-Ala
88/II/3
3.1.) D- und L-Konfiguration bezeichnen die Stellung der Aminogruppe am chiralen
Kohlenstoffatom einer Aminosäure in der Fischerprojektion. Dabei steht D (lat.: Dextro) für
rechts, und L (lat.: Laevus) für links.10
HO
HO
O
H
O
H
H
C*
NH2
H
C*
NH2
C
H
H
C
H
H
C
H
H
C
H
H
L-Alanin
D -Alanin
C
HO
O
C
C*
H
HO
C
C
C
H2N
HO
O
C*
H
H
C
H
H
C
H
H
C
H
H
C
H
H2N
O
D - G lu ta m in s ä u re
NH2
L-Ala n in
3.2.)
CH3NH2
CH2
CH2
H
CH3
H
H
+
N
H
C
H
N
O
CH2
H
C
C
H
N
C
H
C
O
CH2
CH2
COOH
N
H
C
O
O
C
C
H
CH3
O
3.3.)
H
R
N
C
R
O
Die Aminosäuren sind über Säureamidbindungen, so genannten Peptidbindungen,
miteinander verknüpft. Diese Bindungen sind planar und starr. Die Bindungswinkel betragen
120°. Grund dafür ist, dass die C-, N- und O-Atome der Säureamidbindung sp²-hybridisiert
sind. Die Atome sind über Sigmabindungen verknüpft, die freien py-Orbitale überlappen sich
zu einer delokalisierten ð-Elektronenwolke. Die Delokalisation der Elektronen sorgt für die
Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
C
O
Ketoform
+
N
R1
R2
C
O
R2
}
N
R1
R2
C
O
H
Enolform
Auch weist die C-O-Doppelbindung der Peptidbindung einen Einfachbindungscharakter. Sie
ist länger als eine normale Doppelbindung. Die C-N-Einfachbindung dagegen weist
Doppelbindungscharakter auf. Sie ist kürzer als eine normale Einfachbindung.1
3.4.) Das Molekül Biuret zeigt bei einer Reaktion mit Cu²+-Ionen im Basischem eine
charakteristische rot-violette Färbung. Dafür verantwortlich ist die Komplexbildung der Cu²+Ionen mit den Säureamidbindungen, die in Biuret enthalten sind. Auch die Aminosäuren in
Proteinen sind über Säureamidbindungen verknüpft. Diese CO-NH-Bindungen können mit
Cu²+-Ionen ebenfalls einen Komplex bilden, der zu einer rot-violetten Färbung führt.11
89/IV/3
3.1.)
*
N
R1
H
C
H
N
H
C
H
C
O
R2
O
R3
C
C
H
N
*
C
n
O
H
3.2.)
H
C
0,147 nm
C
N
C
O
0,132 nm
Die Bindungswinkel betragen 120°. Die Reste sind in Transform angeordnet. Diese
Konfiguration wird bevorzugt, da die Reste einen großen Raumbedarf haben, und so
möglichst weit voneinander entfernt sind. Die Peptidbindung ist planar und starr, die Bindung
ist also um die eigene Achse nicht frei drehbar. Grund hierfür ist, dass das Kohlenstoffatom,
das Stickstoffatom und das Sauerstoffatom der Peptidbindung sp2-hybridisiert sind. Die
Valenzstriche in der Skizze kennzeichnen die Sigmabindungen zwischen den Atomen. Die
freien py-Orbitale überlappen sich zu einer delokalisierten ð-Elektronenwolke. Die
Delokalisation der Elektronen führt zur Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
C
O
Ketoform
+
N
R1
R2
C
O
R2
}
N
R1
R2
C
O
H
Enolform
Die Bindungsverhältnisse in der Peptidbindung sind somit nicht klar definiert. Als Folge
weist die C-O-Doppelbindung (Länge: 123 pm) der Peptidbindung einen
Einfachbindungscharakter auf, das bedeutet sie ist länger als eine normale Doppelbindung
(Länge: 122 pm). Die C-N-Einfachbindung (Länge: 132 pm) der Peptidbindung hingegen
weist einen Doppelbindungscharakter auf, sie ist kürzer als eine normale Einfachbindung
(Länge: 147 pm). Die Bindung des Stickstoffs mit dem C-Atom das nicht an der
Peptidbindung beteiligt ist entspricht in ihrer Länge einer normalen Einfachbindung (Länge:
147pm). Da dieses C-Atom nicht von der delokalisierten Elektronenwolke betroffen ist,
werden seine Bindungsverhältnisse durch die Ausbildung von Mesoformen nicht beeinflusst.1
3.3.) Verantwortlich für den räumlichen Bau eines Polypeptids ist die Tertiärstruktur.
Stabilisiert wird die Tertiärstruktur durch:2
o Esterbindungen zwischen freien Carboxylgruppen in Seitenketten und freien
Hydroxylgruppen.
O
R1
C
O
R2
o Peptidbindungen zwischen freien Carboxylgruppen in den Seitenketten und freien
Aminogruppen in den Seitenketten.
O
R1
C
N
R2
H
o Disulfidbrücken zwischen Cysteinresten.
R1
S
S
R2
o Ionale Bindungen zwischen COO- -Gruppen und NH3+-Gruppen.
H
O
R1
C
O
H
N
+
R2
H
o Wasserstoffbrückenbindungen zwischen stark positiv polarisierten
Wasserstoffatomen und stark elektronegativen Sauerstoffatomen.
R1
H
O
O
H
R2
o Van-der-Waal´sche Anziehungskräfte zwischen unpolaren Seitengruppen.
H
H
R1
C
H
H
C
R2
H
H
90/IV/3
3.1.) Skizze 1 (von der Seite):
Minus-Pol
Plus-Pol
Elektrode
Gleichstrom
Trägerstreifen
(aus Papier)
Elektrolytlösung
(Pufferlösung)
Skizze 2 (von oben):
Anion
Minus-Pol
Zwitterion
Kation
Startlinie
Plus-Pol
Bei der Elektrophorese dient die Pufferlösung zum Einstellen und Aufrechterhalten eines
konstanten pH-Wertes. Ein Gemisch aus verschiedenen Aminosäuren wird auf die Startlinie
des Trägerstreifens aufgebracht. Je nach dem spezifischen IEP der Aminosäuren, liegen diese
beim eingestellten pH-Wert entweder als Anion, Kation oder Zwitterion vor. Beim
Einschalten des Gleichstroms wandern die Aminosäuren entsprechend ihrer Ladung nun zum
Plus-Pol, Minus Pol oder bleiben auf der Startlinie stehen. Wanderungsrichtung und geschwindigkeit der Aminosäuren hängen von ihrer Ladung, ihrer Größe und der Stärke der
angelegten Spannung ab.4
3.2.) Glutaminsäure hat einen IEP von 3,22. Bei einem pH = 7,0 liegt Glutaminsäure als
doppeltes Anion vor, und wandert bei der Elektrophorese Richtung Plus-Pol.16
1. Schritt:
O
O
HO
C
C
CH2
O
+
O
H2O
CH2
H2C H C
C
O
H2C H C
C
O
+
H3O
+
+
NH3
+
NH3
Zwitterion
O
Anion
Wasser
Säure
2.Schritt:
O
O
C
O
C
CH2
O
H2C H C
C
O
+
H2O
O
O
H2C H C
C
O
+
H3O
+
NH2
+
NH3
Anion
CH2
Wasser
doppeltes Anion
Säure
91/III/4
4.1.) Bei der Spaltung des Peptids wurden zwei verschiedene Enzyme verwendet. Durch die
zwei unterschiedlichen enzymatischen Spaltprozesse entstehen unterschiedliche Teilstücke.
Durch die Überlappung gemeinsamer Sequenzanteile in den Bruchstücken, lässt sich die
ursprüngliche Aminosäuresequenz des Peptids rekonstruieren.
Tryptische Peptidfragmente:
Thr-Tyr-Val-Lys
Ala-Gly-Trp-Gly-Lys
Chymotryptische Peptidfragmente:
Thr-Tyr
Val-Lys-Ala-Gly-Trp
Gly-Lys
Ursprüngliche Peptidsequenz:
Thr-Tyr-Val-Lys-Ala-Gly-Trp-Gly-Lys
4.2.) Unter Kettenkonformation versteht man die Raumstruktur die Proteine einnehmen.17 Die
Kettenkonformation entspricht der Sekundärstruktur. Sie wird durch
Wasserstoffbrückenbindungen stabilisiert. Diese Wasserstoffbrückenbindungen bilden sich
zwischen dem Sauerstoffatom einer CO-Gruppe und dem Wasserstoffatom einer NH-Gruppe
aus.
O
C
R
N
R
H
Wasserstoffbrückenbindung
O
R
C
R
N
H
Man unterscheidet zwischen zwei bevorzugten Raumstrukturen:
o Die α -Helix: Eine schraubenförmige Struktur die durch die Ausbildung von
intramolekularen Wasserstoffbrückenbindungen entsteht.
o Die Faltblattstruktur: Diese Struktur kommt durch die Ausbildung von
intermolekularen Wasserstoffbrückenbindungen zwischen gestreckten
Polypeptidketten zustande.
Ob sich die α -Helix-Struktur oder die Faltblattstruktur ausbildet, hängt ausschließlich von
der Aminosäuresequenz, auch Primärstruktur genannt, des Proteins ab.9
92/III/4.2
4.2.1.) Um die Biuretreaktion durchzuführen, gibt man Kupfersulfat und Natronlauge im
Überschuss zur Naturseide hinzu. Es zeigt sich bei der Reaktion mit Cu²+-Ionen im Basischen
eine charakteristische rot-violette Färbung. Dafür verantwortlich ist die Komplexbildung der
Cu²+-Ionen mit den Säureamidbindungen.
4.2.2.) Da die Biuretreaktion mit Naturseide positiv verläuft, muss Naturseide
Säureamidbindungen aufweisen. Bei Naturseide handelt es sich daher um ein Protein.1
*
N
H
R1
H
C
H
N
C
H
C
O
R2
O
R3
C
C
H
N
H
*
C
n
O
93/III/3
3.1.1.) Die Lösung ist im stark sauren Milieu klar. Durch die Zugabe von Base wird die
Lösung langsam trüb. Es bildet sich ein Niederschlag. Nachdem der neutrale pH = 7
überschritten ist, beginnt sich der Niederschlag aufzulösen. Im stark Basischen ist die Lösung
wieder klar. Grund ist, dass Tyrosin eine Aminosäure mit polarem, ungeladenem Rest ist. Ihr
IEP liegt bei 5,66. Im stark Sauren liegt Tyrosin als Kation vor, welches gut in Wasser löslich
ist. Durch die Zugabe von Base erhöht sich die Zwitterionenkonzentration. Die Zwitterionen
ballen sich zusammen, und fallen als Niederschlag aus. Bei einem neutralen pH-Wert liegt
Tyrosin fast vollständig als Zwitterion vor. Durch eine weitere Basenzugabe bilden sich
Anionen. Die Anionen sind wieder gut in Wasser löslich. Die Lösung klart auf. Im stark
Basischen liegt Tyrosin fast vollständig als Anion vor.3
3.1.2.) Es gilt je kleiner der pKs-Wert, desto stärker ist die Säure.6 Da die COOH-Gruppe den
kleinsten pKs-Wert hat, gibt sie ihr Proton zuerst ab. Darauf erfolgt die Protonenabgabe der
NH3+-Gruppe. Da die OH-Gruppe den größten pKs-Wert hat, ist sie die schwächste
Säuregruppe. Sie gibt ihr Proton zuletzt ab.
O
H
C C C
H2
+
NH3 OH
HO
Kation
HO
Zwitterion
O
H
C C C
H2
+
NH3 O
O
H
C C C
H2
NH2 O
HO
Anion
+
H2O
Anion
H2O
H2O
O
3.2.1.)
+
H3N
H
C
H
CH3
C
C
H
N
H
O
H
N
C
H
C
O
HC
C
O
CH3
CH3
Peptidbindung
3.2.2.)
H
R
C
O
H3O
+
Säure
N
R
+
O
H
C C C
H2
NH2 O
doppeltes Anion
O
+
Wasser
+
+
Zwitterion
H3O
+
+
H2O
Die Bindungswinkel betragen 120°. Die Reste sind in Transform angeordnet. Diese
Konfiguration wird bevorzugt, da die Reste einen großen Raumbedarf haben, und so
möglichst weit voneinander entfernt sind. Die Peptidbindung ist planar und starr, die Bindung
ist also um die eigene Achse nicht frei drehbar. Grund hierfür ist, dass das Kohlenstoffatom,
das Stickstoffatom und das Sauerstoffatom der Peptidbindung sp2-hybridisiert sind. Die
Valenzstriche in der Skizze kennzeichnen die Sigmabindungen zwischen den Atomen. Die
freien py-Orbitale überlappen sich zu einer delokalisierten ð-Elektronenwolke. Die
Delokalisation der Elektronen führt zur Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
C
+
N
R1
R2
R2
C
O
N
R1
}
R2
C
O
O
H
Enolform
Ketoform
Die Bindungsverhältnisse in der Peptidbindung sind somit nicht klar definiert. Als Folge
weist die C-O-Doppelbindung (Länge: 123 pm) der Peptidbindung einen
Einfachbindungscharakter auf, das bedeutet sie ist länger als eine normale Doppelbindung
(Länge: 122 pm). Die C-N-Einfachbindung (Länge: 132 pm) der Peptidbindung hingegen
weist einen Doppelbindungscharakter auf, sie ist kürzer als eine normale Einfachbindung
(Länge: 147 pm).1
93/III/4
Bei der Xanthoproteinreaktion handelt es sich um eine Substitutionsreaktion. Ein
Wasserstoffatom am Benzolring wird durch eine Nitrogruppe ersetzt:12
Die Nitrierung von Phenol:13
o Startreaktion:
NO2 +
2 HNO3
+
NO3
-
+
H 2O
o Bildung des ð-Komplexes:
+
OH
NO2 +
HO
NO2 +
o Addition und Protonenabspaltung:
C
+
HO
+
HO
O2N
+
H
O2N
H
o Regeneration des Ausgangsprodukts:
+
+
H
NO3
-
HNO3
Bevorzugt wird ein Wasserstoffatom in ortho-Stellung substituiert:
HO
O2N
Es entsteht jedoch auch ein para-Nitrophenol:
HO
NO2
94/IV/2
2.1.)
O
O
+
H3N
C
H2
C
O
Zwitterion
H2N
C
H2
C
O
Anion
Bei einem pH = 9,60 gilt: n (Zwitterion) : n (Anion) = 1 : 1
Der pH-Wert steigt von 5,97 auf 9,60 => 9,60 – 5,97 = 3,63
Um den gleichen Wert sinkt der pH-Wert wenn gilt: n (Kation) : n (Zwitterion) = 1 : 1
Der pH-Wert sinkt auf => 5,97 – 3,63 = 2,34
2.2.) Bestimmung von Ks- und KB-Wert:14
Ks (COOH):
Bei einem pH-Wert von 2,34 gilt n (Kation) : n (Zwitterion) = 1 : 1
Vergleichbar der Halbtitration gilt in diesem Fall pH = pKs => pKs = 2,34
Da pKs = – log Ks => Ks = 10 – pKs => Ks = 10 – 2,34 = 4,57 · 10 – 3 mol/l
KB (NH2):
Bei einem pH-Wert von 9,60 gilt n (Zwitterion) : n (Anion) = 1 : 1
In diesem Fall gilt pOH = 14 – pH = pKB => pKB = 4,40
Da pKB = – log KB => KB = 10 – pKB => KB = 10 – 4,40 = 3,98 · 10 – 5 mol/l
2.3.) Die NH2-Gruppe des Glycins übt einen starken –I-Effekt auf die COOH-Gruppe aus.
Dadurch wird das Anion leicht gebildet, und zusätzlich stabilisiert. Bei der Essigsäure hat der
+-I-Effekt der Methylgruppe einen gegenteiligen Effekt. Die Abgabe des Protons wird
erschwert, und das Anion kann nur unter hohem Energieaufwand gebildet werden. Der KSWert von Glycin ist größer als der KS-Wert von Essigsäure. Die COOH-Gruppe von Glycin
ist demnach stärker sauer als die COOH-Gruppe von Essigsäure.6
Glycin:
O
H2N
C
H2
C
OH
Essigsäure:
O
H 3C
C
OH
95/I/3
3.1.) Alanin ist eine Aminosäure mit unpolarem, aliphatischem Rest. Die Acidität der SäureGruppe überwiegt die Basizität der Aminogruppe, der IEP liegt im leicht saueren Bereich.
Auf sie bezieht sich der gegebene IEP von 6,00.3 Asparaginsäure weist eine zweite CarboxylGruppe im Rest auf. Bei der Zugabe von Base zum Zwitterion gibt zuerst diese CarboxylGruppe ein Proton ab, erst danach erfolgt die Protonenabgabe der NH3+-Gruppe. Im
Basischen liegt Asparaginsäure als doppeltes Anion vor. Dadurch ist der IEP von
Asparaginsäure sehr niedrig. Auf sie bezieht sich der gegebene IEP von 2,77.16
3.2.) Skizze 1 (von der Seite):
Minus-Pol
Plus-Pol
Elektrode
Gleichstrom
Trägerstreifen
(aus Papier)
Elektrolytlösung
(Pufferlösung)
Skizze 2 (von oben):
Anion
Minus-Pol
Zwitterion
Plus-Pol
Kation
Startlinie
Bei der Elektrophorese dient die Pufferlösung zum Einstellen und Aufrechterhalten eines
konstanten pH-Wertes. Ein Gemisch aus verschiedenen Aminosäuren wird auf die Startlinie
des Trägerstreifens aufgebracht. Je nach dem spezifischen IEP der Aminosäuren, liegen diese
beim eingestellten pH-Wert entweder als Anion, Kation oder Zwitterion vor. Beim
Einschalten des Gleichstroms wandern die Aminosäuren entsprechend ihrer Ladung nun zum
Plus-Pol, Minus Pol oder bleiben auf der Startlinie stehen. Wanderungsrichtung und geschwindigkeit der Aminosäuren hängen von ihrer Ladung, ihrer Größe und der Stärke der
angelegten Spannung ab.4
Man stellt einen pH = 6 ein. Beim gegebenen pH-Wert liegt Alanin als Zwitterion vor, und
bleibt auf der Startlinie stehen.
Der IEP von Asparaginsäure liegt unterhalb des eingestellten pH-Wertes. Die Aminosäure
gibt ein Proton ab, und wandert als Anion zum Plus-Pol.
3.3.1.) Es gibt sechs mögliche Aminosäuresequenzen, die durch die Verknüpfung von Alanin
und Serin zu einem Tripeptid entstehen können:
Ala-Ala-Ser
Ala-Ser-Ala
Ser-Ala-Ala
Ser-Ser-Ala
Ser-Ala-Ser
Ala-Ser-Ser
3.3.2.) Die Strukturformel eines Tripeptids mit der Aminosäuresequenz Ala-Ala-Ser
OH
+
H3N
CH3
H
C
H
N
C
H
C
O
CH3
O
CH2
C
C
H
N
H
C
O
O
96/I/3
3.1.) Im Abschnitt (I) liegt Glycin als Kation vor:
O
+
H3N
C
H2
C
OH
Im Abschnitt (V) liegt Glycin als Anion vor:
O
H2N
C
H2
C
O
3.2.1.) Ermittlung von Ks und KB-Wert:14
Ks (COOH):
Aus dm 1. Wendepunkt wird ersichtlich, dass bei einem pH-Wert von 2,34 gilt
n (Kation) : n (Zwitterion) = 1 : 1
Vergleichbar der Halbtitration gilt in diesem Fall pH = pKs => pKs = 2,34
Da pKs = – log Ks => Ks = 10 – pKs => Ks = 10 – 2,34 = 4,57 · 10 – 3 mol/l
KB (NH2):
Aus dem 3. Wendepunkt wird ersichtlich, dass bei einem pH-Wert von 9,60 gilt
n (Zwitterion) : n (Anion) = 1 : 1
In diesem Fall gilt pOH = 14 – pH = pKB => pKB = 4,40
Da pKB = – log KB => KB = 10 – pKB => KB = 10 – 4,40 = 3,98 · 10 – 5 mol/l
3.2.2.) Bei einem pH = 2,34 und bei einem pH = 9,60 hat die Aminosäure eine Pufferwirkung.
Der Kurvenverlauf ändert sich in diesen pH-Bereichen nur langsam. Was bedeutet, dass sich
der pH-Wert der Aminosäurelösung in diesen Bereichen durch Zugabe einer begrenzten
Menge an Säure oder Base nur geringfügig ändert.8
3.3.) Isoelektrischer Punkt (IEP) = Der pH-Wert an dem eine Aminosäure fast ausschließlich
in zwitterionischer Form vorliegt.3
Aus dem Diagramm lässt sich ein IEP (Glycin) = 5.97 ablesen. Rechnerisch lässt sich der IEP
mit der Formel
pKs1 + pKs2
bestimmen.14
2
Da pKs1 (Glycin) = 2,34 und pKs2 (Glycin) = 9,60 => IEP (Glycin) =
2,34 + 9,60
= 5,97
2
97/III/2
2.1.) Skizze 1 (von der Seite):
Minus-Pol
Plus-Pol
Elektrode
Gleichstrom
Trägerstreifen
(aus Papier)
Elektrolytlösung
(Pufferlösung)
Skizze 2 (von oben):
Anion
Minus-Pol
Zwitterion
Plus-Pol
Kation
Startlinie
Bei der Elektrophorese dient die Pufferlösung zum Einstellen und Aufrechterhalten eines
konstanten pH-Wertes. Ein Gemisch aus verschiedenen Aminosäuren wird auf die Startlinie
des Trägerstreifens aufgebracht. Je nach dem spezifischen IEP der Aminosäuren, liegen diese
beim eingestellten pH-Wert entweder als Anion, Kation oder Zwitterion vor. Beim
Einschalten des Gleichstroms wandern die Aminosäuren entsprechend ihrer Ladung nun zum
Plus-Pol, Minus Pol oder bleiben auf der Startlinie stehen. Wanderungsrichtung und -
geschwindigkeit der Aminosäuren hängen von ihrer Ladung, ihrer Größe und der Stärke der
angelegten Spannung ab.4
2.2.1.) Isoelektrischer Punkt (IEP) = Der pH-Wert an dem eine Aminosäure fast
ausschließlich in zwitterionischer Form vorliegt.3
H
H
{
O
H2N
C
+
H3N
C
OH
R
O
C
C
O
}
R
Glutaminsäure ist eine saure Aminosäure. Das heißt sie weist eine zweite Säuregruppe, eine
COOH-Gruppe, im Rest auf. Diese zweite Säuregruppe gibt, genau wie die NH3+-Gruppe des
Zwitterions, bei der Zugabe von Base ein Proton ab. Was dazu führt das Glutaminsäure im
Basischen als doppeltes Anion vorliegt. Dadurch ist der IEP von Glutaminsäure ziemlich
niedrig, er beträgt etwa 3,22.16
2.2.2.) Wanderungsrichtung:4
Alanin:
Der IEP von Alanin entspricht dem eingestellten pH-Wert der Pufferlösung, damit liegt
Alanin als Zwitterion vor und bleibt auf der Startlinie stehen.
Glutaminsäure:
Glutaminsäure weist zwei Carboxylgruppen auf. Der IEP von Glutaminsäure liegt unterhalb
des eingestellten pH-Wertes der Pufferlösung. Es wird Base zum Zwitterion hinzugegeben.
Dadurch gibt Glutaminsäure zwei Protonen ab, und liegt als doppeltes Anion vor. Die
Glutaminsäure wandert zum Plus-Pol.
1. Schritt:
O
O
HO
C
C
CH2
O
+
OH
O
CH2
O
H2O
H2C H C
C
O
H2C H C
O
C
+
NH3
+
NH3
Wasser
Anion
Base
Zwitterion
2.Schritt:
O
O
C
O
C
CH2
O
H2C H C
C
O
+
OH
O
CH2
H2C H C
C
O
+
H2O
NH2
+
NH3
Anion
O
Base
doppeltes Anion
Wasser
Lysin:
Lysin weist zwei Aminogruppen auf. Der IEP von Lysin liegt somit über dem eingestellten
pH-Wert der Pufferlösung. Das Zwitterion wird angesäuert und nimmt zwei Protonen auf.
Lysin liegt als doppeltes Kation vor, und wandert zum Minus-Pol.
1. Schritt:
+
H2N
H3N
CH2
H2C
CH2
O
+
+
H3O
CH2
H2C
H2C H C
C
O
CH2
+
O
H2O
H2C H C
C
O
+
+
NH3
NH3
Zwitterion
Kation
Säure
Wasser
2. Schritt:
+
+
H3N
H3N
CH2
H2C
CH2
O
+
+
H3O
CH2
H2C
CH2
+
H2O
H2C H C
C
OH
H2C H C
C
O
+
+
NH3
NH3
Kation
O
Säure
doppeltes Kation
Wasser
2.3.) Am Anfang ist die wässrige Lösung von Phenylalanin trüb. Grund ist, dass Phenylalanin
an ihrem IEP als Zwitterion vorliegt. Die Zwitterionen ballen sich zusammen, und fallen als
Niederschlag aus.
Probe 1: Die Lösung klart auf. Durch die Zugabe von Säure nimmt die COO--Gruppe des
Zwitterions ein Proton auf, und liegt somit als Kation vor. Die Kationen stoßen sich ab, sie
ballen sich nicht mehr zusammen, und sind gut in Wasser löslich.
Probe 2: Die Lösung klart auf. Durch die Zugabe von Base gibt die NH3+-Gruppe des
Zwitterions ein Proton ab, und liegt somit als Anion vor. Die Anionen stoßen sich ab, sie
ballen sich nicht mehr zusammen, und sind gut in Wasser löslich.
Probe 3: Die Lösung bleibt trüb. Phenylalanin liegt weiterhin als Zwitterion vor.3
98/IV/2
2.2.2.) Propansäure besteht aus zwei Teilen. Einem aliphatischem, unpolarem Rest und einer
polaren Carboxyl-Gruppe. Zwischen den Carboxyl-Gruppen der Propansäuremoleküle bilden
sich Wasserstoffbrückenbindungen aus. Diese Wasserstoffbrückenbindungen sind nicht sehr
stabil. Sie können mit relativ niedrigem Energieaufwand gelöst werden. Der Schmelzpunkt
von Propansäure ist sehr niedrig, sie ist bei Raumtemperatur flüssig.15
H H O
H C C C O H
H H
Wasserstoffbrückenbindung
O H H
H O C C C H
H H
Glycin hat eine zwitterionische Struktur. Zwischen den COO--Gruppen und den NH3+Gruppen der Zwitterionen bilden sich ionale Bindungen aus. Dadurch entsteht eine
Kristallgitterstruktur. Dieses Kristallgitter ist sehr stabil, und kann nur unter hohem
Energieaufwand gelöst werden. Der Schmelzpunkt von Glycin ist relativ hoch, die
Aminosäure ist bei Raumtemperatur ein Feststoff.
H
+
N
R1
R2
C
O
ionale Bindung
O
R2
+
N
H
C
R1
2.3.1.)
OH
+
H3N
H
C
H
O
CH2
C
C
H
N
H
O
H
N
C
C
H
C
O
CH3
O
Peptidbindung
2.3.2.) Die längeren C-N-Bindungen entsprechen einer Länge von 0,147 nm. Die kürzeren CN-Bindungen entsprechen einer Länge von 0,132 nm.
H
C
0,147 nm
C
N
C
O
0,132 nm
Die Bindungswinkel betragen 120°. Die Reste sind in Transform angeordnet. Diese
Konfiguration wird bevorzugt, da die Reste einen großen Raumbedarf haben, und so
möglichst weit voneinander entfernt sind. Die Peptidbindung ist planar und starr, die Bindung
ist also um die eigene Achse nicht frei drehbar. Grund hierfür ist, dass das Kohlenstoffatom,
das Stickstoffatom und das Sauerstoffatom der Peptidbindung sp2-hybridisiert sind. Die
Valenzstriche in der Skizze kennzeichnen die Sigmabindungen zwischen den Atomen. Die
freien py-Orbitale überlappen sich zu einer delokalisierten ð-Elektronenwolke. Die
Delokalisation der Elektronen führt zur Ausbildung von mesomeren Grenzformeln.
H
H
N
R1
C
O
+
N
R1
R2
C
R2
}
N
R1
O
Ketoform
R2
C
O
H
Enolform
Die Bindungsverhältnisse in der Peptidbindung sind somit nicht klar definiert. Als Folge
weist die C-O-Doppelbindung (Länge: 123 pm) der Peptidbindung einen
Einfachbindungscharakter auf, das bedeutet sie ist länger als eine normale Doppelbindung
(Länge: 122 pm). Die C-N-Einfachbindung (Länge: 132 pm) der Peptidbindung hingegen
weist einen Doppelbindungscharakter auf, sie ist kürzer als eine normale Einfachbindung
(Länge: 147 pm). Die Bindung des Stickstoffs mit dem C-Atom das nicht an der
Peptidbindung beteiligt ist entspricht in ihrer Länge einer normalen Einfachbindung (Länge:
147pm). Da dieses C-Atom nicht von der delokalisierten Elektronenwolke betroffen ist,
werden seine Bindungsverhältnisse durch die Ausbildung von Mesoformen nicht beeinflusst.1