Proteinbiosynthese2
Werbung

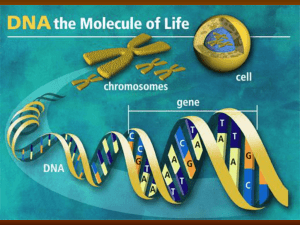
Proteinbiosynthese Transkription Funktion: Herstellung Genkopien, Umschreibung einer DNA Sequenz in RNA Sequenz „genetische Information wird beweglich) Der erste Schritt ist die Herstellung einer Gen-Kopie in Form von mRNA. Die Transkription ist eine „Überschreibung“ einer DNA-Sequenz in eine RNA-Sequenz. Dieser Vorgang wird durch RNA-Polymerase katalysiert. Das Enzym wird an den Promotor(spezifische DNA-Sequenz) gebunden. Hinter der Promotor Sequenz startet die Transkription. Dazu wird die DNA-Doppelhelix durch Entwindung geöffnet. Die RNA-Nucleotide lagern sich an den codogenen Strang an. Die RNA-Polymerase wandert weiter, die frisch synthetisierte RNA löst sich von der DNA-Matrize und der abgelesene DNA-Bereich windet sich in die Doppelhelix Form zurück. Wie die DNA-Polymerasen kann auch die RNA-Polymerase nur in 5` 3` verknüpfen. Sie benötigt keinen Primer und erkennt Start und Stoppsignale auf dem codogenen Strang. Bei der Transkription werden nur kleine DNA-Abschnitte abgelesen, es arbeiten mehrere DNAPolymerasen gleichzeitig. Die entstandene mRNA ist in Prokaryoten nur wenige Minuten haltbar, dann wird sie von zelleigenen Enzymen abgebaut. Stichpunkte - Das Enzym RNA-Polymerase wird an den Promotor(spezifische DNA-Sequenz) in der DNA gebunden - Hinter der Promotor Sequenz startet die Transkription - Die DNA-Doppelhelix wird durch Entwinden geöffnet - Die RNA-Nucleotide lagern sich an den codogenen(der genetische Information-tragende DNA-Strang, dessen Basensequenz bei der Transkription von RNA abgelesen wird) an. - Es wird von 3` 5` abgelesen. (Codogener Strang) - Die RNa-Polymerase wandert weiter, die frisch synthetisierte RNA (mRNA) löst sich von der DNA-Matritze und der abgelesene DNA-Bereich windet sich in die Doppelhelix Form zurück - Die DNA-Polymerase kann nur von 5` 3` verknüpfen. - Die Transkription endet, sobald die RNA-Polymerase auf eine bestimmte Basenabfolge am DNA-Matrizenstrang (Stoppsequenz) stößt. - Das Enzym löst sich vom codogenen Strang und setzt die m-RNA frei. - Bei eukaryothen kann die mRNA den Kern verlassen und von der DNA zu den Ribosomen, den Organellen der Proteinbiosynthese, transportiert werden. Unterschiede RNA-DNA - Die RNA besteht aus nur einem Einzelstrang - Sie ist viel kürzer, da sie nur einen kleinen Ausschnitt der genomischen Information repräsentiert - Anstelle der Desoxyribose enthält sie den Zucker Ribose - Anstelle der Base Thymin findet man die ebenfalls mit Adenin paarende Base Uracil) Translation (Es wird die Basensequenz der m-RNA in die Aminosäuresequenz des Proteins übersetzt) Die Translation findet an den Ribosomen statt, diese bestehen aus zwei Untereinheiten, die erst beim Start der Proteinbiosynthese zu einem funktionsfähigen ganzen Ribosom zusammentreten. Sie ist der zweite Schritt auf dem Wege der Genexpression. Die Proteinsynthese benötigt: 1. Alle Aminosäuren, die Bausteine der Polypeptidketten(Proteinen) sind 2. Ribosomen 3. Enzyme 4. mRNA als Träger der Information über die Aminosäureabfolge 5. die Transfer-TNA (tRNA) Die tRNA besitzt eine kleeblattartige Struktur, was an den drei Schleifen liegt. Die tRNA besitzt zwei Bindungsstellen eine Anlagerungsstelle für das Ribosom und eine Aminosäure Bindungstelle (3`). - Das so genannte Anticodon bindet nach der Basenpaarungsregel komplementär an bestimmte m-RNA-Tripletts - Die Aminosäurebindungsstelle, an der eine bestimmte Aminosäure angelagert wird, und zwar diejenige, die nach den Regeln des genetischen Codes von dem zugehörigen m-RNA-Triplett verschlüsselt wird Die ribosomalen Untereinheiten treten am Startcodon einer m-RNA zusammen. An das Startcodon (AUG) lagert sich nach dem Prinzip der komplementären Basenpaarung die Methionin-t-RNA an. Das t-RNA-Molekühl bindet zudem an die P-Stelle des Ribosoms. An der A-Stelle lagert sich eine weitere beladene t-RNA an, deren Anticodon zum nächsten Codon der m-RNA passt. Sind Codon und Anticodon basenkomplementär, wird die entsprechende t-RNA noch fester gebunden. Wenn sie nicht komplementär sind fallen die Moleküle wieder ab. Bei Basenkomplementarität werden die Aminosäuren der beiden t-RNA-Moleküle unter Energieaufwand chemisch miteinander verknüpft. (Peptidbindung => Wasser wird abgespalten) Damit die komplette Information der mRNA abgelesen, und in die Aminosäurekette eingebunden werden kann, rückt das Ribosom um ein Basentriplett auf der m-RNA in Translationsrichtung nach vorn. Die t-RNA an der P-Stelle rückt aus dem Ribosom heraus und löst sich ab Die verbliebene t-RNA rückt dabei von der A-Stelle zur P-Stelle vor, die A-Stelle ist wieder frei, es kann eine t-RNA mit passendem Anticodon komplementär binden. Dieser Vorgang wird solange durchgeführt, bis das Ribosom auf ein Stoppcodon der m-RNA trifft. Der Abbruch der Translation wird eingeleitet, da es keine passende t-RNA gibt. Der Komplex aus Ribosom und m-RNA zerfällt und die synthetisierte Aminosäurekette wird freigesetzt. T-RNA Hochspezifische Enzyme, die sog. Amino-acyl-t-RNA-Synthetasen, erkennen das Anticodon einer tRNA und beladen diese mit der dazugehörigen Aminosäure Es sind rund 50 verschiedene t-RNA Arten bekannt, also mehr als genug, um jeweils genau eine der 20 Aminosäuren spezifisch zu binden. Die meisten Aminosäuren werden durch mehrere Tripletts codiert, der genetische Code ist degeneriert. Es gibt für eine Aminosäure mehrere Typen von t-RNA-Molekülen.