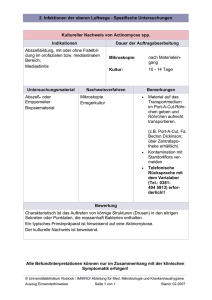
Instrumentelle Bioanalytik
Werbung

Instrumentelle Bioanalytik 7. Mikroskopie 7.1 7.2 7.3 7.4 7.5 7.6 7.7 7-1 Einführung Lichtmikroskopie Fluoreszenzmikroskopie Konfokal-Mikroskopie (CLSM) Elektronenmikroskopie Rasterkraftmikroskopie Optisches Rasternahfeldmikroskop 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.1 Einführung Historie 1600 Drebbel 1. Mikroskop Kepler (erstes M. mit 2 Sammellinsen), Hooke, Leeuwenhoeck, Malpighi 1890 Abbe Theorie 1913 Lehmann, Prowazek Fluoreszenz 1941 Zernike Phasenkontrast Nobelpreis 1955 Nomarski DIC 1957 Minsky Konfokal 1982 Binning, Rohrer STM Nobelpreis 1986 Binning, Gate, Gerber AFM 1990 Hell 4Pi historische Lichtmikroskope Antonie van Leeuwenhoeck, 1660 275 fach, 1,3 mm erste Zeichnungen von Bakterien 7-2 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik verbessertes Design, John Yarwell, 1680 Lichmikroskop von Robert Hooke (mit 2 Linsen) (Zeichnung von Korkzellen) 7. Mikroskopie 7.1 Einführung Größe von biologischen Strukturen vs. Auflösung Der kleinste noch auflösbare Abstand zweier Punkte ist etwa halb so groß wie die Wellenlänge der verwendeten elektromagnetischen Strahlung. Bei einer Wellenlänge von λ = 400 - 700nm (sichtbarer Lichtbereich) entspricht dieses einem Abstand von ca. 200 – 350 nm bzw. ca. 0.2 - 0.35 μm. 7-3 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.2 Lichtmikroskopie Strahlengang (vereinfacht) Objekt zwischen f und 2f : Reelles Bild das auf dem Kopf steht 7-4 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik Objekt zwischen 0 und f : Virtuelles Bild seitenrichtig 7. Mikroskopie 7.2 Lichtmikroskopie Vergößerung Auflösung d: Abstand zwischen zwei Punkten A: numerische Apertur (NA) n: Brechzahl des Mediums zwischen Deckglas und Objektiv . Erhöhung der Auflösung durch Ölimmersion Immersionsöl a bezeichnet den halben Öffnungswinkel zwischen Präparat und Objektiv. Das Auflösungsvermögen des Lichtmikroskops ist im Wesentlichen abhängig von der Wellenlänge des verwendeten Lichtes und dem Winkel a. Eine Verringerung des Arbeitsabstandes vergrößert den Öffnungswinkel. Für eine optimale Auflösung sollte der Winkel a so groß wie möglich sein (im besten Fall ungefähr 70°). Immersionsöl erhöht den Brechungsindex n zwischen Objekt und Objektiv. 7-5 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik Steigerung des Öffnungswinkels ( a ) durch Verwendung von Immersionsöl. Gezeigt sind die Strahlengänge dreier eng benachbart liegender Punkte eines Objektes (1, 2 und 3), die bei Verwendung von Immersionsöl aufgelöst werden (rechts). Ohne Immersionsöl (links) werden die Strahlen 2 und 3 an der Grenzfläche zwischen Deckglas und Luft gebrochen Typische Objektive Billig: Apertur 0.1 Auflösung 2,75μm Hochwertig: Apertur 0,65 Auflösung 0,42μm Öl: Apertur 1,4 Auflösung 0,20μm 7. Mikroskopie 7.2 Lichtmikroskopie Kontraststeigerung (1): Hellfeld Dunkelfeld Hellfeld: Kreisförmige Blende Transmission Dunkelfeld: Ringförmige Blende Sichtbar sind Umrisse und Beugung Kutikulapräparation eines Embryos von Drosophila melanogaster, etwa 22h alt [http://de.wikipedia.org/] 7-6 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.2 Lichtmikroskopie Kontraststeigerung (2): Phasenkontrast-Mikroskop Prinzip 1) Abschattung durch Ringblende vor Eintritt in Objekt 2) Phasendrehung durch λ/2 Plättchen nur des Objektlichtes 3) Mischung Objektlicht und Direktlicht (Interferenz) 7-7 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik Phasenverschiebung beim Durchgang durch ein Objekt mit anderem Brechungsindex als das umgebende Medium 7. Mikroskopie 7.1 Lichtmikroskopie Phasenkontrast-Mikroskop (S) surround wave (D) diffracted wave (P) resulting particle wave Depending upon the configuration and properties of the phase ring positioned in the objective rear focal plane, specimens can be observed either in positive or negative phase contrast. http://www.microscopyu.com/tutorials/java/phasecontrast/ positivenegative/index.html 7-8 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.1 Lichtmikroskopie Kontraststeigerung (3): Differenzial-Interferenzkontrast / DIC-Mikroskop Differentieller Interferenzkontrast, DIC von engl. differential interference contrast Prinzip: Methode nutzt die Unterschiede in der optischen Weglänge im betrachteten Objekt und stellt diese als Helligkeitsunterschiede im Mikroskopiebild dar. Dadurch können transparente Objekte sichtbar gemacht werden. Das Bild entspricht der lokalen Änderung (Gradient) des Brechungsindex der Probe (daher die Bezeichnung "differentieller" Bildkontrast). Auflicht: Bildkontrast gibt die Änderungen der Oberflächenmorphologie wieder. Durchlicht: plastische Bilder des Objekts. Strahlengang der DIC-Mikroskopie [http://de.wikipedia.org/wiki/Differentialinterferenzkontrast] 7-9 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.2 Lichtmikroskopie Kontraststeigerung: Differenzial-Interferenzkontrast 7-10 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Beispiel: Triple-Fluorescence-labeled endothelial cells: Red: Actin-Filaments labeled with Phalloidin Green: Membranes (DiO-C6) Blue: Nuclei (DAPI-staining of DNA) 4′,6-Diamidin-2-phenylindol 7-11 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Epifluoreszenzmikroskopie (Auflichtfluoreszenzmikroskopie) fluoreszenzmikroskopisches Verfahren, bei dem die Fluoreszenz auf derselben Seite nachgewiesen wird, von der aus die Anregung erfolgt. Dieses Verfahren funktioniert analog zur Reflexionsmikroskopie, bei der ebenfalls Anregung und Nachweis in derselben Halbebene bezüglich des Präparates stattfinden. Im Gegensatz dazu finden in der Transmissionsfluoreszenzmikroskopie Anregung und Nachweis in verschiedenen Halbebenen statt. 7-12 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Anregungslichtquellen [http://zeiss-campus.magnet.fsu.edu/articles/basics/fluorescence.html] 7-13 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Farbstoffe (Fluoreszenzsonden) (1) Anregung Emission http://www.invitrogen.com/site/us/en/home/support/Tutorials.html 7-14 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Farbstoffe (Fluoreszenzsonden) (2) interaktiv [http://www.invitrogen.com/site/us/en/home/support/Research-Tools/Fluorescence-SpectraViewer.html] 7-15 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie Epifluoreszenzmikroskopie Filterselektion – Kombination von Filtersets zwecks Optimierung 7-16 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.3 Fluoreszenz-Mikroskopie TIRF Microscopy (TIRFM) Principles of TIRF Chemically Modified TIRF slides and TIRF Microscopy lightguides 29.01.2012 6-17 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.4 Konfokal-Mikroskopie Problem der konventioneller Mikroskopie: Licht, welches von außerhalb des Fokuspunktes kommt (unterhalb bzw. darüber) gelangt ebenfalls zum Detektor verschwommener Fleck, der die Qualität des Bildes herabsetzt. Lösung: Einsatz einer Lochblende: Hintergrundsignal ebenfalls fluoreszierender Bestandteile aus höher oder tiefer liegenden Schichten des Präparats wird ausgeblendet. Durch Scannen übereinander liegender Schichten und anschließende Rekonstruktion der so gewonnenen Einzelbilder können dreidimensionale Bilder erstellt werden. Tiefenauflösung 0,8 mm, abhängig von Lochblendendurchmesser Hohe Anforderungen Laser mit intensiver, kohärenter und paralleler Strahlung Confocal Laser Scanning Microscopy (CLSM) Ergebnis: sehr schafes Bild ohne Unschärfe (Auswaschungen) etwas höhere Auflösung als konventionelle Epifluoreszenz-Mikroskopie. 7-18 20.12.2011 Michael Wörner - Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.4 Konfokal-Mikroskopie Confocal Laser Scanning Microscopy (CLSM) Epifluoreszenz vs Konfokal Zelle mit Teilungsapparat Spectral Imaging Confocal Microscopy (with Emission Curve Analysis) Leica Confocal Microscope TCS SP2: Monochromator in front of the detector AOBS: Acousto-Optical Beam Splitter (instead of dicroic mirror) META System of Zeiss: 32 PMT-detectors every 10.7 nm (400 – 720 nm): simultaneous wavelength analysis. lambda-stack 7-19 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie Prinzip Abbildung eines Objektes mit Hilfe von Elektronen(strahlen). Vorteil: schnelle Elektronen (als Materiewelle) besitzen eine sehr viel kleinere Wellenlänge als sichtbares Licht höheres Auflösungsvermögen derzeit realisiert: etwa 0,1 nm Nachteile: elektronenoptische Bauteile besitzen hohe Aberrationen (Abbildungsfehler) nutzbare Auflösung fast um 2 Größenordnungen kleiner als Elektronenwellenlänge (100 keV 0,0037 nm) Elektronenmikroskopischen Daten (Bilder) werden oftmals falsch „interpretiert“ (digitale Bildverarbeitung) (Aufwändige) Probenpräparation kann zu Artefakten führen. 7-20 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie Instrumentation Elektronenquelle: Erzeugung von freien Elektronen (Glühkathode) und Beschleunigung dier Elektronen durch eine um die Strahlachse liegenden Anode. Die Spannung zwischen Kathode und Anode bestimmt die Energie der Elektronen und liegt je nach Mikroskop zwischen wenigen Kilovolt bis zu 3 Megavolt. Elektronenlinsen, die die Flugbahnen der Elektronen ablenken können. Meist werden magnetische Linsen verwendet, zum Teil auch elektrostatische. Elektronenlinsen haben die gleiche Funktion wie Glaslinsen im Lichtmikroskop. Während die Brennweite der Glaslinsen fest liegt, ist sie bei Elektronenlinsen regelbar. Neben Linsen kommen wie beim Lichtmikroskop auch Blenden zum Einsatz. Vakuumsystem: notwendig für effiziente Erzeugung eines Elektronenstrahls; verhindert die Kollision der Elektronen mit Gasmolekülen. Objekthalterung: garantiert stabile Lage des Objekts. Darüber hinaus sind oft Manipulationsmöglichkeiten erwünscht, von denen je nach Art des Objekthalters unterschiedliche Kombinationen realisiert werden: Verschiebung, Drehung, Kippung, Heizung, Kühlung, Dehnung. Detektoren, die die Elektronen selbst oder sekundäre Signale registrieren. Mikroskopsäule (engl. column) bildet den Rahmen für alle elektronenoptischen Bauteile, schirmt in der Regel magnetisch ab, um die Einflüsse äußerer Magnetfelder auf die Messungen abzuschwächen, und dichtet das im Inneren aufrecht zu erhaltende Vakuum ab. 7-21 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik Strahlengang und Anordnung der Linsen im Transmissionselektronenmikroskop (TEM) im Vergleich zum Lichtmikroskop (LM) 7. Mikroskopie 7.5 Elektronenmikroskopie Aufnahmetechniken (Bilderzeugung) Ruhebildmikroskopie: Objektbereich wird mit einem feststehenden, breiten Elektronenstrahl bestrahlt. Das Bild wird hier erzeugt, indem ein Teil der vom Objekt ausgehenden Elektronen zur Bilderzeugung mittels eines elektronenoptischen Systems verwendet wird. Elastische Streuung eines Elektrons innerhalb eines Atoms Wichtig ist, dass die Elektronen im Objekt - anders als Licht – auch stark inelastisch gestreut werden und Energie verlieren. Generell kann der Ruhebildmodus nur genutzt werden, wenn nach der Wechselwirkung des Elektronenstrahls mit dem Objekt genügend Elektronen mit einer hinreichend schmalen Energieverteilung zur Verfügung stehen und gleichzeitig nicht zu viele Elektronen mit abweichenden Energien auftreten. Dies ist nur für hinreichend dünne Objekte gegeben. Wechselwirkungsvolumen in kompakten Proben in Abhängigkeit von der Beschleunigungsspannung und der Ordnungszahl (Z) Rasterelektronenmikroskopie REM bzw. SEM (scanning electron microscop) Mit Hilfe eines elektronenoptischen Systems elektromagnetischer und elektrostatischer Linsen wird ein feiner Elektronenstrahl erzeugt, der zeilenweise über den zu untersuchenden rechteckigen Objektbereich geführt wird („gerastert“). Das Bild kommt dabei durch die synchrone Registrierung eines vom Elektronenstrahl ausgelösten oder beeinflussten Signals zustande. 7-22 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie Aufnahmetechniken (Geometrie) Transmission: Die schnellen Strahlelektronen werden nach Durchgang durch das Objekt zur Bilderzeugung verwendet werden, wobei in der Regel nur sehr kleine Streuwinkel erfasst werden. Transmissionselektronenmikroskope (TEM) arbeiten meistens nach der Ruhebildmethode. Gelegentlich wird auch die Rastermethode angewendet: RasterTransmissionselektronenmikroskop (STEM, „scanning transmission electron microscopy/microscope“). Die untersuchten Objektbereiche müssen sehr dünn sein (man spricht von Elektronentransparenz, für heute übliche Beschleunigungsspannungen bzw. Elektronenenergien: maximal einige 100 nm Dicke für sehr grobe Auflösung, typisch unter 100 nm Dicke; für Hochauflösung maximal einige 10 nm). „Reflexion“ Werden zur Bilderzeugung hauptsächlich andere Signale als die transmittierten Elektronen eingesetzt, so gibt es dafür keine feststehende Bezeichnung. Die benutzten Signale sind meist Sekundärelektronen, seltener Rückstreuelektronen. Will man kompakte Objekte untersuchen, so ist dies die einzige Möglichkeit. In der Regel kann hierfür nur mit Rasterverfahren (s. o.) gearbeitet werden. 7-23 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie TEM (Transmissionselektronenmikroskopie) Die Elektronen durchstrahlen das Objekt, das zu diesem Zweck entsprechend dünn sein muss ( Probenpräparation). Je nach Ordnungszahl der Atome, aus denen das Objekt besteht, der Höhe der Beschleunigungsspannung und der gewünschten Auflösung kann die sinnvolle Objektdicke von wenigen Nanometern bis zu einigen Mikrometern reichen. (biologische Proben: meist 80 – 120 keV, ≤ 100 nm) In der zu untersuchenden Probe werden die Elektronen gestreut, das heißt ihre Bewegungsrichtung ändert sich. Teilweise verlieren sie dabei auch Bewegungsenergie (inelastische Streuung). Elastisch gestreute Elektronen, die das Objekt unter demselben Winkel verlassen, werden in der hinteren Brennebene der Objektivlinse in einem Punkt fokussiert. Man kann nun in dieser Ebene mit einer Blende (Objektivblende beziehungsweise Kontrastblende) nur die Elektronen passieren lassen, die nicht gestreut wurden. Da Atome mit höherer Ordnungszahl sowie dickere Objektbereiche stärker streuen, wird der entstehende Kontrast Massendickenkontrast genannt. (Hinweis: sehr stark vereinfachte Betrachtungsweise der Bildgebung/entstehung). Dieser Kontrast ermöglicht bei amorphen Festkörpern eine recht einfache Interpretation der erhaltenen Abbildungen. Der Kontrast kristalliner Materialien folgt komplizierteren Gesetzmäßigkeiten und wird als Beugungskontrast bezeichnet. Durch eine Änderung des Projektivlinsensystems kann anstatt des Zwischenbildes auch die Fokusebene der Objektivlinse vergrößert abgebildet werden. Man erhält so ein Elektronenbeugungsbild, mit dessen Hilfe sich die Kristallstruktur der Probe bestimmen lässt. Die Mikroskopie im Vakuum erfordert präparative Maßnahmen für das (biologische) Objekt (Fixieren, Einbetten, Einfrieren), damit es nicht durch Trocknungsartefakte zerstört wird 7-24 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie TEM – Biologische Präparate Präparationsverfahren (1) - Übersicht Ganze Zellen oder Gewebeteile werden in spezielle Harze eingebettet, um sie mit dem Ultramikrotom zu Ultradünnschnitten (≤ 100 nm) zu verarbeiten. Zellwandfragmente, Membranen, Hüllen von Organellen, Proteine und andere makromolekulare Strukturen sind dünn genug. Ein kleiner Tropfen der Probe (2 – 5 ul) wird auf den Objektträger (Trägernetzchen, Grid) pipettiert und nach kurzer Zeit mit Filterpapier abgesaugt. Das noch feuchte Präparat wird durch Eintauchen in ein Kryogen eingefroren oder kontrastiert. Fixierung – um die Probe realistischer darstellen zu können. Verwendet werden Glutardialdehyd zur Vernetzung und damit Verhärtung der Proteine und Osmiumtetroxid, welches Lipide schwarz färbt und gleichzeitig fixiert. Cryo-Fixierung – die Probe wird in flüssigem Ethan bei weniger als −135 °C schockgefroren. Dabei kristallisiert das Wasser nicht, sondern bildet vitrifiziertes (glasartiges) Eis. Bei dieser Methode wird die biologische Probe mit der geringsten Artefaktbildung fixiert. Allerdings ist der Kontrast sehr gering. Dehydratisierung – Wasser wird entfernt und schrittweise durch Ethanol oder Aceton ersetzt. Einbettung – um Gewebe sektionieren zu können. Hierzu werden meist Acrylharze genutzt. Sektionierung – Aufteilen der Probe in dünne Scheiben (Ultradünnschnitte). Diese können auf einem Ultra-Mikrotom mit einer Diamant- oder Glasklinge geschnitten werden. Färbung (Negative Stain) – schwere Atome wie Blei- oder Uran-Atome streuen Elektronen stärker als leichte Atome und erhöhen so den Kontrast. Hier werden Reagenzien wie Uranylacetat oder Bleicitrat verwendet. 7-25 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik A: Probenhalter für Kryoelektronenmikroskopie B: Trägernetzchen (3 mm ) C: Kohlefilm mit Löchern 7. Mikroskopie 7.5 Elektronenmikroskopie TEM – Biologische Präparate Präparationsverfahren (2) - Negativkontrastierung Negativkontrastierung mit Schwermetallsalzen: - einfache und schnelle Methode zur Darstellung von Proteinen, Proteinaggregaten und Membranen. - maximal nutzbare Auflösung von 1,0 bis 1,5 nm (entspricht Proteindomänen mit etwa 5 bis 15 AS) Schematische Darstellung der verschiedenen Kontrastierungsarten für die Abbildung von Makromolekülen im TEM. Die Kontrastierungen mit Schwermetall führen zu sehr unterschiedlichen Dichteverteilungen im EM-Bild und liefern jeweils andere Strukturinformationen. Nur bei der Einbettung der Proteine in (amorphes) Eis stammt der Kontrast vom Objekt selbst, sonst wird das die Moleküle umgebende oder eingelagerte Metall abgebildet. 7-26 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.5 Elektronenmikroskopie TEM – Biologische Präparate Präparationsverfahren (3) – Bedampfung mit Schwermetallen Gefrierbruch/Gefrierätzung (freeze etching) Intakte Gewebe oder Zellen werden eingefroren und im Vakuum geschnitten oder gebrochen; die Oberfläche wird durch Sublimation des Eises freigelegt und mit einer dünnen Metallschicht (meist 1 - 2 nm Pt/C) unidirektionell oder einem Winkel von 20 – 60° bedampft und anschließend mit senkrechter Bedampfung mit 10 – 20 nm Kohle stabilisiert. Die anhaftenden biologischen Teile werden mit oxidierenden/ätzenden Flüssigkeit entfernt und der verbleibende Abdruck im Mikroskop betrachtet (Replikattechnik) ( 3D-Information von Oberflächen und aufgebrochenen Zellen) Direkte Bedampfung Variante, zur Mikroskopie von isolierten Proteinen oder Membranen. Wie bei der Negativkontrasttechnik werden die Objekte auf den Objektträgernetzchen adsorbiert und in der Kälte bedampft. Als kontrastierende Metalle werden entweder Pt/C (1 – 1,5 nm) oder auch Ta/W (etwa 0,5 nm) verwendet EM-Aufnahmen eines zweidimensionalen Proteinkristalls (Oberflächenprotein des Bakteriums Deinnococcus radiodurans) mit verschiedenen Kontrastierung . Die Vergrößerungen stellen Mittelungen über viele Einheitszellen dar. A: Bei Einbettung in amorphes Material (Eis) liefert das Protein selbst den Kontrast und erscheint dunkel. B: Bei Negativkontrastierung erscheint das Protein hell, das Schwermetallsalz (Phosphorwolframat) hingegen dunkel. C: Unidirektionelle direkte Bedampfung mit Metall (Ta/W) liefert zwei Ansichten, von der physiologischen Innen- bzw. Außenseite des Kristalls, die damit leicht unterschieden werden können. 7-27 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Das Rasterkraftmikroskop (AFM atomic force microscope) wurde1986 entwickelt/vorgestellt (durch Gerd Binnig, Calvin Quate und Christoph Gerber). Wichtiges Werkzeug in der Oberflächenchemie, dient zur mechanischen Abtastung von Oberflächen und der Messung atomarer Kräfte auf der Nanometerskala. Gehört zur Familie der Rastersondenmikroskopie (SPM, scanning probe microscopes), welche Oberflächen mit spitzen Sonden abtasten: Art der spezifischen Wechselwirkung der Sonde mit dem Objekt bestimmt die Zugehörigkeit, z.B. Optische Signale Rasternahfeldmikroskop (scanning near-field microscope, SNOM) Tunnelströme Rastertunnelmikroskop (scanning tunneling microscope, STM) Mittlerweise sind über zwanzig verschiedene Messanwendungen für die Rastersondenmikroskopie anorganischer und organischer Proben entwickelt worden. Oberfläche von Natriumchlorid (Kochsalz) mit dem Rasterkraftmikroskop im Nicht-Kontakt-Modus im Ultrahochvakuum aufgenommen. Man sieht die einzelnen Atome als Erhebungen bzw. Vertiefungen in dieser 3-dimensionalen Darstellung. (Messung: Center for Nanotechnology (CeNTech), Universität Münster, Deutschland) 7-28 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Wechselwirkungskräfte Tab. Kräfte zwischen AFM-Sonde und Objekt Kraft Kraftrichtung Reichweite Kraft bei Kontakt der Elektronenhüllen abstoßend extrem kurz (≤ 0,1 nm) Van-der-WaalsWechselwirkungen anziehend sehr kurz (wenige Nanometer) elektrostatische WW anziehend/abstoßend kurz (nm bis um) Kapillarkräfte (Sonde im Wasser) anziehend weit (um bis mm) Vorteile der AFM (bei biologischen Proben): - Oberflächen können in Flüssigkeiten (unter physiologischen Bedingungen) abgebildet werden Signal-zu-Rausch-Verhältnis besser als bei Licht- und Elektronenmikroskopie Auflösung im Subnanometerbereich Probe wird bei Untersuchung nicht beschädigt bzw. Funktionsfähigkeit bleibt erhalten 7-29 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Funktionsprinzip Eine scharfe Messspitze (tip), die sich auf einem elastisch biegsamen Hebelarm (cantilever) befindet, wird als Messsonde (probe) in geringem Abstand in einer Rasterbewegung über die Probenoberfläche geführt. Ein piezoelektrischer Scanner bewegt hierfür entweder die Spitze über die Probe oder die Probe unter der feststehenden Spitze. Die Verbiegungen des Hebelarms, hervorgerufen durch Kräfte zwischen Probe (sample) und Spitze, werden hochaufgelöst gemessen, meist indem ein Laserstrahl auf die Spitze gerichtet und der reflektierte Strahl mit einem Photodetektor aufgefangen wird (Lichtzeigerprinzip). Die WW-Kräfte liegen typischerweise in der Größenordnung von 0,01 bis 100 nN und setzen sich aus repulsiven und attraktiven Beiträgen unterschiedlicher Reichweite zusammen (siehe Tabelle). Ein wichtiges Element eines Rasterkraftmikroskops ist der Controller, der die Bewegung des Scanners und der Probe bzw. Spitze steuert sowie die Signale auswertet. Die Bedienung des Geräts wird erleichtert, wenn die Positionierung des Lasers und der Spitze durch ein lichtoptisches Mikroskop unterstützt werden. Abb. Schematische Darstellung des Rasterkraftmikroskops. Die auf einer Unterlage immobilisierte Probe wird mit einem dreiachsigen piezoelektrischen Element unter der Messspitze bewegt. Während dieser Rasterbewegung wird die Verbiegung der Blattfeder mithilfe eines Laserstrahles und eines Photodetektors gemessen. Die Spannungsdifferenz zwischen den oberen und unteren Segmenten des Photodetektors (V = (A + B) - (C + D)) ist dabei ein direktes Maß für die Verbiegung der Feder. 7-30 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Funktionsprinzip - Betriebsmodi Das Rasterkraftmikroskop kann in verschiedenen Betriebsmodi betrieben werden. Die Betriebsmodi können nach drei Systematiken geordnet werden, je nachdem - ob eine Bildgebung erfolgt: bildgebend spektroskopisch - welche Wechselwirkungen für die Messungen genutzt werden: Kontakt-Modus Nicht-Kontakt-Modus Intermittierender Modus - wie die Bewegung der Nadel geregelt wird: Constant-height-Modus Constant-force/amplitude-Modus Abb. Die drei Modi der AFM: (a) Kontakt-, (b) Nicht-Kontakt- und (c) Intermittent-Modus Kontakt-Modus In allen Kontakt-Messmethoden steht die Messspitze in direktem mechanischem Kontakt mit der zu vermessenden Oberfläche. Zwischen den Elektronenhüllen der Atome an der Oberfläche und der sie berührenden Messspitze entsteht dabei eine starke elektrostatische Abstoßung. - ungeregelt: constant height mode (älteste Messmethode: Topographie aus Auslenksignal der Blattfeder) - geregelt: constant force mode (in z-Richtung wird so nachgeregelt, dass die Kraft zwischen Probe und Spitze konstant bleibt (langsamer, dafür schonender für die Probe) 7-31 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Funktionsprinzip – Betriebsmodi (2) Nicht-Kontakt-Modus (NC-AFM) (non-contact, dynamic mode) Dynamischer Anregungsmodus. Federbalken wird durch ein zusätzliches Piezoelement resonant zur Schwingung angeregt (Schwingkreis). Wenn Kräfte zwischen Spitze und Probe auftreten, ändert sich die Frequenz des Schwingkreises Frequenzverschiebung ist Maß für Kraftwechselwirkung Im (Ultra)Hochvakuum höchste Auflösung im Vergleich zu anderen Betriebsarten Intermittierender Modus (intermittent mode, tapping mode) Ebenfalls dynamischer Anregungsmodus. Externe Anregung des Federbalken bei einer festen Frequenz nahe der Resonanzfrequenz. WW-Kräfte zwischen Spitze und Probe verändern die Resonanzfrequenz des Systems, wodurch sich die Schwingungsamplitude und die Phase (zwischen Schwingung und Anregung) ändern. Als Regelsignal wird meistens die Amplitude genutzt, die über einen Regelkreis konstant gehalten wird, indem der Abstand angepasst wird. Bester Modus unter Umgebungsbedingungen und in Flüssigkeiten. 7-32 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Funktionsprinzip – Betriebsmodi (3) Abb. Abbildungsverfahren der Rasterkraftmikroskopie. A Im Kontaktmodus ist die Spitze in ständigem Kontakt mit der biologischen Probe und folgt daher bei Hindernissen der Oberflächenstruktur der Probe, was zu Verbiegungen der Blattfeder führt. Deshalb wird mithilfe einer Regelschlaufe die Kontaktkraft (Verbiegung der Feder) durch Verändern der Z-Position der Probe konstant gehalten. Aus dem Korrektursignal kann dann auf die Topographie der Probenoberfläche geschlossen werden. B In dynamischen Abbildungsverfahren oszilliert die Blattfeder nahe ihrer natürlichen Resonanzfrequenz mit einer Amplitude von wenigen Nanometern. Somit wird das Objekt nur kurzzeitig berührt, wodurch seitliche Wechselwirkungen reduziert werden. Die Regelschlaufe wird in dynamischen Abbildungsverfahren dazu verwendet, die Amplitude der Oszillation konstant zu halten. C Die seitliche Abtastrichtung der AFM-Spitze kann das biologische Objekt verformen. Dies kann durch die präzise Einstellung der Kontaktkraft, der Rastergeschwindigkeit und der Rückkopplungsparameter verhindert werden. D Zur Elastizitätsbestimmung wird die Spitze gegen ein weiches Objekt (z. B. Zellen) bewegt. Durch das Verhältnis von gefahrener Distanz und der tatsächlichen Federverbiegung lässt sich die Elastizität der des Objektes berechnen. 7-33 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Wechselwirkung zwischen Spitze und Probe Auflösungen bis in atomare Dimensionen erfordern besondere Maßnahmen. Wesentlicher Faktor ist die Dimension der Spitze; die erreichbare Auflösung hängt von der Schärfe der Spitze und den Oberflächeneigenschaften des Objektes ab (die gemessene Topograhpie ist eine nichtlineare Überlagerung des untersuchten Objektes mit der Spitze) Biologische Objekte sind relativ weich (Proteine können z.B. bei größeren Kontaktkräften denaturieren) Abbildungsverfahren, Blattfedern und Abbildungsparameter (Rastergeschwindigkeit, Rückkopplungsschleifen, Bildgröße) müssen optimiert werden. Ionenkonzentration des Puffers ist so zu wählen, dass repulsive (ca. 0,05 nN) und langreichweitige (mehrere nm) WW zustande kommen. Abb. Überlagerungseffekte zwischen Spitze und Probe können die AFM-Topographie verfälschen. A Die Spitze kann aufgrund ihres endlichen Durchmessers sehr scharfen Kanten nicht folgen. Daher stellt die erhaltene Topographie (rote Linie) eine Überlagerung zwischen Spitze und Probe dar. B In vielen Fällen kann die Periodizität von Strukturen unabhängig vom Spitzendurchmesser korrekt aufgelöst werden. 7-34 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Präparationsverfahren Abbildung von biologischen Makromolekülen und Zellen erfordert keine Beschichtung oder Markierung! Probenvorbereitung erfordert lediglich die Immobilisierung auf dem Probenträger. Kompromiss: gut verankertes Objekt (da nur in diesem Fall die genaue Position der Spitze über dem Objekt bekannt ist) bei gleichzeitiger Minmierung der WW zwischen biologischen Objekt und Objektträger (um Denaturierung zu verhindern). Probenträger: Glas, Glimmer, Graphit, metallisierte (z.B. Au-beschichtete) Oberflächen Für hohe Auflösung: atomar flache Oberflächen notwendig Proteine und Nucleinsäuren: Glimmer (Schichtsilikat) ist besonders geeignet (Kristallschichten können leicht gespalten werden, um chemisch relativ inerte und negativ geladene atomar glatte Oberflächen zu erhalten). Immobilisierungsstrategien: Physikalische Kopplung: meist durch Abschirmung abstoßender elektrostatischer WW ( Makromoleküle in geeigneter Pufferlösung hoher Ionenkonzentration aufnehmen und auf frisch gespaltene Glimmeroberfläche aufgeben) Kovalente Verknüpfung: chemische Kopplung der Makromoleküle auf funktionalisierten Substratoberflächen (modifiziert mit reaktiven chemischen Gruppen) 7-35 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie AFM biologischer Makromoleküle (1) Oberflächenstrukturen und auch dynamische Prozesse verschiedenster biologischer Proben unter nativen Bedingungen beobachtbar. Objekte: Proteine, DNA, RNA, supramolekulare Komplexe, Bakterien, Zellverbände, Gewebe höherer Organismen. Höchste Auflösung nur an isolierten Makromolekülen, die immobilisiert wurden; Zellen/Zellverbände maximal nur bis 50 nm. Abb. Cytoplasmatische Oberfläche der nativen Hochaufgelöste Topographien zeigen nicht nur Purpurmembran des Halobacteriums salinarum. einzelne Membranproteine in ihrer nativen Umgebung, A In der AFM Topographie ist ersichtlich, wie sich einzelne Bakteriorhodopsinmoleküle zu Trimeren sondern auch Untereinheiten und Substrukturen anordnen. Die Trimere wiederum ordnen sich zu einem hexagonalen Gitter an. Die Topographie der Membran wurde in physiologischer Pufferlösung bei Raumtemperatur aufgenommen. B Das Diffraktionsbild der gezeigten Topographie beugt bis zur elften Ordnung (eingezeichnete Kreise), was eine laterale Auflösung von 0,49 nm andeutet. C Die gemittelte Topographie (oben) und die dazugehörige Standardabweichung (unten) des Bakteriorhodopsintrimers ermöglicht die strukturelle Korrelation zu Daten, welche mittels Elektronenkristallographie erhalten wurden. Überlagert wurden der Umriss der Bakteriorhododopsinmoleküle sowie die Positionen der sieben transmembranen a-Helices (A–F). 7-36 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie AFM biologischer Makromoleküle (2) Struktur der FoF1 ATPase Untereinheiten a, b, 3 a, 3 b und d bilden den Stator Untereinheiten c, g, und e bilden den Rotator Abb. AFM zur Bestimmung der Proteinanordnung und -funktion. A Na+-getriebene Rotoren der F0F1-ATP-Synthase von Ilyobacter tartaricus. Die hohe Auflösung der Topographie ermöglicht es, die Stöchiometrie und Anordnung der Proteine eines funktionellen Rotors zu charakterisieren. B Extrazelluläre Oberfläche der Kommunikationskanäle (gap junctions) aus Epithelzellen der Rattenleber. Die hexameren Proteine zeigen einen offenen, zentralen Kanal. Das Mittel und die Standardabweichung (SA) lassen Einblicke in die Struktur und Flexibilität zu. Während das Profil des Mittels den Kanaleingang erkennen lässt, ordnet die SA-Karte dem zentralen Kanal eine erhöhte strukturelle Flexibilität zu. C In Anwesenheit von 0,5 mM Ca2+ (bei neutralem pH-Wert) schließen die Hexamere ihren zentralen Kanal. Dieser Vorgang wird im gemittelten Hexamer deutlich und die dazugehörige SA-Karte zeigt, dass der Kanaleingang im geschlossenen Zustand seine Flexibilität verloren hat. Alle Topographien wurden in physiologischer Pufferlösung bei Raumtemperatur aufgenommen. 7-37 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie AFM biologischer Makromoleküle (3) Dynamische Prozesse Entscheidender Faktor: Aufnahmezeit für eine einzelne Topographie: Konventionelles AFM: ein bis fünfzehn Minuten Forschungsprototypen (ultraschnelle AFMs): Aufnahmezeit im Sekundenbereich (und kleiner) Anwendungen für biologische Objekte/Fragestellungen - DNS-Transkription - Proteindiffusion - Konformationsänderungen von Proteinen Untersuchungen zur Elastizität Mehrere Verfahren, z.B. - Objekt wird gegen die AFM-Spitze gedrückt, wobei die Auslenkung der Blattfeder mit dem vom Piezoelement gefahrenen Weg in Beziehung gesetzt wird. - Dynamisches Abbildungsverfahren: Elastizität lässt sich aus der Phasenänderung der Blattfederoszillation ableiten. Problem bei biologischen Objekten (Zellen): Korrelation der Elastizitätsbilder mit strukturellen Komponenten, die mechanische Stabilität verleihen (z.B. Cytoskelett). 7-38 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie AFM einzelner Moleküle (1) Sensitivität von AFMs liegt in der Größenordnung von wenigen Pikonewton Messung der Stärke von biologischen und chemischen Bindungen möglich Untersuchung des Verhaltens einzelner Moleküle unter mechanischer Belastung Spezifische Sonden: (z.B.) Fixierung von Proteinen oder niedermolekularen Liganden an der Spitze. Kraftspektroskopie Ortsaufgelöste Detektion von spezifischen Bindungen und Bestimmung der zwischenmolekularen Kräfte: Proteine – Liganden Proteine – Nucleinsäure Antikörper – Antigene zwischen Zellen Abb. Kraftmessungen an einzelnen Molekülen: Dabei wird die Spitze zum Beispiel mit Liganden beschichtet und mit Rezeptoren, welche an einen Probenträger gebunden sind, in Kontakt gebracht. Zur Bestimmung der molekularen Bindungskräfte werden beide Oberflächen voneinander getrennt. Die Abrisskraft zwischen den Molekülen ist geschwindigkeitsabhängig und nimmt mit steigender Trennungsgeschwindigkeit zu. Die Trennung einzelner Moleküle lässt sich mit einer Zwei-Zustands-Energielandschaft beschreiben. Durch Trennen der Moleküle überschreitet man die Energiebarriere, welche den gebundenen (G) vom ungebundenen (U) Zustand trennt. Hierbei entspricht ΔG† der Aktivierungsenergie, um die beiden Moleküle voneinander zu trennen, k0 der natürlichen Übergangsrate und Δx der Breite des Potenzials. Aus der Geschwindigkeitsabhängigkeit der Abrisskraft können k0 und Δx ermittelt werden. 7-39 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie AFM einzelner Moleküle (2) Tab. Ungefähre Abrisskräfte chemischer und biologischer Bindungen (bei einer Ziehgeschwindigkeit von etwa 500 nm/s) Bindungstyp Abrisskraft Kovalente Bindung 1 nN WW zwischen zwei Zellen 500 pN Biotin-Avidin-Bindung 200 pN Antikörper-Antigen-Bindung < 200 pN Protein-Zucker-Bindung 100 pN Protein/Protein- oder Protein-DNA-WW 10-50 pN Wasserstoffbrücke wenige pN Weitere Anwendungen der Kraftspektroskopie: Messung der Elastizität von einzelnen Polysaccharid- oder Nucleinsäuresträngen Kontrollierte Entfaltung von einzelnen Proteinen Abb. Ein Proteinkonstrukt, welches aus mehreren (hier Immunoglobulin-27-)Domänen besteht, kann mit dem Rasterkraftmikroskop gestreckt werden. Dabei entfaltet jede Domäne in einem einzelnen Schritt und die Abrisskraft kann gemessen werden. Das charakteristische Sägezahnmuster in der Kraftabstandskurve kommt dabei durch die sequenzielle Entfaltung der Domänen zustande. 7-40 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie 7.6 Rasterkraftmikroskopie Adhäsion von Zellen / Wechselwirkung von Zellen (BioAFM) [http://www.jpk.com/] 7-41 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie Supplement Chemische Kraftmikroskopie (CFM ) Die chemische Kraftmikroskopie (chemical force microscopy, CFM ) ermöglicht die nanometergenaue topographische und spezifische chemische Abbildung beliebiger Oberflächen. Dabei verwendet sie chemisch einheitlich modifizierte Sondenspitzen und verschiedene liquide Abbildungsmedien, so dass immer nur eine ganz spezifische Wechselwirkung mit der Oberfläche auftritt. Eine speziell aufbereitete Messsonde (Sondenradius kleiner 3 nm) der Rasterkraftmikroskopie wird mit einer einzigen chemischen Endgruppe, wie zum Beispiel –OH, –CH3, –CF3, –NH2 oder –COOH dicht belegt. Durch die nun chemisch einheitliche Oberfläche der Sonde und durch die Verwendung von Wasser, gepufferten Lösungen oder Lösungsmitteln wie Hexadekan als Abbildungsmedium wird erreicht, dass – im Gegensatz zur normalen Rasterkraftmikroskopie – nur ganz spezifische Wechselwirkungen zwischen CFM-Sonde und der abzubildenden Oberfläche auftreten. Damit wird eine chemische Selektivität der CFM-Sonde erzielt. Die Stärke der an einem Ort der Oberfläche gemessenen spezifischen Wechselwirkung erlaubt Rückschlüsse über die Dichte der spezifisch detektierten chemischen Endgruppen der Oberfläche. Beim zeilenweisen Abtasten der Oberfläche wird die chemische Sonde an jedem Messpunkt mit der Oberfläche in Kontakt gebracht und wieder getrennt (Digitaler Pulsed Force Mode). Bei diesem physikalischen Vorgang werden simultan die Stärke der Wechselwirkung, die Steifigkeit der Oberfläche sowie weitere chemische bzw. physikalische Größen bestimmt und jedem Bildpunkt zugeordnet. 7-42 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie Supplement 7.7 Optisches Rasternahfeldmikroskop / SNOM Ein optisches Rasternahfeldmikroskop (scanning nearfield optical microscope, SNOM, auch NSOM) umgeht die Auflösungsgrenze des optischen Mikroskops, indem es nur Licht auswertet, das zwischen einer sehr kleinen (100 nm oder weniger) Nahfeldsonde und der untersuchten Probe ausgetauscht wird. Mit dem optischen Rasternahfeldmikroskop kann eine räumliche Auflösung von etwa 20 nm erreicht werden. Rastersondenmethode, die Spitze wird ins Nahfeld der Probe gebracht und mittels eines Regelkreises auf konstantem Abstand gehalten. Für Abstandsreglung gibt es mehrere Methoden: - Prinzip des Rasterkraftmikroskopes - Scherkraft (engl. shear force, Resonanzänderung eines Schwingers, der die Spitze trägt) - Messen und Regeln des Tunnelstroms (Rastertunnelmikroskop, nur bei leitfähigen Proben) Übliche Abstände zwischen Spitze und Probe liegen bei 1–10 nm. Die Nachführung der Spitze liefert ein topografisches Bild der Oberfläche, zusätzlich gewinnt man im Rasternahfeldmikroskop jedoch auch eine optische Information der Oberflächenstruktur. Die optische Auflösung hängt von der Feinheit der Spitze ab und übertrifft diejenige abbildender Lichtmikroskope um ein Vielfaches. „Kombi“ aus Raster(kraft)mikroskop und optischem Mikroskop Vorteile gegenüber den nichtoptischen Rastersondenverfahren: bekannte Kontrastmechanismen der konventionellen optischen Mikroskopie genutzt können werden, Probe wird zerstörungsfrei untersucht. Nachteile des SNOM sind - die hohen Kosten, da zusätzlich das Rastersonden-Prinzip angewendet werden muss - Schwierigkeiten bei der Auswertung der erhaltenen Daten (Auftreten von Artefakten) - noch bestehende theoretische Probleme der Beschreibung der Kontrastentstehung 7-43 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik 7. Mikroskopie Supplement 7.7 Optisches Rasternahfeldmikroskop / SNOM Vergleich klassisches Mikroskop und Nahfeldmikroskop Auf beiden Bildern sind Kohlenstoff-Nanoröhren abgebildet. (A) Aufnahme mit einem klassischen Mikroskop (B) Aufnahme mit einem Nahfeldmikroskop Quelle: http://www.schilbe.de/archiv/elektrik.htm 7-44 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik Betriebsarten (Modi) Beleuchtungsmodus Sammelmodus 7. Mikroskopie Supplement 7.7 Optisches Rasternahfeldmikroskop / SNOM Abstandabhängigkeit (Nahfeldeinfluss) Spitzentypen mit Apertur 7-45 29.01.2012 aperturlose Spitze Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik leitfähige Spitze 7. Mikroskopie 7.7 SNOM Supplement Single molecule detection by NSOM A 40 nm optical resolution near-field ‘zoom-in’ on the indicated area (3.2 × 3.2 μm2) in the bright-field image of a fibroblast expressing LFA-1-GFP. GFP excitation is accomplished using 488 nm light (Ar-Kr laser line) linearly polarized along 90°. Fluorescence is collected by a 1.3 numerical aperture oilimmersion objective in combination with standard optical filters. A polarizing beam-splitter cube (Newport, Fountain Valley, CA) is used to split the fluorescence signal into two perpendicular polarized components. Both signals are then detected by photoncounting avalanche photodiode detectors. The red/green colorcoding of the signals reflects the orientation of the GFP molecules in the plane of the sample. Examples of clustered molecules (arrows) as well as examples showing clear singlemolecule detection sensitivity are indicated (circles and squares). The squares show the fast-blinking behavior typical of single molecule GFP fluorescence. The circles present demonstrations of discrete photodissociation phenomena. B Estimation of the resolution in the near-field image. This figure shows a line trace through the feature marked with the hexagon in the near-field image. The full width at half maximum (FWHM; arrows) of such traces can be used to obtain an estimate for the maximal resolution (half the FWHM) in the near-field image. On this basis, we estimate the resolution in the near-field image to be ∼40 nm. [C.G. Figdor et.al., Journal of Cell Science 114, 4153-4160 (2001)] 7-46 29.01.2012 Michael Wörner – Fakultät für Chemieingenieurwesen Instrumentelle Bioanalytik