Jetzt unabhängige Virusfamilie mit 13 Mitgliedern
Werbung

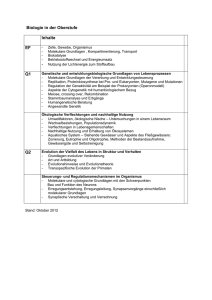
Polyomaviridae Transformation/Polyomavirus Taxonomie Ehemals Unterfamilie der Papovaviridae; Jetzt unabhängige Virusfamilie mit 13 Mitgliedern Molekulare Virologie Ruth Brack-Werner; SS 2008 Mitglieder der Polyomaviridae Molekulare Virologie Virus Wirt Polyomavirus (PyV) Maus K-Papovavirus (KPV) Maus Simian Virus 40 (SV40) Rhesus Affe Lymphotropic Papovavirus (LPV) Afrikanische Grüne Meerkatze Simian agent 12 (SA12) Pavian Stump-tailed macaque virus (STMV) Makake BK Virus Mensch JC Virus Mensch Budgerigar fledgling disease virus Vogel Rabbit kidney vacuolating virus (RKV) Kaninchen Hamster Papovavirus Hamster Transformation/Polyomavirus Ruth Brack-Werner; SS 2008 Entdeckung der Polyomaviren Transformation/Polyomavirus Maus Polyomavirus: entdeckt 1953 von Ludwig Gross bei Untersuchungen zur Übertragung von murinen Leukämien SV4O: entdeckt 1960 von Sweet and Hillemann in Nierenzellen aus der Grünen Afrikanischen Meerkatze. Molekulare Virologie Ruth Brack-Werner; SS 2008 Gemeinsame Merkmale der Polyomaviren Transformation/Polyomavirus 1. Genom: Ringförmiges ds-DNA Molekül (Minichromosom!); ca. 5000 Bp; Ähnliche Genom-Organisation 2. Vermehrung: sehr enges Wirtsspektrum:Vermehrung hauptsächlich in Epithelzellen und Fibroblasten; 3. Unbehüllt: Resistent gegen Lösungsmittel; 4. Maligne Transformation von Zellen in Kultur; 5. Verursachen nicht Tumore in ihren natürlichen Wirten (z.B. SV40 in Rhesus Affen). Molekulare Virologie Ruth Brack-Werner; SS 2008 Einige wichtige Entdeckungen der Zellbiologie, die an Polyaomviren gemacht wurden. Transformation/Polyomavirus 1. Supercoiled DNA 2. Chromatin/Nucleosomen 3. Ursprung der DNA Replikation bei Eukaryonten 4. Enhancer 5. Promoter-Organisation (TATA Box) 6. Alternatives Splicing 7. p53 Molekulare Virologie Ruth Brack-Werner; SS 2008 Struktur von Maus Polyomavirus-Partikel Transformation/Polyomavirus VP1: bildet 72 Pentamere, bestehend aus je 5 Moleküle VP1; VP1 ist das einzige nach aussen gerichtete virale Strukturprotein; VP1 bindet an Oligosaccharide mit SialylSäureresten auf der Zelloberfläche Jedes VP1 Pentamer ist mit einem Molekül VP2 oder VP 3 verbunden. Graphische Nachbildung eines Viruspartikels: Durchmesser ca. 45 nm Aus Fields Virology 4th edition, 2002, Chapter 41, Lippincott, Williams and Wilkins, 2002 Fig. 63-1c Molekulare Virologie Ruth Brack-Werner; SS 2008 Struktur des Polyomavirus-Genoms Transformation/Polyomavirus Nukleosomen: Enthalten Histone Ringförmige Supercoiled DNA (= Eipsom) Nukleosomenfreie Region SV 40 “Minichromosom” Elektronen Mikroskopische Aufnahme von SV40 DNA Aus Fields Virology 4th edition, 2002, Chapter 41, Lippincott, Williams and Wilkins, 2002 Fig. 63-1a, b Molekulare Virologie Ruth Brack-Werner; SS 2008 Verlauf der SV40 Replikation Transformation/Polyomavirus Idealisierte zeitliche Abfolge T-Antigen Virale Genome freigesetzte Viruspartikel Zelluläre DNA Synthese Aus Fields Virology 4th edition, 2002, Chapter 63, Lippincott, Williams and Wilkins, 2002 Fig. 63-5 Molekulare Virologie Ruth Brack-Werner; SS 2008 Zelluläre Rezeptoren für Polyomaviren Transformation/Polyomavirus Virus Rezeptor Moleküle/Zelle Zell-Tropismus Polyomavirus (PyV) N-verknüpfte Glykoproteine mit SialylSäure-Resten 25.000 Weit verbreit Simian Virus 40 (SV40) MHC Klasse I Proteine 90.000 Weit verbreitet Lymphotropic Papovavirus (LPV) O-verknüpfte Glykoproteine mit Sialylsäure-Resten 1.800 B-Lymphozyten BK Virus Glykolipid mit SialylsäureResten unbekannt Weit verbreitet JC Virus N-verknüpfte Glykoproteine mit SialylSäure-Resten 50.000 Oligodendrozyten, Astrozyten, BLymphozyten Molekulare Virologie Ruth Brack-Werner; SS 2008 Sialylsäuren http://www.bme.jhu.edu/~kjyarema/monosaccharides/natural%20si alic%20acids/natural%20sialic_acids.htm Transformation/Polyomavirus Molekulare Virologie Haüfigste Form der Sialylsäure beim Menschen Ruth Brack-Werner; SS 2008 Transformation/Polyomavirus http://www.bme.jhu.edu/~kjyarema/monosaccharides/natural%20si alic%20acids/natural%20sialic_acids.htm Sialylsäuren sind weitverbreitet auf Zelloberflächen Molekulare Virologie Ruth Brack-Werner; SS 2008 Replikationszyklus Replikationszyklus Polyomaviren Principles of Virology, 2004. Flint SJ, Enquist LW, Racaniello VR, Skalka AM, 2nd edition. ASM Press. Appendix, Fig. 16. Expression von SV40 Proteinen: Transformation/Polyomavirus Strategien zur maximalen Nutzung des Genoms 1. Eine Region zur Regulation von verschiedenen Prozessen; 2. Multi-funktionelle virale Regulationsfaktoren; 3. Alternatives Splicing; 4. Multi-cistronische mRNAs; 5. Nutzung von mehreren Leserastern; 6. Zeitliche Koordinierung von Expressionsphasen. Molekulare Virologie Ruth Brack-Werner; SS 2008 Das SV40 Genom Synthese viraler Genome Early Late Aus Fields Virology 4th edition, 2002, Chapter 63, Lippincott, Williams and Wilkins, 2002 Fig. 63-2 Die Regulatorische Region des SV40-Genoms Transformation/Polyomavirus Aus Fields Virology 4th edition, 2002, Chapter 63, Lippincott, Williams and Wilkins, 2002 Fig. 63-3a Molekulare Virologie Ruth Brack-Werner; SS 2008 Eigenschaften des SV40 T-Antigens Transformation/Polyomavirus 1. Sequenzähnlichkeiten zu T-Antigenen aus anderen Polyomaviren verschiedener Viren; 2. Post-translationelle Modifikationen: 1. Phosphorylierung; 2. 0-Glykosylierung ; 3. Poly-ADP-Ribosylierung; 4. Acetylierung am Amino-terminus; 3. Nukleäre Lokalisation;(NLS) 4. 5 x 105 bis 5x106 Moleküle pro lytisch infizierte Zellen Molekulare Virologie Ruth Brack-Werner; SS 2008 Funktionen des SV40 T-Antigens Transformation/Polyomavirus 1. 2. 3. Optimierung der Zelle für die Virusvermehrung: 1. Vorantreiben des Zellzyklus bis zur S-Phase:Befreiung von E2F 2. Verhinderung der Apoptose: Sequestrierung von p53 Transkriptionsregulation: 1. Autoregulation (Effekt Konzentrationsabhängig); 2. Aktivierung der späten Transkription DNA-Replikation: 1. Fördert die Entwindung der DNA Stränge; 2. Rekrutierung von DNA Polymeraseα-Primase für die Synthese der OkazakiFragmente und die DNA Polymerase δ für die Verlängerung der neusynthetisierten DNA Stränge; Molekulare Virologie Ruth Brack-Werner; SS 2008 Domänenstruktur des T-Antigens Transformation/Polyomavirus Aus Fields Virology 4th edition, 2002, Chapter 63, Lippincott, Williams and Wilkins, 2002 Fig. 63-6a Molekulare Virologie Ruth Brack-Werner; SS 2008 Polyomaviren bei Menschen Transformation/Polyomavirus BK-Virus im Urin JC-Virus im infizierten Oligodendrozyten im Gehirn Aus Fields Virology 4th edition, 2002, Chapter 64, Lippincott, Williams and Wilkins, 2002 Fig. 64-1 Molekulare Virologie Ruth Brack-Werner; SS 2008 JC-Virus und BK-Virus Genome Transformation/Polyomavirus Aus Fields Virology 4th edition, 2002, Chapter 64, Lippincott, Williams and Wilkins, 2002 Fig. 64-2 Molekulare Virologie Ruth Brack-Werner; SS 2008 Merkmale der JC- und BK-Virus Infektion Transformation/Polyomavirus 1. Infektion: in der frühen Kindheit 2. Persistenz: Nierenepithel; Lymphozyten; Knochenmark (JC-Virus) 3. Pathogenes Potential: Hauptsächlich bei Immun-Supprimierten Personen 1. BK-Virus? 2. JC-Virus: PML Progressive multifokale Leukoenzephalopathie 4. Onkogenes Potential: 1. Maligne Tumore in Nagetieren (JC und BK Virus); 2. Maligne Gehirntumore in Neuweltaffen nach Inoculation direkt in den Schädel (lange Inkubationszeit); Molekulare Virologie Ruth Brack-Werner; SS 2008 Progressive multifokale Leukoenzephalopathie Transformation/Polyomavirus (PML) 1. Demyelinisierungs-Herde im Gehirn durch den Tod von infizierten Oligodendrozyten; 2. Neuropathologie: Multifokale Plaques: enthalten u.a. Makrophagen, Astrozyten mit anomalen Morphologien, veränderte Oligodendrozyten 3. Klinisches Erscheinungsbild: Muskelschwäche, Geh-, Sehschwierigkeiten, Lähmungen, kognitive Störungen (“Demenz”). 4. Überlebenszeit: Wenige Monate bis zu einem Jahr nach Auftreten von Symptome; 5. Diagnose: endgültige Diagnose nur durch Nachweis von DNA in Gehirnmaterial; Virologie 6.Molekulare Behandlung: deutliche Besserung durch HAART Ruth Brack-Werner; SS 2008 Transformation/Polyomavirus Molekulare Virologie Ruth Brack-Werner; SS 2008