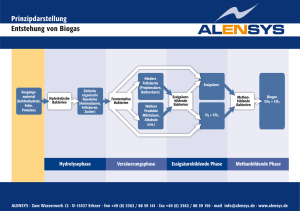
Bewegungsanalyse von Bacillus amyloliquefaciens
Werbung

Bewegungsanalyse von Bacillus amyloliquefaciens durch OnlineMikroskopie mit automatisierter Wegverfolgung vorgelegt von Andreas Ziegler (M.Sc.) geb. in Ludwigshafen am Rhein Von der Fakultät III – Prozesswissenschaften – der Technischen Universität Berlin zur Erlangung des akademischen Grades Doktor der Ingenieurwissenschaften - Dr.-Ing. genehmigte Dissertation Promotionsausschuss: Vorsitzender: Prof. Dr. Peter Neubauer Gutachter: Prof. Dr. Ulf Stahl Gutachter: Prof. Dr. Matthias Rädle Tag der wissenschaftlichen Aussprache: 12. Februar 2015 Berlin 2015 D83 Danksagung Die vorliegende Arbeit entstand in Kooperation des Fachbereichs Angewandte und Molekulare Mikrobiologie der Technischen Universität Berlin mit dem Institut für Prozessmesstechnik und innovative Energiesysteme der Hochschule Mannheim im Zeitraum von August 2010 bis Februar 2015. Mein besonderer Dank gilt meinen Betreuern Prof. Dr. Ulf Stahl und Prof. Dr. Matthias Rädle für die Vergabe des Themas, das entgegengebrachte Vertrauen, die Betreuung über den gesamten Zeitraum, die konstruktiven Diskussionen in zahlreichen Projekttreffen, die stets hilfreichen Vorschläge und für die intensive Unterstützung. Prof. Dr. Peter Neubauer danke ich für die Übernahme des Vorsitzes der wissenschaftlichen Aussprache. Meinen Kollegen vom Institut für Prozessmesstechnik und innovative Energiesysteme danke ich für das ausgezeichnete Arbeitsklima im Institut. Die hohe Hilfsbereitschaft in fachlichen Belangen wie auch der Umgang im privaten gab mir das nötige Umfeld für die lange und angenehme Zusammenarbeit. Besonders hervorheben möchte ich: Patrick Dörnhofer, Viktoria Kapoustina, Alexander Kron, Isabel Medina, Florian Ries, Lukas Schmitt, Daniel SchockKusch und Tobias Teumer. Den Mitarbeitern des Fachbereichs Angewandte und Molekulare Mikrobiologie der Technischen Universität Berlin, im besonderen Sandra Dounia, danke ich für die gute Zusammenarbeit. Weiterhin gilt den zahlreichen Studenten, die an der Durchführung und Auswertung der Fermentationen beteiligt waren, ebenso mein Dank. Neben den hilfreichen Informationen möchte ich mich bei der Firma ABiTEP GmbH vor allem für die Bereitstellung des Produktionsmediums und der Bakteriensporen bedanken. Bei meinem Schwiegervater Eberhard möchte ich mich für das Korrekturlesen bedanken. Ich danke meinen Eltern für die Unterstützung in all meinen Vorhaben. Meiner Frau AnneMarie danke ich besonders für den liebevollen Rückhalt die ganze Zeit über und meinem Sohn Fjonn für die Bereicherung, die er mir brachte. Inhaltsverzeichnis Inhaltsverzeichnis I Bestimmung der Vitalität von Mikroorganismen durch Online-Mikroskopie .......... 1 1 Einführung ........................................................................................................................ 1 2 Online-Mikroskopie ......................................................................................................... 6 2.1 In-Situ-Mikroskopie .................................................................................................... 6 2.2 Durchfluss-Mikroskope: ............................................................................................ 11 2.3 Bildverarbeitung ........................................................................................................ 12 3 Bewegung von Bakterien ............................................................................................... 14 II Bewegungsanalyse von Bacillus amyloliquefaciens ..................................................... 18 4 Einleitung ........................................................................................................................ 18 5 Zielsetzung ...................................................................................................................... 23 6 Material und Methoden ................................................................................................. 24 6.1 Verwendeter Stamm und verwendete Medien........................................................... 24 6.2 Bioreaktor .................................................................................................................. 25 6.3 Versuchsdurchführung............................................................................................... 28 6.4 Online-Mikroskopie mit automatisierter Serienbildaufnahme .................................. 29 6.4.1 Durchflussküvette mit flexibler Schichtdicke .................................................... 30 6.4.2 Messsystem zur Bestimmung der kritischen Scherkräften ................................ 34 6.4.3 Peripherie für den automatisierten Betrieb......................................................... 34 6.4.4 Kamera und Mikroskop ...................................................................................... 37 6.4.5 Auflösungsvermögen der gewählten Komponenten .......................................... 37 6.4.6 Bewegungsunschärfe .......................................................................................... 40 6.4.7 Bildspeicherung .................................................................................................. 41 6.5 Automatisierte Bildserienanalyse .............................................................................. 41 6.5.1 Bildanalyse ......................................................................................................... 41 6.5.2 Bildserie laden .................................................................................................... 42 6.5.3 Indizierte Seriensubtraktion ............................................................................... 43 6.5.4 Bestimmung der Partikelkenndaten ................................................................... 44 6.5.5 Zuordnung der Bakterien zu den Kenndaten...................................................... 45 6.5.6 Nachbearbeitung der Zuordnung ........................................................................ 47 6.5.7 Bereitstellung des Ergebnisses ........................................................................... 47 6.6 Programm zur interaktiven Erfassung der Bewegungsmuster .................................. 48 6.7 Statistische Auswertung ............................................................................................ 49 I Inhaltsverzeichnis 7 Ergebnisse ....................................................................................................................... 51 7.1 7.1.1 Vermessung der Medien..................................................................................... 51 7.1.2 Bestimmung der Scherkräfte .............................................................................. 52 7.1.3 Auslegung der Optik .......................................................................................... 55 7.1.4 Auslegung der Bildanalyse................................................................................. 56 7.1.5 Integration des Gesamtsystems .......................................................................... 57 7.2 8 Technische Ergebnisse .............................................................................................. 51 Ergebnisse aus den Beispielfermentationen .............................................................. 59 7.2.1 Validierung der automatischen Bildanalyse ....................................................... 59 7.2.2 Geschwindigkeit und Anzahl beweglicher Bakterien ........................................ 60 7.2.3 Bewegungsmuster der beweglichen Bakterien .................................................. 73 Diskussion ....................................................................................................................... 78 8.1 Verlauf der Fermentationen ....................................................................................... 78 8.2 Bewegungsanalyse von Bacillus amyloliquefaciens FZB42 ..................................... 82 8.3 Praxistauglichkeit des Online-Mikroskopie-Systems ................................................ 87 9 Ausblick ........................................................................................................................... 90 10 Zusammenfassung .......................................................................................................... 93 11 Verzeichnisse................................................................................................................... 94 11.1 Abkürzungen und Formelzeichen .............................................................................. 94 11.2 Tabellenverzeichnis ................................................................................................... 95 11.3 Abbildungsverzeichnis .............................................................................................. 96 11.4 Literaturverzeichnis ................................................................................................... 97 Anhang .................................................................................................................................. 108 LabVIEW-Unterprogramm „Wegverfolgung“ ................................................................... 108 Statistische Analyse von Fermentation 1............................................................................ 110 Statistische Analyse von Fermentation 2............................................................................ 111 Statistische Analyse von Fermentation 3............................................................................ 112 Statistische Analyse von Fermentation 4............................................................................ 113 Statistische Analyse von Fermentation 5............................................................................ 114 Statistische Analyse von Fermentation 6............................................................................ 115 Fermentation 6 – Verteilung der Bewegungsmuster .......................................................... 116 Fermentation 7 – Verteilung der Bewegungsmuster .......................................................... 116 Vergleich der Wandlung in Grauwerte ............................................................................... 117 II Einführung I 1 Bestimmung der Vitalität von Mikroorganismen durch Online-Mikroskopie Einführung Die Zelldichte und Zellvitalität sind zwei Kernparameter in der Kultivierung von Mikroorganismen. Während die Zelldichte verhältnismäßig einfach zu definieren ist und meist in Anzahl an Zellen pro Volumeneinheit oder pro Gewichtseinheit angegeben wird, ist die Vitalität einer Kultur ein abstrakter Parameter (Kell et al. 1990). Häufig wird als Vitalität die Fähigkeit der einzelnen Zelle verstanden, eine Kolonie auf Nährboden zu bilden. Es sind jedoch Zustände von Bakterien bekannt, in denen keine Koloniebildung auf Nährboden erfolgt, jedoch weiterhin eine Zellenaktivität nachweisbar ist. Der Begriff der Vitalität von Mikroorganismen wird von verschiedenen Quellen unterschiedlich verwendet (Davey 2011). Viele Parameter, angefangen bei der Fähigkeit zur Zellteilung über den Nachweis von Stoffwechselaktivitäten, den Zustand der Zellwand bis hin zur Fähigkeit der aktiven Bewegung oder der Zellform, können zur Bewertung der Vitalität herangezogen werden. Kell et al. (1998) differenzierten in diesem Zusammenhang die verschiedenen physiologischen Stadien von Bakterien. Es werden zwei Modelle vorgeschlagen. In Abbildung 1 werden die beiden Modelle veranschaulicht. Abbildung 1: Physiologische Stadien einer Bakterienzelle Das Kreisdiagramm (links) veranschaulicht vier wichtige physiologische Stadien von Bakterien. Die Pfeile zeigen mögliche Übergänge zwischen den Stadien. Es gibt im Kreisdiagramm kein Feld für nicht tödlich verletzte Zellen. Diese sind mit den vitalen Zellen unter aktiv und kultivierbar zusammengefasst. Das Baumdiagramm (rechts) zeigt eine experimentelle Vorgehensweise zur Einteilung der Zellen in ihre wichtigsten Stadien. Unter selektivem Medium ist ein Medium zu verstehen, welches das Wachstum der meisten Zellen hemmt, z.B. durch Antibiotika oder Gallensalze. Nur unverletzte Zellen sollten sich in diesem Medium vermehren können. Der wachstumshemmende Stoff des selektiven Mediums muss je nach Gattung gewählt werden, da einige wenige Gattungen z.B. durch Antibiotika kaum bis nicht gehemmt werden (Kell et al. 1990) (modifiziert). 1 Einführung Entsprechend der dargestellten Modelle werden zum einen die Zellen nach zwei unabhängigen Parametern beurteilt: aktiv und inaktiv, sowie kultivierbar bzw. nicht kultivierbar. Daraus ergeben sich vier Stadien, in denen sich die Zelle befinden kann. Zum anderen können die Bakterien in fünf Kategorien eingeteilt werden: vital und unverletzt (1), verletzt (2), ruhend (3), aktiv, aber nicht kultivierbar (4) und tot (5). Durch eine experimentelle Vorgehensweise ist eine Einteilung der Bakterien in die fünf wichtigsten physiologischen Stadien möglich. Zunächst wird beurteilt, ob die Zellen morphologisch intakt erscheinen. Ist dies nicht der Fall, werden sie als tot klassifiziert. In der nächsten Untersuchung wird die Kultivierbarkeit unter Normalbedingungen getestet. Ist eine Vermehrung unter Normalbedingungen nicht möglich, werden die Zellen, je nachdem ob Stoffwechselaktivität nachgewiesen wird, als „aktiv, aber nicht kultivierbar“ oder als „tot“ bestimmt. War die Kultivierung unter Normalbedingungen erfolgreich, es konnte aber keine Stoffwechselaktivität nachgewiesen werden, wird die Zelle als „ruhend“ bezeichnet. Ansonsten erfolgt der Versuch, die Zellen auf einem wachstumshemmenden Medium (z.B. durch Zugabe von Gallensalzen oder Antibiotika) zu kultivieren. Ist die Kultivierung dort erfolgreich, sind die Zellen vital und unverletzt. Ist die Vermehrung nicht möglich, werden die Zellen als verletzt eingeordnet (Kell et al. 1990). Über Online-Mikroskopie und automatische Bildauswertung können viele dieser Parameter in Echtzeit erfasst werden. Bei Prozesszeiten von einigen Stunden oder Tagen können auch Systeme mit Messintervallen von einigen Minuten als Echtzeit-Systeme bezeichnet werden. Vor allem die Morphologie (Größe, Form) der Zellen wird unter Verwendung der Mikroskopie häufig zur Beurteilung der Vitalität herangezogen (Pearson et al. 2003; Guez et al. 2010). Auch die Beweglichkeit der Zellen gibt eine Aussage zur Vitalität (Grossart et al. 2001). Häufig wird Ausplattieren und späteres Auszählen der entstandenen Kolonien zum Nachweis und zur Quantifizierung vitaler Bakterien verwendet. Die Methode liefert jedoch erst Stunden oder Tage nach der Probenentnahme Ergebnisse und ist damit nicht zur OnlineProzesskontrolle geeignet (Müller und Nebe-von-Caron 2010; Davey 2011). Die Beurteilung der Vitalität ist auch mit Hilfe der Durchflusszytometrie (Enfors et al. 2001; Davey 2011) möglich. Hierbei wird die zu untersuchende Kulturflüssigkeit bei hoher Geschwindigkeit durch eine enge Messstelle gepumpt. An dieser Engstelle werden die Zellen einzeln von fokussiertem Laserlicht getroffen. Je nach Messprinzip des Durchflusszytometers wird von einem optischen Empfänger das Streulicht oder das Fluoreszenzsignal der Zelle detektiert. Die Signale können so von etwa 1.000 Zellen s-1 erfasst werden (Abu-Absi et al. 2003). Die Intensität des Fluoreszenzsignals bzw. des Streulichtes wird für die Auswertung des Zellzustandes herangezogen. Die Anzahl an Einzelsignalen über eine definierte Zeit lässt Rückschlüsse auf die Zelldichte zu (Hewitt et al. 1999b; Müller und Nebe-von-Caron 2010). Die Ergebnisse aus dem Durchflusszytometer stehen etwa 20 min nach der Probenentnahme zur Verfügung. Der Zeitraum von Probenentnahme bis zum Messergebnis wird vor allem durch die Probenvorbereitung bestimmt. Häufig müssen zur Bestimmung der Vitalität die Bakterien angefärbt werden. Die Färbung kann eine Aussage über den Status der Zelle liefern. 2 Einführung Zur Etablierung dieser Methoden sind z.B. Farbstoffe nötig, die in intakte Zellwände nicht eindringen können. Zellen mit defekter Membran fluoreszieren daher anders als Zellen mit funktionsfähiger Membran. Diese Marker benötigen jedoch eine definierte Einwirkdauer, was eine längere Probenvorbereitung zur Folge hat. Zwei gängige Färbemethoden sind die Vitalfluoreszenzmethode und die 4‘,6-Diamidino-2-phenylindol(DAPI)-Färbung (Hewitt et al. 1999a; Abu-Absi et al. 2003; Müller und Nebe-von-Caron 2010). Bei der Vitalfluoreszenzmethode werden die Farbstoffe Fluorescein-Diacetat (FDA) und Ethidiumbromid verwendet. Lebende Organismen nehmen das FDA auf und wandeln es enzymatisch zu grün fluoreszierendem Fluorescein um. Dieses reichert sich daraufhin in den Zellen an. Bakterien mit beschädigter Zellmembran werden durch das Färbemittel Ethidiumbromid rot eingefärbt (Netuschil 1983; Rehmer 2008). Ein anderer Fluoreszenzmarker wird bei der DAPI-Färbung angewendet. Der Farbstoff reichert sich an Adenin- und Thymin-reichen Regionen der Desoxyribonukleinsäure (DNA) an. Für die Anregung wird ultraviolettes Licht (UV) verwendet. Häufig wird auch Propidiumiodid verwendet, um tote Bakterien zu kennzeichnen. Bei UV-Anregung fluoreszieren die vitalen Bakterien blau, während die nicht vitalen Bakterien gelb fluoreszieren (Ross et al. 1996; Knöfel 2002; Rehmer 2008). Zur Vitalitätsbestimmung sind durch die meist aufwändigen Probenvorbereitungen kaum kürzere Messintervalle als 15 - 30 min realisierbar. Neben den beschriebenen optischen Durchflusszytometern kann an der Messstelle anstelle eines Laserstrahls und eines optischen Empfängers auch eine elektrische Spannung zum Einsatz kommen. Das Messsignal stellt in diesem Fall die Änderung der Leitfähigkeit oder der Kapazität dar. Da sich die Leitfähigkeit bzw. die Kapazität der Zellen von der der Kulturflüssigkeit unterscheiden, kann auf diese Weise ebenfalls die Zelldichte ermittelt werden (Gawad et al. 2001). Eine weitere Methode zur Bestimmung der Vitalität von Bakterien ist die elektrooptische Messung. Der Einfluss eines elektrischen Feldes auf eine Bakteriensuspension hängt von der Polarisierbarkeit der Zellen ab. Zusammensetzung und Struktur der Zellen haben einen messbaren Einfluss auf ihre Polarisierbarkeit (Bunin et al. 2004). Vitale Zellen tendieren zu einer erhöhten Polarisierbarkeit (Dalton et al. 2001). An der Messkammer wird ein elektrisches Feld zwischen 190 und 2100 kHz angelegt und die optische Dichte bei 620 nm gemessen. Das elektrische Feld richtet die polarisierbaren Bakterien aus. Die optische Dichte der Suspension ändert sich je nach Ausrichtung der Bakterien. Die Änderung der optischen Dichte ist abhängig von der Frequenz des elektrischen Feldes und liefert eine Aussage über die Vitalität der Zellen (Junne et al. 2008; Junne et al. 2010). Weiterhin kommen Methoden wie die Messung fokussierter Laserrückstreuung (FBRM) (Pearson et al. 2003; Kougoulos et al. 2005; Höpfner et al. 2010) und die 3-Fold Dynamical Optical Reflectance Measurement (3D-ORM) (Brognaux et al. 2013) für die Qualitätskontrolle in Frage. Beide Methoden sind In-Line-Messungen in Echtzeit. 3 Einführung FBRM liefert eine Partikelgrößenverteilung und kann Partikel ab 0,25 µm detektieren. Das Messverfahren nutzt hierfür die Erfassung von rückwärtigem Laser-Streulicht. Hierzu wird der Laserstrahl über eine rotierende Optik in die Suspension gestrahlt und dort reflektiert sobald er auf einen Partikel trifft. Der Fokuspunkt bewegt sich kreisförmig durch das Medium, das reflektierte Rückstreulicht wird von der Messsonde detektiert (Rudolph et al. 2007). Ausgewertet wird die zeitliche Länge des Reflexes bei bekannter Bewegungsgeschwindigkeit des Lichtfleckes durch die Suspension. Durch dieses Verfahren ist es ebenso möglich bei Bakterien eine Aussage über die Morphologie von Zellverbänden zu treffen. Die Morphologie von einzelnen Bakterien ist jedoch nicht detektierbar (Pearson et al. 2003). 3D-ORM arbeitet ebenso wie FBRM über optische Rückstreuung. Jedoch verfügt die verwendete Optik über eine dynamische Regulierung der Brennweite. Mit Hilfe der rotierenden Optik sowie der dynamischen Brennweite bewegt sich der Fokuspunkt des Lasers wie eine Spirale, nicht kreisförmig, durch das Medium (Brognaux et al. 2013). In Abbildung 2 ist der Aufbau der 3D-ORM-Sonde schematisch dargestellt. Abbildung 2: 3-Fold Dynamical Optical Reflectance Measurement (3D-ORM) Die 3D-ORM-Sonde erzeugt über das rotierende optische System und die dynamische Brennweite einen spiralförmigen Fokuspunkt im Medium. Der Laser und die optische Rückstreuung werden über einen Faserkoppler getrennt. Über einen Photomultiplier als Detektor wird das Signal erfasst. Die Sonde wird über einen DN25-Bioreaktor-Port in das Kulturgefäß eingebracht (Brognaux et al. 2013) (modifiziert). Das reflektierte Signal der 3D-ORM-Sonde wird als Anzahl von Pulsen im zeitlichen Messintervall erfasst. Aus diesem Signal kann die Zelldichte bestimmt sowie Rückschlüsse auf die Vitalität gezogen werden. Die 3D-ORM-Sonde lässt sich über einen DN25Bioreaktor-Port in den Reaktor einführen und ist in-situ autoklavierbar (Brognaux et al. 2013). Durch die Analyse mikroskopischer Aufnahmen ist ebenso eine Auswertung der Vitalität von Bakterien möglich (Singh et al. 1989; Höpfner et al. 2010; Mesquita et al. 2011; Zotta et al. 2012). Etabliert sind z.B. Methoden durch Anfärben der Probe, die durch 4 Einführung Auflichtfluoreszenzmikroskopie untersucht und bildanalytisch ausgewertet werden (Mesquita et al. 2011; Zotta et al. 2012). Die Färbetechniken zur mikroskopischen Untersuchung entsprechen denen der Durchflusszytometrie. Eine zum Teil aufwändige Probenvorbereitung ist somit auch hier erforderlich. Neben der Färbung kann die Beweglichkeit von Bakterien ein Kriterium für die Vitalität sein, da bewegliche Bakterien weder tot noch inaktiv sind (Grossart et al. 2001). Weiterhin können mit Hilfe der Mikroskopie zusätzliche morphologische Parameter bestimmt und für eine genauere Untersuchung herangezogen werden. Die Bestimmung der Vitalität über mikroskopische Aufnahmen ist oft anschaulicher und dadurch weniger abstrakt. Im Fall der Vitalitätsbestimmung über die Beweglichkeit der Bakterien kann eine aufwändige und durch die Farbstoffe zum Teil teure Probenvorbereitung ausbleiben. Durchflusszytometer können z.B. von Becton Dickinson BD Biosciences (Heidelberg, Deutschland), Beckman Coulter (Krefeld, Deutschland) und Partec (Münster, Deutschland) bezogen werden. Eine vollautomatisierte elektrooptische Messung von Bakterienkulturen ist mit dem Messsystem EloTrace von EloSystems (Berlin, Deutschland) durchführbar. Die FBRM-Sonden werden von Mettler-Toledo AutoChem, Inc. (Columbia, USA) und die 3DORM-Sonden von Sequip S+E GmbH (Düsseldorf, Deutschland) vertrieben. 5 Online-Mikroskopie 2 Online-Mikroskopie Ein Messsystem kann als „online“ bezeichnet werden, wenn das Messergebnis schon während hinreichend kleiner Änderungen des Prozesses zur Verfügung steht und somit zur Prozesssteuerung eingesetzt werden kann. Dies wird häufig auch als Echtzeit bezeichnet. Weiterhin werden unter dem Begriff Online-Messsysteme vollautomatisierte Messsysteme verstanden (MacLennan und Kowalski 1996). Mikroskopie-Systeme mit automatisierter Probenentnahme können daher als Online-Messsysteme bezeichnet werden, sobald sie über eine zeitnahe automatische Bildauswertung verfügen. Bei einem In-Situ-Messsystem sind keine Probenentnahmen notwendig, sondern die Messung erfolgt direkt im Medium. 2.1 In-Situ-Mikroskopie Die In-Situ-Mikroskopie ist eine Methode zur direkten Aufnahme mikroskopischer Bilder aus dem Inneren des Bioreaktors (Thomas und Paul 1996; Bluma et al. 2010; Höpfner et al. 2010). Da keine Probenentnahmen notwendig sind, wird die sterile Barriere des Bioreaktors zu keiner Zeit durchbrochen. Die Möglichkeit, den Reaktor mit allen nötigen Einbauten zu autoklavieren, ist ebenso gegeben. Es sind zwei verschiedene Verfahren der In-SituMikroskopie beschrieben, einmal mit theoretischem Probevolumen (Suhr et al. 1995) und einmal mit mechanisch definiertem Probevolumen (Bittner et al. 1998). In der ersten Variante wird über den Fokus des Mikroskops ein theoretisches Probevolumen errechnet und nur Zellen erkannt, die sich in einem definierten Schärfebereich befinden (Suhr et al. 1995; Camisard et al. 2002). Die Variante mit mechanisch definiertem Probevolumen benötigt meist ein mechanisch bewegtes Teil innerhalb der Konstruktion. Beispielsweise wird die Beleuchtung des In-Situ-Mikroskops bewegt, um ein Probevolumen zwischen Beleuchtung und Objektiv zu fixieren (Bittner et al. 1998). Ein Beispiel für ein In-Situ-Mikroskop mit theoretischem Probevolumen ist das In-SituAuflichtfluoreszenzmikroskop von Suhr et al. (1995) (siehe Abbildung 3). Hier findet als Beleuchtung ein gepulster Laser mit einer Wellenlänge von 337 nm Anwendung. Er wird durch einen 45°-Interferenzspiegel reflektiert. Für das Fluoreszenzsignal bei 450 nm ist der Spiegel durchlässig (dichroitischer Spiegel). Mit einem solchen Mikroskop kann interzelluläres NADH und NADPH sichtbar gemacht werden. Das Fluoreszenzsignal ergibt sich aus der Summe beider Stoffe, da diese im selben Bereich angeregt werden (Chance 1979). Ein definiertes Probevolumen ist nötig, um die Zellkonzentration zu bestimmen. Hierzu wird die Schärfeebene des Mikroskops genutzt. Die Bildverarbeitung rechnet unscharfe Objekte heraus, was zu einem theoretisch definierten Probevolumen führt. Eine Kalibrierung mit Standard-Suspensionen ist notwendig (Suhr et al. 1995). 6 Online-Mikroskopie Abbildung 3: In-Situ-Auflichtfluoreszenzmikroskop mit theoretischem Probevolumen Das dargestellte In-Situ-Auflichtfluoreszenzmikroskop (Fluoreszenz-Absorptionswellenlänge(ex): 337 nm, Fluoreszenz-Emissionswellenlänge(em): 450 nm) arbeitet mit einem theoretischen Probevolumen. Über einen Interferenzspiegel wird die Beleuchtung eingekoppelt. Das von der Probe emittierte Licht passiert in die entgegengesetzte Richtung den Interferenzspiegel und wird über einen weiteren Spiegel und einen Bandpassfilter auf die CCD-Kamera abgebildet. Der Fokus kann mit einer Mikrometerschraube manuell justiert werden (Suhr et al. 1995; Bluma et al. 2010) (modifiziert). Ein mechanisch definiertes Probevolumen erfordert einen komplexeren Aufbau des Mikroskops sowie einen tieferen Einbau in den Behälter. In Abbildung 4 ist die Skizze des InSitu-Mikroskops von Bittner et al. (1998) dargestellt. Die Beleuchtungseinheit ist hierbei beweglich und fixiert über eine Silikonmembran das Probevolumen unter dem Deckglas. Die Schärfeebene kann auch hier manuell mit einer Mikrometerschraube eingestellt werden. Für längere Fermentationen ist es sinnvoll, eine Reinigungsmöglichkeit für das Mikroskop vorzusehen. Besonders bei In-Situ-Mikroskopen mit mechanisch definiertem Probevolumen kann eine starke Verschmutzung der Deckgläser zu Störungen führen. Frerichs (2000) integrierte in seiner Arbeit das Intrac®-System von Mettler-Toledo (Gießen) in ein In-SituMikroskop. Mit dieser Technik ist die Reinigung des Proberaums des Mikroskops während des Prozesses möglich. Bei sachgemäßer Verwendung wird die sterile Barriere des Bioreaktors zu keiner Zeit verletzt. Die Reinigungsmöglichkeit erlaubt somit den durchgängigen Betrieb des In-Situ-Mikroskops. 7 Online-Mikroskopie Abbildung 4: In-Situ-Mikroskop mit mechanisch definiertem Probevolumen Die Beleuchtungseinheit mit Kondensor ist bei diesem In-Situ-Mikroskop beweglich. Zur Bildaufnahme fährt der Kondensor nach oben. An dessen Rand befindet sich eine Silikonmembran. Bei der Aufwärtsbewegung des Kondensors wird das Probevolumen zwischen Kondensor und Deckglas eingeschlossen. Die Probe ist somit nicht mehr der Strömung ausgesetzt und kann ruhend aufgenommen werden. Über einen Spiegel wird die Objektebene auf die CCD-Kamera abgebildet. Der Fokus kann über eine Mikrometerschraube manuell eingestellt werden (Bittner et al. 1998; Bluma et al. 2010) (modifiziert). Eine Weiterentwicklung dieses Mikroskops wird von Frerichs (2002) und Joeris (2002) beschrieben. Neben der automatischen Reinigungsmöglichkeit verfügt dieses In-SituMikroskop über eine mittels Motor verstellbare Schärfeebene. Weiterhin lässt sich die Schichtdicke des Proberaums mit Hilfe eines weiteren Motors verstellen (Höpfner et al. 2010). Eine weitere Optimierung dieses Mikroskops wird in der Arbeit von Prediger (2013) präsentiert. Die Genauigkeit der Motoren sowie die Abbildungstreue wurden hier verbessert. Kommerziell erhältliche In-Situ-Mikroskope sind das Particle Vision Microscope (PVM V819) von Mettler-Toledo (Columbia, USA), Particle Image Analysis (PIA) von Sequip S+E GmbH (Düsseldorf, Deutschland) und EnviroCam von EnviroCam Inc. (Colmar, USA) (siehe Tabelle 1). 8 Online-Mikroskopie Tabelle 1: Übersicht von kommerziell verfügbaren In-Situ-Mikroskopen Die Tabelle zeigt eine Übersicht über die kommerziell verfügbaren In-Situ-Mikroskope. PVM V819 von Mettler-Toledo (Columbia, USA), EnviroCam von EnviroCam Inc. (Colmar, USA) und PIA von Sequip S+E GmbH (Düsseldorf, Deutschland). Bei allen drei In-Situ-Mikroskopen kann der Sondenkopf durch Dampf sterilisiert werden. Weiterhin ist bei keinem eine manuelle Fokussierung der Schärfeebene notwendig. PVM V819 Beleuchtung Auflösungsgrenze Bildbereich veröffentlichte Anwendungen Auflicht 2 µm 1075 x 825 µm Kristallisation (Barrett und Glennon 2002; Duffy et al. 2012) Umkristallisation (O'Sullivan et al. 2003; O'Sullivan und Glennon 2005) EnviroCam Durchlicht oder Auflicht 4 bis 30 µm 2048 x 2048 µm bis 15 x 15 mm Größenverteilung von Luftblasen im Fermenter (Junker et al. 2007) PIA Durchlicht 1 bis 10 µm 3586 x 2690 µm Kristallisation (Qu et al. 2006) Die genannten In-Situ-Mikroskope werden alle über einen Standard-Stutzen in den Bioreaktor eingebracht, ähnlich einer pH-Sonde. Weiterhin ist es möglich, durch ein Sichtfenster die Flüssigkeit in einem Reaktor mit einem In-Situ-Mikroskop zu untersuchen, ohne eine zusätzliche Sonde einzubringen. Kougoulos et al. (2005) stellen in ihrer Arbeit eine Möglichkeit des „Process Video Imaging“ (PVI) vor. Hierbei wird mit einem Videomikroskop durch ein Sichtfenster das Innere des Reaktors beobachtet. Die Beleuchtung findet durch ein Stroboskop ebenso von außen statt. Da die verhältnismäßig großen Sichtfenster stabiler sein müssen als die Saphirscheiben der eingebauten In-Situ-Mikroskope ist nur eine geringe Vergrößerung möglich. Die von Voss et al. (2012) entwickelte Immersions-Optik für die horizontale OnlineMikroskopie kann ebenso an einem Sichtfenster eines Bioreaktors integriert werden. Durch die Wasserimmersion ist eine höhere Vergrößerung möglich. Die Immersions-Optik sorgt für einen durchgehenden Wasserfilm zwischen Scheibe und Objektiv. Eine vereinfachte Darstellung der Immersions-Optik ist in Abbildung 5 gegeben. 9 Online-Mikroskopie Abbildung 5: Immersions-Optik für die horizontale Online-Mikroskopie Ein Wasserfilm wird durch die Injektion kleiner Mengen an Wasser zwischen einem Sichtfenster eines Bioreaktors und einem Mikroskop-Objektiv erzeugt. Die überschüssige Menge an Wasser wird kontinuierlich abgesaugt (Voss et al. 2012) (modifiziert). Durch den Wasserfilm kommt es zu einer geringeren Brechung als an der Glas-Luft-GlasGrenze (𝑛𝐺𝐺𝐺𝐺 = 1,46, 𝑛𝐿𝐿𝐿𝐿 = 1,00), da die Differenz der Brechungsindizes an einer GlasWasser-Glas-Grenze (𝑛𝑊𝑊𝑊𝑊𝑊𝑊 = 1,33) deutlich geringer ist. Hierdurch kann eine größere Apertur und damit eine bessere Auflösung bei großem Objektabstand, der durch das dicke Sichtfenster bedingt ist, erlangt werden. Die In-Situ-Mikroskopie hat erhebliche Vorteile gegenüber einer Online-Mikroskopie, die eine Probenentnahme erfordert. Jedoch sieht die Variante mit theoretischem Probevolumen keine Fixierung des Mediums vor. Somit ist keine Nachverfolgung von aktiven Bewegungen der einzelnen Organismen möglich. Außerdem wird über den theoretischen Schärfebereich eine hohe Schichtdicke von etwa 7 – 10 µm (Suhr et al. 1995; Camisard et al. 2002) notwendig. Die Variante mit mechanisch definiertem Probevolumen sieht zwar eine Fixierung der Probe vor, die Schichtdicke bei der beschriebenen Ausführung (Bittner et al. 1998) ist jedoch mit 10 µm ebenso zu hoch für eine exakte Verfolgung von kleinen beweglichen Mikroorganismen wie Bakterien. In der Literatur sind Anwendungen der In-Situ-Mikroskope bei Hefezellen (Suhr et al. 1998; Belini et al. 2013), mikroskopischen Partikeln (z.B. Gipskristalle) (Suhr 2004; Qu et al. 2006), Luftblasen (Junker et al. 2007) sowie tierischen Zellen (Joeris et al. 2002; Guez et al. 2004; Guez et al. 2010; Wiedemann et al. 2011) beschrieben. Die bisherigen Anwendungen mit In-Situ-Mikroskopen zeigen passiv strömende Partikel. Eine aktive Eigenbewegung wird nicht verfolgt. Aussagen zur Vitalität können somit lediglich aus der Morphologie, jedoch nicht aus der Beweglichkeit gezogen werden. 10 Online-Mikroskopie 2.2 Durchfluss-Mikroskope: Neben der In-Situ-Mikroskopie gibt es Verfahren der Online-Mikroskopie mit automatischer Probenentnahme (Ren et al. 1994) oder im Bypass-Betrieb. Die Durchflussküvetten stellen für diese Anwendungen das Kernstück dar und haben Schichtdicken zwischen 20 und 400 µm (Berg und Block 1984; Ren et al. 1994; Gast et al. 2006). Durchflussküvetten für Mikroskope finden Anwendung bei Lebendpräparaten von Geweben und Biofilmen. Hierbei ist das Gewebe oder der Biofilm an der Durchflusskammer fixiert und wird ständig von Nährmedium überspült. Das Nährmedium wird mit einer Peristaltikpumpe durch die Durchflusskammer gefördert. Die Online-Mikroskopie der Gewebeprobe oder des Biofilms ist durch dieses System über einen längeren Zeitraum möglich. Systeme mit automatisierter Probenentnahme bieten häufig zusätzlich die Möglichkeit zur automatisierten Probenvorbereitung, z.B. zum Verdünnen oder Anfärben der Probe (Ren et al. 1994; Zalewski und Buchholz 1996; Akin et al. 2011). Die Kernkomponenten sind schematisch in Abbildung 6 dargestellt. Abbildung 6: Schema eines Systems zur Online-Mikroskopie mit Durchflussküvette Vereinfachtes Schema eines typischen Systems zur Online-Mikroskopie mit einer Durchflussküvette. Die Kernkomponenten sind: automatische Probenentnahme, Probenvorbereitung, Durchflussküvette und Mikroskop. Zu einem Online-MikroskopieSystem gehört zudem die nachgeschaltete automatische Bildauswertung, die hier jedoch nicht dargestellt ist. In der Arbeit von Rehbock et al. (2010) wird die Entwicklung einer Durchflussküvette für ein In-Situ-Mikroskop erläutert. Durch diese Küvette ist das beschriebene In-Situ-Mikroskop nicht nur für den In-Situ-Betrieb an einem 25 mm Stutzen, sondern auch für den BypassBetrieb mit einer Durchflussküvette geeignet. Viele Durchflussküvetten sind jedoch zum Einsatz auf einem herkömmlichen Mikroskop konzipiert (Berg und Block 1984; Ren et al. 1994; Meinhart et al. 1999; Gast et al. 2006). Durchflusssysteme für Lebendpräparate können z.B. von IBI SCIENTIFIC (Peosta, USA) und Bioptechs Inc. (Butler, USA) bezogen werden. Durchflussküvetten zur Untersuchung von 11 Online-Mikroskopie Suspensionen werden z.B. von GeSiM Gesellschaft für Silizium-Mikrosysteme mbH (Großerkmannsdorf, Deutschland) vertrieben (Gast et al. 2006). Weiterhin gibt es einige kommerziell erhältliche Partikelanalysatoren, die Komplettsysteme aus Durchflussküvette, Probenentnahmesystem, Bildaufnahme und Bildanalyse anbieten z.B. Particle Metrix GmbH (Meerbusch, Deutschland), Sympatec GmbH (Clausthal-Zellerfeld, Deutschland) und Occhio SA (Angleur, Belgien). 2.3 Bildverarbeitung Sowohl die In-Situ-Mikroskope als auch die Durchfluss-Mikroskope werden erst durch eine automatische Bildverarbeitung zu einem Online-Mikroskop. Die Bildverarbeitung kann hierbei entweder im Computer, in speziellen Parallelrechnern (Transputer) oder direkt in intelligenten Kameras ausgeführt werden. Durch die Leistungsfähigkeit moderner Computer wird die Bildverarbeitung heute häufig direkt im Computer ausgeführt. Sowohl Programme als auch Hardware zur Entwicklung von Bildverarbeitungsalgorithmen sind in vielfacher Ausführung am Markt erhältlich. Die Bildverarbeitungslösungen im Bereich der Partikelanalyse laufen meist nach einem ähnlichen Schema ab. Zunächst werden die Bilder in einer Vorverarbeitung optimiert. Hierbei werden z.B. Bildrauschen und Hintergrund eliminiert. Anschließend werden die interessierenden Details hervorgehoben, bevor eine Binarisierung der Bilder durchgeführt wird. Die Merkmalsextraktion wie z.B. Partikelgröße, Homogenität und Form findet anschließend aus dem Binärbild statt. Zalewski und Buchholz (1996) beschreiben ein System, in dem die Zellkonzentration und die Morphologie von Hefe ermittelt werden. Die erfassten Klassen im Sinne der Morphologie beinhalten: Einzelzellen, Doppelzellen, Tetrade sowie Zellhaufen jeweils mit und ohne Einschlüsse (z.B. Vakuolen). Es handelt sich um eine Anwendung eines DurchflussMikroskops. Die Bildverarbeitung wird auf einem Transputer durchgeführt und das Ergebnis wird an einen Computer übermittelt. Als Vorverarbeitung kommt eine Kantenschärfung zum Einsatz und über einen Filter werden kleine Lücken in den Kanten geschlossen. Anschließend werden geschlossene Objekte aufgefüllt und eine Binarisierung des Bildes wird vorgenommen. Aus dem entstandenen Binärbild werden die Merkmale extrahiert. Auch bei anderen Bildverarbeitungsanwendungen mit Hefen kommen zur Vorverarbeitung häufig Algorithmen zur Kantenschärfung zum Einsatz (Rehbock et al. 2010; Akin et al. 2011), z.B. Canny-Algorithmus (Canny 1986). Es sind eine ganze Reihe weiterer Online-Mikroskopieanwendungen für Hefen veröffentlicht. Die Ermittlung der Zelldichte steht hierbei oftmals im Vordergrund (Suhr et al. 1995; Akin et al. 2011). Untersucht wird aber auch die Größenverteilung (Camisard et al. 2002; Belini et al. 2013) und die Morphologie (Zalewski und Buchholz 1996; Bittner et al. 1998; Rehbock et al. 2010). Auf die Vitalität der Hefen kann man aus der Größenverteilung, vitale Zellen sind größer (Belini et al. 2013), und aus der Morphologie (Pons et al. 1993) schließen. 12 Online-Mikroskopie Die Online-Bestimmung der Zellkonzentration während der Kultivierung von Säugerzellen wird von Guez et al. (2004) beschrieben. Es wird hier ein In-Situ-Mikroskop mit Durchlichtbeleuchtung eingesetzt. Die Bildverarbeitung wird direkt auf dem Computer ausgeführt. Auch in dieser Anwendung werden zunächst die Kanten hervorgehoben, bevor eine Binarisierung durchgeführt wird. Nach der Binarisierung sucht ein spezieller Algorithmus (Hough-Transformation nach Hough (1962)) nach runden Objekten, die anschließend gezählt und vermessen werden. In weiterführenden Arbeiten wird neben der Zelldichte auch die Messung der Vitalität der Zellen über Online-Mikroskopie diskutiert (Guez et al. 2010; Wiedemann et al. 2011). Eine vollautomatische Bildanalyse zur Bestimmung der Vitalität von Säugerzellen wurde bereits von Tucker et al. (1994) präsentiert. Die Probenentnahme und Vorbereitung wurde hier jedoch noch manuell vorgenommen. Junker et al. (2007) beschreiben einen Bildverarbeitungsalgorithmus zur Online-Bestimmung einer Größenverteilung von Luftblasen. Zum Einsatz kommt hier ein In-Situ-Mikroskop von EnviroCam Inc. (Colmar, USA). Häufig wird zur Elimination des Hintergrundes vor der Bestimmung der Objekte eine Aufnahme ohne Partikel durchgeführt und diese vom aufgenommenen Βild subtrahiert. Die Beleuchtungsbedingungen (Fremdlicht), der Verschmutzungsgrad und der Fokus (leichte Vibrationen) können während der Messdauer variieren. Daher ist es bei längeren Messungen schwer, ein durchgehend gutes Ergebnis mit der Subtraktion eines Hintergrundbildes zu erhalten. Aus diesem Grund wird hier das Hintergrundbild aus dem Durchschnitt von 500 Aufnahmen immer wieder neu generiert. Dies ermöglicht eine stabile Subtraktion vom aktuellen Durchschnittshintergrund. Der weitere Ablauf der Bildverarbeitung ist dem der bereits beschriebenen Anwendungen ähnlich: nach der Kantenschärfung findet die Binarisierung mit anschließender Merkmalsextraktion statt. 13 Bewegung von Bakterien 3 Bewegung von Bakterien Neben der Morphologie und dem Zustand der Zellwand (Anfärben) ist auch durch die Eigenbewegung der Zellen eine Aussage zur Vitalität möglich. Eine Vielzahl von beweglichen Bakterienstämmen wie z.B. Escherichia coli und Bacillus subtilis haben eine hohe Relevanz als Produktionsstämme in Industrie und Forschung, wodurch eine Aussage zur Vitalität von eminenter Bedeutung ist. Durch Vermehrung ist grundsätzlich allen Bakterien eine verhältnismäßig langsame Fortbewegung möglich. Es werden sechs Fortbewegungsarten von Bakterien unterschieden: Verbreiten (sliding, spreading), Stechen (darting), Gleiten (gliding), Kriechen (twitching), Schwimmen (swimming) und Schwärmen (swarming) (Henrichsen 1972; Mattick 2002; Harshey 2003) Verbreiten (sliding, spreading) ist keine aktive Bewegung der einzelnen Zelle, sondern ein Schieben der Koloniefront. Die Ausbreitungsgeschwindigkeiten liegen zwischen 0,03 und 6 µm s-1 (Harshey 2003). Beim Stechen (darting) breitet sich die Kolonie ähnlich wie beim Verbreiten aus, es gibt jedoch keine geschlossene Kolonie, sondern immer wieder sind zufällig verteilte freie Flächen zwischen der Ausbreitungsfläche der Kolonie (Henrichsen 1972). Unter Gleiten (gliding) versteht man eine gleichmäßige axiale Bewegung der Zelle. Typische Geschwindigkeiten bewegen sich zwischen 0,025 und 0,1 µm s-1, Spitzenwerte bis zu 10 µm s-1 erreichen Cyanobakterien (Blaualgen) (Harshey 2003). Beim Kriechen (twitching) handelt es sich um ein ruckartiges Gleiten. Die Bakterien bewegen sich hier mit Hilfe von Fimbrien: es sind dünne Fädchen, die etwa einen Durchmesser von 6 nm aufweisen und bis zu 4 µm lang sein können (Harshey 2003). Die Geschwindigkeit beträgt zwischen 0,06 und 0,3 µm s-1 (Mattick 2002). Schwimmen (swimming) und Schwärmen (schwarming) sind nur bei begeißelten Bakterien zu beobachten. Schwärmen ist eine kollektive Gruppenbewegung. Typische Ausbreitungsgeschwindigkeiten liegen bei 2 bis 10 µm s-1 (Harshey 2003). Schwimmen und Schwärmen existieren nebeneinander. Online kann die Vitalität der Bakterienkultur nur aus der schnellen und aktiven Bewegungsform des Schwimmens geschlossen werden. Betrachtet man die Geschwindigkeit des individuellen Bakteriums, ist Schwimmen die schnellste Fortbewegungsform. Geschwindigkeiten über 40 µm s-1 sind hier leicht möglich (Harshey 2003). Die Fortbewegung beim Schwimmen der Bakterien wird durch Geißeln (Flagellen) ermöglicht (Berg und Anderson 1973). Die Menge und Anordnung der Geißeln unterscheidet sich zum Teil erheblich (Schmitt 1972). Die Geißel stellt im Prinzip einen Mikromotor dar, wobei sie selbst den Rotor, die Zellwand samt Membran den Stator bildet (Silverman und Simon 1974; Berg 2003) Das Geißelfilament ist etwa 5 – 10 µm lang und sein Durchmesser beträgt 18 – 20 nm. Die Filamente sind flexibel und bilden die Form einer Helix. Für die Bakterien stellen sie somit einen schraubenförmigen Propeller dar (Jarosch 1967; Schmitt 1997; Macnab 2003). Die Frequenz eines Geißelbündels von Streptococcus wurde von Lowe et al. (1987) gemessen. Hierbei wurden sowohl eine Temperatur- als auch eine Viskositätsabhängigkeit der Drehzahl 14 Bewegung von Bakterien festgestellt. Die Frequenz des Bündels beträgt bei Streptococcus etwa 3.000 U min-1 bei 10 °C und steigt linear auf etwa 12.000 U min-1 bei 42 °C an. Beim Bewegungsablauf einiger peritrich begeißelten Bakterien wie Salmonella, Escherichia und Bacillus wurde ein ähnliches Bewegungsmuster erkannt. Phasen des axialen GeradeausSchwimmens werden immer wieder unterbrochen durch Taumeln (tumbles) der Zellen. Durch dieses Taumeln kann eine Richtungsänderung vorgenommen werden (Berg und Brown 1972; Macnab und Ornston 1977). In Abbildung 7 ist ein typischer Bewegungsablauf dargestellt. Abbildung 7: Wechsel zwischen Bewegungsphasen von peritrich begeißelten Bakterien Die folgenden Bewegungsphasen wurden bei Salmonella beobachtet. Das Bakterium schwimmt mithilfe eines Geißelbündels zunächst geradeaus (a). Durch Umkehren der Drehrichtung der Geißeln kann das Bündel aufgelöst werden und die Bakterie taumelt (b, c). Nach dem Richtungswechsel der Zelle orientieren sich die Geißeln neu (d) und schließlich stellt sich wieder ein Bündel ein (e) (Macnab und Ornston 1977) (modifiziert). In den Bewegungsabläufen von Bakterien sind sich regelmäßig wiederholende Muster zu erkennen. Bei 2D-Wegverfolgungs-Experimenten von Salmonella konnten von Macnab und Ornston (1977) typische Abstände zwischen Taumeln und Schwimmen ermittelt werden. Weiterhin wurden signifikante Unterschiede in den Bewegungsmustern verschiedener Stämme festgestellt und der Einfluss der Viskosität auf die Geschwindigkeit und die Bewegungsmuster untersucht. In hochviskosen Medien kommt das Taumeln erheblich seltener vor. Charakteristische Schwimmmuster konnten weiterhin bei vielen anderen beweglichen Bakterienstämmen beobachtet werden, z.B. Vibrio alginolyticus (Kudo et al. 2005), B. subtilis (Mendelson et al. 1999) und E. coli (Ping 2012). In 3D-Wegverfolgungs-Experimenten von Pseudomonas aeruginosa fanden Vater et al. (2014) wiederholende Bewegungsabläufe im dreidimensionalen Raum. Eine Klassifikation in fünf charakteristische Schwimmmuster war damit möglich. In 3D-Wegverfolgungen von E. coli unterschieden Wu et al. (2006) die charakteristischen Schwimmmuster von drei Stämmen 15 Bewegung von Bakterien (RP437, RP9535, RP1616). Ebenso wurde der Einfluss von Oberflächen auf die 3DSchwimmmuster von E. coli gezeigt (Frymier et al. 1995; Vigeant und Ford 1997). Bakterien mit der Fähigkeit zu schwimmen haben die Möglichkeit, sich schneller auszubreiten und neue Nährstoffregionen zu erschließen als unbewegliche Bakterien. Hierbei können sie sich nicht nur zufällig durch das Medium bewegen, sondern verfügen unter anderem über die Fähigkeit, Nährstoffgradienten bzw. Nährstoffquellen zu folgen (Berg und Brown 1972; Miller und Koshland 1977; Barbara und Mitchell 2003). Der Nachteil des hohen Energieverbrauchs durch die Bewegung (Grossart et al. 2001) wird in vielen Umgebungen durch den Vorteil der Erschließung neuer Nährstoffquellen ausgeglichen. In Tabelle 2 ist eine Übersicht über typische Schwimmgeschwindigkeiten beweglicher Bakterien zu sehen. Die Geschwindigkeiten liegen meist zwischen 20 µm s-1 und 80 µm s-1. Tabelle 2: Bekannte Schwimmgeschwindigkeiten von Bakterien Angegeben sind eine Reihe bekannter Schwimmgeschwindigkeiten von beweglichen Bakterien in flüssigen Medien. Es werden Quellen, untersuchte Bakterienart und der ermittelte Geschwindigkeitsbereich aufgezeigt. Trotz großer Abweichungen in den Versuchsdurchführungen (z.B. Medium, Temperaturbereich) lässt sich in den Ergebnissen gut die Größenordnung der Schwimmgeschwindigkeiten von Bakterien erkennen. Quelle Bakterienart Geschwindigkeit [µm s-1] (Grossart et al. 2001) Meerwasserbakterien 10 bis 500 , typisch 100 (Schmitt 1972) Vibrio comma Bacillus, Proteus und Enterobakterien Bacillus brevis Escherichia coli und Salmonella typhimurium Bacillus subtilis bis 200 (Schmitt 1972) (Shoesmith 1960) (Harshey 2003) (Mendelson et al. 1999) typisch 50 6 bis 32 bis 40 76 bis 116 (Lowe et al. 1987). Streptococcus (Magariyama et al. 1994; Vibrio alginolyticus Magariyama et al. 2005) Shewanella putrefaciens (Barbara und Mitchell und Pseudoalteromonas 2003) haloplanktis (Shigematsu et al. 1995) Vibrio cholerae 10 bis 30 (Shigematsu et al. 1995) Pseudomonas aeruginosa 51 ± 8 (Harwood et al. 1989) Pseudomonas putida bis 75, typisch 44 (Karim et al. 1998) Campylobacter jejuni 29 bis 53 (Karim et al. 1998) Heliobacter pylori 12 bis 29 (Karim et al. 1998) Escherichia coli 8 bis 18 50 bis 160 bis 440, typisch 100 75 ± 9 16 Bewegung von Bakterien Chemotaxis, speziell Aerotaxis (Folgen von Sauerstoffgradienten) von Bakterien wurde erstmals 1881 beschrieben und in den vergangenen Jahrzehnten ausgiebig untersucht. Neben der Chemotaxis wies Maeda et al. (1976) bei Bakterien auch Thermotaxis (Folgen von Temperaturgradienten) nach. Phototaxis (Folgen von Lichtgradienten) konnte Glagolev (1980) nachweisen. Viele Bakterien können sich somit durch aktive Bewegung in ein Gebiet mit besseren Umweltbedingungen bewegen bzw. von Gebieten mit schlechteren Bedingungen wegbewegen (Adler 1966; Berg 1975). Erst durch diese Fähigkeit bringt die Beweglichkeit von Bakterien einen evolutionären Vorteil, da in einem homogenen Medium die Energieaufnahme von Bakterien durch eine aktive Bewegung nicht signifikant verbessert wird (Berg und Purcell 1977). Die Fähigkeit, Reizen wie Lockmitteln zu folgen bzw. vor Schreckmitteln (Repellent) zurückzuweichen, zeigt die Wahrnehmungsfähigkeit von Bakterien. Die Rezeptoren der Bakterien und ihr Einfluss auf den Geißelmotor ist Schwerpunkt vieler Untersuchungen (Macnab und Koshland 1972; Berg und Purcell 1977; Miller und Koshland 1977; Glagolev 1980; Zhang und Phillips 2003). Durch Chemotaxis, Thermotaxis und Phototaxis ist es den beweglichen Bakterien möglich, die Umgebung mit der für sie besten Energieversorgung zu finden und sich dort anzusammeln (Adler 1966; Berg und Purcell 1977). In homogen gemischten Medien zeigen die Bakterien kein gerichtetes Bewegungsmuster. Geradeaus-Schwimmen wird immer wieder von einem Wechsel der Richtung unterbrochen (Macnab und Koshland 1972). Die Bewegung von Bakterien wurde in zahlreichen Studien untersucht. Häufig werden die Bewegungen der Bakterien in speziellen Küvetten unter dem Mikroskop zweidimensional verfolgt. Barbara und Mitchell (2003) veröffentlichten in einer Studie, wie Shewanella putrefaciens und Pseudoalteromonas haloplanktis eine bewegliche Alge (Pavlova lutheri) verfolgen. Ähnliche Systeme kamen zum Einsatz zur Bewegungs- und Geschwindigkeitsanalyse von Pseudomonas putida (Harwood et al. 1989), Vibrio cholerae und Pseudomonas aeruginosa (Shigematsu et al. 1995), Vibrio alginolyticus (Magariyama et al. 2005) und auch anderer Mikroorganismen wie z.B. von Protisten (Beveridge et al. 2010). Weiterhin wurden zahlreiche 3D-Wegverfolgungssysteme entwickelt. Ein Messsystem zur 3D-Verfolgung einer einzelnen Zelle wurde bereits von Berg (1971) veröffentlicht. Andere Systeme zur parallelen Verfolgung mehrere Zellen im dreidimensionalen Raum folgten z.B. von E. coli (Wu et al. 2006) und Pseudomonas aeruginosa (Vater et al. 2014). Jedoch ist keines der genannten Systeme geeignet, die Bewegungsgeschwindigkeit online auszuwerten, um diese als Vitalitätsparameter zur Prozessverfolgung einzusetzen. Ein solches System benötigt eine automatisierte Probenentnahme und Probenvorbereitung sowie eine daran angeschlossene Durchflussküvette. Weiterhin ist eine automatische Bildaufnahme und Bewegungsverfolgung der Zellen notwendig. Mit Messintervallen von etwa 15 min könnte die Bewegung von Bakterien so als Parameter der Vitalität während des Prozesses zum Monitoring herangezogen werden. 17 Einleitung II Bewegungsanalyse von Bacillus amyloliquefaciens 4 Einleitung Hersteller biotechnologischer Produkte sehen sich immer stärker unter Druck, Qualität und Sicherheit ihrer Produkte nachzuweisen. Zum einen fordern die Kunden häufiger ein ausführliches Qualitätsmanagement, zum anderen gilt es, die Verordnungen wie z.B. die der U.S. Food and Drug Administration (FDA) zu erfüllen. Aus diesem Grund besteht großes Interesse an neuartigen Überwachungsmöglichkeiten der Zellvitalität und Zelldichte. In den letzten Jahren kam es daher zu diversen Entwicklungen in diesem Bereich (Brognaux et al. 2013; Höpfner et al. 2010; Müller und Nebe-von-Caron 2010). Die großtechnische Kultivierung von Bacillus amyloliquefaciens FZB42 (ABiTEP GmbH, Berlin) ist von dieser Entwicklung ebenso betroffen wie viele andere Bioprozesse. Die Kunden der ABiTEP GmbH erwarten eine gleichbleibende Qualität der Produkte. Vitalität und Zelldichte der Bakterien im Verlauf der Fermentation sind hierfür von entscheidender Bedeutung. Entdeckt wurde B. amyloliquefaciens 1943 von Fukomoto (1943a; 1943b). Zunächst galt es jedoch meist als Unterart von B. subtilis. 1987 konnten Priest et al. (1987) das Bakterium unter der Bezeichnung B. amyloliquefaciens sp. nov. nom. rev. als eigenständige Spezies identifizieren. Der Name stammt von seiner starken α-Amylase-Produktion ab. Er setzt sich zusammen aus den Worten „amylo“ (Amylose / Stärke) und „liequefaciens“ (verflüssigend). Näher betrachtet wird hier B. amyloliquefaciens FZB42, das als Unterart B. amyloliquefaciens subsp. plantarum subsp. nov. klassifiziert ist (Borriss et al. 2011). Der Stamm wurde aus einer mit Pflanzenschädlingen infizierten Bodenprobe isoliert (Krebs et al. 1998). Die ABiTEP GmbH stellt die Sporen von B. amyloliquefaciens FZB42 großtechnisch her und verwendet diese in ihrem biologischen Bodenhilfsstoff RhizoVital® 42. Die für Pflanzen wachstumsfördernden Eigenschaften sowie die Unterdrückung von pflanzenschädigenden Organismen durch B. amyloliquefaciens wurden in mehreren Studien nachgewiesen (Krebs et al. 1998; Idriss et al. 2002; Idris et al. 2007; Chen et al. 2009). B. amyloliquefaciens FZB42 ist peritrich begeißelt und dadurch in der Lage, frei zu schwimmen. Da nur lebende Bakterien in der Lage sind zu schwimmen, ist die Analyse der Bewegungsfähigkeit in Hinblick auf die Vitalität und Aktivität von großem Interesse. Es sind verschiedene Bewegungsabläufe von Bakterien bekannt. Das Random-Walk-Model (Berg und Brown 1972), das vor allem bei peritrich begeißelten Bakterien vorherrscht sowie die Run-and-Revers-Strategy (Barbara und Mitchell 2003), die vor allem bei polar begeißelten Meerwasserbakterien nachgewiesen wurde. Das Random-Walk-Model für Bakterienbewegungen wurde von Berg und Brown (1972) am Beispiel von E. coli untersucht und beschrieben. Durch diese Strategie bewegen sich die Zellen langsam auf den Lockstoff zu und sammeln sich nach einiger Zeit um die Quelle an. Berg (1971) entwickelte hierfür ein automatisches Mikroskop zur 3D-Bewegungsverfolgung von Bakterien. Das Mikroskop hat drei bewegliche Achsen mit einer Reichweite von jeweils 18 Einleitung 1 mm. Zur Bewegungsverfolgung wird eine Bakterie in x,y,z-Achse im Fokus gehalten. Die Koordinaten der Achsen können elektronisch ausgelesen werden. Das Mikroskop eignet sich zur Verfolgung einer einzelnen Bakterie im 3D-Raum. Die Bewegungsverfolgung erfolgt automatisch. Eine automatische Probenentnahme sowie die Beobachtung mehrerer Bakterien sind jedoch nicht vorgesehen. Müssen die Bakterien beweglichen Nährstoffquellen folgen, erweist sich diese Strategie als zu langsam. Daher zeigen vor allem Meerwasserbakterien eine andere Strategie, um den Lockstoffen zu folgen. Die Run-and-Revers-Strategy wurde von Barbara und Mitchell (2003) untersucht. Hierzu wurde beobachtet, wie Shewanella putrefaciens und Pseudoalteromonas haloplanktis einer beweglichen Alge (Pavlova lutheri) folgen. Meerwasserbakterien bewegen sich um eine Größenordnung schneller (bis ca. 400 µm s-1) als z.B. E. coli (bis ca. 40 µm s-1), was auf die ständige Bewegung des Meeres zurückzuführen ist. Die Run-and-Revers-Strategy ermöglicht es den Bakterien, sich trotz der Unruhe auf See in Schwärmen in unmittelbarer Nähe (näher als 20 µm) zu Lockstoffen wie Luftblasen oder Nährstoffquellen aufzuhalten. In Abbildung 8 sind Beispieldiagramme zur Run-and-Revers-Strategy dargestellt. Es ist zu sehen, dass es den Bakterien gelingt, den Weg der Alge durch schnelle Richtungswechsel und hohe Geschwindigkeiten gut zu verfolgen. Abbildung 8: Beispiel für Run-and-Revers-Strategy Die Diagramme zeigen je die Wege von einer Bakterie Pseudoalteromonas haloplanktis, die einer beweglichen Alge Pavlova lutheri folgt. Die dargestellten Diagramme zeigen gute Beispiele für die Run-and-Revers-Strategy (aus Barbara und Mitchell 2003). Die Versuche von Barbara und Mitchell (2003) wurden in einer 100 µm tiefen MikroskopieKammer auf einem Labormikroskop mit 200facher Vergrößerung im Dunkelfeld durchgeführt. Die Aufnahmen für das 2D-Tracking erfolgten mit einer Video-Kamera und wurden mit 24 fps aufgezeichnet. Die Wegverfolgung und Geschwindigkeitsanalyse wurden manuell durchgeführt. Eine bekannte Fehlerquelle ist die langsame Aufnahmerate von 24 fps. Die Bakterien können bis zu 20 Richtungswechsel pro Sekunde durchführen, bei 24 fps ist die Abtastung zu gering und Teile der Bewegungen werden nicht erfasst. Geschwindigkeit und zurückgelegte Strecke werden daher tendenziell etwas zu langsam angegeben. Ein weiteres Problem stellt die 100 µm tiefe Mikroskopie-Kammer dar. Zum einen wird nur der mittlere Bereich vom Mikroskop scharf erfasst, zum anderen werden Bewegungen in z-Richtung vernachlässigt. 19 Einleitung Ein ähnliches System wurde zur Bewegungs- und Geschwindigkeitsanalyse von Pseudomonas putida in Abhängigkeit der Zugabe von Benzoesäure verwendet (Harwood et al. 1989). Auch Shigematsu et al. (1995) werten die Schwimmgeschwindigkeiten manuell aus, indem der Pfad der Bakterie mit einer Computermaus auf dem Bildschirm nachverfolgt wird. Die Schwimmgeschwindigkeiten von Vibrio cholerae und Pseudomonas aeruginosa wurden so ermittelt. Die Probenentnahme erfolgt ebenso manuell. Die Messzelle besteht aus einem Objektträger mit einem Deckglas. Der Rand um das Deckglas wurde mit Nagellack abgedichtet. Die Bewegung von Vibrio alginolyticus wurde von Magariyama et al. (2005) untersucht. Auch hier wurde die eigentliche Wegverfolgung manuell durchgeführt. Die Bewegung anderer Mikroorganismen wie z.B. von Protisten wurde mit ähnlichen Systemen untersucht (Beveridge et al. 2010). Durch die „quantitative defocusing method“ konnten Wu et al. (2005) das Problem der Ungenauigkeiten durch Bewegungen in z-Richtung lösen. Die Bakterien können in einer Küvette auf einem herkömmlichen Auflicht-Fluoreszenz-Mikroskop untersucht werden. Unter dem Fluoreszenz-Mikroskop bilden die Partikel außerhalb des Fokus einen Ring um die Partikel aus. Durch den Durchmesser des Rings und des Partikels lässt sich berechnen, wie weit der Partikel außerhalb des Fokus ist. Mit dieser Methode lässt sich ein Volumen von 418 x 335 x 75 µm abbilden. So konnten 3D-Tracking-Experimente mit E. coli durchgeführt werden. Eine Behandlung zur Steigerung der Fluoreszenz ist jedoch notwendig. Die Probenentnahme, Probenvorbereitung und Wegverfolgung erfolgen bei diesem System manuell. Das System wurde bereits zur Bewegungsanalyse verschiedener E. coli Stämme eingesetzt (Wu et al. 2006). Vater et al. (2014) haben in einem 3D-Tracking-Experiment die Bewegung der Bakterien Pseudomonas aeruginosa untersucht. In einem Raum von 480 x 480 x 830 µm wurden die Bewegungen der Bakterien mit Hilfe eines holographischen Mikroskops aufgenommen. Durch die holographische Aufnahme ist vor allem in z-Richtung ein wesentlich größerer Raum abbildbar. Die Aufnahmen erfolgten über 10 min mit einer Abtastrate von 5 fps. Damit sind Bewegungen mit mehr als zwei Richtungswechseln pro Sekunde kaum zu verfolgen. Es werden alle Bakterien im Sichtfeld erfasst, die Bewegungsverfolgung der einzelnen Wege wird jedoch manuell vorgenommen. Eine automatische Probenentnahme ist nicht vorgesehen. Häder und Vogel (1991) haben einen 2D-Tracking Algorithmus zur parallelen Wegverfolgung von mehreren Mikroorganismen entwickelt. Die Mikroorganismen wurden in einer 60 x 14 x 0,5 mm Küvette auf einem Video-Mikroskop kultiviert und beobachtet. Die Bildverarbeitung erfolgte nicht in Echtzeit, jedoch ist die Auswertung bereits vollautomatisch. Ein weiterer automatischer 2D-Tracking Algorithmus wurde von Kang Li et al. (2006) beschrieben. Dieser Bildverarbeitungsalgorithmus eignet sich zur Wegverfolgung mehrerer Zellen in einer Bildserie. Erprobt wurde der Algorithmus an humanen Osteosarkom Zellen. Die Aufnahmen wurden über 10 h mit einem Aufnahmeintervall von 4 min bzw. über 43,5 h mit einem Aufnahmeintervall von 15 min durchgeführt. Beobachtet wurden unter anderem Bewegung, Zellteilung und Sterben der Zellen. Eine Validierung mit manueller Wegverfolgung ergab eine Genauigkeit von 86,4% bis 91,2%. Der Algorithmus besteht aus 20 Einleitung vier Kernmodulen (Cell Detector, Cell Tracker, Cell Arbitrator, Motion Filter), diese sind in Abbildung 9 dargestellt. Abbildung 9: Kommunikation der Kernmodule eines 2D-Tracking Systems Der Cell Detector fügt erkannten Zellen aus den Eingangsbildern eine Beschriftung hinzu. Der Cell Tracker verfolgt die Zellen zwischen den einzelnen Bildern und der Motion Filter unterstützt den Cell Tracker und Cell Arbitrator mit Bewegungsvorhersagen. Der Cell Arbitrator übernimmt die Nachbearbeitung der Zellen. Z.B. werden tote Zellen aussortiert, Zellen, die sich teilen, neu beschriftet und Zellen, die aus dem Sichtbereich verschwinden, ausgetragen (nach Kang Li et al. 2006). Die Eingangsbilder werden an die Module Cell Detector und Cell Tracker übergeben. Die einzelnen erkannten Zellen werden durch den Cell Detector beschriftet. Der Cell Tracker verfolgt die Zellen zwischen den einzelnen Bildern. Die Daten zu den einzelnen beschrifteten Zellen werden zwischen dem Cell Tracker und dem Cell Arbitrator ausgetauscht. Der Cell Arbitrator übernimmt eine Nachbearbeitung der Zuordnungen. Die Nachbearbeitung beinhaltet unter anderem das Zusammenführen unterbrochener Wegverfolgungen, das Aussortieren verschwundener Zellen und die Neubeschriftung nach Zellteilung. Der Motion Filter unterstützt den Cell Tracker und den Cell Arbitrator mit Bewegungsvorhersagen und Bewegungskorrekturen. Das System wurde zur Beobachtung der Zellteilung und der passiven Bewegung von Zellen über mehrere Stunden eingesetzt. Der erfolgreiche Einsatz des Algorithmus für schnell bewegliche Zellen, die mehrere Richtungswechsel vollziehen, wäre zu testen. Karim et al. (1998) untersuchten die Bewegungsgeschwindigkeit und die Form der Bewegung von verschiedenen Bakterien (Helicobacter pylori, Campylobacter jejuni und E. coli). Die Bildserien wurden mit einem Video-Mikroskop aufgenommen. Die Proben befanden sich hierfür in einer Mikroskopie-Kammer mit einer Schichtdicke von 100 µm. Das Programm Hobson BacTracker filtert die bewegten Teile aus den Bildserien und gibt die Koordinaten zu jedem beweglichen Objekt heraus. Diese Koordinaten werden in Echtzeit erfasst. Die Probenentnahme und Probenvorbereitung erfolgen manuell. Das System ist in der Lage, die Bewegungsform und Geschwindigkeiten vieler Bakterien in zwei Dimensionen zu erfassen. 21 Einleitung Die verhältnismäßig hohe Schichtdicke kann bei Bewegungen der Bakterien in z-Richtung zu Fehlern führen. Zusammenfassend lässt sich sagen, dass zur vollautomatischen Messung der aktiven Bewegungen und Geschwindigkeiten von Bakterien bislang kein Online-Mikroskopie-System zur Bioprozesskontrolle publiziert wurde. Den beschriebenen Systemen fehlt mindestens eine der Komponenten, die für Online-Mikroskopie-Systeme notwendig sind, nämlich die vollautomatische Probenentnahme, Probenvorbereitung, Bildaufnahme oder die Bildverarbeitung. 22 Zielsetzung 5 Zielsetzung Es soll ermittelt werden, ob mittels der Bewegungsgeschwindigkeit des Beispielorganismus B. amyloliquefaciens FZB42 eine Aussage über dessen Vitalität getroffen werden kann. Hierfür wird ein Messsystem zur automatisierten Messung der Bewegungsgeschwindigkeit mittels Online-Mikroskopie entwickelt. Die Probenentnahme, Probenvorbereitung und Messung sollen weitgehend automatisch erfolgen. Während des gesamten Prozesses von Probenentnahme bis zur Messung dürfen die Bakterien keinem erhöhten Zellstress, vor allem keinen erhöhten Scherkräften, ausgesetzt werden. Durch Scherung können begeißelte Mikroorganismen ihre Bewegungsfähigkeit verlieren, schon lange bevor eine Beeinträchtigung ihrer Fähigkeit zur Zellteilung eintritt (DePamphilis und Adler 1971; Rosu und Hughes 2006). Die Geißeln sind zwar in der Lage, sich zu regenerieren, dies nimmt zur vollständigen Regeneration jedoch einen Zeitraum von über 60 min in Anspruch und ist damit länger als die angedachte Messzeit (Stocker und Campbell 1959). Es sind Messzyklen (Probenentnahme bis Ergebnis) von etwa 15 min geplant. Das System soll möglichst kompatibel mit verbreiteter Laborausstattung in biotechnologischen Laboren wie Bioreaktoren, Labormikroskopen, Schlauchpumpen usw. betrieben werden. So werden geringe Etablierungskosten ermöglicht. Die Reinigung des Messsystems muss möglichst schnell und unkompliziert erfolgen, ebenso wie die Sterilisierung (z.B. autoklavierbar). Die erhaltenen Daten sollen zum einen als Rohdaten (Bilder) vorliegen und zum anderen auch automatisch ausgewertet werden. Durch die vorliegenden Rohdaten wird das System transparent und überprüfbar. Zudem können die Bilder später auch per Hand auf andere Parameter untersucht werden, sollte es z.B. zu unerwarteten Prozessverläufen kommen. Manuelle Auswertungen von Bilddaten einer ganzen Fermentation dauern jedoch viele Stunden bis zu wenigen Tagen und sind somit nicht durchgängig durchführbar. Zudem stehen die Daten für mögliche Prozesseingriffe nicht zur Verfügung. Ziel für die automatische Bildauswertung sind daher Berechnungszeiten, die kleiner sind als die Dauer eines Messzyklus. An mehreren Beispielfermentationen mit B. amyloliquefaciens FZB42 soll die Funktionsfähigkeit des Messsystems gezeigt werden. Die Prozessführung für die Beispielfermentationen ist auf die Gegebenheiten im Labor anzupassen, welche wiederum so nahe wie möglich an den realen Produktionsbedingungen bei der ABiTEP GmbH liegen sollen. Die in den Beispielfermentationen mit dem Bildanalyse-Messsystem gemessenen Parameter werden anschließend beurteilt und validiert. Hierfür ist es notwendig, weitere Prozessparameter zu messen, um diese als Grundlage zur Beurteilung einsetzen zu können. Auswahl und Implementierung der zusätzlichen Messtechnik ist ebenso Ziel dieser Arbeit. Die Möglichkeit, das automatische Bildanalyse-Messsystem später zur Sicherung der Prozessqualität und somit zum Nachweis der Produktqualität einzusetzen, soll untersucht werden. 23 Material und Methoden 6 6.1 Material und Methoden Verwendeter Stamm und verwendete Medien Als Beispielorganismus findet Bacillus amyloliquefaciens FZB42 (ABiTEP GmbH, Berlin) mit den Chargennummern 20130419 42 2.02 (Fermentation 1 – 4) und 20140310 42 2.02 (Fermentation 5 – 7) Anwendung. Der Stamm FZB42 wird von dem Leibniz Institut DSMZDeutsche Sammlung von Mikroorganismen und Zellkulturen GmbH unter der DSM Nr.: 23117 geführt. Die Bakterien liegen als Sporen in 4 × 1011 cfu g-1 vor. B. amyloliquefaciens subsp. plantarum subsp. nov. sind stäbchenförmige bewegliche Zellen. Das Bakterium ist grampositiv, 0,6 bis 0,9 µm breit und 1,8 – 4 µm lang. Es ist in der Lage, zylindrische Sporen zentral oder parazentral auszubilden. Die Kultivierung erfolgt streng aerob. Optimale Wachstumsbedingungen liegen zwischen 30 und 40 °C, unter 15 °C und über 50 °C findet kein Wachstum statt. Es verfügt über die Fähigkeit, lösliche Cellulose zu metabolisieren und Stärke durch verzuckernde Amylase zu verarbeiten (Borriss et al. 2011). Die Fermentationen 1 – 3 und 7 wurden mit dem Produktionsmedium der ABiTEP GmbH durchgeführt. Das Nährmedium wurde von der ABiTEP GmbH in Pulverform zur Verfügung gestellt. In diesem Medium wird der Stamm auch für die großtechnische Produktion kultiviert. Es handelt sich um ein komplexes Medium auf Stärkebasis und ist stark trüb (partikelbehaftet). Weitere Einzelheiten zur Zusammensetzung des Produktionsmediums dürfen an dieser Stelle nicht veröffentlicht werden. Die Fermentationsbrühe wurde mit 9,5 g l-1 des Produktionsmediums angesetzt, das entspricht einem Viertel der Produktionskonzentration. Die Konzentration wurde auf ein Viertel reduziert, um die Fermentationsdauer im Laborbetrieb auf unter 10 h zu verkürzen. Die Fermentationsbrühe wurde vor Versuchsbeginn mit fünfprozentiger Kalilauge (KOH) auf pH 7,2 eingestellt und im Bioreaktor 30 min autoklaviert. Vor dem Autoklavieren musste der Fermentationsbrühe 1 ml Aspumit AP (Thor GmbH, Speyer) als Antischaummittel zugegeben werden. Dieses Medium wird im Weiteren als ABiTEP-Medium-¼ bezeichnet. Die Fermentationen 4 – 6 wurden mit dem Standard-Nährmedium I (Charge: 503207757, Carl Roth GmbH + Co. KG) durchgeführt. Die Fermentationsbrühe wurde mit 6,25 g l-1 angesetzt. Dies entspricht einem Viertel der vom Hersteller empfohlenen Konzentration. Das Medium wird im Fermenter 15 min autoklaviert. Wie beim für die Fermentationen 1 – 3 und 7 verwendeten Medium wurde die Konzentration reduziert, um die Fermentationszeit auf unter 10 h zu verkürzen. Analog zum ABiTEP-Medium-¼ wird im weiteren Verlauf der Arbeit die Bezeichnung Standard-Nährmedium-I-¼ genutzt. 24 Material und Methoden Dichte und Viskosität der Medien Für spätere Berechnungen wurde die Dichte der Medien ermittelt. Die Messung der Dichte erfolgte mit einem Pyknometer, das vor der Messung im Wasserbad temperiert wurde. Ebenso wie die Dichte wurde auch die Viskosität der beiden Medien ermittelt. Zur Messung wurde ein Kapillarviskosimeter Typ 530 10 / I (SI Analytics GmbH) verwendet. Das Viskosimeter wurde zur Temperierung im Wasserbad betrieben. Zur Bestimmung des Fließverhaltens der beiden Medien kam ein Scherrheometer des Typs Physica UDS 200 (Anton Paar, Österreich) zum Einsatz. Gemessen wurde bei 35 °C mit einem Doppel-Spalt-Zylinder-Messsystem Z1 DIN (double gap). Die Viskosität wurde in Abhängigkeit der Scherrate gemessen. Zur Kalibrierung und Validierung der Messung wurde Wasser bei 35 °C verwendet. 6.2 Bioreaktor Beim verwendeten Bioreaktor handelt es sich um einen 14 l Fermenter (Chemap AG, Typ: 92703). Der Original-Schaltschrank wurde vollständig durch einen neuen ersetzt, welcher die Messdatenerfassung sowie Ansteuerung durch ein eigens für die Anwendung geschriebenes LabVIEW-Programm (National Instruments LabVIEW 2013) ermöglicht. Ein vereinfachtes Fließbild des Bioreaktors ist in Abbildung 10 dargestellt. Die Zuluft (Strom 1) zum Fermenter kann über den Massendurchflussregler FIRC 1.01 (Brooks, Mass-Flow-Controller 5850E) im Bereich von 0 – 2500 mln min-1 geregelt werden. Nach dem Massendurchflussregler befindet sich ein Sterilluftfilter (Midisart 2000, 0,2 µm PTFE, sartorius stedim), um mögliche Kontaminationen aus dem Zuluftsystem zu vermeiden. Dieser Filter ist im vereinfachten Fließbild nicht zu sehen. Der Fermenter B1.1 ist für 3 bar Überdruck und eine Temperatur von 130 °C ausgelegt und kann somit autoklaviert werden. Die Rührerdrehzahl SIRC 1.02 wird erfasst und ist in einem Bereich von 0 – 1200 rpm regelbar. Der Gelöstsauerstoff im Bioreaktor wird durch einen Gelöstsauerstoff-Sensor QIRC 1.03 (InPro 6820/12/320 Mettler Toledo) erfasst und ist über die Zuluftmenge und Rührerdrehzahl regelbar. 25 Material und Methoden Abbildung 10: Vereinfachtes Fließbild des Bioreaktors Der Fermenter B1.1 wird durch einen Doppelmantel temperiert. Temperaturen bis 130 °C sowie ein Überdruck von bis zu 3 bar sind im Behälter zugelassen. Der Fermenter ist somit voll autoklavierbar. Neben der Temperatur (TIRC 1.05) werden der pH-Wert (QIR 1.04) sowie der pO2-Wert (QIRC 1.03) im Bioreaktor gemessen. Weiterhin werden die Rührerdrehzahl (SIRC 1.02) und der Luftvolumenstrom (FIRC 1.01) gemessen. Unter Strom 2 ist die Probenentnahme und Übergabe an die Probenvorbereitung vereinfacht dargestellt. Ebenso werden der pH-Wert QIR 1.04 (405-DPAS-SC-K82/325, Mettler Toledo) und die Temperatur TIRC 1.05 im Bioreaktor erfasst. Dem Abluftstrom (Strom 3) sind nach dem Abluftkühler ein Schaumabscheider und eine Gaswaschflasche zur Abluftbehandlung nachgeschaltet. Das Wasser im Doppelmantel (Strom 4 und 5) wird durch die Temperaturreglung TIC 3.01 temperiert, die Sollwertvorgabe erhält die Regelung durch die Temperaturregelung des Fermenters TIRC 1.05. Die Probe (Strom 2) wird über eine Schlauchpumpe im Bypass befördert und über ein Ventil an die Probenvorbereitung übergeben. Der Bypass und die Entnahme über das Ventil sind im vereinfachten Fließbild des Bioreaktors nicht dargestellt. Die Probenvorbereitung wird in Kapitel 6.4.3 näher erläutert. Die entwickelte Ansteuersoftware erlaubt die Einstellung und Beobachtung der wichtigsten Parameter für die Prozessführung und Probenvorbereitung. Alle Messdaten können in einem Diagramm angezeigt und als Datei (.csv) exportiert werden. Die Bedienoberfläche für den Bioreaktor ist in Abbildung 11 dargestellt. 26 Material und Methoden Abbildung 11: Software Bedienoberfläche des Bioreaktors Die Benutzeroberfläche erlaubt es, die Sollwerte für Temperatur und für den pO2-Wert festzulegen. Die Temperatur- und pO2-Regelungen lassen sich auch in den Handbetrieb schalten. Probenentnahme und –vorbereitung können ebenso von der Oberfläche aus gesteuert werden. Die wichtigsten Prozessparameter können hier überwacht werden. Die Temperatur im Bioreaktor kann über die Temperatur im Doppelmantel geregelt werden. Zur Temperaturregelung wird ein modifizierter PI-Regler eingesetzt. Liegt die Temperatur in einem Bereich von ± 1 K, ist der Regler aktiv, außerhalb dieses Bereichs wird entweder voll gekühlt oder voll geheizt. In jedem Fall wird die Temperaturdifferenz zwischen Doppelmantel und Bioreaktor auf 10 K gehalten. Durch die Begrenzung der Temperaturdifferenz und das Deaktivieren des Reglers außerhalb des Arbeitspunktes wird ein schnelles Ansprechverhalten bzw. ein geringes Überschwingen gewährleistet. Die Stellgröße des Reglers deckt einen Bereich von ± 100 % ab. Im positiven Bereich wird die Heizung mit einer Pulsweitenmodulation (PWM) angesteuert und im negativen die Kühlung. Als Regelparameter haben sich ein Proportionalwert (P-Wert) von 15 und eine Integrationszeit (Ti) von 10 min als optimal erwiesen. Da der Regler nur nahe um den Arbeitspunkt aktiv ist konnten die Parameter auf einen schwingungsarmen Verlauf optimiert werden. Bei größeren Sollwertänderungen sorgen der deaktivierte Regler und das vollständige Heizen bzw. Kühlen für ein schnelles Erreichen des Arbeitspunktes. Die pO2-Regelung wurde ebenso mit einem modifizierten PI-Regler realisiert. Der Regler hat einen Bereich von 0 – 100 % in der Stellgröße. Er wirkt sich auf die Rührerdrehzahl sowie die Zuluftmenge aus. Die Rührerdrehzahl wird je nach Stellgröße in einem Bereich von 250 – 27 Material und Methoden 1200 rpm und die Zuluftmenge mit 100 – 2500 mln min-1 geregelt. Der P-Wert des Reglers beträgt 1,5 und die Integrationszeit (Ti) 10 min. Die Rührerdrehzahl wird selbst bei einem pO2-Wert über dem Sollwert nicht auf 0 gefahren, da eine Mindestdrehzahl des Rührers für die homogene Durchmischung notwendig ist. Die Zuluft bleibt ebenso auf einem Mindestwert von 100, da der leichte Überdruck im System vor Kontamination schützt und ein Zurückziehen der Abluft verhindert. Über die Benutzeroberfläche kann weiterhin die gesamte Probenentnahme und Probenvorbereitung gesteuert werden. Die gesamte Benutzeroberfläche und Programmierung wurde speziell auf die Anforderungen zur Erprobung des Online-Mikroskops optimiert. 6.3 Versuchsdurchführung Im zuvor gereinigten und sterilisierten Βioreaktor wird das Nährmedium (siehe 6.1) mit 8 – 10 l VE-Wasser (vollentsalztes Wasser) angesetzt. Nach dem Autoklavieren und Zuschalten der Belüftung mit 100 mln min-1 erfolgt ein Abkühlen auf 80 °C. Die B. amyloliquefaciens FZB42 Sporen werden zu 150 mg l-1 bei 80 °C zugegeben. Die Temperatur wird nach der Zugabe 30 min auf 80 °C gehalten und anschließend auf 35 °C abgekühlt. Der Temperaturschock begünstigt die spätere Auskeimung der Sporen (vgl. Stewart und Halvorson 1953; Foster und Johnstone 1990). Bei Erreichen der 35 °C wird die pO2-Regelung mit einem Sollwert von 50% gestartet und die Versuchsaufzeichnung beginnt mit 0 min. Der Verfahrensablauf der Versuchsdurchführung ist in Abbildung 12 dargestellt. Während der Versuchsdurchführung werden regelmäßig (Messintervall: 15 – 30 min) Proben in die Probenvorbereitung gepumpt und ggf. verdünnt. Die optische Dichte wird bei einer Wellenlänge von 600 nm (OD600) bestimmt (siehe 6.4.3). Während der Versuchsdurchführung wird ein Probenentnahmeintervall von 15 min angestrebt. Die vorbereiteten Proben werden durch die Durchflussküvette gepumpt, um die Bildstrecken zur automatischen Bildauswertung aufzunehmen. Es wurden zur Geschwindigkeitsmessung zu jedem Probezeitpunkt 10 Bildstrecken mit je 30 Bildern aufgenommen. Dies entspricht jeweils einer Sekunde Aufnahmezeit. Zusätzlich wurde für die Fermentationen 4 – 7 zu jeder Probenentnahme eine Bildstrecke mit 150 Aufnahmen, dies entspricht fünf Sekunden, zur Analyse der Bewegungsmuster aufgenommen. 28 Material und Methoden Abbildung 12: Vereinfachtes Verfahrensfließbild der Versuchsdurchführungen Verfahrensablauf mit ABiTEP-Medium-¼ (links) und mit Standard-Nährmedium-I-¼ (rechts). Der Übergang der einzelnen Verfahrensschritte ist mit Pfeilen von oben nach unten gekennzeichnet. Zu- und Abgänge sind mit Pfeilen von rechts nach links bzw. von links nach rechts gekennzeichnet. Neben der diskontinuierlichen Messung des OD600 und der Bildaufnahme werden kontinuierlich der pH-Wert, die Temperatur, der pO2-Wert, die Rührerdrehzahl und die Begasungsrate gemessen (siehe 6.2). Während Fermentation 4 und 5 wurde zusätzlich nach Bedarf Antischaummittel zugegeben: Dow Corning Antifoam RD Emulsion Charge: 0007473410 (VWR International Ltd.). 6.4 Online-Mikroskopie mit automatisierter Serienbildaufnahme Unter Online-Mikroskopie wird hier die automatische Aufnahme, Darstellung und Auswertung von mikroskopischen Bildern während der Laufzeit des Prozesses verstanden. Die Daten stehen somit für die Prozessführung zur Verfügung. Das Kernstück der OnlineMikroskopie mit automatisierter Serienbildaufnahme stellt eine Durchflussküvette mit flexibler Schichtdicke dar. Die Durchflussküvette wird mit einer Probe aus dem Bioreaktor beschickt. Diese Probe wird durch Wasserzugabe auf eine definierte optische Dichte eingestellt. Die Schichtdicke der Durchflussküvette sollte für die Serienbildaufnahme nicht mehr als 5 µm betragen. Die Küvette kann auf dem Objekttisch eines Labormikroskops (z.B. Olympus CX31) befestigt werden. Die gesamte Probenentnahme mit Bildaufnahme kann voll- und halbautomatisch erfolgen. 29 Material und Methoden 6.4.1 Durchflussküvette mit flexibler Schichtdicke Die Durchflussküvette mit flexibler Schichtdicke wurde von Dipl.-Ing. (FH) Dominik Bopp, Hochschule Mannheim konstruiert. Im Zentrum der Durchflussküvette (siehe Abbildung 13) befindet sich eine Silikonmembran mit eingearbeitetem Polycarbonat-Objektträger (Durchmesser 16 mm, Stärke 2 mm). Oberhalb der Membran wird ein Deckglas (Durchmesser 20 mm, Stärke ca. 0,15 mm) zwischen zwei Silikondichtungen geklemmt. Wird die Probe durch die Küvette gepumpt, liegt die Membran an dem unteren Gehäuse an und eine Durchströmung mit neuer Probe ist möglich. Abbildung 13: Konstruktionszeichnung der Durchflussküvette mit flexibler Schichtdicke Als Vollschnitt (links) und in isometrischer Darstellung (rechts) (konstruiert von Dipl.-Ing. (FH) Dominik Bopp, Hochschule Mannheim). Die Küvette besteht aus fünfzehn Bauteilen: - dreiteiliger Edelstahlküvettenkörper (oben, mittig, unten), Silikonmembran mit eingearbeitetem Polycarbonat-Objektträger, Deckglas (20 mm, Stärke 0,15 mm), zwei Silikondichtringe für das Deckglas, zwei Edelstahlschlauchtüllen, zwei Silikondichtringe für die Schlauchtüllen, vier Edelstahlschrauben. Die Küvette kann zum Reinigen leicht zerlegt und wieder montiert werden. Zwischen fünf und zehn min sind für eine komplette Reinigung inkl. Zerlegen und Montieren nötig. Zum Zusammenbau wird die Silikonmembran auf das Küvettenkörperunterteil gelegt und anschließend der Mittelteil aufgelegt. Die beiden Schlauchtüllen werden zusammen mit den Dichtringen in die passenden Vertiefungen eingesetzt. Ebenso wird das Deckglas mit beiden Silikondichtringen in die vorgesehene Nut platziert. Anschließend wird das Oberteil des Küvettenkörpers aufgelegt und mit den vier Schrauben befestigt. Alle Teile sind geklemmt oder geschraubt und leicht austauschbar. Die verwendeten Materialien sind gegen die gängigen Reinigungs- und Desinfektionsmittel beständig und somit auch im Aufbau zu reinigen und sterilisieren. Weiterhin ist die Küvette autoklavierbar. Bei hoher Schichtdicke kann die Probe mit geringem Druck durch die Küvette gepumpt werden. Durch den verhältnismäßig großen Durchmesser der Zuleitung und des Proberaums sind die Strömungsgeschwindigkeit und somit die Scherkräfte gering. Es ist wichtig, dass die Bakterien möglichst ohne großen Zellstress in den Proberaum gefördert werden. Die 30 Material und Methoden Fähigkeit, sich selbstständig zu bewegen, geht den Bakterien bei großen Scherkräften verloren. Der Verlust der Bewegungsfähigkeit ist auf das Abscheren der Geißeln zurückzuführen. Diese gehen schon bei Scherkräften verloren, die für die Zellen selbst unproblematisch sind. Die Geißeln können sich zwar regenerieren, jedoch nimmt dies eine längere Zeit in Anspruch (DePamphilis und Adler 1971; Witman 1975; Grossart et al. 2001; Näther 2007). Das Abscheren der Geißeln muss für eine exakte Messung auf jeden Fall vermieden werden. In Abbildung 14 ist die Funktion der Durchflussküvette vereinfacht dargestellt. Abbildung 14: Schema der Durchflussküvette mit flexibler Schichtdicke I: p1 < p2, δI < 5 µm, bereit für die Bildaufnahme. II: p1 > p2, δII > 1,2 mm, bereit für die Probenentnahme. 1: Edelstahlgehäuse, 2: Deckglas, 3: Objektträger, 4: Membran, 5: Bildaufnahme, 6: Beleuchtung. Nachdem die Küvette mit Probe beschickt ist, kann durch Unterdruck die Membran an das Deckglas gezogen (p1 < p2) werden. Sobald die gewünschte Schichtdicke (δI < 5 µm) erreicht ist, kann die Serienbildaufnahme gestartet werden. Beim Durchfluten mit neuer Probe steigt der Druck in der Küvette über den Umgebungsdruck (p1 > p2) und die Schichtdicke wird vergrößert (δII > 1,2 mm). 31 Material und Methoden Um eine Beschädigung der Geißeln und somit die Bewegungsunfähigkeit der Bakterien auszuschließen, sollte eine Berechnung der Scherkräfte in der Küvette erfolgen. Zur Berechnung der Scherkräfte wird eine vereinfachte Geometrie herangezogen. Der Flüssigkeitsraum in der Küvette wird als Scheibe mit 36 mm Durchmesser und 1 mm Höhe angenommen. Nahe am Rand, mit 1 mm Abstand, befindet sich der Zu- und Ablauf mit je 2 mm Durchmesser. Der vereinfachte Strömungsraum der Küvette ist in Abbildung 15 zu sehen. Abbildung 15: Strömungsraum in der Küvette (vereinfachte Darstellung) Der Strömungsraum in der Küvette wird vereinfacht als durchströmte Scheibe mit 1 mm Höhe und 36 mm Durchmesser betrachtet. Zu- und Ablauf haben einen Durchmesser von 2 mm. Die Scherkräfte werden unter der Annahme von schlanken Kanälen berechnet. Ein Kanal gilt als schlank, wenn die durchströmte Strecke wesentlich länger ist als die Breite und/oder Höhe des Kanals (Herwig 2008). Weiterhin wird für die Berechnung eine laminare Strömung angenommen und eine ausreichende Einlauflänge zur Ausbildung der Strömung vorausgesetzt (Surek und Stempin 2007). Die Berechnung erfolgt für den Zu- bzw. Abfluss sowie für einen Spalt mit der Breite der Küvette kurz nach dem Einlass und an der breitesten Stelle der Küvette. Die herangezogenen Geometrien sind in Abbildung 16 verdeutlicht. Die Berechnungen der beiden Spalten erfolgt in vereinfachter Form. Die Randeffekte an den beiden schmalen Kanten der Spalte werden vernachlässigt. 32 Material und Methoden Abbildung 16: Geometrie der berechneten schlanken Kanäle Die Scherkräfte werden exemplarisch an drei schlanken Kanälen berechnet: einer Leitung mit 2 mm Innendurchmesser für Zu- und Abfluss, einem Spalt mit der Breite des Durchmessers der Küvette und einem Spalt mit der Breite der Küvette kurz nach dem Einlass. Die folgenden Gleichungen (6.1) bis (6.6) stammen aus Surek und Stempin (2007) und 𝑢 Herwig (2008). Das Geschwindigkeitsprofil 𝑢 , wobei 𝑢 die Geschwindigkeit an der Stelle ℎ 𝑚 bzw. 𝑟 und 𝑢𝑚 die querschnittsgemittelte Geschwindigkeit darstellen, ist für einen Spalt mit gegebener Höhe 𝐻 wie folgt definiert: 2 (6.1) 2 (6.2) 𝑢(ℎ) 3 ℎ = �1 − � � � 𝐻� 𝑢𝑚 2 2 Für ein Rohr mit gegebenem Durchmesser 𝐷 folgt: 𝑢(𝑟) 𝑟 = 2 �1 − � � � 𝐷� 𝑢𝑚 2 Der Geschwindigkeitsgradient 𝛾̇ ergibt sich aus der Geschwindigkeitsänderung (∆𝑢) senkrecht zur Bewegungsrichtung (∆𝑦) . 𝛾̇ = ∆𝑢 d𝑢 = ∆𝑦 d𝑦 (6.3) 33 Material und Methoden Die Schubspannung 𝜏 ergibt sich aus der Scherkraft 𝐹 pro Fläche 𝐴. Die dynamische Viskosität 𝜂 multipliziert mit dem Geschwindigkeitsgradienten ergibt ebenso die Schubspannung. 𝜏= 𝐹 = 𝜂 ∙ 𝛾̇ 𝐴 (6.4) Die Reynoldszahl 𝑅𝑅 ist eine dimensionslose Kennzahl, die eine Aussage über das Turbulenzverhalten der Strömung zulässt. Mit der kinematischen Viskosität 𝜈 gilt für einen Spalt: Die Definition für ein Rohr lautet: 𝑅𝑅 = 𝑢𝑚 𝐻 𝜈 (6.5) 𝑢𝑚 𝐷 (6.6) 𝜈 Liegt die Reynoldszahl unter einem kritischen Wert, kann von einer laminaren Strömung ausgegangen werden. Da der Übergang zwischen stark turbulenter und laminarer Strömung nicht abrupt erfolgt, sondern ein großer Übergangsbereich existiert und die kritische Reynoldszahl von vielen Parametern abhängt, muss dieser Wert als Richtwert angesehen werden. Es wird hier eine kritische Reynoldszahl von 2300 angenommen (Surek und Stempin 2007). Ist die Reynoldszahl kleiner als 2300 können die oben eingeführten Gleichungen für laminare Strömungen zur Anwendung kommen. 𝑅𝑅 = 6.4.2 Messsystem zur Bestimmung der kritischen Scherkräften Um die kritischen Scherkräfte bzw. Schubspannungen für B. amyloliquefaciens FZB42 zu ermitteln, wurden mehrere Versuchsreihen durchgeführt. Die Bakterien wurden hierzu im Standard-Nährmedium-I-¼ kultiviert und in der Wachstumsphase werden pro Versuch durch drei 20 ml Spritzen Bakteriensuspension entnommen. Mit einer Spritzenpumpe (Fusion 400, Chemyx Inc., USA) wurde die entnommene Suspension mit 4 ml min-1 durch eine Referenzkapillare mit 1 mm Durchmesser und 0,6 m Länge sowie durch zwei Messkapillaren mit 0,25 mm sowie 0,5 mm Durchmesser und je 0,6 m Länge gepumpt. Die behandelten Proben wurden anschließend mehrmals abwechselnd unter dem Mikroskop untersucht und je das Verhältnis von beweglichen zu unbeweglichen Bakterien ermittelt. 6.4.3 Peripherie für den automatisierten Betrieb Für einen reibungslosen automatischen Betrieb der Durchflussküvette über den gesamten Zeitraum der Fermentation sind Peripheriegeräte notwendig. Der Verfahrensablauf von Probenentnahme bis zur Bildaufnahme ist vereinfacht in Abbildung 17 dargestellt. Da die Zelldichte während der Wachstumsphase stark ansteigt, wird die Probe vor der Zuführung auf eine konstante optische Dichte eingestellt. Die Messung der optischen Dichte erfolgt kontinuierlich während der Probenentnahme sowie der Verdünnung mit VE-Wasser. Die Anzahl an Bakterien pro Aufnahme kann somit reduziert werden. 34 Material und Methoden Abbildung 17: Vereinfachtes Verfahrensfließbild der Bildaufnahme mit Peripherie Die Probe wird je nach Fermentationsstadium mit VE-Wasser verdünnt. Die verdünnte Probe wird schließlich zur Durchflussküvette gepumpt, wo die Bildaufnahme durchgeführt wird. Zum Einstellen der Schichtdicke wird Vakuum benötigt. Eine anschließende Belüftung sowie die Entsorgung von Abwasser sind für den Dauerbetrieb ebenso notwendig. Die Möglichkeit, die Probe zu verdünnen, erlaubt es, die Anzahl der Bakterien im Sichtfeld der Kamera für die Bewegungsverfolgung optimal einzustellen. Um die optimale Anzahl an Bakterien pro Bild zu ermitteln, wurde der zu erwartende Fehler der Durchschnittsgeschwindigkeit 𝑆𝑆𝑆𝑣𝑣 abgeschätzt. 𝑆𝑆𝑆𝑣𝑣 = 𝑆𝑆𝑣𝑣 (𝑛𝑣𝑣 − 𝑛𝑒𝑒 ) + 𝑆𝑆𝐹 𝑛𝑒𝑒 𝑛𝑣𝑣 �𝑛𝑣𝑣 (6.7) Der erwartete Fehler (6.7) kann mit der erwarteten Standardabweichung 𝑆𝑆𝑣𝑣 , der erwarteten Anzahl an Einzelgeschwindigkeiten 𝑛𝑣𝑣 , der erwarteten Anzahl an fehlerhaften Sprüngen zwischen nahen Bakterien 𝑛𝑒𝑒 und der Standardabweichung für die Berechnung auf Grundlage von fehlerhaften Sprüngen 𝑆𝑆𝐹 abgeschätzt werden. Kommen sich zwei Bakterien zu nahe, besteht die Möglichkeit, dass die automatische Wegverfolgung die beiden Bakterien während der Indizierung vertauscht. Somit kann es zu einem Fehler in der Bestimmung der Durchschnittsgeschwindigkeit bei beiden Wegstrecken kommen. Die erwartete Anzahl an Einzelgeschwindigkeiten 𝑛𝑣𝑣 ergibt sich aus der Anzahl an Bakterien pro Bild 𝑛, der Anzahl an Bildern pro Bildserie 𝑓𝑛 , der Anzahl an Bildserien pro Messpunkt 𝑏𝑛 , der erwarteten Anzahl an Berührungen zwischen den Bakterien 𝑛𝑒𝑒 sowie der Wahrscheinlichkeit für das Auftreten von kleinen Strecken 𝑝𝑘𝑘 . Durch eine Berührung zweier Bakterien während ihrer Bewegung werden beide Wege mit der Länge (𝑏𝑛 𝑓𝑛 − 1) getrennt und es entstehen vier kürzere Wege. Hierdurch gehen vier Einzelgeschwindigkeiten verloren. Ist einer der vier Wege durch die Trennung nun kürzer als die Mindestlänge fällt dieser für die Auswertung komplett weg. 𝑛𝑣𝑣 = 𝑛 (𝑏𝑛 𝑓𝑛 − 1) − 4 𝑛𝑒𝑒 𝑝𝑘𝑘 (6.8) Die erwartete Anzahl an Berührungen zwischen den Bakterien 𝑛𝑒𝑒 wird aus der eingenommenen Fläche aller Bakterien 𝐴𝑖𝑖𝑖 , der Anzahl an Bakterien pro Bild 𝑛 und der Fläche des Bildes 𝐴𝐵 berechnet. 35 Material und Methoden 𝑛 4 𝐴𝑖𝑖𝑖 𝐴𝐵 (6.9) 5 𝑝𝑒𝑒 𝐴𝑖𝑖𝑖 𝐴𝐵 (6.10) 𝑛𝐵 = � 𝑖=0 Die erwartete Anzahl an fehlerhaften Sprüngen ergibt sich aus der Wahrscheinlichkeit für einen fehlerhaften Sprung zwischen nahen Bakterien 𝑝𝑒𝑒 , der eingenommenen Fläche aller Bakterien 𝐴𝑖𝑖𝑖 , der Fläche des Bildes 𝐴𝐵 sowie der Anzahl an Bakterien pro Bild 𝑛. 𝑛 𝑛𝑒𝑒 = � 𝑖=0 Ebenso wird das Gewicht der Probe während des gesamten Prozesses protokolliert, wodurch das Verdünnungsverhältnis zu jeder einzelnen Probe festgelegt ist. Die Probenentnahme beträgt ca. 40 ml bei etwa 0,4 ml Totvolumen, die Größe des Totvolumens beträgt somit etwa 1% der Probenentnahme. In Abbildung 18 ist das vereinfachte Fließbild der Probenvorbereitung und der Messung zu sehen. Abbildung 18: Vereinfachtes Fließbild der Probenvorbereitung Die Probenvorbereitung wird benötigt, um bei hohen Zelldichten die Probe zu verdünnen. Der OD600 (QIRC 2.02) wird bei der Verdünnung auf einen definierten Wert eingestellt. Der Grad der Verdünnung wird hierbei über die Gewichtsmessung (WIR 2.03) erfasst. Weiterhin sind die Durchflussküvette sowie die Vakuum- und Abwasserbehandlung zu sehen. Das Vakuum ist notwendig, um die Schichtdicke der Durchflussküvette einzustellen. Während die neue Probe durch die Küvette gespült wird, muss ein geringer Gegendruck herrschen, um die Küvette komplett zu öffnen und rückstandsfrei zu durchströmen. Der Durchfluss durch die Küvette während der Durchspülung beträgt etwa 30 ml min-1. Nachdem die Küvette mit neuer Probe vollständig durchströmt wurde, wird Unterdruck angelegt, um die Schichtdicke zu minimieren. Anschließend kann die Aufnahme der Bildserie erfolgen. 36 Material und Methoden 6.4.4 Kamera und Mikroskop Für die Analyse der Bakterienbewegung wurde die Küvette auf einem herkömmlichen Labormikroskop vom Typ CX31 (Olympus Corporation, Japan) eingesetzt. Die Bildaufnahme erfolgte im Durchlicht mit einem 40x-Objektiv des Typs PLCN40XPH (Olympus Corporation, Japan). Bei der eingesetzten Kamera handelt es sich um eine GigE Farb-Kamera des Typs DFK 23G445 (The Imaging Source Europe GmbH). Die Auflösung der Kamera beträgt 1280 x 960 Pixel bei einer Bildrate bis zu 30 fps. Jedes Pixel besteht aus drei 8 bit Werten mit jeweils 8 bit Werten für Rot, Grün und Blau. Es ist ein 1/3“ Sensor in der Kamera verbaut. Die Lichtverstärkung der Kamera, die Belichtungszeit (1 µs – 30 s) sowie das Farbverhältnis sind online im System parametrierbar. Um die reale Größe eines Pixels bestimmen zu können, wurde ein Objektmikrometer (2 mm in 200 Skalenteile, 10008.04.005, POG Präzisionsoptik Gera GmbH) zur Kalibrierung verwendet. Das Objektmikrometer besitzt eine Miniaturskala mit 2 mm Länge in 200 Skalenteilen. Der Abstand zweier Skalenteile beträgt somit 10 µm. Das Objektmikrometer wurde auf das Mikroskop aufgebracht und scharf gestellt. Anschließend wurde manuell ein Bild mit der verwendeten Kamera aufgenommen. Das Bild wurde mit dem Programm Vision Assistant Version 2013 (National Instruments) geöffnet und die Strecken von Skalenteil zu Skalenteil vermessen, um anschließend den Mittelwert zu bilden. Der Mittelpunkt der Linien von den Skalenteilen wird automatisch vom Programm ermittelt. Da der Mittelpunkt der Linie aus dem grafischen Schwerpunkt ermittelt wird, können sich Komastellen ergeben, obwohl Pixel eine diskrete Einheit darstellen. 6.4.5 Auflösungsvermögen der gewählten Komponenten Das maximale Auflösungsvermögen eines Lichtmikroskops ist durch die Wellennatur des Lichtes begrenzt. Die Gleichungen eines Gauß-Strahls, der als transversales Profil einer Gauß-Kurve beschrieben wird, sollen hier als Berechnungsgrundlage dienen. In Abbildung 19 ist die Ausbreitung eines Gauß-Strahls dargestellt. Folgend sei der Strahlradius als 𝑤(𝑧) definiert. Die Strahltaille stellt den gewünschten Fokuspunkt des Mikroskops dar. Der Strahlradius wird durch die 𝑒 −2 –Begrenzung der GaußKurve bestimmt. An dieser Begrenzung ist die Amplitude der Gauß-Kurve auf 𝑒 −1 gefallen. Da die Lichtintensität aus der Amplitude zum Quadrat berechnet wird, beträgt die Lichtintensität an dieser Stelle 𝑒 −2 . Der Strahlradius an dieser Stelle wird als 𝑤0 bezeichnet (Pedrotti et al. 2005). 37 Material und Methoden Abbildung 19: Gauß-Strahl, der sich in z-Richtung ausbreitet Die Strahltaille des Gauß-Strahls entspricht dem Fokuspunkt des Mikroskops. Die 1/e2-Begrenzung der Gaußkurve bestimmt den Strahlradius w(z) (Pedrotti et al. 2005, S. 649) (modifiziert). Die folgenden Gleichungen (6.11) bis (6.18) stammen aus Pedrotti et al. (2005). Nach der Schärfentiefe (𝑧𝑅 ) ist der Strahlradius um den Faktor √2 vergrößert. Die Schärfentiefe wird häufig auch Rayleighlänge genannt. 𝜋 𝑤0 2 𝑧𝑅 = 𝜆 (6.11) Wie in (6.11) zu sehen wird die Schärfentiefe durch den Strahlradius an der Strahltaille sowie durch die Wellenlänge des Lichts (λ) bestimmt. 𝑤0 𝜆 𝜃0 = 𝑎𝑎𝑎𝑎𝑎𝑎 � � = 𝑎𝑎𝑎𝑎𝑎𝑎 � � 𝑧𝑅 𝜋 𝑤0 (6.12) Aus der Gleichung (6.12) geht hervor, dass der Tangens des halben Strahlöffnungswinkels (𝜃0 ) dem Verhältnis von Strahlradius an der Strahltaille zur Schärfentiefe entspricht. Weiterhin ist er aus Wellenlänge und Strahlradius an der Strahltaille berechenbar. Die Auflösungsgrenze (𝑦𝑚𝑚𝑚 ) kann durch den Mindestabstand zweier Punkte bestimmt werden, welche in der Abbildung getrennt auflösbar sind. Für die folgende Betrachtung wird der Strahlradius als Abgrenzung herangezogen. Die mit diesem Kriterium berechnete Auflösungsgrenze wird hier mit 𝑦min 𝑒 −2 benannt. Hierbei ist nicht der kleinste Strahlradius an der Strahltaille (𝑤0 ) heranzuziehen, sondern die Schärfentiefe zu berücksichtigen. Der dortige Strahlradius (𝑤(𝑧𝑅 )) muss für die Berechnung verwendet werden. 38 Material und Methoden 𝑦𝑚𝑚𝑚 1 = 2 𝑤(𝑧𝑅 ) = 2 √2 𝑤0 𝑒2 (6.13) Stellt man (6.12) nach w0 um und setzt in (6.13) ein, ergibt sich folgende Abhängigkeit: 𝑦𝑚𝑚𝑚 1 = 𝑒2 2 √2 𝜆 𝜋 𝑡𝑡𝑡(𝜃0 ) (6.14) Das Auflösungsvermögen ist nach (6.14) somit nur vom Strahlöffnungswinkel und der Wellenlänge abhängig. Je kürzer die Wellenlänge ist, desto kleinere Strukturen sind auflösbar. Der Öffnungswinkel verhält sich umgekehrt proportional, je größer der Öffnungswinkel ist, desto besser die Auflösung. Wie in (6.12) zu sehen ist, hat ein hoher Öffnungswinkel jedoch eine geringe Schärfentiefe zur Folge. Je weiter der Öffnungswinkel ist, desto größer wird das Verhältnis von kleinstem Strahlradius an der Strahltaille zu der Schärfentiefe. Der maximal erreichbare Strahlöffnungswinkel ist eine charakteristische Größe bei Mikroskopobjektiven und wird meist als numerische Apertur (𝐴𝑁 ) angegeben. 𝐴𝑁 = 𝑛𝑖 𝑠𝑠𝑠(𝜃0 ) (6.15) Die numerische Apertur wird als Produkt aus dem Brechungsindex des Immersionsmediums (𝑛𝑖 ) und dem halben objektseitigen Strahlöffnungswinkel (𝜃0 ) definiert. Aus (6.14) und (6.15) ergibt sich folgender Zusammenhang: 𝑦𝑚𝑚𝑚 1 = 𝑒2 2 √2 𝜆 𝐴 𝜋 𝑡𝑡𝑡 �𝑎𝑎𝑎𝑎𝑎𝑛 𝑛𝑁 � 𝑖 (6.16) Die elektrische Feldstärkenverteilung zweier Punkte mit dem Abstand 𝑦min 𝑒 −2 sowie die daraus resultierende Intensitätsverteilung sind in Abbildung 20 dargestellt. Die Verteilung der elektrischen Feldstärke erweckt zunächst den Eindruck, dass die Lichtpunkte schwer als getrennte Punkte wahrnehmbar sind. Die Intensität verhält sich jedoch proportional zur quadrierten Amplitude der Feldstärke. Dadurch ist ein deutlicher Kontrast der beiden Maxima zum Minimum zwischen den Punkten zu erkennen. 𝐼 ∝ 𝐸2 (6.17) 39 Material und Methoden Abbildung 20: Verteilung der elektrischen Feldamplitude zweier Lichtpunkte Oben: Verteilung der elektrischen Feldamplitude (E) von zwei Lichtpunkten an den Positionen (y1, y2) mit dem Abstand ymin1/e². Die Verteilung wird an den charakteristischen Stellen z0 und zR abgebildet. Mit EØ wird die resultierende Verteilung der elektrischen Feldamplitude (EØ1 + EØ2) ermittelt. Unten: Verteilung der normierten Feldamplitudenverteilung (EØ1 + EØ2) sowie die daraus resultierende Intensitätsverteilung (IØ1 + IØ2). In den vorangegangen Erläuterungen wurde gezeigt, dass die Verwendung eines Objektivs mit hoher numerischer Apertur sowie der Einsatz von kurzwelligem Licht die Auflösung positiv beeinflussen. 6.4.6 Bewegungsunschärfe Einen weiteren schärfebeeinflussenden Faktor stellt die Bewegungsunschärfe dar. Die Belichtungszeit der Kamera CCD-Sensoren kann nicht unendlich klein sein, daher bewegt sich der Partikel während der Aufnahmezeit weiter fort. Somit ist eine Bewegungsunschärfe im Bild zu beobachten. Die Bewegungsunschärfe (𝑈𝐵 ) ergibt sich aus dem Produkt der Objektgeschwindigkeit (𝑣𝑂 ) und der Belichtungszeit (𝑡𝐵 ). 𝑈𝐵 = 𝑣𝑂 ∙ 𝑡𝐵 (6.18) Als nicht sichtbarer Schärfeverlust wird hier eine Unschärfe von halber Seitenlänge eines Pixels definiert. 40 Material und Methoden 6.4.7 Bildspeicherung Die Bildaufnahmefunktion ist in der Kontrollsoftware des Fermenters integriert. In den Grundeinstellungen besteht eine Bildserie aus 30 Aufnahmen. Mit einer Bildrate von 30 fps stellt dies ein Zeitfenster von einer Sekunde dar. Die aufgenommene Bildserie wird nicht direkt ausgewertet, sondern zunächst im PNGFormat auf die Festplatte des Kontrollrechners gespeichert. Die Bildserien sind so dokumentiert und können später jederzeit begutachtet werden. Das PNG-Format ist ein weit verbreitetes Datenformat für Grafiken. Es verfügt über eine verlustfreie Komprimierung und ist lizenzfrei. Die Abspeicherung von RGB-Grafiken mit 8 bit pro Farbkanal ist problemlos möglich. Die automatisierte Bildserienauswertung lädt die Bildserien und wertet die Kenndaten automatisch aus. Die Bildserienauswertung kann von der Fermenter-Kontrollsoftware direkt nach der Messung gestartet werden, eine nachträgliche Auswertung einer beliebigen, gespeicherten Bildserie kann jedoch auch jederzeit manuell gestartet werden. Für manuelle Analysen, z.B. der Bewegungsmuster, können ebenso Bildserien über längere Zeitfenster aufgenommen werden. Die Obergrenze wird hierbei hauptsächlich vom Arbeitsspeicher und der Festplattengeschwindigkeit gesetzt. Bildserien über fünf Sekunden (150 Aufnahmen pro Bildserien) stellen auch für Standard Labor- oder Bürorechner (Pentium Dual-Core 2,6 GHz, 4 GB-Arbeitsspeicher, Windows 7) kein Problem dar. 6.5 Automatisierte Bildserienanalyse Durch die Bildauswertung werden die Anzahl der beweglichen Bakterien sowie deren Bewegungsgeschwindigkeit erfasst. Weiterhin können Aussagen über die Form (Länge, Breite) und das Bewegungsmuster der Bakterien aus den Daten getroffen werden. 6.5.1 Bildanalyse Unschärfe, wechselnde Belichtung, leicht schwankende Schichtdicken, Partikel aus dem Nährmedium und nicht voraussagbares Verhalten der Bakterien stellen große Herausforderungen an eine automatisierte Bildauswertung. Die automatisierte Bildauswertung wurde in NI LabVIEW 2013 mit NI LabVIEW Vision Development Module (National Instruments) programmiert. Die vereinfachte Softwarestruktur der Bildserienauswertung ist in Abbildung 21 dargestellt. 41 Material und Methoden Abbildung 21: Schematischer Programmablauf der Bildauswertung Zunächst wird eine Bildserie, bestehend aus 30 Aufnahmen, geladen. Nach Vorfilterung und indizierter Seriensubtraktion werden die Partikelkenndaten bestimmt. Aus den Partikelkenndaten der einzelnen Aufnahmen können die Bakterien zugeordnet und verfolgt werden. Bevor das Ergebnis bereitgestellt wird, muss eine Nachbearbeitung vorgenommen werden. Außerdem müssen die Daten aus dem Pixelraum in den metrischen Raum transformiert werden. Im ersten Schritt wird die Bildserie geladen und die beweglichen Partikel werden in jedem einzelnen Bild durch die indizierte Seriensubtraktion hervorgehoben. Dies ist ähnlich dem Vorgehen von Junker et al. (2007), die aus den laufenden Aufnahmen stetig ein Durchschnittshintergrundbild generieren und dieses von der aktuellen Aufnahme abziehen. In den nächsten Schritten werden die Partikelkenndaten bestimmt und den Bakterien zugeordnet. Diese Zuordnung muss gegebenenfalls mehrmals überarbeitet werden, bevor das Ergebnis auf den metrischen Raum transformiert und bereitgestellt wird. Das Programm zur automatischen Auswertung der Bildserien inkl. Benutzeroberfläche kann autark arbeiten oder als Unterprogramm von überlagerten Programmen aufgerufen werden. Dies ermöglicht zum einen die Untersuchung ausgewählter Bildserien oder das Ausführen direkt aus dem Steuerungsprogramm des Fermenters, wie auch das Ausführen aus übergeordneten Programmen, die z.B. mehrere Bildserien ganzer Fermentationsverläufe abarbeiten. 6.5.2 Bildserie laden Die Bildanalyse ist auf die Auswertung von digitalen Bildern im PNG-Format mit den Abmessungen 1280 x 960 Pixel optimiert. Der Programmstart mit dem Laden der Bildserie kann manuell auf eine beliebige, gespeicherte Bildserie angewandt werden oder direkt automatisch, z.B. nach der Bildaufnahmefunktion, ausgeführt werden. In jedem Fall bleiben die gespeicherten Bilder im Original erhalten und werden nicht durch die Bildauswertung manipuliert. 42 Material und Methoden 6.5.3 Indizierte Seriensubtraktion Vor der indizierten Seriensubtraktion werden die Farbbilder in Grauwertbilder gewandelt. Es wird hierfür die integrierte LabVIEW-Funktion „IMAQ ExtractSingleColorPlane“ genutzt. Mit der Funktion ist es möglich, die Werte verschiedener Farbebenen zu extrahieren und in ein Grauwertbild zu wandeln. Hierzu wird dem zur gewählten Ebene gehörenden Wert der einzelnen Pixel ein Grauwert von 0 (schwarz) bis 255 (weiß) zugeordnet. In Tabelle 3 sind die verschiedenen Ebenen, die aus dem Farbbild extrahiert werden können, angegeben. Tabelle 3: Wandlungsmöglichkeiten von einem Farb- zu einem Grauwertbild Die Einstellmöglichkeiten zur Wandlung eines RGB-Bildes in ein Grauwertbild der LabVIEW-Funktion „IMAQ ExtractSingleColorPlane“. Red Der Rot-Wert des Pixels wird extrahiert (RGB-Farbraum) Green Der Grün-Wert des Pixels wird extrahiert (RGB-Farbraum) Blue Der Blau-Wert des Pixels wird extrahiert (RGB-Farbraum) Hue Der Farbwert des Pixels wird extrahiert (HSL-Farbraum) Saturation Die Farbsättigung des Pixels wird extrahiert (HSL-Farbraum) Luminance Die Farbhelligkeit des Pixels wird extrahiert (HSL-Farbraum) Value Die Helligkeit des Pixels wird extrahiert (HSV-Farbraum) Intensity Die Lichtintensität des Pixels wird extrahiert (HSI-Farbraum) Zum näheren Verständnis der weiteren Operationen sind folgende Definitionen notwendig: Der Matrizenraum bestehe aus dem Tripel [ℳ, +,∙] über dem Körper ℝ𝑚×𝑛 , mit der Menge ℳ, der Abbildung der komponentenweisen Addition ⟨+⟩ ∶ ℳ × ℳ → ℳ (𝐴, 𝐵) ↦ 𝐴 + 𝐵 = (𝑎𝑖𝑖 + 𝑏𝑖𝑖 ) und der Abbildung der komponentenweisen skalaren Multiplikation. ⟨∙⟩ ∶ ℝ × ℳ → ℳ (𝜆, 𝐴) ↦ 𝜆 ∙ 𝐴 = (𝜆 ∙ 𝑎𝑖𝑖 ). Zusätzlich ist der komponentenweise Absolutwert definiert durch ⟨𝑎𝑎𝑎⟩ ∶ ℳ → ℳ (𝐴) ↦ 𝑎𝑎𝑎(𝐴) = (�𝑎𝑖𝑖 �). Seien nun die digitalen Bilder (Matrizen) 𝐴, 𝐵 ∈ ℳ mit der Auflösung (Dimensionen) 𝑚 × 𝑛, bestehend aus Bildpunkten (Komponenten) 𝑎𝑖𝑖 , 𝑏𝑖𝑖 , so kann die indizierte Seriensubtraktion wie folgt ausgedrückt werden: 43 Material und Methoden 𝑙−1 30 𝑘=1 𝑘=𝑙+1 𝐵𝑙 = � 𝑎𝑎𝑎(𝐴𝑘 − 𝐴𝑙 ) + � 𝑎𝑎𝑎(𝐴𝑘 − 𝐴𝑙 ) (6.19) Aus den 30 Aufnahmen einer Bildserie 𝐴 werden über die indizierte Seriensubtraktion (6.19) 30 Summen-Differenzbilder 𝐵 erstellt. Die aktuell indizierte Aufnahme 𝐴𝑙 wird hierbei von allen anderen Bildern der Serie 𝐴𝑘 komponentenweise subtrahiert und der komponentenweise Absolutwert (Pixel = Komponenten) wird gebildet. Aus den einzelnen Ergebnissen wird ein Summen-Differenzbild 𝐵𝑙 über eine komponentenweise Addition erzeugt. Anschließend wird aus dem Summen-Differenzbild das Bildrauschen reduziert sowie der Kontrast erhöht. Alleine durch die Erstellung eines Summenbildes über 30 Aufnahmen wird das Rauschen minimiert. Da das Rauschen von Bild zu Bild in einem Bereich um den wahren Wert schwankt, mitteln sich die positiven und negativen Ausschläge weitgehend heraus. Da es sich jedoch um Differenzbilder handelt, sind auch kleine Differenzen, die durch das Rauschen zurückbleiben, störend. Zur Unterdrückung des verbleibenden Rauschens kommt die Anwendung eines Tiefpassfilters in Frage. Dadurch verliert das Bild aber an Schärfe. Da das Rauschen vor allem dort stört, wo keine Bewegung stattgefunden hat, das Bild somit schwarz sein sollte (Pixelwert 0), ist es auch möglich, von der Bildmatrix einen konstanten Wert abzuziehen, oder aber Pixel unterhalb eines Schwellwerts auf 0 zu setzen. Weiterhin wird durch die Differenzbildung der Dynamikbereich des Bildes nicht voll ausgenutzt. Haben sich z.B. nur Werte zwischen 0 – 100 aus der Differenzbildung ergeben, können diese durch die Multiplikation mit 2,55 auf einen Bereich von 0 – 255 gespreizt werden, um den Kontrast in den Bildern zu erhöhen. In der gegebenen Anwendung wurden durch folgende Berechnung das Rauschen eliminiert und der Kontrast erhöht: 𝐵𝑙 = (𝐵𝑙 − 2 𝑛 ∑𝑚 𝑖=0 ∑𝑗=0 𝑏𝑖𝑖 𝑚+𝑛 ) 255 max max 𝑏𝑖𝑖 (6.20) 𝑖→𝑚 𝑗→𝑛 Der zweifache Mittelwert über die Pixel wird abgezogen, um kleine Differenzen, die aufgrund von Bildrauschen entstehen, zu entfernen. Der Kontrast wird anschließend durch eine Multiplikation auf den Wertebereich von 0 – 255 gespreizt. Gleichung (6.20) würde mathematisch auch negative Pixelwerte erzeugen. Aufgrund der internen Datentypwandlung im LabVIEW-Programm werden an dieser Stelle jedoch negative Werte auf Null gesetzt. 6.5.4 Bestimmung der Partikelkenndaten Die Funktion der Partikelkenndatenbestimmung basiert auf der Grundlage des Unterprogramms „IMAQ Particle Analysis“ aus dem LabVIEW NI Vision Development Module. Hier werden für jedes Summen-Differenzbild mehrere gefundene Partikel mit zugehörigen Partikelmerkmalen (z.B. Schwerpunkt in X,Y-Bildkoordinaten, Durchmesser, Fläche, Orientierung, etc.) in einer Partikelmatrix 𝐷 gespeichert. 44 Material und Methoden Vor der Bestimmung der Partikelmerkmale werden sehr kleine Partikel herausgefiltert. Dies ist ein gängiges Vorgehen in Arbeiten mit dem Schwerpunkt der Bildanalyse aus Mikroskopie-Aufnahmen (Pons et al. 1993; Tucker et al. 1994). Um kleine Objekte (Bildstörungen, Kleinstpartikel im Medium) aus den Bildern zu eliminieren, ist es zum einen möglich, die Grauwertbilder vor der Binarisierung mit einem Tiefpassfilter (z.B. Mittelwertfilter) oder einem morphologischen Filter (Erosion) zu behandeln. Eine weitere Möglichkeit bietet die Anwendung der Erosion direkt auf das Binärbild. Durch die Filter werden jedoch auch die größeren Objekte in ihrer Erscheinung verändert. Die Anwendung eines gegensätzlich wirkenden Filters, nachdem die kleinen Objekte eliminiert wurden, kann die verbleibenden großen Objekte wieder an ihre ursprüngliche Erscheinung angleichen, jedoch bleibt das Ergebnis nur eine Annäherung an das Original. Die Dilatation, nach der Erosion auf ein Binärbild angewendet, führt zu der beschriebenen annäherungsweisen Wiederherstellung der ursprünglichen Größe. Soll eine Veränderung der großen Partikel durch das Programm vermieden werden sind komplexere Algorithmen notwendig. Im LabVIEW NI Vision Development Module steht hierfür eine eigene Funktion zur Verfügung („IMAQ RemoveParticle“). In diesem Unterprogramm wird eine zweifache Erosion des Bildes durchgeführt. Partikel, die durch die Erosion verschwinden, werden zur Partikelkenndatenbestimmung nicht herangezogen. Im Gegensatz zur Anwendung einer gewöhnlichen zweifachen Erosion werden die Partikel, die nicht verschwinden, durch die Funktion jedoch nicht in Größe und Form verändert. 6.5.5 Zuordnung der Bakterien zu den Kenndaten Um die Bakterien den verschiedenen Kenndaten zuzuordnen, wird zunächst die Fehlermatrix 𝐸 gebildet. 6 𝑒𝑙𝑙𝑙 = � 𝑤𝑞 ∙ �𝑑𝑙𝑙𝑙 − 𝑑𝑙+1𝑟𝑟 � (6.21) 𝑞=1 In (6.21) wird die Summe der Merkmalsdifferenzen aller gefundener Partikel in Bild 𝑙 zu allen gefundenen Partikeln in Bild 𝑙 + 1 berechnet. 𝑒𝑙𝑙𝑙 ist hier eine Komponente der dreidimensionalen Fehlermatrix 𝐸, wobei der Index 𝑙 die Nummer des Bildes, 𝑝 die Nummer des Partikels in Bild 𝑙 und 𝑟 die Nummer des Partikels in Bild 𝑙 + 1 darstellt. Ebenso ist 𝑤 eine Komponente des Gewichtungsarrays 𝑊 und 𝑑 eine Komponente der Partikelmatrix 𝐷. Der Index 𝑞 steht hier für die Nummer des indizierten Merkmals (z.B. Schwerpunkt, Fläche, etc.). Zum besseren Verständnis ist die Berechnung in Abbildung 22 in einem vereinfachten Programmablaufplan gezeigt. 45 Material und Methoden Abbildung 22: Vereinfachter Programmablaufplan für die Zuordnung der Kenndaten Die Fehlermatrix wird in einer dreifachen Schleife erzeugt. Es wird die Summe der gewichteten Merkmalsdifferenzen zwischen jedem Partikel aus Bild 𝑙 und jedem Partikel aus Bild 𝑙+1 gebildet. Es ist zu sehen, dass die dreidimensionale Fehlermatrix 𝐸 in einer dreifachen Schleife erzeugt wird. Die Summe der gewichteten Merkmalsdifferenzen aller gefundenen Partikel in je zwei aufeinanderfolgenden Bildern wird in der Fehlermatrix 𝐸 gespeichert. Auf Grundlage der Fehlermatrix 𝐸 und der Partikelmatrix 𝐷 wird die Zuordnung der einzelnen Partikel zueinander realisiert. Dies geschieht in einem komplexen Zuordnungsalgorithmus, der über die kleinsten Merkmalsdifferenzen die zueinander gehörenden Partikel findet und in einer Bakterienmatrix 𝐶 speichert. Hier findet die Wegverfolgung der einzelnen Bakterien statt. Der Algorithmus verhindert Mehrfachzuweisungen, wenn sich mehrere Bakterien kreuzen oder nahe beieinander schwimmen. Weiterhin registriert der Algorithmus das Herausschwimmen von Bakterien aus dem Aufnahmebereich sowie das Hineinschwimmen in den Aufnahmebereich. Sobald kein Partikel im folgenden Bild gefunden wird, dessen Merkmalsdifferenz kleiner als ein Schwellenwert ist, wird dem Bakterium kein weiterer Punkt zugeordnet. Das LabVIEWUnterprogramm „Wegverfolgung“ ist im Anhang (Seite 108) dargestellt. 46 Material und Methoden Es ist möglich, dass sich die Partikelmerkmale eines Bakteriums von einem Bild zum nächsten stark unterscheiden. Ist die Summe der Merkmalsdifferenz größer als die zulässige Schwelle, wird der Weg des Bakteriums beendet und das Bakterium wird im nächsten Bild als ein neues Bakterium geführt. Die möglichen Ursachen für große Merkmalsdifferenzen werden in Kapitel 8.3 diskutiert. 6.5.6 Nachbearbeitung der Zuordnung Wie in der Arbeit zur 2D-Wegverfolgung von Kang Li et al. (2006), bei der im Modul „Cell Arbitrator“ eine Nachbearbeitung der Zuordnungen durchgeführt wird, ist auch hier eine Nachbearbeitung der Zuordnung nötig. Ähnlich dem Modul des „Cell Arbitrator“ wurde ein eigenes Unterprogramm zur Nachbearbeitung der Zuordnung erstellt. In diesem Unterprogramm der Bildanalysesoftware werden zunächst mögliche Unterbrechungen in der Wegverfolgung einzelner Bakterien zusammengeführt. Hierfür werden für gefundene Wege, die mitten in der Bildserie enden, passende Wege gesucht, die mit dem folgenden Bild beginnen. Die identifizierten Teilwege, die zu dem vorzeitig beendeten Weg passen könnten, werden anhand einiger Kriterien auf Übereinstimmung zu dem vorangegangenen Teilweg überprüft. Hierfür werden Richtung und Geschwindigkeit der ersten Teilstrecke berechnet. Anschließend überprüft das Programm, ob einer der möglichen Anknüpfungspunkte sich in der Nähe des nächsten erwarteten Wegpunktes befindet. Passt der Anfangspunkt einer der Teilstrecken, werden noch morphologische Merkmale (z.B. Länge, Fläche) der Bakterien miteinander verglichen. Sind auch diese Merkmale ausreichend ähnlich, werden die beiden passenden Teilstrecken zu einem neuen Weg zusammengeführt. Dieser Vorgang wird solange ausgeführt, bis keine Teilstrecken mehr zueinander passen. Im nächsten Schritt werden kurze Wege, die nur in wenigen Bildern gefunden wurden, aus der Zuordnung gelöscht. Zuordnungen von sehr kurzen Wegen stellen häufig falsch erkannte Partikel dar, oder aber Bakterien, die nur kurz den Bildaufnahmebereich gestreift haben. Warum Partikel mit kurzen Wegstrecken im Ergebnis nicht als bewegliches Bakterium gewertet werden sollten, wird in Kapitel 8.3 erörtert. 6.5.7 Bereitstellung des Ergebnisses Die Daten liegen nach der Nachbereitung der Zuordnung als dreidimensionales Array im Pixelraum vor. Der erste Index des dreidimensionalen Arrays entspricht der Nummer der erkannten Bakterien. Für jedes Bakterium ist eine Spalte pro Aufnahme, in der das Bakterium erkannt wurde, vorhanden. In den Zeilen sind die x,y-Koordinaten, die Orientierung, der Umfang, die Fläche, die Länge und die Breite, die im jeweiligen Bild ermittelt wurden, aufgetragen. Im ersten Schritt der Ergebnisbereitstellung werden diese dreidimensionalen Arrays in ein Array aus Datenclustern gewandelt. Hierfür wird für jedes Bakterium jeweils der Durchschnitt aus den Werten für Umfang, Fläche, Länge und Breite berechnet. Weiterhin werden die einzelnen x,y-Koordinaten mit Orientierung im Cluster gespeichert. Neben dem Datenclusterarray wird ein Array aus den Einzelgeschwindigkeiten erstellt. Alle Daten befinden sich hier zunächst noch im Pixelraum (siehe Abbildung 23). 47 Material und Methoden Abbildung 23: Ablauf der Ergebnisbereitstellung Zunächst stehen die reinen Pixeldaten der Wegverfolgung der einzelnen Bakterien in einem dreidimensionalen Array zur Verfügung. Aus diesem wird ein Array aus Einzelgeschwindigkeiten gebildet sowie ein Array aus Datenclustern. Anschließend werden die Daten aus dem Pixelraum in das metrische System umgerechnet. Im letzten Schritt werden die Daten aus dem Pixelraum in das metrische System skaliert. Die Pixel werden in µm umgerechnet und die Geschwindigkeiten, die zunächst in Pixeldifferenzen zwischen zwei Bildern ausgedrückt wurden, werden in µm s-1 umgerechnet. Als Ergebnis stehen schließlich ein Array aus Datenclustern sowie ein Array aus Einzelgeschwindigkeiten bereit. Im Datencluster sind für jedes Bakterium die Durchschnittswerte der morphologischen Daten (Umfang, Fläche, Länge, Breite) aus den einzelnen Aufnahmen, die Durchschnittsgeschwindigkeiten sowie die einzelnen Koordinaten und Orientierungen eingetragen. Im Array aus Einzelgeschwindigkeiten sind die Einzelgeschwindigkeiten in µm s-1 angegeben. Die Daten können zum einen in verschiedene Dateiformate (.txt, .csv, .xls) exportiert werden, um diese mit beliebigen Programmen auszuwerten, oder direkt an ein überlagertes Programm übergeben werden. 6.6 Programm zur interaktiven Erfassung der Bewegungsmuster Zur Verfolgung und Analyse der Bewegungsmuster der Bakterien wurde ein interaktives Auswerteprogramm entwickelt. Dieses Programm wurde ebenso mit NI LabVIEW 2013 inkl. NI LabVIEW Vision Development Module (National Instruments) programmiert. Im Programm können die Aufnahmen einer Bildserie nacheinander aufgerufen werden. Die Länge der Bildserie ist variabel. Je länger der Aufnahmezeitraum ist, desto genauer können die Bewegungsformen erfasst werden. Auf der linken Seite des Programms werden die Aufnahmen der gewählten Bildserie dargestellt. Bakterien, deren Bewegung nachverfolgt wird, werden dort mit einer Nummer versehen. Der Verlauf der Mikroorganismen wird interaktiv von Bild zu Bild mit Hilfe des Mauszeigers und der Eingabetaste dokumentiert. Damit ist die Bewegung des Bakteriums in jeder Aufnahme durch die x- und y-Koordinate festgelegt. Diese Daten können für jedes Bakterium exportiert werden (z.B. Excel). In Abbildung 24 ist ein Beispielbild aus dem Programm zur interaktiven Erfassung der Bewegungsmuster dargestellt. 48 Material und Methoden Abbildung 24: Aufnahme aus dem Programm zur Erfassung der Bewegungsmuster Exemplarisches Bild von Minute 225 aus Fermentation 4 (B. amyloliquefaciens FZB42 in Standard-Nährmedium-I-¼), dargestellt im Programm zur interaktiven Erfassung der Bewegungsmuster. Zu sehen sind die Bakterien mit Nummer und die Nachverfolgung der Bewegungen. Die Stelle, an der die Nummer steht, ist der Startpunkt bei Bild 0. Mit dem interaktiven Programm sollen charakteristische Bewegungsmuster identifiziert und die Anzahl des Auftretens der einzelnen Muster ermittelt werden. Zusätzlich zur Erfassung der Bewegungsmuster gibt es die Möglichkeit, die zurückgelegte Strecke und die Geschwindigkeit von Punkt zu Punkt zu erfassen. Dies ist für die Auswertung der Bewegungsformen nicht von Bedeutung, jedoch konnte das interaktive Programm so auch für die Validierung der Geschwindigkeitsbestimmung aus der automatischen Bildanalyse verwendet werden. Den einzelnen charakteristischen Bewegungsmustern wurden dann Kategorien zugewiesen und nach Anzahl der Beobachtungen ausgewertet. Aus der Anzahl der Beobachtungen wurden Kreisdiagramme erstellt, um das Auftreten der einzelnen Bewegungsmuster zu visualisieren. 6.7 Statistische Auswertung Die Einzelgeschwindigkeitsverteilung der vier charakteristischen Fermentationszeitpunkte (exponentielle Wachstumsphase durch Kohlenhydratverbrauch und Säurebildung, Diauxiestufe, Wachstum durch Säureverbrauch und Sporulation) aus Fermentation 1 – 3 wurde auf Normalverteilung überprüft. Die Überprüfung wurde mit Shapiro-Wilk-, Anderson-Darlingund Cramér-von-Mises-Test durchgeführt. Nachdem die Einzelgeschwindigkeitsverteilung als nicht normalverteilt befunden wurde, konnte der 49 Material und Methoden Wilcoxon-Rangsummentest (auch Mann-Whitney-U-Test) (Wilcoxon 1945; Mann und Whitney 1947) zur Überprüfung auf signifikante Unterschiede in den Einzelgeschwindigkeitsverteilungen eingesetzt werden. Hierzu wurde die Einzelgeschwindigkeitsverteilung zur Diauxiestufe gegen die Verteilung der anderen drei charakteristischen Fermentationszeitpunkte getestet. Beim Wilcoxon-Rangsummentest wird überprüft, ob zwei Messreihen der gleichen Grundgesamtheit angehören. Kann die Nullhypothese abgelehnt werden, kann eine signifikante Differenz zwischen den Messreihen angenommen werden. Als Ergebnis aus dem Wilcoxon-Rangsummentest erhält man den pWert. Der p-Wert ist ein Wahrscheinlichkeitswert und kann Werte zwischen 0 und 1 annehmen. Je kleiner der p-Wert, desto eher kann die Nullhypothese verworfen werden. Die Grenze, um die Nullhypothese sicher abzulehnen, wurde auf 0,05 gesetzt. Die Tests auf Normalverteilung sowie der Wilcoxon-Rangsummentest wurden mit dem Statistikprogramm R durchgeführt. Ebenso wurden die Einzelgeschwindigkeitsverteilungen der sechs charakteristischen Fermentationszeitpunkte (exponentielles Wachstum durch Glukoseverbrauch und Säurebildung, Diauxiestufe I, exponentielles Wachstum durch Stickstoff- und Säureverbrauch, Diauxiestufe II, Wachstum durch reinen Säureverbrauch, Sporulation) aus Fermentation 4 – 6 statistisch untersucht. Die nicht normalverteilten Einzelgeschwindigkeitsverteilungen wurden wie bei Fermentation 1 – 3 mit dem Wilcoxon-Rangsummentest auf signifikante Unterschiede untersucht. Es wurden jeweils die beiden Diauxiestufen gegen die fünf anderen Geschwindigkeitsverteilungen getestet. Die Messdaten aus dem Experiment zur Ermittlung des Einflusses der Scherkraft auf die Beweglichkeit der Bakterien wurden ebenso mit dem Statistikprogramm R ausgewertet. Mit Hilfe des Shapiro-Wilk-, Anderson-Darling- und Cramér-von-Mises-Tests wurden die Daten auf Normalverteilung überprüft. Liegt das Ergebnis aller drei Tests über 0,05, können die Daten als normalverteilt angenommen werden. Signifikate Unterschiede der Mittelwerte wurden mit dem Zweistichproben-t-Test und der Varianzen mit dem F-Test untersucht. Auch bei diesen Tests wurde ein Signifikanzniveau von 0,05 gewählt, liegt der Wert über 0,05 kann nicht von signifikanten Unterschieden ausgegangen werden. 50 Ergebnisse 7 Ergebnisse Die Ergebnisse sind in zwei Unterkapitel gegliedert. Zunächst werden die technischen Ergebnisse, die zur Entwicklung des Messsystems notwendig waren erläutert. Zum anderen werden die mit dem entwickelten Messsystem erhaltenen Messergebnisse aus den Beispielanwendungen gezeigt. 7.1 Technische Ergebnisse Die Ergebnisse, die zur Entwicklung und Optimierung des in Kapitel 6 beschriebenen Messsystems geführt haben, sind im Folgenden aufgeführt. 7.1.1 Vermessung der Medien Die Dichte, kinematische und dynamische Viskosität des Standard-Nährmediums-I-¼ und des ABiTEP-Mediums-¼ bei 35 °C sind in Tabelle 4 zu sehen. Tabelle 4: Eigenschaften des Standard-Nährmediums-I-¼ und ABiTEP-Mediums-¼ Viskosität und Dichte der verwendeten Medien bei 35 °C. Dichte [g l-1] kinematische Viskosität [mm2 s-1] dynamische Viskosität [Pa s] StandardNährmedium-I-¼ 996,5 0,785 0,7818 ABiTEP-Medium-¼ 997,9 0,805 0,8028 Das Fließverhalten der beiden Medien ist in Abbildung 25 dargestellt. Abbildung 25: Fließverhalten von Standard-Nährmedium-I-¼ und ABiTEP-Medium-¼ Das Fließverhalten des Standard-Nährmediums-I-¼ (gestrichelte Linie) und des ABiTEPMediums-¼ (durchgezogen Linie) bei 35 °C wurden mit dem Rheometer Physica UDS 200 (Anton Paar, Österreich) mit dem Messsystem Z1 DIN gemessen. Zur Kalibrierung der Messung wurde Wasser bei 35 °C verwendet. 51 Ergebnisse Das Kapillarviskosimeter liefert genauere Viskositätswerte als das Scherrheometer. Daher werden die Fließkurven der Medien nur zur Beurteilung des Fließverhaltens und nicht zur Bestimmung der absoluten Viskosität herangezogen. Die dynamische Viskosität bleibt bei steigender Scherrate annähernd konstant, daher kann für beide Medien newtonsches Fließverhalten angenommen werden. 7.1.2 Bestimmung der Scherkräfte Der Durchfluss durch die Küvette beträgt etwa 30 𝑚𝑚 𝑚𝑚𝑚−1 (siehe Kapitel 6.4.3). Nach (6.6) ergeben sich für das ABiTEP-¼-Medium (𝜈 = 0,805 𝑚𝑚2 𝑠 −1 ) und das StandardNährmedium-I-¼ (𝜈 = 0,785 𝑚𝑚2 𝑠 −1 ) die in Tabelle 5 gezeigten Strömungsverhältnisse. Tabelle 5: Strömungsverhältnisse im Messsystem Die Strömungsverhältnisse (querschnittgemittelte Strömungsgeschwindigkeit (um) und Reynoldszahl (Re)) im Messsystem mit Standard-Nährmedium-I-¼ und ABiTEP-Medium¼ bei 35 °C und einem Durchfluss von 30 ml min-1. StandardNährmedium-I-¼ ABiTEPMedium-¼ Rohr D = 2 mm Spalte 24 x 1 mm Spalte 36 x 1 mm Rohr D = 2 mm Spalte 24 x 1 mm Spalte 36 x 1 mm um [mm s-1] 159 20,8 13,9 159 20,8 13,9 Re 405,5 26,5 17,7 395,4 25,9 17,3 Damit liegen die Reynoldszahlen weit unter der kritischen Reynoldszahl von 2300 und die Gleichungen für laminare Strömungen sind gültig. Bei bekanntem Durchfluss (30 𝑚𝑚 𝑚𝑚𝑚−1 ) und bekannter Geometrie (Rohr 𝐷 = 2 𝑚𝑚 , Spalt 𝐻 = 1 𝑚𝑚, 𝐵 = 24 𝑚𝑚 und Spalt 𝐻 = 1 𝑚𝑚, 𝐵 = 36 𝑚𝑚) können mit Hilfe der Gleichungen (6.1) bzw. (6.2) die Geschwindigkeitsprofile im Rohr und in den Spalten berechnet werden. Unter Berücksichtigung der kinematischen Viskosität des ABiTEP-Mediums-¼ (𝜂 = 0,8028 𝑃𝑃 𝑠) und des Standard-Nährmediums-I-¼ (𝜂 = 0,7818 𝑃𝑃 𝑠) sowie der Gleichungen (6.3) und (6.4) sind die Schubspannungen berechenbar. In Abbildung 26 sind die Geschwindigkeiten und die Schubspannungen in Abhängigkeit vom Durchmesser bzw. von der Höhe dargestellt. Es ist jeweils nur der halbe Durchmesser bzw. Höhe dargestellt, die andere Hälfte verhält sich symmetrisch. Die höchsten Schubspannungen treten mit 500 Pa am Rand der Zu- und Ableitungen auf. Die Scherkräfte sind proportional zu den Schubspannungen. 52 Ergebnisse Abbildung 26: Geschwindigkeiten und Schubspannungen von Standard-Nährmedium-I¼ und ABiTEP-Medium-¼ in der Durchflussküvette Dargestellt sind die Geschwindigkeitsprofile für eine laminare Strömung bei einem Durchfluss von 30 ml min-1 in einem Rohr mit 2 mm Durchmesser (links oben), in einem Spalt mit 1 mm Höhe und 24 mm Breite (links Mitte) und einem Spalt mit 1 mm Höhe und 36 mm Breite (links unten). Das Geschwindigkeitsprofil bildet sich bei dem ABiTEPMedium-¼ (durchgezogene Linie) und Standard-Nährmedium-I-¼ (gestrichelte Linie) jeweils gleich aus. Weiterhin sind die Schubspannungen mit zwei unterschiedlichen Flüssigkeiten ABiTEP-Medium-¼ (η = 0,803 Pa s) und Standard-Nährmedium-I-¼ (η = 0,782 Pa s) für das Rohr (rechts oben), den 24 mm-Spalt (rechts mitte) und den 36 mmSpalt (rechts unten) berechnet. Die Strömungsverhältnisse im Referenzsystem zur Bestimmung der kritischen Schubspannungen (siehe Kapitel 6.4.2) sind in Tabelle 6 gezeigt. Zu jeder Kapillare wurden fünfzehn Proben vermessen. Der prozentuale Anteil an beweglichen Bakterien pro Bildstrecke in Abhängigkeit des Kapillarinnendurchmessers wurde ermittelt. 53 Ergebnisse Tabelle 6: Strömungsverhältnisse in den Messkapillaren bei Standard-Nährmedium-I-¼ Die wichtigsten Kennwerte zu den Strömungsverhältnissen mit Standard-Nährmedium-I-¼ bei 35 °C (η = 0,782 Pa s, 𝜈 = 0,785 𝑚𝑚2 𝑠 −1) in den Kapillaren im Messsystem zur Bestimmung der kritischen Schubspannungen von B. amyloliquefaciens FZB42. Kapillarinnendurchmesser [mm] 1,0 0,5 0,25 Reynoldszahl 108 216 432 Mittlere Geschwindigkeit um [mm s-1] 84,8 339,5 1.358 Maximale Geschwindigkeit umax [mm s-1] 169,8 679 2.716 Maximale Schubspannung 𝜏max [Pa] 525,5 4.161,8 33.294 Die Mittelwerte des Anteils der beweglichen Zellen betragen 28,46% ± 13,66% bei 0,25 mm Kapillarinnendurchmesser, 28,14% ± 11,34% bei 0,5 mm und 28,88% ± 12,70% bei 1 mm. In Tabelle 7 sind die Ergebnisse aus der statistischen Auswertung zusammengefasst. Tabelle 7: Statistische Auswertung des Anteils der beweglichen Bakterien von Bacillus amyloliquefaciens FZB42 nach Durchströmung der Messkapilaren Die Messreihen mit verschiedenen Kapillarinnendurchmessern wurden mit dem Statistikprogramm R auf Normalverteilung (Shapiro-Wilk-Test, Anderson-Darling-Test und Cramér-von-Mises-Test) geprüft. Weiterhin wurden die Mittelwerte (Zweistichprobent-Test) und Varianzen (F-Test) auf signifikante Unterschiede geprüft. In der ersten Spalte ist die Testmethode angegeben und in den folgenden Spalten die p-Werte für die einzelnen Datensätze. Liegt das Ergebnis aller drei Tests auf Normalverteilung über 0,05, können die Daten als normalverteilt angenommen werden. Liegt der Wert des Zweistichproben-t-Tests und des F-Tests über 0,05 sind signifikante Unterschiede in den Messreihen nicht nachweisbar. p-Wert D = 1 mm D = 0,5 mm D = 0,25 mm Shapiro-Wilk-Test 0,08746 0,6425 0,2445 Anderson-Darling-Test 0,2243 0,5279 0,1717 Cramér-von-Mises-Test 0,4548 0,4001 0,143 D = 1 mm vs D = 1 mm vs D = 0,5 mm vs D = 0,5 mm D = 0,25 mm D = 0,25 mm Zweistichproben-t-Test 0,9479 0,9337 0,8734 F-Test 0,4947 0,7863 0,6796 54 Ergebnisse 7.1.3 Auslegung der Optik Die reale Größe eines Pixels wurde mit ein Objektmikrometer (2 mm in 200 Skalenteilen, 10008.04.005, POG Präzisionsoptik Gera GmbH) unter Verwendung des Vision Assistant Version 2013 (National Instruments) bestimmt. Die Messung ergab, dass 10 µm der Skaleneinteilungen durchschnittlich 106,05 Pixel entsprechen, die Standardabweichung beträgt 1,00 Pixel. Da die Linienmittelpunkte vom Vision Assistant Version 2013 (National Instruments) mathematisch bestimmt werden, kommen bei den Distanzen Kommazahlen als Abstände zustande. Mit den gewählten Komponenten entspricht ein Pixel im digitalen Bild somit einem Quadrat von 94,3 nm Seitenlänge (± 0,9 nm) in der Objektebene. Bei einer Bildauflösung von 1280 x 960 Pixel wird eine Fläche von 120,7 μm x 90,5 μm in der Aufnahme abgebildet. Die numerische Apertur des eingesetzten Objektivs beträgt 0,65 und wird für die folgende Berechnung herangezogen. Als Wellenlänge wird grünes Licht bei 550 nm gewählt. Aus (6.16) ergibt sich eine theoretische Auflösung (𝑦𝑚𝑚𝑚, 𝑒 −2 ) von 579 nm. Daraus ergibt sich mit (6.13) und (6.11) eine Schärfentiefe (𝑧𝑅 ) von 239 nm. Die Dicke der Schärfeebene beträgt damit 478 nm, da der Bereich über und unter dem Fokuspunkt (±𝑧𝑅 ) betrachtet wird. Ein Quadrat mit der Seitenlänge von 94,3 nm in der Objektebene wird auf einen Pixel abgebildet. In Abbildung 27 ist die Pixel-Auflösung in Bezug auf die resultierende Intensitätsverteilung bei 550 nm dargestellt (vgl. Abbildung 20). Abbildung 27: Pixel-Auflösung in Bezug auf die Intensitätsverteilung Dargestellt ist die Pixel-Auflösung der Kamera in der Objektebene in Bezug auf die Intensitätsverteilung zweier Lichtpunkte bei 550 nm mit dem Abstand der definierten Auflösungsgrenze ymin,e-². Je dunkler die Pixel eingefärbt sind, desto höher ist die Beleuchtungsintensität auf diesem Bildpunkt. Bei der gewählten Konstellation liegen etwa sechs Pixel zwischen zwei Maxima mit dem Abstand der definierten Auflösungsgrenze 𝑦min, 𝑒 −2 . Der Kompromiss zwischen Kontrast und resultierende Größe des Bildausschnittes ist hiermit gut auf die theoretische Auflösungsgrenze angepasst. 55 Ergebnisse Aus (6.18) geht hervor, dass Bakterien mit Geschwindigkeiten bis zu 50 μm s-1, ohne sichtbaren Schärfeverlust mit einer maximalen Belichtungszeit von 0,942 ms zu visualisieren sind. Wird eine höhere Belichtungszeit gewählt, wird die Bewegungsunschärfe sichtbar. Je höher die Belichtungszeit ist, desto stärker ist die Bewegungsunschärfe. 7.1.4 Auslegung der Bildanalyse Die optimale Anzahl an Bakterien pro Bild für die Bildanalyse wurde mit Hilfe von (6.7), (6.8), (6.9) und (6.10) abgeschätzt. Folgende Werte wurden zur Berechnung herangezogen: - erwartete Standardabweichung (𝑆𝑆𝑣𝑣 ) = 25 µ𝑚 𝑠 −1 , Standardabweichung bei fehlerhaften Sprüngen (𝑆𝑆𝐹 ) = 66 µ𝑚 𝑠 −1 , Anzahl an Bildern pro Bildserie (𝑓𝑛 ) = 30, Anzahl an Bildserien pro Messpunkt (𝑏𝑛 ) = 10, Wahrscheinlichkeit für das Auftreten von kleinen Strecken (𝑝𝑘𝑘 ) = 1,153, Fläche eines Bakteriums (𝐴𝐵𝐵 ) = 884,53 𝑝𝑝𝑝𝑝𝑝, Fläche des Bildes (𝐴𝐵 ) = 1.228.800 𝑝𝑝𝑝𝑝𝑝, Wahrscheinlichkeit für einen fehlerhaften Sprung naher Bakterien (𝑝𝑒𝑒 ) = 0,33. Der erwartete Fehler der Durchschnittsgeschwindigkeit in Abhängigkeit der Anzahl an Bakterien pro Bild ist in Abbildung 28 aufgetragen. Abbildung 28: Standardfehler in Abhängigkeit der Anzahl an Bakterien pro Bild Die Abschätzung des erwarteten Standardfehlers der mittleren Geschwindigkeit ist über der Anzahl an Bakterien pro Bild aufgetragen. Der optimale Bereich liegt zwischen fünf und fünfzehn Bakterien pro Bild. Davor wirkt sich die statistische Streuung stark aus, danach häufen sich die Berührungen zwischen den Bakterien. Für die Bildanalyse ist eine Anzahl von fünf bis fünfzehn vitalen Bakterien pro Bild optimal. Bei mehr als fünfzehn Bakterien pro Bild neigt die automatische Bewegungsverfolgung zu 56 Ergebnisse Fehlern und das Ergebnis wird ungenauer. Bei weniger als fünf Bakterien pro Bild wird durch die starke Streuung die statistische Betrachtung ungenauer. Dies entspricht einer Dichte an beweglichen Zellen von 1,45 1011 𝑙 −1 bis 4,35 1011 𝑙 −1. Die Ergebnisse der verschiedenen Grauwertwandlungen sind in Abbildung 29 dargestellt. Abbildung 29: Vergleich der Grauwertwandlungen bei einer Mikroskopaufnahme von Bacillus amyloliquefaciens FZB42 Vergleich der Grauwertwandlungen mit Beispielbild und zugehörigem Linienhistogramm längs durch das Bakterium (B. amyloliquefaciens FZB42). Die Grauwertwandlung wurde anhand der Luminance (Farbhelligkeit eines Pixels im HSLFarbraum) durchgeführt. Hierbei wurden über mehrere Bildstrecken die besten Ergebnisse erzielt. Im Anhang (S. 117) sind die Ergebnisse der Grauwertwandlung noch einmal detaillierter dargestellt. 7.1.5 Integration des Gesamtsystems Die Durchflussküvette mit flexibler Schichtdicke wurde wie geplant gefertigt (siehe Kapitel 6.4.1) und konnte erfolgreich auf dem Mikroskop getestet werden. Die entwickelte Durchflussküvette ist in Abbildung 30 zu sehen. 57 Ergebnisse Abbildung 30: Entwickelte Durchflussküvette Die entwickelte Durchflussküvette mit flexibler Schichtdicke einzeln (links) und auf dem Mikroskop (rechts). Das Gesamtsystem mit Bioreaktor, Probenentnahme, Probenaufbereitung, Mikroskop und Steuerungssoftware ist in Abbildung 31 dargestellt. Abbildung 31: Gesamtsystem für die Beispielfermentationen Das Gesamtsystem zur Durchführung der Beispielfermentationen mit den Modulen: Bioreaktor, Probenentnahme, Probenaufbereitung, Mikroskop, Durchflussküvette und Steuerungssoftware. Das System wurde speziell für die Anwendung angepasst und optimiert, das gesamte System ist durch die entwickelte Steuerungssoftware regelbar. Um die vollständige Fernsteuerung und Datenerfassung zu ermöglichen, musste ein neuer Schaltschrank geplant und ausgestattet 58 Ergebnisse werden. Mit dem Gesamtsystem war die Durchführung der geplanten Beispielfermentationen möglich. 7.2 Ergebnisse aus den Beispielfermentationen Durch die automatisierte Bildanalyse können Geschwindigkeit und Bewegungsmuster der Bakterien erfasst werden. Neben der Durchschnittsgeschwindigkeit der Bakterien wird die Anzahl der beweglichen Bakterien pro Bild ermittelt. Ebenso wurden Bewegungsmuster der einzelnen Bakterien charakterisiert. 7.2.1 Validierung der automatischen Bildanalyse Für die Validierung der automatischen Bildanalyse in Bezug auf die Bewegungsgeschwindigkeit wurden die Bildserien der vier charakteristischen Fermentationszeitpunkte (Exponentielles Wachstum durch Kohlenhydratverbrauch und Säurebildung (1), Diauxiestufe, Umstellung von Kohlenhydrat- auf Säureverstoffwechselung (2), Wachstum durch Säureverbrauch (3) und Sporulation (4)) von Fermentation 1 herangezogen. Hierzu wurden 7.922 Einzelgeschwindigkeiten in 930 Bildern manuell ermittelt und die Ergebnisse mit den Werten aus der automatischen Bildauswertung verglichen. Die Durchschnittsgeschwindigkeit mit Standardfehler aus automatischer und manueller Auswertung ist in Abbildung 32 zu sehen. Abbildung 32: Validierung der automatischen Bildauswertung bei Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ Die Bestimmung der Bewegungsgeschwindigkeiten aus der automatischen Bildauswertung wurde an den vier charakteristischen Fermentationszeitpunkten (Exponentielles Wachstum durch Kohlenhydratverbrauch und Säurebildung (232 min), Diauxiestufe, Umstellung von Kohlenhydrat- auf Säureverstoffwechselung (292 min), Wachstum durch Säureverbrauch (352 min) und Sporulation (382 min)) von Fermentation 1 (ABiTEP-Medium-¼, B. amyloliquefaciens FZB42) durch manuelle Messung validiert. Die Mittelwerte der Einzelgeschwindigkeiten mit Standardfehler sind für die automatische Auswertung und für die manuelle Auswertung aufgetragen. Insgesamt wurden 930 Bilder mit 7.922 Einzelgeschwindigkeiten manuell ausgewertet. 59 Ergebnisse Die manuelle Messung wurde mit dem Programm zur interaktiven Erfassung der Bewegungsmuster durchgeführt (siehe Kapitel 6.6). Die Geschwindigkeits-Mittelwerte an allen vier Messpunkten der automatischen und der manuellen Auswertung liegen im Bereich von 45 – 52 µm s-1. An den vier Messpunkten liegt die Differenz zwischen manueller Messung und automatischer Messung im Bereich von 0,1 - 0,55 µm s-1. Wie in Abbildung 32 gezeigt, gibt es an jedem Messpunkt eine große Überschneidung der Fehlerindikatoren des Standardfehlers. Der Vertrauensbereich der manuellen Auswertung überschneidet sich komplett mit dem der automatischen Auswertung. Der Standardfehler der manuellen Auswertung liegt zwischen ± 0,28 und ± 0,83 µm s-1, die automatische Bildserienauswertung erreicht Standardfehler von ± 0,37 bis ± 1,34 µm s-1. 7.2.2 Geschwindigkeit und Anzahl beweglicher Bakterien In den Fermentationen 1 – 3 waren bezüglich Durchschnittsgeschwindigkeit und Anzahl der beweglichen Bakterien jeweils charakteristische Verläufe zu beobachten. Die Anzahl der beweglichen Bakterien nimmt mit fortschreitender Fermentationsdauer zu bis zu einem Abfall bei Beginn der Sporulation. Ähnliches ist bei der optischen Dichte (OD600) zu beobachten, jedoch nicht so spezifisch, da zu Beginn der Feststoffanteil des Mediums und später die Sporen die Absorption des Lichtes beeinflussen. Die Durchschnittsgeschwindigkeit der Bakterien in den Fermentationen 1 – 3 bewegt sich über den gesamten Fermentationsverlauf im Bereich von 20 – 60 µm s-1. Im Zeitraum zwischen den charakteristischen Fermentationszeitpunkten vor der Diauxiestufe bis zum Beginn der Sporulation liegt die Durchschnittsgeschwindigkeit in einem Bereich von 40 – 60 µm s-1. An den vier charakteristischen Fermentationszeitpunkten ist ein charakteristischer Verlauf zu erkennen. Während der Diauxiestufe ist die Durchschnittsgeschwindigkeit geringer als davor und danach und mit Beginn der Sporulation fällt die Durchschnittsgeschwindigkeit ab. In Abbildung 33 ist der Fermentationsverlauf von Fermentation 3 als Beispiel dargestellt. Es ist zu sehen, dass mit zunehmender Zelldichte die Gelöstsauerstoffsättigung (pO2) abnimmt. Die Belüftung (100 mln min-1) und die Rührerdrehzahl (225 rpm) sind hierbei jeweils auf ihrem Minimalwert. Nach ca. 250 min Fermentationsdauer erreicht die pO2-Sättigung 50%. Ab diesem Punkt greift die Regelung des pO2 auf einen Sollwert von 50%, Belüftung und Rührerdrehzahl werden über ihren Minimalwert erhöht und der pO2 bei 50% gehalten. 60 Ergebnisse Abbildung 33: Kultivierung von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium¼ bei 35 °C Daten aus Fermentation 3: 180 min bis 480 min. A) Verlauf der Gelöstsauerstoffsättigung sowie der Belüftung und Rührerdrehzahl. B) pH-Wert Kurve und Durchschnittsgeschwindigkeiten der beweglichen Bakterien mit Standardfehler (SEM). Als zusätzliche Angabe ist die Anzahl der ermittelten Einzelgeschwindigkeiten an jedem Messpunkt zu sehen. Zur Ermittlung der Durchschnittsgeschwindigkeit wurden insgesamt 18.484 Einzelgeschwindigkeiten herangezogen. C) Optischen Dichte bei 600 nm (OD600) und Anzahl der beweglichen Bakterien pro Bild mit Standardabweichung (SD). Zu diesem Zeitpunkt steigen auch die Anzahl der beweglichen Bakterien sowie die optische Dichte (OD600). Die Durchschnittsgeschwindigkeit der Bakterien liegt bei etwa 58 µm s-1. Der pH-Wert fällt über diesen Zeitraum immer weiter ab. Bei ca. 315 min ist der minimale pHWert (ca. 6,8) erreicht. Hier ist der Zeitpunkt der Diauxiestufe. Danach steigt der pH-Wert wieder an. Zum Zeitpunkt der Diauxiestufe ist ein lokales Minimum der Durchschnittsgeschwindigkeit der Bakterien zu sehen (ca. 50 µm s-1). Bei etwa 400 min ist die höchste Zelldichte erreicht. Die Anzahl an beweglichen Bakterien pro Bild (etwa 17) und die OD600 haben hier ihren jeweiligen Maximalwert. Kurz vor diesem Punkt zeigt die Durchschnittsgeschwindigkeit der Bakterien ein lokales Maximum (ca. 52 µm s-1). Nach Erreichen der maximalen Zelldichte werden die Bakterien immer unbeweglicher. Die Anzahl der beweglichen Bakterien und die OD600 sowie auch die Durchschnittsgeschwindigkeit nehmen ab. Erste Sporen sind bei manueller Sichtung der Aufnahmen zu sehen. Sowohl Rührerdrehzahl als auch Belüftung steigen nicht weiter an bzw. fallen minimal ab. Für die Fermentationen 1 – 3 sind die Ergebnisse der vier charakteristischen Fermentationszeitpunkte in Tabelle 8 zusammengefasst. 61 Ergebnisse Tabelle 8: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ Übersicht über die Durchschnittsgeschwindigkeit (vØ) von B. amyloliquefaciens FZB42 mit Standardfehler (SEM), die Anzahl an beweglichen Bakterien pro Bild (nmB) und den pHWert in den vier charakteristischen Fermentationsphasen aus den Fermentationen 1 – 3. Die jeweils dazugehörigen Fermentationszeitpunkte (tp) sind in Minuten angegeben Exponentielles Wachstum; Kohlenhydratverbrauch und Säurebildung Diauxiestufe; Kohlenhydrat- auf Säureverbrauch (min. pH) Wachstum durch Säureverbrauch Sporulation Ferm. 1 Ferm. 2 Ferm. 3 tp [min] vØ ± SEM [µm s-1] nmB 232 304 305 50,74 ± 1,34 51,03 ± 2,11 58,04 ± 2,92 1,07 2,43 3,31 pH-Wert 6,85 6,78 6,79 tp [min] vØ ± SEM [µm s-1] nmB 292 338 335 47,71 ± 0,64 46,17 ± 0,82 50,17 ± 1,01 5,95 2,9 7,53 pH-Wert 6,68 6,77 6,79 tp [min] vØ ± SEM [µm s-1] nmB 352 360 365 51,88 ± 0,37 59,56 ± 0,39 52,39 ± 1,26 13,69 14,57 8,8 pH-Wert 6,75 6,80 6,81 tp [min] vØ ± SEM [µm s-1] nmB 382 391 425 46,16 ± 0,44 44,95 ± 0,38 40,76 ± 0,41 9,41 13,62 10,43 pH-Wert 6,82 6,84 6,85 Mit der Umstellung von Kohlenhydrat- auf Säureverbrauch geht ein globales Minimum des pH-Werts einher. Die steigende Anzahl an beweglichen Bakterien pro Bild ist ebenso in jeder der drei Fermentationen aufgezeigt. Zur besseren Visualisierung sind die Durchschnittsgeschwindigkeiten und pH-Werte aus Tabelle 8 in Abbildung 34 grafisch über die vier charakteristischen Fermentationszeitpunkte aufgetragen. 62 Ergebnisse Abbildung 34: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ Übersicht über die Durchschnittsgeschwindigkeit (vØ) von B. amyloliquefaciens FZB42 und den pH-Wert an den vier charakteristischen Fermentationszeitpunkten (Exponentielles Wachstum durch Kohlenhydratverbrauch und Säurebildung (I), Diauxiestufe; Kohlenhydrat- auf Säureverstoffwechselung (II), Wachstum durch Säureverbrauch (III) und Sporulation (IV)) aus den Fermentationen 1 – 3. Im Minimum des pH-Werts ist jeweils ein lokales Minimum der Durchschnittsgeschwindigkeit zu beobachten. Die Durchschnittsgeschwindigkeiten sind als durchgehende Linie mit schräggestellten Quadraten und der pH-Wert als gestrichelte Linie mit Dreiecken dargestellt. Die OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien für die Fermentationen 1 – 3 mit ABiTEP-Medium-¼ ist in Abbildung 35 dargestellt. Durch den Feststoffanteil im ABiTEP-Medium-¼ beginnt die OD600 bereits mit einem Wert über 1 und zeigt im Verlauf der Fermentation nur eine Änderung von 0,2 bis 0,3. Mit steigender OD600 ist in den drei Fermentationen ein Anstieg in der Anzahl der beweglichen Bakterien zu beobachten, bis die Sporulation einsetzt. Mit Einsetzen der Sporulation nimmt die Anzahl der beweglichen Bakterien ab, die OD600 bleibt jedoch auf hohem Niveau. 63 Ergebnisse Abbildung 35: OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien (Bacillus amyloliquefaciens FZB42) pro Bild in ABiTEP-Medium-¼ Die OD600 ist aufgetragen in Abhängigkeit der ermittelten Anzahl an beweglichen Bakterien pro Bild für die Fermentationen 1 – 3 (B. amyloliquefaciens FZB42) mit dem ABiTEP-Medium-¼. Alle Messpunkte vom Start der Fermentation bis zum Beginn der Sporulation werden verwendet. Das Bestimmtheitsmaß (R2) und die lineare Näherung sind im Diagramm angegeben. Daten nach Beginn der Sporulation wurden nicht berücksichtigt. Für die Fermentationen 1 – 3 werde die Einzelgeschwindigkeitsverteilung zur Diauxiestufe jeweils den Einzelgeschwindigkeitsverteilungen der drei anderen charakteristischen Fermentationszeitpunkte gegenübergestellt. Die Überprüfung erfolgt durch den WilcoxonRangsummentest. Die Ergebnisse sind in Tabelle 9 aufgetragen. Tabelle 9: Vergleich der Geschwindigkeitsverteilungen für Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ Die Einzelgeschwindigkeitsverteilung bei der Diauxiestufe (Kohlenhydrat- auf Säureverstoffwechselung) wird mit den Einzelgeschwindigkeitsverteilungen der drei anderen charakteristischen Fermentationszeitpunkte verglichen. Dies wird mit dem Wilcoxon-Rangsummentest für die Fermentationen 1 – 3 (B. amyloliquefaciens FZB42, ABiTEP-Medium-¼) durchgeführt. Es soll bewiesen werden, dass die beiden verglichenen Verteilungen signifikant unterschiedlich sind. Bei p-Werten unter 0,05 ist die Aussage mit hoher Wahrscheinlichkeit (über 95%) richtig. Vergleich der Geschwindigkeitsverteilungen von zwei definierten Fermentationszeitpunkten Diauxiestufe gegen exponentielles Wachstum durch Kohlenhydratverbrauch und Säurebildung Diauxiestufe gegen Wachstum durch Säureverbrauch Diauxiestufe gegen Beginn der Sporulation p-Werte Ferm. 1 Ferm. 2 Ferm. 3 0,1120 * < 0,0001 < 0,0001 < 0,0001 < 0,0001 0,0068 < 0,0001 < 0,0001 < 0,0001 * p-Wert > 0,05; signifikanter Unterschied konnte nicht bewiesen werden 64 Ergebnisse In acht von neun Vergleichen ergibt der Wilcoxon-Rangsummentest p-Werte unter 0,05. Damit existiert mit hoher Wahrscheinlichkeit (über 95%) ein signifikanter Unterschied zwischen den ermittelten Einzelgeschwindigkeitsverteilungen. Lediglich in Fermentation 1 ergibt die Gegenüberstellung der Messdaten der Diauxiestufe zu den Daten der exponentiellen Wachstumsphase (Kohlenhydratverbrauch und Säurebildung) einen p-Wert von 0,1120. Die detaillierten Ergebnisse der statistischen Auswertung von Fermentation 1 – 3 sind im Anhang (Seite 110) beigefügt. Die Verteilungen der Einzelgeschwindigkeiten an den vier charakteristischen Fermentationszeitpunkten von Fermentation 1 sind in Abbildung 36 dargestellt. Abbildung 36: Histogramme der Einzelgeschwindigkeiten von Bacillus amyloliquefaciens FZB42 aus Fermentation 1 mit ABiTEP-Medium-¼ Die Histogramme zeigen die Geschwindigkeitsverteilung der Einzelgeschwindigkeiten an den vier charakteristischen Fermentationszeitpunkten von Fermentation 1. Exponentieller Wachstum durch Kohlenhydratverbrauch und Säurebildung (links oben), Diauxiestufe; Kohlenhydrat- auf Säureverstoffwechselung (rechts oben), Wachstum durch Säureverbrauch (links unten) und Sporulation (rechts unten). Die Daten wurden mit der automatischen Bildanalyse ermittelt. Die Einzelgeschwindigkeiten liegen, mit Ausnahme weniger Ausreißer, in einem Bereich von 5 – 100 µm s-1. Die Balken zeigen die absolute Häufigkeit der Geschwindigkeitsbereiche und die Linie kennzeichnet die kumulierte Häufigkeit in %. 65 Ergebnisse Die Verteilungen sind unimodal (ein Gipfel) mit einem positiven Exzess (steilgipflig). Mit Ausnahme weniger Ausreißer bewegen sich die Einzelgeschwindigkeiten in einem Bereich von 5 – 100 µm s-1. Die Fermentationen 4 – 6 mit Standard-Nährmedium-I-¼ zeigen ebenso bezüglich Durchschnittsgeschwindigkeit und Anzahl der beweglichen Bakterien charakteristische Verläufe. Die Durchschnittsgeschwindigkeit der Bakterien nimmt zu, während sich die Kultur in der exponentiellen Wachstumsphase befindet. Dies geht einher mit einem Anstieg des OD600 sowie mit steigendem Sauerstoffbedarf der Kultur, was an steigender Zuluftrate und Rührerdrehzahl zu erkennen ist. In dieser Fermentationsphase ist ein Abfallen des pH-Werts (von ca. 7,2 auf ca. 6,1) zu erkennen. Im Minimum des pH-Werts ist ein Einbruch der Durchschnittsgeschwindigkeit wie auch im Sauerstoffbedarf (Rührerdrehzahl und Zuluftrate bricht ein) zu beobachten. Die Steigung des OD600–Verlaufs wird flacher. Dieser Zeitpunkt ist als Diauxiestufe I (Umstellung von Glukose- auf Säureverstoffwechselung mit Stickstoffverbrauch) definiert. Hier können in allen drei Fermentationen ähnliche charakteristische Verläufe gezeigt werden. Danach steigt der pH-Wert langsam an, der Sauerstoffbedarf nimmt nach dem Einbruch ebenso wieder zu. Die Durchschnittsgeschwindigkeit der Bakterien nimmt in dieser Phase ebenso zu. Dieser charakteristische Fermentationszeitpunkt (Diauxiestufe II; Umstellung von Stickstoffverbrauch auf reine Säureverstoffwechselung) ist an einem Knick in der Steigung des pH-Wertverlaufs deutlich zu erkennen. Der zunächst langsame Anstieg des pH-Werts nimmt nun rasant zu. An dieser Stelle ist ein leichter Abfall des Sauerstoffbedarfs zu beobachten, die Durchschnittsgeschwindigkeit bricht hier, wie bei der Diauxiestufe I, ein. Nach der Diauxiestufe II steigt der pH-Wert rasant an, der Sauerstoffbedarf steigt nochmals ein wenig und der OD600 erreicht seinen Höchstwert. Die Durchschnittsgeschwindigkeit steigt nochmals etwas an. Nach Erreichen des Maximums der OD600 sinken die Durchschnittsgeschwindigkeit und der Sauerstoffbedarf. Die Sporulation der Kultur setzt ein. Der Anstieg des pH-Werts verlangsamt sich mit einsetzender Sporulation. In Abbildung 37 ist der Fermentationsverlauf von Fermentation 6 dargestellt. 66 Ergebnisse Abbildung 37: Kultivierung von Bacillus amyloliquefaciens FZB42 in StandardNährmedium-I-¼ Daten aus Fermentation 6: 60 min bis 330 min. A) Verlauf der Gelöstsauerstoffsättigung sowie der Belüftung und Rührerdrehzahl. B) pH-Wert und die Durchschnittsgeschwindigkeiten der beweglichen Bakterien mit Standardfehler (SEM). Der Ermittlung der Durchschnittsgeschwindigkeiten liegen insgesamt 7.493 Einzelgeschwindigkeiten zu Grunde. C) Optischen Dichte bei 600 nm (OD600) und der Anzahl beweglichen Bakterien pro Bild. Gut zu sehen ist der Einbruch der Durchschnittsgeschwindigkeiten zum Zeitpunkt der beiden Diauxiestufen (Umstellung von Glukose- auf Säureverbrauch mit Stickstoffverbrauch (pH-Minimum) sowie die Umstellung auf reinen Säureverbrauch (rasanter Anstieg des pH-Werts)). Eine Zusammenfassung der charakteristischen Fermentationszeitpunkte ist in Tabelle 10 zu sehen. Bei Fermentation 4 und 5 wurde in der Endphase Antischaummittel zugesetzt. Die Verläufe nach Zusatz des Antischaummittels zeigen Unterschiede zu den charakteristischen Verläufen. In Fermentation 4 stimmt der Zeitpunkt der Diauxiestufe I nicht mit dem Zeitpunkt einer Probenentnahme überein. Die betroffenen Messungen sind in Tabelle 10 markiert. 67 Ergebnisse Tabelle 10: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in Standard-Nährmedium-I-¼ Übersicht über die Durchschnittsgeschwindigkeit (vØ) mit Standardfehler (SEM), die Anzahl an beweglichen Bakterien pro Bild (nmB) und den pH-Wert in den sechs charakteristischen Fermentationszeitpunkten aus den Fermentationen 4 – 6. Die jeweils dazugehörigen Fermentationszeitpunkte (tp) sind in Minuten angegeben. Messungen, die nach der ersten Zugabe von Antischaummittel durchgeführt wurden, sind markiert. Bei Fermentation 4 lag die Diauxiestufe I (pH-Minimum) zwischen zwei Probenentnahmen. Daher wird dieser Fermentationszeitpunkt nicht gut in der Tabelle repräsentiert. Die betroffene Stelle ist in der Tabelle markiert. Ferm. 4 Ferm. 5 Ferm. 6 142 165 165 46,03 ± 2,82 58,32 ± 2,89 53,39 ± 1,98 nmB 0,4 0,27 0,92 Exponentielles Wachstum durch Glukose- und Stickstoffverbrauch und Säurebildung tp [min] pH-Wert 6,62 6,34 6,22 Diauxiestufe I; Glukose- auf Stickstoff- und Säureverbrauch (pH-Minimum) tp [min] 162 180 180 42,95 ± 2 50,16 ± 1,62 39,26 ± 1,32 0,97 3,65 6,02 6,09 Exponentielles Wachstum durch Stickstoff- und Säureverbrauch Diauxiestufe II; Stickstoff- auf reinen Säureverbrauch (pH-Knick) vØ ± SEM [µm s-1] vØ ± SEM [µm s-1] 1,4 nmB pH-Wert 6,21 tp [min] 185 210 195 46,06 ± 0,69 54,57 ± 1,11 55,1 ± 1,25 nmB 3,12 1,05 2,8 pH-Wert 6,08 6,13 6,13 205 225 210 39,28 ± 0,64 47,63 ± 0,67 44,1 ± 0,88 nmB 8,11 3,24 2,64 pH-Wert 6,17 6,3 6,19 245 255 225 46,19 ± 1,02 45,06 ± 0,62 47,44 ± 0,65 nmB 15,77 3,86 5,09 pH-Wert 6,76 6,76 325 315 305 38,37 ± 1,55 36,91 ± 1,54 36,93 ± 0,95 4,23 1,64 0,83 vØ ± SEM [µm s-1] tp [min] -1 vØ ± SEM [µm s ] tp [min] Wachstum durch Säureverbrauch -1 vØ ± SEM [µm s ] tp [min] Beginn der Sporulation *1 -1 vØ ± SEM [µm s ] nmB *2 6,38 7,27 *2 7,13 *2 7,05 *1 Diauxiestufe I (pH-Minimum) liegt zwischen zwei Probenentnahmen. Das tatsächliche pH-Minimum bei Fermentation 4 beträgt pH 6,03 in der 171. Minute. *2 Messungen nach Zugabe von Antischaummittel pH-Wert 68 Ergebnisse Alle drei Fermentationen zeigen die beschriebenen charakteristischen Verläufe. Diese sind für den pH-Wert und die Durchschnittsgeschwindigkeit in Abbildung 38 dargestellt. Der Zeitpunkt der Zugabe von Antischaummittel bei Fermentation 4 und 5 ist in Abbildung 38 markiert, da bei Zugabe des Antischaummittels schlagartige Einbrüche des pO2-Werts und auch Einbrüche der Durchschnittsgeschwindigkeit zu beobachten sind. Abbildung 38: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 mit Standard-Nährmedium-I-¼ Übersicht über die Durchschnittsgeschwindigkeit (vØ) von B. amyloliquefaciens FZB42 und den pH-Wert an den sechs charakteristischen Fermentationszeitpunkten aus den Fermentationen 4 – 6. Im Minimum des pH-Werts ist jeweils ein lokales Minimum der Durchschnittsgeschwindigkeit zu beobachten. An der Stelle des pH-Knicks (Steigung des pH-Werts ändert sich erheblich) ist ein zweites lokales Minimum der Durchschnittsgeschwindigkeit zu sehen. Bei Fermentation 4 lag die Diauxiestufe I (pHMinimum) zwischen zwei Probenentnahmen. Daher ist hier ein zusätzlicher Messwert im pH-Wert angegeben (nicht ausgefülltes Dreieck). Bei den Fermentationen 4 und 5 ist zusätzlich der Bereich ab Zugabe von Antischaummittel markiert. Die Durchschnittsgeschwindigkeiten sind als durchgehende Linie mit schräggestellten Quadraten und der pH-Wert als gestrichelte Linie mit Dreiecken dargestellt. 69 Ergebnisse Das Minimum des pH-Werts wurde bei Fermentation 4 als zusätzlicher Messpunkt hinzugefügt, da der Zeitpunkt der Diauxiestufe I nicht gut von einer Probenentnahme erfasst wurde. Die OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien für die Fermentationen 4 – 6 mit Standard-Nährmedium-I-¼ ist in Abbildung 39 dargestellt. Abbildung 39: OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien (Bacillus amyloliquefaciens FZB42) in Standard-Nährmedium-I-¼ Die OD600 ist aufgetragen in Abhängigkeit der ermittelten Anzahl an beweglichen Bakterien (B. amyloliquefaciens FZB42) pro Bild für die Fermentationen 4 – 6 mit dem Standard-Nährmedium-I-¼. Alle Messpunkte vom Start der Fermentation bis zum Beginn der Sporulation werden verwendet. Das Bestimmtheitsmaß (R2) und die lineare Näherung sind im Diagramm angegeben. Daten nach Beginn der Sporulation sind nicht berücksichtigt. Die OD600 beginnt mit einem Wert von ca. 0,4 und zeigt im Verlauf der Fermentation eine Änderung von 0,4 bis 0,6. Mit steigender OD600 ist in den drei Fermentationen ein Anstieg in der Anzahl der beweglichen Bakterien zu beobachten. Mit Einsetzen der Sporulation nimmt die Anzahl der beweglichen Bakterien ab, die OD600 bleibt jedoch auf hohem Niveau. Für die Fermentationen 4 – 6 werden die Einzelgeschwindigkeitsverteilungen zu den Diauxiestufen jeweils den Einzelgeschwindigkeitsverteilungen der fünf anderen charakteristischen Fermentationszeitpunkte gegenübergestellt. Die Überprüfung erfolgt durch den Wilcoxon-Rangsummentest. Die Ergebnisse sind in Tabelle 11 aufgetragen. In 23 von 27 Vergleichen ergibt der Wilcoxon-Rangsummentest p-Werte unter 0,05. Die pWerte der Gegenüberstellung zwischen den Diauxiestufen, sowie zwischen der Diauxiestufen und der Sporulation sind in einigen Fällen erhöht. Erhöhte p-Werte zeigen Ähnlichkeiten in den Einzelgeschwindigkeitsverteilungen. Die detaillierten Ergebnisse der statistischen Auswertung der Fermentationen 4 – 6 sind im Anhang (S. 113) beigefügt. 70 Ergebnisse Tabelle 11: Vergleich der Geschwindigkeitsverteilungen von Bacillus amyloliquefaciens FZB42 in Standard-Nährmedium-I-¼ Die Einzelgeschwindigkeitsverteilung bei beiden Diauxiestufen (Umstellung Glukose- auf Säureverstoffwechselung und Stickstoffverbrauch (I) und Umstellung Stickstoff- auf reinen Säureverbrauch(II)) wird je mit den Einzelgeschwindigkeitsverteilungen der fünf anderen charakteristischen Fermentationszeitpunkten verglichen. Dies wird mit dem WilcoxonRangsummentest für die Fermentationen 4 – 6 (B. amyloliquefaciens FZB42, StandardNährmedium-I-¼) durchgeführt. Es soll bewiesen werden, dass die beiden verglichenen Verteilungen signifikant unterschiedlich sind. Bei p-Werten unter 0,05 ist die Aussage mit hoher Wahrscheinlichkeit (über 95%) richtig. Erhöhte p-Werte zeigen Ähnlichkeiten in den Einzelgeschwindigkeitsverteilungen. Vergleich der Geschwindigkeitsverteilungen von zwei definierten Fermentationszeitpunkten Diauxiestufe I gegen exponentielles Wachstum durch Glukose- und Stickstoffverbrauch und Säurebildung Diauxiestufe I gegen exponentielles Wachstum durch Stickstoffverbrauch und Säureverbrauch Diauxiestufe I gegen Diauxiestufe II Diauxiestufe I gegen Wachstum durch Säureverbrauch Diauxiestufe I gegen Beginn der Sporulation Diauxiestufe II gegen exponentielles Wachstum durch Glukose- und Stickstoffverbrauch und Säurebildung p-Werte Ferm. 4 Ferm. 5 Ferm. 6 0,0028 0,0002 < 0,0001 < 0,0001 < 0,0001 < 0,0001 0,0524 * 0,3676 * < 0,0001 0,0004 0,0008 < 0,0001 0,5754 * < 0,0001 0,0921 * 0,0168 < 0,0001 < 0,0001 Diauxiestufe II gegen Diauxiestufe I 0,0524 * 0,3676 * < 0,0001 Diauxiestufe II gegen exponentielles Wachstum durch Stickstoffverbrauch und < 0,0001 < 0,0001 < 0,0001 Säureverbrauch Diauxiestufe I gegen Wachstum durch 0,0290 0,0002 < 0,0001 Säureverbrauch Diauxiestufe I gegen Beginn der 0,0138 < 0,0001 < 0,0001 Sporulation * p-Wert > 0,05; signifikanter Unterschied kann nicht bewiesen werden Die Histogramme der Geschwindigkeitsverteilung an den sechs charakteristischen Fermentationszeitpunkten aus Fermentation 6 sind in Abbildung 40 dargestellt. Die Verteilungen für Fermentation 6 sind in Näherung ebenso unimodal (ein Gipfel) wie die Verteilungen von Fermentation 3. Zur Kurtosis und Schiefe kann keine allgemeingültige Aussage getroffen werden, da diese zwischen den einzelnen Histogrammen variieren. Mit Ausnahme weniger Ausreißer bewegen sich die Einzelgeschwindigkeiten in einem Bereich von 5 – 100 µm s-1. 71 Ergebnisse Abbildung 40: Histogramme der Einzelgeschwindigkeiten von Bacillus amyloliquefaciens FZB42 aus Fermentation 6 mit Standard-Nährmedium-I-¼ Die Histogramme zeigen die Geschwindigkeitsverteilung der Einzelgeschwindigkeiten an den sechs charakteristischen Fermentationszeitpunkten von Fermentation 6: Exponentielles Wachstum durch Glukose- und Stickstoffverbrauch und Säurebildung (links oben), Diauxiestufe I; Glukose- auf Säureverstoffwechselung und Stickstoffverbrauch (rechts oben), exponentielles Wachstum durch Stickstoff- und Säureverbrauch (links Mitte), Diauxiestufe II; Stickstoff- auf reine Säureverstoffwechselung (rechts Mitte), Wachstum durch Säureverbrauch (links unten), Beginn der Sporulation (rechts unten). Die Daten wurden mit der automatischen Bildanalyse ermittelt. Die Einzelgeschwindigkeiten liegen mit Ausnahme weniger Ausreißer in einem Bereich von 5 – 100 µm s-1. Die Balken zeigen die absolute Häufigkeit der Geschwindigkeitsbereiche und die Linie kennzeichnet die kumulierte Häufigkeit in %. 72 Ergebnisse Die Fermentationen 1 – 3 und Fermentationen 4 – 6 zeigen einen grundsätzlich ähnlichen Verlauf. Im Standard-Nährmedium-I-¼ der Fermentationen 4 – 6 kann neben dem pHMinimum jedoch ein weiterer Knick in der Steigung des pH-Wert-Verlaufs beobachtet werden. Die Geschwindigkeiten bewegen sich über die Fermentationen etwa in derselben Größenordnung. In Abbildung 41 ist ein Vergleich der Durchschnittsgeschwindigkeiten über den gesamten Fermentationsverlauf mit zugehöriger Standardabweichung angegeben. Abbildung 41: Vergleich der Geschwindigkeiten von Bacillus amyloliquefaciens FZB42 im ABiTEP-Medium-¼ und Standard-Nährmedium-I-¼ Die Fermentationen 1 – 3 werden mit ABiTEP-Medium-¼ und die Fermentationen 4 – 6 mit Standard-Nährmedium-I-¼ durchgeführt. Die Durchschnittsgeschwindigkeiten (vØ) werden jeweils über alle Einzelgeschwindigkeiten im Zeitraum zwischen dem exponentiellen Wachstum mit Säurebildung und dem Beginn der Sporulation gebildet. Die Standardabweichungen der Durchschnittsgeschwindigkeit sind als Fehlerbalken angegeben. Die Durchschnittsgeschwindigkeiten werden aus den Einzelgeschwindigkeiten von jeweils drei Fermentationen (Ferm. 1 – 3 und 4 – 6) gebildet. Es wird jedoch nur der Zeitraum zwischen dem exponentiellen Wachstum mit Säurebildung bis zum Beginn der Sporulation berücksichtigt. Für die Fermentationen 1 – 3 werden 42.268 Einzelgeschwindigkeiten und für die Fermentationen 4 – 6 werden 19.273 Einzelgeschwindigkeiten berücksichtigt. Der Durchschnittswert liegt bei den Fermentationen 1 – 3 bei 48,5 µm s-1 und bei den Fermentationen 4 – 6 bei 44 µm s-1, die Standardabweichung ist bei beiden ähnlich und beträgt ± 28 bei den Fermentationen 1 – 3 und ± 29,5 bei den Fermentationen 4 – 6. 7.2.3 Bewegungsmuster der beweglichen Bakterien In Anlehnung an die Definitionen verschiedener 3D-Bewegungsmuster aus der Arbeit von Vater (2014) werden acht Kategorien zur Einteilung der Bewegungsmuster festgelegt: Richtungsänderung ohne Stoppphasen (Orientation), wellenförmige Bewegungen (Undulation), gerade Strecken (Straight path), Richtungsänderung mit längeren Schwimmbewegungen und kurzen Taumelphasen (Tumble), Richtungswechsel mit kurzen Schwimmbewegungen und längeren Stoppphasen ohne Eigenbewegung (Orientation and 73 Ergebnisse Stop), bogenförmige Strecken (Curve), Richtungsänderung aufgrund von Zusammenstößen (Collision) und sonstige Bewegungen (Another). In Abbildung 42 sind Beispiele für die charakteristischen Bewegungsmuster dargestellt. Diese Beispiele wurden aus dem interaktiven Programm zur Erfassung der Bewegungsmuster exportiert (siehe Kapitel 6.6). Abbildung 42: Typische Bewegungsmuster von Bacillus amyloliquefaciens FZB42 Beispiele typischer Bewegungsmuster von B. amyloliquefaciens FZB42 im StandardNährmedium-I-¼. Die Bewegungen wurden fünf Sekunden lang mit 30 fps aufgenommen. Der Startpunkt jeder Wegstrecke ist hervorgehoben. Es sind Beispiele für folgende typische Bewegungsmuster dargestellt: Richtungsänderung ohne Stoppphasen (Orientation), wellenförmige Bewegungen (Undulation), gerade Strecken (Straight path), Richtungsänderung mit längeren Schwimmbewegungen und kurzen Taumelphasen (Tumble), Richtungswechsel mit kurzen Schwimmbewegungen und längeren Stoppphasen ohne Eigenbewegung (Orientation and Stop), bogenförmige Strecken (Curve). Die Daten wurden aus dem Programm zur interaktiven Erfassung der Bewegungsmuster exportiert. 74 Ergebnisse Die Bewegungsmuster für die Fermentationen 4 – 7 wurden auch auf charakteristische Bewegungsmuster hin untersucht. Die Aufnahme einer zusätzlichen Bildstrecke über 150 Aufnahmen, dies entspricht fünf Sekunden, zu jedem Probezeitpunkt ermöglicht diese Auswertung. Die Fermentationen 4 – 6 wurden mit dem Standard-Nährmedium-I-¼ und Fermentation 7 mit dem ABiTEP-Medium-¼ durchgeführt. Die Auswertung erfolgte über die Summe der Probenentnahmen der einzelnen Fermentationen. Die Daten, in Kreisdiagramme überführt, sind in Abbildung 43 dargestellt. Abbildung 43: Summarische Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 über den gesamten Probezeitraum Ferm. 4 – 6 wurden mit Standard-Nährmedium-I-¼ und Ferm. 7 mit ABiTEP-Medium-¼ durchgeführt. Die Bewegungsmuster sind in acht Kategorien eingeteilt: Richtungsänderung ohne Stoppphasen (Orientation), wellenförmige Bewegungen (Undulation), gerade Strecken (Straight path), Richtungsänderung mit längeren Schwimmbewegungen und kurzen Taumelphasen (Tumble), Richtungswechsel mit kurzen Schwimmbewegungen und längeren Stoppphasen ohne Eigenbewegung (Orientation and Stop), bogenförmige Strecken (Curve), Richtungsänderung aufgrund von Zusammenstößen (Collision) und sonstige Bewegungen (Another). Die summarischen Verteilungen sind über den gesamten Probezeitraum ermittelt. Die Bakterien wurden je über 150 Bilder bei 30 fps beobachtet (5 s). Insgesamt werden für die vier Fermentationen 912 Wegstrecken kategorisiert: Ferm. 4: 267, Ferm. 5: 290, Ferm. 6: 186, Ferm. 7: 169. 75 Ergebnisse Die Kreisdiagramme der Fermentationen 4 – 6 zeigen eine ähnliche Verteilung. Etwa die Hälfte der Bakterien (51%, 52% und 56%) bewegen sich geradlinig (Straight path). Eine Richtungsänderung ohne Stoppphase (Orientation) wird bei 9%, 12% und 13% vollzogen. Das Taumeln (Tumble) wird bei 7%, 8% und 10% der Beobachtungen erkannt. Eine Kurve (Curve) haben 4%, 5% und 8% der Zellen zurückgelegt. Ein Richtungswechsel nach einer Stoppphase, jedoch ohne Taumeln, (Orientation and Stop) wurde bei 1%, 4% und 5% beobachtet. Selten (1%, 2% und 3%) konnte eine wellenförmige Bewegung (Undulation) erkannt werden. Aufgrund von Kollisionen (Collision) einzelner Bakterien können 9%, 12% und 19% der Beobachtungen keinem der Muster zugewiesen werden. Weiterhin ist es bei wenigen (1%, 4% und 4%) Beobachtungen nicht möglich, diese einem der charakteristischen Muster zuzuordnen (Another). Die Verteilung der beobachteten Bewegungsmuster aus Fermentation 7 mit ABiTEPMedium-¼ unterscheidet sich an einigen Punkten signifikant von denen aus den Fermentationen 4 – 6. So ist nur bei 37% der Beobachtungen eine geradlinige Bewegung (Straight path) zu erkennen. Das Taumeln (Tumble) tritt mit 21% im Gegensatz dazu mehr als doppelt so häufig auf. Alle anderen Bewegungsmuster bewegen sich in einer ähnlichen Größenordnung wie bei Fermentation 4 – 6: Orientation 6%, Curve 5%, Orientation and Stop 4%, Undulation 2%, Collision 19% und Another 6%. Das Auftreten der Bewegungsmuster im zeitlichen Verlauf von Fermentation 6 ist in Abbildung 44 aufgetragen. Die Werte sind im Anhang (Seite 116) tabellarisch aufgetragen. Abbildung 44: Zeitliche Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 in Fermentation 6 (Standard-Nährmedium-I-¼) Die Sichtung der verschiedenen Bewegungsformen (Richtungsänderung ohne Stoppphasen (Orientation), wellenförmige Bewegungen (Undulation), gerade Strecken (Straight path), Taumeln (Tumble), Richtungswechsel mit kurzen Schwimmbewegungen und längeren Stoppphasen ohne Eigenbewegung (Orientation and Stop), bogenförmige Strecken (Curve), Richtungsänderung aufgrund von Zusammenstößen (Collision) und sonstige Bewegungen (Another)) im zeitlichen Verlauf der Fermentation 6. Die Bakterien wurden in 150 Bildern mit einer Auflösung von 30 fps beobachtet (Beobachtungszeitraum: 5 s). 76 Ergebnisse Mit Zunahme der Biomasse über die Zeit nimmt die Anzahl der beweglichen Bakterien zu. Vor allem das Bewegungsmuster gerade Strecken (Straight path) nimmt zu Beginn stark zu. Bei Minute 225 sind Bewegungen der Kategorie Richtungsänderung ohne Stoppphasen (Orientation) in einem relevanten Anteil vertreten, Taumeln ist vor allem bei Minute 260 zu beobachten. Die Kollisionen sind bei Minute 225 besonders hoch. Die Verteilung der einzelnen Bewegungsmuster im zeitlichen Verlauf der Fermentation 7 ist in Abbildung 45 dargestellt. Die Werte sind im Anhang (S. 116) tabellarischer aufgetragen. Abbildung 45: Zeitliche Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 in Fermentation 7 (ABiTEP-Medium-¼) Die Sichtung der verschiedenen Bewegungsformen (Richtungsänderung ohne Stoppphasen (Orientation), wellenförmige Bewegungen (Undulation), gerade Strecken (Straight path), Richtungsänderung mit längeren Schwimmbewegungen und kurzen Taumelphasen (Tumble), Richtungswechsel mit kurzen Schwimmbewegungen und längeren Stoppphasen ohne Eigenbewegung (Orientation and Stop), bogenförmige Strecken (Curve), Richtungsänderung aufgrund von Zusammenstößen (Collision) und sonstige Bewegungen (Another)) im zeitlichen Verlauf der Fermentation 7. Die Bakterien wurden in 150 Bildern mit einer Auflösung von 30 fps beobachtet (Beobachtungszeitraum: 5 s). Bei Fermentation 7 sind vor allem Bewegungen der Kategorien Taumeln (Tumble) und gerade Strecken (Straight path) in der Wachstumsphase der Fermentation zu sehen. In Minute 305 übersteigt der Wert der taumelnden Bakterien sogar den der geradlinigen Bewegung, ansonsten liegt das Taumeln immer hinter den Bewegungen der Kategorie gerade Strecken (Straight path). 77 Diskussion 8 Diskussion Die Fermentationsverläufe werden zunächst anhand der Daten der kommerziell verfügbaren Sensoren (Temperatur, pH-Wert und pO2) diskutiert. Anschließend erfolgt die Interpretation der Ergebnisse aus der Bewegungsanalyse. Weiterhin wird die Praxistauglichkeit des entwickelten Messsystems bewertet. 8.1 Verlauf der Fermentationen Bei der Kultivierung von sporenbildenden Bakterien der Gattung Bacillus durchlaufen diese stets ähnliche Veränderungen im Metabolismus. Nach der Auskeimung werden zunächst Glukose oder andere Kohlenhydrate verbraucht und es kommt zu einer exponentiellen Wachstumsphase. In dieser Wachstumsphase werden organische Säuren produziert, was sich durch eine Absenkung des pH-Werts bemerkbar macht. Nachdem die Kohlenhydrate aufgebraucht sind, werden die organischen Säuren metabolisiert (Diauxie). Dies ist durch einen Anstieg des pH-Werts nachvollziehbar. In dieser Phase wächst die Kultur weiter, jedoch setzt bald danach die Sporulation ein (Nakata und Halvorson 1960; Deutscher und Kornberg 1968). Bakterien sind in der Lage, durch Katabolitrepression in einem komplexen Medium zunächst die Nährstoffquellen zu verarbeiten, welche das stärkste Wachstum ermöglichen. Es ist bekannt, dass grampositive Bakterien wie Bacillus Glukose und andere Zucker als Kohlenstoffquelle bevorzugen. Solange Zucker vorhanden sind, werden von den Bakterien nur die Enzyme produziert, die notwendig sind, um diese zu verarbeiten (Stülke und Hillen 1999). Der typische Verlauf des pH-Werts während der Kultivierung von Bacillus wird hierdurch verständlich und entspricht dem charakteristischen Fermentationsverlauf mit dem ABiTEP-Medium-¼ (Fermentationen 1 – 3 und 7). In Abbildung 46 ist ein typischer Verlauf von Glukose, organischen Säuren, pH-Wert und Wachstum dargestellt, hier vom sporenbildenden Bacillus cereus. 78 Diskussion Abbildung 46: Diauxischer Kultivierungsverlauf von Bacillus cereus Kultivierung von B. cereus bei 30 °C in einem komplexen Medium (siehe Nakata und Halvorson 1960). A) Die verbleibende Glukose im Medium sowie der Verlauf der Konzentration von Essig- und Brenztraubensäure in Abhängigkeit der Zeit. B) Der pHWert, die Gesamtkonzentration an Essig- und Brenztraubensäure und die Zelldichte in Abhängigkeit der Zeit. (aus Nakata und Halvorson 1960) (modifiziert). Der Abbau der Glukose und die gleichzeitige Bildung der organischen Säuren - hier wurde die Essig- und die Brenztraubensäurekonzentration ermittelt - ist deutlich zu sehen. Auch das gleichzeitige Absinken des pH-Werts ist dargestellt. Im Tal des pH-Werts zum Ende des Glukoseverbrauchs ist auch eine kurze Stagnation des Zellwachstums zu beobachten. Nach der Umstellung von Glukose- auf Säureverbrauch, hier nach vier Stunden Fermentationsdauer, ist der Beginn der Sporenbildung zu beobachten (Nakata und Halvorson 1960). Einen vergleichbaren Verlauf konnten auch Hageman et al. (1984) bei B. subtilis in einem chemisch definierten Medium aufzeigen. Die Fermentationen werden bis zum Ende der Sporenbildung durchgeführt. Im Anschluss des pH-Wert-Anstiegs ist eine Stagnation und ein leichtes Abfallen des pH-Werts zu beobachten. Dieser Verlauf der Kultivierung kann auch beim verwendeten B. amyloliquefaciens FZB42 unter dem Einsatz des ABiTEP-Medium-¼ beobachtet werden. Die Messwerte der konventionellen Sensoren (Rührerdrehzahl, pO2, pHWert und OD600) sind in Abbildung 47 zu sehen. Die beschriebenen Fermentationsstadien lassen sich anhand dieser Messwerte gut erkennen. 79 Diskussion Abbildung 47: Zeitlicher Kultivierungsverlauf von Bacillus amyloliquefaciens FZB42 auf ABiTEP-Medium-¼ Fermentation 1 zeigt den typischen Verlauf einer Kultivierung mit ABiTEP-Medium-¼. Es sind die Messwerte von Rührerdrehzahl, pO2, pH-Wert und OD600 im zeitlichen Verlauf aufgetragen. Die Fermentation ist in fünf Stadien eingeteilt: Auskeimung und Latenzphase (I), exponentielles Wachstum durch Kohlenhydratverbrauch und Säurebildung (II), Diauxiestufe; Kohlenhydrat- auf Säureverstoffwechselung (III), Wachstum durch Säureverbrauch (IV), Sporulation (V). Zunächst erfolgt die Auskeimung (I) der Sporen. Hierbei ist kaum Aktivität der Kultur zu beobachten. Daher steigt bei konstanter Rührerdrehzahl die Gelöstsauerstoffsättigung (pO2) an. Da das ABiTEP-Medium-¼ trüb ist, liegt die OD600 bereits oberhalb des Werts 1. Nach der Auskeimung beginnt die exponentielle Wachstumsphase (II), die Sauerstoffkonzentration fällt ab und während die Kohlenhydrate verbraucht werden, sinkt der pH-Wert langsam, während die OD600 ansteigt. Die stärkste Wachstumsphase ist geprägt durch einen rasanten Abfall der Sauerstoffsättigung und des pH-Werts, ebenso steigt die OD600 nun deutlich. Die Rührerdrehzahl ist inzwischen gestiegen und die pO2 wird dadurch stabil auf 50% geregelt. Der pH-Wert erreicht nun seinen Tiefpunkt und steigt ab hier wieder zügig an. Die Rührerdrehzahl ist bis dahin stetig gestiegen, stagniert jedoch in dieser Phase. Die OD600 steigt weiter an. Diese Phase stellt eine Stoffwechselumstellung von Kohlenhydrat- auf Säureverbrauch (III) dar. An dieser Stelle sind die Kohlenhydrate im Medium. Während dieser Stoffwechselumstellung steigt der Sauerstoffbedarf nicht weiter an. Die nachfolgende Phase des Wachstums durch Säureverbrauch (IV) ist von einem rasanten Anstieg des pHWerts und des Sauerstoffbedarfs geprägt. Die OD600 steigt noch einmal deutlich an. Sobald die Nährstoffquellen aufgebraucht sind, sinkt der Sauerstoffbedarf schlagartig, der pH-Wert steigt weniger schnell. Weiterhin fällt nun die OD600 rasch ab. Dies stellt den Beginn der Sporulation (V) dar. Die Beobachtungen aus den Fermentationen mit ABiTEP-Medium-¼ decken sich somit mit den Erkenntnissen aus der Literatur. 80 Diskussion Der typische Verlauf mit Standard-Nährmedium-I-¼ (Fermentationen 4 – 6) zeigt eine ähnliche Charakteristik wie mit ABiTEP-Medium-¼. Bei genauer Beurteilung des Fermentationsverlaufs muss hier jedoch eine feinere Untergliederung der Fermentationsphasen vorgenommen werden. Der pH-Wert wechselt weniger parabelförmig seine Richtung und zeigt weiterhin einen deutlichen Knick in der Steigung. Auch die pO2 zeigt hier eine stärkere Änderung. Anstelle einer Stagnation bricht beim pH-Minimum der Sauerstoffbedarf stark ein. Beim Knick in der Steigung des pH-Werts ist wiederum eine Stagnation zu beobachten, ähnlich wie beim Minimum bei der Fermentation mit ABiTEPProduktionsmedium. In Abbildung 48 ist der Verlauf der Kultivierung aus Fermentation 6 zu sehen. Dies ist ein typischer Verlauf für die Kultivierung von B. amyloliquefaciens FZB42 in diesem Medium. Abbildung 48: Zeitlicher Kultivierungsverlauf von Bacillus amyloliquefaciens FZB42 auf Standard-Nährmedium-I-¼ Fermentation 6 zeigt den typischen Verlauf einer Kultivierung mit Standard-NährmediumI-¼. Es sind die Messwerte von Rührerdrehzahl, pO2, pH-Wert und OD600 im zeitlichen Verlauf aufgetragen. Die Fermentation ist in sieben Stadien eingeteilt: Auskeimung und Latenzphase (I), exponentielles Wachstum durch Glukose- und Stickstoffverbrauch und Säurebildung (II), Diauxiestufe I; Glukose- auf Säureverstoffwechselung und Stickstoffverbrauch (III), exponentielles Wachstum durch Stickstoff- und Säureverbrauch (IV), Diauxiestufe II; Stickstoff- auf reine Säureverstoffwechselung (V), Wachstum durch Säureverbrauch (VI), Sporulation (VII). Da das Standard-Nährmedium-I-¼ klar ist, beginnt die OD600 bei deutlich niedrigen Werten als bei den Fermentationen mit dem ABiTEP-Medium-¼. Die Phase der Auskeimung (I) verläuft ähnlich, jedoch findet die Auskeimung schneller statt. Nach der Auskeimung und Latenzphase beginnt die exponentielle Wachstumsphase durch Glukose- und Stickstoffverbrauch mit Säurebildung (II). Der Abfall des pH-Werts (pH 7,3 zu Fermentationsbeginn auf 6,1 im pH-Tiefpunkt) ist stärker als beim ABiTEP-Medium-¼ (pH 7 bei Fermentationsbeginn auf 6,8 im pH-Tiefpunkt). Die anderen Fermentationen des jeweiligen Mediums verhalten sich vergleichbar. Ebenso ist der Sauerstoffverbrauch höher, 81 Diskussion der Rührer wird schon in der Wachstumsphase stark hochgeregelt. Die OD600 steigt deutlich an. Die erste Diauxiestufe (IV) wird vom Minimum des pH-Werts geprägt, die Umkehrung der Steigung im pH-Wert-Verlauf erfolgt sehr plötzlich. Die Glukose- wird hier auf Säureverstoffwechselung umgestellt. Zu diesem Zeitpunkt kommt es auch zu einem deutlichen Einbruch des Sauerstoffbedarfs. Die OD600 steigt weiter an. Die exponentielle Wachstumsphase mit Stickstoff- und Säureverbrauch (IV) zeigt einen langsamen Anstieg des pH-Werts. Die OD600 und der Sauerstoffbedarf steigen hier an. Ein Knick in der Steigung des pH-Wert-Verlaufs sowie eine Stagnation des Sauerstoffbedarfs sind die Anzeichen für die zweite Diauxiestufe (V). Eine Umstellung zum reinen Säureverbrauch findet statt. Die OD600 steigt hier weiter an, jedoch mit deutlich geringerer Steigung. Während des Wachstums durch Säureverbrauch (VI) erreicht die OD600 ihr Maximum, der pH-Wert steigt stark an, der Sauerstoffbedarf steigt leicht. Das Absinken des Sauerstoffbedarfs sowie der schnelle Abfall der OD600 zeigen den Beginn der Sporulation (VII). Der pH-Wert steigt hier weiter an. Die Fermentationen mit dem ABiTEP-Medium-¼ dauern von Fermentationsbeginn bis zum Einsetzen der Sporulation etwa 1,5 h länger als im Standard-Nährmedium-I-¼ (Ferm. 1: 382 min, Ferm. 2: 391 min, Ferm. 3: 425 min, Ferm. 4: 325 min, Ferm. 5: 315 min, Ferm. 6: 305 min). Im ABiTEP-Medium-¼ liegen die Kohlenhydrate zum Teil als Stärke vor, die erst in Glukose gespalten werden muss. Durch das Aufspalten der Stärke ist die Steigungsänderung im pH-Minimum nicht so plötzlich wie im Standard-Nährmedium-I-¼. Dort liegen die Kohlenhydrate als reine Glukose vor. Ist diese aufgebraucht, wird die starke pHWertänderung beobachtet. Parallel zur Glukose werden im Standard-Nährmedium-I-¼ Stickstoffquellen verarbeitet. 8.2 Bewegungsanalyse von Bacillus amyloliquefaciens FZB42 Die Vitalität von Mikroorganismen während ihrer Kultivierung ist ein bedeutender Fermentationsparameter. Wie Grossart et al. (2001) schon formuliert haben, kann die Beweglichkeit von Bakterien ein Kriterium für ihre Vitalität sein, da Bakterien, die sich aktiv bewegen, weder tot noch inaktiv sind. Durch das entwickelte System kann die Beweglichkeit begeißelter Bakterien beobachtet und ausgewertet werden. Der Abstand der einzelnen Probenentnahmen beträgt bei allen Versuchen zwischen 10 und max. 30 min (typisch 15 – 20 min). Bei Prozessen mit Fermentationsdauern von mehreren Stunden sind Prozesseingriffe aufgrund der in dieser Messfrequenz erhaltenen Ergebnisse möglich. Vor allem im Hinblick auf Optimierungsmöglichkeiten, die eine Messfrequenz von 5 min ermöglichen, kann das entwickelte System als onlinefähig angesehen werden. Die Optimierung der Probenentnahme wird in Kapitel 8.3 näher diskutiert. Da nur Partikel erfasst werden, die sich aktiv bewegen, kann das Messsystem auch in partikelbehafteten Medien Anwendung finden. Trübe Medien stellen vor allem für elektrooptische Messsysteme ein Problem dar (Junne et al. 2008; Junne et al. 2010). Die Trübung durch Stärke stellt einen Offset für die optische Dichte dar, der im Laufe der Fermentation abnimmt. Diese Einschränkung ist mit dem entwickelten Messsystem nicht gegeben, da unbewegliche Partikel nicht berücksichtigt werden. Voraussetzung ist, dass Partikelgröße und 82 Diskussion Beschaffenheit nicht hinderlich bei der Minimierung der Schichtdicke sind. Starre Partikel in der Suspension, die größer als 3 µm sind, führen zu einer erhöhten Schichtdicke. Die Funktionsfähigkeit wurde mit B. amyloliquefaciens FZB42 im Produktionsmedium für die industrielle Kultivierung (ABiTEP GmbH) in ¼-Konzentration und mit StandardNährmedium-I (Carl Roth GmbH + Co. KG) in ¼-Konzentration gezeigt. In allen Versuchen wurde ein ähnlicher Verlauf der durchschnittlichen Bewegungsgeschwindigkeit während der verschiedenen Fermentationsphasen beobachtet. Dem Abfall der Geschwindigkeit während der Diauxiestufen folgt ein Anstieg nach abgeschlossener Stoffwechselumstellung. Im ABiTEP-Medium-¼ kam es zu einer Diauxiestufe (Umstellung von Kohlenhydrat- auf Säureverstoffwechselung), im Standard-Nährmedium-I-¼ kam es zu zwei Diauxiestufen (Umstellung von Glukose- auf Säure- und Stickstoffverbrauch (I) und Umstellung von Stickstoff- auf reine Säureverstoffwechselung (II)). Nachdem die maximale Zelldichte erreicht ist, fällt die Durchschnittsgeschwindigkeit erneut ab, bis keine beweglichen Bakterien mehr detektierbar sind. Bei den Fermentationen 1 – 3 (ABiTEP-Medium-¼) beträgt die durchschnittliche Bewegungsgeschwindigkeit in der exponentiellen Wachstumsphase zwischen 50,7 und 58 µm s-1. In der Diauxiestufe (pH-Minimum) wird der Stoffwechsel von Kohlenhydrat- auf Säureverbrauch umgestellt. Der Verlauf der berechneten Bewegungsgeschwindigkeit zeigt an dieser Stelle ebenso ein lokales Minimum, welches zwischen 47,7 und 50,2 µm s-1 liegt. Es wird vermutet, dass die Bewegungsgeschwindigkeit aufgrund des Wechsels der Energiegewinnung (von Kohlenhydrat- auf Säureverbrauch) reduziert ist, da zu diesem Zeitpunkt weniger Energie für den Geißelantrieb zur Verfügung steht. Nach der Diauxiestufe, während des Wachstums durch Säureverbrauch, zeigt die Bewegungsgeschwindigkeit bei den Fermentationen 1 – 3 jeweils einen Aufwärtstrend mit Durchschnittsgeschwindigkeiten von 51,9 bis 59,6 µm s-1. Der pH-Wert steigt weiter an während Rührerdrehzahl und Belüftung bei konstantem pO2 stagnieren bzw. leicht abfallen. Die Bewegungsgeschwindigkeit liegt zwischen 40,8 und 46,2 µm s-1 und sinkt anschließend ab, da die Nährstoffquellen aufgebraucht sind. Dieser Punkt zeigt den Beginn der Sporulation an. Bei den Fermentationen 4 – 6 (Standard-Nährmedium-I-¼) ist ein ähnlicher Verlauf der Bewegungsgeschwindigkeiten zu sehen. In der exponentiellen Wachstumsphase mit Glukoseverbrauch und Säurebildung sind die Bakterien am schnellsten mit Durchschnittsgeschwindigkeiten zwischen 46,03 und 58,32 µm s-1. Da die Glukose im Standard-Nährmedium-I-¼ am effektivsten zu verarbeiten ist, steht hier für den Geißelantrieb und die Bewegung die meiste Energie zur Verfügung. Während der ersten Diauxiestufe, wenn die Glukose aufgebraucht ist (im pH-Minimum), sinkt die Durchschnittsbewegungsgeschwindigkeit in den drei Fermentationen auf einen Bereich zwischen 39,26 und 50,16 µm s-1 ab. Die Bakterien verwenden während der Umstellung von Säurebildung auf Säureverbrauch vermutlich weniger Energie für die Bewegung. In der darauffolgenden exponentiellen Wachstumsphase ist ein erneuter Anstieg der Bewegungsgeschwindigkeit zu beobachten (46,06 bis 55,1 µm s-1), bevor sie während der zweiten Diauxiestufe wieder abfällt (39,28 bis 47,63 µm s-1). Die Bakterien versuchen stets, die optimale Energieversorgung zu 83 Diskussion erreichen. Sinkt die Konzentration des Nährstoffs, der zu dieser Zeit zur hauptsächlichen Energieversorgung dient, unter ein kritisches Niveau, ist der Verbrauch einer alternativen Energiequelle sinnvoll. Anstelle durch Bewegung die optimale Konzentration des Nährstoffs zu finden, wird der Metabolismus umgestellt. Daher sinkt die Bewegungsgeschwindigkeit in diesen Phasen und steigt anschließend wieder an. Dieser Anstieg nach der zweiten Diauxiestufe erreicht bei den Fermentationen 4 – 6 nochmal Werte zwischen 45,06 und 47,44 µm s-1. Zu Beginn der Sporulation, wenn die Nährstoffe aufgebraucht sind, liegt die durchschnittliche Bewegungsgeschwindigkeit noch zwischen 36,91 und 38,37 µm s-1 und sinkt stetig bis zur vollständigen Sporulation. Bei den Fermentationen 4 und 5 wurde gegen Ende der Fermentation Antischaummittel zugegeben. Es ist nach jeder Zugabe von Antischaummittel zu einem Absinken der Durchschnittsgeschwindigkeit gekommen. Daher wurde in Fermentation 6 kein Antischaummittel zugegeben. Das Absinken der Durchschnittsgeschwindigkeit durch die Zugabe von Antischaummittel wird später diskutiert (S. 85). Die gemessenen durchschnittlichen Bewegungsgeschwindigkeiten liegen an allen Messpunkten zwischen 35 und 60 µm s-1 mit Höchstgeschwindigkeiten bis ca. 100 µm s-1. Dieser Bereich passt gut zu den Literaturwerten gemessener Bakteriengeschwindigkeiten (siehe Tabelle 2). Die Durchschnittsgeschwindigkeit im ABiTEP-Medium-¼ (Fermentationen 1 – 3) beträgt 48,5 µm s-1. Im Standard-Nährmedium-I-¼ (Fermentationen 4 – 6) ist die berechnete Durchschnittsgeschwindigkeit mit 44 µm s-1 etwas geringer. Da das StandardNährmedium-I-¼ im Gegensatz zum ABiTEP-Medium-¼ keine Partikel (Stärke) beinhaltet, sind die Nährstoffe homogener im Medium verteilt. Vermutlich regt die inhomogene Verteilung im ABiTEP-Medium-¼ die Bakterien zu einer höheren Beweglichkeit an. Die Geschwindigkeitsverteilung um die Mittelwerte ist in beiden Medien vergleichbar. Die Standardabweichung beträgt 28 µm s-1 beim ABiTEP-Medium-¼ und 29,5 µm s-1 beim Standard-Nährmedium-I-¼. Die geringen Unterschiede könnten aus der unterschiedlichen Dauer der Fermentationsphasen und den Unterschieden im Fermentationsverlauf resultieren. Die statistische Auswertung der Fermentationen 1 – 3 zeigt für die charakteristischen Fermentationszeitpunkte signifikante Unterschiede in den Bewegungsgeschwindigkeiten. Acht von neun statistischen Vergleichen zeigen p-Werte unterhalb des Signifikanzniveaus von 0,05. Lediglich in Fermentation 1 ergibt die Gegenüberstellung der Daten bei einem Vergleich einen p-Wert von 0,1120. Bei Fermentation 1 sind die Messintervalle von max. 30 min länger als bei den Fermentationen 2 und 3 (Fermentation 2: max. 20 min, Fermentation 3: max. 15 min). Es ist daher wahrscheinlich, dass die Probenentnahme nicht genau den Punkt der Diauxiestufe getroffen hat. Dies könnte den p-Wert oberhalb des Signifikanzniveaus erklären. Da die acht anderen Untersuchungen jedoch p-Werte deutlich unter 0,05 ergeben, kann davon ausgegangen werden, dass signifikante Unterschiede zwischen den Messdaten vorliegen. Neben dem statistischen Beweis über die Signifikanz der Messdaten zeigt die Untersuchung, dass unter den untersuchten Kultivierungsbedingungen einige Fermentationsphasen (z.B. die Diauxiestufe) nicht mit einem Messintervall von 30 min darstellbar sind. Die Zeit zwischen zwei Messungen sollte daher deutlich unter 30 min liegen. 84 Diskussion Die p-Werte der statistischen Untersuchung von Fermentation 4 – 6 zeigen zum größten Teil (23 von 27 Vergleichen) Werte unterhalb des Signifikanzniveaus von 0,05. Der Vergleich der ersten und zweiten Diauxiestufe bei Fermentation 4 und 5 ergeben p-Werte von 0,0524 bzw. 0,3676. Die Vergleichbarkeit der Ergebnisse der beiden Diauxiestufen und damit verbunden die hohen p-Werte zeigen, dass ähnliche Gründe für die Reduktion der Durchschnittsgeschwindigkeit vorliegen, nämlich die Umstellung des Metabolismus auf eine andere Nährstoffquelle (z.B. von Glukose- auf Säureverbrauch und von Stickstoff- auf reinen Säureverbrauch). Bei den Fermentationen 4 und 6 überschreitet der p-Wert für den Vergleich der ersten Diauxiestufe gegen den Beginn der Sporulation den Grenzwert von 0,05. Die pWerte liegen bei 0,5754 bei Fermentation 4 und bei 0,0921 für Fermentation 6. Da auch zu Beginn der Sporulation ein Rückgang der Durchschnittsgeschwindigkeit aufgrund der Verknappung von Nährstoffen stattfindet, ist die Ähnlichkeit zwischen den Messwerten gegeben. Die Messintervalle von Fermentation 4 liegen bei 20 min, die bei den Fermentationen 5 und 6 bei jeweils 15 min. Die Ursachen für unterschiedliche Messintervalle werden in Kapitel 8.3 diskutiert. Das entwickelte Messsystem kann somit die Änderungen der Bewegungsgeschwindigkeit von B. amyloliquefaciens FZB42 während einer Batch-Fermentation verfolgen. Die Änderungen der Geschwindigkeit korrelieren mit bekannten Veränderungen im Stoffwechsel der Bakterien (Nakata und Halvorson 1960; Deutscher und Kornberg 1968), die durch pH-Wert, pO2 und OD600 validiert wurden. Das System eignet sich somit zur Online-Erfassung der Vitalität von beweglichen Bakterien. Untersuchungen weiterer Stressfaktoren neben der Nährstofflimitierung (z.B. Temperaturschwankungen, Sauerstoffmangel oder Infektionen mit Bakteriophagen) sind zum Beweis jedoch nötig. Bevor eine allgemeingültige Aussage getroffen werden kann, sind ebenso weitere Untersuchungen anderer beweglicher Bakterienstämme notwendig. Bei den Fermentationen 4 und 5 ist ein Absinken der Beweglichkeit nach Zugabe von Antischaummittel aufgetreten. Da das Antischaummittel einen Einfluss auf die Sauerstoffsättigung im Medium hat, ist der beobachtete Einfluss auf die Bewegungsgeschwindigkeit möglicherweise hierauf zurückzuführen. Ein weiterer Grund für das Absinken der Geschwindigkeit könnte die Änderung der Oberflächenspannung sein. Der Grund für das Absinken der Geschwindigkeit nach Zugabe von Antischaummittel muss in weiteren Messreihen separat untersucht werden. Neben der durchschnittlichen Geschwindigkeit lässt sich über die Anzahl der beweglichen Bakterien eine Aussage über die Zelldichte (beweglicher Bakterien) der Kultur treffen. Anders als beim OD600 werden tote, inaktive und unbewegliche Bakterien sowie Sporen und Trübstoffe nicht ermittelt. Aus der ermittelten Zelldichte beweglicher Bakterien könnten Trends in der Entwicklung der Kultur somit schneller als durch die OD600 erkennbar sein. Die Ergebnisse der Versuchsfermentationen diesbezüglich sind vielversprechend. Besonders in trüben Medien zeigen die Kurven der Anzahl beweglicher Bakterien spezifischere Verläufe als die OD600 (siehe Abbildung 33). Die Verläufe der beweglichen Bakterien zeigen in den Fermentationen 1 – 3 mit dem ABiTEP-Medium-¼ trotz der Trübung eine gute Korrelation 85 Diskussion zur OD600 (siehe Abbildung 35). Nach Einsetzen der Sporulation bleibt die OD600 hoch, die Anzahl der beweglichen Bakterien sinkt jedoch. Ein vergleichbares Verhalten nach Einsetzen der Sporulation kann auch im Standard-Nährmedium-I-¼ gezeigt werden. Vor der Sporulation ist die Korrelation zwischen der OD600 und die der Anzahl der beweglichen Bakterien ebenso gut (siehe Abbildung 39). Versuche in weiteren Medien, Fed-BatchFermentationen, die Kultivierung mit anderen Bakterienstämmen sowie das parallele Ausplattieren von Proben als Referenz sind noch notwendig, um die Aussagekraft des Parameters (Anzahl an beweglichen Bakterien pro Bild) zu beweisen. Neben der automatischen Erfassung der Bewegungsgeschwindigkeit und der Anzahl der beweglichen Bakterien konnten durch eine interaktive Auswertung von Bildserien charakteristische Bewegungsmuster identifiziert werden. Die Bildstrecken zur Auswertung der Bewegungsmuster haben eine Länge von 150 Aufnahmen, dies entspricht fünf Sekunden. Bei den Fermentationen 4 – 6 (Standard-Nährmedium-I-¼) schwammen 53% der Bakterien geradeaus, 5% schwammen eine Kurve und nur 2% der Bakterien bewegten sich wellenförmig vorwärts. Die wellenförmige Bewegung konnte hauptsächlich bei Ketten aus zwei Zellen beobachtet werden. Einen Richtungswechsel vollzogen etwas mehr als 20% der Bakterien, davon knapp 10% durch Taumeln, knapp 5% nach einem Stopp ohne Taumelbewegung und ungefähr 10% ohne Taumel- und Stopp-Phase. Etwa 15% der Beobachtungen konnten keinem der Bewegungsmuster zugewiesen werden, ein Großteil davon aufgrund von Kollisionen. In Fermentation 7 (ABiTEP-Medium-¼) wurden ebenso die Bewegungsmuster ausgewertet. Im Gegensatz zu den Fermentationen 4 – 6 schwammen nur etwa ein Drittel (37%) der Zellen geradeaus, eine Kurve schwammen etwa 5% und 2% in einer wellenförmigen Bewegung. Auch hier handelte es sich bei den wellenförmigen Bewegungen vornehmlich um die Bewegung zweier Ketten. Etwa ein weiteres Drittel (31%) wechselte in den beobachteten Bildserien die Richtung, 21% durch Taumeln, 4% nach einer Stopp-Phase und 6% ohne Stopp- und Taumel-Phase. 25% der Beobachtungen konnten keinem charakteristischen Bewegungsmuster zugewiesen werden, 19% davon wegen Kollisionen. Die Anzahl der Kollisionen steigt durch den Feststoffanteil im Medium. Während bei Standard-NährmediumI-¼ die Kollisionen ausschließlich zwischen Bakterien stattfanden, kam es hier auch zu Kollisionen zwischen Bakterien und Partikeln aus dem Medium. Neben den Kollisionen liegen die Unterschiede in der Verteilung der Bewegungsmuster zwischen Standard-Nährmedium-I-¼ und ABiTEP-Medium-¼ hauptsächlich bei den Mustern geradeaus Schwimmen (Straight path) und Taumeln (Tumble). Die Ursache hierfür könnte die Verteilung der Nährstoffe im Nährmedium sein. Während beim Standard-Nährmedium-I-¼ die Nährstoffe gelöst und gut verteilt sein dürften, könnten durch die Feststoffe im ABiTEPMedium-¼ Nährstoffgradienten durch den Stärkeabbau entstehen. Da Bakterien die Fähigkeit besitzen, Nährstoffgradienten zu folgen (Adler 1966; Macnab und Koshland 1972; Berg 1975) wird vermutlich im ABiTEP-Medium-¼ häufiger ein Taumeln beobachtet, um einer erhöhten Nährstoffkonzentration entgegen zu schwimmen. Dies ist ein weiterer Hinweis, dass sich die Durchschnittsgeschwindigkeit im Verlauf der Fermentation im ABiTEP-Medium-¼ 86 Diskussion durch das Auftreten von Nährstoffgradienten erhöht. Bakterien könnten während der gerichteten Nahrungssuche schneller sein als bei der Bewegung im homogenen Medium. Die zeitliche Abhängigkeit der Verteilung der Bewegungsmuster zu beurteilen, ist mit der Anzahl der erhobenen Daten nur schwer zu realisieren. Natürlich nehmen die Kollisionen mit steigender Bakterienkonzentration und Aktivität zu. Um weitere Abhängigkeiten zu den einzelnen Fermentationsphasen eindeutig nachzuweisen, sind weitergehende und ausführlichere Messungen notwendig. Die gewonnenen Messwerte lassen jedoch vermuten, dass sich die Richtungsänderungen im Bereich der Diauxiestufen häufen. Da bei einer Nahrungsknappheit das aktive Suchen nach neuen Nahrungsquellen notwendig ist, erscheint diese Tendenz plausibel. 8.3 Praxistauglichkeit des Online-Mikroskopie-Systems Um die Ergebnisse der automatischen Bildauswertung in Bezug auf die Durchschnittsgeschwindigkeit auf Plausibilität zu überprüfen, wurde eine manuelle Wegverfolgung zu den vier charakteristischen Fermentationszeitpunkten von Fermentation 1 durchgeführt. Wie in Kapitel 7.2.1 gezeigt, konnte eine gute Übereinstimmung der Messergebnisse erreicht werden. Eine geringfügig zu hohe Schichtdicke kann dazu führen, dass die Bakterien sich leicht in Richtung z-Achse drehen können, oder etwas außerhalb des Fokus schwimmen. Die Veränderung in den Merkmalen der betreffenden Bakterien kann zu einer Unterbrechung der Wegverfolgung führen. Diese Unterbrechungen werden zum Teil durch eine spätere Nachbearbeitung zusammengeführt, dies ist dem Algorithmus jedoch nicht immer möglich. Der entstehende Fehler ist jedoch gering, da alle Durchschnittsgeschwindigkeiten über die Anzahl der erkannten Aufnahmen gewichtet sind. Durch den beschriebenen Fehler kann aus einem Bakterium, das manuell über 30 Aufnahmen verfolgt wird, zwei Bakterien (z.B. ein Bakterium über 10 Aufnahmen und eines über 19 Aufnahmen) werden. Die Ergebnisse der Durchschnittsgeschwindigkeit sind in diesen Fällen jedoch nahezu identisch. Bakterien, die nur über wenige Aufnahmen verfolgt werden konnten werden vom System nicht gewertet, da die Messwerte in der Regel nicht repräsentativ sind, da z.B. das Bakterium nur an einer Ecke den Bildausschnitt streift und in zwei von vier Aufnahmen nicht vollständig auf dem Bild dargestellt wird. Starke Änderungen der Merkmale der einzelnen Bakterien können neben erhöhter Schichtdicke auch auf Kollisionen mit anderen Bakterien oder Partikeln zurückgeführt werden. Diese Ungenauigkeiten können in der manuellen Wegverfolgung ausgeglichen werden, daher sind die Standardfehler der manuellen Wegverfolgung geringfügig kleiner. Diese potentiell höhere Genauigkeit der manuellen Wegverfolgung muss jedoch vor dem Hintergrund der Zeitersparnis sowie der Online-Tauglichkeit des automatischen Systems betrachtet werden. Wenige Minuten Berechnungszeit stehen vielen Arbeitsstunden bzw. -tagen manueller Auswertung gegenüber. Die Validierung gegenüber der manuellen Auswertung konnte somit die Plausibilität der automatisch ermittelten Durchschnittsgeschwindigkeiten zeigen. 87 Diskussion Die Stabilität des Messsystems steigt mit der Bildqualität. Angestrebte Optimierungen im Messsystem könnten dies erreichen. Mit einer steigenden Stabilität wäre neben der Durchschnittsgeschwindigkeit auch eine Beurteilung der verschiedenen Bewegungsmuster möglich. Dies könnte als weiterer Parameter eine Aussage über den Status der Bakterien ermöglichen. Viele Richtungswechsel könnten z.B. auf eine intensive Nahrungssuche und bevorstehende Stoffwechseländerung hinweisen. Hohe Scherkräfte können zum Abriss von Geißeln führen. Dadurch können Bakterien, die hohen Scherkräften ausgesetzt wurden, sich nicht mehr bewegen. Die Fähigkeit zur Regeneration der Geißeln und auch zur Zellteilung muss dadurch nicht verloren gehen (Grossart et al. 2001). Um die Geißeln während der Probenentnahme und der Vermessung nicht zu beschädigen, müssen die Scherkräfte minimiert werden. Daraus resultierte die Notwendigkeit einer Küvette mit variabler Schichtdicke. Bei gegebenem Durchfluss werden die Strömungsgeschwindigkeiten und somit die Scherkräfte durch eine hohe Schichtdicke minimiert. Es konnte gezeigt werden, dass in der Wachstumsphase ein hoher Anteil an Bakterien beweglich war. Der Einfluss der Scherkräfte bei der Probenentnahme und Messung scheint daher keine offensichtliche Beschädigung der Geißeln zu verursachen trotz schneller Durchspülung der Messzelle. Zudem wurden die Schubspannungen in der Küvette näherungsweise berechnet. Die höchsten Schubspannungen treten demnach mit ca. 500 Pa in der Zuleitung auf. Durch Versuche mit vielfach höheren Schubspannungen (bis ca. 33.000 Pa) konnte gezeigt werden, dass keine Beschädigung der Geißeln zu befürchten ist. Der Anteil der beweglichen Bakterien, nachdem sie mit 4 ml min-1 durch Kapillaren mit verschiedenen Innendurchmessern (0,25 mm, 0,5 mm und 1 mm) gepumpt wurden, erwies sich in der mikroskopischen Untersuchung als unabhängig vom Innendurchmesser. Die beweglichen Bakterien machten im Mittel einen Anteil von 28 % ± 12 % aus. Da schon die Probenentnahme aus dem Fermenter etwa die Schubspannung der 1 mm dicken Kapillare erzeugt (die Entnahme erfolgt ebenso über einen dünnen Schlauch), dient diese Messung als Referenz. Eine Referenzmessung ganz ohne Schubspannung wurde nicht durchgeführt. Da auch die Entnahme über eine Pipette Schubspannungen erzeugt, wurde die definierte Schubspannung in der 1 mm Kapillare als Referenz vorgezogen. Da kein Unterschied in der Anzahl an beweglichen Bakterien nach den drei unterschiedlichen Behandlungen gefunden wurde, ist davon auszugehen, dass die verwendeten Schubspannungen keinen Einfluss auf die Bewegungsfähigkeit von B. amyloliquefaciens FZB42 haben. Für andere Mikroorganismen sollte vor Verwendung des Messsystems ebenso der Einfluss von Schubspannungen auf die Bewegungsfähigkeit untersucht werden. In seltenen Fällen wurde die Verschmutzung der Küvette während der Fermentation zu stark, um eine ausreichend kleine Schichtdicke in der Küvette zu erreichen. Gerade durch die verhältnismäßig großen Partikel im ABiTEP-Medium-¼ konnten in diesen Fällen Probleme auftreten. Die Reinigung erfolgte während der Fermentation nur bei Bedarf und wird manuell durchgeführt. Durch diese nötigen, außerplanmäßigen Reinigungen konnten die Probeintervalle von z.B. 15 min nicht immer eingehalten werden. Dies erklärt die Uneinheitlichkeit der Messintervalle bei manchen Fermentationen. 88 Diskussion Die aktuell umgesetzte Probenentnahme und Probenvorbereitung benötigen aufgrund ihrer Ausführung im Prototyp eine große Menge an Probe pro Messung. Dies ist einer der Gründe, weshalb die Anzahl der Probenentnahme mit dem aktuellen Aufbau nicht wesentlich erhöht werden kann. Um den Einfluss der Probenentnahme auf die Fermentation gering zu halten, wurden für Versuchsmaßstäbe relativ große Fermentationsvolumen (8 – 10 l) gewählt. Weiterhin sind bei geringen Zelldichten kaum ausreichend viele bewegliche Bakterien zu erfassen, um eine stabile Durchschnittsbildung der Geschwindigkeit zu ermöglichen. Neben dem Verdünnen der Probe wäre es daher sinnvoll, eine Erhöhung der Konzentration, z.B. durch Filterung, zu ermöglichen. Trotz der aufgezeigten Einschränkungen des Prototyps konnte die Praxistauglichkeit und das Potential des Messsystems, der Durchflussküvette mit flexibler Schichtdicke und der automatisierten Probenentnahme und Auswertung aufgezeigt werden. Nötige und empfohlene Optimierungen für den Einsatz des Messsystems folgen im Ausblick (Kapitel 9). Eine Aussage über die Aktivität und Vitalität der Zellen ist durch das entwickelte Messsystem möglich. Je höher die Durchschnittsgeschwindigkeit ist, desto höher ist die Aktivität der Zelle. Bei Nahrungsknappheit bzw. Stoffwechseländerungen nimmt die Durchschnittsgeschwindigkeit ab. Der Einfluss anderer Fermentationsparameter auf die Durchschnittsgeschwindigkeit ist wahrscheinlich. Beim Einsatz von Antischaummittel konnte dies bereits beobachtet werden. 89 Ausblick 9 Ausblick Mit dem Prototyp konnte die Abhängigkeit der Durchschnittsgeschwindigkeit von den verschiedenen Fermentationsstadien (Diauxie) von B. amyloliquefaciens FZB42 gezeigt werden. Nach den Erfahrungen mit dem Prototyp der Durchflussküvette gibt es an dieser Stelle mehrere Ansätze für Verbesserungsmöglichkeiten. Im aktuellen Prototyp kommt, wie in Kapitel 6.4.1 beschrieben ist, ein Polycarbonat-Objektträger zum Einsatz. Dieser kreisförmige Objektträger hat einen Durchmesser von 16 mm und eine Stärke von 2 mm. Durch eine Kerbe im Objektträger kann dieser zwischen eine Silikonmembran gespannt werden (siehe Abbildung 13). Anstelle des Polycarbonats sollte Quarzglas oder besser Saphirglas für den Objektträger zum Einsatz kommen. Quarzglas oder Saphirglas sind zum einen beständiger gegen Chemikalien und zum anderen kratzfest. Eine bessere Oberflächengüte verhindert das Anhaften von Bakterien und Schmutz. Durch die bessere Chemikalienbeständigkeit sind weitere Reinigungsschritte möglich, z.B. der Einsatz von Natronlauge, Methanol oder Kalilauge. Tabelle 12 zeigt die Beständigkeit von Polycarbonat, hier Makrolon (Bayer MaterialScience AG, Leverkusen), gegen einige Chemikalien. Tabelle 12: Beständigkeit von Makrolon gegen einige Chemikalien Die Beständigkeit von Polycarbonat, hier Makrolon, gegen einige Chemikalien. + = beständig, - = nicht beständig (aus Makrolon Technische Information, Datei-Nr.: KU28057-0409, Ausgabe 2004-09-28). Quarz- und Saphirglas sind gegenüber allen den in der Tabelle genannte Chemikalien beständig. 6 Tage / 23 °C Aceton Benzin (aromatenfrei) Chloroform quillt an + löst Essigsäure, 10%ig in Wasser + Ethanol (rein) + Isopropanol (rein) + Kaliumhydroxid (Kalilauge), 1%ig in Wasser - Methanol - Natriumhydroxid (Natronlauge), 1%ig in Wasser - Phosphorsäure, 1%ig in Wasser + Wasserstoffperoxid, 30%ig in Wasser + Zitronensäure, 10%ig in Wasser + Weiterhin wäre eine Verkleinerung des Durchmessers von Objektträger und Deckglas sinnvoll, da damit das Volumen der Küvette reduziert wird, was ein schnelleres Einstellen der 90 Ausblick erwünschten Schichtdicke, sowie ein geringeres Probevolumen zur Folge hätte. Bei einer Verkleinerung des Objektträgers und des Deckglases und somit der gesamten Geometrie muss weiterhin die Kompatibilität mit gängigen Labormikroskopen berücksichtigt werden. Es wäre ebenfalls sinnvoll, die Schlauchtüllen durch Anschlüsse für Kapillaren, sowie die Schläuche durch dünne Edelstahlkapillaren zu ersetzen. In diesem Zuge müsste auch die Schlauchpumpe durch z.B. eine Spritzenpumpe ersetzt werden. Die Schläuche sind flexibel und wirken sich beim Anlegen des Unterdrucks negativ auf die nötige Zeit, bis die erwünschte Schichtdicke erreicht wird, aus. Zudem wirkt sich die starke Flexibilität auch negativ auf die Stabilität der Schichtdicke aus und die Strömung in der Probe dauert länger an. Bei einem Wechsel von Schlauch auf Kapillare sollte auch der Innendurchmesser reduziert werden, da ein geringeres Probevolumen beim Anlegen des Unterdrucks schneller zur Ruhe kommt und auch die erwünschte Schichtdicke sich schneller einstellt. Ein geringeres Probevolumen ermöglicht ebenso Optimierungen und Verkleinerungen bei der Probenentnahme und der Probenvorbereitung. Eine Möglichkeit, die Durchflussküvette zu temperieren, sollte in einer überarbeiteten Version ebenso vorgesehen werden. Hierzu können z.B. Peltierelemente oder Heizwiderstände zum Einsatz kommen. Die automatische Probeentnahmeeinheit sollte im nächsten Schritt überarbeitet werden. Das Totvolumen bei der Entnahme und die nötige Probenmenge sollten reduziert werden. Eine Möglichkeit, die Probe aufzukonzentrieren und nicht nur zu verdünnen, würde eine stabile Messung schon bei kleinen Zelldichten ermöglichen. Es sollten dann auch Funktionen zur Reinigung der Probenvorbereitungseinheit und der Küvette integriert werden. Eine Spülung der Küvette mit einer Reinigungslösung nach jedem Messvorgang würde die Bildqualität wie auch die Stabilität der Schichtdicke positiv beeinflussen. Weiterhin sollte für zukünftige Anwendungen die Probenvorbereitung temperiert erfolgen. Gerade durch die Reduktion der Probenmenge, was die Anwendung des Systems auch bei kleineren Fermentationsvolumina möglich macht, ist ein stärkerer Einfluss der Umgebungstemperatur zu erwarten. Die Temperierung der Probenvorbereitungseinheit zusammen mit der angedachten Temperierung der Durchflussküvette eliminiert den Temperatureinfluss auf die gemessenen Geschwindigkeiten der Bakterien. Die aktuell eingesetzte Kamera DFK 23G445 (The Imaging Source Europe GmbH) ist eine Industriekamera im Niedrigpreissegment. Durch den Einsatz einer hochwertigen Kamera für den Mikroskopie-Einsatz wie z.B. die ZEISS Axiocam 503 color (Carl Zeiss Microscopy GmbH) könnten die Ergebnisse und die Stabilität des Messsystems erheblich verbessert werden. In Tabelle 13 ist ein Vergleich der Kernspezifikationen der beiden genannten Kameras aufgeführt. 91 Ausblick Tabelle 13: Vergleich Kamera DFK 23G445 und ZEISS Axiocam 503 color Die Kernspezifikationen der momentan eingesetzten Industriekamera DFK 23G445 (The Imaging Source Europe GmbH) aus dem Niedrigpreissegment im Vergleich mit der ZEISS Axiocam 503 color (Carl Zeiss Microscopy GmbH). DFK 23G445 Axiocam 503 color Belichtungszeit 1 µs – 30 s 250 µs – 60 s Sensor 1/3 “ 2/3 “ Auflösung 1280 x 960 Pixel 1936 x 1460 Pixel Dynamikbereich 8 bit 14 bit Bildrate 30 fps 38 fps Kühlung keine Peltierkühler Eine integrierte Kühlung vermindert das Bildrauschen. Nur dann ist auch eine Erhöhung des Dynamikbereichs sinnvoll. Bei hohem Rauschen kann ein großer Dynamikbereich nicht ausgenutzt werden. Je höher der effektive Dynamikbereich ist, desto besser können später die feinen Kontraste wahrgenommen werden. Je höher die Sensorfläche ist, desto größer ist der Bildausschnitt, was die Verfolgung einer größeren Anzahl an Bakterien je Bild ermöglicht. Ebenso können die Bakterien über eine längere Strecke verfolgt werden. Bei einer höheren Auflösung können Bildfilter besser angewandt werden. Durch eine höhere Bildrate sind die Bakterien von Bild zu Bild näher aneinander und die Wegverfolgung kann stabiler funktionieren. Der zukünftige Einsatz einer besseren Kamera ist empfehlenswert. Ebenso wie die Kamera steckt auch in der Verwendung eines besseren Mikroskops erhebliches Potential. Das verwendete Labormikroskop CX31 (Olympus) ist für die Lehre und für Routineanwendungen konzipiert. Mikroskope, die für Forschungsaufgaben konzipiert wurden, können durch eine bessere und robustere Optik kontrast- und detailreichere Abbildungen erzeugen. Für weitere Untersuchungen und zur Weiterentwicklung des Systems ist daher die Verwendung eines besseren Mikroskops empfehlenswert. Neben der Anwendung zur Untersuchung von B. amyloliquefaciens FZB42 kommen alle beweglichen Bakterienstämme für die Analyse in Frage. Mit E. coli und B. subtilis sind zwei für die Industrie und die Forschung hochinteressante Arten potentielle Kandidaten für das entwickelte Messsystem. Die Möglichkeit der Analyse anderer beweglicher Mikroorganismen wie z.B. bewegliche Algen (z.B. Chlamydomonas), Archaeen (z.B. Pyrococcus furiosus) oder Ciliata (z.B. Coleps sp.) ist ebenso denkbar. 92 Zusammenfassung 10 Zusammenfassung Im Rahmen der Arbeit wurde ein neuartiges, automatisiertes Messsystem zur Bestimmung der Durchschnittsgeschwindigkeit von sich aktiv bewegenden Mikroorganismen entwickelt. Die Probenentnahme, -vorbereitung und Messung sowie die Auswertung können vollautomatisch erfolgen. Als Beispielorganismus wird Bacillus amyloliquefaciens FZB42 in zwei verschiedenen Medien untersucht. Die Durchschnittsgeschwindigkeit der Bakterien wird in Intervallen von 15 bis 30 min während mehrerer Beispielfermentationen bestimmt. Das Messsystem arbeitet mit Hilfe einer eigens entwickelten Mikroskop-Durchflussküvette mit flexibler Schichtdicke. Dadurch ist es möglich, einen extrem geringen Fluidfilm von etwa 5 µm während der Bildaufnahme zu realisieren, um alle Mikroorganismen im Tiefenschärfebereich zu halten und eine Seitenansicht der einzelnen Bakterien zu garantieren. Da bei sehr engen Kanälen, wie hier der Fall, die Scherkräfte bei Durchströmung sehr hoch sind, muss die Schichtdicke während der Probenentnahme erheblich erweiterbar sein. Die auftretenden Kräfte bei Durchspülung der Küvette wurden berechnet und über Vergleich sichergestellt, dass die Geißeln nicht abreißen und das Bakterium dadurch geschädigt wird. Die mikroskopischen Aufnahmen werden anschließend in den programmierten Bildverarbeitungsalgorithmen automatisch ausgewertet. Neben der durchschnittlichen Geschwindigkeit der Bakterien kann auch die Anzahl der beweglichen Bakterien pro Bildserie bestimmt werden. Ein Monitoring von Durchschnittsgeschwindigkeit und Anzahl der beweglichen Bakterien pro Bild wird neben den gängigen Parametern pO2, pH-Wert, Temperatur und optische Dichte an den Beispielfermentationen durchgeführt. Weiterhin ist es mit dem Messsystem möglich, weitere mikroskopische Untersuchungen durchzuführen. Als Beispiel hierfür werden die typischen Bewegungsmuster von Bacillus amyloliquefaciens FZB42 in zwei verschiedenen Medien halbautomatisch ausgewertet. Erstmals liefert das neue Verfahren damit die Möglichkeit, die über die Geschwindigkeit korrelierte Aktivitäten von Bakterien zu monitoren und wertvolle Hinweise für einen optimal zu betreibenden Fermentationsprozess zu liefern. 93 Verzeichnisse 11 Verzeichnisse 11.1 Abkürzungen und Formelzeichen OD600 pO2 SEM 𝑆𝑆𝑆𝑣𝑣 SD 𝑆𝑆𝑣𝑣 𝑆𝑆𝐹 𝑛𝑣𝑣 𝑛𝑒𝑒 𝑓𝑛 𝑏𝑛 𝑝𝑘𝑘 𝑝𝑒𝑒 𝐴𝐵𝐵 𝐴𝐵 fps DNA DAPI FDA ex em UV CCD 2D 3D RGB HSL HSV HSI rpm cfu VE-Wasser 𝑢 𝑢𝑚 ̇ ) 𝛾̇ (Gamma 𝜏 (Tau) Optische Dichte bei einer Wellenlänge von 600 nm Sauerstoffsättigung in Prozent Standardfehler des arithmetischen Mittelwerts erwarteter Standardfehler der mittleren Geschwindigkeiten Standardabweichung erwartete Standardabweichung der mittleren Geschwindigkeiten erwartete Standardabweichung bei Fehlern in der Bildverarbeitung erwartete Anzahl an Einzelgeschwindigkeiten erwartete Anzahl fehlerhafter Zuweisungen zwischen nahen Bakterien Anzahl der Bilder pro Bildserie Anzahl an Bildserien pro Messpunkt Wahrscheinlichkeit für das Auftreten kleiner Strecken Wahrscheinlichkeit fehlerhafter Zuweisungen zwischen nahen Bakterien Fläche eines Bakteriums Fläche eines Bildes frames per second / Bilder pro Sekunde Desoxyribonukleinsäure 4‘,6-Diamidin-2-phenylindol Fluorescein-Diacetat Fluoreszenz-Absorptionswellenlänge Fluoreszenz-Emissionswellenlänge Ultraviolett charge-coupled device / ladungsgekoppeltes Bauteil zweidimensional dreidimensional Rot-Grün-Blau Hue(Farbwert)–Saturation(Farbsättigung)–Luminance(Farbhelligkeit) Hue-Saturation-Value(Helligkeit) Hue-Saturation-Intensity(Lichtintensität) revolutions per minute / Umdrehungen pro Minute colony forming unit / koloniebildende Einheit vollentsalztes Wasser Strömungsgeschwindigkeit [mm s-1] querschnittgemittelte Strömungsgeschwindigkeit [mm s-1] Strömungsgeschwindigkeitsgradient [mm s-1 mm-1] Schubspannung [Pa] 94 Verzeichnisse 𝐹 η (Eta) 𝑅𝑅 𝜈 (Ny) 𝑤(𝑧) 𝑧𝑅 𝜆 (Lambda) 𝜃0 (Theta0) 𝐴𝑁 𝑛𝑖 R2 𝑈𝐵 𝑣𝑂 𝑡𝐵 v vØ tp nmB E EØ I IØ ∝ 𝐴, 𝐵, … 𝑎𝑖𝑖 , 𝑏𝑖𝑖 , … (Scher)Kraft [N] dynamische Viskosität [Pa s] Reynolds-Zahl kinematischen Viskosität [mm2 s-1] Strahlradius [nm] Schärfentiefe [nm] Wellenlänge des Lichts [nm] halber Strahlöffnungswinkel [Grad] numerische Apertur Brechungsindex des Immersionsmediums Bestimmtheitsmaß Bewegungsunschärfe [µm s-1] Objektgeschwindigkeit [µm s-1] Belichtungszeit [ms] (Bakterien)Geschwindigkeit [µm s-1] Durchschnittsgeschwindigkeit (der Bakterien) [µm s-1] Fermentationszeitpunkt [min] Anzahl beweglicher Bakterien pro Bild elektrische Feldamplitude [V m-1] resultierende elektrische Feldamplitude [V m-1] Intensität resultierende Intensität proportional zu Matrix oder Array Element der Matrix A bzw. B an der Stelle i,j 11.2 Tabellenverzeichnis Tabelle 1: Übersicht von kommerziell verfügbaren In-Situ-Mikroskopen ................................ 9 Tabelle 2: Bekannte Schwimmgeschwindigkeiten von Bakterien ........................................... 16 Tabelle 3: Wandlungsmöglichkeiten von einem Farb- zu einem Grauwertbild ...................... 43 Tabelle 4: Eigenschaften des Standard-Nährmediums-I-¼ und ABiTEP-Mediums-¼ ........... 51 Tabelle 5: Strömungsverhältnisse im Messsystem................................................................... 52 Tabelle 6: Strömungsverhältnisse in den Messkapillaren bei Standard-Nährmedium-I-¼ ..... 54 Tabelle 7: Statistische Auswertung des Anteils der beweglichen Bakterien von Bacillus amyloliquefaciens FZB42 nach Durchströmung der Messkapilaren .............................. 54 Tabelle 8: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ ................................................................................................... 62 Tabelle 9: Vergleich der Geschwindigkeitsverteilungen für Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ ................................................................................................... 64 Tabelle 10: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in Standard-Nährmedium-I-¼ ......................................................................................... 68 95 Verzeichnisse Tabelle 11: Vergleich der Geschwindigkeitsverteilungen von Bacillus amyloliquefaciens FZB42 in Standard-Nährmedium-I-¼ ............................................................................ 71 Tabelle 12: Beständigkeit von Makrolon gegen einige Chemikalien ...................................... 90 Tabelle 13: Vergleich Kamera DFK 23G445 und ZEISS Axiocam 503 color ........................ 92 11.3 Abbildungsverzeichnis Abbildung 1: Physiologische Stadien einer Bakterienzelle ....................................................... 1 Abbildung 2: 3-Fold Dynamical Optical Reflectance Measurement (3D-ORM) ...................... 4 Abbildung 3: In-Situ-Auflichtfluoreszenzmikroskop mit theoretischem Probevolumen .......... 7 Abbildung 4: In-Situ-Mikroskop mit mechanisch definiertem Probevolumen .......................... 8 Abbildung 5: Immersions-Optik für die horizontale Online-Mikroskopie .............................. 10 Abbildung 6: Schema eines Systems zur Online-Mikroskopie mit Durchflussküvette ........... 11 Abbildung 7: Wechsel zwischen Bewegungsphasen von peritrich begeißelten Bakterien ...... 15 Abbildung 8: Beispiel für Run-and-Revers-Strategy ............................................................... 19 Abbildung 9: Kommunikation der Kernmodule eines 2D-Tracking Systems ......................... 21 Abbildung 10: Vereinfachtes Fließbild des Bioreaktors .......................................................... 26 Abbildung 11: Software Bedienoberfläche des Bioreaktors .................................................... 27 Abbildung 12: Vereinfachtes Verfahrensfließbild der Versuchsdurchführungen .................... 29 Abbildung 13: Konstruktionszeichnung der Durchflussküvette mit flexibler Schichtdicke.... 30 Abbildung 14: Schema der Durchflussküvette mit flexibler Schichtdicke .............................. 31 Abbildung 15: Strömungsraum in der Küvette (vereinfachte Darstellung) ............................. 32 Abbildung 16: Geometrie der berechneten schlanken Kanäle ................................................. 33 Abbildung 17: Vereinfachtes Verfahrensfließbild der Bildaufnahme mit Peripherie.............. 35 Abbildung 18: Vereinfachtes Fließbild der Probenvorbereitung ............................................. 36 Abbildung 19: Gauß-Strahl, der sich in z-Richtung ausbreitet ................................................ 38 Abbildung 20: Verteilung der elektrischen Feldamplitude zweier Lichtpunkte ...................... 40 Abbildung 21: Schematischer Programmablauf der Bildauswertung ...................................... 42 Abbildung 22: Vereinfachter Programmablaufplan für die Zuordnung der Kenndaten .......... 46 Abbildung 23: Ablauf der Ergebnisbereitstellung ................................................................... 48 Abbildung 24: Aufnahme aus dem Programm zur Erfassung der Bewegungsmuster ............. 49 Abbildung 25: Fließverhalten von Standard-Nährmedium-I-¼ und ABiTEP-Medium-¼ ...... 51 Abbildung 26: Geschwindigkeiten und Schubspannungen von Standard-Nährmedium-I-¼ und ABiTEP-Medium-¼ in der Durchflussküvette ............................................................... 53 Abbildung 27: Pixel-Auflösung in Bezug auf die Intensitätsverteilung .................................. 55 Abbildung 28: Standardfehler in Abhängigkeit der Anzahl an Bakterien pro Bild ................. 56 Abbildung 29: Vergleich der Grauwertwandlungen bei einer Mikroskopaufnahme von Bacillus amyloliquefaciens FZB42 ................................................................................. 57 Abbildung 30: Entwickelte Durchflussküvette ........................................................................ 58 Abbildung 31: Gesamtsystem für die Beispielfermentationen ................................................ 58 Abbildung 32: Validierung der automatischen Bildauswertung bei Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ ....................................................................................... 59 96 Verzeichnisse Abbildung 33: Kultivierung von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ bei 35 °C ......................................................................................................................... 61 Abbildung 34: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 in ABiTEP-Medium-¼ ....................................................................................... 63 Abbildung 35: OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien (Bacillus amyloliquefaciens FZB42) pro Bild in ABiTEP-Medium-¼ ......................................... 64 Abbildung 36: Histogramme der Einzelgeschwindigkeiten von Bacillus amyloliquefaciens FZB42 aus Fermentation 1 mit ABiTEP-Medium-¼ ..................................................... 65 Abbildung 37: Kultivierung von Bacillus amyloliquefaciens FZB42 in StandardNährmedium-I-¼ ............................................................................................................ 67 Abbildung 38: Charakteristische Fermentationszeitpunkte von Bacillus amyloliquefaciens FZB42 mit Standard-Nährmedium-I-¼ .......................................................................... 69 Abbildung 39: OD600 in Abhängigkeit der Anzahl der beweglichen Bakterien (Bacillus amyloliquefaciens FZB42) in Standard-Nährmedium-I-¼ ............................................. 70 Abbildung 40: Histogramme der Einzelgeschwindigkeiten von Bacillus amyloliquefaciens FZB42 aus Fermentation 6 mit Standard-Nährmedium-I-¼ .......................................... 72 Abbildung 41: Vergleich der Geschwindigkeiten von Bacillus amyloliquefaciens FZB42 im ABiTEP-Medium-¼ und Standard-Nährmedium-I-¼ .................................................... 73 Abbildung 42: Typische Bewegungsmuster von Bacillus amyloliquefaciens FZB42 ............. 74 Abbildung 43: Summarische Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 über den gesamten Probezeitraum ........................................ 75 Abbildung 44: Zeitliche Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 in Fermentation 6 (Standard-Nährmedium-I-¼) ................................................ 76 Abbildung 45: Zeitliche Verteilung der Bewegungsmuster von Bacillus amyloliquefaciens FZB42 in Fermentation 7 (ABiTEP-Medium-¼) ........................................................... 77 Abbildung 46: Diauxischer Kultivierungsverlauf von Bacillus cereus ................................... 79 Abbildung 47: Zeitlicher Kultivierungsverlauf von Bacillus amyloliquefaciens FZB42 auf ABiTEP-Medium-¼ ....................................................................................................... 80 Abbildung 48: Zeitlicher Kultivierungsverlauf von Bacillus amyloliquefaciens FZB42 auf Standard-Nährmedium-I-¼ ............................................................................................. 81 11.4 Literaturverzeichnis Abu-Absi, N. R.; Zamamiri, A.; Kacmar, J.; Balogh, S. J.; Srienc, F. (2003): Automated flow cytometry for acquisition of time-dependent population data. Cytometry 51 (2), S. 87– 96. Adler, J. (1966): Chemotaxis in Bacteria. Science 153 (3737), S. 708–716. Akin, M.; Prediger, A.; Yuksel, M.; Höpfner, T.; Demirkol, D. O.; Beutel, S.; Timur, S.; Scheper, T. (2011): A new set up for multi-analyte sensing: At-line bio-process monitoring. Biosensors and Bioelectronics 26 (11), S. 4532–4537. 97 Verzeichnisse Barbara, G. M.; Mitchell, J. G. (2003): Bacterial tracking of motile algae. FEMS Microbiology Ecology 44 (1), S. 79–87. Barrett, P.; Glennon, B. (2002): Characterizing the Metastable Zone Width and Solubility Curve Using Lasentec FBRM and PVM. Chemical Engineering Research and Design 80 (7), S. 799–805. Belini, V. L.; Wiedemann, P.; Suhr, H. (2013): In situ microscopy: A perspective for industrial bioethanol production monitoring. Journal of Microbiological Methods 93 (3), S. 224–232. Berg, H. C. (1971): How to Track Bacteria. Review of Scientific Instruments 42 (6), S. 868. Berg, H. C.; Brown, D. A. (1972): Chemotaxis in Escherichia coli analysed by Threedimensional Tracking. Nature 239 (5374), S. 500–504. Berg, H. C.; Anderson, R. A. (1973): Bacteria Swim by Rotating their Flagellar Filaments. Nature 245 (5425), S. 380–382. Berg, H. C. (1975): Chemotaxis in Bacteria. Annual Review of Biophysics and Bioengineering 4 (1), S. 119–136. Berg, H. C.; Purcell, E. M. (1977): Physics of chemoreception. Biophysical Journal 20 (2), S. 193–219. Berg, H. C.; Block, S. M. (1984): A Miniature Flow Cell Designed for Rapid Exchange of Media Under High-power Microscope Objectives. Microbiology 130 (11), S. 2915– 2920. Berg, H. C. (2003): The Rotary Motor of Bacterial Flagella. Annual Review of Biochemistry 72 (1), S. 19–54. Beveridge, O. S.; Petchey, O. L.; Humphries, S. (2010): Mechanisms of temperaturedependent swimming: the importance of physics, physiology and body size in determining protist swimming speed. The Journal of Experimental Biology 213 (Pt 24), S. 4223–4231. Bittner, C.; Wehnert, G.; Scheper, T. (1998): In situ microscopy for on-line determination of biomass. Biotechnology and Bioengineering, 60(1) S. 24–35. Bluma, A.; Höpfner, T.; Lindner, P.; Rehbock, C.; Beutel, S.; Riechers, D.; Hitzmann, B.; Scheper, T. (2010): In-situ imaging sensors for bioprocess monitoring: state of the art. Analytical and Bioanalytical Chemistry 398 (6), S. 2429–2438. Borriss, R.; Chen, X.-H; Rueckert, C.; Blom, J.; Becker, A.; Baumgarth, B.; Fan, B.; Pukall, R.; Schumann, P.; Sproer, C.; Junge, H.; Vater, J.; Puhler, A.; Klenk, H.-P. (2011): Relationship of Bacillus amyloliquefaciens clades associated with strains DSM 7T and FZB42T: a proposal for Bacillus amyloliquefaciens subsp. amyloliquefaciens subsp. nov. and Bacillus amyloliquefaciens subsp. plantarum subsp. nov. based on complete genome sequence comparisons. International Journal of Systematic and Evolutionary Microbiology 61 (8), S. 1786–1801. 98 Verzeichnisse Brognaux, A.; Bugge, J.; Schwartz, F. H.; Thonart, P.; Telek, S.; Delvigne, F. (2013): Realtime monitoring of cell viability and cell density on the basis of a three dimensional optical reflectance method (3D-ORM): investigation of the effect of sub-lethal and lethal injuries. Journal of Industrial Microbiology & Biotechnology 40 (7), S. 679–686. Bunin, V. D.; Ignatov, O. V.; Guliy, O. I.; Zaitseva, I. S.; O’Neil, D.; Ivnitski, D. (2004): Electrooptical analysis of the Escherichia coli–phage interaction. Analytical Biochemistry 328 (2), S. 181–186. Camisard, V.; Brienne, J.-P.; Baussart, H.; Hammann, J.; Suhr, H. (2002): Inline characterization of cell concentration and cell volume in agitated bioreactors using in situ microscopy: Application to volume variation induced by osmotic stress. Biotechnology and Bioengineering 78 (1), S. 73–80. Canny, J. (1986): A Computational Approach to Edge Detection. IEEE Transactions on Pattern Analysis and Machine Intelligence 8 (6), S. 679–698. Chance B.; Schoener B.; Oshino R.; Itshak F.; Nakase Y. (1979): Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. The Journal of Biological Chemistry 254(11), S.4764-4771 Chen, X.H; Scholz, R.; Borriss, M.; Junge, H.; Mögel, G.; Kunz, S.; Borriss, R. (2009): Difficidin and bacilysin produced by plant-associated Bacillus amyloliquefaciens are efficient in controlling fire blight disease. Journal of Biotechnology 140 (1-2), S. 38–44. Dalton, C.; Goater, A. D.; Pethig, R.; Smith, H. V. (2001): Viability of Giardia intestinalis Cysts and Viability and Sporulation State of Cyclospora cayetanensis Oocysts Determined by Electrorotation. Applied and Environmental Microbiology 67 (2), S. 586–590. Davey, H. M. (2011): Life, Death, and In-Between: Meanings and Methods in Microbiology. Applied and Environmental Microbiology 77 (16), S. 5571–5576. DePamphilis, M. L.; Adler, J. (1971): Purification of intact flagella from Escherichia coli and Bacillus subtilis. Journal of Bacteriology 105 (1), S. 376–383. Deutscher, M. P.; Kornberg, A. (1968): Biochemical studies of bacterial sporulation and germination. 8. Patterns of enzyme development during growth and sporulation of Bacillus subtilis. The Journal of Biological Chemistry 243 (18), S. 4653–4660. Duffy, D.; Cremin, N.; Napier, M.; Robinson, S.; Barrett, M.; Hao, Hongxun; Glennon, Brian (2012): In situ monitoring, control and optimization of a liquid–liquid phase separation crystallization. Chemical Engineering Science 77, S. 112–121. Enfors, S.-O.; Jahic, M.; Rozkov, A.; Xu, B.; Hecker, M.; Jürgen, B.; Krüger, E.; Schweder, T.; Hamer, G.; O'Beirne, D.; Noisommit-Rizzi, N.; Reuss, M.; Boone, L.; Hewitt, C.; McFarlane, C.; Nienow, A.; Kovacs, T.; Trägårdh, C.; Fuchs, L.; Revstedt, J.; Friberg, P.C; Hjertager, B.; Blomsten, G.; Skogman, H.; Hjort, S.; Hoeks, F.; Lin, H.-Y; Neubauer, P.; van der Lans, R.; Luyben, K.; Vrabel, P.; Manelius, Å. (2001): 99 Verzeichnisse Physiological responses to mixing in large scale bioreactors. Journal of Biotechnology 85 (2), S. 175–185. Foster, S. J.; Johnstone, K. (1990): Pulling the trigger: the mechanism of bacterial spore germination. Molecular Microbiology 4 (1), S. 137–141. Francis, N. R.; Sosinsky, G. E.; Thomas, D.; DeRosier, D. J. (1994): Isolation, Characterization and Structure of Bacterial Flagellar Motors Containing the Switch Complex. Journal of Molecular Biology 235 (4), S. 1261–1270. Frerichs, J.-G.; Joeris, K.; Konstantinov, K.; Scheper, T. (2002): Einsatz eines In-situMikroskops zur Online-Beobachtung von Tierzellkultivierungen. Chemie Ingenieur Technik 74 (11), S. 1629–1633. Frerichs, J.-G. (2000): Entwicklung eines In-situ-Mikroskops zur bildgestützten OnlineÜberwachung von Bioprozessen. Dissertation. Universität Hannover, Hannover. Fachbereich Chemie. Frymier, P. D.; Ford, R. M.; Berg, H. C.; Cummings, P. T. (1995): Three-dimensional tracking of motile bacteria near a solid planar surface. Proceedings of the National Academy of Sciences of the United States of America 92 (13), S. 6195–6199. Fukomoto, J. (1943a): Studies on the production of bacterial amylase. I. Isolation of bacteria secreting potent amylase and their distribution. Journal of the Agricultural Chemical Society of Japan (in Japanese) 19, S. 487–503. Fukomoto, J. (1943b): Studies on the production of bacterial amylase. II. Bacterial and physiological nature. Journal of the Agricultural Chemical Society of Japan (in Japanese) 19, S. 643–650. Gast, F.-U.; Dittrich, P. S.; Schwille, P.; Weigel, M.; Mertig, M.; Opitz, J.; Queitsch, U.; Diez, S.; Lincoln, B.; Wottawah, F.; Schinkinger, S.; Guck, J.; Käs, J.; Smolinski, J.; Salchert, K.; Werner, C.; Duschl, C.; Jäger, M. S.; Uhlig, K.; Geggier, P.; Howitz, S. (2006): The microscopy cell (MicCell), a versatile modular flowthrough system for cell biology, biomaterial research, and nanotechnology. Microfluid Nanofluid 2 (1), S. 21– 36. Gawad, S.; Schild, L.; Renaud, Ph. (2001): Micromachined impedance spectroscopy flow cytometer for cell analysis and particle sizing. Lab on a Chip 1, S. 76-82. Glagolev, A. N. (1980): Reception of the energy level in bacterial taxis. Journal of Theoretical Biology 82 (2), S. 171–185. Grossart, H. P.; Riemann, L.; Azam, F. (2001): Bacterial motility in the sea and its ecological implications. Aquatic Microbial Ecology 25, S. 247–258. Guez, J. S.; Cassar, J. Ph.; Wartelle, F.; Dhulster, P.; Suhr, H. (2004): Real time in situ microscopy for animal cell-concentration monitoring during high density culture in bioreactor. Journal of Biotechnology 111 (3), S. 335–343. 100 Verzeichnisse Guez, J. S.; Cassar, J. Ph.; Wartelle, F.; Dhulster, P.; Suhr, H. (2010): The viability of animal cell cultures in bioreactors: Can it be estimated online by using in situ microscopy? Process Biochemistry 45 (2), S. 288–291. Häder, D.-P.; Vogel, K. (1991): Simultaneous tracking of flagellates in real time by image analysis. Journal of Mathematical Biology 30 (1), S. 63–72. Hageman, J. H.; Shankweiler, G. W.; Wall, P. R.; Franich, K.; McCowan, G. W.; Cauble, S. M.; Grajeda, J.; Quinones, C. (1984): Single, chemically defined sporulation medium for Bacillus subtilis: growth, sporulation, and extracellular protease production. Journal of Bacteriology 160 (1), S. 438–441. Harshey, R. M. (2003): Bacterial Motility on a Surface. Many Ways to a Common Goal. Annual Review of Microbiology 57 (1), S. 249–273. Harwood, C. S.; Fosnaugh, K.; Dispensa, M. (1989): Flagellation of Pseudomonas putida and analysis of its motile behavior. Journal of Bacteriology 171 (7), S. 4063–4066. Henrichsen, J. (1972): Bacterial surface translocation: a survey and a classification. Bacteriological Reviews 36 (4), S. 478–503. Herwig, H. (Hg.) (2008): Strömungsmechanik. Einführung in die Physik von technischen Strömungen. Wiesbaden: Vieweg+Teubner, S. 163 – 169. Hewitt, C. J.; Nebe-Von Caron, G.; Nienow, A. W.; McFarlane, C. M. (1999a): The use of multi-parameter flow cytometry to compare the physiological response of Escherichia coli W3110 to glucose limitation during batch, fed-batch and continuous culture cultivations. Journal of Biotechnology 75 (2-3), S. 251–264. Hewitt, C. J.; Nebe-Von Caron, G.; Nienow, A. W.; McFarlane, C. M. (1999b): Use of multistaining flow cytometry to characterise the physiological state of Escherichia coli W3110 in high cell density fed-batch cultures. Biotechnology and Bioengineering 63 (6), S. 705–711. Höpfner, T.; Bluma, A.; Rudolph, G.; Lindner, P.; Scheper, T. (2010): A review of noninvasive optical-based image analysis systems for continuous bioprocess monitoring. Bioprocess and Biosystems Engineering 33 (2), S. 247–256. Hough, P. V. C. (1962): Method and means for recognizing complex patterns. Patentnummer: US3069654 A. Idris, E. E.; Iglesias, D. J.; Talon, M.; Borriss, R. (2007): Tryptophan-Dependent Production of Indole-3-Acetic Acid (IAA) Affects Level of Plant Growth Promotion by Bacillus amyloliquefaciens FZB42. Molecular Plant-Microbe Interactions 20 (6), S. 619–626. Idris, E. E.; Makarewicz, O.; Farouk, A.; Rosner, K.; Greiner, R.; Bochow, H.; Richter, T.; Borriss, R. (2002): Extracellular phytase activity of Bacillus amyloliquefaciens FZB45 contributes to its plant-growth-promoting effect. Microbiology (148), S. 2097–2109. Jarosch, R. (1967): Studien zur Bewegungsmechanik der Bakterien und Spirochäten des Hochmoores. Österreichische Botanische Zeitschrift 114 (3), S. 255–306. 101 Verzeichnisse Joeris, K.; Frerichs, J.-G.; Konstantinov, K.; Scheper, T. (2002): In-situ microscopy: Online process monitoring of mammalian cell cultures. Cytotechnology 38 (1/3), S. 129–134. Junker, B.; Maciejak, W.; Darnell, B.; Lester, M.; Pollack, M. (2007): Feasibility of an in situ measurement device for bubble size and distribution. Bioprocess and Biosystems Engineering 30 (5), S. 313–326. Junne, S.; Klein, E.; Angersbach, A.; Goetz, P. (2008): Electrooptical measurements for monitoring metabolite fluxes in acetone–butanol–ethanol fermentations. Biotechnology and Bioengineering 99 (4), S. 862–869. Junne, S.; Cruz-Bournazou, M. N.; Angersbach, A.; Götz, P. (2010): Electrooptical monitoring of cell polarizability and cell size in aerobic Escherichia coli batch cultivations. Journal of Industrial Microbiology & Biotechnology 37 (9), S. 935–942. Kang L.; Miller, E. D.; Weiss, L. E.; Campbell, P. G.; Kanade, T. (2006): Online Tracking of Migrating and Proliferating Cells Imaged with Phase-Contrast Microscopy. Proceedings of the 2006 Conference on Computer Vision and Pattern Recognition Workshop. New York City, IEEE Computer Society Conference, S. 65. Karim, Q. N.; Logan, R. P.; Puels, J.; Karnholz, A.; Worku, M. L. (1998): Measurement of motility of Helicobacter pylori, Campylobacter jejuni, and Escherichia coli by real time computer tracking using the Hobson BacTracker. Journal of Clinical Pathology 51 (8), S. 623–628. Kell, D. B.; Kaprelyants, A. S.; Weichart, D. H.; Harwood, C. R.; Barer, M. R. (1998): Viability and activity in readily culturable bacteria: a review and discussion of the practical issues. Antonie van Leeuwenhoek 73 (2), S. 169–187. Kell, D. B.; Markx, G. H.; Davey, C. L.; Todd, R. W. (1990): Real-time monitoring of cellular biomass: Methods and applications. Trends in Analytical Chemistry 9 (6), S. 190–194. Knöfel, R. (2002): Bewertung mikrobieller Kontaminationen in Prozeßwässern mit Hilfe von Farbklassifikation in Fluoreszenzmikroskopie-Bildern. Workshop Farbbildverarbeitung. Workshop Farbbildverarbeitung. Ilmenau, German Color Group Kougoulos, E.; Jones, A. G.; Jennings, K. H.; Wood-Kaczmar, M. W. (2005): Use of focused beam reflectance measurement (FBRM) and process video imaging (PVI) in a modified mixed suspension mixed product removal (MSMPR) cooling crystallizer. Journal of Crystal Growth 273 (3-4), S. 529–534. Krebs, B.; Höding, B.; Kübart, S.; Workie, M. A.; Junge, H.; Schmiedeknecht, G.; Grosch, R.; Bochow, H.; Hevesi, M. (1998): Use of Bacillus subtilis as biocontrol agent. I. Activities and characterization of Bacillus subtilis strains. Zeitschrift für Pflanzenkrankheiten und Pflanzenschutz 105, S. 181–197. Kudo, S.; Imai, N.; Nishitoba, M.; Sugiyama, S.; Magariyama, Y. (2005): Asymmetric swimming pattern of Vibrio alginolyticus cells with single polar flagella. FEMS Microbiology Letters 242 (2), S. 221–225. 102 Verzeichnisse Lowe, G.; Meister, M.; Berg, H. C. (1987): Rapid rotation of flagellar bundles in swimming bacteria. Nature 325 (6105), S. 637–640. MacLennan, F.; Kowalski, B. R. (Hg.) (1995): Process analytical chemistry. Springer Netherlands, S. 1-13. Macnab, R. M.; Koshland, D. E. (1972): The gradient-sensing mechanism in bacterial chemotaxis. Proceedings of the National Academy of Sciences of the United States of America 69 (9), S. 2509–2512. Macnab, R. M. (2003): How Bacteria Assemble Flagella. Annual Review of Microbiology 57 (1), S. 77–100. Macnab, R. M.; Ornston, M. K. (1977): Normal-to-curly flagellar transitions and their role in bacterial tumbling. Stabilization of an alternative quaternary structure by mechanical force. Journal of Molecular Biology 112 (1), S. 1–30. Maeda, K.; Imae, Y.; Shioi, J. I.; Oosawa, F. (1976): Effect of temperature on motility and chemotaxis of Escherichia coli. Journal of Bacteriology 127 (3), S. 1039–1046. Magariyama, Y.; Sugiyama, S.; Muramoto, K.; Maekawa, Y.; Kawagishi, I.; Imae, Y.; Kudo, S. (1994): Very fast flagellar rotation. Nature 371 (6500), S. 752. Magariyama, Y.; Ichiba, M.; Nakata, K.; Baba, K.; Ohtani, T.; Kudo, S.; Goto, T. (2005): Difference in Bacterial Motion between Forward and Backward Swimming Caused by the Wall Effect. Biophysical Journal 88 (5), S. 3648–3658. Mann, H. B.; Whitney, D. R. (1947): On a Test of Whether one of Two Random Variables is Stochastically Larger than the Other. The Annals of Mathematical Statistics 18 (1), S. 50–60. Mattick, J. S. (2002): Type IV pili and twitching motility. Annual Review of Microbiology 56 (1), S. 289–314. Meinhart, C. D.; Wereley, S. T.; Santiago, J. G. (1999): PIV measurements of a microchannel flow. Experiments in Fluids 27 (5), S. 414–419. Mendelson, N. H.; Bourque, A.; Wilkening, K.; Anderson, K. R.; Watkins, J. C. (1999): Organized Cell Swimming Motions in Bacillus subtilis Colonies: Patterns of ShortLived Whirls and Jets. Journal of Bacteriology 181 (2), S. 600–609. Mesquita, D. P.; Amaral, A.L; Ferreira, E. C. (2011): Characterization of activated sludge abnormalities by image analysis and chemometric techniques. Analytica Chimica Acta 705 (1-2), S. 235–242. Miller, J. B.; Koshland, D. E. (1977): Sensory electrophysiology of bacteria: relationship of the membrane potential to motility and chemotaxis in Bacillus subtilis. Proceedings of the National Academy of Sciences of the United States of America 74 (11), S. 4752– 4756. 103 Verzeichnisse Müller, S.; Nebe-von-Caron, G. (2010): Functional single-cell analyses: flow cytometry and cell sorting of microbial populations and communities. FEMS Microbiology Reviews 404, S. 1211–1237. Nakata, H. M.; Halvorson, H. O. (1960): Biochemical changes occurring during growth and sporulation of Bacillus cereus. Journal of Bacteriology 80, S. 801–810. Näther, D. (2007): Untersuchung der Flagellen von Pyrococcus furiosus. Dissertation. Universität Regensburg, Regensburg. Naturwissenschaftlichen Fakultät III. Netuschil, L. (1983): Vitalfärbung von Plaque-Mikroorganismen mit Fluoresceindiacetat und Ethidiumbromid. Deutsche zahnärztliche Zeitschrift 38 (10), S. 914–917. O'Sullivan, B.; Barrett, P.; Hsiao, G.; Carr, A.; Glennon, B. (2003): In Situ Monitoring of Polymorphic Transitions. Organic Process Research & Development 7 (6), S. 977–982. O'Sullivan, B.; Glennon, B. (2005): Application of in Situ FBRM and ATR-FTIR to the Monitoring of the Polymorphic Transformation of d -Mannitol. Organic Process Research & Development 9 (6), S. 884–889. Pearson, A. P.; Glennon, B.; Kieran, P. M. (2003): Comparison of morphological characteristics of Streptomyces natalensis by image analysis and focused beam reflectance measurement. Biotechnology Progress 19 (4), S. 1342–1347. Pedrotti, F. L.; Pedrotti, L. S.; Bausch, W.; Schmidt, H. (2005): Optik für Ingenieure. Grundlagen. 3., bearbeitete und aktualisierte Auflage. Berlin, Heidelberg: SpringerVerlag Berlin Heidelberg, S. 459 – 480, S. 641 – 665. Ping, L. (2012): Cell orientation of swimming bacteria: From theoretical simulation to experimental evaluation. Science China Life Sciences 55 (3), S. 202–209. Pons, M. N.; Vivier, H.; Remy, J. F.; Dodds, J. A. (1993): Morphological characterization of yeast by image analysis. Biotechnology and Bioengineering 42 (11), S. 1352–1359. Prediger, A. (2013): Entwicklung von In-situ- und Durchfluss-Mikroskopen für die Bioprozesstechnik. Dissertation. Universität Hannover, Hannover. TCI. Priest, F. G.; Goodfellow, M.; Shute, L. A.; Berkeley, R. C. W. (1987): Bacillus amyloliquefaciens sp. nov., nom. rev. International Journal of Systematic Bacteriology 37 (1), S. 69–71. Qu, H.; Louhi-Kultanen, M.; Kallas, J. (2006): In-line image analysis on the effects of additives in batch cooling crystallization. Journal of Crystal Growth 289 (1), S. 286– 294. Rehbock, C.; Riechers, D.; Höpfner, T.; Bluma, A.; Lindner, P.; Hitzmann, B.; Beutel, S.; Scheper, T. (2010): Development of a flow-through microscopic multitesting system for parallel monitoring of cell samples in biotechnological cultivation processes. Journal of Biotechnology 150 (1), S. 87–93. 104 Verzeichnisse Rehmer, O. (2008): Detektion und Visualisierung von Mikroorganismen in der In-situPellikel mit verschiedenen Verfahren. Dissertation. Universität Freiburg, Freiburg. Medizinische Fakultät. Ren, J.; Reid, J. F.; Litchfield, J. B. (1994): Knowledge-based supervision and control of bioprocess with a machine vision-based sensing system. Journal of Biotechnology 36 (1), S. 25–34. Ross, J.; Boon, P. I.; Sharma, R.; Beckett, R. (1996): Variations in the fluorescence intensity of intact DAPI-stained bacteria and their implications for rapid bacterial quantification. Letters in Applied Microbiology 22 (4), S. 283–287. Rosu, V.; Hughes, K. T. (2006): 28-Dependent Transcription in Salmonella enterica is independent of Flagellar Shearing. Journal of Bacteriology 188 (14), S. 5196–5203. Rudolph, G.; Brückerhoff, T.; Bluma, A.; Korb, G.; Scheper, T. (2007): Optische InlineMessverfahren zur Zellzahl- und Zellgrößenbestimmung in der Bioprozesstechnik. Chemie Ingenieur Technik 79 (1-2), S. 42–51. Schmitt, R. (1972): Bakteriengeißeln: Bewegliche Biopolymere. Biologie in unserer Zeit 2 (3), S. 82–91. Schmitt, R. (1997): Molekulare Propeller: Bakteriengeißeln und ihr Antrieb. Biologie in unserer Zeit 27 (1), S. 40–47. Shigematsu, M.; Meno, Y.; Misumi, H.; Amako, K. (1995): The Measurement of Swimming Velocity of Vibrio cholerae and Pseudomonas aeruginosa using the Video Tracking Method. Microbiology and Immunology 39 (10), S. 741–744. Shoesmith, J. G. (1960): The Measurement of Bacterial Motility. Microbiology 22 (2), S. 528–535. Silverman, M.; Simon, M. (1974): Flagellar rotation and the mechanism of bacterial motility. Nature 249 (5452), S. 73–74. Singh, A.; Pyle, B. H.; McFeters, G. A. (1989): Rapid enumeration of viable bacteria by image analysis. Journal of Microbiological Methods 10 (2), S. 91–101. Stewart, B. T.; Halvorson, H. O. (1953): Studies on the spores of aerobic bacteria. I. The occurrence of alanine racemase. Journal of Bacteriology 65 (2), S. 160–166. Stocker, B. A. D.; Campbell, J. C. (1959): The Effect of Non-Lethal Deflagellation on Bacterial Motility and Observations on Flagellar Regeneration. Journal of General Microbiology 20 (3), S. 670–685. Stülke, J.; Hillen, W. (1999): Carbon catabolite repression in bacteria. Current Opinion in Microbiology 2 (2), S. 195–201. Suhr, H. (2004): In Situ Mikroskopie an schwebenden Mikropartikeln, insbesondere in gerührten Bioreaktoren. Horizonte 24, S. 38–43. Suhr, H.; Volz, J.; Hammann, J. (1998): In Situ Mikroskopie. BIOforum 21, S. 595–600. 105 Verzeichnisse Suhr, H.; Wehnert, G.; Schneider, K.; Bittner, C.; Scholz, T.; Geissler, P.; P.; Jähne, B.; Scheper, T. (1995): In situ microscopy for on-line characterization of cell-populations in bioreactors, including cell-concentration measurements by depth from focus. Biotechnology and Bioengineering 47 (1), S. 106–116. Surek, D.; Stempin, S. (Hg.) (2007): Angewandte Strömungsmechanik. Für Praxis und Studium. Wiesbaden: Vieweg+Teubner, S. 71 - 106. Thomas, C. R.; Paul, G. C. (1996): Applications of image analysis in cell technology. Current Opinion in Biotechnology 7 (1), S. 35–45. Tucker, K. G.; Chalder, S.; Al-Rubeai, M.; Thomas, C. R. (1994): Measurement of hybridoma cell number, viability, and morphology using fully automated image analysis. Enzyme and Microbial Technology 16 (1), S. 29–35. Vater, S. M.; Weiße, S.; Maleschlijski, S.; Lotz, C.; Koschitzki, F.; Schwartz, T.; Obst, U.; Rosenhahn, A.; van Veen, H. W. (2014): Swimming Behavior of Pseudomonas aeruginosa Studied by Holographic 3D Tracking. PLoS ONE 9(1). Vater, S. M. (2014): Holographische Motilitätanalyse des Schwimm- und Besiedlungsverhaltens flagellierter Mikroorganimsmen. Dissertation. Ruprecht-Karls Universität, Heidelberg. Mathematisch-Naturwissenschaftliche Gesamtfakultät. Vigeant, M. A.; Ford, R. M. (1997): Interactions between motile Escherichia coli and glass in media with various ionic strengths, as observed with a three-dimensional-tracking microscope. Applied and Environmental Microbiology 63 (9), S. 3474–3479. Voss, K.-O.; Fischer, B. E.; Jakob, B.; Merk, B. (2012): Immersion Optics for the Horizontal Online Microscope at the GSI Microbeam. GSI SCIENTIFIC REPORT 2011, S. 425. Wiedemann, P..; Guez, J. S.; Wiegemann, H. B.; Egner, F.; Quintana, J. C.; AsanzaMaldonado, D.; Filipaki, M.; Wilkesman, J.; Schwiebert, C.; Cassar, J. P.; Dhulster, P.; Suhr, H. (2011): In situ microscopic cytometry enables noninvasive viability assessment of animal cells by measuring entropy states. Biotechnology and Bioengineering 108 (12), S. 2884–2893. Wilcoxon, F. (1945): Individual comparisons by ranking methods. Biometrics 1 (6), S. 80–83. Witman, G. B. (1975): The site of in vivo assembly of flagellar microtubules. Annals of the New York Academy of Sciences 253 (1), S. 178–191. Wu, M.; Roberts, J. W.; Buckley, M. (2005): Three-dimensional fluorescent particle tracking at micron-scale using a single camera. Experiments in Fluids 38 (4), S. 461–465. Wu, M.; Roberts, J. W.; Kim, S.; Koch, D. L.; DeLisa, M. P. (2006): Collective Bacterial Dynamics Revealed Using a Three-Dimensional Population-Scale Defocused Particle Tracking Technique. Applied and Environmental Microbiology 72 (7), S. 4987–4994. Zalewski, K.; Buchholz, R. (1996): Morphological analysis of yeast cells using an automated image processing system. Journal of Biotechnology 48 (1-2), S. 43–49. 106 Verzeichnisse Zhang, W.; Phillips, G. N. (2003): Structure of the Oxygen Sensor in Bacillus subtilis. Structure 11 (9), S. 1097–1110. Zotta, T.; Guidone, A.; Tremonte, P.; Parente, E.; Ricciardi, A. (2012): A comparison of fluorescent stains for the assessment of viability and metabolic activity of lactic acid bacteria. World Journal of Microbiology and Biotechnology 28 (3), S. 919–927. 107 Anhang Anhang LabVIEW-Unterprogramm „Wegverfolgung“ 108 Anhang 109 Anhang Statistische Analyse von Fermentation 1 110 Anhang Statistische Analyse von Fermentation 2 111 Anhang Statistische Analyse von Fermentation 3 112 Anhang Statistische Analyse von Fermentation 4 113 Anhang Statistische Analyse von Fermentation 5 114 Anhang Statistische Analyse von Fermentation 6 115 Anhang Fermentation 6 – Verteilung der Bewegungsmuster Minut e 120 135 150 165 195 210 225 240 260 275 290 305 320 Orientatio Undulatio Straigh Tumbl Orientatio Curv Collisio Anothe n n t path e n and Stop e n r 0 0 1 0 0 0 0 0 0 0 1 0 1 0 0 0 1 1 6 1 0 0 0 0 1 0 1 1 1 0 0 0 1 1 15 1 2 1 1 3 2 2 18 2 1 2 1 0 7 0 16 2 0 3 7 2 2 0 8 1 0 0 1 0 2 1 17 6 2 0 4 2 3 0 5 0 0 0 1 0 1 0 4 0 0 1 1 1 1 0 3 0 0 2 0 0 1 0 10 0 0 0 0 0 Fermentation 7 – Verteilung der Bewegungsmuster Minut e 210 225 240 255 270 285 305 325 345 380 405 425 445 Orientatio n 0 0 0 0 2 0 0 2 2 4 0 0 0 Undulatio n 0 0 0 0 0 0 1 1 0 1 0 0 0 Straigh t path 0 2 4 3 7 8 7 9 11 8 2 2 0 Tumbl e 1 0 3 1 1 6 8 7 6 2 0 0 0 Orientatio n and Stop 0 0 1 1 0 2 1 0 0 1 0 0 0 Curv e 0 0 0 0 1 0 1 3 4 0 0 0 0 Collisio n 1 1 0 2 3 7 3 1 4 7 1 1 1 Anothe r 0 0 0 0 0 1 0 0 6 2 2 0 0 116 Anhang Vergleich der Wandlung in Grauwerte 117 Anhang 118 Anhang 119