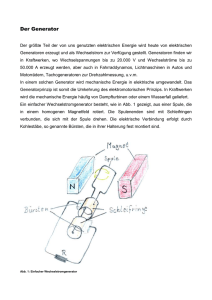
Lokalisierung und funktionelle Charakterisierung von
Werbung

Lokalisierung und funktionelle Charakterisierung von Interaktionsdomänen der EGR-Zinkfinger-Proteine Dissertation zur Erlangung des akademischen Grades doctor rerum naturalium (Dr. rer. nat.) vorgelegt dem Rat der Biologisch-Pharmazeutischen Fakultät der Friedrich-Schiller-Universität Jena von Dipl. Biol. Nina Nehmann, geboren am 13. Dezember 1974 in Hamburg Gutachter: Herr Professor. Dr. Peter F. Zipfel Herr Professor Dr. Edgar Serfling Herr Professor Dr. Frank Große Tag der Verteidigung: 23. Februar 2004 Inhaltsverzeichnis Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis IV Tabellenverzeichnis V Abkürzungen VI Zusammenfassung VIII 1 Einleitung 1 1.1 Das Immunsystem 1 1.1.1 Zellen des Immunsystems 1 1.1.2 Mediatoren des Immunsystems 2 1.2 Entzündungsreaktion als Beispiel einer Immunantwort 4 1.3 Transkriptionsfaktoren als Regulatoren induzierbarer Genexpression 6 1.4 „Immediate early“-Gene 8 1.5 „Early growth response”-Proteine 9 1.5.1 EGR-Proteinstruktur 11 1.5.2 Regulation der EGR-Proteine 12 1.5.3 Funktionen von EGR-Proteinen 16 1.6 Die NFAT-Proteine 17 1.7 Die NFκB-Proteine 19 1.8 Fragestellung 21 2. Material und Methoden 22 2.1 Allgemein verwendete Materialien, Puffer, Medien und Lösungen 22 2.2 DNA-Arbeitstechniken zur Herstellung von Expressionsvektoren 23 2.2.1 Polymerase-Kettenreaktion (PCR) 24 2.2.2 Restriktion und Ligation von DNA 26 2.2.3 Herstellung und Transformation kompetenter Bakterienzellen 27 2.2.4 Plasmidpräparation 28 2.2.5 DNA-Sequenzierung 29 2.3 Expression eukaryotischer und prokaryotischer Proteine 2.3.1 Zellkultur und Expression eukaryotischer Proteine 2.3.1.1 Transiente Transfektion und Luciferase-Reportergenassay -I- 30 30 30 Inhaltsverzeichnis 2.3.1.2 Expression nativer EGR-Proteine 32 2.3.1.3 Expression rekombinanter EGR-Proteine 32 2.3.2 Extraktion eukaryotischer Proteine 33 2.3.3 Affinitätschromatographie zur Aufreinigung rekombinanter EGR-Proteine 34 2.3.4 Expression prokaryotischer Proteine 35 2.3.5 Affinitätschromatographien zur Aufreinigung bakterieller Proteine 35 2.4 Proteinchemische Methoden 36 2.4.1 SDS-PAGE (Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese) 36 2.4.2 Proteinnachweis durch Silberfärbung 37 2.4.3 Proteintransfer 38 2.4.4 Immunodetektion membrantransferierter Proteine (Westernblot) 38 2.4.5 in vitro-Proteinbindungsassay 39 2.4.6 Peptidspotanalyse 40 2.4.7 Plasmonresonanz-Studie 41 3. Ergebnisse 42 3.1 Expression rekombinanter und nativer Proteine 42 3.1.1 Expression rekombinanter und nativer EGR-Proteine 42 3.1.2 Expression rekombinanter und nativer NFAT- und NFκB-Proteine 44 3.2 Interaktion von EGR- und NFAT-Proteinen 44 3.2.1 Interaktion von EGR mit immobilisierten NFAT-Proteinen 45 3.2.2 Interaktion von NFAT mit immobilisierten EGR-Proteinen 47 3.3 Salzeffekt bei EGR-Interaktionen 49 3.4 Charakterisierung der EGR/NFAT-Interaktionen mit der Plasmonresonanz 51 3.5 Lokalisierung von EGR-3-Interaktionsdomänen 54 3.5.1 Expression von EGR-3-Deletionsmutanten 54 3.5.2 Zwei Domänen von EGR-3 vermitteln die Interaktion mit NFAT 55 3.5.3 Interaktion von EGR-3-Deletionsmutanten mit der Plasmonresonanz 56 3.6 Lokalisierung von EGR-4-Interaktionsdomänen 57 3.6.1 Expression von EGR-4-Deletionsmutanten 58 3.6.2 Zwei Domänen von EGR-4 vermitteln die Interaktion mit NFAT 59 3.6.3 Interaktionen von EGR-4-Deletionsmutanten mit der Plasmonresonanz 60 - II - Inhaltsverzeichnis 3.6.4 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit NFATp 61 3.6.5 EGR-4/NFAT-Interaktion vermittelnde Aminosäuren 62 3.6.6 Inhibition der EGR-4/NFAT-Interaktion 65 3.7 Interaktion von EGR- und NFκB-Proteinen 66 3.7.1 Interaktion von EGR mit immobilisierten NFκB-Proteinen 67 3.7.2 Interaktion von NFκB mit immobilisierten EGR-Proteinen 68 3.7.3 EGR/NFκB-Interaktionen mit der Plasmonresonanz 69 3.7.4 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit p65 71 3.7.5 EGR/NFκB-Interaktion vermittelnde Aminosäuren 72 3.7.6 Inhibition der EGR/NFκB-Interaktion 74 3.8 Lokalisierung von Bindungsdomänen in NFAT- und NFκB-Proteinen 75 4. Diskussion 77 4.1 Expression der EGR-Proteine 77 4.2 Interaktion von EGR- und NFAT-Proteinen 79 4.2.1 Salzeffekt bei EGR-Interaktionen 80 4.2.2 Charakterisierung der EGR/NFAT-Interaktionen mit der Plasmonresonanz 81 4.3 Lokalisierung von EGR-Interaktionsdomänen für die NFAT-Interaktion 82 4.3.3 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit NFATp 83 4.3.4 EGR-4/NFAT-Interaktion vermittelnde Aminosäuren 84 4.4 Interaktion von EGR- und NFκB-Proteinen 86 4.4.1 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit p65 87 4.4.2 EGR/NFκB-Interaktion vermittelnde Aminosäuren 88 4.5 Lokalisierung von Bindungsmotiven in NFAT- und NFκB-Proteinen 89 4.6 Strukturelle Domänen von EGR-4 89 5 Literaturverzeichnis 91 Publikationen i Selbständigkeitserklärung ii Lebenslauf iii - III - Abbildungsverzeichnis Abbildungsverzeichnis Abb. 1: Auslösung einer Entzündungsantwort 6 Abb. 2: Regulatorische Elemente von IL-2- und TNF-α-Promotoren 7 Abb. 3: Struktur von EGR-Proteinen 10 Abb. 4: Nachweis rekombinanter und nativer EGR-Proteine 43 Abb. 5: Nachweis rekombinanter und nativer NFAT- und NFκB-Proteine 44 Abb. 6: Immobilisierung von NFAT-Proteinen und EGR-4-Proteinbindungsassay 46 Abb. 7: Bindung von EGR-3 und EGR-4 an immobilisierte NFAT-Proteine 47 Abb. 8: Immobilisierung von EGR-Proteinen 48 Abb. 9: Bindung von NFATc und NFATp an immobilisierte EGR-Proteine 49 Abb. 10: Salzeffekt bei der EGR-Interaktionen 51 Abb. 11: Untersuchung der Proteininteraktion mit der Plasmonresonanz 52 Abb. 12: Bestimmung der NFAT/EGR-Interaktionen mit der Plasmonresonanz 53 Abb. 13: Bestimmung der EGR/NFAT-Interaktionen mit der Plasmonresonanz 54 Abb. 14: Struktur und Expression der EGR-3-Deletionsmutanten 55 Abb. 15: Interaktion von EGR-3-Deletionsmutanten und NFAT-Proteinen 56 Abb. 16: Bestimmung der Interaktion von EGR-3-Deletionsmutanten mit der Plasmonresonanz 57 Abb. 17: Struktur und Expression der EGR-4-Deletionsmutanten 58 Abb. 18: Interaktion von EGR-4-Deletionsmutanten und NFAT-Proteinen 59 Abb. 19: Bestimmung der Interaktion von EGR-4-Deletionsmutanten mit der Plasmonresonanz 60 Abb. 20: Funktionale Interaktion von EGR-4 und NFATp 62 Abb. 21: EGR-4/NFAT-Interaktion vermittelnde Aminosäurebereiche 63 Abb. 22: EGR-4/NFAT-Interaktion vermittelnde Aminosäuren 65 Abb. 23: Einfluss eines inhibitorischen Peptids auf die EGR-4/NFATc-Interaktion 66 Abb. 24: Immobilisierung der NFκB-Proteine p50 und p65 67 Abb. 25: Bindung von EGR-3 und EGR-4 an immobilisierte NFκB-Proteine 68 Abb. 26: Bindung von p50 und p65 an immobilisierte EGR-Proteine 69 Abb. 27: Bestimmung der NFκB/EGR-Interaktionen mit der Plasmonresonanz 70 Abb. 28: Funktionale Interaktion von EGR-4 und p65 71 Abb. 29: EGR/NFκB-Interaktion vermittelnde Aminosäurebereiche 73 Abb. 30: Einfluss eines inhibitorischen Peptids auf die EGR-4/p65-Interaktion 75 Abb. 31: Domänen von NFAT- und NFκB-Proteinen für die Interaktion mit EGR-4 76 Abb. 32: Interaktionsdomänen und transkriptionell regulative Domänen von EGR-4 90 - IV - Tabellenverzeichnis Tabellenverzeichnis Tab. 1: Übersicht von Transkriptionsfaktoren bei Entzündungsprozessen 8 Tab. 2: Eukaryotische Expressionsplasmide für EGR-Proteine 23 Tab. 3: Bakterielle Expressionsplasmide für NFAT- und NFκB-Proteine 23 Tab. 4: Bakterielle Expressionsplasmide für deletionale EGR-Proteine 24 Tab. 5: PCR-Primer 25 Tab. 6: Reporterplasmide für die transiente Zelltransfektion 31 Tab. 7: Expressionsplasmide für die transiente Zelltransfektion 31 Tab. 8: Primäre und sekundäre Antikörper 39 Tab. 9: EGR-4/NFAT-Interaktion vermittelnde Aminosäurebereiche 63 Tab. 10: Vergleich der EGR-4/NFAT-Interaktion vermittelnden Aminosäuren 65 Tab. 11: Vergleich der EGR/p65-Interaktion vermittelnden Aminosäuren 74 Tab. 12: Bindungsmotive in NFAT und NFκB-Proteinen für die Interaktion mit EGR-4 76 -V- Abkürzungsverzeichnis Abkürzungsverzeichnis A ADP Amp AP-1 APS BCR bHLH bp BSA C cAMP CBP CD CRE CREB CsA CSF Da ddH2O DNA dNTP DTT E. coli E3/I-IV E4/I-X EDTA EGF EGR FCS FGF g G G-CSF GF GM-CSF GST HEPES HLA IκB ICAM-1 IE IFN Ig IKK IL IPTG IRE IRF-1 kb kDa Krox LB LFA-1 LH-β LPS MAP MCP MCS M-CSF Adenosin Adenosindiphosphat Ampizillin Aktivatorprotein 1 Ammoniumpersulfat B-Lymphozytenantigenrezeptor basische „Helix-Loop-Helix“ Basenpaar Rinderserumalbumin (bovine serum albumine) Cytosin zyklisches Adenosindiphosphat CREB-bindendes Element „cluster of differentiation“, „cluster determinant“, Differenzierungsantigen „cAMP response“-Element CRE-bindendes Protein Cyclosporin A Kolonie-stimulierender Faktor (colony-stimulating factor) Dalton doppelt destilliertes Wasser Desoxyribonukleinsäure Desoxyribonukleosidtriphosphat Dithiothreitol Escherichia coli EGR-3-Deletionsmutanten 1-4 EGR-4-Deletionsmutanten 1-4 Ethylendiamintetraessigsäure Wachstumsfaktor der Epidermis (epidermal growth factor) „early growth response” fötales Kälberserum (fetal calf serum) Fibroblasten-Wachstumsfaktor (fibroblast growth factor) Erdbeschleunigung Guanosin Granulozyten-CSF growth-Faktor Granulozyten/Makrophagen-CSF Glutathion-S-Transferase N-2-Hydroxyethylpiperazin-N´-2-Ethansulfonsäure humanes Leukozytenantigen Inhibitor von NFκB Interzelluläres-Adhäsionsmolekül 1 „immediate early“ Interferon Immunglobulin IκB-Kinase Interleukin Isopropyl-β-D-thiogalactopyranosid „interferon response“-Element „interferon responsive“-Faktor 1 Kilobasenpaare Kilodalton Krüppelbox „Luria Bertani“-Kulturmedium Lymphozyten-Funktions-assoziiertes Antigen 1 Luteinisierendes Hormon Lipopolysaccharid mitogen-aktivierte Proteinkinase Monozyten-chemotaktisches Protein Multiple Klonierungsstelle (multiple cloning site) Makrophagen-CFS - VI - Abkürzungsverzeichnis MHC MIP MMP MOI NAB NFκB NFAT NGF NGFI NHR NK-Zellen OD ori PAGE PBS PCR PDGF PHA PKC PMA PMSF RANTES RLU RNA rpm RPMI RSD RT RU SDS SDS-PAGE SF-1 Sf9 SRE STAT T TAF TBE TBS TBST TEMED TGF TNF TRAF Tris U UV v/v VCAM w/v X-Gal zif268 ZIP Haupt-Histokompatibilitätskomplex (most histocompatibility complex) Makrophagen inflammatorisches Protein Matrixmetalloprotein Infektionsrate (multiplicity of infection) NGFI-A bindendes Protein Nuklearer Faktor kappa B Nuklearer Faktor von aktivierten T-Zellen „nerve growth”-Faktor NGF-induziertes Protein NFAT-Homologie-Region natürliche Killerzellen Optische Dichte Replikationsstartpunkt (origin of replication) Polyacrylamidgel-Elektrophorese Phosphat-gepufferte Salzlösung (phosphat buffered saline) Polymerase-Kettenreaktion (polymerase chain reaction) „platelet-derived growth“-Faktor Phytohämagglutinin Proteinkinase C Phorbol 12-myristat 13-acetat Phenylmethylsulfonylfluorid reguliert durch Aktivierung normalerweise in T-Zellen exprimiert und sekretiert Relative Licht Einheiten für Lumineszenz (relative light units) Ribonucleinsäure Umdrehungen pro Minute (rounds per minute) „Rosewell Park Memorial Institute“-Zellkulturmedium „Rel-Similarity“-Domäne Raumtemperatur relative Resonanzeinheit Natriumdodecylsulfat (sodiumdodecylsulfat) Natriumdodecylsulfat Polyacrylamidgel-Elektrophorese steroidogene Faktor 1 Spodoptera fruiperda 9 (Ovarzellen von Insektenzellen) „serum-response“-Element Signaltransduktor und Aktivatoren der Transkription Thymin „TATA-box“-assoziierter Faktor Tris-Borat-EDTA-Lösung Tris-gepufferte Salzlösung (tris buffered saline) Tween enthaltende TBS-Lösung N, N, N´, N´-Tetramethylethylendiamin „transforming growth“-Faktor Tumornekrosefaktor TNF-assoziierter Faktor Tris-(hydroxymethyl)-aminomethan Unit (Enzymeinheit) Ultraviolettes Licht Volumen/Volumen (Volumenprozent) vaskuläres Zelladhäsionsmolekül weight/volume (Massenprozent) 5-Bromo-4-Chloro-3-Inodyl-β-D-galaktosid Zinkfingerklon 268 Zinkfingerprotein-Binderegion - VII - Zusammenfassung Zusammenfassung Die Mitglieder der EGR-Familie werden durch eine Vielzahl unterschiedlicher Stimuli in diversen Zellen des Immunsystems exprimiert und sind an der induzierbaren Genexpression während der Immunantwort beteiligt. Bei der Initiierung der inflammatorischen Antwort aktivieren EGR-Proteine das Expressionsprogramm pro- und antiinflammatorischer Schlüsselzytokine im Zusammenspiel mit anderen regulatorischen Transkriptionsfaktoren. Die Proteine der EGR-Familie interagieren mit den Transkriptionsfaktoren der NFAT- Familie. Die Proteininteraktionen können sowohl direkt als auch funktional sein. Die physikalischen Interaktionen von Proteinbindungsassays, EGR-3 mit und der EGR-4 mit den Plasmonresonanz-Studien NFAT-Proteinen und in wurden in Peptidspotanalysen demonstriert. EGR-3 und EGR-4 binden mit zwei Regionen unterschiedlicher Affinität die NFAT-Proteine. Die Domänen, die für eine direkte Interaktion verantwortlich sind, vermitteln auch die funktionale Kooperation der EGR- und NFAT-Proteine. Zusätzlich wurden in Transfektionsstudien funktionale Repressor- und Aktivierungsdomänen von EGR-4 nachgewiesen. Eine genauere Charakterisierung einer EGR-4-Interaktionsdomäne mit der Peptidspotanalyse zeigte, dass EGR-4 in der Region mehrere Aminosäurebereiche für die Bindung von NFAT-Proteinen besitzt. In den Aminosäurebereichen sind hauptsächlich positiv geladene Aminosäuren für die Interaktion mit den NFAT-Proteinen verantwortlich. Ein Austausch dieser positiv geladenen Aminosäuren reduziert die Bindung von NFAT-Proteinen deutlich. Weiterhin konnte die Interaktion von intaktem EGR-4 und NFATp in Plasmonresonanz-Studien durch den Einsatz inhibitorischer Peptide gehemmt bzw. reduziert werden. Da EGR-4 mehrere Domänen für die Interaktion besitzt, konnte die Interaktion von EGR-4 und NFATc nicht vollständig gehemmt werden. EGR-3 und EGR-4 gehen weiterhin direkte und funktionale Interaktionen mit den Proteinen der NFκB-Familie ein. Die direkten Interaktionen wurden in Proteinbindungsassays, mit der Plasmonresonanz-Studie und in Peptidspotanalysen dargestellt. In Transfektionsstudien wurden mit Hilfe von Deletionsmutanten für EGR-4 funktionelle Interaktions-, Aktivierungs- und Repressordomänen lokalisiert. Die Zinkfingerregion ist eine Aktivierungsdomäne und für die zentrale Rolle von EGR-Proteinen wichtig, da sie sowohl die DNA-Bindung als auch die Interaktion mit p65 vermittelt. EGR-Proteine binden das NFκB-Protein p65 mit jeweils einem - VIII - Zusammenfassung Zinkfinger. Die Interaktion von EGR-4 und p65 konnte in Plasmonresonanz-Studien mit einem inhibitorischem Peptid der EGR-4-Zinkfingerregion vermindert werden. Auch hier konnte die Interaktion nicht vollständig gehemmt werden, da es noch weitere Kontaktpunkte für p65 im EGR-4-Protein gibt. Im Rahmen dieser Arbeit konnten für EGR-4 Interaktionsdomänen und transkriptionell regulative Domänen identifiziert werden. EGR-4 besitzt drei Aktivierungs- und zwei Repressordomänen. Zwei Aktivierungsdomänen wurden im N-Terminus (aa 1-150) und in der mittleren Region (aa 171-325) von EGR-4 identifiziert. Die Zinkfingerregion (aa 379 bis 463) stellt die dritte Aktivierungsdomäne im C-Terminus von EGR-4 dar, die für die Bindung an die DNA verantwortlich ist. Die Repressordomänen befinden sich N-terminal (aa 326 bis 378) und C-terminal (aa 464 bis 487) direkt neben der Zinkfingerdomäne. EGR-4 besitzt jeweils drei Interaktionsdomänen für die Bindung von NFAT- und NFκB-Proteinen. Die Interaktionsdomänen für die Bindung von NFAT-Proteinen (aa 173-197, aa 224-238 und aa 306 -324) liegen N-terminal vom Zinkfinger in einer Aktivierungsdomäne. Die Interaktionsdomäne für die p65-Bindung ist in der Aktivierungsdomäne der Zinkfingerregion im Bereich der Aminosäuren 397-465 lokalisiert. Die Ergebnisse zeigen, dass die EGR-Proteine bei der transkriptionellen Regulation von Entzündungsgenen in T-Zellen eine wichtige Rolle spielt. Die Charakterisierung der Interaktionsdomänen deutet daraufhin, dass EGR-4 ein Multidomänenprotein ist und wahrscheinlich als Teil eines spezifischen regulatorischen Promotorkomplexes die Transkription inflammatorischer Gene kontrolliert und mit dem der basale Transkriptionskomplex verbunden ist. - IX - Einleitung 1 Einleitung 1.1 Das Immunsystem Der Körper reagiert auf das Eindringen von Pathogenen mit einer Immunantwort, die sich in angeborener und adaptiver (erworbener) Immunität unterscheidet. Die angeborene Immunität wird direkt durch Krankheitserreger aktiviert, wobei die Reaktionsfähigkeit konstant bleibt. Zu der angeboren Immunität zählen sowohl physikalische Barrieren wie Haut und Mukosa, chemische Barrieren durch lösliche Plasmaproteine (Mediatoren, Komplement) als auch die zelluläre Abwehr von Makrophagen, Granulozyten und natürliche Killerzellen. Die adaptive Immunantwort ist die Reaktion antigenspezifischer Lymphozyten auf ein Antigen mit der Ausbildung eines sogenannten immunologischen Gedächtnisses und einer langandauernden Immunität, die als Folge einer Impfung oder Erkrankung entstehen kann. Adaptive Immunantworten entstehen durch Selektion bestimmter Lymphozytenklone und können nach einer wiederholten Infektion schneller und effizienter reagieren. Die adaptive Immunität besitzt durch T-Zellen und B-Zellen zelluläre und humorale Antwortfähigkeiten. Die angeborene und adaptive Immunität bilden zusammen ein außerordentlich wirksames Abwehrsystem, das miteinander verbunden ist. So tragen Komplement, Interleukine, Interferone, Makrophagen und natürliche Killerzellen sowohl zur angeborenen als auch zur adaptiven Immunität bei. 1.1.1 Zellen des Immunsystems Immunreaktionen werden durch die Leukozyten vermittelt, einer großen Gruppe weißer Blutzellen, zu der die Lymphozyten, polymorphkernige Leukozyten und Monozyten zählen. Die Lymphozyten sind die weißen Blutzellen, die variable Rezeptoren für Antigene auf der Zelloberfläche tragen. Die zwei Hauptklassen der Lymphozyten, B-Lymphozyten (B-Zellen) und T-Lymphozyten (T-Zellen), sind für die humorale bzw. zelluläre Immunität verantwortlich. Die B- und T-Lymphozyten entwickeln sich in den primären lymphatischen Organen (Knochenmark und Thymus) und wandern als reife Lymphozyten ins Blut. Lymphozyten zirkulieren ständig vom Blutsystem zu den peripheren, sekundären lymphatischen Organen und über die Lymphgefäße wieder zurück ins Blut. Bei Kontakt mit einem Antigen differenzieren sich die Lymphozyten zu antigenspezifischen Effektorzellen. Adaptive Immunantworten werden im peripheren lymphatischem Gewebe in den meisten Fällen durch T-Zellen ausgelöst, die -1- Einleitung spezifische Antigene erkennen, die von dendritischen Zellen präsentiert werden. Zu der Gruppe polymorphkerniger Leukozyten gehören neutrophile, eosinophile und basophile Granulozyten, die im Blut zirkulieren und nach der Aktivierung als Effektorzellen an Infektions- und Entzündungsorte wandern. Die Monozyten sind die Vorläuferzellen der gewebeständigen Makrophagen, die bei der angeborenen Immunität und in frühen, nichtadaptiven Phase der Immunantwort eine Rolle spielen. Sie können als antigenpräsentierende Zellen sowie als Effektorzellen bei humoralen und zellulären Immunreaktionen fungieren. Während der Immunantwort werden sie von T-Zellen aktiviert und sind für die Abwehr und Zerstörung von Pathogenen und der Aktivierung weiterer phagozytotischer, neutrophiler Zellen von großer Bedeutung. Makrophagen und Mastzellen differenzieren im Gewebe und lösen dort als erste Mediatoren Entzündungen aus. Mastzellen sind große exozytotische Zellen, die über den ganzen Körper verteilt im Bindegewebe (Submukosa, Oberhaut) vorkommen. Sie enthalten große Granula, in denen eine Vielzahl von Mediatormolekülen gespeichert sind wie z. B. die vasoaktive Substanz Histamin. Mastzellen sind wahrscheinlich bei der parasitären Abwehr und der Auslösung von allergischen Reaktionen beteiligt. Weiterhin aktivieren Mastzellen eosinophile und basophile Zellen. Dendritische Zellen entwickeln sich aus Vorläuferzellen im Knochenmark und sammeln sich als unreife Formen in den Bereichen lymphatischer Gewebe, die viele T-Zellen enthalten. Sie sind verzweigt und wirken als die stärksten Stimulatoren der TZell-Reaktion. In nichtlymphatischen Geweben werden T-Zell-Antworten erst dann ausgelöst, wenn dendritische Zellen aktiviert wurden und zu den lymphatischen Geweben wandern. Dendritische Zellen dringen als unreife Phagozyten in Gewebe ein und sind darauf spezialisiert Antigene aufzunehmen. Anschließend wandern sie als antigenpräsentierende Zellen in das Lymphgewebe. 1.1.2 Mediatoren des Immunsystems Im Rahmen der Immunantwort wird die Aktivität der beteiligten Zellen durch verschiedene Mediatoren reguliert. Die aktivierten Zellen exprimieren wiederum Mediatoren, die sowohl auf sie selbst wie auf andere Zellen einwirken. Zu den wichtigsten Mediatoren zählen die Zytokine, Chemokine und Adhäsionsmoleküle. Zytokine werden auf einen Stimulus von Zellen exprimiert, binden an spezifische Rezeptoren und steuern so das Verhalten ihrer Zielzellen. Die Zytokinproteine können autokrin auf Zellen, -2- Einleitung die sie produzieren, parakrin auf benachbarte Zellen oder endokrin auf weiter entfernte Zellen wirken. Die Zytokine können aufgrund der Struktureigenschaften in strukturelle Familien eingeteilt werden, zu denen die Hämatopoetine, die Chemokine, die Interferone und die Familie der Tumornekrosefaktoren (TNF) zählen. Die wichtigsten Vertreter der Hämatopoetine sind viele Interleukine. Da Interleukin eine übergeordnete Bezeichnung aller von Leukozyten produzierten Zytokine ist, kommen diese auch in den anderen Zytokinfamilien vor. Die Hämatopoetin-Familie stellt eine große Familie aus Wachstumshormonen und zahlreichen Interleukinen dar. Einige Mitglieder sind neben IL-2, IL-3, IL-4, IL-5, IL-6, IL-7, IL-9, IL-11, IL-13, IL-15 auch die Zytokine G-CSF und GM-CSF. Sie werden in T-Zellen, Epithelzellen, Mastzellen, Makrophagen, Fibroblasten, Monozyten und anderen Zellen exprimiert. Sie besitzen vielfältige Funktionen bei der T-Zell-Proliferation, B-Zellaktivierung und Stimulation erythroider Vorläuferzellen. Weiterhin sind Hämatopoetine an der Differenzierung, der Entwicklung und beim Wachstum vieler verschiedener Zellen beteiligt. Chemokine sind Zytokine, die als Chemoattraktoren die Wanderung und Aktivierung von Phagozyten und Lymphozyten stimulieren. Sie werden in den ersten Phasen einer Infektion in dem betroffenen Gewebe freigesetzt und wirken auf Leukozyten, Monozyten, Makrophagen, Granulozyten, dendritische Zellen und andere Effektorzellen im Blut und signalisieren ihnen eine vorhandene Entzündung. Immunmodulatorische Chemokine sind z. B. Interleukin-8 und RANTES. Weiterhin zählt zu den Zytokinen die Gruppe der Interferone, die in der Zelle bei einer Vireninfektion induziert werden und als antivirale Effektormoleküle die Resistenz von Zellen gegen die Replikation von Viren bewirken können. Die Interferone werden von Leukozyten (IFN-α), Fibroblasten (IFN-β), von T-Zellen und natürlichen Killerzellen (IFN-γ) exprimiert. Die wichtigste Aufgabe von Interferon-γ ist die Aktivierung von Makrophagen. Die TNFFamilie spielt sowohl bei der adaptiven als auch bei der erworbenen Immunität eine bedeutende Rolle. Die Mitglieder der TNF-Familie, zu denen neben TNF-α und TNF-β noch LT-β, CD4Ligand, Fas-L, CD-27 L und CD30L zählen, werden alle von T-Zellen produziert. Weiterhin exprimieren B-Zellen, Mastzellen, Monozyten und Osteoblasten einige Mitglieder dieser Familie. Zytokine der TNF-Familie sind in viele Ereignisse der Immunantwort involviert und spielen bei lokalen Entzündungen, bei der Entwicklung von Lymphknoten und bei Apoptose eine Rolle. Außerdem sind sie an der Zellaktivierung, Proliferation und Kostimulierung von B- und T-Zellen beteiligt. Die Mitglieder der TNF-Familie wirken entweder als zellassoziierte oder sezernierte Proteine, die mit den Rezeptoren der Familie der TNF-Rezeptoren in -3- Einleitung Wechselwirkung treten. Diese wiederum interagieren mit dem Inneren der Zelle über Proteine, die man als TNF-assoziierte Faktoren (TRAFs) bezeichnet. Zelladhäsionsmoleküle vermitteln bei der angeborenen Immunantwort die Wanderung der Leukozyten aus den Blutgefäßen (Extravasation), die in vier Schritten (rollende Adhäsion, feste Bindung, Diapedese und Wanderung im Gewebe) eingeteilt werden kann. Während einer Entzündungsreaktion wird die Expression von Adhäsionmolekülen auf der Oberfläche von Leukozyten und Endothelzellen induziert. Diese können miteinander interagieren und letztendlich zu einer lokalen Leukozyteneinwanderung ins entzündliche Endothel führen. Zu den Zelladhäsionsmolekülen zählen Selektine, Integrine und die Immunglobulin-Superfamilie. Auf der Endotheloberfläche werden Selektine (P- und E-Selektin) exprimiert, die Kohlehydrate auf der Leukozytenoberfläche binden können und so eine Leukozyten-Endothel-Assoziation vermitteln. Die selektinvermittelte Adhäsion ist schwach und ermöglicht den Leukozyten, an der Epitheloberfläche entlang zu rollen. Bei Entzündungsprozessen wird das Chemokin IL-8 von Zellen am Infektionsherd ausgeschüttet. Die Bindung von IL-8 an spezifische Oberflächenrezeptoren der Leukozyten induziert die Aktivierung von Integrinproteinen auf der Leukozytenoberfläche. Eine festere Bindung der Leukozyten wird durch die Mitglieder der Immunglobulin-Superfamilie (ICAM-1, ICAM-2, VCAM-1 und PECAM) auf Endotheloberfläche vermittelt. Durch die Bindung von Zelladhäsionsmolekülen an heterodimere Integrinproteine (LFA-1, CD-11a/CD18 oder Mac-1) auf der Leukozytenoberfläche wird der Leukozyt fest gebunden und kann nun in das Gewebe eindringen. 1.2 Entzündungsreaktion als Beispiel einer Immunantwort Mikroorganismen, die in den Körper eindringen, werden sofort von Zellen und Molekülen des angeborenen Immunsystems angegriffen. Phagozytotische Makrophagen steuern die Abwehr von Bakterien mit Hilfe von Oberflächenrezeptoren, die allgemein vorkommende bakterielle Oberflächenproteine erkennen und binden können. Die Makrophagen nehmen die Fremdkörper auf und induzieren die Sekretion der biologisch aktiven Zytokine und Chemokine. Eine Entzündung wird durch die charakteristischen Zustände Wärme, Schmerz, Rötung und Schwellung beschrieben und ist auf die Auswirkungen von Zytokinen und anderen Entzündungsmediatoren auf lokale Blutgefäße zurückzuführen. Zytokine beeinflussen die Adhäsionskraft des Gefäßendothels, so dass Leukozyten aus dem Blut in die betroffenen Gewebe -4- Einleitung einwandern können. An frühen Entzündungsreaktionen sind neutrophile Zellen und Makrophagen, die sogenannte Entzündungszellen darstellen, beteiligt. Neutrophile Zellen besitzen wie Makrophagen Oberflächenrezeptoren und können Fremdstoffe sowie Eindringlinge phagozytieren. Monozyten wandern kurz nach der Entzündung ins Gewebe ein und differenzieren sich zu Makrophagen. Die Entzündungsreaktion erhöht die Flußrate der Lymphflüssigkeit, die Antigene und antigentragende Zellen in das lymphatische Gewebe transportiert. Makrophagen und dendritische Zellen aktiveren Lymphozyten und setzen die adaptive Immunantwort und damit die spezifische Bekämpfung des Pathogens in Gang. Bei der Antwort auf eine Entzündung werden viele Zellen aktiviert und moduliert (Abb. 1). Nach dem Eindringen von Pathogenen erkennen Makrophagen mit Hilfe ihrer Oberflächenrezeptoren allgemein vorkommende Bestandteile auf der bakteriellen Oberfläche. Daraufhin können die Makrophagen die Pathogene phagozytieren und induzieren die Produktion und Sekretion der Zytokine IL-1, IL-6, IL-12, TNF-α und des Chemokins IL-8. Das Chemokin IL-8 besitzt eine ständige chemotaktische Aktivität und lockt andere immunmodulatorische Zellen wie neutrophile Leukozyten und Monozyten an den Ort des Geschehens. Die Zytokine induzieren lokal eine Erweiterung und erhöhte Durchlässigkeit der Blutgefäße, so dass es zu charakteristischen Entzündungsmerkmalen kommt. Außerdem wirken Zytokine auf die Adhäsionskraft des Gefäßendothels, damit Leukozyten aus dem Blut am entzündlichen Gewebe haftenbleiben und eindringen können. Aktivierte Leukozyten exprimieren wiederum IL-8 und das Chemokin RANTES, die auf Monozyten, T-Zellen und auf andere Leukozyten einwirken. Die Interleukine IL-6 und IL-1 werden von Makrophagen exprimiert und stimulieren das Wachstum und die Differenzierung von T- und B-Zellen. IL-1 induziert zusätzlich weitere Makrophagen, während IL-12 natürliche Killerzellen aktiviert. Die T-Lymphozyten werden von Makrophagen über die Ausschüttung von IL-6 und IL-1 aktiviert. Die T-Zellen exprimieren während der Entzündungsantwort ebenfalls Zytokine und können somit auf die B-ZellAktivierung (IL-4), das Wachstum und die Differenzierung eosinophiler Zellen (IL-5), dendritischer Zellen (GM-CSF) und der myelomonocytischen Zelllinie wirken. Weiterhin wirkt das Zytokin IL-2 von T-Zellen auf die eigene T-Zell-Proliferation und zusammen mit IL-10 inhibitorisch auf die Makrophagenfunktion. Mit der Produktion von IL-8 können auch T-Zellen weitere immunmodulatorische Zellen anlocken. Endothelzellen exprimieren nach der Aktivierung durch TNF-α vorwiegend Zelladhäsionsmoleküle (E-Selektin, P-Selektin, ICAM-1 und VCAM-1) und sorgen damit für die Einwanderung von Leukozyten. Die eosinophilen Leukozyten werden im Rahmen der erworbenen Immunantwort von Lymphozyten aktiviert. Sie -5- Einleitung produzieren wiederum neben IL-3 und IL-5 den Wachstumsfaktor TGF-β, der als Inhibitor bei Entzündungsreaktionen wirkt. Die Freisetzung von TNF-α aus Makrophagen, eosinophilen Leukozyten und T-Zellen führt zu vielfältigen Reaktionen. Der allgemeine Einfluß von TNF-α verursacht in Blutgefäßen einen gesteigerten Blutfluß, eine erhöhte Durchlässigkeit für Proteine und Zellen sowie eine stärkere Adhäsion von Leukozyten und Blutplättchen. Später bilden sich in engen Gefäßen Blutgerinsel, so dass sich die Infektion nicht über das Blutsystem ausbreiten kann. Flüssigkeit und Zellen werden in regionale Lymphknoten abgeleitet um dort adaptive Immunreaktionen zu initiieren. Abb. 1: Auslösung einer Entzündungsantwort Aktivierte vaskuläre Endothelien im Gewebe exprimieren verschiedene Mediatoren, die zirkulierende Leukozyten an den Ort der Entzündung führen. Aktivierte Leukozyten exprimieren verschiedene Zytokine, Chemokine und Wachstumsfaktoren, die zu der Auslösung der Entzündung beitragen. Störungen in dem Genexpressions-Programm sind für chronische Erkrankungen verantwortlich (verändert nach Manning and Rao, 1999). 1.3 Transkriptionsfaktoren als Regulatoren induzierbarer Genexpression Bei Entzündungen werden eine große Anzahl von Zytokinen und Zelladhäsionsmolekülen mobilisiert, die in immunmodulatorischen Zellen exprimiert werden. Die Expression der Gene, die für Zytokine und Zelladhäsionsmoleküle kodieren, ist induzierbar und stellt damit eine wichtige Regulationsstufe während der Entzündungsantwort dar. Die Transkription eines Gens ist definiert als das Umschreiben der Gen-kodierenden DNA in mRNA, die wiederum als Matrize für die darauf folgende Translation dient. Der Transkriptionsprozess wird durch Transkriptionsfaktoren, den RNA-Polymerasekomplex und -6- Einleitung verschiedene Koaktivatoren kontrolliert, die an spezifische DNA-Elemente der Gene binden (Tjian and Maniatis, 1994). Die regulatorischen Regionen besitzen einen Promotorbereich, an den ein basaler Transkriptionskomplex bindet, und eine oder mehrere weiter entfernt liegende „Enhancer“-Elemente. Die spezifischen DNA-Elemente besitzen eine charakteristische Abfolge von Bindungsstellen für Transkriptionsfaktoren und bestimmen somit die Genexpression. Beispiele regulatorischer Elemente von Promotoren sind anhand der IL-2- und TNF-αPromotoren in Abbildung 2 dargestellt. Die Interaktion von Transkriptionsfaktoren an proximale oder distale „Enhancer“-Regionen fördert direkt oder über Koaktivatoren die Bindung des basalen Transkriptionskomplexes und moduliert so die Aktivität der RNA-Polymerase II. Die gesamte Aktivität der Transkriptionsfaktoren, die an regulatorische Domänen eines Gens binden, führt jeweils zu einer spezifischen Transkriptionsrate der mRNA. Abb. 2: Regulatorische Elemente von IL-2- und TNF-α-Promotoren Dargestellt sind Bindungsstellen für die verschiedenen Transkriptionsfaktoren, die an Promotoren des Tumornekrosefaktors-α und des Interleukins-2 binden. In einigen Fällen binden Transkriptionsfaktoren wie NFATProteine und AP-1 bzw. NFAT- und EGR-Proteine über benachbarte Bindungsstellen. Die Proteine der NFAT- und NFκB-Familie können oft über ähnliche Sequenzmotive an dieselben Promotorelemente binden. Transkriptionsfaktoren können als Monomere, Dimere oder höher geordnete Multimere an spezifische DNA-Motive binden, die normalerweise 7-20 Basenpaare lang sind. In vielen Fällen bilden die Transkriptionsfaktoren Heterodimere mit anderen Transkriptionsfaktoren der eigenen Familie und kooperative Multimere mit Mitgliedern anderer Familien. Individuelle Mitglieder einer Familie können in bestimmten Geweben und Zellen spezifisch exprimiert werden und dort die Expression von zell- und gewebespezifischen Genen regulieren. Die Fähigkeit der Transkriptionsfaktoren, an DNA zu binden und somit die Gentranskription zu modulieren, wird in der Zelle streng reguliert. Spezifische intrazelluläre Signaltransduktionswege kontrollieren die Aktivität von Transkriptionsfaktoren über Phosphorylierungen an Schlüsselpositionen (Karin -7- Einleitung and Hunter, 1995). Die Tabelle 1 liefert eine Übersicht verschiedener Transkriptionsfaktoren, die bei Entzündungsprozessen eine Rolle spielen. Tabelle 1: Übersicht von Transkriptionsfaktoren bei Entzündungsprozessen Familie Aktivatoren regulierte Gene NGF, PMA, PHA, Calciumionophore, IL-2-Rβ, FasL, NFκB1, BP-1, nur 77, EGR-Familie: Mitogene, neuronale Erregung EGR-1, EGR-2, Proliferation-, Entwicklungs- und Synapsin I und II, hoxB2, PNMT, bFGF, EGR-3, EGR-4 Differenzierungssignale, LH-β,TF, PDGF A, PDGF B, p53, TGF- Gewebe- oder Strahlenschädigung, NFκB/Rel-Familie: TNF, IL-1, UV, H2O2, LPS p65, p50/105, CD44, ICAM-1, CD23, Fas, SOD, β1, Bcl-2, EGR-1, ADA, EGR-4 IL-1, IL-2, IL-6, IL-8, TNF-α, E- Selektion, VCAM-1; ICAM-1, MIP-1, Rel , Rel B MCP-1, Eotaxin, RANTES, COX2, Myc, NFAT-Familie: T-Zellrezeptor, B-Zellrezeptor, FCε- IL-2, IL-3, IL-4, IL-5, IL-8, IL-13, TNF-α, NFATp, NFATc, Rezeptorprotein, Histamin- und GM-CSF, IFN-γ, CD40L, FASL, ICAM-1 NFAT3, NFATx, NFAT5 Thrombinrezeptoren AP-1: Aktivatorprotein, bFGF: „basic fibroblast growth-“Faktor, CSF: „colony-stimulating“-Faktor, FasL: FasLigand, GF: „growth“-Faktor, GM-CSF: „granulocyte macrophage colony-stimulating“-Faktor, hox: Homöobox, ICAM: interzelluläres Adhäsionsmolekül, IFN: Interferon, IL: Interleukin, LPS: Lipopolysaccharid, MCP: Monozyten-chemotaktisches Protein, MIP: Makrophagen-inflammatorisches Protein, MMP: Matrixmetalloprotein, NFκB: nuklearer Transkriptionsfaktor κB, NFAT: nuklearer Faktor von aktivierten T-Zellen, PDGF: „platelet derived growth“-Faktor, PNMT: Phenylethanolamin N-Methhyltransferase, RANTES: reguliert durch Aktivierung, normalerweise in T-Zellen exprimiert und sekretiert, SOD: Superoxid-Dismutase, STAT: Signaltransduktor und Aktivator der Transkription, TGF-β1: „transforming growth”-Faktor, TF: „tissue”-Faktor, TNF: Tumornekrosefaktor, UV: Ultraviolettes Licht, VCAM: vaskuläres Adhäsionsmolekül. 1.4 „Immediate early“-Gene Die „immediate early“ (IE)-Gene werden nach einem Stimulus als erste Gruppe von Zielgenen durch verschiedene intrazelluläre Mediatorsysteme aktiviert. Die „immediate early“-Gene sind durch die schnelle Induktion der Transkription definiert, die von der de novo-Proteinsynthese unabhängig ist und den Übergang einer Zelle aus der ruhenden Phase in die Wiederaufnahme in den Zellzyklus charakterisiert (Herschman, 1989). Eine Unterklasse der „immediate early“-Gene kodiert für induzierbare Transkriptionsfaktoren und kann folglich als Mediator im Zellkern wirken. Zu den bekannten „immediate early“-kodierenden Transkriptionsfaktoren gehören die „early growth response“-Proteine EGR-1, EGR-2, EGR-3 und EGR-4. Weiterhin zählt zu den „immediate early“-kodierenden Proteinen das Aktivatorprotein AP-1, das ein Heterodimer aus den Mitgliedern der Fos- und Jun-Familien ist (Beckmann and Wilce, 1997). -8- Einleitung 1.5 „Early growth response“-Proteine Die EGR-Proteine sind Transkriptionsfaktoren, die aufgrund ihrer charakteristischen DNAbindenden Domäne zu einer Familie zusammengefasst werden (Abb. 3). Die Familie besteht aus den Mitgliedern EGR-1, EGR-2, EGR-3 und EGR-4. Die DNA-bindende Domäne der EGRProteine besteht aus drei aufeinanderfolgenden Zinkfinger-Motiven des Cys2His2-Typs, die innerhalb der EGR-Familie eine Homologie von über 90 % zeigen (Abb. 3). Die geringe basale Expressionsrate der EGR-Proteine im Zytoplasma ruhender Zellen wird auf verschiedenen Niveaus reguliert. Die Regulation schließt sowohl transkriptionelle, translationelle als auch posttranslationelle Regulationsstufen wie Glykosylierung, Phosphorylierung und Redoxreaktionen mit ein. EGR-1 wurde erstmals als „immediate-early“-Gen nach der Stimulation von PC12-Zellen der Ratte mit Nerven-Wachstumsfaktoren (NGF) entdeckt (NGFI-A; Milbrandt, 1987). Kurz darauf wurden von mehreren Forschergruppen weitere EGR-1-Homologe und andere Mitglieder der EGR-Familie aus verschiedenartig stimulierten Zellen identifiziert. Die weiteren EGR-1Homologen neben dem NGFI-A waren das murine TIS8 (Lim et al., 1987 und 1989), das humane und murine Egr-1 (Sukhatme et al., 1988), das murine Krox-24 (Lemaire et al., 1988) und der murine Zinkfingerklon zif268 (Christy et al., 1988). EGR-2 wurde mit einer cDNASonde der Zinkfingerregion des Krüppelgens aus Drosophila in murinen Zellen identifiziert und Krox-20 genannt (Chavrier et al., 1988). Humanes EGR-2 wurde aus aktivierten T-Zellen (pAT591; Zipfel et al., 1989) und Fibroblasten isoliert (Joseph et al., 1988). EGR-3 wurde aus serum-stimulierten humanen 3T3 Zellen isoliert (Patwardhan et al., 1991) und als transkriptioneller Regulator bei der T-Zell-spezifischen Expression von Zytokin-kodierenden Genen identifiziert, weshalb es auch PILOT genannt wird (Mages et al., 1993). EGR-3Homologe wurden weiterhin aus der Maus (Egr-3; Patwardhan et al., 1991) und der Ratte isoliert (Yamagata et al., 1994). Mit der Identifizierung von EGR-4 (pAT133, NGFI-C) wurde das vierte Mitglied der EGR-Familie beschrieben (Milbrandt, 1987; Zipfel et al., 1989; Müller et al., 1991; Crosby et al., 1991). Alle Mitglieder der EGR-Familie erkennen ein DNA-Konsensusmotiv mit der Sequenz GCG(G/T)GGGCG, das auch als GSG-Motiv oder EBS bezeichnet wird (Christy and Nathans, 1989 a; Cao et al., 1990; Lemaire et al., 1990; Chavrier et al., 1988; Patwardhan et al., 1991; Crosby et al., 1991). Alle Mitglieder der EGR-Familie können aufgrund der hoch konservierten -9- Einleitung Zinkfingerregion an ein GC-reiches Konsensusmotiv binden und verschiedene EGR-regulierte Gene transaktivieren (Lemaire et al., 1990; Chavrier et al., 1990; Patwardhan et al., 1991; Crosby et al., 1991). Die Zinkfinger der EGR-Proteine weisen eine Homologie von 70-90 % zu dem Drosophila-Segmentierungsprotein Krüppel und eine Homologie von 50 % zu dem Zinkfingerprotein Wilms´ Tumor 1 (WT1) auf, das vier Zinkfinger des Cys2His2-Typs im Cterminalen Bereich besitzt (Call et al., 1990; Gessler et al., 1990; Müller et al., 1991; Mages et al., 1993; Frommer et al., 1996). WT1 besitzt wie die EGR-Proteine hohe Serin-, Prolin- und Glycinanteile, zeigt jedoch im N-Terminus keine weiteren Homologien (Call et al., 1990; Gessler et al., 1990). Die Zinkfinger von WT-1 werden im Gegensatz zu denen der EGRProteine von separaten Exons kodiert (Haber et al., 1991) und entsprechen damit der genomischen Organisation anderer Transkriptionsfaktoren wie TFIIIA (Tso et al., 1986). Abb. 3: Struktur von EGR-Proteinen (A) EGR-Proteine besitzen neben der konservierten Zinkfingerregion Domänen, die vorwiegend aus Serin-, Glycin-, Alanin- und Prolinresten bestehen. Die drei Zinkfinger des Typs Cys2His2 binden je ein Zinkion und vermitteln die DNA-Bindung der EGR-Proteine, die am Beispiel von EGR-4 in (B) dargestellt ist. - 10 - Einleitung 1.5.1 EGR-Proteinstruktur Die DNA-Bindungsregion der EGR-Proteine besitzt drei Zinkfinger des Typs Cys2His2. Diese Zinkfinger sind sich wiederholende Regionen aus 28-30 Aminosäuren mit je zwei Cystein- und zwei Histidinresten an konservierten Positionen. Die konservierten Cystein- und Histidinreste bilden ein Tetraeder um ein Zinkion, so dass ein finger-artiges Gebilde entsteht. (Miller et al., 1985; Berg, 1988). Die EGR-Zinkfingerregionen vermitteln die Bindung an die DNA (Pavletich and Pabo, 1991; Gashler et al., 1993; Vesque and Charnay, 1992). Die Regionen außerhalb der Zinkfinger können Einfluß auf die DNA-Bindungsaktivität haben. So kann z. B. die Deletion der N-terminal vom Zinkfinger liegenden Regionen die DNA-Bindungsaktivität von EGR-1 erhöhen (Carman and Monroe, 1995; Huang et al., 1994). Eine Erhöhung der DNA-Bindungsaktivität bei EGR-2 wurde ebenfalls durch die Deletion N-terminaler Sequenzen kurz vor dem Zinkfinger beobachtet (Vesque and Charnay, 1992). Das intakte EGR-1 bindet an die DNA mit höherer Affinität als die Zinkfingerdomäne alleine, so dass es vermutlich für die DNA-Bindung weitere regulatorische Domänen gibt (Pavletich und Pabo, 1991). Die Struktur der Zinkfinger wurde durch Röntgenstrukturanalysen und Methylase-InterferenzStudien mit einem EGR-1/DNA-Komplex aufgeklärt (Christy and Nathans, 1989 a; Lemaire et al., 1990; Pavletich and Pabo, 1991). Die drei Zinkfinger sind in einer semizirkulären (c-shaped) Struktur arrangiert, die sich in die große Furche der B-DNA einfügt. Jede Zinkfingerdomäne besteht aus einem antiparallelen β-Faltblatt und einer α-Helix. Je zwei Cysteine des β-Faltblatts und zwei Histidine der α-Helix koordinieren ein Zinkionen (Abb. 3). Jeder Finger bindet drei Basenpaare der DNA, wobei der Hauptkontakt zum GC-reichen Strang besteht. Die drei Zinkfinger werden in die zwei Strukturformen I und II eingeteilt. Der erste und dritte Zinkfinger besitzt jeweils die Struktur I und bindet die Sequenzen gcggggGCG bzw. GCGggggcg. Der zweite Zinkfinger der Struktur II bindet DNA mit der Sequenz gcgGGGgcg. Weiterhin sind die Zinkfingerstrukturen I und II durch homologe Aminosäuren gekennzeichnet. Der erste und dritte Zinkfinger besitzt an denselben Positionen Argininreste, mit denen ein Kontakt zur DNA ausgebildet wird (Abb. 3 B). Der zweite Zinkfinger bindet die DNA mit einem Arginin- und einem Histidinrest. Einzelne EGR-Proteine zeigen Homologien auch außerhalb der DNA-Bindedomäne (Crosby et al., 1992). Bisher wurden konservierte Regionen beschrieben, die einen besonders hohen Anteil positiv oder negativ geladener Aminosäurereste besitzen (Lemaire et al., 1988; Crosby et al., - 11 - Einleitung 1992; Vesque and Charnay, 1992; Russo et al., 1993; Gashler et al., 1993; Carman and Monroe, 1995; Russo et al., 1995; Svaren et al., 1996). Alle EGR-Proteine enthalten im N-terminalen Proteinbereich Regionen, die vorwiegend aus den Aminosäureresten Prolin, Alanin, Serin und Glycin bestehen. Die verschiedenen EGR-Proteine besitzen außerdem Regionen, bei denen die Ladungsverteilung konserviert ist. EGR-1, EGR-2 und EGR-3 besitzen negativ geladene Aminosäuren an homologen Positionen relativ zu der Sequenzhomologie (Vesque and Charnay, 1992). 1.5.2 Regulation der EGR-Proteine EGR-Proteine spielen eine wichtige Rolle bei der Kontrolle von Entwicklungs-, Wachstums- und Proliferationsprozessen in Zellen und Geweben (Sukhatme et al., 1988; Cao et al., 1990). Induzierbare Transkriptionsfaktoren wie die EGR-Proteine kommen mit sehr niedrigen Expressionsraten in unstimulierten Zellen vor und spiegeln laufende physiologische Prozesse in der Zelle wieder. Die Induktion der EGR-Gene und die Expression der EGR-Proteine in Zellkultursystemen ist ausführlich von Sukhatme (1990, 1992) und Gashler (1995) beschrieben worden. Die Regulation der Expression und Funktion der EGR-Proteine findet auf verschiedenen Ebenen statt. Bei der Transkription werden die EGR-Gene durch sogenannte cis-aktivierende Elemente in der 5´-Promotorregion reguliert, die mehrere Kilobasen von oder in den Introns lokalisiert sein können. Die Produkte der EGR-mRNA können durch verschiedene posttranslationale Prozesse und Modifikationen reguliert werden. Posttranslationale Prozesse sind z. B. Phosphorylierung, Glykosylierung und/oder Redoxpotentierung. Die Aktivitäten eines Proteins können durch allosterische Mechanismen und Interaktionen mit anderen zellulären Komponenten und/oder Proteinen verändert werden. Die Expression der EGR-Genfamilie wird auf allen Stufen reguliert. EGR-Proteine können nach einer Stimulation koordinierte oder diskrete Regulationsstufen durchlaufen, in denen bestimmte Induktionszeiten und/oder Gewebe- und Zellspezifität der Proteine eine wichtige Rolle spielen. Zunächst hängt es vom Promotorkontext der EGR-Gene ab, dass EGR-Proteine auf einen gegebenen Stimulus in einem Zelltyp exprimiert werden. Für die Regulation auf der Ebene der Transkription besitzen die EGR-Gene besondere Genstrukturen und spezifische regulatorische Promotorelemente. - 12 - Einleitung Im Genom des Menschen sind die Gene, die für EGR-1, EGR-2, EGR3 und EGR-4 kodieren, als Einzelgenkopien vorhanden. Die EGR-kodierenden Gene liegen in den Chromosomenloci 5q31 (EGR-1), 10q21 (EGR-2), 8q21 (EGR-3) und 2p13 (EGR-4) (Sukhatme et al., 1988; Tsai-Morris et al., 1988; Changelian et al., 1989; Janssen-Timmen et al., 1989; Sakamoto et al., 1991; Joseph et al., 1988; Chavrier et al., 1989; Rangnekar et al., 1990; Patwardhan et al., 1991; Crosby et al., 1992; Holst et al., 1993). Obwohl ein Intron in der 5´-Region des Zinkfinger-kodierenden Bereiches liegt, werden die EGR-Proteine nicht in sichtbare Domänen geteilt, so dass die drei Zinkfinger von einem Exon kodiert werden. Die ist nicht immer der Fall, wie das TFIIIkodierende Gen zeigt, bei dem die ersten sechs der neun Zinkfinger von separaten Exons kodiert werden (Tso et al., 1986). Das EGR-2-Gen besitzt im Gegensatz zu den anderen EGR-Genen ein zweites Intron (Chavrier et al., 1989; Rangnekar et al., 1990). Die Translation der beiden EGR2-mRNAs führt zu zwei Proteinvarianten, von denen eine Variante am N-Terminus zusätzlich 50 Aminosäuren besitzt (Chavrier et al., 1989; Vesque and Charnay, 1992). Die kurze mRNAVariante kodiert für ein Protein mit geringerer transkriptioneller Aktivität als das Protein der längeren mRNA-Variante (Vesque and Charnay, 1992). Diverse Stimuli aktivieren Kaskaden intrazellulärer Sekundärmessengersysteme, welche die Transkription von EGR-Genen beeinflussen. Die Promotoren der EGR-Gene enthalten viele regulatorische Elemente, an denen Sekundärmessenger binden können. Die EGR- Promotorregionen besitzen „serum response“-Elemente (SRE), Sp-1-ähnliche Motive, mehrere „calcium response“-Elemente (CRE), CCAATT-Sequenzen und verschiedene AP-ähnliche Bindungssequenzen an konservierten Positionen (Christy et al., 1988; Tsai-Morris et al., 1988; Changelian et al., 1989; Chavrier et al., 1989; Janssen-Timmen et al., 1989; Rangnekar et el., 1990; Sakamoto et al., 1991; Holst et al., 1993). Weiterhin wurden GC-reiche EGRKonsensussequenzen im Promotor von EGR-1 gefunden (Christy and Nathans, 1989 a; Cao et al., 1990). Die Expression der mRNA von EGR-1 kann durch jedes „serum response“-Element (SRE), das für die Serum- und PMA-Antwortfähigkeit wichtig ist, erhöht werden (Christy and Nathans, 1989 b; Janssen Timmen et al., 1989). Die Expression der EGR-Proteine wird in vitro durch das Calciumionophor A23187, Dibutyryl-cAMP und Zytokin-vermittelte Mechanismen induziert (Day et al., 1990; Müller et al., 1991; Vaccarino et al., 1993). Alle vier EGR-Proteine werden in der Jurkat-T-Zelllinie durch Phytohämagglutinin (PHA) und Phorbol 12-myristat 13acetat (PMA) induziert (Skerka et al., 1997). EGR-2 und EGR-3 werden weiterhin über eine Cyclosporin A-sensitive Calcineurinphosphorylierung induziert (Shao et al., 1997; Miyazaki and Lemonnier, 1998). Die Expression von EGR-1 wird durch IL-1β, IL-1α, TGF-β und INF-γ - 13 - Einleitung induziert, während TNF-α und FGF keinen Einfluß auf die Expression haben (Nikam et al., 1995). Eine weitere Stufe der Regulation von EGR-Proteinen ist die differenzierte Translation der mRNA. In NGF-stimulierten Zellen wurden zwei Varianten von EGR-1-Proteinen mit molekularen Massen von 73 und 75 kDa beschrieben (Waters et al., 1990). Die zwei EGR-1Translationsprodukte besitzen unterschiedliche Stabilität und könnten durch die Verwendung verschiedener Startkodons oder durch posttranslationelle Modifikationen zustande kommen (Lemaire et al., 1990). Nach einer Stimulation mit PHA oder einem Calciumionophor wurden zwei Formen von EGR-1 mit Mobilitäten von 84 kDa und 54 kDa gefunden (Day et al. 1990). Das 54 kDa-Protein ist eine zytoplasmatische Variante von EGR-1, während die 84 kDa-Form im Kern lokalisiert ist. Weiterhin sind dephosphorylierte und phosphorylierte Formen der 84 kDa-Variante von EGR-1 gefunden worden (Day et al., 1990). Zwei EGR-2-Varianten (79 und 55 kDa) wurden im Kernextrakt von Zellen des Gehirngewebes gefunden (Mack et al., 1992; Day et al. 1990). Die EGR-2-Varianten werden unterschiedlich in den Kern transloziert, so dass die 79-kDa-Variante im Kern erscheint, während die andere im Zytoplasma bleibt (Herdegen et al., 1993). Von verschiedenen EGR-3-oder EGR-4-Varianten wurde bisher nichts berichtet. Die Aktivität der Transkriptionsfaktoren wird ebenfalls durch posttranslationelle Modifikationen reguliert (Hunter and Karin, 1992). Die EGR-Transkriptionsfaktoren können auf mehreren Ebenen posttranslationeller Modifikation verändert werden, zu denen Phosphorylierung, Glykosylierung und Änderungen von Redoxstufen zählen. EGR-Proteine besitzen mehrere Konsensussequenzen für Phosphorylierungen (Milbrandt, 1987). Neben der Serin/Threoninphosphatase könnten potentielle Phosphorylierungsenzyme der EGR-Proteine die Kinase C, eine Caseinkinase II und/oder eine Tyrosinkinase sein (Lemaire et al., 1990; Russo et al., 1993). Die Phosphorylierung von EGR-1 verstärkt zwar die DNA-Bindungsaktivität, hat aber keinen Einfluß auf die Transaktivierungsfähigkeit (Huang and Adamson, 1994). Im zweiten Zinkfinger aller EGR-Proteine ist eine potentielle Glykosylierungsstelle vorhanden, die wahrscheinlich im Zusammenhang mit der Regulation der DNA-Bindungsaktivität steht (Müller et al., 1991). Die Funktion der verschiedenen Glykosylierungen bei Transkriptionsfaktoren ist bisher noch nicht vollständig verstanden. Die Kerntranslokation stellt einen weiteren wichtigen Mechanismus für die Kontrolle der EGRFunktion dar. Für einen Kerntransport besitzen die EGR-Proteine Kerntranslokationssignale, die - 14 - Einleitung von den flankierenden Sequenzen beeinflußt werden können. EGR-1 kann in der Zelle in zwei Varianten auftreten, die sich in der Proteingröße unterscheiden. Die kurze EGR-1-Variante ist vor dem Zinkfinger um die Region verkürzt, die das Kernlokalisationssignal enthält und bleibt im Zytoplasma, während die längere EGR-1-Variante in den Kern transloziert wird (Matheny et al., 1994). Die Region, die das Kernlokalisationssignal enthält, ist in allen EGR-Proteinen homolog (Crosby et al., 1992; Vesque and Charnay, 1992; Matheny et al., 1994). Interessanter Weise besitzt das Kernlokalisationssignal eine potentielle Glykosylierungsstelle, die wahrscheinlich Einfluß auf die Kerntranslokation hat. Die Sekundärstruktur der Zinkfinger spielt im Kernlokalisationsprozeß eine wichtige Rolle. Mutationen der Cysteinreste, die das Zinkion koordinieren, führen zu einer Ansammlung von EGR-1 im Zytoplasma. Eine Zerstörung der Histidin-Cystein-Verbindung im zweiten und dritten Zinkfinger führt zu einem Verlust der Kernlokalisation (Gashler et al., 1993). Die Aktivität der EGR-Transkriptionsfaktoren kann weiterhin über Redoxstufenpotentiale reguliert werden. EGR-Proteine binden an die DNA in Abhängigkeit von Zn2+-, Fe2+-oder Mn2+Ionen (Cao et al., 1990). Die Aktivität von EGR-Proteinen, an die DNA zu binden, könnte somit durch zelluläre Komponenten oder regulatorische Proteine kontrolliert werden, die das zelluläre Redoxstadium durch das Zurückhalten von Zinkionen verändern und dadurch möglicherweise eine oxidierte EGR-Proteinform inaktivieren (Huang and Adamson, 1993). Die transkriptionelle Aktivität von EGR-Proteinen kann durch die Interaktion mit anderen Transkriptionsfaktoren über DNA-Protein- und/oder Protein-Protein-Interaktionen reguliert werden. Hier spielt die Anordnung regulatorischer Elemente in den Promotorregionen EGRregulierter Gene eine große Rolle. Die EGR-Proteine regulieren die Transaktivierung verschiedener Gene, indem sie kooperative Interaktionen mit anderen Transkriptionsfaktoren eingehen. Die meisten der identifizierten Proteine, die mit EGR-1 interagieren, werden in gewebsspezifischer Weise exprimiert und binden als Kernproteine an benachbarte oder nahegelegene EGR-Bindungsstellen. In vielen Promotoren EGR-regulierter Gene überlappen die DNA-Bindungsstellen für EGR-Proteine mit denen für Sp-1 (Krämer et al., 1994; Skerka et al., 1995; Lin und Leonard, 1997; Khachigian et al., 1996; Decker et al., 1998 und 2003). Weiterhin kooperieren EGR-Proteine mit Transkriptionsfaktoren, deren DNA-Bindungsstellen nicht der EGR-Bindungsstelle benachbart sind. Die Transkription des Adhäsionsmolekül-kodierenden Gens ICAM-1 wird von EGR-Proteinen mit den Proteinen der NFAT- und der NFκB-Familie induziert (Zipfel et al., 2003). Ein weiterer Faktor, der mit den EGR-Proteinen kooperiert ist die - 15 - Einleitung NFκB-Untereinheit RelA, welcher die Transkription von NFκB-1-Genen beeinflußt (Chapman and Perkins, 2000; Cogswell et al., 1997). Das Homöobox-Protein PTx-1 und der steroidogene Faktor 1 (SF-1) regulieren zusammen mit EGR-1 die Expression des Luteinisierenden Hormons LH-β (Lee et al., 1996; Tremblay and Drouin, 1999). Das CREB („cAMP-response element binding protein)-bindende Protein (CBP/300) induziert mit EGR-Proteinen die Genexpression der Lipogenase (Silverman et al., 1998). Der Tumorsuppressor p53 bildet einen physikalischen Komplex mit EGR-1 (Liu et al., 2001). EGR-Proteine interagieren mit NFATc bei der FasLPromotoraktivität (Li-Weber et al., 1999) und den Repressorproteinen NAB1 und NAB2 (Russo et al., 1995; Svaren et al., 1996). Der „macrophage stimulating“-Faktor (M-CSF) bindet in Kooperation mit Sp-1 funktional an EGR-1 (Srivastava et al., 1998). Interaktionen von EGR-1 wurden weiterhin mit mehreren viralen Proteinen wie HBx, IE2, tax und tat beobachtet (Yoo et al., 1996 a und b; Trejo et al., 1997; Yang et al., 2002). 1.5.3 Funktionen von EGR-Proteinen Die EGR-Proteine sind in den überwiegenden Fällen aktivatorische Regulatoren der Transkription verschiedener Gene in unterschiedlichen Zellen und Geweben (Tabelle 1). In TZellen werden durch die EGR-Proteine die Gene des Interleukin-2-Rezeptors β (IL-2-Rβ), des Fas-Liganden (FasL) und von NFκB-1 aktiviert (Lin and Leonard, 1997; Mittelstadt and Ashwell, 1998 und 1999; Cogswell et al., 1997; Chapman and Perkins, 2000). Nach der Stimulierung des B-Zellrezeptors exprimieren reife B-Zellen EGR-1 und EGR-2 (Seyfert et al., 1989; Newton et al., 1996). In B-Zellen werden die Gene von BP-1, CD44 und ICAM-1 durch EGR-Proteine aktiviert, während die Gene von CD23 und Fas durch EGR-Proteine reprimiert werden (Maltzman et al., 1996 a und 1996 b; Dinkel et al., 1997 und 1998). EGR-1 transaktiviert den Transkriptionsfaktor nur77, der eine entscheidende Rolle bei der Apoptose von Thymozyten spielt (Woronicz et al., 1994; Williams and Lau, 1993). In Monozyten aktiveren die EGRProteine z. B. das Gen der Superoxid-Dismutase (SOD) (Minc et al., 1999). Im Nervensystem sind die EGR-Proteine bei Differenzierungs- und Proliferationsprozessen beteiligt. Dort aktivieren EGR-Proteine z. B. die Gene des Synapsins I und Synapsins II, der Homöobox B2 (hoxB2), der Phenylethanolamin-N-Methyltransferase (PNMT) und des „basic fibroblast growth“-Faktors (bFGF) (Thiel et al., 1994; Petersohn et al., 1995; Sham et al., 1993; Wong et al., 1998; Biesiada et al., 1996). In der Hypophyse regulieren EGR-Proteine die Gene des Luteinisierenden Hormons-β (Lee et al., 1996; Tremblay and Drouin, 1999). Der „tissue“-Faktor - 16 - Einleitung (TF) in der Epidermis gehört ebenfalls zu EGR-regulierten Genen (Cui et al., 1996). Im Epithel werden die Gene des „platelet derived growth“-Faktors A und B (PDGF A und PDGF B) von EGR-Proteinen transaktiviert (Khachigian et al., 1996). EGR-Proteine aktivieren weiterhin die p53-Gene in Melanomzellen und das pgp2/mdr1β-Gen in Hepatomzellen (Nair et al., 1997; Thottassery et al., 1999). In Fibrosarkomzellen werden die Gene des „transforming growth“Faktors-β1 (TGF-β1) durch EGR-Proteine aktiviert, während das Bcl-2-Gen reprimiert wird (Liu et al., 1996; Huang et al., 1998). Repressorfunktionen üben die EGR-Proteine auf das EGR-1Gen in Fibroblasten und auf das Adenosin-Desaminasegen (ADA) in der C1-1D-Zelllinie der Maus aus (Cao et al., 1993; Ackermann et al., 1991). EGR-4 aus Jurkat T-Zellen besitzt autoinhibitorische Fähigkeiten auf sein eigenes Gen (Zipfel et al., 1997). 1.6 Die NFAT-Proteine Die Mitglieder der „nuclear factor of activated T cells“ (NFAT)-Familie spielen eine zentrale Rolle bei der induzierbaren Genexpression während der Immunantwort (Rao et al., 1997). Die Proteine der NFAT-Familie NFATp (NFAT1), NFATc (NFAT2), NFAT3, NFATx (NFAT4) und NFAT5 werden von fünf Genen kodiert und kommen jeweils in mehreren Isoformen vor (McCaffrey et al., 1993; Northrop et al., 1994; Chuvpilo et al., 1999; Hoey et al., 1995; Masuda et al., 1995; Rao et al., 1997; López-Rodríquez et al., 1999). Obwohl NFAT-kodierende Gene in nahezu jedem Gewebe transkribiert werden, ist die Expression der NFAT-Proteine auf spezifische Gewebe beschränkt (Hoey et al., 1995; Rao et al., 1997; Ranger et al., 1998 a und b). NFATp wird in mehreren Klassen von Immunzellen wie T-Zellen, B-Zellen, Mastzellen und natürlichen Killerzellen exprimiert (Shaw et al., 1988; Ho et al., 1994; Choi et al., 1994; Venkataraman et al., 1994; Prieschl et al., 1995; Aramburu et al., 1995, Loh et al., 1996 a und b). NFATc wird mit hoher Expressionsrate in Skelettmuskeln, im Endokard und in aktivierten lymphoiden Zellen exprimiert, während NFAT3 vorwiegend außerhalb des Immunsystems und NFATx im Thymus exprimiert werden (Northrop et al., 1994; Masuda et al., 1995; Hannum et al., 1995; Hoey et al., 1995; de la Pompa et al., 1998; Ranger et al., 1998 a). In ruhenden Zellen sind NFAT-Proteine phosphoryliert und bleiben im Zytoplasma. In einer stimulierten Zelle aktiviert intrazelluläres Calcium die Calmodulin-abhängige Phosphatase Calcineurin, die im Zytoplasma NFAT-Proteine dephosphoryliert (Timmerman et al., 1996). Die NFAT-Proteine exponieren daraufhin ihre Kernlokalisationssequenz und induzieren damit ihre Translokation in den Kern (Aramburu et al.1998; Luo et al., 1996 a und b; Beals et al., 1997; Masuda et al., 1998; - 17 - Einleitung Zhu et al., 1998). NFAT-Proteine besitzen eine regulatorische NFAT-Homologie-Region (NHR), die alle Calcineurin-abhängigen Funktionen der NFAT-Proteine. Dazu zählen die Zugänglichkeit des konservierten Kernlokalisationssignals (NLS) und der N-terminalen Transaktivierungsdomäne sowie die stimulationsabhängige Zunahme der DNA-Bindung (Shibasaki et al., 1996; Luo et al., 1996 a und b). Neben der NHR-Domäne besitzen die NFATProteine eine DNA-Bindungsdomäne, die strukturell den Rel-Domänen der Rel/NFκB-Proteine gleicht und aufgrund der hohen Homologie (65-70 %) auch als „Rel-similarity“-Domäne (RSD) bezeichnet wird (Jain et al., 1995; Chytil and Verdine, 1996; Rao et al., 1997). Die NFATProteine binden nicht wie die NFκB-Proteine als Dimere, sondern als Monomere an die DNA. Einige NFAT-Bindungsstellen in den regulatorischen Regionen induzierbarer Gene sind Elemente mit der NFAT-Konsensussequenz GGAAAANN und einer benachbarten AP-1Bindungsseite mit der Konsensussequenz TGAC/GTCA (Rao et al., 1997). Im Kern wird die Funktion der NFAT-Proteine durch kooperative Interaktionen reguliert. Bekannte Kooperationspartner von NFAT-Proteinen sind die Proteine der AP-1-Familie. NFAT-Proteine besitzen die Fähigkeit, mit Dimeren der AP-1-Familie (Fos und Jun) einen stabilen, kooperativen DNA-Bindungskomplex zu bilden (Jain et al., 1992; Chytil and Verdine, 1996; Rao et al., 1997; Chen et al., 1998). Die Struktur des NFAT/Fos/Jun-DNA-Komplexes wurde durch eine Röntgenstrukuranalyse aufgeklärt und zeigt die Bindung eines Multiproteinkomplexes aus einem NFAT-Protein und einem Fos/Jun-Heterodimer an die DNA (Chen et al., 1998). In vielen anderen Genen binden NFAT-Proteine mit hoher Affinität regulatorische Elemente, in deren Nähe keine AP-1-Elemente gefunden worden sind. Die NFAT-Transkriptionsfaktoren regulieren die Expression vieler Zytokine wie z. B. IL-2, IL-3, IL-4, IL-5, IL-8, IL-13 und TNF-α (Tabelle 1). Weiterhin werden induzierbare Gene wie die von GM-CSF, IFN-γ, CD4OL, FASL und ICAM-1 während der Immunantwort von NFAT-Proteinen kontrolliert (McCaffrey et al., 1993; Northrop et al., 1994; Masuda et al., 1995; Hoey et al., 1995; Xanthoudakis et al., 1996; Luo et al., 1996 c; Rao et al., 1997; Stranick et al., 1997; Ranger et al., 1998 a und b; Oukka et al., 1998; Hodge et al., 1996). Die Aktivierung von NFAT-Proteinen kann durch die immunsupprimierenden Agenzien Calcineurin A (CsA) und FK506 inhibiert werden (Emmel et al., 1989; Granelli-Piperno et al., 1990; Liu, 1993). Die Inhibition der Expression vieler Zytokingene, der T-Zell-Aktivierung und NFAT-Aktivierung durch die Immunsuppressiva CsA und FK506 ist schon lange bekannt (Schreiber and Crabtree, 1992; Crabtree and Clipstone, 1994; Liu, 1993). CsA und FK506 bilden inhibitorische Komplexe mit intrazellulären Rezeptoren (Immunophiline), die in hoher Anzahl in Zellen vorkommen. In der Zelle binden CsA und FK506 mit hoher Affinität an ihre intrazellulären Rezeptoren Cyclophilin (CysPs) und FK- 18 - Einleitung Bindeprotein (FKBP). Die Medikament-Immunophilinkomplexe binden als aktive immunsupprimierende Agenzien Calcineurin und inhibieren dessen Fähigkeit, NFAT zu dephosphorylieren und führen somit zu einer Hemmung der NFAT-Aktivierung. In den letzten 15 Jahren hat sich die klinische Anwendung von CsA und FK506 bei Organtransplantationen etabliert, da sie die Abstoßungen von Transplantaten minimieren (Wiederrecht et al., 1993). Da CsA und FK506 Calcineurin auch außerhalb betroffener Zellen und Gewebe inhibieren (Dumont et al., 1992), werden sie aufgrund dieser Toxizität nur selten bei anderen Krankheiten wie Asthma, Entzündung und Autoimmunkrankheiten verwendet. 1.7 Die NFκB-Proteine NFκB wurde ursprünglich als B-Zellen spezifischer Faktor identifiziert, der an ein kurzes DNASequenzmotiv der „Enhancer“-Region der leichten Kette des Immunglobulins-κ bindet. NFκBProteine werden jedoch in allen Zelltypen exprimiert und besitzen dort wichtige regulatorische Funktionen bei der Transkription vieler Gene (Baeuerle and Henkel, 1994; Siebenlist et al., 1994). Der NFκB-Prototyp ist der aus p50- und p65-Proteinen bestehende Heterodimerkomplex. Die bisher identifizierten Mitglieder der NFκB-Familie können in zwei Gruppen eingeteilt werden, von denen eine auf den p105- und p100-Vorläuferproteinen basiert, die proteolytisch in die Proteine p50 (NFκB-1) und p54 (NFκB-2) prozessiert werden (Bours et al., 1990; Ghosh and Baltimore, 1990; Kieran et al., 1990; Meyer et al., 1991; Schmidt et al., 1991; Bours et al., 1992 b; Mercurio et al., 1992). Die zweite Gruppe setzt sich aus den Proteinen Rel (c-Rel), v-Rel, Rel A (p65) und Rel B zusammen (Brownell et al., 1989; Nolan et al., 1991; Ruben et al., 1991; Ballard et al., 1992; Ryseck et al., 1992). Die Mitglieder der NFκB-Familie besitzen eine hoch konservierte N-terminale Rel-Domäne, welche die Homo-/Heterodimerisierung der verschiedenen Mitgliedern der NFκB-Familie und die DNA-Bindung vermittelt (Bours et al., 1992 a und b; Ryseck et al., 1992; Ip et al., 1993). Neben den klassischen NFκB-Dimeren bestehend aus p65 (RelA) und p50 (NFκB-1) treten viele Variationen von Dimeren anderer Relenthaltender Proteine auf (Baeuerle and Henkel, 1994; Baeuerle and Baltimore, 1996; Baldwin, 1996). NFκB spielt eine wichtige Rolle bei der Expression vieler zentraler Gene in verschiedenen Geweben und Zellen während der Immunantwort (Tabelle 1, Baldwin, 1996; Baeuerle and Baichwal, 1997; Manning and Anderson, 1994). NFκB-Proteine regulieren die Expression von TNF-α, IL-6, E-Selektin, VCAM-1 und ICAM-1 (Ziegler-Heitbrock et al., 1993; - 19 - Einleitung Chen C. C. et al., 1995; Fujisawa et al., 1996). Eine NFκB-Inhibierung verhindert oder vermindert zumindest die Adhäsion von Leukozyten und deren Transmigration (Collins et al., 1995; Chen C. C. et al., 1995). Aktiviertes NFκB wird in vivo während vieler inflammatorischer Reaktionen gefunden, z. B. bei Artheriosklerose, Sepsis, rheumatoidem Synovium und bei UVgeschädigter Haut (Brand et al., 1996; Handel et al., 1996; Fisher et al., 1996). NFκB wird durch eine aussergewöhnlich große Anzahl verschiedener Signale aktiviert (Baeuerle and Henkel, 1994; Baeuerle and Baichwal, 1997). NFκB-Proteine liegen im Zytoplasma ruhender Zellen in inaktiver Form als Komplex mit den inhibitorischen Proteinen IκB vor. Die Mitglieder der IκBFamilie regulieren die DNA-Bindung und die Translokation der NFκB-Proteine durch die Maskierung des NFκB-Kernlokalisationssignals nahe des C-Terminus der Rel- Homologiedomäne (Henkel et al., 1992). Extrazelluläre Stimuli initiieren eine Signalkaskade, die zur Aktivierung von IκB-Kinasen (IKK-1 und IKK-2) führt, die wiederum die IκB-Proteine im Komplex mit NFκB phosphorylieren (Regnier et al., 1997; DiDonato et al., 1997; Cow et al., 1997; Stancovski and Baltimore, 1997; Maniatis, 1997; Alkalay et al., 1995; Yaron et al., 1997). Phosphoryliertes und ubiquitiniliertes IκB, das noch immer mit NFκB assoziiert ist, wird selektiv durch ein 26S-Proteasom degradiert (Chen Z. et al., 1995). Dieser Prozeß setzt das Kernlokalisationssignal von NFκB frei, so dass NFκB in den Zellkern transloziert wird und dort die Transkription von Zielgenen induzieren kann. - 20 - Einleitung 1.8 Fragestellung Die Mitglieder der EGR-Familie werden durch viele verschiedene Stimuli in diversen Zellen des Immunsystems exprimiert und regulieren die induzierbare Genexpression während der Immunantwort. Bei der Initiierung der inflammatorischen Antwort aktivieren EGR-Proteine das Expressionsprogramm pro- und antiinflammatorischer Schlüsselzytokine im Zusammenspiel mit anderen regulatorischen Transkriptionsfaktoren. Die regulatorische Funktion der EGR-Proteine wird sowohl durch Bindung an die DNA als auch durch Protein-Proteininteraktionen kontrolliert, so dass es sich bei den EGR-Proteinen möglicherweise um Multidomänenproteine handelt. Um eine mögliche Spezifität der EGR-Proteine bei der Interaktion mit anderen Transkriptionsfaktoren nachzuweisen, war es von Interesse, den molekularen Mechanismus der kooperativen Interaktionen aufzuklären. Daher sollten in dieser Arbeit die Proteininteraktion von EGR-3 und EGR-4 mit den NFAT-Proteinen (NFATc und NFATp) näher charakterisiert werden. In Proteinbindungsassays wurden Deletionsmutanten von EGR-3 und EGR-4 eingesetzt, um mögliche Interaktionsdomänen von EGR-Proteinen zu lokalisieren. Die Interaktionsdomänen von EGR-3 und EGR-4 sollten daraufhin auf potentielle Bindungsmotive untersucht werden. Weiterhin sollte die Funktionalität der gefundenen Interaktionsdomänen an EGR-regulierten Genen überprüft werden. Außerdem stellte sich die Frage, ob die Interaktion zwischen EGR- und NFAT-Proteinen einen generellen Mechanismus darstellt und auch bei der ProteinProteininteraktion mit den NFκB-Proteinen zustande kommt. - 21 - Material und Methoden 2 Material und Methoden 2.1 Allgemein verwendete Materialien, Puffer, Medien und Lösungen Die nicht gesondert aufgeführten verwendeten Chemikalien und Reagenzien wurden im Reinheitsgrad zur Analyse von den Firmen Biomol (Hamburg), Merck (VWR International, Dresden), Roth (Karsruhe), Eppendorf (Hamburg), Greiner (Frickenhausen), Quiagen (Hilden), Nunc (Mannheim), Amersham Pharmacia Biotech (München), PALL Life Sciences (Dreieich), Invitrogen (Karlsruhe), BD Biosciences (Heidelberg) und Sigma (Sigma-Aldrich VertriebsGmbH, Dreisenhofen) bezogen. Für Reaktionen in Volumina 0,5 bis 2 ml wurden EppendorfReaktionsgefäße verwendet. Bei der Zellkultur wurden Kulturflaschen, Zellschaber, Pipetten, und „12well“-Platten (3,5 cm2/well, NunclonTM Multidishes) von Nunc (NuncTM Brand Products, Mannheim) und Falconröhrchen von Greiner (Frickenhausen) verwendet. Zentrifugationen erfolgten in den Eppendorf-Tischzentrifugen 5415D, in der Eppendorf-Kühlzentrifuge 5417R (Eppendorf, Hamburg) und einer Beckman AllegraTM 6KR Zentrifuge (Beckman Coulter, Unterschleißheim). Es wurde weiterhin der Thermomixer comfort (Eppendorf, Hamburg), der Heizmagnetrührer IKA RCT basic (Merck, Dresden), das Schüttelgerät Vortex Genie2 (Scientific Industries, Merck, Dresden), der Taumel-Rollenmischer SRT1 (Stuart scientific, Merck, Dresden) und das Schüttelgerät Unimax 2010 (Heidolph, Merck, Dresden) verwendet. Photometrische Bestimmungen der optischen Dichte von Medien, DNA- und Proteinkonzentrationen erfolgten in einem BioPhotometer (Eppendorf, Hamburg) und in dem UV/visible Spectrophotometer Ultrospec 2100 pro (Amersham Pharmacia Biotech, München). Allgemein verwendete Puffer, Medien und Lösungen wurden mit zweifach destilliertem Wasser (ddH2O) angesetzt und ggf. bei 121 °C und 2 x 105 Pa 20 min autoklaviert. Phosphat-gepufferte Salzlösung (PBS; 3,3 mM NaH2PO4 x H2O, 6,7 mM Na2HPO4, 145 mM NaCl, pH 7,5) und Trisgepufferte Salzlösung (TBE; 1 M Tris, 1 M Borsäure, 25 mM Na2-EDTA (TitriplexIII), pH 8,0) wurden verändert nach Sambrook et al. (1989) hergestellt. Für Phosphat-gepufferte, mit DTT und PMSF versetzte Salzlösung (PBS-DP) wurde dem PBS-Puffer (verändert nach Sambrook et al., 1989) kurz vor Gebrauch 1 mM Dithiothreitol (Cleland´s Reagent, DTT, GibcoBRL), 0,5 mM Phenylmethylsulfonylfluorid (PMSF, Roth, Karlsruhe) zugesetzt. Die Tris-gepufferte, mit Ca2+ versetzte Salzlösung (TBE-Ca2+, 10 mM Tris, 150 mM NaCl, 1 mM CaCl2, pH 7,5) und die Tris-gepufferte, mit Ca2+ und Tween-20 versetzte Salzlösung (TBST-Ca2+, 10 mM Tris, 150 mM NaCl, 1 mM CaCl2, 0,05 % Tween-20, pH 7,5) wurden nach Roche (2000) hergestellt. Das - 22 - Material und Methoden Luria-Bertani (LB)-Medium enthielt 10 g pankreatisch verdautes Trypton/Pepton aus Casein, 5 g Hefeextrakt, 10 g NaCl, ad 1 l ddH2O. Luria-Bertani (LB)-Nährböden wurden aus Luria-BertaniMedium mit einem Zusatz von 15 g Agar (DIFCO) hergestellt. Für eine Ampizillin-Selektion wurden die LB-Medien 100 µg/ml Ampizillin (Binotal, Grünenthal) versetzt. 2.2 DNA-Arbeitstechniken zur Herstellung der Expressionsvektoren Für die Charakterisierung von Proteininteraktionen wurden die Sequenzen verschieden kodierender Proteine in eukaryotische und bakterielle Expressionsvektoren kloniert. In dieser Arbeit hergestellte Vektoren, erworbene Klonierungsvektoren bzw. zur Verfügung gestellte Plasmide sind in Tabelle 2, 3 und 4 dargestellt. Tabelle 2: Eukaryotische Expressionsplasmide für EGR-Proteine Plasmid pBSV-8His pBSV-EGR-4 Struktur Referenz r Größe: 9755 bp, Marker: Ap , Replikon: prok. ori, Promotor: Kühn and Zipfel, polyhedrin, kodierend für 8 x Histidin 1995 Vektor: pBSV-8His, Insert humane EGR-4-cDNA, gesamt diese Arbeit kodierender Bereich, 8 x Histidin pBSV-EGR-3 Vektor: pBSV-8His, Insert humane EGR-3-cDNA, gesamt Zipfel et al., 2003 kodierender Bereich, 8 x Histidin Tabelle 3: Bakterielle Expressionsplasmide für NFAT- und NFκB-Proteine Plasmid pGEX-1 Struktur r Referenz q Größe: 4,9 kb, Marker: Ap , lacl , Replikon: prok. ori, Promotor: Smith und Johnsen, tac, kodierend für 26 kD Glutathion-S-Transferase (GST) aus 1988 Schistosoma japonicum. pGEX-NFATc pGEX-NFATp pGEX-p50 pGEX-p65 Vektor: pGEX-1, Insert: NFATc-cDNA, Serfling, gesamt kodierender Bereich, GST persönl. Mitteilung Vektor: pGEX-1, Insert: NFATp-cDNA-Fragment Serfling, (+629/+2065, muriner NFATp-Klon pBS-q1b1), GST persönl. Mitteilung Vektor: pGEX-1, Insert: humane p50-cDNA, Siebenlist, gesamt kodierender Bereich, GST persönl. Mitteilung Vektor: pGEX-1, Insert: humane p65-cDNA, Siebenlist, gesamt kodierender Bereich, GST persönl. Mitteilung - 23 - Material und Methoden Tabelle 4: Bakterielle Expressionsplasmide für deletionale EGR-Proteine Plasmid pBX Struktur Referenz r q Größe: 1399 bp, Marker: Ap , lacl , Replikon: prok. ori, Roche, Promotor: trc, kodierend für Protein C pBX-EGR-4-I Vektor: pBX, Insert humanes Mannheim EGR-4-cDNA-Fragment, Nehmann, kodierender Bereich: +4/+472, Protein C pBX-EGR-4-II Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, Nehmann kodierender Bereich: +474/+975, Protein C pBX-EGR-4-III Vektor: pBX, Insert: humanes Vektor: pBX, Insert: humanes unveröffentlicht EGR-4-cDNA-Fragment, Nehmann kodierender Bereich: +973/+1458, Protein C pBX-EGR-4-IV unveröffentlicht unveröffentlicht EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +1135/+1389, Protein C pBX-EGR-4-VI Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +235/+723, Protein C pBX-EGR-4-VII Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +355/+603, Protein C pBX-EGR-4-VIII Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +415/+543, Protein C pBX-EGR-4-IX Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +4/+381, Protein C pBX-EGR-4-X Vektor: pBX, Insert: humanes EGR-4-cDNA-Fragment, diese Arbeit kodierender Bereich: +577/+975, Protein C pBX-EGR-3-I Vektor: pBX, Insert: humanes EGR-3-cDNA-Fragment, diese Arbeit kodierender Bereich: +4/+291, Protein C pBX-EGR-3-II Vektor: pBX, Insert: humanes EGR-3-cDNA-Fragment, diese Arbeit kodierender Bereich: +292/+504, Protein C pBX-EGR-3-III Vektor: pBX, Insert: humanes EGR-3-cDNA-Fragment, diese Arbeit kodierender Bereich: +505/+780, Protein C pBX-EGR-3-IV Vektor: pBX, Insert: humanes EGR-3-cDNA-Fragment, diese Arbeit kodierender Bereich: +781/+1161, Protein C 2.2.1 Polymerase-Kettenreaktion (PCR) Die Deletionsmutanten von EGR-3 und EGR-4 wurden anhand der Matrizen pSG5-EGR-4 und pSG5-EGR-3 mit Hilfe von PCR-Reaktionen amplifiziert und in den bakteriellen Expressionsvektor pBX kloniert. Die für die PCR benötigten Oligonukleotide wurden von - 24 - Material und Methoden Invitrogen (Karlsruhe) bezogen und sind mit ihren Restriktionsschnittstellen in Tabelle 5 dargestellt. Sofern nicht anders angegeben, sind die inserierten Sequenzen humanen Ursprungs. Tabelle 5: PCR-Primer Name Sequenz Restriktionssseite Position pBX-E4-IV-F aaaagcttgccttcgcttgcccggtggagagttgt Hind III + 1135 pBX-E4-IV-R aagaattcctgcttgaggtgcaccttgctgtgccg Eco RI + 1389 pBX-E4-VI-F aaaagcttggcctcagctacagcggtagcttcttc Hind III + 235 pBX-E4-VI-R aagaattcctcaatcttggtccctattacgggaaa Eco RI + 723 pBX-E4-VII-F aaaagcttgcagcagcgtccagatccccgctggat Hind III + 355 pBX-E4-VII-R aagaattcgaaggcagagacagcgtccagcgctgg Eco RI + 603 pBX-E4-VIII-F aaaagcttcgggtccgccggacctttactccccgg Hind III + 415 pBX-E4-VIII-R aagaattcgtcgggcggggagagctgaggctcata Eco RI + 543 pBX-E4-IX-F aaaagcttctccaccttagcgagttttccgaaccc Hind III +4 pBX-E4-IX-R aagaattccatccagcggggatctggacgctgctgc Eco RI + 381 pBX-E4-X-F aaaagcttccagcgctggacgctgtctctgccttc Hind III + 577 pBX-E4-X-R aagaattcagggaagtccgccgcggcggcgctgcg Eco RI + 975 pBX-E3-I-F aaaagcttaccggcaaactcgccgagaagctgccggtg Hind III +4 pBX-E3-I-R aagaattcccagttggaaggggagtcgaaggcgaactt Eco RI + 291 pBX-E3-II-F aaaagctttctttccacgacccccagggcaatcccggg Hind III + 292 pBX-E3-II-R aagaattccccgggattgccctgggggtcgtggaaaga Eco RI + 505 pBX-E3-III-F aaaagcttctcgcctattccccccaggattaccaatcg Hind III + 505 pBX-E3-III-R aagaattccttgcggggccggatgggcttgag Eco RI + 780 pBX-E3-IV-F aaaagctttaccccaaccggcctagcaagacaccgctctc Hind III + 781 pBX-E3-IV-R aagaattcggcgcaggtggtgaccacgggggccag Eco RI + 1161 pBSV-EGR-4-F aaagatcttataaatatgctccaccttagcgagttttcc Bgl II +1 pBSV-EGR-4-F cccggggaattcgagagaagcgaaggagaggcccag Eco RI + 1458 Die Primer wurden lyophilisiert geliefert und mit ddH2O auf die gewünschte Konzentration eingestellt. Der PCR-Reaktionsansatz wurde mit 10 x PCR-Puffer (15 mM MgCl2, 0,5 M KCl, 0,1 M Tris pH 9,0; Amersham Pharmacia Biotech, München), 200 µM dNTP-Mix, 100 ng DNAMatrize, 25 pmol 5´-Primer, 25 pmol 3´-Primer und 0,5 µl Taq-Polymerase (2,5 U; Amersham Pharmacia Biotech, München) in einem Volumen von 100 µl ddH2O in 200 µl PCRReaktionsgefäßen angesetzt. Denaturierungstemperatur von Die 94 PCR-Reaktion °C, fand in 25 Extensionstemperatur Zyklen von 72 bei °C einer und Hybridisierungstemperatur von 52 °C, jeweils für 1 min, im Gene-Amp-PCR-System 9700 - 25 - Material und Methoden (Applied Biosystems, Darmstadt) satt. Das Ergebnis der Reaktion wurde durch Agarosegelelektrophorese überprüft. Die elektrophoretische Auftrennung der DNA erfolgte gemäß der Größe der zu trennenden Fragmente mit 1-2 % Agarosegelen (Agarose NA, speziell aufgereinigt für Gelelektrophorese von Nukleinsäuren; Amersham Pharmacia Biotech, München) in 1 x TBE mit 0,5 µg/ml Ethidiumbromid in horizontalen Flachgelapparaturen (wide mini-sub Cell GT, Bio-Rad, Krefeld) in 1 x TBE-Puffer als Laufpuffer bei maximal 10 V/cm Gellänge (Electrophoresis Power Supply EPS 301, Amersham Pharmacia Biotech, München). Die DNA-Proben wurden mit 5 x DNA-Probenpuffer (Sambrook et al., 1989; 0,25 % (w/v) Bromphenol, 0,25 % (w/v) Xylencyanol FF, 30 % (v/v) Glycerol in H2O) versetzt. Als Längenvergleich diente dabei ein Markergemisch aus λ-DNA-Hind III und ΦX174 DNA-Hae III (ready-to-use-Marker, Finnzymes, Espoo, Finnland). Die Auftrennung wurde mit Hilfe der Geldokumentationsanlage GeneGenius Bio Imagingsystem (Syngene, Merck, Dresden) dokumentiert. Anschließend wurde der gesamte PCR-Reaktionsansatz aufgereinigt. Die Aufreinigung von PCR-Produkten erfolgte direkt nach einer PCR-Reaktion mit dem „QIAquick PCR Purification Kit“ (Quiagen, Hilden) nach Angaben des Herstellers. Die Elution erfolgte in 15-30 µl ddH2O. 2.2.2 Restriktion und Ligation von DNA Der DNA-Restriktionsverdau wurde mit den Restriktionsenzymen Eco RI, Hind III, Xba I und Bgl II (Amersham Pharmacia Biotech, München) unter Verwendung des „One-Phor-All Plus“Puffers (10 x, 10 mM Tris-HCl, pH 7,5, 50 % Glycerol, 100 mM KCl, 1 mM DTT, 0,1 mM EDTA, 300 µg BSA/ml, Amersham Pharmacia Biotech, München) durchgeführt, wobei das Volumen der insgesamt eingesetzten Restriktionsenzyme maximal 10 %, das Puffervolumen 1/10 des Gesamtansatzes einnahm. Die Reaktionsansätze wurden für 1-3 h je nach eingesetztem Gesamtvolumen (10-180 µl) bei 37 °C im Thermomixer inkubiert und bei 4 °C bzw. auf Eis beendet. Nach einer Aufreinigung wurde die restringierte DNA elektrophoretisch überprüft (2.2.1). - 26 - Material und Methoden Ligationen restringierter DNA-Fragmente und restringierter Vektor-DNA wurden mit 1 U T4DNA-Ligase (Roche, Mannheim) und dem mitgelieferten Puffer (10 x Puffer: 660 mM Tris-HCl, 50 mM MgCl2, 10 mM Dithioerythrit, 10 mM ATP, pH 7,5) in einem Gesamtvolumen von 12 µl über Nacht bei 14 °C im Thermomixer durchgeführt. Das Verhältnis von Vektor- zu Insert-DNA betrug 1:3 bis 1:5. Das Ergebnis der Ligation wurde elektrophoretisch (2.2.1) überprüft. 2.2.3 Herstellung und Transformation kompetenter Bakterienzellen Als Bakterienstamm wurde der Escherichia coli-Stamm DH5α (Hanahan, 1983) verwendet. Er besitzt den Genotyp supE44, ∆lacU169 (Φ80, lacZ∆M15), hsaR17, recA1, endA, gyr96, thi1 und relA1. Zur Anzucht einer Bakterienkultur wurde eine entsprechende Menge LB- oder LB-AmpMedium mit einer Bakterienkolonie angeimpft und über Nacht bei 37 °C im Schüttelinkubator (Infors) angezogen (Übernachtkultur). Die Herstellung kompetenter E. coli DH5α-Zellen erfolgte nach Hanahan (1983). 100 ml LBMedium wurden mit 1 ml einer Übernachtkultur angeimpft und bei 37 °C im Schüttelinkubator bis zu einer optischen Dichte (OD600) von 0.45-0.55 inkubiert. Nach der Zentrifugation (5 min bei 4 °C und 3000 x g) wurde das Pellet in 30 ml eiskaltem TFBI-Puffer (30 mM Na-Acetat, pH 6,0, 50 mM MnCl2, 100 mM NaCl, 10 mM CaCl2, 15 % Glycerin) vorsichtig resuspendiert, für 10 min auf Eis inkubiert und erneut zentrifugiert (10 min bei 4 °C und 3000 x g). Nach vorsichtigem Resuspendieren des Zellsedimentes in 4 ml eiskaltem TFBII-Puffer (10 mM MOPS, 75 mM CaCl2, 10 mM NaCl, 15 % Glycerin) wurden die Zellen in Aliquots bei -80 °C gelagert. Die Transformation von Bakterien wurde nach der Methode von Dagert und Ehrlich (1979) durchgeführt. Für die Transformation wurden zu 0,5 µl Plasmid-DNA 20-100 µl kompetente E. coli DH5α-Zellen hinzugefügt, durch Schnippen gemischt, 30 min auf Eis inkubiert und für den Hitzeschock 45 s bei 42 °C im Thermomixer (300-400 rpm) geschüttelt. Anschließend wurde der Ansatz mit 200 µl LB-Medium versetzt und für 45-60 min bei 37 °C im Thermomixer (300-400 rpm) geschüttelt und 50-100 µl bei gegebener Ampizillinresistenz auf LB-Amp-Platten ausgestrichen. Die Inkubation des ausplattierten Transformationsansatzes erfolgte über Nacht bei 37 °C (Heraeus B6420, Heraeus, Hanau). Die Transformation mit Ligationsansätzen wurde wie oben beschrieben durchgeführt. Davon abweichend wurden 6 µl Ligationsansatz und 100 µl - 27 - Material und Methoden kompetente E. coli-DH5α-Zellen verwendet. Zudem wurde der gesamte Transformationsansatz bei gegebener Ampizillinresistenz auf LB-Amp-Platten ausgestrichen. 2.2.4 Plasmidpräparation Die Plasmidisolierung in kleinem Maßstab erfolgte aus 1-1,5 ml Übernachtkulturen transformierter E. coli DH5α-Kolonien mit dem „GFX TM Micro Plasmid Prep Kit“ (Amersham Pharmacia Biotech, München) nach Angaben des Herstellers. Die Zellkulturen wurden zentrifugiert (1 min bei RT, 14 000 rpm), das Zellsediment in 150 µl Lösung I (100 mM Tris pH 7,5, 10 mM EDTA, 400 µg/ml RNase I) resuspendiert, durch Zugabe von 150 µl Lösung II (1 M NaOH, 5,3 % SDS ad 26 ml ddH2O) durch Schwenken lysiert und mit 300 µl Lösung III (Puffer mit Acetat und Chaotrop) in Lösung gebracht. Nach 5 min Zentrifugation bei 14000 rpm wurde die Plasmid-DNA auf „MicroSpinTM“-Säulen mit einer Glasfibermatrix 1 min inkubiert, zentrifugiert (30 s bei 14 000 rpm und RT) und mit 400 µl Waschpuffer (Tris-EDTA in 48 ml absoluten Ethanol) gewaschen. Die Elution erfolgte durch Zentrifugation (1 min bei 14 000 rpm und RT) mit 50 µl ddH2O. Die Präparation von größeren Plasmidmengen für den Einsatz in transienten Transfektionen wurde mit dem „Plasmid Megakit“ (Quiagen, Hilden) durchgeführt. Nach Anzucht einer E. coli DH5α-Kolonie in 1-2 ml LB-Amp-Medium für 8 h bei 37 °C im Schüttelinkubator, wurden 500 µl der Schüttelkultur in 500 ml LB-Amp-Medium bei denselben Bedingungen über Nacht inkubiert. Das Medium wurde durch Zentrifugation (15 min bei 4 °C 6000 rpm; Sorvall Super T21) entfernt, das Zellsediment mit 50 ml eiskalter Lösung P1 (50 mM Tris-HCl, pH 8,0, 10 mM EDTA, 100 g/ml RNase A) resuspendiert und nochmals zentrifugiert (30 min bei 4 °C und 11000 rpm, Sorvall Super T21). Nach Zugabe von 50 ml Lösung P2 (200 mM NaOH, 1 % SDS) wurden die Zellen für 5 min bei RT lysiert und mit 50 ml eiskalter Lösung P3 (3 mM Kaliumacetat, pH 5,5) für 30 min auf Eis neutralisiert. Vor dem Auftragen des Lysates auf die Säule wurde die Säule mit 35 ml Lösung QBT (750 mM NaCl, 50 mM MOPS (3-(NMorpholino)-propansulfonsäure), pH 7,0, 15 % Isopropanol, 0,15 % Triton X-100) äquilibriert und das Lysat durch eine Mullkompresse (5 x 5 cm, 8-fach) filtriert. Anschließend wurde die Säule mit 200 ml Lösung QC (1 mM NaCl, 50 mM MOPS, pH 7,0, 15 % Isopropanol) gewaschen. Nach der Elution der Plasmide von der Säule mit 35 ml QF-Lösung (1,25 M NaCl, - 28 - Material und Methoden 50 mM Tris-HCl, pH 8,5, 15 % Isopropanol) wurde die DNA mit 0,7 Volumen Isopropanol präzipitiert, zentrifugiert (30 min bei RT und 3500 rpm), mit 5 ml 70 % Ethanol gewaschen und mit 1 ml 70 % Ethanol in 1,5 ml Eppendorf-Reaktionsgefäße überführt. Durch weitere Zentrifugation wurde das Ethanol von der DNA entfernt und das Pellet getrocknet. Die DNA wurde in ddH2O aufgenommen und nach der Konzentrationsbestimmung bei-20 °C gelagert. 2.2.5 DNA-Sequenzierung Die Sequenzierungen wurden nach der „Cycle-Sequenzierungs“-Methode von Sanger (1977) mit dem „BigDye Terminator v3.1 Cycle Sequencing Kit“ in einem ABI-Prism 3100 (Genetic Analyser, Applied Biosystems, Darmstadt) durchgeführt. Für den Reaktionsansatz der Kettenabbruchreaktion wurden 200 ng DNA, 4 µl „BigDye Ready-Reaction-Mix” (2,5 x, AmpliTaq-Polymerase FS, thermostabile Pyrophosphatase, dNTP, dITP, BigDye-markierte ddNTP), 2 µl „BigDye-sequencing“-Puffer (5 x), 3,2 pmol Primer mit HPLC-H2O auf ein Gesamtvolumen von 20 µl gebracht. Die Kettenabbruchsreaktion (BigDye terminator reaction) erfolgte im Gene-Amp-PCR-System 9700 (Applied Biosystems, Darmstadt) in 25 Zyklen, die jeweils aus 10 s Denaturierung bei 96 °C, 5 s Hybridisierung bei 50 °C und 4 min Extension bei 60 °C bestanden. Die Reaktionsprodukte wurden durch Zugabe von 80 µl 75 % Isopropanol gefällt, nach 15 min für 45 min bei 2800 x g zentrifugiert, mit 150 µl 70 % Ethanol gewaschen und in 10 µl Hi-Di Formamid aufgenommen. Die Sequenzierungen der in dieser Arbeit hergestellten Klone im pBX-Vektor wurden mit den Primern pBX-Forward (5´-gaccggaattatcgattaactt) und pBX-Reverse (5´-atagggagtcccttacttacca) durchgeführt. Für die Sequenzüberprüfung der pBSV-8His-Expressionsvektoren wurden die Primer pBSV-8His-Forward (5´-tttactgttttcgtaacagttt) und pBSV-8His-Reverse (5´- ttccatccaacgacaagcttca) verwendet. Die Sequenzen wurden mit Hilfe des „standard nucleotidenucleotide Blast“-Programmes mit der Datenbank verglichen. Weiterhin wurde das „GenoDB“Programm von Heiko Richter für Sequenzvergleiche genutzt. - 29 - Material und Methoden 2.3 Expression eukaryotischer und prokaryotischer Proteine 2.3.1 Zellkultur und Expression eukaryotischer Proteine Die Zellkulturarbeiten wurden unter sterilen Bedingungen an der Sterilwerkbank HeraCell (Heraeus, Hanau) durchgeführt. Für mikroskopische Analysen wurde das Auflicht-Binokular Axiovert 25 (inverses Mikroskop, Carl Zeiss, Jena) verwendet. 2.3.1.1 Transiente Transfektion und Luciferase-Reportergenassay Die transiente Transfektion von humanen 293T-Zellen (primäre embryonale Nierenzelllinie) erfolgte in „12well“-Platten bei etwa 40-60 % Konfluenz mit der „multi-komponenten“Verbindung FuGENETM 6 für die Transfektion eukaryotischer Zellen (Roche, Mannheim). Die in transienten Transfektionen verwendeten Plasmide sind in Tabelle 6 und Tabelle 7 dargestellt. Für die transiente Transfektion wurden humane 293T-Zellen in 20 ml RPMI 1640-Medium (Invitrogen, Karlsruhe) inklusive 10 % hitzeinaktiviertem FCS (PAA Laboratories GmbH, Cölbe) und 1 % w/v L-Glutamin (200 mM; BioWhittaker Cambrex, Verviers, Belgien) in 80 cm2 Zellkulturflaschen im Brutschrank (Heraeus, Hanau) bei 37 °C und 5 % CO2 bei ausreichender Luftfeuchtigkeit kultiviert. Der Medienwechsel und die Zellpassage erfolgten dreimal die Woche. Die Zellen wurden bei einer Konfluenz von 70 % vom Flaschenboden abgespült, in 50 ml Falconröhrchen überführt und das verbrauchte Medium von den Zellen durch Zentrifugation (5 min bei RT und 300 x g) entfernt. Die Zellen wurden mit frischem Kulturmedium im Verhältnis 1:10 bis 1:20 auf neue Zellkulturflaschen verteilt. Die transiente Transfektion wurde nach Angaben des Herstellers (Roche, Mannheim) durchgeführt. Für jeden Transfektionsansatz wurden 4 x 105 Zellen mit 0,2 µg Reporterplasmid, 0,6 µg der jeweiligen Expressionsplasmide und 0,032 µg pRL-SV40 verwendet (Tabelle 6 und 7). Die Gesamtkonzentration der DNA wurde mit pSG5-Plasmid-DNA konstant gehalten. Nach der Transfektion wurden die Zellen über Nacht bei 37 °C inkubiert. - 30 - Material und Methoden Tabelle 6: Reporterplasmide für die transiente Zelltransfektion Plasmid pGL-2 pZNA3-Luc pTNF-Luc Struktur Referenz r Größe: 5,6 kb, Marker: Ap , Replikon: colE1, f1, Promega, kodierend für die „Firefly“-Luciferase aus Photinus pyralis Mannheim Vector: pGL-2-Basic, Insert: IL-2 Promotor-Fragment Decker et al., kodierender Bereich: -302/-257, 3x und -63/+51 1998 Vector: pGL-2-Basic, Insert: TNF-α-Promotor-Fragment, kodierender Zipfel et al., Bereich: -174/-143, 3x 2003 Tabelle 7: Expressionsplasmide für die transiente Zelltransfektion Plasmid Struktur Referenz pSG5 Größe: 3,7 kb, Marker: Apr, Replikon: prok., euk. ori, Promotor: Stratagene, T7, SV 40 (früh) La Jolla, USA Vektor: pSG5, Insert: humane EGR-4-cDNA, gesamt kodierender Decker et al., 2003 pSG5-EGR-4 Bereich +1/1461 pSG5-EGR-4 MI Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kodierender Bereich: +450/+1461 pSG5-EGR-4 MII Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kodierender Bereich: +975/+1461 pSG5-EGR-4 MIII Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kodierender Bereich: +1131/+1461 pSG5-EGR-4 MIV pSG5-EGR-4 MV Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel, kodierender Bereich: +1/+1392 unveröffentlicht Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kodierender Bereich: +1131/+1392 pSG5-EGR-4 MVI Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kodierender Bereich: +513/+975 pSG5-EGR-4 MVII Vector: pGL-2-Basic, Insert: humanes EGR-4-cDNA-Fragment, Zipfel et al., 2003 kod. Bereich: 513/+975, +1131/+1392 pMTZT-p50 pMTZT-p65 pRSV-NFATc pSH210 pRL-SV40 Vektor: pMTZT, Insert: humane p50-cDNA, Siebenlist, gesamt kodierender Bereich persönl. Mitteilung Vektor: pMTZT, Insert: humane p65-cDNA, Siebenlist, gesamt kodierender Bereich persönl. Mitteilung Vektor: pRSV, Insert: humane NFATc-cDNA, Serfling, gesamt kodierender Bereich persönl. Mitteilung Vektor: pBJ5, Insert: NFATp-cDNA, kodierend für die 755 C- Crabtree, terminalen Aminosäuren von NFATp persönl. Mitteilung Vektor: pRL, Insert: Renilla-Luciferase-cDNA (Rluc), kloniert aus Promega, Renilla reniformis. Mannheim - 31 - Material und Methoden Transfizierte 293T-Zellen wurden nach 18-24 Stunden geerntet, zwei mal mit 1 x PBS gewaschen, mit 250-400 µl „Passiv Lysis Buffer“ (Promega) lysiert und 10 s bei 12.500 g zentrifugiert. Die Zelllyse und die Messung der Luciferaseaktivität wurden mit dem „DualLuciferase TM “-Reporter-Assay-System durchgeführt. Die Lichtreaktion wurde hervorgerufen, indem 5 µl Zelllysat (1:20 in 1 x PBS) mit 25 µl „Luciferase Assay Reagent II“ (Promega) in Mikrotiterplatten (EG&G Berthold) vermischt wurden. Anschließend wurde das Gesamtintegral der Firefly-Luciferase-Lichtemission über 10 s bei RT in einem Luminometer (LuMistarl; BMG, Labtechnologies) gemessen. Durch die Zugabe von 25 µl „Stop &GloTM Reagent“ (Promega) wurde die Aktivität der Firefly-Luciferase inhibiert und zugleich die Renilla-Luciferase aktiviert. Die resultierende Lichtemission wurde wiederum über einen Zeitraum von 10 s gemessen. Von allen Lysaten wurden Dreifachbestimmungen vorgenommen. 2.3.1.2 Expression nativer EGR-Proteine Die humane Jurkatzelllinie (T-Zell-Hybridom) exprimiert nach mitogener Stimulation EGRProteine. Die Jurkat-T-Zellen wurden in 300 ml RPMI 1640-Medium (Invitrogen, Karlsruhe) inklusive 10 % hitzeinaktiviertes FCS (PAA Laboratories GmbH, Cölbe) und 1 % w/v LGlutamin (200 mM; BioWhittaker Cambrex, Verviers, Belgien) in 185 cm2 Zellkulturflaschen im Brutschrank (Heraeus, Hanau) bei 37 °C, 5 % CO2 und ausreichender Luftfeuchtigkeit kultiviert. Nach Bestimmung der Zellzahl wurden die Zellen zentrifugiert (5 min bei RT und 300 x g) und in entsprechender Zellzahl auf neue Zellkulturflaschen verteilt, so dass die Dichte der Jurkatzellen zwischen 0,5 x 105 Zellen pro ml und 1 x 106 Zellen pro ml betrug. Jurkatzellen wurden zur Gewinnung von nativen EGR-Proteinen für 2-3 h bei 37 °C durch Zugabe von 1 µg/ml PHA (Phytohämagglutinin; Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) und 20 ng/ml PMA (Phorbol 12-myristat 13-acetat; Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) bei einer Dichte von 1 x 106 Zellen pro ml stimuliert. 2.3.1.3 Expression rekombinanter EGR-Proteine Rekombinante Proteine von EGR-3 und EGR-4 wurden im Baculovirus-Expressionssystem exprimiert. Hierbei werden Zellen mit den entsprechenden Expressionsvektoren und einer linearisierten „BaculoGoldTM-Baculovirus“-DNA (PharMingen, BD) kotransfiziert. Durch - 32 - Material und Methoden homologe Rekombination des Expressionsvektors und der viralen DNA entstehen Viren, welche die entsprechenden Proteine von der Wirtszelle produzieren lassen. Für die Expressionsvektoren wurde in dem 9755 bp großen Vektor pBSV-8His (Kühn and Zipfel, 1995) die gesamt kodierende Region der humanen EGR-4-cDNA (Zipfel et al., 1997) und die gesamt kodierende Region der humanen EGR-3-cDNA (Zipfel et al., 2003) zwischen die Restriktionsstellen von Bgl II und Eco RI inseriert. Die Insektenzelllinie Sf9 (Intestinalzellen von Spodoptera frugiperda) IPLB-Sf21-AE wurde in 35 ml Insect-Xpress-Medium (BioWhittaker Cambrex, Verviers, Belgien) inklusive 4 % hitzeinaktiviertem FCS (PAA Laboratories GmbH, Cölbe) als Monolayer in 185 cm2 Zellkulturflaschen im Brutschrank (Heraeus, Hanau) bei 27 °C und 95 % Luftfeuchtigkeit kultiviert. Der Medienwechsel erfolgte dreimal die Woche, indem die Zellen mit einem Zellschaber vom Flaschenboden gelöst und zentrifugiert (5 min bei RT und 300 x g) wurden. Die Zellen wurden daraufhin im Verhältnis 1:3 oder 1:4 in 15 ml Grace´s-Insect-Medium (BioWhittaker Cambrex, Verviers, Belgien) aufgenommen, auf neue Kulturflaschen verteilt und für 30 min zum Adhärieren im 27 °C-Brutschrank inkubiert. Danach erfolgte ein Ersatz des Mediums durch Kulturmedium. Zur Expression rekombinanter Proteine und Gewinnung von Virusstocklösungen wurden Sf9Zellen bei ca. 60 -70 % Konfluenz nach dem Ersatz des Mediums mit rekombinanten Viren bei einer „multiplicity of infection“ (MOI) von 5-7 infiziert. Zur Gewinnung von Virusstocklösung wurden die Sf9-Zellen für 10-11 Tage, zur Proteinexpression für drei Tage im Brutschrank bei 27 °C inkubiert. 2.3.2 Extraktion eukaryotischer Proteine Für die Extraktion nativer und rekombinanter EGR-Proteine aus Jurkat-T-Zellen und Sf9Insektenzellen wurden Ganzzellextrakte hergestellt. Jurkatzellen der Dichte 1 x 106 wurden direkt nach 2,5 h mitogener Stimulierung (2.3.1.2) bzw. Sf9-Zellen einer 185 cm2 Zellkulturflasche 3 Tage nach der Infektion (2.3.1.3) geerntet und abzentrifugiert (5 min bei 4° und 300 x g). Die Zellsedimente wurden mit 50 ml eiskaltem PBS-DP gewaschen und in 1,5 ml eiskaltem hypotonischem RSB-Puffer (10 mM Tris-HCl pH 7,4, 10 mM NaCl, 1,5 mM MgCl2; kurz vor Gebrauch zugesetzt: 10 mM NaF, 0,5 mM DTT, 0,1 mM PMSF) für 10 min auf Eis - 33 - Material und Methoden inkubiert. Die Zelllyse erfolgte mit einer dem Sedimentvolumen entsprechenden Menge eiskaltem Puffer C (20 mM HEPES pH 7,9, 420 mM NaCl, 0,2 mM EDTA, 25 % Glycerin, 1,5 mM MgCl2; kurz vor Gebrauch zugesetzt: 10 mM NaF, 0,5 mM DTT, 0,1 mM PMSF) für 45 min auf Eis. Die Lysate wurden abzentrifugiert (10 min bei 14 000 rpm) und die Proteinenthaltenden Überstände bei -80 °C gelagert. Die Zellextrakte wurden für die weitere Verwendung mit Puffer C ohne NaCl auf eine Salzkonzentration von 120 mM verdünnt. Der Erfolg der Proteinexpression wurde nach der Auftrennung der Proteine in der SDS-PAGE (2.4.1), dem Proteintransfer (2.4.3) mit spezifischen Antikörpern (2.4.4) überprüft. Für die Verwendung in den Plasmonresonanz-Studien (2.4.7) wurden die Zellextrakte mit der NickelAffinitätschromatographie (2.3.3) aufgereinigt, mit Entsalzungssäulen PD-10 (desalting column; Amersham Pharmacia Biotech, München) oder im Mikrodialysiersystem (QuixSep micro dialyser; Roth, Karlsruhe) nach Angaben des Herstellers in 1 x PBS überführt. Gegebenenfalls wurden die aufgereinigten und dialysierten Proteine mit den Konzentratoren Ultrafree-0.5 (centrifugal filter device Biomax-5 und Biomax-10; Millipore, Schwalbach) nach Angaben des Herstellers einkonzentriert. 2.3.3 Affinitätschromatographie zur Aufreinigung rekombinanter EGR-Proteine Rekombinante EGR-Proteine wurden in Sf9-Insektenzellen als Histidin-Fusionsproteine exprimiert und konnten mit Hilfe der Nickel-Affinitätschromatographie aufgereinigt werden. Die Affinitätschromatographie wurde nach Angaben des Herstellers (QIAexpressionist 2001; Quiagen, Hilden) modifiziert. 1-3 ml Nickel-NTA-Agarose wurden mit 3 ml „Charge“-Puffer (400 mM NiSO4) für 30 min inkubiert und mit 12-14 ml eiskaltem „Binding“-Puffer (5 mM Imidazol in 1 x PBS, pH 7,5) bei 4 °C gewaschen. Nach der Zentrifugation (5 min bei RT und 500 x g) wurden 1-3 ml Zellextrakt bei 4 °C über Nacht mit der Nickel-NTA-Agarose inkubiert, dreimal mit 12-14 ml und einmal mit 1 Säulenvolumen eiskaltem „Binding“-Puffer gewaschen. Die aufgereinigten Proteine wurden mit 1-2 ml eiskaltem Elutionspuffer (4 M Imidazol in 1 x PBS, pH 7,5) für 30 min bis 1 h bei 4 °C von der Säule gelöst. Die Elutionsfraktionen wurden bei -80 °C gelagert oder für analytische Versuche weiter aufbereitet. Sämtliche Fraktionen der Aufreinigung wurden mit spezifischen Antikörpern analysiert. Zusätzlich wurde die Proteinkonzentration im Biophotometer (Eppendorf, Hamburg) bestimmt. - 34 - Material und Methoden 2.3.4 Expression prokaryotischer Proteine Die Expression prokaryotischer Proteine erfolgte modifiziert nach einer von Smith und Johnsen (1988) beschriebenen Methode. Rekombinante EGR-4-Deletionsmutanten, NFAT- und NFκBProteine wurden in E. coli DH5α exprimiert. Eine 10 ml Übernachtkultur mit entsprechend transformierten Zellen wurde 1:100 in 100-200 ml LB-Selektionsmedium (0,2 % Glukose, 50 µg/ml Ampizillin) aufgenommen und bei 37 °C bis zu einer optischen Dichte (OD600) von 0,60,8 inkubiert. Die Proteinexpression wurde durch 0,4 mM IPTG (Isopropyl-1-thio-β-DGalactosid; Stratagene, La Jolla, USA) 1-2 h bei 37 °C induziert. Alle folgenden Schritte wurden bei 4 °C bzw. auf Eis durchgeführt. Die Zellen wurden für 15 min bei 4 °C und 2800 x g zentrifugiert und mit 50 ml eiskaltem PBS-DP gewaschen. Die Zelllyse erfolgte in 2-4 ml eiskaltem PBS-DP durch Ultraschallbehandlung (1 min, Stufe 10 im Ultraschall-Desintegrator Sonifer II; Branson, Dietzenbach). Die Lysate wurden bei 10.000 x g für 10 min bei 4 °C sedimentiert und die im Überstand befindlichen, rekombinanten Proteine im Westernblot überprüft. Die restlichen Lysate wurden in 30µl- bzw. 200 µl-Aliquots bei-80 °C gelagert oder zur Proteinaufreinigung direkt einer „GST“- bzw. Protein C-Affinitätschromatographie unterzogen (2.3.5). 2.3.5 Affinitätschromatographien zur Aufreinigung bakterieller Proteine Die rekombinanten Glutathion-S-Transferase (GST)-gekoppelten NFAT-Fusionsproteine wurden nach einer modifizierten Methode von Smith und Johnsen (1988) aus Zellextrakten über die Bindung der 26 kD Glutathion-S-Transferase aus Schistosoma japonicum an ihr Substrat Glutathion aufgereinigt. Die Glutathionagarose (Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) wurde nach Angaben des Herstellers aufbereitet und gelagert. Für eine Aufreinigung wurden 1-3 ml Glutathionagarose dreimal mit 12-14 ml PBS-DP gewaschen und zentrifugiert (5 min bei RT und 500 x g). 1-5 ml Zellextrakt wurde bei 4 °C über Nacht mit der Glutathionagarose inkubiert. Nach Zentrifugation (5 min bei 4 °C und 500 x g) wurde das Säulenmaterial dreimal mit 10 Säulenvolumen eiskaltem PBS-DP und einmal mit 1 Säulenvolumen PBS-DP gewaschen. Die Elutionsfraktionen wurden bei -80 °C gelagert oder für analytische Versuche weiter aufbereitet. Sämtliche Fraktionen der Aufreinigung wurden im Westernblot (2.4.4) analysiert. Zusätzlich wurde die Proteinkonzentration im Biophotometer (Eppendorf, Hamburg) bestimmt. - 35 - Material und Methoden Die EGR-4-Deletionsmutanten wurden rekombinant mit einem C-terminalen Protein C exprimiert und mit einer „Anti-Protein C-Affinitätsmatrix“ (Roche, Mannheim) nach Angaben des Herstellers aufgereinigt. 100 µl „Anti-Protein C-Affinitätsmatrix“ wurden in ein 2 ml Eppendorf-Reaktionsgefäß überführt, für 15 min mit 10 Volumen Äquilibrierungspuffer (20 mM Tris, 0,1 M NaCl, 1 mM CaCl2, pH 7,5; kurz vor Gebrauch zugesetzt: 0,5 mM DTT, 0,1 mM PMSF) bei RT inkubiert, zentrifugiert (5 min bei RT und 2000 rpm) und für 30 min bei 4 °C mit 10 Volumen Puffer erneut äquilibriert. Die Inkubation von 1,5 ml Protein, das üblicherweise aus 150 ml Bakterienkultur (2.3.4) gewonnen wurde, erfolgte bei 4 °C für 1-3 h. Nach Zentrifugation (5 min bei 4 °C und 2000 rpm) wurden viermal für 10 min mit 10 Volumen eiskaltem Waschpuffer (20 mM Tris, 1M NaCl, 1 mM CaCl2, pH 7,5; kurz vor Gebrauch zugesetzt: 0,5 mM DTT, 0,1 mM PMSF) und einmal für 30 min mit 10 Volumen Äquilibrierungspuffer gewaschen. Die Elution der Proteine erfolgte dreimal für je 30 min bei 4 °C mit EDTA-Elutionspuffer (20 mM Tris, 1M NaCl, 5 mM EDTA, pH 7,5; kurz vor Gebrauch zugesetzt: 0,5 mM DTT, 0,1 mM PMSF). Die Eluate wurden aliquotiert und bei -80 °C für die Lagerung eingefroren. Die aufgereinigten Proteine wurden bei Bedarf mit den Entsalzungssäulen PD-10 (desalting column; Amersham Pharmacia Biotech, München) oder in einem Mikrodialysesystem (QuixSep micro dialyser; Roth, Karlsruhe) nach Angaben des Herstellers gegen 1 x PBS dialysiert. Die Proteine wurden weiterhin bei Bedarf mit den Konzentratoren Ultrafree-0.5 (centrifugal filter device Biomax-5 und Biomax-10; Millipore, Schwalbach) nach Angaben des Herstellers einkonzentriert. 2.4 Proteinchemische Methoden 2.4.1 SDS-PAGE (Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese) Die analytische Elektrophorese wurde nach Laemmli et al. (1970) mit dem Elektrophorese„Minigel Twin“-System (Biometra, Göttingen) durchgeführt. Für die Auftrennung und Darstellung der Proteine wurde die Konzentration der Trenngele nach Größe der zu trennenden Proteine zwischen 10 % und 15 % gewählt. Zur Herstellung eines SDS-Trenngels wurden 5,5 ml des Trenngelansatzes (Trenngelpuffer (91 g Tris, 0,4 % SDS in 0,5 l H2O, pH 8,8) der Gelkonzentration von 10 %-15 % entsprechend, 25 µl 10 % APS (Ammoniumpersulfat; Merck, - 36 - Material und Methoden Dresden), 5 µl TEMED (Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) mit der entsprechenden Konzentration von 30 % Acrylamid (Acrylamide/Bis Solution (37,5:1); Invitrogen, Karlsruhe) zwischen präparierte Gelplatten (10,5 cm x 9,8 cm, 1 mm Spacer) gefüllt und für die Polymerisation mit H2O-gesättigtem Isobutanol überschichtet. Nach 30 min wurde das Isobutanol verworfen und das Trenngel mit H2O gewaschen. Danach wurde das Trenngel mit 2 ml Sammelgellösung (325 µl 30 % Acrylamid (Acrylamide/Bis Solution (37,5:1); Invitrogen, Karlsruhe), 625 µl Sammelgelpuffer (6,05 g Tris, 0,4 % SDS in 0,1 l H2O, pH 6,8), 1,5 ml H2O, 12,5 µl 10 % APS (Ammoniumpersulfat; Merck, Dresden) und 2,5 µl TEMED (Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) überschichtet und ein Silikon-Kamm luftblasenfrei eingesetzt, so dass das Gel auspolymerisieren konnte. Die Proteinlösungen wurden im Verhältnis 1:5 mit SDS-Probenpuffer (nach Sambrook et al. 1989; 5x, 125 mM Trisbase, 5 % SDS, 50 % Glycerin, 1 mg Bromphenolblau, 40 mg Orange G ad 100 ml ddH2O, pH 6,8) aufgenommen. Die analytische Elektrophorese wurde in vertikalen Elektrophorese-Gelkammern (Minigel Twin; Biometra, Göttingen) bei 10 mA (Consort E844 und E865, Consort, Merck, Dresden) pro Gel zum Einlaufen und 15-20 mA für den Trennvorgang durchgeführt. Es wurden weiterhin 1 x SDS-Elektrophoresepuffer (nach Sambrook et al. 1989; 3,04 g Trisbase, 1,0 g SDS, 14,4 g Glycerin ad 1l ddH2O) und als Molekulargewichts-Marker 5 µl „Benchmark“-Standard (Invitrogen, Karlsruhe) verwendet. 2.4.2 Proteinnachweis durch Silberfärbung Nach einer SDS-PAGE-Auftrennung (2.4.1) wurden Proteine mit der Silberfärbemethode nach Heukeshoven und Dernick (1986) nachgewiesen. Das SDS-Polyacrylamidgel (9 x 6 cm) wurde in Lösung 1 (30 % Ethanol absolut, 10 % Eisessig) für 10 min fixiert, anschließend in Lösung 2 (30 % Ethanol absolut, 0,5 M Natriumacetat, 0,5 % Glutardialdehyd, 0,2 % Natriumthiosulfat) für 10 min inkubiert und mindestens 3 bis 5 mal ausgiebig mit ddH2O gewaschen. Nach der Inkubation in Lösung 4 (0,1 % AgNO3, 0,02 % Formaldehyd) für 10 min wurde das Gel in Lösung 5 (2,5 % Natriumcarbonat, 0,01 % Formaldehyd) entwickelt. Mit Lösung 6 (0,1 M EDTA) wurde die Reaktion nach Erreichen der gewünschten Farbintensität des Gels gestoppt. Anschließend wurde das Silbergel in 30 ml Trocknungslösung (5 % Glycerin,10 % Ethanol) überführt und mit dem „DryEase Mini-Gel Drying“ System von Invitrogen (Karlsruhe) getrocknet. - 37 - Material und Methoden 2.4.3 Proteintransfer Die im SDS-PAGE (2.4.1) aufgetrennten Proteine wurden mit Hilfe einer elektrophoretischen Transfereinheit nach dem „Semi-Dry-Blotting“-Verfahren (Kyhse-Andersen, 1984) auf eine Nitrozellulose-Membran (6,5 x 9 cm, ProtranBA, Schleicher & Schüll, Merck, Dresden) unter Verwendung einer „Semi-Dry-Blotkammer mit Graphitelektroden (Keutz, Reiskirchen) transferiert. Das Gel wurde nach der Elektrophorese für 10-15 min in Kathodenlösung (40 mM Aminohexansäure, 20 % Methanol, pH 7,6) inkubiert. Währenddessen wurden 6 Lagen Filterpapier (6,5 x 9 cm, Whatman, Merck, Dresden) in Anodenlösung 1 (300 mM Tris, 20 % Methanol, pH 10,4) inkubiert und auf die Anodenplatte der Blot-Kammer gelegt. Nacheinander folgten drei Lagen Filterpapier und eine Nitrozellulosemembran, die vorher in Anodenlösung 2 (25 mM Tris, 20 % Methanol, pH 10,4) inkubiert wurden. Schließlich wurden das Gel und neun in Kathodenlösung äquilibrierte Filter luftblasenfrei aufgelegt. Der Transfer wurde für 1-1,5 h bei 0,8 mA/cm2 (Consort E844 und E865, Consort, Merck, Dresden) durchgeführt. 2.4.4 Immunodetektion membrantransferierter Proteine (Westernblot) Die spezifische Detektion der nach dem „Semi-Dry-Blotting“-Verfahren (2.4.3) transferierten Proteine erfolgte durch Peroxidase-Reaktionen an den Antikörpern. Unspezifische Bindungsstellen auf der Nitrozellulose-Membran wurden durch 30 min Inkubation in 5 % Milchpulver in 1 x PBS bzw. in 2 % BSA in 1 x TBST gesättigt. Die Nitrozellulosemembran wurde nach der Blockierungsreaktion mit den entsprechenden primären Antikörper (Tabelle 8) in 2,5 % Milchpulver in 1 x PBS bzw. in 1 % BSA in 1 x TBS über Nacht bei 4 °C oder für 1-3 h bei RT inkubiert. Anschließend wurde die Membran dreimal mit 1 x PBS in einem Volumen von 30 ml gewaschen und für 3 h bei RT mit dem entsprechenden sekundären Antikörper (Tabelle 8) in 2,5 % Milchpulver in 1 x PBS bzw. in 1 % BSA in 1 x TBS inkubiert. Die immunogenen Proteinbanden wurden nach erneutem dreimaligen Waschen in 1 x PBS mit Hilfe des Peroxidase-Nachweises durch Schwenken in Entwicklerlösung (0,1 % H2O2, 0,3 % 4-Chloro-1Naphtol in 10 % Methanol in 1 x PBS bzw. 1 x TBS) detektiert. Die Antikörper wurden von den Firmen Alexis (Grünberg), Biomol (Hamburg), DAKO (Glostrup, Dänemark), Roche (Mannheim), Santa Cruz (Heidelberg), Sigma (Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) und Upstate (Biomol, Hamburg) bezogen. - 38 - Material und Methoden Tabelle 8: Primäre und sekundäre Antikörper Antikörper Herkunft Konzentration Quelle Anti-EGR-3, polyklonal Kaninchen 1:400 Santa Cruz, Anti-EGR-4, polyklonal Kaninchen 1:400 Skerka et al., 1997 Anti-Protein C, monoklonal Maus 1:400 Roche Anti-Protein C-Peroxidase, monoklonal Maus 1:1000 Roche Anti-NFATc1 (NFATc), monoklonal Maus 1:1000 Alexis Anti-NFAT1 (NFATp), polyklonal Kaninchen 1:1000 Upstate Anti-NFκB p50, polyklonal Ziege 1:1000 Santa Cruz Anti-NFκB p65, polyklonal Kaninchen 1:1000 Biomol Anti-Glutathion-S-Transferase, polyklonal Kaninchen 1:1000 Sigma Anti-polyhistidin, monoklonal Maus 1:1000 Sigma Anti-Kaninchen, HRP-konjugiert Schwein 1:400 Dako Anti-Ziege, HRP-konjugiert Kaninchen 1:400 Dako Anti-Maus, HRP-konjugiert Kaninchen 1:400 Dako 2.4.5 in vitro-Proteinbindungsassay Für die in vitro-Proteinbindungsassays wurden Histidin-Fusionsproteine der EGR-Proteine und die GST-Fusionsproteine der NFAT bzw. NFκB-Proteine an einer entsprechenden Matrix immobilisiert und mit Zellextrakten inkubiert, die mögliche Interaktionspartner enthielten. In dem „GST“-Proteinbindungsassay (GST-pull down-Assay)wurden 100 µl NFAT bzw. NFκB enthaltender Zellextrakt mit 400 µl Glutathionagarose für minimal 1 h (maximal über Nacht) bei 4 °C inkubiert. Nach Zentrifugation (5 min bei 4 °C und 500 x g) und ausgiebigem Waschen mit eiskaltem 1 x PBS wurde die Säule mit 100-200 µl EGR-Protein enthaltender Sf9-Zellextrakt (1:4 verdünnt in Protein C ohne NaCl) oder 500 µl Jurkat-T-Zellextrakt (1:4 verdünnt in Protein C ohne NaCl) bei 4 °C für 1-3 h inkubiert. Der Überstand wurde entfernt und nochmals ausgiebig gewaschen. Die Elution erfolgte mit 150 µl Elutionspuffer (10 mM Gluthation in 50 mM Tris; kurz vor Verwendung hinzugefügt: 0,5 mM DTT und 0,1 mM PMSF). Für den „Nickel“-Proteinbindungsversuch (Nickel-pull down-Assay) wurde Zellextrakt von Sf9 Zellen, die mit rekombinanten EGR-Viren infiziert wurden, mit 400 µl Ni2+-NTA Agarose (Quiagen, Hilden) für 1-2 h bei 4 °C inkubiert. Das Säulenmaterial wurde durch Zentrifugation - 39 - Material und Methoden (5 min bei 4 °C und 500 x g) präzipitiert und viermal mit eiskaltem 1 x „Binding“-Puffer (5 mM Imidazol in 1 x PBS, pH 7,5) gewaschen. Daraufhin wurden 100 µl NFAT oder NFκB enthaltender Zellextrakt für 1-2 h bei 4 °C hinzugefügt, ausgiebig mit 1 x „Binding“-Puffer gewaschen und mit 150 µl eiskaltem Elutionspuffer (4 M Imidazol in 1 x PBS, pH 7,5) eluiert. 2.4.6 Peptidspotanalyse Für die Untersuchung der Interaktionsdomänen der verschiedenen Proteine auf Peptidebene wurden Peptide mit einer Länge von 15 bzw. 13 Aminosäuren und einer Überlappung von 13 bzw. 10 Aminosäuren synthetisiert und auf Whatman 50 Zellulosemembranen gekoppelt (Jerini Peptide Technologies, Berlin). Die Peptide entsprachen der mittlere Region von EGR-4 (aa159331 und aa 159-333), die Zinkfingerregionen von EGR-1 (aa 335-426), EGR-2 (aa 337-426), EGR-3 (aa 272-361), EGR-4 (aa 376-465) und Sp1 (aa 534-625), ebenso wie die Regionen aa 406-462 von NFATc, aa 388-444 von NFATp, aa 41-80 von p50, aa 37-77 von p52 und aa 17-57 von p65. Jede Membran wurde mit 5 % Albumin (Sigma-Aldrich Vertriebs-GmbH, Dreisenhofen) in Peptidpuffer I (50 mM TRIS, pH 8,0, 137 mM NaCl, 2,7 mM KCl) über Nacht bei Raumtemperatur geblockt, dreimal mit Peptidpuffer II (50 mM TRIS, pH 8,0, 137 mM NaCl, 2,7 mM KCl, 0,05 % Tween) gewaschen und mit entsprechend aufgereinigten EGR-, NFAT- oder NFκB-Proteinen für 3 h bei 4 °C inkubiert. Nach dreimaligem Waschen mit Peptidpuffer II wurden die Membranen zuerst mit spezifischen ersten Antikörper (Tabelle 8, 1:2000 in 2,5 % Albumin in Peptidpuffer I), danach mit dem HRP-gekoppelten sekundären Antikörper (Tabelle.8, 1:2000 in 2,5 % Albumin in Peptidpuffer I) über Nacht bei 4 °C inkubiert. Die Membranen wurden dreimal sowohl mit Peptidpuffer I als auch mit Peptidpuffer II gewaschen. Die Entwicklung wurde mit dem ECLTM (Amersham Pharmacia Biotech, München) durchgeführt, auf einem X-Ray Film (Amersham Pharmacia Biotech, München) exponiert und im CURIX 60 (AGFA-Gevaert, Mortsel, Belgien) entwickelt. - 40 - Material und Methoden 2.4.7 Plasmonresonanz-Studie Eine weitere Methode für die Untersuchung von Protein-Protein-Interaktionen ist die Verwendung der Plasmonresonanz im Biacore-System (Biacore AB, Upsala, Schweden). Hierbei wird der eine Reaktand (Ligand) auf der Oberfläche eines Sensorchips immobilisiert. Der zweite Reaktand (Analyt) strömt über die Oberfläche des Chips und ruft durch Massenänderungen an der Oberfläche des Sensors eine Änderung des Brechungsindexes hervor, woraufhin eine Antwortfunktion der Plasmonresonanz detektierbar ist. Rekombinante EGR-, und NFAT-Proteine wurde exprimiert (2.3.1.3, 2.3.4), aufgereinigt (2.3.3, 2.3.5) und gegen 10 mM Acetatpuffer (pH 5,5) dialysiert. Die Proteine wurden mit Hilfe der Amin-Kopplungs-Einheit (Amine Coupling Kit; Biacore AB, Upsala, Schweden) bei einer Flußrate von 5 µl/min immobilisiert. Nach der Aktivierung der Oberfläche des CM5-Sensorchips mit 35 µl N-Hydroxysuccinimid (NHS), 11,5 mg/ml) und 35 µl 1-Ethyl-3-(3- dimethylaminopropyl) carbiimid hydrochlorid (EDC; 75 mg/ml), wurde soviel rekombinantes Protein (1 µg/ml) verwendet, dass eine Resonanzantwort von 3000-5000 Resonanzeinheiten (resonance units, RU) erreicht wurde. Die Oberfläche des Sensorchips wurde anschließend mit 35 µl 1 M Ethanolaminhydrochlorid-NaOH (pH 8,5) deaktiviert. Für jeden Versuch wurde eine Kontrollflußzelle mit Acetatpuffer immobilisiert. Die Analytenproteine wurde exprimiert (2.3.1.3, 2.3.4), aufgereinigt (2.3.3, 2.3.5), gegen 0,5 x PBS (pH 7,5) dialysiert und gegebenenfalls einkonzentriert (2.3.2 und 2.3.5). Jedes Analyt wurde gekühlt bei 4 °C separat in die Flußzellen bei einer Flußrate von 5 µl/min bei 25 °C injiziert. Die einzelnen Versuche wurden mit dem Biacore-„wizard“-Programm durchgeführt und mit dem Biacore- Evaluationsprogramm analysiert. Für die Interaktionsversuche mit EGR-4 und NFATc bzw. p65, bei denen die Interaktion gehemmt werden sollte, wurden inhibitorische Peptide (Jerini Peptide Technologies, Berlin) eingesetzt. Die inhibitorischen Peptide entsprechen den EGR-4Aminosäureregionen aa 451-465 (NFκB-Peptid: EKKRHSKVHLKQKAR) und aa 306-320 (NFAT-Peptid: TQPQLSPLGLRSAAA). - 41 - Ergebnisse 3 Ergebnisse 3.1 Expression rekombinanter und nativer Proteine Für die Untersuchung der EGR-Interaktionen mit NFAT- und NFκB-Proteinen wurden sowohl rekombinante als auch native EGR-, NFAT- und NFκB-Proteine exprimiert. Rekombinante EGR-Proteine wurden mit Hilfe des Baculovirus-Systems in Sf9-Insektenzellen, die rekombinanten Proteine der NFAT-Familie (NFATc und NFATp) und der NFκB-Familie (p50 und p65) in E. coli exprimiert. Die nativen Proteine EGR-3, EGR-4, NFATc, NFATp, p50 und p65 wurden aus mitogen stimulierten Jurkat-T-Zellen erhalten. Die jeweiligen Proteine wurden durch SDS-PAGE aufgetrennt, auf Nitrozellulosemembranen transferiert und mit spezifischen Antikörpern detektiert. 3.1.1 Expression rekombinanter und nativer EGR-Proteine Die EGR-Proteine EGR-3 und EGR-4 wurden rekombinant in Sf9-Insektenzellen exprimiert, während native EGR-Proteine aus stimulierten Jurkat-T-Zellen isoliert wurden. Das rekombinante und native EGR-4 zeigte nach SDS-PAGE und einem Transfer im Westernblot eine Proteinbande mit einer Mobilität von 69 kDa (Abb. 4 A, Spuren 1-2). Die bei der Zelllyse erforderliche Salzkonzentration von 420 mM wirkt sich auf die Mobilität der EGR-Proteine aus (Abb. 4 B). EGR-4 zeigte bei einer Konzentration von 420 mM Natriumchlorid Banden unterschiedlicher Mobilität. Um die Banden von EGR-4 zu charakterisieren, wurde das Laufverhalten der EGR-4-Banden bei Salzkonzentrationen von 420 mM bis 60 mM Natriumchlorid untersucht. Bei gleichen Proteinmengen nahm die Intensität der 69 kDa-Bande von EGR-4 während der Erniedrigung des Salzgehaltes zu (Abb. 4 B). Die EGR-Banden, die höhere Mobilitäten besitzen, konnten bei geringeren Salzkonzentrationen nicht mehr nachgewiesen werden. Bei einer Salzkonzentration von 220 mM trat eine weitere EGR-4-Bande von 140 kDa auf, deren Intensität bei niedrigeren Salzkonzentrationen zunahm (Abb. 4 B, Spuren 3-5). Die Veränderung des Laufverhaltens der EGR-4-Proteine während der Abnahme der Salzkonzentration war reversibel. Bei einer Erhöhung der Salzkonzentration von 60 mM auf 420 mM Natriumchlorid zeigte die EGR-4-Bande von 69 kDa wieder eine geringere Intensität und es traten erneut EGR-4-Banden höherer Mobilität auf (Abb. 4 B, Spuren 5-6). Die bei den unterschiedlichen Salzkonzentrationen auftretenden EGR-4-Banden konnten aufgereinigt werden (Abb. 8). Eine Veränderung im Laufverhalten wurde auch bei nativen EGR-4-Proteinen aus - 42 - Ergebnisse stimulierten Jurkatzellen beobachtet (Abb. 4 C). Im Zellextrakt mit einer Salzkonzentration von 420 mM Natriumchlorid zeigte natives EGR-4 niedermolekulare Banden, aber keine 69 kDaBande. Bei einer Salzkonzentration von 120 mM trat die 69 kDa-Bande von EGR-4 wieder auf (Abb. 4 C, Spur 2). Durch die Erhöhung der Salzkonzentration von 120 mM auf 420 mM Natriumchlorid konnte der Verlust der 69 kDa-Bande und das Wiederauftreten der EGR-4Bande höherer Mobilität gezeigt werden. Das Laufverhalten von EGR-4 verändert sich in Abhängigkeit zur Salzkonzentration und stellt Konformationsänderungen von EGR-4 dar. Bei hohen Salzkonzentrationen treten neben der 69 kDa-Bande verschiedene Konformationen im SDS-PAGE und Westernblot auf, die bei einer Erniedrigung der Salzkonzentrationen nicht mehr nachgewiesen werden konnten. Die Konformationsänderungen bei sinkenden Salzkonzentrationen waren reversibel und konnten durch eine Erhöhung der Salzkonzentration rückgängig gemacht werden. EGR-3 wurde rekombinant in Sf9-Insektenzellen und nativ in stimulierten Jurkat-T-Zellen exprimiert. Die EGR-3-Proteine wurden durch SDS-PAGE getrennt, auf eine Nitrozellulosemembran transferiert und im Westernblot detektiert. Rekombinantes EGR-3 zeigte eine Bande mit einer Mobilität von 59 kDa (Abb. 4 D, Spur 1). Natives EGR-3 zeigte neben einer 59 kDa-Bande noch eine zusätzliche 45 kDa-Bande (Abb. 4 D, Spur 2). Abb. 4: Nachweis rekombinanter und nativer EGR-Proteine (A) Expression von EGR-4-Proteinen. Das Zinkfingerprotein EGR-4 wurde rekombinant in Sf9-Insektenzellen (Spur 1) und in stimulierten Jurkat-T-Zellen exprimiert (Spur 2). (B) Die Salzkonzentration beeinflusst die Mobilität von EGR-4. Die Intensität der 69 kDa-Bande von EGR-4 nimmt bei fallendem Salzgehalt zu. Bei hohen Salzkonzentrationen tritt neben der 69 kDa-Bande eine weitere Bande höherer Mobilität auf, die bei fallenden Salzkonzentrationen nicht mehr nachgewiesen werden kann. Derselbe Effekt ist auch bei nativen EGR-4-Proteinen aus stimulierten Jurkat-T-Zellen zu beobachten (C). (D) Expression von EGR-3-Proteinen. Rekombinantes EGR-3 wurde in Sf9-Insektenzellen (Spur 1), natives EGR-3 in stimulierten Jurkat-T-Zellen (Spur 2) exprimiert. Sämtliche Proteine wurde durch SDS-PAGE getrennt, und nach dem Transfer auf eine Nitrozellulosemembran im Westernblot mit spezifischen Antikörpern detektiert. rek: rekombinant. - 43 - Ergebnisse 3.1.2 Expression rekombinanter und nativer NFAT- und NFκB-Proteine Die rekombinanten NFAT- und NFκB-Proteine wurden als Glutathion-S-Transferase (GST)Fusionsproteine in E. coli exprimiert und mit der „GST“-Affinitätschromatographie aufgereinigt. Native NFAT- und NFκB-Proteine wurden aus stimulierten Jurkat-T-Zellextrakten isoliert. Die verschiedenen Proteine wurden durch SDS-PAGE getrennt und nach einem Proteintransfer im Westernblot nachgewiesen. Rekombinantes und natives NFATc zeigte im Westernblot Banden mit einer Mobilität von 95 kDa (Abb. 5 A, Spuren 1-2). Rekombinantes NFATp wurde mit einer Bande von 89 kDa nachgewiesen und entsprach damit der Bande von nativem NFATp (Abb. 5 B, Spuren 3-4). Sowohl rekombinante als auch native Proteine der NFκB-Familie p50 und p65 zeigten jeweils Banden mit Mobilitäten von 100 kDa (Abb. 5 B, Spuren 1-2) und 120 kDa (Abb. 5 B, Spuren 3-4). Abb. 5: Nachweis rekombinanter und nativer NFAT- und NFκB-Proteine (A) Expression von rekombinantem und nativem NFATc und NFATp. Rekombinantes NFATc (Spur 1) und NFATp (Spur 3) wurden als „GST“-Fusionsproteine in E. coli exprimiert und aufgereinigt. Sowohl natives NFATc (Spur 2) als auch natives NFATp (Spur 4) wurde im Zellextrakt stimulierter Jurkat-T-Zellen detektiert. (B) Expression der rekombinanten und nativen NFκB-Proteine p50 und p65. Die NFκB-Proteine p50 (Spur 1) und p65 (Spur 3) wurden als „GST“-Fusionsproteine rekombinant in E. coli exprimiert und aufgereinigt. Native NFκB-Proteine wurden in stimulierten Jurkat-T-Zellen exprimiert. Die Proteine wurden durch SDS-PAGE getrennt, auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. rek: rekombinant. 3.2 Interaktion von EGR- und NFAT-Proteinen Die EGR-Proteine interagieren mit verschiedenen Transkriptionsfaktoren, zu denen auch die Proteine der NFAT-Familie gehören. Die EGR/NFAT-Interaktion wurde in Bindungsversuchen (GST-pull down-Assays) untersucht, bei denen ein Interaktionspartner auf einer Glutathionmatrix immobilisiert und anschließend mit dem anderen Interaktionspartner inkubiert wird. Zunächst wurden NFATc und NFATp immobilisiert und mit rekombinanten EGR- 44 - Ergebnisse Proteinen inkubiert. Zusätzlich wurde das Bindungsverhalten von nativem EGR-3 und EGR-4 an die NFAT-Proteine untersucht. Im Weiteren wurde überprüft, ob die Interaktion auch in umgekehrter Weise stattfindet. Deshalb wurden EGR-3 und EGR-4 immobilisiert und die Bindung an rekombinante und native NFAT-Proteine untersucht. 3.2.1 Interaktion von EGR mit immobilisierten NFAT-Proteinen Für den Nachweis der EGR/NFAT-Interaktion wurden rekombinante EGR-Proteine in Sf-9Insektenzellen und NFAT-Proteine in E. coli exprimiert. Die nativen EGR- und NFAT-Proteine wurden aus Zellextrakt stimulierter Jurkat-T-Zellen isoliert. In einem „GST-pull down"-Assay wurden NFATc und NFATp immobilisiert und mit EGR-Proteinen inkubiert. Die Proteine in den verschiedenen Fraktionen wurden durch SDS-PAGE getrennt und im Westernblot mit spezifischen Antikörpern nachgewiesen. Die Abbildung 6 A zeigt die Immobilisierung von NFATc und NFATp auf einer Glutathionmatrix. NFATc enthaltender Zellextrakt (ze; Abb. 6 A, Spur 1) wurde nach Inkubation mit der Glutathionagarose weder in der Fraktion „Überstand nach Bindung“ (ünb; Abb. 6 A, Spur 2) noch in den Waschfraktionen nachgewiesen (w; Abb. 6 A, Spuren 3-4). NFATc wurde auf der Glutathionmatrix immobilisiert, da es in den Elutionsfraktionen nachgewiesen werden konnte (Abb. 6 A, Spuren 5-7). NFATp bindet vollständig an die Glutathionmatrix und konnte in den Elutionsfraktionen (Abb. 6 B, Spuren 6-8), aber nicht in der Fraktion „Überstand nach Bindung“ (ünb; Abb. 6 B, Spur 2) und in den Waschfraktionen (w; Abb. 6 B, Spur 3-5) nachgewiesen werden. In Abbildung 6 C ist die Interaktion von nativem EGR-4 mit immobilisiertem NFATc dargestellt. EGR-4 bindet vollständig an NFATc, da es zusammen mit NFATc eluiert werden konnte (Abb. 6 C, Spuren 6-7). EGR-4 konnte nicht in der Fraktion „Überstand nach Bindung“ und in den Waschfraktionen nachgewiesen werden (Abb. 6 C, Spuren 2-4). Natives EGR-4 bindet spezifisch an NFATc, da EGR-4 nicht an der Glutathionagarose gebunden werden konnte (Abb. 7 C). - 45 - Ergebnisse Abb. 6: Immobilisierung von NFAT-Proteinen und EGR-4-Proteinbindungsassay (A) Immobilisierung von NFATc auf der Glutathionmatrix. In E. coli rekombinant exprimiertes NFATc wurde mit der Glutathionagarose inkubiert (Spuren 1-2), gewaschen (Spuren 3-4) und mit Glutathion eluiert (Spuren 5-7). (B) Immobilisierung von rekombinantem NFATp aus E. coli-Zellextrakten auf der Glutathionmatrix (Spuren 1-2). Die immobilisierten Proteine wurden gewaschen (Spuren 3-5) und mit Glutathion eluiert (Spuren 6-8). (C) Interaktion von nativem EGR-4 mit NFATc. EGR-4 enthaltender Zellextrakt stimulierter Jurkat-T-Zellen wurde mit immobilisiertem NFATc inkubiert (Spuren 1-2) und gewaschen (Spuren 3-5). EGR-4 konnte zusammen mit NFATc eluiert werden (Spuren 6-7). Kontrolle siehe Abb. 7. Die Proteine wurden nach SDS-PAGE auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. el: Eluat, ünb: Überstand nach Bindung, w: Waschfraktion, ze: Zellextrakt. Für den Nachweis der Interaktion von EGR- und NFAT-Proteinen wurde rekombinantes NFATc bzw. NFATp auf einer Glutathionmatrix immobilisiert und mit rekombinantem EGR-3 bzw. EGR-4 inkubiert. Zusätzlich wurde das Interaktionsverhalten von nativem EGR-3 und EGR-4 an NFAT-Proteinen untersucht. Da eine Interaktion durch die gemeinsame Elution der Interaktionspartner nachgewiesen wird, werden in den folgenden Abbildungen die Interaktionen durch Zellextrakt- und Elutionsfraktionen dargestellt. In Abbildung 7 A sind die Interaktionen von rekombinantem und nativem EGR-3 bzw. EGR-4 mit immobilisiertem NFATc dargestellt. Sowohl rekombinantes als auch natives EGR-3 bindet an NFATc und wurde zusammen mit NFATc eluiert (Abb. 7 A, Spuren 1-4). EGR-4 bindet ebenfalls an NFATc, da rekombinantes und natives EGR-4 in den Elutionsfraktionen nachgewiesen wurde (Abb. 7 A, Spuren 5-8). Der Nachweis für die Interaktion von EGR-Proteinen an NFATp erfolgte nach der Immobilisierung von NFATp auf einer Glutathionmatrix. Sowohl rekombinantes als auch natives EGR-3 wurde in den Elutionsfraktionen detektiert, so dass die Interaktion von EGR-3 mit NFATp nachgewiesen werden konnte (Abb. 7 B, Spuren 1-4). EGR-4 bindet in rekombinanter und nativer Form an NFATp, da es zusammen mit NFATp eluiert werden konnte (Abb. 7 B, Spuren 5-8). EGR-3 und EGR-4 binden sowohl an NFATc als auch an NFATp, da die EGR-Proteine selbst nicht an die Glutathionagarose binden (Abb. 7 C). Unspezifische Interaktionen von EGR- mit den NFATProteinen können somit ausgeschlossen werden. - 46 - Ergebnisse Abb. 7: Bindung von EGR-3 und EGR-4 an immobilisierte NFAT-Proteine EGR-3 und EGR-4 wurden rekombinant in Sf9-Insektenzellen und nativ in stimulierten Jurkat-T-Zellen exprimiert. Die EGR-Proteine enthaltenden Zellextrakte wurden mit immobilisierten NFAT-Proteinen inkubiert, gewaschen und mit Glutathion eluiert. Die Proteine wurden durch SDS-PAGE aufgetrennt, auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung von EGR-Proteinen an immobilisiertes NFATc. Rekombinantes EGR-3 (Spur 1) wurde gemeinsam mit NFATc eluiert (Spur 2). Natives EGR-3 (Spur 3) aus Jurkat-T-Zellextrakt konnte an NFATc gebunden werden (Spur 4). Die Interaktion von immobilisierten NFATc mit rekombinantem EGR-4 ist in den Spuren 5-6, mit nativem EGR-4 in den Spuren 7-8 dargestellt. (B) Bindung von EGR-3 und EGR-4 an immobilisiertes NFATp. Rekombinantes EGR-3 (Spuren 1-2) und rekombinantes EGR-4 (Spuren 5-6) binden an NFATp, da sie in den Elutionsfraktionen nachgewiesen wurden. Sowohl natives EGR-3 (Spuren 3-4) als auch natives EGR-4 (Spuren 7-8) bindet an NFATp. (C) Inkubation der EGR-Proteine mit Glutathionagarose. Eine unspezifische Bindung von rekombinantem oder nativem EGR-3 (Spuren 1-4) bzw. EGR-4 (Spuren 5-8) mit den NFAT-Proteinen konnte ausgeschlossen werden, da keines der EGR-Proteine an der Glutathionagarose gebunden wurde. el: Eluat,rek: rekombinant, ze: Zellextrakt. Die Proteinassays mit immobilisierten NFAT-Proteinen zeigten direkte Interaktionen zwischen EGR- und NFAT-Proteinen. Die Interaktion von rekombinantem und nativem EGR-3 bzw. EGR-4 an NFATc und NFATp führt zur Komplexbildung. 3.2.2 Interaktion von NFAT mit immobilisierten EGR-Proteinen Nachdem die EGR-Interaktionen mit immobilisierten NFAT-Proteinen nachgewiesen wurden, stellte sich die Frage, ob die Interaktionen auch in umgekehrter Weise stattfinden. EGR-3 und EGR-4 wurden deshalb rekombinant als Histidin-Fusionsproteine exprimiert und auf einer Nickelmatrix immobilisiert (Abb. 8). Die Proteine wurden nach der Auftrennung im SDS-PAGE und einem Transfer mit spezifischen Antikörpern im Westernblot detektiert. EGR-3 aus dem Zellextrakt bindet an die Nickel-Agarose und kann eluiert werden (Abb. 8 A). EGR-3 bindet nicht vollständig, da es in der Fraktion „Überstand nach Bindung“ und in einer Waschfraktion nachgewiesen werden konnte. EGR-4 bindet ebenfalls an die Nickelagarose, da es in den Elutionsfraktionen detektiert wurde (Abb. 8 B). Die Bindung von EGR-4 an die Matrix ist nicht - 47 - Ergebnisse vollständig. Dies zeigte sich durch den Nachweis von EGR-4 in der Fraktion „Überstand nach Bindung“ (Abb. 8 B, Spur 2). Mit immobilisierten EGR-Proteinen wurden weiterhin die Interaktionen von EGR- und NFAT-Proteinen untersucht. Abb. 8: Immobilisierung von EGR-Proteinen (A) Immobilisierung von EGR-3 auf einer Nickelmatrix. EGR-3 wurde in Sf9-Insektenzellen rekombinant als Histidin-Fusionsprotein exprimiert und mit Nickelagarose inkubiert (Spuren 1-2), gewaschen (Spuren 3-5) und mit Imidazol eluiert (Spuren 6-7). (B) Immobilisierung von rekombinantem EGR-4 aus Sf-9-Zellextrakten (Spuren 1-2) an einer Nickelmatrix. Die immobilisierten EGR-4-Proteine wurden gewaschen (Spuren 3-5) und mit Imidazol eluiert (Spuren 6-7). Die Proteine wurden durch SDS-PAGE getrennt und nach dem Transfer auf eine Nitrozellulosemembran mit spezifischen Antikörpern detektiert. el: Eluat, ünb: Überstand nach Bindung, w: Waschfraktion, ze: Zellextrakt. Für den Nachweis der EGR/NFAT-Interaktion wurden EGR-3 und EGR-4 immobilisiert und mit rekombinantem NFATc und NFATp inkubiert. Zusätzlich wurden immobilisierte EGR-Proteine mit Zellextrakt stimulierter Jurkat-T-Zellen inkubiert, um das Bindungsverhalten nativer NFATProteine zu untersuchen. Die Proteine sämtlicher Fraktionen wurden nach SDS-PAGE im Westernblot mit spezifischen Antikörpern detektiert. Die Abbildung 9 A zeigt die Bindungen von NFATc und NFATp an immobilisiertes EGR-3. Rekombinantes und natives NFATc binden an EGR-3, da sie sowohl in der Zellextrakt- als auch in der Elutionsfraktion nachgewiesen wurden (Abb. 9 A, Spuren 1-4). NFATp bindet an EGR-3 und konnte in rekombinanter und nativer Form mit EGR-3 eluiert werden (Abb. 9 A, Spuren 5-8). Die Interaktionen von rekombinantem und nativem NFATc mit EGR-4 sind in Abbildung 9 B dargestellt. Sowohl rekombinantes als auch natives NFATc bindet an EGR-4 (Abb. 9 B, Spuren 1-4). Rekombinantes NFATp wurde in der Elutionsfraktion nachgewiesen und zeigt somit eine Bindung an EGR-4 (Abb. 9 B, Spuren 5-6). Natives NFATp bindet ebenfalls an EGR-4 (Abb. 9 B, Spuren 7-8). Die Interaktionen zwischen den EGR- und NFAT-Proteinen sind spezifisch, da rekombinante und native NFAT-Proteine nach Inkubation mit der Nickelmatrix nicht in den Elutionsfraktionen nachzuweisen sind (Abb. 9 C). - 48 - Ergebnisse Abb. 9: Bindung von NFATc und NFATp an immobilisierte EGR-Proteine NFATc und NFATp wurden rekombinant in E. coli und nativ in stimulierten Jurkat-T-Zellen exprimiert. NFATProteine enthaltende Zellextrakte wurden mit immobilisierten EGR-Proteinen inkubiert, gewaschen und mit Imidazol eluiert. Die Proteine wurden durch SDS-PAGE getrennt, auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung von NFAT-Proteinen an immobilisiertes EGR-3. Rekombinantes NFATc (Spur 1) wurde gemeinsam mit EGR-3 eluiert (Spur 2). Natives NFATc (Spur 3) aus Jurkat-T-Zellextrakt konnte an EGR-3 gebunden werden (Spur 4). Die Bindung von immobilisiertem EGR-3 an rekombinantes NFATp ist in den Spuren 5-6, an natives NFATp in den Spuren 7-8 dargestellt. (B) Bindung von NFATc und NFATp an immobilisiertes EGR-4. Rekombinantes NFATc (Spuren 1-2) und rekombinantes NFATp (Spuren 5-6) interagierten mit EGR-4. Sowohl natives NFATc (Spuren 3-4) als auch natives NFATp (Spuren 7-8) wurde an EGR-4 gebunden. (C) Inkubation der NFAT-Proteine mit Nickelagarose. Eine unspezifische Bindung von rekombinantem oder nativem NFATc (Spuren 1-4) bzw. NFATp (Spuren 5-8) mit den EGR-Proteinen konnte ausgeschlossen werden, da die NFAT-Proteine nicht an die Nickelmatrix binden. el: Eluat,rek: rekombinant, ze: Zellextrakt. Die EGR- und NFAT-Proteine interagieren miteinander und bilden stabile Komplexe. EGR-3 und EGR-4 binden sowohl an NFATc als auch an NFATp, wenn die NFAT-Proteine immobilisiert werden. Die Interaktionen von EGR- und NFAT-Proteinen finden auch in umgekehrter Weise statt, da die Bindung von NFATc und NFATp an immobilisierte EGRProteinen nachgewiesen werden konnte. 3.3 Salzeffekt bei EGR-Interaktionen EGR-Proteine interagieren mit den Proteinen der NFAT-Familie. Da EGR-Proteine Konformationen unterschiedlicher Mobilität besitzen (3.1.1), stellte sich die Frage, ob und in wie weit sich die Salzkonzentration auf das Bindungsverhalten der EGR-Proteine auswirkt. Deshalb wurden die EGR/NFAT-Interaktionen bei verschiedenen Salzkonzentrationen untersucht. Eine Probe EGR-4-Zellextrakt wurde so verdünnt, dass drei Reaktionsansätze mit je einem Salzgehalt von 320 mM, 220 mM und 120 mM Natriumchlorid im Vergleich zur Ursprungsprobe mit 420 mM Natriumchlorid in Proteinbindungsassays mit immobilisiertem NFAT-Proteinen eingesetzt - 49 - Ergebnisse werden konnten. Die vier Proben unterschieden sich im Proteingehalt, so dass Proteinanteile von 75 % (Abb. 10 B), 50 % (Abb. 10 C) oder 25 % (Abb. 10 D) im Vergleich zu 100 % in der Ursprungsprobe (Abb. 10 A) eingesetzt wurden. Die Proteine der einzelnen Fraktionen wurden durch SDS-PAGE getrennt und im Westernblot mit spezifischen Antikörpern detektiert. Die Effekte auf das Bindungsverhalten von EGR-Proteinen durch eine Änderung der Salzkonzentration sind in Abbildung 10 am Beispiel für EGR-4 und NFATc dargestellt. EGR-4 bindet bei einem Salzgehalt von 420 mM Natriumchlorid nicht an NFATc (Abb. 10 A). In den Fraktionen Zellextrakt und „Überstand nach Bindung“ wurden EGR-4-Konformationen unterschiedlicher Mobilität nachgewiesen (Abb. 10 A, Spuren 1-2), von denen keine zusammen mit NFATc eluiert werden konnte (Abb. 10 A, Spuren 6-7). Bei einem Salzgehalt von 320 mM Natriumchlorid bindet EGR-4 ebenfalls nicht an NFATc, da es nur in der Fraktion Zellextrakt und „Überstand nach Bindung“, nicht aber in den Elutionsfraktionen nachgewiesen werden konnte (Abb. 10 B). EGR-4 bindet an NFATc sobald die Salzkonzentration auf 220 mM Natriumchlorid reduziert wurde (Abb. 10 C). Im Zellextrakt zeigte EGR-4 wiederum mehrere Konformationsbanden, von denen nur die 69 kDa-Bande in den Elutionsfraktionen detektiert wurde. EGR-4 bindet bei 120 mM Natriumchlorid vollständig an NFATc, da in der Fraktion „Überstand nach Bindung“ und in den Waschfraktionen kein EGR-4 detektiert werden konnte, dafür aber in den Elutionsfraktionen (Abb. 10 D). EGR-4 besitzt bei einem Salzgehalt von 120 mM nur die Konformation der Mobilität von 69 kDa, da in sämtlichen Fraktionen keine weiteren Konformationbanden nachgewiesen werden konnten. EGR-4 besitzt in Abhängigkeit von der Salzkonzentration mehrere Konformationen, die unterschiedliche Bindungsverhalten zu NFAT-Proteinen zeigen. Die Konformation, die mit einer 69 kDa-Bande im Westernblot nachgewiesen wurde, bindet an NFATc (Abb. 10). EGR-4 bindet NFATp ebenfalls nur mit der 69 kDa-Konformation (nicht dargestellt). Sämtliche EGR-4Konformationen, die mit Banden höherer Mobilität detektiert wurden, konnten nicht an NFATProteine gebunden werden. - 50 - Ergebnisse Abb. 10: Salzeffekt bei EGR-Interaktionen EGR-Proteine besitzen Konformationen unterschiedlicher Mobilität, die eine Abhängigkeit vom Salzgehalt zeigen. Deshalb wurden die EGR/NFAT-Interaktionen bei Salzkonzentrationen von 420 mM, 320 mM, 220 mM und 120 mM untersucht. Aufgrund der Verdünnungsreihe des EGR-4-Zellextraktes, der 420 mM Natriumchlorid enthielt, unterscheiden sich die EGR-4-Proteinanteile in A, B, C und D. In (A) wurde der EGR-4-Proteinanteil im Zellextrakt mit 420 mM Natriumchlorid gleich 100% gesetzt. Dementsprechend ist in (B) bei 320 mM Natriumchlorid der EGR-4-Proteinanteil 75 %, in (C) bei 220 mM Natriumchlorid 50 % und in (D) bei 120 mM Natriumchlorid 25 %. Die Proteine der verschiedenen Fraktionen wurden durch SDS-PAGE getrennt, auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. el: Eluat, ünb: Überstand nach Bindung, w: Waschfraktionen, ze: Zellextrakt. 3.4 Charakterisierung der EGR/NFAT-Interaktionen mit der Plasmonresonanz Die Interaktionen von EGR-3 und EGR-4 mit den NFAT-Proteinen NFATc und NFATp wurden in Proteinbindungsassays nachgewiesen und charakterisiert (3.2-3.3). Für eine nähere Charakterisierung wurden die EGR/NFAT-Interaktionen mit der Plasmonresonanz im BiacoreSystem untersucht. Mit dem System können Proteininteraktionen (Ligand/Analyt) in Echtzeit verfolgt werden. Rekombinantes EGR-4 und EGR-3 wurden aufgereinigt und aufgrund ihres basischen isoelektrischen Punktes für die Immobilisierung in einen sauren Acetatpuffer (pH 5,5) - 51 - Ergebnisse aufgenommen. Die Immobilisierung der EGR-Proteine auf der Gold-Dextran-Oberfläche eines CM-5-Chips erreichte durch 20-40 µl Proteinlösung relative Resonanzwerte von 3000-7000 RU (relative Resonanzeinheiten). Für die Untersuchung der EGR/NFAT-Interaktionen wurden NFATc und NFATp rekombinant in E. coli exprimiert, aufgereinigt, in 0,5 x PBS aufgenommen und in der Flüssigphase zu immobilisierten EGR-Proteinen gegeben. Die Abbildung 11 zeigt einen gesamten Reaktionslauf, bei dem die Bindung von NFATc an immobilisiertem EGR-4 untersucht wurde. Die Kurve der Proteinbindung zeigt eine Assoziations- und eine Dissoziationsphase. In der Assoziationsphase, die mit der Injektion von NFATc beginnt, werden die NFAT-Proteine an die EGR-Proteine gebunden. In der frühen Dissoziationsphase werden nicht oder nur schwach gebundene NFAT-Proteine aus dem System entfernt. Die späte, stabile Dissoziationsphase charakterisiert die Bindung von NFATc an EGR-4. Die anschließende Regeneration mit EDTA- und NaCl-Elutionspuffern führt zur vollständigen Dissoziation des NFATc-Proteins von EGR-4, so dass die Bindungskurve auf die basalen Resonanzwerte zurückgeht. Abb. 11: Untersuchung der Proteininteraktion mit der Plasmonresonanz Die EGR-4/NFATc-Interaktion wurde mit der Plasmonresonanz im Biacore-System (Biacore 3000) untersucht. EGR-4 wurde in Sf9-Insektenzellen exprimiert und auf der Oberfläche eines Chips immobilisiert. Wird NFATc in der Flüssigphase hinzugegeben, ändert sich die von EGR-4 allein hervorgerufene Plasmonresonanz, so dass die Proteininteraktion gemessen werden kann. Der Anstieg der Kurve zeigt die Assoziationsphase, in der gereinigtes NFATc an immobilisiertes EGR-4 bindet. Der Abfall der Kurve zeigt die Dissoziationsphase, bei der in der frühen Phase NFATc aus dem System entfernt wird. Die Interaktionsfähigkeit der Proteine wird durch den Verlauf der stabilen Kurve in der späten Dissoziationsphase gezeigt. Nach der zweifachen Elution in der Regenerationsphase geht die Kurve auf den anfänglichen Basiswert zurück, der von immobilisiertem EGR-4 alleine hervorgerufen wird. Die Werte der Bindungskurven wurden im Evaluationsprogramm von Kontrollwerten abgezogen. Die EGR/NFAT-Interaktionen wurden mit verschiedenen NFAT-Proteinkonzentrationen an immobilisiertem EGR-3 und EGR-4 mit der Plasmonresonanz im Biacore-System untersucht. Rekombinantes NFATc bindet konzentrationsabhängig an EGR-3 (Abb. 12 A). Die - 52 - Ergebnisse konzentrationsabhängigen Bindungen von NFATc an EGR-3 riefen relative Resonanzwerte von 500, 650 und 700 RU hervor. Die Interaktionen von NFATp an EGR-3 zeigten konzentrationsabhängige Bindungen mit relativen Resonanzwerten von 525, 750 und 850 RU (Abb. 12 B). NFATc interagiert mit EGR-4, da die Bindung in der Dissoziationsphase in Abhängigkeit zur Konzentration stabile Resonanzwerte von 575, 700 und 800 RU hervorrief (Abb. 12 C). Die Assoziation von NFATp an EGR-4 war geringer als die von NFATc an EGR-4. Die Interaktion von NFATp mit EGR-4 zeigte konzentrationsabhängig relative Resonanzwerte von 375, 600 und 700 RU (Abb. 12 D). Abb. 12: Bestimmung der NFAT/EGR-Interaktionen mit der Plasmonresonanz Die EGR/NFAT-Interaktionen wurden mit der Plasmonresonanz im Biacore-System untersucht, um die einzelnen Bindungen in Echtzeit verfolgen zu können. Die rekombinant in E. coli exprimierten und aufgereinigten NFATProteine wurden in der Flüssigphase zu immobilisierten EGR-Proteinen hinzugegeben. (A) Die Interaktionen von 10 µg/ml, 5 µg/ml und 2 µg/ml NFATc mit immobilisiertem EGR-3 zeigen eine dosisabhängige Bindung. Die konzentrationsabhängige Interaktion von NFATp (10 µg/ml, 5 µg/ml und 2 µg/ml) mit EGR-3 ist in (B) dargestellt. An immobilisiertes EGR-4 wurden jeweils 15 µg/ml, 10 µg/ml und 5 µg/ml NFATc (C) bzw. NFATp (D) gebunden. Als Kontrolle diente ein E. coli-Zellextrakt, der zuvor mit einem Vektor ohne Insert transformiert und parallel mit den Zellextrakten der NFAT-Proteine aufbereitet wurde. Die einzelnen Werte der verschiedenen Bindungskurven wurden mit dem Evaluationsprogramm des Biacore-Systems von den Kontrollwerten abgezogen. NFAT-Proteine binden im Flüssigphasesystem an immobilisierte EGR-Proteine. Die EGR/NFAT-Interaktionen finden auch in umgekehrter Weise mit immobilisierten NFATProteinen statt. NFATc und NFATp wurden rekombinant exprimiert, auf der Oberfläche eines CM-5-Chips immobilisiert und die Interaktionen mit EGR-3 und EGR-4 mit der Plasmonresonanz in Echtzeit verfolgt. In Abbildung 13 ist für die EGR-Interaktion an - 53 - Ergebnisse immobilisierten NFAT-Proteinen beispielsweise die Interaktion von EGR-4 an NFATc und NFATp dargestellt. EGR-4 bindet im Flüssigphasesystem sowohl an NFATc als auch an NFATp. Die Interaktion von EGR-4 mit immobilisiertem NFATc zeigte konzentrationsabhängige Bindungen mit relativen Resonanzwerten von 550, 600 und 625 RU (Abb. 13 A). Die Resonanzwerte, die durch die Interaktion von EGR-4 an NFATp hervorgerufen wurden, waren niedriger als die der EGR-4/NFATc-Interaktion (500-600 RU, Abb. 13 B). Abb. 13: Bestimmung der EGR/NFAT-Interaktionen mit der Plasmonresonanz Die Interaktion von EGR-4 an immobilisierte NFAT-Proteine wurde mit der Plasmonresonanz im Biacore-System untersucht. EGR-4 aus Sf9-Insektenzellen wurde aufgereinigt und in der Flüssigphase zu immobilisierten NFATProteinen hinzugegeben. (A) Die Interaktion von 1 µg/ml, 2 µg/ml und 4 µg/ml EGR-4 an immobilisiertes NFATc führten zu dosisabhängigen Bindungen, die Resonanzwerte zwischen 550 und 625 RU hervorriefen. (B) Die Bindungen von EGR-4 an immobilisiertem NFATp riefen Resonanzwerte von 550-600 RU hervor. Die einzelnen Werte der Bindungskurven wurden im Evaluationsprogramm von Kontrollwerten abgezogen. 3.5 Lokalisierung von EGR-3-Interaktionsdomänen EGR-3 interagiert mit Proteinen der NFAT-Familie und bildet mit NFATc bzw. NFATp Proteinkomplexe. Es stellte sich nun die Frage, über welche Domäne im EGR-3-Protein die Interaktion mit Interaktionsdomäne NFAT-Proteinen wurden vermittelt wird. EGR-3-Deletionsmutanten Für als die Charakterisierung Protein der C-Fusionsproteine rekombinant in E. coli exprimiert und das Bindungsverhalten an NFAT-Proteinen untersucht. 3.5.1 Expression von EGR-3-Deletionsmutanten Für die Lokalisation der EGR-3-Domäne, welch die Bindung von EGR-3 mit NFAT Proteinen vermittelt, wurden vier EGR-3-Deletionsmutanten hergestellt und in E. coli rekombinant exprimiert. Die Abbildung 14 A gibt einen Überblick über die Struktur der vier EGR-3- 54 - Ergebnisse Deletionsmutanten I, II, III und IV. Die Deletionsmutanten besitzen die Regionen aa 1-97 (EGR3-I), aa 98-168 (EGR-3-II), aa 169-260 (EGR-3-III) und aa 261-387 (EGR-3-IV). Die EGR-3Deletionsproteine wurden im SDS-PAGE aufgetrennt und im Westernblot mit spezifischen Antikörpern detektiert. Die Deletionsmutanten zeigten Banden mit Mobilitäten von 11,0 kDa (EGR-3-I), 8,0 kDa (EGR-3-II), 11,5 kDa (EGR-3-III) und 16,0 kDa (EGR-3-IV) (Abb. 14 B). Abb. 14: Struktur und Expression der EGR-3-Deletionsmutanten (A) Struktur von EGR-3 mit der Zinkfingerdomäne (grau) im Vergleich zu den Deletionsmutanten EGR-3-I (aa 197), EGR-3-II (aa 98-168), EGR-3-III (aa 169-260) und EGR-3-IV (aa 261-387). (B) Expression der rekombinanten EGR-3-Deletionsmutanten. Die Proteine wurden in E. coli exprimiert, aufgereinigt, durch SDS-PAGE getrennt und im Westernblot mit spezifischen anti-Protein C-Antikörpern detektiert. EGR-3-I zeigte eine Bande mit einer Mobilität von 11 kDa, die Deletionsmutanten EGR-3-II, EGR-3-III und EGR-3-IV zeigten Banden mit Mobilitäten von 8 kDa, 11,5 kDa und 16 kDa. Kontrolle siehe Abb. 18. 3.5.2 Zwei Domänen von EGR-3 vermitteln die Interaktion mit NFAT Die einzelnen EGR-Deletionsmutanten wurden als Protein C-Fusionsproteine in E. coli exprimiert und in Interaktionsstudien mit NFAT-Proteinen untersucht (Abb. 15). Rekombinantes NFATc und NFATp wurden an einer Glutathionmatrix immobilisiert und mit Zellextrakten der verschiedenen EGR-3-Deletionsmutanten inkubiert. Die Proteine der verschiedenen Fraktionen wurden im SDS-PAGE getrennt und im Westernblot mit spezifischen Antikörpern detektiert. Die Deletionsmutante EGR-3-II bindet an NFATc, da es in den Fraktionen Zellextrakt und Eluat nachgewiesen wurde (Abb. 15 A, Spuren 3-4). EGR-3-IV interagiert ebenfalls mit NFATc und wurde zusammen mit NFATc eluiert (Abb. 15 A, Spuren 7-8). Die Deletionsmutanten EGR-3-I und EGR-III binden nicht an NFATc (Abb. 15 A, Spuren 1-2, 5-6). EGR-3-I und EGR-3-III wurden in der Zellextraktfraktionen, aber nicht in der Elutionsfraktionen nachgewiesen. An NFATp binden die Deletionsmutanten EGR-3-II und EGR-IV, da sie sowohl in den Zellextraktfraktionen als auch in den Elutionsfraktionen detektiert wurden (Abb. 15 B, Spuren 34, 7-8). Eine Interaktion von EGR-3-I mit NFATp konnte nicht nachgewiesen werden (Abb. 15 - 55 - Ergebnisse B, Spuren 1-2). EGR-3-III wurde nicht in der Elutionsfraktion detektiert, weshalb eine Bindung an NFATp nicht nachgewiesen werden konnte (Abb. 15 B, Spuren 5-6). Die Interaktion von NFATc und NFATp findet in den EGR-3-Regionen aa 98-168 (EGR-3-II) und aa 261-387 (EGR-3-IV) statt. Abb. 15: Interaktion von EGR-3-Deletionsmutanten und NFAT-Proteinen Die EGR-3-Deletionsmutanten EGR-3-I, EGR-3-II, EGR-3-III und EGR-3-IV wurden rekombinant in E. coli exprimiert, mit immobilisierten NFAT-Proteinen inkubiert, gewaschen und mit Glutathion eluiert. Die Proteine der verschiedenen Fraktionen wurden nach einer SDS-PAGE auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung der EGR-3-Deletionsmutanten an immobilisiertes NFATc. Die Deletionsmutanten EGR-3-II (Spuren 3-4) und EGR-3-IV (Spuren 7-8) wurden gemeinsam mit NFATc eluiert. Rekombinantes EGR-3-I (Spuren 1-2) bzw. EGR-3-III (Spuren 5-6) konnte nicht an NFATc gebunden werden. (B) Bindung von EGR-3-Deletionsmutanten an immobilisiertes NFATp. Während die Deletionsmutanten EGR-3-II (Spuren 3-4) und EGR-3-IV (Spuren 7-8) an NFATp binden, konnte eine Interaktion von EGR-3-I (Spuren 1-2) und EGR-3-III (Spuren 5-6) mit NFATp nicht nachgewiesen werden. Kontrolle siehe Abb. 19. el: Eluat, rek: rekombinant, ze: Zellextrakt. 3.5.3 Interaktion von EGR-3-Deletionsmutanten mit der Plasmonresonanz Die Interaktionsdomänen von EGR-3 wurden in Proteinbindungsassays in den Regionen aa 98168 (EGR-3-II) und aa 261-387 (EGR-3-IV) lokalisiert. Es stellte sich die Frage, ob die NFATProteine an den EGR-3-Interaktionsdomänen mit unterschiedlichen Affinitäten gebunden werden. Deshalb wurden die EGR-3-Deletionsmutanten in Plasmonresonanz-Studien mit immobilisierten NFAT-Proteinen eingesetzt und das Interaktionsverhalten der Deletionsmutanten charakterisiert. Die Bindungen der vier EGR-3-Deletionsmutanen an NFATProteine wurde in Echtzeit verfolgt und mit der Bindung des intakten EGR-3-Proteins verglichen (Abb. 16). - 56 - Ergebnisse Die Deletionsmutanten EGR-3-II und EGR-3-IV binden an NFATc, wobei EGR-3-II stärker als EGR-3-IV an NFATc gebunden wurde (Abb. 16 A). Bei der Interaktion von EGR-3-II mit NFATc wurde ein Resonanzwert von 575 RU erreicht und war damit um 100 RU höher als der Resonanzwert von EGR-3-IV bei der Bindung an NFATc. Eine Interaktion von EGR-3-I bzw. EGR-3-III mit NFATc konnte nicht nachgewiesen werden, da die Bindungskurven keine Resonanzwerte hervorriefen (Abb. 16 A). An NFATp binden die Deletionsmutanten EGR-3-II und EGR-3-IV mit unterschiedlicher Intensität (Abb. 16 B). EGR-3-II bindet mit 70 RU stärker als EGR-IV an NFATp, da diese Interaktion zu relativen Resonanzwerten von 450 RU führte. Das intakte EGR-3 bindet stärker an NFAT-Proteine als die EGR-3-Deletionsmutanten EGR-3-II und EGR-IV (Abb. 16 A und B). Abb. 16: Bestimmung der Interaktion von EGR-3-Deletionsmutanten mit der Plasmonresonanz Die Bindungen von intaktem EGR-3 und den EGR-3-Deletionsmutanten EGR-3-I, EGR-3-II, EGR-3-III und EGR3-IV an NFAT-Proteine wurden mit der Plasmonresonanz im Biacore-System untersucht. Die rekombinant in E. coli exprimierten EGR-3-Deletionsmutanten und das rekombinant in Sf-9-Insektenzellen exprimierte EGR-3 wurden aufgereinigt und in der Flüssigphase zu immobilisierten NFAT-Proteinen hinzugegeben. (A) Bindung der EGR-3Deletionsmutanten im Vergleich zum intaktem EGR-3 an immobilisiertes NFATc. Das intakte EGR-3 bindet stärker an NFATc als die Deletionsmutanten EGR-3-II und EGR-3-IV, während EGR-3-I und EGR-3-III nicht an NFATc gebunden werden konnten. (B) An immobilisiertes NFATp bindet das intakte EGR-3 stärker als die Deletionsmutanten EGR-3-II und EGR-3-IV. Für die Deletionsmutanten EGR-3-I und EGR-3-III konnten keine Bindungen an NFATp nachgewiesen werden. Alle Werte der Bindungskurven wurden im Evaluationsprogramm von den Kontrollwerten abgezogen. 3.6 Lokalisierung von EGR-4-Interaktionsdomänen Für die Charakterisierung der EGR-Interaktion mit den NFAT-Proteinen war es weiterhin von Interesse, die Interaktionsdomänen von EGR-4 zu lokalisieren und zu überprüfen, ob EGR-3 und EGR-4 in gleicher Weise mit den NFAT-Proteinen interagieren. Von EGR-4 wurden verschiedene Deletionsmutanten in E. coli exprimiert und die Interaktionsfähigkeit mit NFATProteinen untersucht. - 57 - Ergebnisse 3.6.1 Expression von EGR-4-Deletionsmutanten Zur Lokalisation der Interaktionsdomäne von EGR-4, die für die Bindung von NFAT-Proteinen verantwortlich ist, wurden zusätzlich zu den vorhandenen drei EGR-4-Deletionsmutanten sechs weitere Mutanten hergestellt. Die Struktur der EGR-4-Deletionsmutanten ist in Abbildung 17 A dargestellt. Die bereits vorhandenen Deletionsmutanten besitzen von EGR-4 die N-terminale Region aa 1-158 (EGR-4-I), die mittlere Region aa 158-325 (EGR-4-II) und die C-terminale Region aa 325-487 (EGR-4-III). Die zusätzlichen Deletionsmutanten weisen die Bereiche aa 379-463 (EGR-4-IV), aa 79-241 (EGR-4-VI), aa 119-201 (EGR-4-VII), aa 139-181 (EGR-4VIII), aa 1-127 (EGR-4-IX) und aa 193-325 (EGR-4-X) auf. Die in E. coli exprimierten Proteine wurden durch SDS-PAGE getrennt und mit spezifischen Antikörpern detektiert. Die Deletionsmutanten EGR-4-I, EGR-4-II, EGR-4-III und EGR-4-IV zeigten bei der Proteinauftrennung Banden mit Mobilitäten von 15,0 kDa, 19,0 kDa, 18,0 kDa und 10,0 kDa (Abb. 17 B). Die weiteren EGR-4-Deletionsmutanten wurden mit relativen Massen von 17,4 kDa (EGR-4-VI), 9,4 kDa (EGR-4-VII), 5,4 kDa (EGR-4-VIII), 14 (EGR-4-IX) und 15,0 kDa (EGR4-X) identifiziert (Abb. 17 B). Abb. 17: Struktur und Expression der EGR-4-Deletionsmutanten EGR-4 und EGR-4- Deletionsmutanten sind strukturell in (A) dargestellt. EGR-4-I besitzt den N-terminalen Bereich von EGR-4 (aa 1-158), EGR-4-II die mittlere Region (aa 158-325), EGR-4-III die C-terminale Region (aa 325-487) und EGR-4-IV die Zinkfingerdomäne (aa 379-463) von EGR-4. Weitere Deletionsmutanten sind EGR-4-VI (aa 79241), EGR-4-VII (aa 119-201), EGR-4-VIII (aa 139-181), EGR-4-IX (aa 1-127) und EGR-4-X (aa 193-325). (B) Expression der EGR-4-Deletionsmutanten. Die EGR-4-Deletionsmutanten wurden rekombinant in E. coli als Protein C-Fusionsproteine exprimiert und aufgereinigt. Die Proteine wurden durch SDS-PAGE getrennt, im Westernblot detektiert und zeigen Banden mit Mobilitäten von 15 kDa (I), 19 kDa (II), 18 kDa (III), 10 kDa (IV), 17,4 kDa (VI), 9,4 kDa (VII), 5,4 kDa (VIII), 14 (IX) und 15 kDa (X). Als Kontrolle (co; Spur 10) diente ein E. coli-Zellextrakt, der zuvor mit einem Vektor ohne Insert transformiert wurde. - 58 - Ergebnisse 3.6.2 Zwei Domänen von EGR-4 vermitteln die Interaktion mit NFAT Für die Lokalisierung der EGR-4-Interaktionsdomäne, die für die Bindung von NFAT-Proteinen verantwortlich ist, wurden die EGR-4-Deletionsmutanten in Proteinbindungsassays mit NFATc und NFATp eingesetzt. In Abbildung 18 sind Bindungen der EGR-4-Deletionsmutanten EGR-4I, EGR-4-II, EGR-4-III, EGR-4-IV, EGR-4-VI, EGR-4-VII, EGR-4-VIII, EGR-4-IX und EGR4-X an NFATc und NFATp dargestellt. Die Deletionsmutanten EGR-4-I und EGR-4-II binden an NFATc (Abb. 18 A, Spuren 1-4), da sie zusammen mit NFATc eluiert werden konnten. EGR4-III und EGR-4-IV konnten zwar in den Zellextraktfraktionen, aber nicht in den Elutionsfraktionen nachgewiesen werden und binden somit nicht an NFATc (Abb. 18 A, Spuren 5-8). An NFATp binden ebenfalls EGR-4-I und EGR-II (Abb. 18 B, Spuren 1-4), deren Proteine in den Elutionsfraktionen detektiert werden konnten. Alle weiteren EGR-4-Deletionsmutanten konnten weder an NFATc noch an NFATp gebunden werden (Abb. 18). Als Kontrolle wurde ein parallel zu den Deletionsmutanten behandelter Zellextrakt einer E. coli-Kultur verwendet, die zuvor mit dem Expressionsvektor ohne Insert transformiert wurde (Abb. 18 B, Spuren 19-20). Abb. 18: Interaktion von EGR-4-Deletionsmutanten und NFAT-Proteinen Die EGR-4-Deletionsmutanten EGR-4-I, EGR-4-II, EGR-4-III, EGR-4-IV, EGR-4-VI, EGR-4-VII, EGR-4-VIII, EGR-4-IX und EGR-4-X wurden rekombinant E. coli exprimiert, aufgereinigt, mit immobilisierten NFAT-Proteinen inkubiert, gewaschen und mit Glutathion eluiert. Die Proteine wurden nach einer SDS-PAGE auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung von EGR-4-Deletionsmutanten an NFATc. Die Deletionsmutanten EGR-4-I und EGR-4-II binden an NFATc (Spuren 1-4). Für die weiteren EGR-4Deletionsmutanten konnten keine Bindungen an NFATc nachgewiesen werden (Spuren 5-18). (B) Bindung von EGR-4-Deletionsmutanten an NFATp. An immobilisiertes NFATp binden sowohl EGR-4-I als auch EGR-4-II (Spuren 1-4). Auch hier zeigten die weiteren Deletionsmutanten EGR-4-III, EGR-4-IV, EGR-4-VI, EGR-4-VII, EGR-4-VIII, EGR-4-IX und EGR-4-X keine Bindung an NFATp (Spuren 5-18). Eine unspezifische Bindung der Deletionsmutanten EGR-4-I und EGR-4-II an die NFAT-Proteine konnte ausgeschlossen werden, da weitere Deletionsmutanten und ein Zellextrakt von E. coli, der mit einem Vektor ohne Insert transformiert wurde, nicht an die Matrix gebunden werden konnten (co; Spuren 19-20). el: Eluat, ze: Zellextrakt. - 59 - Ergebnisse 3.6.3 Interaktionen von EGR-4-Deletionsmutanten mit der Plasmonresonanz Für die Bindung von EGR-4 an NFAT-Proteine wurden mit EGR-4-Deletionsmutanten zwei Interaktionsdomänen im EGR-4-Protein lokalisiert (3.6.2). Es stellte sich nun die Frage, ob es Unterschiede bei der Bindung der NFAT-Proteine an die beiden EGR-4-Domänen gibt. Deshalb wurde die Bindung der EGR-4-Deletionsmutanten EGR-4-I, EGR-4-II, EGR-4-III und EGR-4IV an NFAT-Proteine mit der Plasmonresonanz im Biacore-System untersucht. EGR-4-II bindet an NFATc stärker als EGR-4-I (Abb. 19 A). Der relativen Resonanzen der Interaktion von EGR-4-II mit NFATc (RU 625) waren stärker als die Resonanzwerte der Bindung von EGR-4-I (RU 550). Mit den Deletionsmutanten EGR-4-III bzw. EGR-IV konnte keine Interaktion an NFATc nachgewiesen werden. In Abbildung 19 B sind die Bindungen von intaktem EGR-4 und den Deletionsmutanten EGR-4-I, EGR-4-II, EGR-4-III und EGR-4-IV an NFATp dargestellt. Die Bindung von EGR-4-I an NFATp führte zu Resonanzwerten von 575 RU (Abb. 19 B). Die Assoziation von EGR-4-II an NFATp rief Resonanzwerte von 650 RU hervor und war somit stärker als die von EGR-4-I an NFATp. EGR-4-III und EGR-4-IV konnten nicht an NFATp gebunden werden. Die Deletionsmutanten EGR-4-I und EGR-4-II binden sowohl NFATc als auch NFATp schwächer als das intakte EGR-4-Protein (Abb. 19 A und B). Abb. 19 Bestimmung der Interaktion von EGR-4-Deletionsmutanten mit der Plasmonresonanz Die Bindung von EGR-4 und EGR-4-Deletionsmutanten an NFAT-Proteine wurde mit der Plasmonresonanz im Biacore-System untersucht. Die rekombinant in E. coli exprimierten und aufgereinigten Deletionsmutanten EGR-4I, EGR-4-II, EGR-4-III und EGR-4-IV wurden im Vergleich zum intakten EGR-4 aus Sf9-Insektenzellen zu immobilisiertem NFAT-Proteinen hinzugegeben. (A) Die Bindung von intaktem EGR-4 an NFATc ist stärker als die Bindung von EGR-4-I und EGR-4-II an NFATc. EGR-4-III und EGR-4-IV konnten nicht an NFATc gebunden werden. Die Interaktionen von NFATp mit intaktem EGR-4 und den EGR-4-Deletionsmutanten sind in (B) dargestellt. Die Deletionsmutanten EGR-4-I und EGR-4-II binden ebenso wie das intakte EGR-4 an NFATp, während die Deletionsmutanten EGR-4-III und EGR-4-IV nicht an NFATp gebunden werden konnten. Im Evaluationsprogramm wurden die Werte der verschiedenen Kurven von den Werten der Kontrollkurven abgezogen. - 60 - Ergebnisse In Proteinbindungsstudien konnten die EGR-Domänen, die für die Interaktion mit NFATProteinen verantwortlich sind, lokalisiert werden. EGR-3 bindet NFAT-Proteine mit zwei Domänen, die in den Regionen aa 98-168 und aa 261-387 lokalisiert sind. EGR-4 besitzt die zwei Dömanen aa 1-158 und aa 158-325 für die Interaktion mit NFAT-Proteinen. In Plasmonresonanz-Studien konnte gezeigt werden, dass jeweils die intakten EGR-Proteine stärker an NFAT-Proteine binden als die einzelnen EGR-Deletionsmutanten. EGR-Proteine binden NFAT-Proteine mit unterschiedlichen Affinitäten an die Domänen. Die EGR-3-Domäne der Region aa 98-168 bindet NFAT-Proteine stärker als die Domäne der Region aa 261-387. Sowohl NFATc als auch NFATp werden von der EGR-4-Domäne der Region aa 158-325 stärker gebunden als von der Domäne in der Region aa 1-158. 3.6.4 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit NFATp EGR-Proteine können kooperativ mit NFAT-Proteinen die Transkription verschiedener Gene regulieren (Decker et al., 2003; Zipfel et al., 2003). Nachdem die Domänen lokalisiert wurden, die für direkte Interaktionen mit den NFAT-Proteinen verantwortlich sind, war es von Interesse, die Funktionalität der EGR-Domänen zu untersuchen. Deshalb wurden in transienten Transfektionen EGR-4-Deletionsmutanten eingesetzt und die funktionelle Kooperation mit NFATp am Promotor des humanen Interleukins 2 untersucht. EGR-4 und NFATp verstärken kooperativ die Transkription des IL-2-Gens (Abb. 20). In den Transfektionsexperimenten zeigte EGR-4 alleine keine transkriptionelle Aktivität, während NFATp die Transkription IL-2-Reporterkonstrukts um das 18-fache aktivierte. Die Kombination von EGR-4 und NFATp führte zu einer 80-fachen Aktivierung des IL-2-Promotors. Um herauszufinden, welche EGR-4-Domäne für die synergistische Interaktion verantwortlich ist, wurden weiterhin die Deletionsmutanten EGR-4-MI (aa 150-487), EGR-4 MII (aa 325-487), EGR-4 MIII (aa 379-487) und EGR-4 MIV (aa 1-463) eingesetzt. Die Deletion der N-terminalen 150 Aminosäuren (EGR-4 MI) zeigte keinen Effekt auf die Transkription des IL-2-Gens. Die Deletion der mittleren EGR-4-Region aa 150-325 führte bei der Kooperation mit NFATp zu einer reduzierten Transkription um 75 %. Die weitere Deletion (E4 MIII) reduzierte die Transaktivierung noch einmal, während die Deletion der C-terminalen Aminosäuren von EGR-4 keinen Einfluss auf die Reportergenaktivität hatte. - 61 - Ergebnisse Abb. 20: Funktionale Interaktion von EGR-4 und NFATp Humane 293T-Zellen wurden mit einem Reportergenkonstrukt transfiziert, das die Promotorregionen bp -302 bis 257 (3x) des humanen Interleukins-2 und ein Luciferasegen enthält. Die Bindungsstellen des Reportergenkonstrukts für die entsprechenden Transkriptionsfaktoren sind unten im Bild dargestellt. Das Reporterkonstrukt wurde zusammen mit den Expressionsvektoren, die für die EGR-Proteine und NFATp kodieren, in den dargestellten Kombinationen kotransfiziert. Die Aktivierungswerte repräsentieren Mittelwerte und Standardabweichungen von mindestens drei unabhängigen Experimenten. ZIP: Zinkfingerprotein-Binderegion, EGR-Bindungsstelle. In den Transfektionsstudien wurde die Funktionalität der Interaktion von EGR- und NFATProteinen untersucht. EGR-4 und NFATp aktivieren in einer synergistischen Kooperation die Transkription des IL-2-Gens. Die synergistische Kooperation wird durch die Interaktion von NFATp in der EGR-4-Region aa 150-325 vermittelt. 3.6.5 EGR-4/NFAT-Interaktion vermittelnde Aminosäuren Für die weitere Charakterisierung der EGR-4-Interaktionsdomäne war es von Interesse, die Interaktionsbereiche auf Peptidebene zu untersuchen. In der EGR-4-Region aa 158-325 werden sowohl direkte als auch funktionale Interaktionen mit den NFAT-Proteinen vermittelt. Für die Lokalisierung von Bindungsmotiven im EGR-4-Protein wurden von der Region aa 158-325 Peptide synthetisiert, auf einer Membran immobilisiert und in Peptidspotanalysen mit NFATProteinen untersucht. EGR-4 bindet NFATc in der Region aa 158-325 an fünf lineare Aminosäurebereiche (D1c-D5c) (Abb. 21 A). Die Aminosäuresequenzen der jeweiligen Bereiche sind in Abbildung 21 B dargestellt. Die Aminosäurebereiche unterscheiden sich bei der Bindung von NFATc sowohl in der Länge als auch in der Intensität. EGR-4 bindet NFATc neben den Aminosäurebereichen - 62 - Ergebnisse D1c-D4c am stärksten im linearen Bereich D5c. Die Bindung von NFATp an EGR-4 findet ebenfalls über fünf lineare Aminosäurebereiche (D1p-D5p) statt, von denen D1p die Bindung von NFATp am stärksten vermittelt (Abb. 21 C). Die Sequenzen der Aminosäurebereiche D1pD5p sind in Abbildung 21 D angegeben. Die in Abbildung 21 dargestellten EGR-4-Domänen für die Interaktion mit NFATc und NFATp sind im Vergleich in Tabelle 9 dargestellt. Abb. 21: EGR-4/NFAT-Interaktion vermittelnde Aminosäurebereiche Für die Lokalisierung von Aminosäuren, die in der mittleren EGR-4-Domäne die NFAT-Interaktion vermitteln, wurden Peptide der EGR-4-Region (aa 158-325) synthetisiert, auf einer Membran immobilisiert und mit NFATProteinen inkubiert. Die Detektion erfolgte mit spezifischen Antikörpern. (A) Bindung von NFATc an EGR-4. NFATc bindet an fünf EGR-4-Aminosäurebereiche (D1c-D5c), dessen Aminosäuresequenzen in (B) angegeben sind. (C, D) Bindung von NFATp an EGR-4. NFATp bindet ebenfalls an fünf Aminosäurebereiche von EGR-4 (D1p-D5p). Die Aminosäuren der EGR-4-Peptide, welche die stärksten Intensitäten durch die Bindung von NFATProteinen zeigten, sind in Kästen dargestellt. Tabelle 9: EGR-4/NFAT-Interaktion vermittelnde Aminosäurebereiche Darstellung und Vergleich der EGR-4-Aminosäurebereiche, die die Interaktionen mit NFATc und NFATp vermitteln. Die Aminosäuresequenzen der Peptide, welche die stärksten Interaktionen zeigten (Abb. 21) sind hervorgehoben. Weiterhin ist die Position der Peptide im EGR-4-Protein angegeben. Bereich D1c D1p Sequenz YEPQLSPPDVKPGLRRPPASPALDAVS PQLSPPDVKPGLRRPPASPALDAVSAF Position (aa) 173-199 175-201 D2c AVSAFKGPYAPWELLSVGAPNC 197-218 D2p AVSAFKGPYAPWELLSVGAPN 197-217 D3c D3p CGSQGDYQAAPEARFPVIGTKIE VGAPNCGSQGDYQAAPEARFPVIGTK 218-240 213-238 D4c VPANRLYPSGAYDAFPLAPGDLGEG 254-278 D4p VPANRLYPSGAYDAFPLAPGD 254-273 D5c FLASTQPQLSPLGLRSAAAADFP 302-324 D5p FLASTQPQLSPLGLRSAAAADFPKPLV 302-328 - 63 - Ergebnisse NFATc und NFATp binden in der EGR-4-Region aa 158-325 dieselben fünf Aminosäurebereiche, allerdings mit unterschiedlichen Affinitäten. NFATc bindet stark an das Peptid 8 des ersten Aminosäurebereiches D1, während NFATp das Peptid 13 am stärksten bindet. NFATc bindet im fünften Aminosäurebereich (D5) am stärksten an das Peptid 75, NFATp hingegen an das Peptid 77. Das Peptid 34 des Aminosäurebereichs D3 wurde von NFATc und NFATp gleichermaßen gebunden, so dass dieses Peptid für weitere Charakterisierungen mit einbezogen wurde. Um die relevanten Aminosäuren zu identifizieren, die den Kontakt zwischen EGR-4 und den NFAT-Proteinen ausbilden, wurden einzelne Aminosäuren in einer „Glycine-walk“Peptidspotanalyse ausgetauscht. Der Austausch relevanter Aminosäuren gegen Glycin (bzw. Alanin) führte zu einem Verlust der Bindung von NFAT-Proteinen an EGR-4 (Abb. 22). EGR-4 bindet NFATc vorwiegend mit den positiv geladenen Aminosäuren Lysin und Arginin (Abb. 22 A). Nach dem Austausch der Aminosäuren Lysin183 und Arginin187 gegen Glycin bindet NFATc nicht mehr an EGR-4 (Abb. 22 A, GW 8). Weiterhin wird NFATc nicht mehr von EGR-4 gebunden, wenn die Aminosäuren Arginin231 und Lysin238 ausgetauscht werden (Abb. 22 A, GW 34). NFATc wird von EGR-4 über die Aminosäuren Serin312, Prolin312, Lysin313, Arginin316 und Glycin314 gebunden, da ein Austausch dieser Aminosäuren zu einem Verlust der Bindung führte (Abb. 22 A, GW 75). EGR-4 bindet NFATp ebenfalls über die positiv geladenen Aminosäuren Lysin und Arginin (Abb. 22 B). NFATp kann nicht mehr an EGR-4 binden, nachdem die Argininreste der Positionen 187 und 188 gegen Glycin ausgetauscht wurden (Abb. 22 B, GW 13). Die Bindung von NFATp findet weiterhin an die Aminosäuren Arginin231 und Lysin238 von EGR-4 statt, da durch den Austausch die Bindung von NFATp an EGR-4 gehemmt wurde (Abb. 22 B, GW 34). Die Aminosäuren Lysin313/315, Arginin316 und Alanin318 von EGR-4 konnten für die NFATp-Bindung durch einen Austausch lokalisiert werden (Abb. 22 B, GW 77). - 64 - Ergebnisse Abb. 22: EGR-4/NFAT-Interaktion vermittelnde Aminosäuren Um die relevanten Aminosäuren zu identifizieren, die den Kontakt zwischen EGR-4 und den NFAT-Proteinen ausbilden, wurden einzelne Aminosäuren in einer „Glycine-walk“-Peptidspotanalyse ausgetauscht. In den Peptiden 8, 34, 75 (NFATc) und 13, 34, 77 (NFATp) wurden schrittweise einzelne Aminosäuren gegen Glycin (bzw. Alanin) ausgetauscht. Die Peptide wurden auf einer Membran immobilisiert und mit NFATc (A) bzw. NFATp (B) inkubiert. Die Detektion erfolgte mit spezifischen Antikörpern. Die Aminosäuren, die nach einem Austausch nicht mehr von NFATc oder NFATp gebunden werden, sind hervorgehoben. Siehe Tabelle 10. Die Tabelle 10 gibt einen Überblick über die Aminosäuredomänen, Peptide und relevanten Aminosäuren von EGR-4, die für Interaktion mit den NFAT-Proteinen verantwortlich sind. Tabelle 10: Vergleich der EGR-4/NFAT-Interaktion vermittelnden Aminosäuren Darstellung und Vergleich der Aminosäuren, die die Interaktionen mit NFATc und NFATp vermitteln. Die EGR-4Aminosäuren, die nach einem Austausch NFAT-Proteine nicht mehr binden (Abb. 22) sind hervorgehoben. Weiterhin ist die Position der Aminosäuren im EGR-4-Protein angegeben. Bereich D1c D1p Sequenz YEPQLSPPDVKPGLR KPGLRRPPASPALDA Peptid Position (aa) 8 173-187 13 183-197 D3c YQAAPEARFPVIGTK 34 224-238 D3p YQAAPEARFPVIGTK 34 224-238 D5c TQPQLSPLGLRSAAA 75 306-320 77 310-324 D5p LSPLGLRSAAAADFP 3.6.6 Inhibition der EGR/NFAT-Interaktion Die Interaktion von EGR-4 und NFATc wird von der EGR-4-Region (aa 158-325) durch fünf kurze Aminosäurebereiche vermittelt. EGR-4 bindet NFATc stark im Bereich aa 306-320 (Abb. 21-22). Es stellte sich die Frage, ob die Bindung von NFATc an diesem Aminosäurebereich im intakten EGR-4 gehemmt werden kann. Deshalb wurden Peptide, die der EGR-4-Region aa 306320 (TQPQLSPLGLRSAAA) entsprechen, synthetisiert und der Einfluss auf die EGR4/NFATc-Interaktion mit Hilfe der Plasmonresonanz untersucht. Die Abbildung 23 zeigt die - 65 - Ergebnisse Auswirkung des inhibitorischen NFAT-Peptids auf die Interaktion von EGR-4 und NFATc. Das inhibitorische NFAT-Peptid hat einen hemmenden Einfluss auf die EGR-4/NFATc-Interaktion. Die Bindung von NFATc an EGR-4 zeigte ohne Peptide relative Resonanzwerte von 575 RU, während die Zugabe der inhibitorischen Peptide die Bindung auf 375 RU reduzierte. Ein Kontrollpeptid besaß keinen Einfluss auf die EGR-4/NFATc-Interaktion. Die EGR-4/NFATcInteraktionen mit und ohne Kontrollpeptid riefen Resonanzwerte von 575 RU hervor. Abb. 23: Einfluss eines inhibitorischen Peptids auf die EGR-4/NFATc-Interaktion Die Interaktion von EGR-4 und NFATc wurde in Anwesenheit inhibitorischer Peptide mit der Plasmonresonanz im Biacore-System untersucht. Rekombinant exprimiertes und aufgereinigtes NFATc (1 µg/ml) wurde mit 10 µg/ml NFAT-Peptiden (TQPQLSPLGLRSAAA) oder 10 µg/ml Kontrollpeptiden (EKKRHSKVHLKQKAR) inkubiert und zu immobilisiertem EGR-4 hinzugegeben. Als Kontrolle wurde die Interaktion von EGR-4 und NFATc ohne Peptide untersucht. Die einzelnen Werte der verschiedenen Kurven wurden im Evaluationsprogramm von den Werten der Kontrollkurven abgezogen. 3.7 Interaktion von EGR- und NFκB-Proteinen EGR-Proteine interagieren während der Immunantwort im Zellkern auch mit anderen Transkriptionsfaktoren, zu denen z. B. die Mitglieder der NFκB-Familie p50 und p65 gehören. Die NFκB- und NFAT-Proteine besitzen wichtige regulatorische Funktionen bei der Immunantwort und zeigen in bestimmten Regionen eine hohe Homologie zueinander. Die NFκB-Proteine besitzen eine hoch konservierte Rel-Domäne, welche die Fähigkeit zur Homobzw. Heterodimerisierung und die DNA-Bindung vermittelt (Bours et al., 1992 a und b). Die stark konservierten DNA-Bindungsdomänen der NFAT-Proteine gleichen strukturell den RelDomänen der Rel/NFκB-Proteine und werden aufgrund der hohen Homologie (65-70 %) auch als Rel Similarity“-Domänen (RSD) bezeichnet (Jain et al., 1995). Es stellte sich die Frage, ob - 66 - Ergebnisse die Interaktionen zwischen EGR- und NF-κB-Proteinen mit den EGR/NFAT-Interaktionen vergleichbar sind. Die Bindung von EGR-3 und EGR-4 wurde mit den Proteinen p50 und p65 aus der NFκB-Familie in Proteinbindungsassays untersucht und mit der Plasmonresonanz charakterisiert. Für die Bindungsstudien wurden EGR-Proteine rekombinant aus Sf9Insektenzellextrakten und die NFκB-Proteine rekombinant in E. coli exprimiert (Abb. 4-5). Die nativen EGR- und NFκB-Proteine wurden aus stimulierten Jurkat-T-Zellen isoliert (Abb. 4-5). 3.7.1 Interaktion von EGR mit immobilisierten NFκB-Proteinen Die Mitglieder der NFκB-Familie p50 und p65 wurden rekombinant in E. coli als „GST“Fusionsproteine exprimiert und auf einer Glutathionmatrix immobilisiert (Abb. 24). Das NFκBProtein p50 bindet vollständig an die Glutathionmatrix, da es in allen Elutionsfraktionen nachgewiesen wurde (Abb. 24 A). Das NFκB-Protein p65 bindet an die Glutathionmatrix und konnte nach der Immobilisierung wieder eluiert werden (Abb. 24 B). Abb. 24: Immobilisierung der NFκB-Proteine p50 und p65 (A) Immobilisierung von p50 auf der Glutathionmatrix. Rekombinantes, in E. coli exprimiertes p50 wurde mit der Glutathionagarose inkubiert (Spuren 1-2), gewaschen (Spuren 3-5) und mit Glutathion eluiert (Spuren 6-8). (B) Immobilisierung von rekombinantem p65 aus E. coli-Zellextrakten (Spuren 1-2). Die immobilisierten p65-Proteine wurden gewaschen (Spuren 3-4) und eluiert (Spuren 5-8). Die Proteine wurden nach SDS-PAGE auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. el: Eluat, ünb: Überstand nach Bindung, w: Waschfraktion, ze: Zellextrakt. Für den Nachweis der EGR/NFκB-Interaktion wurden die NFκB-Proteine immobilisiert und mit EGR-3 und EGR-4 inkubiert. Sowohl rekombinante als auch native EGR-Proteine binden an die NFκB-Proteine p50 und p65 (Abb 25). Rekombinantes EGR-3 bindet an p50, da es in den Elutionsfraktionen nachgewiesen wurde (Abb. 25 A, Spuren 1-2). Natives EGR-3, das ebenfalls an p50 bindet, wurde zusammen mit p50 eluiert (Abb. 25 A, Spuren 3-4). EGR-4 bindet sowohl - 67 - Ergebnisse in rekombinanter als auch in nativer Form an das NFκB-Protein p50 (Abb. 25 A, Spuren 5-8). Die Interaktion von p65 mit rekombinantem EGR-3 sowie mit nativem EGR-3 wurde durch den Nachweis von EGR-3 in den Elutionsfraktionen gezeigt (Abb. 25 B, Spuren 1-4). EGR-4 bindet an p65, da sowohl rekombinantes als auch natives EGR-4 zusammen mit p65 eluiert werden konnte (Abb. 25 B, Spuren 5-8). EGR-3 und EGR-4 binden an p50 und p65, da die EGRProteine nach der Inkubation mit Glutathionagarose nicht in den Elutionsfraktionen nachgewiesen werden konnten (Abb. 7 C). Abb. 25: Bindung von EGR-3 und EGR-4 an immobilisierte NFκB-Proteine EGR-3 und EGR-4 wurden rekombinant in Sf9-Insektenzellen und nativ in stimulierten Jurkat-T-Zellen exprimiert. Die EGR-Proteine wurden mit immobilisierten NFκB-Proteinen inkubiert, gewaschen und mit Glutathion eluiert. Die Proteine wurden nach SDS-PAGE auf eine Nitrozellulosemembran transferiert und mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung von EGR-Proteinen an immobilisiertes p50. Rekombinantes EGR-3 (Spur 1) wurde gemeinsam mit p50 eluiert (Spur 2). Natives EGR-3 (Spur 3) aus Jurkat-T-Zellextrakt konnte an p50 gebunden werden (Spur 4). Die Interaktion von immobilisiertem p50 mit rekombinantem EGR-4 ist in den Spuren 5-6 und mit nativem EGR-4 in den Spuren 7-8 dargestellt. (B) Bindung von EGR-3 und EGR-4 an immobilisiertes p65. Rekombinantes EGR-3 (Spuren 1-2) und rekombinantes EGR-4 (Spuren 5-6) interagierten mit p65. Sowohl native EGR-3-Proteine (Spuren 3-4) als auch EGR-4-Proteine (Spuren 7-8) wurden an p65 gebunden. Kontrolle siehe Abb. 7. rek: rekombinant, ze: Zellextrakt. 3.7.2 Interaktion von NFκB mit immobilisierten EGR-Proteinen EGR-Proteine interagieren mit den Mitgliedern der NFκB-Familie, wenn diese immobilisiert werden. Für den Nachweis, dass die EGR/NFκB-Interaktion auch in umgekehrter Weise stattfindet, wurden weiterhin EGR-Proteine immobilisiert (Abb. 8) und mit p50 bzw. p65 inkubiert. Die Abbildung 26 A zeigt die Interaktionen von rekombinantem und nativem p50 und p65 mit immobilisiertem EGR-3. Rekombinantes und natives p50 bindet an EGR-3 und wurde in den Elutionsfraktionen nachgewiesen (Abb. 26 A, Spuren 1-4). Das NFκB-Protein p65 bindet - 68 - Ergebnisse ebenfalls an EGR-3, da sowohl rekombinantes als auch natives p65 in den Elutionsfraktionen nachgewiesen wurde (Abb. 26 A, Spuren 5-8). Die Interaktionen von rekombinantem und nativem p50 mit EGR-4 ist in Abbildung 26 B dargestellt. EGR-4 bindet sowohl rekombinantes als auch natives p50 (Abb. 26 B, Spuren 1-4). Rekombinantes p65 wurde in den Elutionsfraktionen nachgewiesen und zeigt eine Bindung an EGR-4 (Abb. 26 B, Spuren 5-6). Natives p65 bindet ebenfalls an EGR-4 (Abb. 26 B, Spuren 78). Die Bindung von EGR- und NFκB-Proteinen ist spezifisch, da die NFκB-Proteine nicht an die Nickelmatrix binden (Abb. 26 C). Abb. 26: Bindung von p50 und p65 an immobilisierte EGR-Proteine Die NFκB-Proteine p50 und p65 wurden rekombinant in E. coli exprimiert und nativ aus stimulierten Jurkat-TZellen isoliert. Die NFκB-Proteine wurden mit immobilisiertem EGR-3 bzw. EGR-4 inkubiert, gewaschen und eluiert. Die Proteine wurden durch SDS-PAGE getrennt und im Westernblot mit spezifischen Antikörpern detektiert. Dargestellt sind jeweils die Fraktionen Zellextrakt (ze) und Eluat 1 (el). (A) Bindung von NFκBProteinen an immobilisiertes EGR-3. Rekombinantes p50 (Spur 1-2) bzw. natives p50 (Spur 3-4) wurde gemeinsam mit EGR-3 eluiert. Die Interaktion von immobilisiertem EGR-3 mit rekombinantem p65 ist in den Spuren 5-6 und mit nativem p65 in den Spuren 7-8 dargestellt. (B) Bindung von p50 und p65 an immobilisiertes EGR-4. Rekombinantes p50 (Spuren 1-2) und rekombinantes p65 (Spuren 5-6) binden an EGR-4. Sowohl native p50Proteine (Spuren 3-4) als auch native p65-Proteine (Spuren 7-8) wurden an EGR-4 gebunden. (C) Inkubation der NFκB-Proteine mit der Nickelagarose. Eine unspezifische Bindung von rekombinantem oder nativem p50 (Spuren 1-4) bzw. p65 (Spuren 5-8) an EGR-Proteine konnte ausgeschlossen werden, da die NFkB-Proteine nicht an die Nickelmatrix binden. el: Eluat, rek: rekombinant, ze: Zellextrakt. 3.7.3 EGR/NFκB-Interaktionen mit der Plasmonresonanz Die Interaktionen von EGR-Proteinen mit den NFκB-Proteinen p50 und p65 wurde mit der Plasmonresonanz im Biacore-System untersucht, um die Bindungen in Echtzeit verfolgen und miteinander vergleichen zu können. NFκB-Proteine wurden in E. coli exprimiert und in der Flüssigphase zu immobilisierten EGR-Proteinen gegeben. - 69 - Ergebnisse Die Abbildung 27 zeigt das Bindungsverhalten der NFκB-Proteine an EGR-3 und EGR-4. Das NFκB-Protein p50 bindet an EGR-3, so dass durch die Interaktion konzentrationsabhängige Resonanzwerte von 75, 100 und 150 RU hervorgerufen wurden (Abb. 27 A). p65-Proteine binden an EGR-3 stärker als p50-Proteine, da die Interaktion von p65 mit EGR-3 zu relativen Resonanzwerten von 450, 500 und 550 RU führten (Abb. 27 B). Die Bindungen von p50 an EGR-4 zeigten sich in stabilen Resonanzwerten von 750, 1500 und 1800 RU (Abb. 27 C). Das NFκB-Protein p65 bindet EGR-4 schwächer als p50, da für die Bindung Resonanzwerte von 250, 750 und 1250 RU nachgewiesen wurden (Abb. 27 D). Die von den NFκB/EGR-4Interaktionen hervorgerufenen starken Assoziations- und Dissoziationsresonanzen sind auf die hohen Proteinkonzentrationen zurückzuführen. Abb. 27: Bestimmung der NFκB/EGR-Interaktionen mit der Plasmonresonanz Die Interaktionen von NFκB-Proteinen mit immobilisierten EGR-Proteinen wurden mit der Plasmonresonanz im Biacore-System untersucht. Die NFκB-Proteine p50 und p65 wurden in E. coli exprimiert, aufgereinigt und in unterschiedlichen Konzentrationen zu immobilisierten EGR-Proteinen hinzugegeben. (A, B) Bindungen von p50 und p65 an EGR-3, die Resonanzwerte in Abhängigkeit von der Konzentration hervorriefen. Die dosisabhängigen Interaktionen von immobilisiertem EGR-4 mit p50 ist in (C) und mit p65 in (D) dargestellt. Im Evaluationsprogramm wurden die Resonanzwerte der Interaktionskurven von Kontrollwerten abgezogen. - 70 - Ergebnisse 3.7.4 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit p65 In transienten Transfektionen wurde die kooperative Interaktion von EGR-4 und des NFκBProteins p65 bei der Transkription des humanen Tumornekrosefaktors-α untersucht. Für die Lokalisierung funktioneller Domänen wurden die EGR-4-Deletionsmutanten EGR-4 MI (aa 150487), EGR-4 MII (aa 325-487) EGR-4 MIII (aa 379-487), EGR-4 MV (aa 379-463), EGR-4 MVI (aa 171-325) und EGR-4 MVII (aa 171-325 und 379-463) eingesetzt. Das intakte EGR-4 aktivierte in Kooperation mit p65 die Transkription des TNF-α-Promotors 105-fach, während p65 alleine oder in Kombination mit p50 die Transkription 25- bzw. 67-fach aktivierte (Abb. 28). Die Deletion der N-terminalen 149 (EGR-4 MI) bzw. 324 Aminosäuren (EGR-4 MII) von EGR4 führte bei der Kooperation mit p65 zu einer Reduktion der Aktivität um 60 % bzw. 80 %. Ein zusätzlicher Verlust N-terminaler Aminosäuren (EGR-4 MIII) verstärkte wiederum die Transkription auf das 160-fache. EGR-4 MV, das die Zinkfingerregion besitzt, zeigte eine 130fache Aktivität. Die Deletion der C-terminalen EGR-4-Region (EGR-4 MVI) hatte bei der Kooperation mit p65 keinen Effekt auf die Transkription. Die gemeinsame Deletion der EGR-4Regionen aa 1-170 und aa 326-378 führte in Kombination mit p65 zu einer 273-fachen transkriptionellen Aktivierung. Abb. 28: Funktionale Interaktion von EGR-4 und p65 In Transfektionen mit humanen 293T-Zellen wurden Expressionsvektoren, die für EGR-4 und p65 kodieren, zusammen mit dem Reportergenkonstrukt kotransfiziert. Für die Lokalisierung funktionaler EGR-4-Domänen wurden EGR-4-Deletionsmutanten eingesetzt. Das Reportergenkonstrukt ist unten im Bild dargestellt und besitzt zusätzlich zum TNF-α-Promotorbereich (bp -174 bis -143, 3x) ein Luciferasegen. Die Expressionsvektoren wurden in den dargestellten Kombinationen mit dem Reporterplasmid kotransfiziert. Die Aktivierungswerte repräsentieren Mittelwerte und Standardabweichungen von mindestens drei unabhängigen Experimenten. - 71 - Ergebnisse In der Transfektionsstudie wurde für EGR-4 eine synergistische Kooperation mit dem NFκBProtein p65 bei der Transkription des humanen Tumornekrosefaktors-α nachgewiesen. Mit EGR-4-Deletionsmutanten wurden für EGR-4 funktionale Interaktions-, Aktivierungs- und Repressordomänen lokalisiert. EGR-4 besitzt Aktivierungsdomänen in der Region aa 171-325 und in der Zinkfingerregion (379-463). Die Repressordomänen befinden sich im EGR-4-Protein in den Regionen aa 325-378 und aa 463-487. EGR-4 bindet p65 über die Zinkfingerdomäne, um die Transkription von TNF-α kooperativ zu aktivieren. 3.7.5 EGR/NFκB-Interaktion vermittelnde Aminosäuren In Transfektionsstudien mit EGR-4-Deletionsmutanten und dem NFκB-Protein p65 wurde der Zinkfingerdomäne von EGR-4 eine Beteiligung an der funktionellen Interaktion mit p65 nachgewiesen. Es stellte sich die Frage, ob die Zinkfingerdomäne der EGR-Proteine auch eine direkte Interaktion mit den NFκB-Proteinen eingehen kann. Deshalb wurden lineare Peptide der Zinkfingerregionen von EGR-1 (aa 335-426), EGR-2 (aa 337-426), EGR-3 (aa 272-361) und EGR-4 (aa 376-465) synthetisiert und auf einer Membran immobilisiert. Zusätzlich wurden Peptide der Zinkfingerregion (aa 534-625) des Transkriptionsfaktors Sp1 synthetisiert, der p65 (RelA) mit der Zinkfingerregion bindet (Perkins et al., 1994; Sif and Gilmore, 1994). Die Peptide der EGR- und Sp-1-Zinkfingerregionen wurde mit aufgereinigtem p65 inkubiert und mit spezifischen Antikörpern detektiert. In Abbildung 29 sind die p65-Interaktionen mit den Zinkfingerregionen von EGR-Proteinen und Sp-1 dargestellt. Die EGR-Proteine binden das NFκB-Protein p65 mit drei Domänen, die in je einem der drei Zinkfinger lokalisiert sind. Das NFκB-Protein p65 wurde von EGR-1 im Zinkfinger 1 (aa 347368), im Zinkfinger 2 (aa 374-386) und im Zinkfinger 3 (aa 404-424) gebunden. EGR-2 bindet p65 mit den drei Zinkfingerregionen aa 349-370, aa 376-388 und aa 406-426. Die p65-Bindung wurde bei EGR-3 durch je eine Region im ersten Zinkfinger (aa 284-305), im zweiten Zinkfinger (aa 311-323) und im dritten Zinkfinger (aa 341-361) vermittelt. EGR-4 bindet p65 mit der Region aa 397-409 im ersten Zinkfinger und mit den Regionen aa 415-427 und aa 445-465 des zweiten und dritten Zinkfingers. Das Transkriptionsfaktor Sp-1 bindet p65 ebenfalls an drei Regionen in je einem Zinkfinger (aa 546-567, aa 576-588 und aa 603-625). - 72 - Ergebnisse Die Aminosäuren der EGR-Proteine und Sp-1, die eine Interaktion mit p65 vermittelten, sind in Tabelle 11 dargestellt. Für den Vergleich der Aminosäurebereiche einzelner EGR-Proteine wurden die Strukturen der Zinkfinger mit einbezogen. Die EGR-Zinkfinger 1 und 3 besitzen die Zinkfingerstruktur I, während der Zinkfinger 2 der Struktur II entspricht. Die Sp-1Zinkfingerstrukturen sind dementsprechend zugeordnet worden (Tabelle 11). Abb. 29: EGR/NFκB-Interaktion vermittelnde Aminosäurebereiche Die Interaktion von EGR-Proteinen mit p65 wurde in einer Peptidspotanalyse untersucht. Peptide der Zinkfingerregion von EGR-1 (aa 335-426), EGR-2 (aa 337-426), EGR-3 (aa 272-361), EGR-4 (aa 376-465) und Sp1 (aa 534-625) wurden synthetisiert, auf einer Membran immobilisiert, mit gereinigtem p65 inkubiert und mit spezifischen Antikörpern detektiert. Hervorgehoben sind die Regionen, an die p65 gebunden wurde. - 73 - Ergebnisse Tabelle 11: Vergleich der EGR/p65-Interaktion vermittelnden Aminosäuren Darstellung und Vergleich der Aminosäuren der Zinkfingerregionen von EGR-Proteinen und Sp-1, die für die Interaktionen mit p65 verantwortlich sind (Abb 29). Homologe Aminosäuren sind hervorgehoben. Die EGRZinkfinger (ZF) 1 und 3 besitzen die Struktur I, während der Zinkfinger 2 der Struktur II entspricht. Die Aminosäuren, welche die DNA-Bindung vermitteln, sind grau unterlegt. Protein ZF Sequenz Position (aa) EGR-1 1 (I) RRFSRSDELTRHIRIHTGQKPF 347-368 EGR-2 1 (I) RRFSRSDELTRHIRIHTGHKPF 349-370 EGR-3 1 (I) RRFSRSDELTRHLRIHTGHKPF 284-305 EGR-4 1 (I) RSFARSDELNRHLRIHTGHKPF 389-409 EGR-1 3 (I) KFARSDERKRHTKIHLRQKDK 404-424 EGR-2 3 (I) KFARSDERKRHTKIHLRQKER 406-426 EGR-3 3 (I) KFARSDERKRHAKIHLKQKEK 341-361 EGR-4 3 (I) RFARSDEKKRHSKVHLKQKAR 445-465 Sp1 2 (I) EGR-1 2 (II) MRNFSRSDHLTTH 374-386 EGR-2 2 (II) MRNFSRSDHLTTH 376-388 EGR-3 2 (II) MRSFSRSDHLTTH 311-323 EGR-4 2 (II) LRNFSRSDHLTTH 415-427 Sp1 1 (II) KVYGKTS HLRAHLRWHTGERPF 546-567 Sp1 3 (II) PKRFMRSDHLSKHIKTHQNKKGG 603-625 KRFTRSDELQRHK 576-588 3.7.6 Inhibition der EGR/NFκB-Interaktion Die Bindung des NFκB-Proteins p65 wird bei den EGR-Proteinen durch die Zinkfingerregion vermittelt. Bei einem Vergleich der Interaktionsdomänen innerhalb der EGR-Familie stellte sich heraus, dass die Bindung von p65 durch Argininreste vermittelt werden kann, die ebenfalls für die DNA-Bindung verantwortlich sind (Abb. 3). Um herauszufinden, dass die Argininreste im intakten EGR-4 für die Interaktion mit p65 relevant sind, wurde Peptide der EGR-4-Region aa 451-465 synthetisiert und dessen Einfluß auf die EGR-4/p65-Interaktion in PlasmonresonanzStudien untersucht. Die Interaktion von EGR-4 und p65 kann durch inhibitorische Peptide gehemmt werden (Abb. 30). Die Bindung von p65 an EGR-4 rief relative Resonanzwerte von 540 RU hervor. Durch die Zugabe des inhibitorischen NFκB-Peptids wurden die Resonanzwerte der EGR-4/p65Interaktion auf 340 RU reduziert, so dass dem Peptid ein hemmender Einfluss zugesprochen - 74 - Ergebnisse wird. Die Interaktion von EGR-4 und p65 wurde durch ein Kontrollpeptid nur gering beeinflusst (525 RU). Abb. 30: Einfluss eines inhibitorischen Peptids auf die EGR-4/p65-Interaktion Die Interaktionen von EGR-4 und p65 wurde in Anwesenheit inhibitorischer Peptide mit der PlasmonresonanzStudie an der Biacore 3000 untersucht. 10 µg/ml inhibitorische NFκB-Peptide (EKKRHSKVHLKQKAR) oder Kontrollpeptide (TQPQLSPLGLRSAAA) wurden mit aufgereinigtem p65 inkubiert und zu immobilisiertem EGR-4 hinzugegeben. Als Kontrolle wurde die Interaktion von EGR-4 und p65 ohne Peptide untersucht. Die verschiedenen Kurvenwerte wurden im Evaluationsprogramm von den Kontrollwerten abgezogen. 3.8 Lokalisierung von Bindungsdomänen in NFAT- und NFκB-Proteinen Für die Lokalisierung möglicher Interaktionsdomänen in den Proteinen der NFAT- und NFκBFamilie für die Bindung von EGR-4 wurde die Interaktion von EGR-4 mit Rel-enthaltenden Proteinen in einer Peptidspotanalyse untersucht. Peptide der Rel-Regionen von p50 (aa 41-80), p52 (aa 37-77), p65 (aa 17-57), NFATc (aa 406-462) und NFATp (aa 388-444) wurden synthetisiert, auf einer Membran immobilisiert und mit EGR-4-Proteinen inkubiert. Die NFAT- und NFκB-Proteine binden EGR-4 mit der Rel-Domäne (Abb. 31). EGR-4 bindet an die NFκB-Proteine in den Rel-Regionen aa 41-71 (p50), aa 37-67 (p52) und aa 18-48 (p65). An NFATc und NFATp bindet EGR-4 in den Regionen aa 423-454 und aa 406-434. EGR-4 wurde an Peptide gebunden, deren Aminosäuresequenzen innerhalb der NFAT- und NFκB-Proteine homolog sind. - 75 - Ergebnisse Abb. 31: Domänen von NFAT- und NFκB-Proteinen für die Interaktion mit EGR-4 Für die Lokalisierung von Domänen in NFAT- und NFκB-Proteinen, die für die Interaktion mit EGR-4-Proteinen verantwortlich sind, wurde eine Peptidspotanalyse durchgeführt. Peptide der homologen Rel-Domäne von p50 (aa 41-80), p52 (aa 37-77), p65 (aa 17-57), NFATc (aa 406-462) und NFATp (aa 388-444) wurden synthetisiert und auf einer Membran immobilisiert. Die Membran wurde mit gereinigtem EGR-4 inkubiert und mit spezifischen Antikörpern detektiert. Hervorgehoben sind Peptidsequenzen, die EGR-4-Proteine binden. Bei einem Homologievergleich von NFAT- und NFκB-Proteinen konnte ein potentielles Bindungsmotiv der Sequenz xGxYx3ExQPKx4Rx2Yx2EGxSxGx für die Bindung von EGR-4 identifiziert werden (Abb. 31 und Tabelle 12). Das potentielle Bindungsmotiv wurde für p50 in der Region aa 42-68, für p52 und p65 in den Regionen aa 37-63 und aa 19-45 lokalisiert. NFATProteine besitzen das potentielle Motiv für die EGR-4-Bindung in den Regionen aa 423-448 (NFATc) und aa 406-431 (NFATp). Tabelle 12: Bindungsmotive in NFAT und NFκB-Proteinen für die Interaktion mit EGR-4 Darstellung und Vergleich der Rel-Domänen von NFAT- und NFκB-Proteinen, die eine Bindung an EGR-4 vermitteln. Homologe Aminosäuren von p50, p52, p65, NFATc und NFATp, die bei der Interaktion mit EGR-4 eine Rolle spielen, sind hervorgehoben. Protein Sequenz Position (aa) p50 GPYLQILE QPKQRGFRFRYVCEGPSHGGLPG 42-72 p52 GPYLVIVE QPKQRGFRFRYGCEGPSHGGLPG 37-67 p65 GPYVEIIE QPKQRGMRFRYKCEGRSAGSIPG 19-49 NFATc GPYELRIEVQPKSHH RAHYETEG SRGAVKA 423-452 NFATp GSYELRIEVQPKPHH RAHYETEG SRGAVKA 406-435 Konsensus G-Y----E-QPK----R--Y--EG-S-G---- - 76 - Diskussion 4 Diskussion Die Mitglieder der EGR-Familie werden durch viele verschiedene Stimuli in diversen Zellen des Immunsystems exprimiert und sind an der induzierbaren Genexpression während der Immunantwort beteiligt. Bei der Initiierung der inflammatorischen Antwort aktivieren EGRProteine das Expressionsprogramm pro- und antiinflammatorischer Schlüsselzytokine im Zusammenspiel mit anderen regulatorischen Transkriptionsfaktoren. Die Proteine der EGRFamilie interagieren mit den Transkriptionsfaktoren der NFAT- und NFκB-Familie. In dieser Arbeit wurden die Interaktionen der EGR-Proteine mit den NFAT- und NFκB-Proteinen charakterisiert. 4.1 Expression der EGR-Proteine Die EGR-Proteine sind Transkriptionsfaktoren, die von den „immediate early“-Genen kodiert werden und in vielen Zellen und Geweben exprimiert werden. Diverse Stimuli, die bei Wachstums-, Differenzierungs- und Immunantwortprozessen auftreten, aktivieren die Expression der EGR-Proteine innerhalb kurzer Zeit (Sukhatme et al., 1988; Milbrandt, 1987). So können natives EGR-3 und EGR-4 nach 2 Stunden in stimulierten Jurkat-T-Zellen nachgewiesen werden (Abb. 4). Für die Charakterisierung der Interaktion von EGR- und NFAT-Proteinen wurden EGR-3 und EGR-4 rekombinant in Sf9-Insektenzellen exprimiert. Rekombinantes EGR-4 zeigt nach einer SDS-PAGE im Westernblot eine Mobilität von 69 kDa (Abb. 4 A). Natives EGR-4 besitzt dieselbe molekulare Größe von 69 kDa (Abb. 4). Die EGR-Proteine besitzen verschiedene Konformationen in Abhängigkeit von der Salzkonzentration (Abb. 4 und 8). Im EGR-4-Zellextrakt treten bei hohen Salzkonzentrationen neben einem geringen Anteil der 69 kDa-Konformation hauptsächlich Konformationen höherer Mobilität auf. Der Anteil der 69 kDa-Konformation nimmt bei einer Erniedrigung des Salzgehalt im Zellextrakt zu, während Konformationen höherer Mobilitäten nicht mehr nachgewiesen werden können (Abb. 4 B). Wird die Salzkonzentration von 120 mM auf 420 mM Natriumchlorid wieder erhöht, treten im Zellextrakt wiederum mehrere Konformationen auf. Dies zeigt, dass die Konformationsänderungen reversibel sind (Abb. 4 - 77 - Diskussion B). Die auftretenden EGR-4-Konformationen sind spezifisch, da die EGR-4-Varianten aufgereinigt werden können (Abb. 8). Das Phänomen der Konformationsänderung ist sowohl bei rekombinantem EGR-4 als auch bei nativem EGR-4 zu beobachten (Abb. 4 C). Im Zellextrakt von Jurkat-T-Zellen treten bei hohen Salzkonzentrationen EGR-4-Konformationen höherer Mobilität auf, während die 69 kDa-Konformation nicht oder nur schwach auftritt. EGR-Proteine sind Proteine, die in der Zelle in verschiedenen Konformationen auftreten, die in vitro durch die Abhängigkeit von der Salzkonzentration charakterisiert werden können. Von welchen Faktoren die EGR-Konformationen in der Zelle abhängig sind, ist momentan ungeklärt. Für EGR-1 und EGR-2 wurden bereits verschiedene Proteinvarianten beschrieben (Lemaire et al., 1990; Day et al. 1990; Mack et al., 1992). Die EGR-1- und EGR-2-Varianten besitzen unterschiedliche Stabilitäten, Phosporylierungsstufen und Kerntranslokationen (Lemaire et al., 1990; Day et al., 1990; Herdegen et al., 1993). Ob die hier vorgestellten EGR-3- und EGR-4-Varianten ebenfalls unterschiedlich phosphoryliert und in den Kern transloziert werden, ist bisher nicht bekannt. Das Auftreten unterschiedlicher Konformationen könnte der Regulation der EGR-Proteine dienen. Die Funktionen von EGR-Proteinen sind von der Anwesenheit bestimmter Ionen abhängig und könnten somit durch zelluläre Komponenten oder regulatorische Proteine kontrolliert werden, die das zelluläre Redoxstadium durch das Zurückhalten von Ionen verändern und dadurch möglicherweise eine EGR-Variante inaktivieren (Cao et al., 1990; Huang and Adamson, 1993). EGR-4 zeigt bei Salzkonzentrationen von 60-220 mM eine zusätzliche Bande, die einem molekularem Gewicht von 140 kDa entspricht (Abb. 4 B). Bei der Erhöhung des Salzgehaltes von 60 mM auf 420 mM Natriumchlorid tritt diese 140 kDa-Bande nicht mehr auf. Die Intensität der 140 kDa-Bande nimmt bei Abnahme des Salzgehaltes zu und könnte ein Homodimer der EGR-4-Proteine repräsentieren. Es ist sehr wahrscheinlich, dass EGRProteine die Fähigkeit zur Homo- bzw. Heterodimerisierung besitzen, da viele Transkriptionsfaktoren Heterodimere mit anderen Transkriptionsfaktoren der eigenen Familie und kooperative Multimere mit Mitgliedern anderer Familien bilden können (Manning and Rao, 1999). Homo- und Heterodimere werden oft für die Ausübung regulatorischer Funktionen gebildet, z. B. zwischen den NFκB-Proteinen und anderen Rel-enthaltenden Proteinen (Baeuerle and Henkel, 1994). NFAT-Proteine gehen stabile und kooperative Trimere mit Dimeren der AP-1-Familie (Fos und Jun) ein (Jain et al., 1992). Die Fähigkeit - 78 - Diskussion der EGR-Proteine, Homo- und Heterodimer zu bilden, liefert einen wichtigen Beitrag zur Aufklärung der regulatorischen Funktion dieser Proteine. Rekombinantes und natives EGR-3 zeigt im SDS-PAGE und Westernblot eine molekulare Größe von 59 kDa (Abb. 4 D). Im Zellextrakt mit rekombinantem und nativem EGR-3 treten ebenfalls mehrere Konformationen auf, die vom Salzgehalt abhängig sind (Abb. 8). Die Konformationen von EGR-3 unterscheiden sich von den EGR-4-Konformationen. Der Unterschied zwischen EGR-3 und EGR-4 bei der Konformationsbildung wird besonders bei der Untersuchung von Zellextrakten stimulierter Jurkat-T-Zellen deutlich. EGR-3 zeigt im Zellextrakt mit hohen Salzkonzentrationen öfter die Hauptkonformation (59 kDa) als EGR-4, deren Hauptkonformation (69 kDa) selten bei hohen Salzkonzentrationen nachgewiesen werden kann. Dies deutet daraufhin, dass EGR-3 und EGR-4 möglicherweise verschiedene Proteinzustände bzw. Konformationen einnehmen, die sich außerdem in der Stabilität unterscheiden könnten. Der Unterschied bei der Konformationsbildung könnte durch verschiedene Dissoziationsraten von EGR-3 und EGR-4 erklärt werden. EGR-4 besitzt in der Zelle eine schnellere Dissoziationsrate als die anderen EGR-Proteine, die von einer basischen Region N-terminal des Zinkfingers vermittelt werden könnte, da sie nicht in EGR-1, EGR-2, EGR-3 konserviert ist (Swirnoff and Milbrandt, 1995). 4.2 Interaktion von EGR- und NFAT-Proteinen Die Mehrzahl der Transkriptionsfaktoren binden sequenzspezifisch an regulatorische DNASequenzen und üben ihre Funktion in Kooperation mit verschiedenen anderen Faktoren aus. In dieser Arbeit konnte gezeigt werden, dass EGR-Proteine mit den NFAT-Proteinen physikalische Interaktionen eingehen. Die Interaktionen von EGR-3 und EGR-4 mit NFATc und NFATp wurden in Proteinbindungsassays nachgewiesen (Abb. 6-10) und mit Plasmonresonanz-Studien näher charakterisiert (Abb. 11-13). EGR-3 und EGR-4 interagieren mit den NFAT-Proteinen NFATc und NFATp. Sowohl rekombinantes als auch natives EGR-3 und EGR-4 bindet an NFAT-Proteine, wenn NFATc bzw. NFATp auf einer Matrix immobilisiert werden (Abb. 6-7). Die EGR/NFAT-Interaktion findet auch in umgekehrter Anordnung statt, bei der EGR-Proteine an einer Matrix - 79 - Diskussion immobilisiert werden (Abb. 8-9). Rekombinante und native NFAT-Proteine binden sowohl EGR-3 als auch EGR-4 und gehen stabile Proteinkomplexe mit den EGR-Proteinen ein (Abb. 9). Die EGR- und NFAT-Proteine bilden stabile Heterodimere. EGR-Proteine regulieren die Transaktivierung verschiedener Gene, indem sie kooperative Interaktionen mit anderen Transkriptionsfaktoren eingehen (Lee et al., 1996; Cogswell et al., 1997; Li-Weber et al., 1999; Tremblay and Drouin, 1999; Chapman and Perkins, 2000; Silverman et al., 1998; Liu et al., 2001). Die Transkription von IL-2, TNF-α und ICAM-1 wird durch synergistische Kooperationen von EGR- und NFAT-Proteinen reguliert (Decker et al., 1998). Hier wurde gezeigt, dass die EGR-Proteine eine direkte Interaktion mit den NFAT-Proteinen eingehen und stabile Komplexe bilden. Die Fähigkeit zur Heterodimerisierung könnte dazu dienen, die EGR-Funktion bei der Regulation verschiedener Gene zu kontrollieren. 4.2.1 Salzeffekt bei EGR-Interaktionen Die EGR-Proteine besitzen in Abhängigkeit von der Salzkonzentration verschiedene Konformationen. Da die EGR-Proteine stabile Komplexe mit den NFAT-Proteinen eingehen, war es von Interesse, den Einfluss der Salzkonzentration auf die EGR/NFAT-Interaktion zu untersuchen. Aus diesem Grund wurde die Interaktion von EGR-4 mit den NFAT-Proteinen bei unterschiedlichen Salzkonzentrationen untersucht (Abb. 10). EGR-4 bindet NFAT-Proteine nur, wenn EGR-4 in einer bestimmten Konformation vorliegt (Abb. 10 C und D). Diese Konformation tritt nur bei niedrigen Salzkonzentrationen auf und besitzt nach einer SDS-PAGE im Westernblot eine Mobilität von 69 kDa. EGR-4 bindet die NFAT-Proteine bei Konzentrationen von 120-220 mM Natriumchlorid, während bei hohen Konzentrationen (320-420 mM) keine Interaktion nachgewiesen werden konnte. Dies deutet daraufhin, dass die Konformationen, die von EGR-4 bei hohen Salzkonzentrationen gebildet werden, nicht mit den NFAT-Proteinen interagieren können. Das Ergebnis unterstützt die These, dass die Existenz mehrere Konformationen der Regulation der EGR-Proteinen dienen könnte. Es ist möglich, dass die Konformationen und - 80 - Diskussion damit die Funktionen der EGR-Proteine durch Änderungen im zellulären Milieu kontrolliert werden. Die Affinität von EGR-Proteinen zur DNA und somit die EGR-Aktivität wird z.B. durch Änderungen der Redoxstufenpotentiale kontrolliert (Cao et al., 1990). In der Zelle kommen Na+- und Cl--Ionen in Konzentrationen von 120-140 mM vor und veranlassen EGRProteine interaktionsfähige Konformation einzunehmen. Sowohl die Bildung als auch die Interaktionsfähigkeit der Konformationen könnten somit durch Veränderungen des zellulären Salz- bzw. Ionenhaushaltes in der Zelle kontrolliert werden. Einen Hinweis auf eine mögliche Regulation der Interaktionsfähigkeit verschiedener Konformationen lieferte die Untersuchung der Interaktion von EGR-4 und NFATc bei einem Salzgehalt von 220 mM Natriumchlorid. Hier tritt EGR-4 in mehreren Konformationen einschließlich der Konformation auf, die im SDS-PAGE und Westernblot eine Mobilität von 69 kDa besitzt. Diese kann aber NFATProteine nicht binden. Dies Die Aktivität der EGR-Proteine könnte durch zelluläre Komponenten oder regulatorische Proteine kontrolliert werden, welche die Konzentration zellulärer Ionen verändern können (Huang and Adamson, 1993). 4.2.2 Charakterisierung der EGR/NFAT-Interaktionen mit der Plasmonresonanz Für die weitere Charakterisierung der Interaktionen von EGR-3 und EGR-4 mit den NFATProteinen wurden Plasmonresonanz-Studien durchgeführt, um die Bindungen in Echtzeit verfolgen zu können. In der Plasmonresonanz-Studie kann die EGR/NFAT-Interaktion mit einer Assoziations- und einer Dissoziationsphase dargestellt werden (Abb. 11). Die Bindung von EGR- und NFAT-Proteinen führt zur Bildung stabiler Proteinkomplexe. Die Assoziationsphasen zeigen eine stetige Zunahme der Bindung von NFAT- an EGR-Proteinen, die in den seltensten Fällen gesättigt wurde. Die Bindungskurven fallen in den Dissoziationsphasen zunächst relativ stark ab, stabilisieren sich aber im Verlauf der Zeit. Ein weiterer Hinweis auf die Stabilität der Bindung ist die Verwendung einer zweifachen Elution. In anderen Interaktionsstudien mit Plasmonresonanzen reicht eine einfache und kürzere Elution aus (Hilyard et al. 1994). Die Verwendung zu hoher NFAT-Proteinkonzentrationen führt zu einer starken Interaktion, so dass EGR- und NFAT-Proteine nicht mehr getrennt werden konnten. Im Weiteren wurde festgestellt, dass die Bindungskurven erst nach 4 Stunden auf den Basisresonanzwert zurückgehen, der von den immobilisierten EGRProteinen alleine hervorgerufen wird (nicht gezeigt). Die Plasmonresonanz-Studien im - 81 - Diskussion Biacore-System demonstrieren starke Komplexbildungen zwischen EGR- und NFATProteinen. Die EGR-Proteine binden sowohl NFATc als auch NFATp mit hoher Affinität und bilden stabile Komplexe (Abb. 12-13). Die Interaktion der NFAT-Proteine an EGR-3 und EGR-4 ist abhängig von der Konzentration der eingesetzten Proteine. Die Affinitäten der EGR-Proteine zu NFATc und NFATp sind vergleichbar. NFAT-Proteine rufen zwar bei der Bindung an EGR-Proteine unterschiedlich starke Assoziationsphasen hervor, stabilisieren sich jedoch in der späten Dissoziationsphase. In dieser Arbeit konnte zum ersten Mal gezeigt werden, dass die Untersuchung von Interaktionen zwischen den EGR- und NFAT-Proteinen mit der Plasmonresonanz möglich ist. Expression und Funktion der EGR-Proteine während der Immunantwort sind bisher ausführlich beschrieben worden (Christy et al., 1988; Changelian et al., 1989; Chavrier et al., 1989; Day et al., 1990; Rangnekar et el., 1990; Sukhatme, 1990; Müller et al., 1991; Sakamoto et al., 1991; Holst et al., 1993; Gashler and Sukhatme, 1995). Wenig ist jedoch über den molekularen Mechanismus der Regulation bei der Interaktion mit anderen Transkriptionsfaktoren bekannt. Mit der Plasmonresonanz im Biacore-System können Affinitäten von EGR-Proteinen zu anderen Transkriptionsfaktoren untersucht werden und damit einen wichtigen Beitrag zur Aufklärung des Regulationsmechanismus dieser Proteine liefern. 4.3 Lokalisierung von EGR-Interaktionsdomänen für die NFAT-Interaktion Nachdem die direkten Interaktionen zwischen EGR-3 bzw. EGR-4 und den Proteinen der NFAT-Familie nachgewiesen wurde, stellte sich die Frage, welche Domänen des EGRProteins für die Interaktionen verantwortlich sind. Zu diesem Zweck wurden Deletionsmutanten von EGR-3 und EGR-4 rekombinant exprimiert und sowohl in Proteinbindungsassays als auch in Plasmonresonanz-Studien eingesetzt (Abb. 14-19). Die Interaktion von EGR-3 und NFAT-Proteinen wird durch zwei EGR-3-Domänen vermittelt (Abb.15-16). Die EGR-3-Interaktionsdomänen sind in der Region aa 98-168 und aa - 82 - Diskussion 261-387 lokalisiert, von denen letztere auch die Zinkfingermotive enthält. In Plasmonresonanz-Studien konnten diese zwei Interaktionsdomänen bestätigt werden (Abb. 16). EGR-3 bindet NFAT-Proteine an die Regionen mit unterschiedlicher Affinität. Die erste Interaktionsdomäne (aa 98-168) bindet an die NFAT-Proteine stärker als die zweite Domäne (aa 261-387). Das intakte EGR-3 bindet stärker als die beiden Deletionsmutanten und zeigt, dass beide Domänen für die Interaktion mit den NFAT-Proteinen wichtig sind (Abb. 15 ). EGR-4 bindet sowohl NFATc als auch NFATp mit zwei Domänen (Abb. 18-19). Die EGR-4Interaktionsdomänen liegen im N-terminalen Bereich aa 1-158 und in der mittleren Region aa 158-325 (Abb. 18). Die Untersuchung mit weiteren Deletionsmutanten zeigte, dass es sich nicht um eine, sondern um zwei Interaktionsdomänen handelt. Dies deutet daraufhin, dass die Aminosäuresequenz der beiden Interaktionsdomänen für die Interaktion mit NFAT-Proteinen wichtig ist. EGR-4 bindet NFAT-Proteine an die Interaktionsdomäne aa 1-158 stärker als an der Domäne aa 158-325 (Abb. 19). EGR-3 und EGR-4 binden NFAT-Proteine mit zwei Domänen. Die Interaktionsdomänen von EGR-3 bzw. EGR-4 besitzen unterschiedliche Affinitäten zu den NFAT-Proteinen und tragen zu einer vollständigen NFAT-Bindung bei. Die Interaktionsdomänen von EGR-3 weisen zu denen von EGR-4 keine eindeutige Homologie auf. Bisher wurden konservierte geladene Regionen beschrieben, die einen besonders hohen Anteil positiv oder negativ geladener Aminosäurereste besitzen (Lemaire et al., 1988; Crosby et al., 1992; Vesque and Charnay, 1992; Russo et al., 1993; Gashler et al., 1993; Carman and Monroe, 1995; Russo et al., 1995; Svaren et al., 1996). Alle EGR-Proteine enthalten im N-terminalen Proteinbereich Regionen, die vorwiegend aus den Aminosäureresten Prolin, Alanin, Serin und Threonin bestehen. EGRProteine binden die NFAT-Proteine wahrscheinlich nicht über ein homologes Bindungsmotiv, sondern über Regionen mit konservierten Aminosäurepositionen oder Ladungsverteilungen. 4.3.3 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit NFATp Da die Kooperation der EGR- und NFAT-Proteine bei der Regulation verschiedener Gene eine wichtige Rolle spielt, sollte die Funktionalität der lokalisierten Interaktionsdomänen in Transfektionsstudien untersucht werden. Deshalb wurde die Interaktion von EGR-4 und - 83 - Diskussion NFATp bei der Regulation des Interleukin-2-Promoters mit EGR-4-Deletionsmutanten untersucht (Abb. 20). EGR-4 und NFATp regulieren die IL-2-Transkription durch eine synergistische Kooperation, bei der NFATp in der Region aa 150-325 von EGR-4 gebunden wird (Abb. 20). Weiterhin konnte gezeigt werden, dass EGR-4 ein Multidomänenprotein ist. EGR-4 besitzt eine Interaktions- und Aktivierungsdomäne in der Region 150-325, welche die Bindung von NFAT-Proteinen vermittelt. Die Deletion der EGR-4-Region aa 325-377 vermindert die kooperative Aktivität von EGR-4 und NFATp. Dies deutet daraufhin, dass in der Region aa 325-377 zusätzlich eine Interaktionsdomäne oder eine Repressordomäne vorhanden sein könnte. Da in Proteinbindungsassays die EGR-4-Region aa 325-487 keine Interaktion mit NFAT-Proteinen vermitteln konnte (Abb. 18 und 19), handelt es sich bei der Region aa 325377 wahrscheinlich um eine Repressordomäne. Für EGR-1 und EGR-2 wurden bereits Aktivierungs- und Repressordomänen beschrieben, die in der Serin- bzw. Prolin-reichen EGR-Region lokalisiert sind (Russo et al., 1993; Gashler et al., 1993; Carman and Monroe, 1995; Vesque and Charney, 1992; Russo et al., 1995; Svaren et al, 1996). Die Aktivierungsdomäne von EGR-4 in der Region aa 150-325 ist reich an Prolin- und Glutaminresten. Dies wurde auch bei der Aktivierungsdomäne EGR-1 beobachtet, die große Homologie zu der N-terminale Aktivierungsdomäne von EGR-2 besitzt, wenn man die Verteilung der geladenen Aminosäuren mit einbezieht (Russo et al., 1993; Vesque and Charney, 1992). EGR-1 besitzt eine Repressordomäne N-terminal vor dem Zinkfinger, die Interaktionen mit den zellulären Inhibitoren NAB1 und NAB2 vermitteln (Russo et al., 1993; Russo et al., 1995; Svaren et al, 1996). Die Deletion der C-terminalen Region aa 463-487 von EGR-4 hat keinen Einfluss auf die Transaktivierung von IL-2. Eine homologe Region von EGR-1 wurde beschrieben, die ebenfalls keine transkriptionelle Aktivierung zeigte (Carman and Monroe, 1995). 4.3.4 EGR-4/NFAT-Interaktion vermittelnde Aminosäuren Die Domänen, die für eine direkte Interaktion mit den NFAT-Proteinen verantwortlich sind, spielen auch bei der funktionalen Kooperation von EGR- und NFAT-Proteinen eine Rolle. In Transfektionsstudien wurden im Bereich der EGR-4-Region aa 150-325 sowohl direkte als auch funktionale Interaktionen mit NFATp nachgewiesen (Abb. 18 und 20). Eine genauere - 84 - Diskussion Charakterisierung der EGR-4-Interaktionsdomäne mit der Peptidspotanalyse zeigte, dass EGR-4 in dieser Domäne die NFAT-Proteine an mehrere Aminosäurebereiche bindet (Abb. 21, Tabelle 9). Sowohl NFATc als auch NFATp binden an dieselben linearen Aminosäurebereiche, aber mit unterschiedlicher Affinität. EGR-4 bindet NFATc hauptsächlich in der Region 302-328, während NFATp stärker an die Region aa 175-201 gebunden wird (Abb. 21). Innerhalb einer Region binden die einzelnen NFAT-Proteine wiederum mit unterschiedlicher Affinität an die jeweiligen Aminosäuren. So bindet z. B. NFATc in der Region 302-328 besonders stark an die Aminosäuren 306-320, während NFATp in der Region aa 175-201 stark an die Aminosäuren 183-197 bindet. Um die relevanten Aminosäuren zu identifizieren, die den Kontakt zwischen EGR-4 und den NFATProteinen vermitteln, wurden einzelne Aminosäuren ausgetauscht (Abb. 22). In den Aminosäurebereichen sind vorwiegend positiv geladene Aminosäuren für die Interaktion mit den NFAT-Proteinen verantwortlich. Der Austausch für die Bindung relevanter Aminosäuren führt zu einem Verlust der Bindung von NFAT-Proteinen an EGR-4. EGR-4 bindet NFATc und NFATp hauptsächlich mit den Aminosäuren Lysin, Leucin, Prolin und Arginin (Abb. 22). Bei einem Homologievergleich der Aminosäurenbereiche, welche die Bindung vermitteln, ist ein potentielles Bindungsmotiv der Sequenz xPx3P(L/x)GLRx erkennbar. Die EGR4/NFATc-Interaktion konnte mit einem inhibitorischen Peptid, welches die Sequenz des Bindungsmotives besitzt, zumindest reduziert werden (Abb. 23). Die Interaktion konnte nicht vollständig gehemmt werden, da das intakte EGR-4 mehrere Domänen für die Bindung von NFAT-Proteinen besitzt. Da alle EGR-Proteine NFATc und NFATp binden, ist es wahrscheinlich, dass auch EGR-1, EGR-2 und EGR-3 die NFAT-Proteine über ein ähnliches Bindungsmotiv binden. Bei einem Homologievergleich ist das potentielle Bindungsmotiv Px3PGL in EGR-1 (aa 269-277), EGR-2 (aa 213-219) und EGR-3 (aa 163-169) gefunden worden. Ähnliche Motive sind in weiterhin in EGR-1 (aa 258-266), EGR-2 (aa 312-318) und EGR-3 (aa 146-153) gefunden worden, die als potentielle Bindungmotive in Frage kommen. EGR-Proteine besitzen konservierte Regionen, die einen besonders hohen Anteil positiv oder negativ geladener Aminosäurereste besitzen und die oft Aktivierungsdomänen darstellen (Lemaire et al., 1988; Crosby et al., 1992; Vesque and Charnay, 1992; Russo et al., 1993; Gashler et al., 1993; Carman and Monroe, 1995). Es ist deshalb sehr wahrscheinlich, dass die Interaktionen von EGR-Proteinen mit den NFAT-Proteinen durch positiv geladene Aminosäuren vermittelt werden. - 85 - Diskussion 4.4 Interaktion von EGR- und NFκB-Proteinen Die NFκB- und NFAT-Proteine besitzen hoch konservierte DNA-Bindungsdomänen (RelDomänen). Die „Rel-similarity“-Domänen (RSD) der NFAT-Proteine zeigen bis zu 70 % Homologie zu den Rel-Domänen der NFκB-Proteine (Jain et al., 1995). Bei den NFκBProteinen vermitteln die Rel-Domänen neben der DNA-Bindung die Fähigkeit zur Homo/Heterodimerisierung (Bours et al., 1992 a und b). Da die EGR-Proteine Interaktionen mit den NFAT-Proteinen eingehen, die homologe Regionen zu den NFκB-Proteinen besitzen, wurde die Interaktion von EGR- und NFκB-Proteinen untersucht (Abb. 24-30). EGR-3 und EGR-4 binden die NFκB-Proteine p50 und p65. Die Interaktion von rekombinanten und nativen EGR-Proteinen findet sowohl mit p50 oder als auch mit p65 statt, wenn die NFκB-Proteine immobilisiert werden (Abb. 24 und 25). Die Umkehrung der Interaktion, bei der die EGR-Proteine immobilisiert werden, bestätigt die Interaktionsfähigkeit von EGR-Proteinen an NFκB-Proteinen (Abb. 26). Hier binden p50 und p65 in rekombinanter und nativer Form an EGR-3 und EGR-4. Die EGR-Proteine binden die NFκB-Proteine p50 und p65 mit unterschiedlichr Affinität. EGR-3 bindet p50 mit einer geringeren Affinität als p65 (Abb. 27 A). EGR-4 hingegen bindet p65 stärker als p50 (Abb. 27 B). Die Bindungen von NFκB-Proteinen sowohl an EGR-3 als auch an EGR-4 sind konzentrationsabhängig. Die physikalische Interaktion von EGR- und NFκB-Proteinen ist wahrscheinlich für die synergistische Kooperation bei der Transaktivierung verschiedener inflammatorischer Gene verantwortlich. Zusätzliche Faktoren, die mit den EGR-Proteine interagieren, wurden bereits identifiziert. Die NFκB-Untereinheit RelA interagiert mit EGR-Proteinen bei der Transkription des NFκB-1-Gens (Chapman and Perkins, 2000; Cogswell et al., 1997). Weiterhin regulieren das Homeobox-Protein PTx-1 und der steroidogene Faktor 1 (SF-1) zusammen mit EGR-1 die Expression des Luteinisierenden Hormons LH-β (Lee et al., 1996; Tremblay and Drouin, 1999). Das „cAMP-respone element binding protein-binding“-Protein (CBP/300) bindet EGR-Proteine und induziert die Genexpression der Lipogenase (Silverman et al., 1998). Der Tumorsuppressor p53 bildet ebenfalls einen physikalischen Komplex mit EGR-1 (Liu et al., 2001). - 86 - Diskussion 4.4.1 Funktionale Domänen im EGR-4-Protein bei der Interaktion mit p65 In transienten Transfektionen wurde die Funktionalität der EGR/NFκB-Interaktion am Promotor des humanen Tumornekrosefaktor-α untersucht und EGR-4 mit Deletionsmutanten als Multidomänenprotein charakterisiert. (Abb. 28). EGR-4 kooperiert mit den NFκB-Proteinen und reguliert die Transkription von TNF-α in synergistischer Weise. EGR-4 verstärkt mit p65 die Transkription von TNF-α 105-fach, während p65 alleine oder in Kombination mit p50 die Transkription 25- bzw. 67-fach aktivierte (Abb. 28). Die Verwendung der Deletionsmutanten charakterisiert EGR-4 als ein Multidomänenprotein, das drei Aktivierungs- und zwei Repressordomänen besitzt. Eine Aktivierungsdomäne ist in der Zinkfingerregion aa 379-463 lokalisiert. Die Zinkfingerregion ist für die zentrale Rolle von EGR-4 wichtig, da sie die DNA-Bindung vermittelt. EGR-4 besitzt zwei zusätzliche Aktivierungsdomänen in der N-terminalen Region (aa 1-150) und in der mittleren Region (aa 171-325). Neben der Zinkfingerdomäne befinden sich direkt Nterminal bzw. C-terminal die Repressordomänen der Regionen aa 326-378 und aa 464-487. Eine N-terminale Repressordomäne wurde bereits für EGR-1 beschrieben, die Bindung mit dem Repressorprotein NAB1 und NAB2 vermittelt (Russo et al., 1995; Svaren et al., 1996). Die funktionale Interaktionsdomäne von EGR-4 für die p65-Bindung ist die Zinkfingerregion, die schon als DNA-bindende und Aktivierungsdomäne charakterisiert wurde. In früheren Untersuchungen wurde gezeigt, dass EGR-1 und das verwandte Zinkfingerprotein Sp-1 mit ihren Zinkfingerregionen p65 (RelA) binden (Sif and Gilmore, 1994; Chapman and Perkins, 2000). Es ist wahrscheinlich, dass eine Interaktion der EGR/NFκB-Komplexe mit zusätzlichen Faktoren zur Bildung eines Multiproteinkomplexes führt. EGR-1 und p65 z.B. interagieren mit den Basalfaktoren CBP/p300 (Gerritsen et al., 1997; Zhong et al., 1998). Das NFκBProtein p65 interagiert weiterhin mit den „TATA-box“-assoziierten Faktoren (TAF) und der Histonacetylase (Yamit-Hezi et al., 2000; Zhong et al., 2002). Diese Proteine sind wahrscheinlich ein Teil eines spezifischen regulatorischen Promotorkomplexes, der die Transkription kontrolliert und der mit dem basalen Transkriptionskomplex verbunden ist. - 87 - Diskussion 4.4.2 EGR/NFκB-Interaktion vermittelnde Aminosäuren EGR-4 bindet das NFκB-Protein p65 mit der Zinkfingerdomäne und kann durch diese Interaktion die Transkription von TNF-α regulieren (Abb. 28). Für die Lokalisierung Bindungs-relevanter Aminosäuren wurden Peptide der Zinkfingerregionen von EGR-1 (aa 335-426), EGR-2 (aa 337-426), EGR-3 (aa 272-361) und EGR-4 (aa 376-465) in Peptidspotanalysen eingesetzt (Abb. 29, Tabelle11). Zusätzlich wurden Peptide der Zinkfingerregion (aa 534-625) des transkriptionellen Zinkfingerproteins Sp1 synthetisiert, von dem bekannt ist, dass es p65 (RelA) mit der Zinkfingerregion bindet (Perkins et al., 1994; Sif and Gilmore, 1994). EGR-Proteine binden p65 mit drei konservierten Domänen, die in je einem der drei Zinkfinger lokalisiert sind. Die Bindung von p65 findet vor allem in einem linearen Aminosäurebereich im ersten Zinkfinger und einem langen Aminosäurebereich im dritten Zinkfinger statt. Bei einem Homologievergleich wird ein potentielle Motiv xFxRSDx3RHx für die Bindung von p65 erkennbar. Im ersten und dritten Zinkfinger wird p65 von einem Aminosäurebereich gebunden, das dieses potentielle Bindungsmotiv besitzt. Im zweiten Zinkfinger, der p65 schwächer bindet, fehlt dem Bindungsmotiv ein Argininrest. Betrachtet man die p65-Bindung genauer, so wird erkennbar, dass p65 im ersten Zinkfinger unterschiedlich stark gebunden wird. Die p65-Interaktion wird stark durch das Motiv xRHx und schwächer durch das Motiv xFxRSDx vermittelt. Dies bestärkt die Annahme, dass der Argininrest für die die p65-Bindung wichtig ist. Die Relevanz des Argininrestes für die EGRNFκB-Interaktion wurde mit einem inhibitorischen Peptid untersucht (Abb. 30). Das inhibitorische Peptid, welches das Motiv xRHx besitzt, reduziert die Interaktion von EGR-4 und p65. EGR-Proteine binden mit der Zinkfingerdomäne sowohl an die DNA als auch an das NFκBProtein p65 (Abb. 3 und 32). Die an der DNA-Bindung beteiligten Aminosäurereste sind innerhalb der EGR-Proteine konserviert (Gashler and Sukhatme, 1995; Swirnoff and Milbrandt, 1995). Je zwei Argininreste der ersten und dritten Zinkfinger bzw. ein Argininund ein Histidinrest des zweiten Fingers bilden über Wasserstoffbrückenbindungen direkte Kontakte zu je zwei Guaninen des Nukleotidtripletts aus (Pavletich and Pabo, 1991). Diese Argininreste spielen ebenfalls bei der Bindung von p65 eine Rolle. Wie die gemeinsame Bindung von DNA und p65 stattfinden kann, muß noch geklärt werden. - 88 - Diskussion 4.5 Lokalisierung von Bindungsmotiven in NFAT- und NFκB-Proteinen Die NFκB- und NFAT-Proteine sind in den stark konservierten DNA-Bindungsdomänen (Rel- und Rel-similarity domain) homolog. Da sowohl NFAT- als auch NFκB-Proteine an EGR-Proteine binden, stellte sich die Frage, ob die EGR-Bindung über diese hochkonservierten Domänen stattfindet. Deshalb wurden Peptide der Rel-Regionen von p50, p52, p65, NFATc und NFATp synthetisiert und die Bindung an EGR-4 untersucht. EGR-4 bindet die NFAT- und NFκB-Proteine in den konservierten Rel-domänen. Die NFκBProteine binden EGR-4 in den Rel-Domänen in Aminosäurenbereichen aa 43-67 (p50), aa 3963 (p52) und aa 20-44 (p65). NFATc und NFATp binden EGR-4 in den „Rel-similarity“Domänen mit den Bereichen aa 426-449 und aa 408-431. In einem Homologievergleich zwischen NFAT- und NFκB-Proteinen konnte ein potentielles Motiv der Sequenz xGxYx3ExQPKx3Rx2Yx2EGxSxGx für die Bindung von EGR-4 identifiziert werden (Abb. 31 und Tabelle 12). Die hoch konservierten Rel-Domänen vermitteln sowohl bei NFAT- als auch bei den NFκB-Proteinen die DNA-Bindung (Jain et al., 1995; Luo et al., 1996 a und b; Chytil and Verdine, 1996; Rao et al., 1997). Bei den Proteinen der NFκB-Familie finden außerdem Homo- bzw. Heterodimerisierungen über die Rel-Domäne statt (Bours et al., 1992 a und b; Ryseck et al., 1992; Ip et al., 1993). Es ist deshalb sehr wahrscheinlich, dass die NFκBProteine über die Rel-Region auch andere Transkriptionsfaktoren können. 4.6 Strukturelle Domänen von EGR-4 In dieser Arbeit konnten für EGR-4 strukturellen Domänen identifiziert werden, die bei der Interaktion mit NFAT- und NFκB-Proteinen eine wichtige Rolle spielen (Abb. 32). EGR-4 besitzt drei Aktivierungs- und zwei Repressordomänen. Zwei Aktivierungsdomänen wurden im N-Terminus (aa 1-150) und in der mittleren Region (aa 171-325) identifiziert (Abb. 20 und 28). Die dritte Aktivierungsdomäne liegt in der Zinkfingerregion (aa 379-463), die für die Bindung an die DNA verantwortlich ist. Die Repressordomänen befinden sich direkt Nterminal (aa 326-378) und C-terminal (aa 464-487) neben der Zinkfingerdomäne. EGR-4 besitzt je drei Aminosäurebereiche in der Region aa 158-325 für die Bindung von NFAT- und NFκB-Proteinen. Die Aminosäurebereiche für die Bindung von NFAT-Proteinen (aa 173-197, - 89 - Diskussion aa 224-238 und aa 306-325) liegen N-terminal vom Zinkfinger in einer Aktivierungsdomäne (Abb. 18-22). Die Interaktionsdomäne für die p65-Bindung ist in einer Aktivierungsdomäne der Zinkfingerregion im Bereich der Aminosäuren 397-465 vorhanden (Abb. 29). Die regulatorische Funktion der EGR-Proteine wird sowohl durch Bindung an die DNA als auch durch Protein-Proteininteraktionen kontrolliert (Pavletich and Pabo, 1991; Gashler et al., 1993; Vesque and Charnay, 1992; Krämer et al., 1994; Skerka et al., 1995; Lin und Leonard, 1997; Khachigian et al., 1996; Decker et al., 1998 und 2003). Abb. 32 Interaktionsdomänen und transkriptionell regulative Domänen von EGR-4 In der Abbildung sind die EGR-Domänen, die in dieser Arbeit charakterisiert wurden, zusammengefasst. Die EGR-4-Regionen, die Interaktionen mit NFAT-Proteinen vermitteln, sind im EGR-4-Protein schwarz gestreift dargestellt und die verantwortlichen Aminosäurereste angegeben. In der Zinkfingerdomäne von EGR-4 (hellgrau) sind drei Regionen schwarz hervorgehoben, welche das NFκB-Proteine p65 binden. Im Ausschnitt darüber sind die an der Bindung von EGR-4 und p65 beteiligten Aminosäuren hervorgehoben. Aminosäuren, welche p65 stark binden, sind schwarz unterlegt. Die Aktivierungs- (AD) und Repressordomänen (RD) sind unter dem Protein als Balken dargestellt. Die Positionen der Domänen sind unter dem Protein angedeutet. Die Ergebnisse deuten darauf hin, dass EGR-4 ein Multidomänenprotein ist und wahrscheinlich als Teil eines spezifischen regulatorischen Promotorkomplexes die Transkription inflammatorischer Gene kontrolliert Transkriptionskomplex verbunden ist. - 90 - und mit dem der basale Literaturverzeichnis 5 Literaturverzeichnis Ackerman, S. L., Minden, A. G., Williams, G. T., Bobonis, C. and Yeung, C. Y. (1991): Functional significance of an overlapping consensus motif for Sp-1 and Zif268 in the murine deaminase gene promoter. Proc. Natl. Acad. Sci. USA 88: 7523-7527. Alkalay, I., Yaron, A., Hatzubai, A., Orian, A., Ciechanover, A. and Ben-Neriah, Y. (1995): Stimulation-dependent IκB phosphorylation marks the NF-κB inhibitor for degradation via the ubiquitin-proteasome pathway. Proc. Natl. Acad. Sci. USA 92: 10599-10603. Aramburu, J., Azzoni, L., Rao, A. and Perussia, B. (1995): Activation and expression of the nuclear factor of activated T cells, NFATp and NFATc, in human natural killer cells: regulation upon CD16 ligand binding. J. Exp. Med. 182: 801-810. Aramburu, J., Garcia-Cozar, F., Raghavan, A., Okamura, H., Rao, A. and Hogan, P. G. (1998): Selective inhibition of NFAT activation by a peptide spanning the calcineurin targeting site of NFAT. Mol. Cell. 1 (5): 627-637. Baeuerle, P. A. and Henkel, T. (1994): Function and activation of NF-κB in the immune system. Annu. Rev. Immunol. 12: 141-179. Baeuerle, P. A. and Baltimore, D. (1996): NF-κB: ten years after. Cell 87: 13-20. Baeuerle, P. A. and Baichwal, V. R. (1997): NF-κB as a frequent target for immuno-supressive and antiinflammatory molecules. Adv. Immunol. 65: 111-137. Baldwin, A. S. (1996): The NF-κB and IκB proteins: new discoveries and insights. Annu. Rev. Immunol. 14: 649681. Ballard, D. W., Dixon, E. P., Peffer, N. J., Bogerd, H. and Doerre, S. (1992): The 65-kDa subunit of human NF-κB functions as a potent transcriptional activator and a target for v-Rel-mediated repression. Proc. Natl. Acad. Sci. USA 89: 1875-1879. Beals, C. R., Clipstone, N. S., Ho, S. N. and Cabtree, G. R. (1997): Nuclear localization of NF-ATc by calcineurindependent, cyclosporine-sensitive intramolecular interaction. Genes Dev. 11: 824-834. Beckmann, M. A. and Wilce, P. A. (1997): Egr transcription factors in the nervous system. Neurochem. Int. 31: 477510. Biesiada, E., Razandi, M. and Levin, E. R. (1996): Egr-1 activates basic fibroblast growth factor transcription. J. Biol. Chem. 271: 18576-18581. Bours, V., Villalobos, J., Burd, P. R., Kelly, K. and Siebenlist, U. (1990): Cloning of a mitogen-inducible gene encoding a κB DNA-binding protein with homology to the rel oncogene and to cell-cycle motifs. Nature 348: 76-80. Bours, V., Fransoso, G., Brown, K., Park S., Azarenko, V., Tomita-Yamaguchi, M., Kelly, K. and Siebenlist, U. (1992 a): Lymphocyte activation and the family of NF-κB transcription factor complexes. Curr. Top. Microbiol. Immunol. 182: 411-420. Bours, V., Burd, P. R., Brown, K., Villalobos, J., Park, S. Ryseck, R. P., Bravo, R., Kelly, K. and Siebenlist, U. (1992 b): A novel mitogen- inducible gene product related to p50-p105-NF-κB participates in transactivation trough a κB site. Mol. Cell. Biol. 12: 685-695. Brand, K., Page, S., Rogler, G., Bartsch, A., Brandl, R., Knuechel, R., Page, M., Kaltschmidt, C., Baeuerle, P. A. and Neumeier D. (1996): Activated transcription factor NF-κB is present in the atherosclerotic lesion. J. Clin. Invest. 97: 1715-1722. Brownell, E., Mittereder, N. and Rice, N. R. (1989): A human rel protooncogene cDNA containing an Alu fragment as a potential coding exon. Oncogene 4: 935-942. Call, K. M., Glaser, T., Ito, C. Y., Buckler, A. J., Pelletier, J., Haber, D. A., Rose, E. A., Yeger, H., Lewis, W. H., Jones, C. and Housman, D. E. (1990): Isolation and characterization of a zinc finger polypeptide gene at the human chromosome 11 Wilms´tumor locus. Cell 60: 509-520. Cao, X., Koski, R. A., Gashler, A., McKiernan, M., Morris, C. F., Gaffney, R., Hay, R. V. and Sukhatme, V. P. (1990): Identification and characterization of the Egr-1 gene product, a DNA-binding zinc finger protein induced by differentiation and growth signals. Mol. Cell Biol. 10: 1931-1939. - 91 - Literaturverzeichnis Cao, X., Guy, G. R., Sukhatme, V. P. and Tan, Y. H. (1993): Regulation of the EGR-1 gene by tumor necrosis factor and interferons in primary human fibroblasts. J. Biol. Chem. 267: 1345-1349. Carman, J. A. and Monroe, J. G. (1995): The EGR1 protein contains a discrete transcriptional regulatory domain whose deletion results in a truncated protein that blocks EGR1-induced transcription. DNA Cell Biol. 14: 581-589. Changelian, P. S., Feng, P., King, T. and Milbrandt, J. (1989): Structure of the NGFI-A gene and detection of upstream sequences responsible for its transcriptional induction by nerve growth factor. Proc. Natl. Acad. Sci. USA 86: 377-381. Chapman, N. R. and Perkins, N. D. (2000): Inhibition of RelA (p65) NF-kappaB subunit by Egr-1. J. Biol. Chem. 275: 4719-4725. Chavrier, P., Zerial, M., Lemaire, P., Almendral, J., Bravo, R. and Charnay, P. (1988): A gene encoding a protein with zinc fingers is activated during G0/G1 transition in cultured cells. EMBO J. 7: 29-35. Chavrier, P., Janssen-Timmen, U., Mattei, M. G., Zerial, M., Bravo, R. and Charnay, P. (1989): Structure, chromosome localization, and expression of the mouse zinc finger gene krox-20: multiple gene products and coregulation with the proto-oncogene c-fos. Mol. Cel. Neurosci. 9: 787-797. Chavrier, P., Vesque, C., Galliot, B., Vigneron, M., Dollé, P., Duboule, D. and Charnay, P. (1990): The segmentspecific gene Krox-20 encodes a transcription factor with binding sites in the promoter region of the Hox-A4 gene. EMBO J. 9: 1209-1218. Chen, C. C., Rosenbloom, C. L., Anderson, D. C. and Manning, A. M. (1995): Selective inhibition of E-selectin, vascular cell adhesion molecule-1, and intercellular adhesion molecule-1 expression by inhibitors of IκB-α phosphorylation. J. Immunol. 155: 3538-3545. Chen, L., Glover, J. N. M., Hogan, P. G., Rao, A. and Harrison, S. C. (1998): Structure of the DNA-binding domains from NFAT, Fos and Jun bound specifically to DNA. Nature 392: 42-48. Chen, Z., Hagler, J., Palombella, V. J., Melandri, F., Scherer, D., Ballard, D. and Maniatis, T. (1995): Signalinduced site- specific phosphorylation targets IkB α to the ubiquitin-proteasome pathway. Genes Dev. 9: 1586-1597. Choi, M. S. K., Brines, R. D., Holman, M. J. and Klaus, G. G. B. (1994): Induction of NF-AT in normal B lymphocytes by antiimmunoglobulin or CD40 ligand in conjunction with IL-4. Immunity 1: 179-187. Christy, B. A., Lau, L. F. and Nathans, D. (1988): A gene activated in mouse 3T3 cells by serum growth factors encodes a protein with “zinc finger” sequences. Proc. Natl. Acad. Sci. USA 85: 7857-7861. Christy, B. and Nathans, D. (1989 a): DNA binding site of the growth factor- inducible protein Zif2688. Proc. Natl. Acad. Sci. USA 86: 88737-8741. Christy, B. and Nathans, D. (1989 b): Functional serum response elements upstream of the growth factor-inducible gene zif268. Mol. Cell. Biol. 9: 4889-4895. Chuvpilo, S., Avots, A., Berberich-Siebelt, F., Glockner, J., Fischer, C., Kerstan, A., Escher, C., Inashkina, I., Hlubek, F., Jankevics, E., Brabletz, T. and Serfling, E. (1999): Multiple NF-ATc isoforms with individual transcriptional properties are synthesized in T lymphocytes. J. Immunol. 162 (12): 7294-301. Chytil, M. and Verdine, G. L. (1996) The Rel family of eukaryotic transcription factors. Curr. Opin. Struct. Biol. 6: 91-100. Cogswell, P. C., Mayo, M. W. and Baldwin, A. S. (1997): Involvement of EGR-1/RelA synergy in distinguishing T cell activation from tumor necrosis factor-α-induced NFκB1 transcription. J. Exp. Med. 185: 491-497. Collins, T., Read, M. A., Neish, A. S., Whitley, M. Z., Thanos, D. and Maniatis, T. (1995): Transcriptional regulation of endothelial cell adhesion molecules: NF-κB and cytokine-inducible enhancers. FASEB J. 9: 899-909. Cow, C. W., Rincon, M., Cavanagh, J., Dickens, M. and Davis, R. J. (1997): Nuclear accumulation of NFAT4 opposed by JNK signal transduction pathway. Science 278: 1638-1641. Crabtree, G. R. and Clipstone, N. A. (1994): Signal transmission between the plasma membrane and nucleus of Tlymphocytes. Annu. Rev. Biochem. 63: 1045-1083. - 92 - Literaturverzeichnis Crosby, S. D., Puetz, J. J., Simburger, K. S., Fahmer, T. J. and Milbrandt, J. (1991): The early response gene NGFIC encodes a zinc finger transcriptional activator and is member of the GCGGGGGCG (GSG) element-binding protein family. Mol. Cell. Biol. 11: 3835- 3841. Crosby, S. D., Veile, R. A., Donis-Keller, H., Baraban, J. M., Bhat, R. V., Simburger, K. S. and Milbrandt, J. (1992): Neural-specific expression, genomic structure, and chromosomal localization of the gene encoding the zincfinger transcription factor NGFI-C. Proc. Natl. Acad. Sci. USA 89: 4739-4743. Cui, M.-Z., Party, G. C. N., Oeth, P., Larson, H., Smith, M., Huang, R.-P., Adamson, E. D. and Mackman, N. (1996): Transcriptional regulation of the tissue factor gene in human epithel cells is mediated by Sp1 and EGR-1. J. Biol. Chem. 271: 2731-2739. Dagert, W. S. and Ehrlich, S. D. (1979): Prolonged incubation in calcium chlorid improves the competence of E. coli cells. Gene 6: 23-28. Day, M. L., Wu, S. and Basler, J. W. (1990): Prostatic nerve growth factor inducible A gene binds a novel element in the retinoblastoma gene promoter. Cancer Res. 53: 5597-5599. de la Pompa, J. L., Timmerman, L. A., Takimoto, H., Yoshida, H., Elia, A. J., Samper, E., Potter, J., Wakeham, A., Marengrere, L., Langille, B. L., Crabtree, G. R. and Mak, T. W. (1998): Role of the NF-ATc transcription factor in morphogenesis of cardiac valves and septum. Nature 392: 182-186. Decker, E., Skerka, C. and Zipfel, P. F. (1998): The early growth response protein (EGR-1) regulates interleukin-2 transcription by synergistic interaction with the nuclear factor of activated T cells. J. Biol. Chem. 273: 26923-26930. Decker, E. L. , Nehmann, N., Kampen, E., Eibel, H., Zipfel, P. F. and Skerka, C. (2003): Early growth response proteins (EGR) and nuclear factors of activated T cells (NFAT) form heterodimers and regulate proinflammatory cytokine gene expression. Nucl. Acids Res. 31 (3): 911-921. DiDonato, J. A., Hayakawa, M., Rothwarf, D. M., Zandi, E. and Karin, M. (1997): A cytokine-responsive IB kinase that activates the transcription factor NF-κB. Nature 388: 853-862. Dinkel, A., Aichler, W. K., Haas, C., Zipfel, P. F., Peter, H. H. and Eibel, H. (1997): Transcription factor Egr-1 activity down-regulates Fas and CD23 expression in B cells. J. Immunol. 159: 2678-2684. Dinkel, A., Warnatz, K., Ledermann, B., Rolnik, A., Zipfel, P. F., Bürki, K. and Eibel, H. (1998): The transcription factor early growth response 1 (Egr-1) advances differentiation of pre-B and immature B cells. J. Exp. Med. 188: 2215-2224. Dumont, F. J., Staruch, M. J., Koprak, S. L., Siekierka, J. J., Lin, C. S., Harrison, R., Sewell, T., Kindt, V. M., Beattie, T. R. and Wyvratt, M. (1992): The immunosuppressive and toxic effects of FK-506 are mechanistically related: pharmacology of a novel antagonist of FK-506 and rapamycin. J. Exp. Med. 176: 751-760. Emmel, E. A., Verweij, C. L., Durand, D. B., Higgins, K. M., Lacy, E. and Crabtree, G. R. (1989): Cyclosporin A specifically inhibits function of nuclear proteins involved in T cell activation. Science 246: 1617-1620. Fisher, G. J., Datta, S. C., Talwar, H. S., Wang, Z. Q., Varani, J., Kang, S. and Voorhees, J. J. (1996): Molecular basis of sun-induced premature skin aging and retinoid antagonism. Nature 379: 335-339. Frommer, G., Vorbrüggen, G., Pasca, G., Jäckle, H. and Volk, T. (1996): Epidermal egr-like zinc finger protein of Drosophila participates in myotube guidance. EMBO J. 15: 1642-1649. Fujisawa, K., Aono, H., Hasunuma, T., Yamamoto, K., Mita, S. and Nishioka, K. (1996): Activation of transcription factor NF-κB in human synovial cells in response to tumor necrosis factor α. Arthritis Rheum. 39: 197-203. Gashler, A. L., Swaminathan, S. and Sukhatme, V. P. (1993): A novel repression module, an extensive activation domain, and a bipartite nuclear localization signal defined in the immediate-early transcription factor Egr-1. Mol. Cell. Biol. 13: 4556-4571. Gashler, A. and Sukhatme, V. P. (1995): Early Growth Response Protein 1 (Egr-1): Prototyp of a Zinc-finger Family of Transcription Factors. Prog. Nucl. Acid Res. Mol. Biol. 50: 191-224. Gerritsen, M. E., Williams, A. J., Neish, A. S., Moore, S., Shi, Y. and Collins, T. (1997): CREB-binding protein/p300 are transcriptional coactivators of p65. Proc. Natl. Acad. Sci. USA 94 (7): 2927-2932. Gessler, M., Poustka, A., Cavenee, W., Neve, R. L., Orkin, S. H. and Bruns, G. A. P. (1990): Homozygous deletion in Wilms tumours of a zinc-finger gene identified by chromosome jumping. Nature 343: 774-778. - 93 - Literaturverzeichnis Ghosh, S. and Baltimore, D. (1990): Activation in vitro of NF-κB by phosphorylation o its inhibitor IκB. Nature 344: 678-682. Granelli-Piperno, A., Nolan, P., Inhaba, K. and Steinman, R. M. (1990): The effect of immunosuppressive agents on the induction of nuclear factors that bind to sites on the interleukin 2 promoter. J. Exp. Med. 172: 1869-1872. Haber, D. A., Sohn, R. L., Buckler, A. J., Pelletier, J., Call, K. A. and Housman, D. E (1991): alternative splicing and genomic structure of Wilms tumor gene WT1. Proc. Natl. Acad. Sci. USA 88: 9618-9622. Hanahan, D. (1983): Studies on transformation of E. coli with Plasmids. J. Mol. Biol. 166: 557-580. Handel, M. L., McMorrow, L. B. and Gravallese, E. M. (1996): NF-κB in rheumatoid synovium: localization of p50 and p65. Arthritis Rheum. 38: 1762-1770. Hannum, C., Arai, K.-I. and Arai, N. (1995): NFATx: a novel member of the NFAT family that is predominantly expressed in the thymus. Mol. Cell. Biol. 15: 2697-2706. Henkel, T., Zabel, U., Van Zee, K., Muller, J. M., Fanning, E. and Bauerle, P. (1992): Intramolecular masking of the nuclear localization signal and dimerization domain in the precursor for the p50 NF-κB subunit. Cell 68: 1121-1133. Herdegen, T., Bravo, R., Waxman, S. G. and Kocsis, J. D. (1993): Jun, Fos, Krox and CREB protein expression is different in adult dorsal root ganglion neurons studied in in vivo and in vitro. Soc. Neurosci. Abstr. 19: 711-715. Herschman, H. R. (1989): Extracellular signals, transcriptional responses and cellular specificity. Trends Biochem. Sci. 14: 455-458. Heukeshoven, J. and Denick, R. (1986): Simplified method for silver staining of proteins in polyacrylamide gels and the mechanism of silver staining. Electrophoresis 6: 103-112. Hilyard, K., Reyburn, H., Chung, S., Bell., J. I. and Strominger, J. L. (1994): Binding of soluble natural ligands to a soluble human T-cell receptor fragment produced in Escherichia coli. Proc. Natl. Acad. Sci. USA 91: 9057-9061. Ho, A. M., Jain, J., Rao, A. and Hogan, P. G. (1994): Expression of the transcription factor NFATp in a neuronal cell line and the murine nervous system. J. Biol. Chem. 269: 28181-28186. Hodge, M. R., Ranger, A. M., de la Brousse, F. C., Hoey, T., Grusby, M. and Glimcher, L. H. (1996): Hyperproliferation and dysregulation of IL-4 expression in NF-ATp-deficient mice. Immunity 4: 397-405. Hoey, T., Sun, Y.-L., Williamson, K. and Xu, X. (1995): Isolation of two new members of the NF-AT gene family and functional characterization of the NF-AT proteins. Immunity 2: 461-472. Holst, C., Skerka, C., Lichter, P., Bialonski, A. and Zipfel, P. F. (1993): Genomic organization, chromosomal localization and promoter function of the human zinc- finger gene pAT133. Human Mol. Gen. 2: 367-372. Huang, R. P. and Adamson, E. D. (1993): Characterization of the DNA-binding properties of the early growth response-1 (Egr-1) transcription factor: evidence for modulation by redox mechanism. DNA Cell Biol. 12: 265-273. Huang, R. P. and Adamson, E. D. (1994): The phosphorylated forms of the transcription factor, Egr-1, bind to DNA more efficiently than non-phosphorylated. Biochem. Biophys. Res. Comm. 200: 1271-1276. Huang, R. P., Darland, T., Okamura, D., Mercola, D. and Adamson, E. D. (1994): Supression of v-sis-dependent transformation by transcription factor Egr-1. Oncogene 9: 1367-1377. Huang, R.-P., Fan, Y., Peng, A., Zeng, Z.-L., Reed, J. C., Adamson, E. D. and Boynton, A. L. (1998): Suppression of human fibrosarcoma cell growth by transcription factor, Egr-1, involves down-regulation of Bcl-2. Int. J. Cancer 77: 880-886. Hunter, T. and Karin, M. (1992): The regulation of transcription of phosphorylation. Cell 70: 374-387. Ip, Y. T., Reach, M., Engstrom, Y., Kadalayil, L., Cai, H., Gonzalez-Crespo, S., Tatei, K. and Levine, M. (1993): Dif, a dorsal- related gene that mediates an immune response in Drosophila. Cell 75: 753-763. Jain, J., McCaffrey, P. G., Valge-Archer, V. E. and Rao, A. (1992): Analysis of the AP-1 sites in the IL-2 promoter. Nature 356: 801-804. Jain, J., Burgeon, E., Badalian, T. M., Hogan, P. G. and Rao, A (1995): A similar DNA-binding motif in NFAT family proteins and the rel homology domain. J. Biol. Chem. 270: 4138-4145. - 94 - Literaturverzeichnis Janssen-Timmen, U., Lemaire, P., Mattéi, M. G., Revelant, O. and Charnay, P. (1989): Structure, chromosome mapping and regulation of the mouse zinc-finger gene Krox-24: evidence for a common regulatory pathway for immediate-early serum response genes. Oncogene 80: 325-336. Joseph, L. J., Le Beau, M. M., Jamieson, G. A. Jr., Acharya, S., Shows, T. B., Rowley, J. D. and Sukhatme, V. P. (1988): Molecular cloning, sequencing and mapping of EGR2, a human early growth response gene encoding a protein with “zinc-binding finger” structure. Proc. Natl. Acad. Sci. USA 85: 7164-7168. Karin, M. and Hunter, T. (1995): Transcriptional control by protein phosphorylation: signal transmission from the cell surface to the nucleus. Curr. Biol. 5: 747-757. Khachigian, L. M., Lindner, V., Williams, A. J. and Collins, T. (1996): Egr-1 induced endothelial gene expression: a common theme in vascular injury. Science 271: 1427-1430. Kieran, M., Blank, V., Logeat, F., Vandekerckhove, J., Lottspeich, F., Le Bail, O., Urban, M. B., Kourilsky, P., Baeuerle, P. A. and Israel, A. (1990): The DNA binding subunit of NF-κB is identical to factor KBF1 and homologous to the rel oncogene product. Cell 62: 1007-1018. Krämer, B., Meichle, A., Hensel, G., Charnay, P. and Krönke, M. (1994): Characterization of an Krox-24/Egr-1 responsive element in the human tumor necrosis factor promoter. Biochim. Biophys. Acta 1219: 413-421. Kühn, S. and Zipfel, P. F. (1995): The baculovirus expression vector pBSV-8His directs secretion of histidinetagged proteins. Gene 162: 225-229. Kyhse-Andersen, J. (1984): Electroblotting of multiple gels: a simple apparatus without buffer tank for rapid transfer of proteins from polyacrylamide ti nitrocellulose. J. Biophys. Methods 10:203-209. Laemmli, U. K. (1970): Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature 227: 680-685. Lee, S. L., Sadovsky, Y., Swirnoff, H., Polish, J. A., Goda, P., Gavrilina, G. and Milbrandt, J. (1996): Luteinizing hormone deficiency and female infertility in mice lacking the transcription factor NGFI-A (Egr-1): Science 273: 1219-1221. Lemaire, P., Revelant, O., Bravo, R. and Charnay, P. (1988): Two mouse genes encoding potential transcription factors with identical DNA-binding domains are activated by growth factors in cultured cells. Proc. Natl. Acad. Sci. USA 85: 4691-4695. Lemaire, P., Vesque, C., Schmitt, J., Stunnenberg, H., Frank, R. und Charnay, P. (1990): The serum-inducible mouse gene Krox-24 encodes a sequence-specific transcriptional activator. Mol. Cell. Biol. 10: 3456-3467. Lim, R. W., Varnum, B. C. and Herman, H. R. (1987): Cloning of tetradecanoyl phorbol ester-induced “primary response” sequences and their expression in density-arrested Swiss 3T3 cells and a TPA non-proliferative variant. Oncogene 1: 263- 270. Lim, R. W., Varnum, B. C., O´Brian, T. G. and Herman, H. R. (1989): Induction of tumor promoter-inducible genes in murine Swiss 3T3 cell lines and tetradecanoyl phorbol acetate-nonproliferative 3T3 variants can occur through protein kinase C-dependent and -independent pathways. Mol. Cell. Biol. 9: 1790-1793. Lin, J.-X. and Leonard, W. J. (1997): The immediate-early gene product Egr-1 regulates the human interleukin-2 receptor b-chain promoter trough noncanonical Egr and Sp1 binding sites. Mol. Cell. Biol. 17: 3714-3722. Liu, C., Adamson, E. and Mercola, D. (1996): Transcription factor EGR-1 suppresses the growth and transformation of human HAT-1080 fibrosarcoma cells by induction of transforming growth factor b1. Proc. Natl. Acad. Sci. USA 93: 11831-11836. Liu, J. (1993): FK506 and cyclosporin, molecular probes for studying intracellular signal transduction. Immunol. Today 14: 290-295. Liu, J., Grogan, L., Nau, M. M., Allegra, C. J., Chu, E. and Wright, J. J. (2001): Physical interaction between p53 and primary response gene Egr-1. Int. J. Oncol. 18: 863-870. Li-Weber, M., Laur, O. and Krammer, P. H. (1999): Novel Egr/NFAT composite sites mediate activation of the CD95 (APO/Fas): ligand promoter in response to T cell stimulation. Eur. J. Immunol. 29: 3017-3027. Loh, C., Carew, J. A., Kim, J., Hogan, P. G. and Rao, A. (1996 a): T-cell receptor stimulating elicits an early phase of activation and a later phase of deactivation of the transcription factor NFAT1. Mol. Cell. Biol. 16: 3945-3954. - 95 - Literaturverzeichnis Loh, C., Shaw, K. T. Y., Carew, J. A., Viola, J. P., Luo, C., Perrino, B. A. and Rao, A. (1996 b): Calcineurin binds the transcription factor NFAT1 and reversibly regulates its activity. J. Biol. Chem. 271: 10884-10891. Lόpez-Rodriguez, C., Aramburu, J., Rakeman, A. S. and Rao, A. (1999): NFAT5, a constitutively nuclear NFAT protein that does not cooperate with Fos and Jun. Proc. Natl. Acad. Sci. USA 96: 7214-7219. Luo, C., Burgeon, E., Carew, J. A., McCaffrey, P. G., Badaiian, T. M., Lane, W. S., Hogan, P. G. and Rao, A. (1996 a): Recombinant NFAT1 (NFATp) is regulated by calcineurin in T cells and mediates transcription of several cytokine genes. Mol. Cell. Biol. 16: 3955-3966. Luo, C., Shaw, K. T. Y., Ragghaven, A., Aramburu, J., Garcia-Cozar, F., Perrino, B. A., Hogan, P. G. and Rao, A. (1996 b): Interaction of calcineurin with a domain of the transcription factor NFAT1 that controls nuclear import. Proc. Natl. Acad. Sci. USA 93: 8907-8912. Luo, C., Burgeon, E. and Rao, A. (1996 c) Mechanisms of transactivation by nuclear factor of activated T Cell-1. J. Exp. Med. 184: 141-147. Mack, K. J., Cortner, J., Mack, P. and Farnham, P. J. (1992): krox 20 messenger RNA and protein expression in the adult central nervous system. Mol. Brain Res. 14: 177-123. Mages, H. W., Stamminger, T., Rilke, O., Bravo, R. and Kroczek, R. A. (1993): Expression of PILOT, a putative transcription factor, requires two signal and is cyclosporin a sensitive in T cells. Intern. Immunol. 5: 63-70. Maltzman, J. S., Carman, J. A. and Monroe, J. G. (1996 a): Role of EGR-1 in regulation of stimulus-dependent CD44 transcription in B lymphocytes. Mol. Cell. Biol. 16: 2283-2294. Maltzman, J. S., Carman, J. A. and Monroe, J. G. (1996 b): Transcriptional regulation of the ICAM-1 gene in antigen receptor- and phorbol ester-stimulated B lymphocytes: role for transcription factor EGR1. J. Exp. Med. 183: 1747-1759. Maniatis, T. (1997): Catalysis by multiprotein IκB kinase complex. Science 278: 818-819. Manning, A. M. and Anderson, D. C., (1994): Transcription factor NF-κB: an emerging regulator of inflammation. Annu. Rep. Med. Chem. 29: 235-244. Manning, A. M. and Rao, A. (1999): Agents targeting transcription factors. Inflammation: Basic Principles and Clinical Correlates. 3rd ed., edited by John. I. Gallin and Ralph Snyderman. Lippincott Williams & Wilkins, Philadelphia. Masuda, E. S., Naito, Y., Tokumitsu, H., Campbell, D., Saito, F., Hannum, C., Arai, K.-I. and Arai, N. (1995): NFATx, a novel member of the nuclear factor of activated T cells family that is expressed predominantly in the thymus. Mol. Cell. Biol. 15: 2697- 2706. Masuda, E. S., Imamura, R., Amaski, Y., Arai, K. and Arai, N. (1998): Signaling into the T- cell nucleus: NFAT regulation. Cell. Signal. 10: 599-611. Matheny, C. Day, M. L. and Milbrandt, J. (1994): The nuclear localization signal of NGFI-C is located within the zinc finger DNA binding domain. J. Biol. Cehm. 269: 8176-8181. McCaffrey, P. G., Luo, C., Kerppola, T. K., Jain, J., Badalian, T. M., Ho, A. M., Burgeon, E., Lane, W. S., Lambert, J. N., Curran, T., Verdine, G. L., Rao, A. and Hogan, P. G. (1993): Isolation of the cyclosporin-sensitive T cell transcription factor NFATp. Science 262: 750-754. Mercurio, F., Didonato, J., Rosette, C. and Karin, M. (1992): molecular cloning and characterization of a novel Rel/N-κB family member displaying structural and functional homology to NF-κB p50-p105. DNA Cell. Biol. 11523-537. Meyer, R., Hatada, E. N., Hohmann, H. P., Haiker, M., Bartsch, C., Rothlisberger, U., Lahm, H. W., Schlaeger, E. J., van Loon, A. P. and Scheidereit, C. (1991): Cloning of the DNA-binding subunits of human nuclear factor κB: the level of mRNA is strongly regulated by phorbol ester or tumor necrosis factor α. Proc. Natl. Acad. Sci. USA 88: 966-970. Milbrandt, J. (1987): A nerve growth factor-induced gene encodes a possible transcriptional regulatory factor. Science 238: 797-799. Miller, J., McLachlan, A. D. and Klug, A. (1985): Repetitive zinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes. EMBO J. 4: 1609-1614. - 96 - Literaturverzeichnis Minc, E., de Coppet, P., Masson, P., Thiery, L., Dutertre, S., Amor-Guéret, M. and Jaulin, C. (1999): The human copper-zinc superoxide dismutase gene (SOD1) proximal promoter is regulated by Sp1, Egr-1 and WT1 via noncanonical binding sites. J. Biol. Chem. 274: 503-509. Mittelstadt, P. R. and Ashwell, J. D. (1998): Cyclosporin A-sensitive transcription factor Egr-3 regulates Fas ligand expression. Mol. Cell. Biol. 18: 3744-3751. Mittelstadt, P. R. and Ashwell, J. D. (1999): Role of Egr-2 in up-regulation of Fas ligand in normal T cells and aberrant double-negative Ipr and gld T cells. J. Biol. Chem. 274: 3222-3227. Miyazaki, T. and Lemonnier, F. A. (1998): Modulation of thymic selection by expression of an immediate early gene, early growth response 1 (Egr-1). J. Exp. Med. 188: 715-723. Müller, H.-J., Skerka, C., Bialonski, A. and Zipfel, P. F. (1991): Clone pAT133 identifies a gene that encodes another human member of a class of growth factor-induced genes with almost identical zinc-finger domains. Proc. Natl. Acad. Sci. USA 88: 10079-10083. Nair, P., Muthukkumar, S., Sells, S. F., Han, S.-S., Sukhatme, V. P. and Rangnekar, V. M. (1997): Early growth response-1-dependent apoptosis is mediated by p53. J. Biol. Chem. 272: 20131-20138. Newton, J. S., Li, J., Ning, Z.-Q., Schoendorf, D. E., Norton, J. D. and Murphy, J. J. (1996): B cell early response gene expression coupled to B cell receptor, CD40 and interleukin-4 receptor costimulation: evidence for a role of the egr-2/krox20 transcription factor in B cell proliferation. Eur. J. Immunol. 26: 811-816. Nikam, S. S., Tennekoon, G. I., Christy, B. A., Yoshino, J. E. and Rutkowski, J. L. (1995): The zinc finger transcription factor zif268 Egr-1 is essential for Swann cell expression of the p75NGF receptor. Mol. Cell Neurosci. 6: 337-348. Nolan, G. P., Ghosh, S., Liou, H.-C., Tempst, P. and Baltimore, D. (1991): DNA binding and IκB inhibition of the cloned p65 subunit of NF-κB, a rel-related polypeptide. Cell 64: 961-969. Northrop, J. P., Ho, S. N., Chen, L., Thomas, D. J., Timmerman, L. A., Nolan, G. P., Admon, A. and Crabtree, G. R., (1994): NF-AT components define a family of transcription factors targeted in T cell activation. Nature 369: 497-502. Oukka, M., Ho, I. C., de la Brousse, F. C., Hoey, T., Grusby, M. J. and Glimcher, L. H. (1998): The transcription factor NFAT4 is involved in the generation and survival of T cells. Immunity 9 (3): 295-304. Patwardhan, S., Gashler, A., Siegel, M. G., Chang, L. C., Joseph, L. J., Shows, T. B., Le Beau, M. M. and Sukhatme, V. P. (1991): EGR3, a novel member of the Egr family of genes encoding immediate-early transcription factors. Oncogene 6: 917-928. Pavletich, N. P. and Pabo, C. O. (1991): Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex of 2.1 Å. Science 252: 806-817. Perkins, N. D., Arganoff, A. B., Pascal, E. and Nabel, G. J. (1994): An interaction between the DNA-binding domains of RelA (p65) and Sp 1 mediates human immunodeficiency virus gene activation. Mol. Cell Biol. 14: 65706583. Petersohn, D., Schoch, S., Brinkmann, D. R. and Thiel, G. (1995): The human synapsin II gene promoter. Possible role for the transcription factor zif268/egr-1, polyoma enhancer activator 3, and AP 1. J. Biol. Chem. 270: 24612469. Prieschl, E. E., Gooilleux-Gruart, V., Walker, C, Harrer, N. E. and Baumruker, T. (1995): A nuclear factor of activated T cell-like transcription factor in mast cells is involved in IL-5 gene regulation after IgE plus antigen stimulation. J. Immunol. 154: 6112-6119. Ranger, A. M., Grusby, M. J., Hodge, M. R., Gravallese, E. M., de la Brousse, F. C., Hoey, T., Mickanin, C., Baldwin, H. S. and Glimcher, L. H. (1998 a): The transcription factor NF-ATc is essential for cardiac valve formation. Nature 392: 186-190. Ranger, A. M., Oukka, M., Rengarajan, J. and Glimcher, L. H. (1998 b): Inhibitory function of two NFAT family members in lymphoid homeostasis and Th2 development. Immunity 9: 627-635. - 97 - Literaturverzeichnis Rangnekar, V. M., Aplin, A. C. and Sukhatme, V. P. (1990): The serum and TPA responsive promoter and intronexon structure of EGR2, a human early growth response gene encoding a zinc finger protein. Nucl. Acids Res. 18: 2749-2757. Rao, A., Luo, C. and Hogan, P. G. (1997): Transcription factors of the NFAT family: regulation and function. Annu. Rev. Immunol. 15: 707-747. Regnier, C. H., Song, H., Gao, H., Goeddel, D. V., Cao, Z. and Rothe, M. (1997): Identification and characterization of an IκB kinase. Cell 90: 373-383. Ruben, S. M., Dillon, P. J., Schreck, R., Henkel, T., Chen, C. H., Maher, M., Baeuerle, P. A. and Rosen, C. A. (1991): Isolation of a rel-related human cDNA that potentially encodes the 65-kD subunit of NF-κB. Science 251: 1490-1493. Russo, M. W., Matheny, C. and Milbrandt, J. (1993): Transcriptional activity of the zinc finger protein NGFI-A is influenced by its interaction with cellular factor. Mol. Cell. Biol. 13: 6858-6865. Russo, M. W., Sevetson, B. R. and Milbrandt, J. (1995): Identification of NAB1, a repressor of NGFI-A- and Krox20-mediated transcription. Proc. Natl. Acad. Sci. USA 92: 6873-6877. Ryseck, R. P., Bull, P., Takamiya, M., Bours, V. and Siebenlist, U. (1992): RelB, a new rel family transcription activator that can interact with p50-NF-κB. Mol. Cell. Biol. 12: 674-684. Sakamoto, K. M., Bardeleben, C., Yates, K. E., Raines, M. A., Golde, D. W. and Gasson J. C. (1991): 5´upstream sequence and genomic structure of the human primary response gene, EGR-1/TIS8. Oncogene 6: 867-871. Sambrook, J., Fritch, E. F. and Maniatis, T. (1989): Molecular Cloning- A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York. Sanger, F., Nicklen, S. and Coulson, A. R. (1977): DNA-Sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. USA 74: 5463-5467. Schmidt, R. M., Perkins, N. D., Duckett, C. S., Andrews, P. C. and Nabel, G. J. (1991): Cloning of an NF-κB subunits which stimulates HIV transcription in synergy with p65. Nature 352: 733-736. Schreiber, S. L. and Crabtree, G. R. (1992): The mechanism of action of cyclosporin A and FK506. Immunol. Today 13: 136-142. Seyfert, V. L., Sukhatme, V. P. and Monroe, J. G. (1989): Differential expression of a zinc finger-encoding gene in response to positive versus negative signaling through receptor immunoglobulin in murine B lymphocytes. Mol. Cell. Biol. 9: 2083-2088. Sham, M. H., Vesque, C., Nonchev, S., Marshall, H., Frain, M., da Gupta, R., Whiting, J., Wilkinson, D., Charnay, P. and Krumlauf, R. (1993): The zinc finger gene Krox20 regulates HoxB2 (Hox2.8) during hindbrain segmentation. Cell 72: 183-196. Shao, B. H., Kono, D. H., Chen, L.-Y., Rubin, E. M. and Kaye, J. (1997): Induction of the early growth response (Egr) family of transcription factors during thymic selection. J. Exp. Med. 185: 731-744. Shaw, J.-P., Utz, P. J., Durand, D. B., Tröle, J. J., Emmel, E. A. and Crabtree, G. R. (1988): Identification of a putative regulator of early T cell activation genes. Science 241: 202-205. Shibasaki, F., Price, E. R., Milan, D. and Mckeon, F. (1996): Role of kinases and the phosphatase calcineurin in the nuclear shuttling of transcription factor NF-AT-4. Nature 382: 370-373. Siebenlist, U., Franzoso, G. and Brown, K. (1994): Structure, regulation and function of NF-κB. Annu. Rev. Cell Biol. 10: 405-455. Sif, S. and Gilmore, T. D. (1994): Interaction of the v-Rel oncoprotein with cellular transcription factor Sp1. J. Virol. 68: 7131-7138. Silverman, E. S., Du, J., Williams, A. J., Wadgaonkar, R., Drazen, J. M. and Collins T. (1998): cAMP-responseelement-binding-protein-binding-protein (CBP): and p300 are transcriptional co-activators of early growth response factor-1 (Egr-1). Biochim. J. 336: 183-189. Skerka, C., Decker, E. L. and Zipfel, P. F. (1995): A regulatory element in the human Interleukin 2 gene promoter is a binding site for the zinc finger proteins Sp1 and EGR-1. J. Biol. Chem. 38: 22500-22506. - 98 - Literaturverzeichnis Skerka, C., Decker, E. L. and Zipfel, P. F. (1997): Coordinate expression and distinct DNA-binding characteristics of the four EGR-zinc finger proteins in Jurkat T lymphocytes. Immunobiology 198: 179-191. Smith, D. B. and Johnsen, K. S. (1988): Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene 67: 31-40. Srivastava, S., Weitzmann, M. N., Kimble, R. B., Rizzo, M., Zahner, M., Milbrandt, J., Ross, F. P. and Pacifici, R. (1998): Estrogen blocks M-CSF gene expression and osteoclast formation by regulating phosphorylation of Egr-1 and its interaction with Sp1. J. Clin. Invest. 102: 1850-1859. Stancovski, I. and Baltimore, D. (1997): NF-κB-activation: the IκB kinase revealed? Cell 91: 299-302. Stranick, K. S., Zambas, D. N., Schettino-Us, A., Egan, R. W., Billah, M. M. and Umland, S. P. (1997): Identification of transcription factor binging sites important in the regulation of human interleukin-5 gene. EMBO J. 12: 3551-3558. Sukhatme, V. P., Cao, X., Chang, L. C., Tsai-Morris, C.-H., Stamenkovich, D., Ferreira, P. C. P., Cohen, D. R., Edwards, S. A., Shows, T. B., Curran, T., Le Beau, M. M. and Adamson, E. D. (1988): A zinc finger-encoding gene coregulated with c-fos during the growth and differentiation, and after cellular depolarisation. Cell 53: 37-43. Sukhatme, V. P. (1990): Early transcriptional events in cell growth: the Egr family. J. Am. Nephrol. 1: 859-866. Sukhatme, V. P. (1992): The EGR transcription factor family: From signal transduction to kidney differentiation. Kidney International 41: 550-553. Svaren, J., Sevetson, B. R., Apel, E. D., Zimonjic, D. B., Popescu, N. C. and Milbrandt, J. (1996): NAB2, a corepressor of NGFI-A (Egr-1): and Krox20, is induced by proliferative and diferentiative stimuli. Mol. Cell. Biol. 16: 3545-3553. Swirnoff, A. H. and Milbrandt, J. (1995): DNA-binding specificity of NGFI-A and related zinc finger transcription factors. Mol. Cell. Biol. 15: 2275-2287. Thiel, G., Schoch, S. and Petersohn, D. (1994): Regulation of synapsin I gene expression by the zinc finger transcription factor zif268/egr-1. J. Biol. Chem. 269: 15294-15301. Thottassery, J. V., Sun, D., Zambetti, G. P., Troutman, A., Sukhatme, V. P., Schuetz, E. G. and Schuetz, J. D. (1999): Sp1 and egr-1 have opposing effects on the regulation of the rat Pgp/2/mdr1b gene. J. Biol. Chem. 274: 3199-206. Timmerman, L. A., Clipstone, N. A., Ho, S. N., Northrop, J. P. and Cabtree, G. R. (1996): Rapid shuttling of NF-AT in discrimination of Ca2+ signals and immunosupression. Nature 383: 837-840. Tjian, R. and Maniatis T. (1994): Transcriptional activation complex puzzle with few easy pieces. Cell 77: 5-8. Trejo, S. R., Fahl, W. E. and Ratner, L. (1997): The Tax protein of human T-cell leukemia virus type 1 mediates the transactivation of the c-sis/ platelet-derived growth factor-B promoter through interactions with the zinc finger transcription factors Sp1 and NGFI-A/Egr-1. J. Biol. Chem. 272: 27411-27421. Tremblay, J. J. and Drouin, J. (1999): Egr-1 is a downstream effector of GnRH and synergizes by direct interaction with Ptx1 and SF-1 to enhance luteinizing hormone b gene transcription. Mol. Cell. Biol. 19: 2567-2576. Tsai-Morris, C. H., Cao, X. and Sukhatme, V. P. (1988): 5´flanking sequence and genomic structure of Egr-1, a murine mitogen inducible zinc finger encoding gene. Nucl. Acids Res. 16: 8835-8846. Tso, J. Y., Van Den Berg, D. J., and Korn, L. J. (1986): Structure of the gene for Xenopus transcription factor TFIIIA. Nucl. Acids Res. 14: 2187-2200. Vaccarino, F. M., Hayward, M. D., Le, H. N., Hartigan, D. J., Duman, R. S. and Nestler, E. J. (1993): Induction of immediate early genes by cyclic AMP in primary cultures of neurons from rat cerebral cortex. Mol. Brain Res. 19: 76-82. Venkataraman, L., Francis, D. A., Wang, Z., Liu, J., Rothstein, T. L. and Sen R. (1994): Cyclosporin A-sensitive induction of NF-AT in murine B cells. Immunity 1: 189-196. Vesque, C. and Charnay, P. (1992): Mapping of functional regions of the segment specific transcription factor Krox20. Nucl. Acids Res. 20: 2485-2492. - 99 - Literaturverzeichnis Waters, c. M., Hancock, D. C and Evan, G. I. (1990): Identification and characterization of the egr-1 gene product as an inducible, short-lived, nuclear phosphoprotein. Oncogene 5: 669.674. Wiederrecht, G., Lam, E., Hung, S., Martin, M. and Sigal, N. (1993): The mechanism of action of FK-506 and cyclosporin A. Ann. NY Acad. Sci. 696: 9-19. Williams, G. T. and Lau, L. F. (1993): Activation of the inducible orphan receptor gene nur77 by serum growth factors: dissociation of immediate-early and delayed-early responses. Mol. Cell. Biol. 13: 6124-6136. Wong, D. L., Siddall, B. J., Ebert, S. N., Bell, R. A. and Her, S. (1998): Phenylethanolamin N-methyltransferase gene expression: synergistic activation by Egr-1, AP-2 and the glucocorticoid receptor. Mol. Brain Res. 61: 154161. Woronicz, J., Calnan, B., Ngo, V. and Winoto, A. (1994): Requirement for the orphan steroid receptor nur77 in apoptosis of T-cell hybridisms. Nature 367: 277-281. Xanthoudakis, S., Viola, J. P. B., Shaw, K. T. Y., Luo, C., Wallace, J. D., Bozza, P. T., Curran, T. and Rao A (1996): An enhanced immune response in mice lacking the transcription factor NFAT1. Science 272: 892-895. Yamagata, K., Kaufmann, W. E., Lanahan, A., Papapavlou, M., Barnes, C. A., Andreasson, K. I. and Worley, P. F. (1994): Egr3/Pilot, a zinc finger transcription factor, is rapidly regulated by activity in brain neurons and colocalizes with Egr1/zif268. Learn Mem. 1 (2): 140-52. Yamit-Hezi, A., Nir, S., Wolstein, O. and Dikstein, R. (2000): Interaction of TAFII105 with selected p65/RelA dimers is associated with activation of subset of NF-kappa B genes. J. Biol. Chem. 275 (24): 18180-18187. Yang, Y., Dong, B., Mittelstadt, P. R., Xiao, H. and Ashwell, J. D. (2002): HIV Tat binds Egr proteins and enhances Egr-dependent transactivation of the Fas ligand promotor. J. Biol. Chem. 277: 19482-19487. Yaron, A., Alkalay, I., Hatzubai, A., Jung, S., Beyth, S., Mercurio, F., Manning, A. M., Ciechanover, A. and BenNeriah, Y. (1997): Inhibition of NF-κB cellular function via specific targeting of the IkB ubiquitin ligase. EMBO J. 16: 6486-94. Yoo, Y. D., Ueda, H., Park, K., Flanders, K. C., Lee, Y. I., Jay, G. and Kim, S.-J. (1996 a): Regulation of transforming growth factor-b1 expression by Hepatitis B virus (HBV): X transactivator. J. Clin. Invest. 97: 388-395. Yoo, Y. D., Chiou, C.-J., Choi, K. S., Yi, Y., Michelson, S., Kim, S., Hayward, G. S. and Kim, S.-J. (1996 b): The IE2 regulatory protein of human cytomegalovirus induces expression of the human transforming growth factor b1 gene through an Egr-1 binding site. J. Virol. 70: 7062-7070. Zhong, H., Voll, R. E. and Ghosh, S. (1998): Phosphorylation of NF-kappa B p65 by PKA stimulates transcriptional activity by promoting a novel bivalent interaction with the coactivator CBP/p300. Mol. Cell. 1 (5): 661-671. Zhong, H., May, M. J., Jimi, E. and Ghosh, S. (2002): The phosphorylation status of nuclear NF-kappa B determines its association with CBP/p300 or HDAC-1. Mol. Cell. 9 (3): 625-636. Zhu, J., Shibasaki, F., Price, R., Guillemot, J. C., Yano, T., Dotsch, V., Wagner, G., Ferrara, P. and McKeon, F. (1998): Intramolecular masking of nuclear import signal on NF-AT4 by casein kinase I and MEKK1. Cell 93: 851861. Ziegler-Heitbrock, H. W., Sternsdorf, T., Liese, J., Belohradsky, B., Weber, C., Wedel, A., Schreck, R., Bauerle, P. and Strobel, M. (1993): Pyrrolidine dithiocarbamate inhibits NF-kappa B mobilization and TNF production in human monocytes. J. Immunol. 151 (12): 6986-6993. Zipfel, P. F., Irving, S. G., Kelly, K. and Siebenlist, U. (1989): Complexity of the primary genetic response to mitogenic activation of human T cells. Mol. Cell. Biol. 9: 1041- 1048. Zipfel, P. F., Decker, E., Holst, C. and Skerka, C. (1997): The human zinc finger protein EGR- 4 acts as autoregulatory transcriptional repressor. Biochim. Biophys. Acta 1354: 134-144. Zipfel, P. F., Nehmann, N., Eibel, H., Siebenlist, U. and Skerka, C. (2003): Early Growth Response Protein EGR-4 interacts with immune inflammatory mediators NF-κB p65 and p50. J. Biol. Chem., eingereicht. - 100 - Publikationen Publikationen Aus dieser Arbeit sind während der Promotionszeit folgende Publikationen hervorgegangen: Decker, E. L., Nehmann, N., Kampen, E., Eibel, H., Zipfel, P. F. and Skerka, C. (2003): Early growth response proteins (EGR) and nuclear factors of activated T cells (NFAT) form heterodimers and regulate proinflammatory cytokine gene expression. Nucl. Acids Res. 31 (3): 911-921. Zipfel, P. F., Nehmann, N., Eibel, H., Siebenlist, U. and Skerka, C. (2003): Early Growth Response Protein EGR-4 interacts with immune inflammatory mediators NF-κB p65 and p50. J. Biol. Chem, in Revision. Nehmann, N., Skerka, C. and Zipfel, P. F. (2003): Localization of the interaction domain of the human zinc finger Protein EGR-4 with NFATc and NFATp. Manuskript in Vorbereitung. Posterpräsentationen und Vorträge Nehmann, N., Skerka, C., Kampen, E. and Zipfel, P. F. (2000): The human EGR-4 Transcription factor: Localization of Protein Domains required for Functional and Physical Interaction with NFAT proteins. 31th Annual Meeting of the German Society of Immunology, Immunobiology 203 (1-3): 120-121. Nehmann, N., Kröncke, K. D., Zipfel, P. F. and Skerka, C. (2001): The synergistic interaction of EGR-4 with both NFATp and NFATc regulates ICAM-1 transcription. Spring-Meeting of the German Society of Immunology. Nehmann, N., Skerka, C., Kröncke, K. D. and Zipfel, P. F. (2001): ICAM-1 gene transcription depends on the cooperation of EGR-3 and NFkappaB p65. 32th Annual Meeting of the German Society of Immunology, Immunobiology 204 (1-2): 24. Skerka, C., Nehmann, N., Kampen, E., Eibel, H. and Zipfel, P. F. (2001): The early growth response protein EGR-4 forms heterodimers with NF-κB proteins and functions as strong activator of gene transcription. 32th Annual Meeting of the German Society of Immunology, Immunobiology 204 (1-2): 29. Nehmann, N., Löschmann, I., Skerka, C. and Zipfel, P. F. (2002): Two domains of the EGR-3 and EGR-4 proteins are required for the interaction with NFATp. 33th Annual Meeting of the German Society of Immunology, Immunobiology 206 (1-3): 137-138. Nehmann, N., Zipfel, P. F., Eibel, H., Siebenlist, U. and Skerka, C. (2003): Early Growth Response Protein EGR-4 interacts with immune inflammatory mediators NF-κB p65 and p50. 34th Annual Meeting of the German Society of Immunology, Immunobiology 208: 122-123. Richter, H., Nehmann, N., Skerka, C. and Zipfel, P. F. (2003): Localization of the interaction domain of the human zinc finger Protein EGR-4 with NFATc and NFATp. 34th Annual Meeting of the German Society of Immunology, Immunobiology 208: 124-125. -i- Selbständigkeitserklärung Selbständigkeitserklärung Hiermit erkläre ich, dass mir die geltende Promotionsordnung der Biologisch-Pharmazeutischen Fakultät der Friedrich-Schiller-Universität Jena bekannt ist, ich die vorliegende Dissertation selbst angefertigt habe und alle benutzten Hilfsmittel, persönlichen Mitteilungen und verwendeten Quellen angegeben habe. Alle Personen, die mich bei der Auswahl des Materials sowie bei der Herstellung des Manuskripts unterstützt haben, habe ich benannt. Ich habe nicht die Hilfe eines Promotionsberaters in Anspruch genommen. Dritte Personen haben weder unmittelbar noch mittelbar geldwerte Leistungen von mir für Arbeiten erhalten, die im Zusammenhang mit dem Inhalt der vorgelegten Dissertation stehen. Die vorliegende Arbeit wurde weder in der Vergangenheit noch gegenwärtig als Dissertation bei einer anderen Hochschule, als Prüfungsarbeit für eine staatliche oder andere wissenschaftliche Prüfung eingereicht. Jena, den 8.Oktober 2003 - ii - Lebenslauf Lebenslauf Persönliche Daten: Schulausbildung: 1981-1985: 1985-1994: Nina Nehmann, geb. 13.12.1974 Witthof 12 22305 Hamburg Grundschule Peter-Petersen, Hamburg Gymnasium Grootmoor, Hamburg Abschluß: Abitur Berufsausbildung: 1994-2000: Studium Diplom-Biologie, Universität Hamburg Hauptfach: Mikrobiologie, Nebenfächer: Genetik, Zoologie Diplomarbeit angefertigt am Bernhardt-Nocht-Institut, Hamburg Abschluß: Dipl. Biol. 2000-2003: Promotionsstudium, Universität Jena Doktorarbeit angefertigt am Hans-Knöll-Institut, Jena voraussichtlicher Abschluß: Dr. rer. nat. Arbeitsverhältnisse: 2001-2002: seit 2002: Wissenschaftliche Angestellte, Universität Hamburg Wissenschaftliche Angestellte, Hans-Knöll-Institut, Jena Praktika: 1998: 1998: Praktikum am Bernhardt-Nocht-Institut, Hamburg Praktikum in der Diagnostik, Bundeswehrkrankenhaus, Hamburg Jena, den 8. Oktober 2003 - iii - Danksagungen Mein besonderer Dank gilt Herrn Prof. Dr. Peter F. Zipfel für die Bereitstellung, Unterstützung und Ermöglichung dieser Arbeit. Mein Dank gilt den namentlich nicht erwähnten Kollegen, die mich auf die eine oder andere Weise unterstützt haben. Bei Frau Dipl. Biol. Monika Wetzker und Gerlinde Heckrodt bedanke ich mich insbesondere für ihre Hilfsbereitschaft bei Problemen im Laboralltag. Ina „Frau“ Löschmann danke ich für die Zusammenarbeit, die ich sehr vermissen werde. Dr. Gerhard Wieland danke ich dafür, dass ich meine Nerven behalten habe. Tamara Manuelian, Frau Dr. Mihály Jόzsi, Sandra Majno und Lars Schmiedeberg danke ich sowohl für die thematischen als auch privaten Diskussionen. Morten Pöhls danke insbesondere ich für die liebevolle Unterstützung während der letzten 1004 Tage.