Optophysiologische Methoden in der Neurobiologie
Werbung

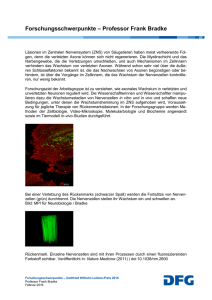
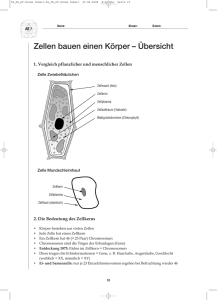
730_775_BIOsp_0707.qxd:730_775 746 07.11.2007 14:11 Uhr Seite 746 MET H ODE N & AN WE N DU NGEN Optophysiologie Optophysiologische Methoden in der Neurobiologie ANDRÉ FIALA THEODOR-BOVERI-INSTITUT, LEHRSTUHL FÜR GENETIK UND NEUROBIOLOGIE, JULIUS-MAXIMILIANS-UNIVERSITÄT WÜRZBURG In den letzten Jahren wurde eine Reihe von Proteinen beschrieben, die es ermöglichen, neuronale Aktivität und molekulare Veränderungen in genetisch definierten Populationen von Nervenzellen optisch darzustellen. Mehr noch: Kürzlich wurden lichtsensitive Proteine entdeckt, die es erlauben, Nervenzellen durch Beleuchtung zu aktivieren oder zu inhibieren. In Anlehnung an den Begriff Elektrophysiologie umschreibt der Begriff Optophysiologie die Möglichkeit, durch diese neuartigen optischen Werkzeuge der Funktionsweise neuronaler Verschaltungen nachzugehen. Anhand transgener Modellorganismen wie der Taufliege Drosophila melanogaster wird mit diesen Methoden versucht, das komplexe Zusammenspiel von Nervenzellen in einem Gehirn zu entschlüsseln. ó Wie arbeiten komplexe Verbände von Nervenzellen in Gehirnen zusammen, um ein sinnvolles Verhalten des Tiers zu erzeugen? Um dieser Frage nachzugehen, verfolgen Neurophysiologen in der Regel zwei komplementäre Ansätze. Zum einen wird die Aktivität von Nervenzellen während eines Verhaltens oder auf einen verhaltensauslösenden Reiz hin beobachtet. Zum anderen wird Aktivität in Nervenzellen induziert, um deren Funktion aufzuklären. Das klassische Werkzeug, die Aktivität von Neuronen und Neuronenverbänden zu untersuchen, ist seit Jahrzehnten die Elektrode. Mithilfe von Ableitelektroden können Veränderungen von Membranpotenzialen mit optimaler zeitlicher Auflösung und hoher räumlicher Präzision in ein- ¯ Abb. 1: Visualisierung neuronaler Aktivität im Gehirn der Taufliege Drosophila. A, Der Calciumsensor Cameleon[2]. Ein enhanced cyan fluorescent protein (ECFP) und ein enhanced yellow fluorescent protein (EYFP) wurden mit einer Calmodulin-Sequenz und dem Calmodulin-Zielpeptid M13 so fusioniert, dass CalciumBindung zu einer Konformationsänderung des Proteins führt. Bei Anregung des ECFP (∼ 436 nm Wellenlänge) ergeben sich für den Calciumfreien und den Calcium-bindenden Zustand des Proteins unterschiedlich starke Emissionen der Fluorochrome, sodass der intrazelluläre Calciumspiegel beobachtet werden kann. B, Die Taufliege wird so unter dem Mikroskop befestigt werden, dass durch ein kleines, in den Kopf geschnittenes Fenster die den Fluoreszenzsensor exprimierenden Nervenzellen beobachtet werden können. C, Der Sensor kann im Drosophila-Gehirn in den Nervenzellen zur Expression gebracht werden, die einer genaueren Untersuchung zugrunde liegen sollen. In dieser dreidimensionalen Rekonstruktion des Drosophila-Gehirns sind Nervenzellen rot markiert, die bei der Duftprozessierung eine Rolle spielen. D, Die Verzweigungen dieser Zellen lassen sich unter dem Mikroskop durch die Fluoreszenz gut sichtbar machen. E, Die durch einen Duftstoff evozierte neuronale Aktivität wird durch die Fluoreszenzänderung des Calciumsensors sichtbar. Hier werden Bereiche hoher Aktivität rot, Bereiche niedriger Aktivität dunkelblau dargestellt. BIOspektrum | 07.07 | 13. Jahrgang 730_775_BIOsp_0707.qxd:730_775 07.11.2007 zelnen Nervenzellen oder Verbänden von Nervenzellen gemessen werden. Umgekehrt dienen Stimulationselektroden zur Induzierung von Nervenzellaktivität. Diese Methoden bringen allerdings eine Einschränkung mit sich. In der Regel sind viele Nervenzellen unterschiedlicher Typen und Funktionen – aktivierende, inhibierende und modulatorische Neurone – in dichten Netzen und Schaltkreisen miteinander verbunden. Damit ist die Anwendung von elektrophysiologischen Methoden durch die Präzision und die Anzahl an Ableitorten limitiert. Dies ist insofern problematisch, als Information in Nervenzellverbänden häufig als Aktivitätsmuster von bestimmten Neuronenpopulationen kodiert wird, die in ein dichtes neuronales Netz eingebettet sind. Lange Zeit war es ein Traum der Neurowissenschaftler, die Aktivität von vielen Neuronen zu detektieren und zu manipulieren, die durch eine gemeinsame Funktion gekennzeichnet sind. Eine Lösung dieses Problems zeichnete sich durch die Verbindung von neurophysiologischen Fragen mit genetischen Methoden ab. Nervenzellen eines bestimmten Typs sind oft durch ein gemeinsames Muster an Genexpression gekennzeichnet. Unter diesem Gesichtspunkt erstaunt es nicht, dass die Entwicklung genetisch kodierter Sonden zur Visualisierung neuronaler Aktivität mit großer Begeisterung aufgenommen wurde[1]. Der entscheidende Aspekt dieser Sonden beruht darauf, dass sie als Proteine DNA-kodiert sind und vom Organismus selbst exprimiert werden können, unter Umständen in ganz klar definierten Nervenzelltypen. Visualisierung neuronaler Aktivität mithilfe DNA-kodierter Sonden Die ersten beschriebenen DNA-kodierten Sonden waren Calciumsensoren und Fluoreszenzproteine, die eine Visualisierung synaptischer Transmission erlauben[1]. Ein mittlerweile klassischer Fluoreszenzsensor ist in Abbildung 1A dargestellt, der Calciumindikator Cameleon[2]. Dieser besteht aus Varianten des grün fluoreszierenden Proteins (GFP), einer gelb fluoreszierenden Variante (EYFP) und einer cyan fluoreszierender Variante (ECFP). Durch die Fusion dieser beiden Fluorochrome mit einer Calcium-bindenden Calmodulin-Sequenz und einem Calmodulin-bindenden Peptid entsteht ein BIOspektrum | 07.07 | 13. Jahrgang 14:11 Uhr Seite 747 Molekül, das seine Konformation und damit seine Fluoreszenzeigenschaften in einer Calcium-abhängigen Weise verändert. Da neuronale Aktivität mit einem Anstieg der intrazellulären Calciumkonzentration einhergeht, spiegeln die relativen Emissionsintensitäten der zwei Fluorochrome neuronale Aktivität wider, wenn der Sensor im Zytoplasma exprimiert wird. Mittlerweile wurde eine ganze Reihe solcher genetisch kodierter Calciumsensoren beschrieben, die meist auf ähnlichen Prinzipien beruhen. Wir verwenden Drosophila melanogaster als Modellorganismus, um die Aktivität spezifischer Nervenzellen in einem lebenden Organismus zu visualisieren[3, 4]. Die Vorteile der Taufliege gegenüber anderen neurobiologischen Modellorganismen sind vielfältig. Erstens besteht das DrosophilaGehirn aus nur etwa 100.000 Nervenzellen – eine sehr überschaubare Größenordnung im Vergleich zu den Milliarden an Nervenzellen des Mäuse- oder Rattengehirns. Zweitens lassen sich Transgene, wie z. B. Fluoreszenzsensoren, sehr einfach in fast jedem beliebigen Neuronentyp zur Expression bringen, was beispielsweise bei Mäusen mit erheblich größerem Aufwand verbunden ist. Und drittens befähigt das winzige Drosophila-Gehirn das Tier zu erstaunlich komplexen Verhaltensleistungen, die z. B. auf Lernvorgängen und Gedächtnisbildung beruhen – Prozesse, die bislang nur rudimentär verstanden sind. Abbildung 1B zeigt eine Taufliege, die unter einem Mikroskop fixiert wurde. Indem man ein kleines Fenster in die Kopfkapsel der Fliege schneidet, bekommt man einen optischen Zugang zu der Population von Nervenzellen, die im Fokus des Interesses steht. Wie Abbildung 1C verdeutlicht, sind weite Bereiche des DrosophilaGehirns anatomisch schon gut beschrieben. Als Beispiel sieht man in Abbildung 1D die Verzweigungen einer Gruppe von Nervenzellen, die an der Verarbeitung von Duftinformation beteiligt sind. Präsentiert man nun der Taufliege einen Duftstoff, kann man die Aktivität dieser Nervenzellen durch die Fluoreszenzänderung des Calciumsensors in sehr hoher räumlicher Auflösung verfolgen, wie es in Abbildung 1E anhand eines Falschfarben-kodierten Aktivitätsmusters dargestellt ist. Der Einsatz genetisch kodierter Fluoreszenzsensoren erlaubt es also, die Aktivität von Neuronen direkt im lebenden Tier zu 730_775_BIOsp_0707.qxd:730_775 748 07.11.2007 14:11 Uhr Seite 748 MET H ODE N & AN WE N DU NGEN ˚ Abb. 2: Optische Manipulation neuronaler Aktivität durch lichtabhängige Proteine. A, Channelrhodopsin-2 bildet einen Kanal aus sieben Transmembran-Domänen, der sich in die Zellmembran integriert. Bei Anwesenheit des Kofaktors all-trans- Retinal wird dieser Kanal lichtsensitiv, d. h. er öffnet bei Beleuchtung mit blauem Licht. Da der Kanal durchlässig für Kationen ist, kann damit die Zellmembran depolarisiert und die Nervenzelle aktiviert werden[5]. B, Umgekehrt wirkt die lichtsensitive Chloridpumpe Halorhodopsin. Der lichtabhängige Anstieg der Chloridkonzentration im Zellinneren führt zu einer Hyperpolarisation der Zellmembran, wodurch die Nervenzelle inhibiert wird[9]. C, Während man mit elektrophysiologischen Techniken mittels einer Stimulationselektrode mehr oder minder weite Bereiche innerhalb eines Nervengewebes aktivieren kann, erlauben es einem diese neuen optophysiologischen Zugänge, ganz gezielt nur die Nervenzellen zu stimulieren, die das lichtabhängige Protein exprimieren. beobachten. Damit ist aber nur eine Seite der Medaille beschrieben. Welche Funktion hat diese neuronale Aktivität für das Verhalten des Tiers? Diese Frage kann dadurch geklärt werden, dass man die Neuronen direkt aktiviert und die Auswirkung auf das Verhalten des Tiers beobachtet. Manipulation neuronaler Aktivität: lichtinduzierte Aktivierung und Inhibition Neben Sonden zur Beobachtung neuronaler Aktivität wurden Proteine beschrieben, die eine lichtabhängige Manipulation neuronaler Aktivität erlauben. Konzeptionell wurde diese Idee schon früh formuliert. Es blieb aber lange ein Traum der Neurobiologen, durch einen Reiz, wie z. B. Licht, ganz gezielt bestimmte Nervenzellgruppen zu aktivieren oder stillzulegen. Der Durchbruch des Konzepts wurde mit der Beschreibung des lichtsensitiven Proteins Channelrhodopsin-2 erreicht[5]. Dieses Protein, das aus der einzelligen Grünalge Chlamydomonas reinhardtii isoliert wurde[5], besteht aus sieben Transmembran-Domänen, die einen selektiven Kanal für Kationen bilden (Abb. 2A). Eine Bindungsdomäne für all-trans-Retinal vermittelt die lichtabhängige Öffnung des Kanals. Exprimiert man dieses Protein in kultivierten Neuronen, lässt sich das Membranpotenzial durch Lichtblitze manipulieren: Es kommt zu einer sehr schnellen Depolarisation und damit zur Generierung von Aktionspotenzialen[6]. Man kann also Neuronen fast wie mit einer Fernbedienung anund ausschalten. Die Funktionalität dieser Methode wurde kürzlich anhand von larvalen und adulten Taufliegen nachgewiesen[7, 8]. So konnte die Rolle bestimmter Neuronen bei dem Prozess des assoziativen Duftlernens aufgeklärt werden[7]. Zudem wurde kürzlich ein weiteres lichtsensitives Protein beschrieben, das antagonistisch wirkt[9]. Dabei handelt es sich um die Chloridpumpe Halorhodopsin, das aus dem Halobakterium Natronomonas pharaonis isoliert wurde. In diesem Fall führt die Beleuchtung des Proteins zu einer Erhöhung der Chloridkonzentration im Zellinneren (Abb. 2B). Die Folge ist ein Abfall des Membranpotenzials, eine Hyperpolarisation, und damit eine Inhibition der Nervenzellaktivität[9]. Diese Proteine bieten die interessante Aussicht, Nervenzellaktivität bidirektional durch Licht zu manipulieren, und zwar in definierten Populationen innerhalb eines neuronalen Verbunds[9]. Während konventionelle elektrophysiologische Techniken eine Stimulation von Nervenzellen über ein mehr oder minder ausgedehntes Areal des Gewebes erlauben (Abb. 2C), könnte es in Zukunft möglich sein, durch gezielte Expression lichtsensitiver Proteine Aktivität in solchen Nervenzellen zu stimulieren oder zu inhibieren, welche durch eine gemeinsame genetische Identität und damit möglicherweise eine gemeinsame Funktion gekennzeichnet sind. Zukunftsaussichten Die beschriebenen Techniken der optophysiologischen Registrierung und Manipulation neuronaler Aktivität lassen sich natürlich gut an einfachen Testsystemen, wie dem Gehirn von Drosophila oder dem Nervensystem des Fadenwurms C. elegans, testen. In Zukunft wird es spannend sein zu sehen, inwieweit transgene Mäuse für derartige Techniken zugänglich sind. Neurobiologische Modellorganismen, die „näher am Menschen“ sind, lassen es in Zukunft möglich erscheinen, die derzeit entwickelten optphysiologischen Methoden zur Klärung neuronaler Erkrankungen zu nutzen[10]. Danksagung Ich danke Herrn Prof. Dr. Erich Buchner und Herrn Prof. Dr. Martin Heisenberg für die Unterstützung meiner Forschung. ó Literatur [1] Miyawaki, A. (2003): Fluorescence imaging of physiological activity in complex systems using GFP-based probes. Curr. Opin. Neurobiol. 13: 591–596. [2] Miyawaki, A., Llopis, J., Heim, R., McCaffery, J. M., Adams, J. A., Ikura, M., Tsien, R. Y. (1997): Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature 388: 882–887. [3] Fiala, A., Spall, T., Diegelmann, S., Eisermann, B., Sachse, S., Devaud, J. M., Buchner, E., Galizia, C. G. (2002): Genetically expressed cameleon in Drosophila melanogaster is used to visualize olfactory information in projection neurons. Curr. Biol. 12: 1877–1884. [4] Fiala, A., Spall, T. (2003): In vivo calcium imaging of brain activity in Drosophila by transgenic cameleon expression. Sci STKE 174, pl 6. [5] Nagel, G., Szellas, T., Huhn, W., Kateriya, S., Adeishvili, N., Berthold, P., Ollig, D., Hegemann, P., Bamberg, E. (2003): Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proc. Natl. Acad. Sci. U S A 100: 13940– 13945. [6] Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. (2005): Millisecond-timescale, genetically targeted optical control of neural activity. Nat. Neurosci. 8: 1263– 1268. [7] Schroll, C., Riemensperger, T., Bucher, D., Ehmer, J., Völler, T., Erbguth, K., Gerber, B., Hendel, T., Nagel, G., Buchner, E., Fiala, A. (2006): Light-induced activation of distinct modulatory neurons triggers appetitive or aversive learning in Drosophila larvae. Curr. Biol. 16: 1741–1747. [8] Suh, G. S., Ben-Tabou de Leon, S., Tanimoto, H., Fiala, A., Benzer, S., Anderson, D. J. (2007): Light activation of an innate olfactory avoidance response in Drosophila. Curr. Biol. 17: 905–908. [9] Zhang, F., Wang, L. P., Brauner, M., Liewald, J. F., Kay, K., Watzke, N., Wood, P. G., Bamberg, E., Nagel, G., Gottschalk, A., Deisseroth, K. (2007): Multimodal fast optical interrogation of neural circuitry. Nature 446: 633–639. [10] Zhang, F., Aravanis, A. M., Adamantidis, A., de Lecea, L., Deisseroth, K. (2007): Circuit-breakers: optical technologies for probing neural signals and systems. Nat. Rev. Neurosci. 8: 577–581. Korrespondenzadresse: Dr. André Fiala Theodor-Boveri-Institut Lehrstuhl für Genetik und Neurobiologie Biozentrum der Julius-Maximilians-Universität Würzburg Am Hubland D-97074 Würzburg Tel.: 0931-888 4482 Fax: 0931-888 4452 [email protected] burg.de BIOspektrum | 07.07 | 13. Jahrgang