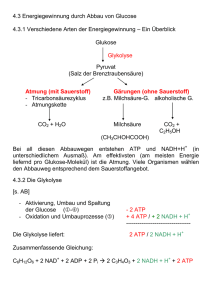
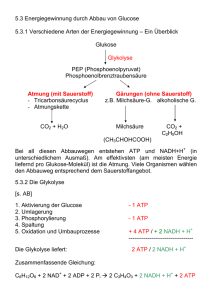
ATP-Synthese
Werbung

Biochemie für Lehramtsstudierende Inhalt 1. Einführung 2. Die Zelle 3. Aminosäuren 4. Peptide, Proteine, Enzyme 5. Kohlenhydrate (Sacharide) 6. Nukleinsäuren 7. Lipide Literatur 1. W. Müller-Esterl, Biochemie – Eine Einführung für Mediziner und Naturwissenschaftler, 2. Aufl., Spektrum-Verlag 2010 2. J. M. Berg, J. L. Tymoczko, L. Stryer, Stryer Biochemie, 7. Aufl., Spektrum-Verlag 2014 3. M. Königshoff, T. Brandenburger, Kurzlehrbuch Biochemie, 3. Aufl., Thieme-Verlg 2012 4. B. Alberts, A. Johnson, J. Lewis, M. Raff, K. Roberts, P. Walter, Molekularbiologie der Zelle, 5. Aufl., Wiley-VCH 2011 5. D. J. Voet, J. G. Voet, C. W. Pratt, Lehrbuch der Biochemie, 2. Aufl., Wiley-VCH 2010 Vorlesung: Beginn: 17.10.2017 (nicht am 31.10.17 und am 02.01.18) Klausur: 27. Februar 2018 Nachklausur: 20.März 2018 Voraussetzung: bestandene OC01 Vorlesung wird im WS 17/18 zum letzten Mal angeboten; Prüfungen auch danach! 1. Einführung Biochemie ist die Chemie des Lebens. Sie untersucht, wie Lebewesen auf molekularer Ebene funktionieren. 1.1 Themengebiete der Biochemie - Biomoleküle (Synthese, Bereitstellung, Wechselwirkung) - Stoffwechsel, Bioenergetik, Biokatalysatoren - Informationsaustausch in und zwischen Organismen 1.2 Wesentliche Stoffgruppen (Biomoleküle) Proteine, Kohlenhydrate (nebst Derivaten), Lipide, Nukleinsäuren 2. Die Zelle 2.1 Die tierische Zelle Aufbau einer tierischen Zelle 1. Nucleolus; 2. Zellkern; 3. Ribosomen; 4. Vesikel; 5. Rauhes Endoplasmatisches Retikulum; 6. Golgi-Apparat; 7. Mikrotubuli; 8. Glattes Endoplasmatisches Retikulum; 9. Mitochondrien; 10. Lysosomen; 11. Cytoplasma (Cytosol); 12. Peroxysomen; 13. Zentriolen 2. Die Zelle 2.1 Die tierische Zelle Zellkern: enthält Großteil des Erbmaterials in Form von DNA (Bauplan der Zelle) Mitochondrien: Kraftwerke der Zelle, Ort der Energiegewinnung Zellmembran / Cytoplasmamembran: Hydrophobe Barriere um die Zelle, bestehend aus Lipiddoppelschicht und Membranproteinen Ribosomen: Komplexe, die Proteinsynthese der Zelle durchführen 2. Die Zelle 2.2 Die pflanzliche Zelle → im Wesentlichen ähnlicher Bau wie tierische Zelle → augenfälligste Unterscheide: Zellwand, Zellsaftvakuole, Chloroplasten Chrloroplasten: Ort der Photosynthese Vakuole: Stoffspeicherung und Zellstabilisierung Zellwand: Zellstabilisierung 2. Die Zelle 2.3 Die Bakterienzelle → deutlich unterschiedlicher Zellaufbau im Vergleich zu Tieren / Pflanzen → augenfälligste Unterscheide: kein Zellkern, keine Organellen → nahezu alle zellulären Prozesse im Cytoplasma 3. Aminosäuren 3.1 Konstitution der Aminosäuren In (fast) allen Aminosäuren, die für die Biochemie wichtig sind, ist ein zentrales CAtom mit einer Aminogruppe –NH2, mit einer Carboxylgruppe –COOH, mit einem Wasserstoffatom –H und mit einer Seitenkette –R verknüpft. Zur Erinnerung: Die Konstitution einer Substanz beschreibt nur die Verknüpfungsverhältnisse zwischen den Atomen, ohne auf die räumliche Struktur einzugehen. NH2 R C COOH Allgemeine Konstitutionsformel für Aminosäuren H -> α – Aminosäuren: Aminogruppe in α – Stellung zur Carboxylgruppe Alle bekannten Aminosäuren unterscheiden sich in der Seitenkette R. R kann ein Alkylrest sein, ein Arylrest oder ein Heterocyclus. In der Natur kommen ca. 20 verschiedene Aminosäuren extrem häufig vor (nämlich die Protein-bildenden), weitere 10 sind eher selten, spielen aber in gewissen Bereichen der Biochemie eine große Rolle. 3.2 Konfiguration der Aminosäuren Alle Aminosäuren können prinzipiell in zwei verschiedenen Konfigurationen vorkommen: COOH H2N H = C COOH R H C NH2 H2N H = C Fischer-Projektion Keil-Strich-Formel D-Aminosäure = C R COOH CIP: R-Aminosäure COOH H NH2 H2N COOH R R C H R Fischer-Projektion L-Aminosäure H NH2 = C R COOH Keil-Strich-Formel CIP: S-Aminosäure Zur Erinnerung: Die Konfiguration einer Substanz beschreibt die räumliche Anordnung der Atome im Molekül. Eine Substanz ist chiral, wenn die Moleküle keine Spiegelebene und keine Inversionszentren besitzen. Fischer - Projektion - Methode zur eindeutigen 2-dimensionalen Darstellung chiraler Verbindungen - Chiralität: Eigenschaft eines Objektes, keine Drehspiegelachse zu besitzen - Beispiel: C – Atom mit vier verschiedenen Substituenten: NH2 R C COOH H Stereozentrum (Chiralitätszentrum) -Regeln der Fischer-Projektion: - Kette von C-Atomen von oben nach unten zeichnen, dabei das am stärksten oxidierte C-Atom nach oben - Horizontale Linien zeigen aus der Projektionsebene - Vertikale Linien laufen hinter die Projetionsebene, vom Betrachter weg Fischer - Projektion z.B. Glyzerinaldehyd: L-Enantiomer: (L für laevus, links) funtionelle Gruppe mit höherer Priorität (hier –OH) im untersten Stereozentrum zeigt nach links D-Enantiomer: (D für dexter, rechts) funtionelle Gruppe mit höherer Priorität (hier –OH) im untersten Stereozentrum zeigt nach rechts Fischer - Projektion z.B. Glyzerinaldehyd: Vorsicht: aus den Bezeichnungen L und D kann NICHT auf die Eigenschaften rechtsdrehend bzw. linksdrehend geschlossen werden! Die Begriffe rechtsdrehend und linksdrehend beziehen sich auf die Änderung der Polarisationsebene von linear polarisiertem Licht und wird mit (+) bzw. (-) gekennzeichnet: a) L-(-) –Glyzerinaldehyd und D-(+)-Glyzerinaldehyd b) L-(+)-Weinsäure und D-(-) Weinsäure D-(-)-Weinsäure 3.3 Übersicht über die wichtigsten Aminosäuren Aminosäuren mit Alkylseitenkette (in der Fischer-Projektion) COOH H2N H H Glycin Gly COOH H2N H COOH H2N H COOH H2N H COOH H2N H H Alanin Ala Valin Val Leucin Leu Isoleucin Ile Aminosäuren mit Arylseitenkette (in der Fischer-Projektion) COOH H2N H COOH H2N H HO Phenylalanin Phe Tyrosin Tyr D oder L? Aminosäuren mit heterocyclischen Seitenkette (in der Fischer-Projektion) COOH H2N H H N COOH H N COOH H N OH N COOH H2N H N H Histidin His Prolin Pro Hydroxyprolin Tryptophan Trp Aminosäuren mit OH in der Seitenkette (in der Fischer-Projektion) COOH H2N H HO Serin Ser COOH H2N H H OH Threonin Thr Aminosäuren mit Schwefel in der Seitenkette (in der Fischer-Projektion) COOH H2N H HS COOH H2N H S S H Cystein Cys COOH H2N H SCH3 NH2 COOH Cystin Methionin Met Aminosäuren mit Stickstoff in der Seitenkette (in der Fischer-Projektion) COOH H2N H COOH H2N H H NH2 Lysin Lys H N N NH2 Arginin Arg Aminosäuren mit Carboxylgruppen in der Seitenkette (in der Fischer-Projektion) COOH H2N H HOOC COOH H2N H COOH H2N H COOH H2N H H2NOC HOOC Asparaginsäure Asp Asparagin Asn H2NOC Glutaminsäure Glu Glutamin Gln Essentielle Aminosäuren sind Aminosäuren, die der Mensch nicht selbst erzeugen kann. Diese müssen unbedingt mit der Nahrung in ausreichender Menge aufgenommen werden, um Mangelerscheinungen vorzubeugen. Arg, His, Ile, Leu, Lys, Met, Phe, Thr, Trp, Val Neben den ca. 20 sehr häufig vorkommenden Aminosäuren gibt es noch über 100 sehr viel seltener vorkommende Aminosäuren, die oft in verschiedensten Lebewesen ganz spezielle Aufgaben übernehmen. 3.4 Säure-Base-Eigenschaften von Aminosäuren Das ideale Lösungsmittel lebender Organismen ist Wasser. Aminosäuren lösen sich gut in Wasser, in dem sie als Zwitterionen vorliegen: Die protonierte Aminogruppe kann hier als Säure (Protonendonator), die Carboxalatgruppe als Base (Protonenakzeptor) dienen. Die Ladungsverhältnisse in einem Aminosäuremolekül hängen vom pH-Wert ab: Kation Zugabe Säure Neutrales Molekül Anion Zugabe Base Isoelektrischer Punkt: = pH-Wert, bei dem gleich viele Säuregruppen negativ, wie Aminogruppen positiv geladen sind - jede Aminosäure hat einen charakteristischen isoelektrischen Punkt Beispiel Glycin: Abhängigkeit der Nettoladung vom pH-Wert Isoelektrische Punkt pI oder pHI : znetto = 0 pI = ½(pK1+pK2) Glycin: pI = 5,97; d.h. bei pH 5,97 wandert die Aminosäure Glycin nicht in einem elektrischen Feld. Säure-Base-Verhalten bei proteinogenen Aminosäuren: Im Protein interessiert im Wesentlichen das Säure-Base-Verhalten des Restes R Im Protein Peptidbindung zur nächsten AS Im Protein Peptidbindung zur nächsten AS „freier“ Rest R z.B. Tetrapeptid Ala – Ser – Gly - Leu R1 R3 R2 R4 Über pKS-Werte (oft kurz pK-Wert) kann der Säure-Base-Charakter der funktionellen Gruppen von Aminosäuren charakterisiert werden. Der pK-Wert ist der pH-Wert, bei dem eine titrierbare funktionelle Gruppe in gleichen Teilen protoniert und deprotoniert vorkommt. z.B. L-Glutaminsäure: pKR 4,25 pK1 2,19 pK2 9,67 3.5 Nicht-proteinogene Aminosäuren = Aminosäuren, die nicht im Zuge der Translation in Proteine eingebaut werden - diese Aminosäuren besitzen keinen DNA-Code a) Bildung durch posttranslationale Modifikation: z.B. L-Cystin -> wichtiger Bestandteil des Keratin (Faserproteine, aus denen Haare, Horn, Hufe... gebildet werden b) Nicht-proteinogene Aminosäuren mit Stoffwechselfunktion z.B. das Schilddrüsenhormon L-Thyroxin Bei den nicht-proteinogenen Aminisäuren kommen auch D-Aminosäuren vor, z.B. β-Alanin (Abbauprodukt von Nukleinsäuren): 3.6 Biosynthese von Aminosäuren Zur Biosynthese von Aminosäuren wird Sticksoff benötigt, der in der Luft zwar reichlich vorhanden ist, aber: • Nur wenige Lebewesen, z.B. Knöllchenbakterien bei Erbsen, sind in der Lage, N2 aus der Luft in NH3 umzuwandeln (108 t/a (!)). NADH +H+ + ATP + H2O N N NH3 –NAD+ – ADP – P Tiere und Pflanzen können Stickstoff nur in reduzierter oder oxidierter Form verwerten, z.B. NH4+, NO3-, NO2-, organische N-Verbindungen. Pflanzen können alle proteinogenen Aminosäuren selbst synthestisieren, Tiere nur die nicht-essentiellen • Die zentrale Rolle bei der Biosynthese von Aminosäuren spielen Glutaminsäure Glu und Glutamin Gln. O HOOC NH3 COOH α-Ketoglutarsäure (aus Citratcyclus) – H2O H+ +H– NH HOOC COOH Glutamat-Dehydrogenase +NADPH HOOC COOH – H2O COOH L-Glutaminsäure NH4+ aus Harnstoffzyklus -> Entgiftung NH3 NH2 HOOC NH2 NH2 H2NOC L-Glutaminsäure Glutamin-Synthase +ATP COOH L-Glutamin • Pflanzen können alle Aminosäuren aus den entsprechenden Vorstufen und NH3 aufbauen. • Alle nicht-essentiellen Aminosäuren baut der tierische Organismus mit Hilfe von Glutaminsäure und Glutamin als NH3-Quelle entsprechend den aus Glykolyse angegebenen mechanistischen Details auf. aus Citrat-Cyclus O O COOH Brenztraubensäure HOOC OH O COOH Oxalessigsäure HOOC COOH Ketoglutarsäure (Pyruvat) (HO)2P(O)O COOH Glycerinsäure-3-Phosphat (3-Phosphoglycerat) aus Glykolyse NH2 Valin COOH NH2 NH2 HOOC COOH HOOC COOH NH2 HO COOH Leucin Alanin Asparaginsäure Lysin Cystein Methionin Isoleucin Threonin CONH2 Asparagin Phenylalanin Tyrosin Serin Glutaminsäure NH2 HOOC Tryptophan NH2 HOOC Glutamin CONH2 H2N COOH Glycin Arginin Prolin Sehr gute Beschreibung: McMurry et al., „Organische Chemie der biologischen Stoffwechselwege“, Kap. 5. 3.7 Funktion von Aminosäuren • Bausteine für Proteine (aus AS aufgebaute Polymere) • Neurotransmitter (dienen der Reizleitung an Synapsen; z.B. Glutaminsäure) • Vorstufen für die Synthese von Nukleinbasen (Bausteine v. DNA und RNA) • Vorstufen für die Synthese von Polyaminen wie Spermidin (dienen häufig der Stabilisierung von Nukleinsäuren) • Vorstufen für die Synthese von Glutathion (dient als Redox-Puffer, Cystein-Speicher und zur Entgiftung) • Vorstufe für die Synthese von Kreatin (Energieversorgung v. Muskeln) • Vorstufe für die Synthese von Porphyrinen (z.B. Chlorophyll, Häm) • Vorstufe für die Synthese von Alkaloiden • Vorstufe für die Synthese von N-haltigen sekundären Naturstoffen Spermidin Glutathion Kreatin 4. Peptide, Proteine, Enzyme 4.1 Peptidbindung und Peptide • Zwei Aminosäuren können prinzipiell unter Wasserabspaltung miteinander reagieren. H H N O R O 1 H H H N O O R 2 H H H N – H2O R2 O R N H 1 O H O Amidbindung = Peptidbindung • Die neu geknüpfte Bindung nennt man Amidbindung oder (eher in der Biochemie) Peptidbindung. • Die neu entstandene Verbindung aus zwei Aminosäuren nennt man Dipeptid. H H N R2 O R1 N H H N O Tripeptid O O R3 H H H N R2 O R1 N H H N O R4 O R3 N H Tetrapeptid O O H Allgemein: H H N Ri O R1 N H H N O O H i = 2, 3, …, n Rn+2 O n N-Terminus C-Terminus • n = 0,…,7 : Oligopeptide • n = 8,…,98 : Polypeptide • n > 98 : Proteine • Enzyme = Proteine, die als Katalysatoren für biochemische Reaktionen wirken Peptide: (von ‚peptos‘, verdaut) - Organische Verbindung, bei der bis zu 100 Aminosäuren über Peptidbindungen meist linear zu einer Kette verbunden sind - In der Regel α-Peptidbindung wie in Proteinen: Verknüpfung von LAminosäuren über α-Amino- und α-Carboxylgruppe - Ausnahmen: z.B. Peptide mit ω-Peptidbindung: Verknüpfung über endständige Amino- bzw. Carboxalgruppen, z.B. mit L-Lysin oder L-Glutaminsäure Peptide: (von ‚peptos‘, verdaut) ω-Peptidbindung z.B. Gluthation: α -Peptidbindung = Pseudo-Tripeptid aus L-Glutaminsäure, L-Cystein und Glycin (u.a. entgiftendes Antioxodans; Cystein-Reserve) Peptid-Synthese a)Ribosomale Synthese an Ribosomen (-> Proteinsynthese) - auf der Basis eines genetischen Codes werde L-Aminosäuren durch α-Peptidbindungen verknüpft Peptid-Synthese b) Nichtribosomale Synthese mittels Enzymen - manche Peptide werden nicht mittels Ribosomen, sondern über enzymatisch katalysierte Reaktionen synthetisiert - auf diesem Weg können auch D-Aminosäuren eingebaut werden, ω-Peptidbindungen geknüpft werden oder cyclische Peptide synthetisiert werden - z.B. Gluthationsynthese über die Enzyme Glutamatcysteinligase und Glutationsynthase Cyclische Peptide, Cyclopeptide -ringförmige Peptide -chemisch sind sie Lactame (cyclische Amide) -oft stabiler gegen Hitze und enzymatischen Abbau als lineare Peptide -z.B. Amanitine im Knollenblätterpilz: 4.2 Proteine – Synthese und Struktur Allgemeines: - Proteine (Eiweißstoffe) sind aus Aminosäuren aufgebaute biologische Makromoleküle - die meisten Proteine (proteios – grundlegend) bestehen aus 100-300 L-Aminosäuren - größtes Protein beim Mensch: Muskelprotein Connectin, über 30.000 AS - die molekulare Größe von Proteinen wird meist in Kilodalton (kDa) angegeben: Atomare Masseneinheit: 1 Da = 1 u = 1/12 der Masse eines 12C-Atoms z.B. Connectin: 3600 kDa pro Aminosäure rechnet man im Schnitt mit 110 Da 2.2.2. Proteine – Synthese und Struktur Proteinbiosynthese: - wird später ausführlich dargestellt - für alle Lebewesen der zentrale Prozess der Genexpression: DNA Transkription im Zellkern mRNA Translation Protein im Cytoplasma Modifikation ‚reifes‘ Protein 4.2 Proteine – Synthese und Struktur Proteinbiosynthese: - die Reihenfolge der Aminosäuren im Protein wird durch den genetischen Code der DNA festgelegt - die Basenfolge der DNA wird in zwei Schritten in eine Folge von Aminosäuren übersetzt - je drei aufeinanderfolgende Basen kodieren eine Aminosäure: 4.2 Proteine – Synthese und Struktur Proteinstruktur • Um die Struktur von Peptiden, Proteinen und Enzymen zu bestimmen, muss man vier verschiedene Fragen beantworten: a) in welcher Reihenfolge liegen die Aminosäuren im Protein vor? Aminosäuresequenz = Primästruktur b) welche Konformation (räumliche Anordnung drehbarer Bindungen) nimmt die Aminosäurekette an (z.B. auf Grund von Wasserstoffbrückenbindungen zwischen einzelnen Aminosäuren) Konformation der Aminosäurekette = Sekundärstruktur c) welche Faltung nimmt die Aminosäurekette an (z.B. auf Grund von Disulfidbrücken zwischen Cystein-Molekülen innerhalb der Kette)? Faltung der Aminosäurekette = Tertiärstruktur d) Lagern sich mehrere Aminosäureketten zu einem größeren Proteinkomplex zusammen? Proteinkomplex aus mehreren Proteinen = Quartärstruktur a) Primärstruktur von Proteinen - als Primärstruktur von Proteinen bezeichnet man die Abfolge (Sequenz) der einzelnen Aminosäuren in der Polypeptidkette -> Aminosäuresequenz - vereinbarungsgemäß schreibt man die Aminosäuresequenz beginnend mit dem N-Terminus (freie α–Aminogruppe) und endend mit dem C-Terminus (freie α–Carboxylgruppe), z.B. C-Terminus N-Terminus Drei-Buchstaben-Code: Val - Ein-Buchstaben-Code: V Gly - Ser G S - Ala A Aufklärung der Primärstruktur von Proteinen: Die Aufklärung der Primärstruktur (Sequenzanalyse) wird in mehreren Schritten durchgeführt. i) Totalhydrolyse des Proteins in einzelne Aminosäuren und qualitative und quantitative Analyse des erhaltenen Aminosäuregemisches. Beispiel: Menschliches Insulin HCl konz. Insulin 120 °C / 6 h Gly Ile Val Glu Gln Cys Ser 4 2 4 4 3 6 3 Leu Tyr Asn Phe His Ala Arg Thr Pro Lys 6 4 3 3 2 1 1 3 1 1 Man kennt nun die qualitative und quantitative Zusammensetzung von Insulin (51 Aminosäuren), kann aber noch nichts über die Reihenfolge der Aminosäuren aussagen. ii) Sequenzanalyse durch Edmann-Abbau (erfolgt immer vom N-Terminus) (≠ Thiocyanat, R-S-C≡N) R1 H N H2N O N C S O Phenylisothiocyanat R 2 R1 S N H pH 8 N H H N O O R2 Thioamid N Cyclisierung S H N O R N C S O R2 1 HCl ... H2N S pH 8 N H N O R1 Analyse, z.B. mittels HPLC (Flüssigchromat.) Phenylthiohydantoin der Aminosäure 1 - Edmann-Abbau: Sequenzierung von 20-50 Aminosäuren - bei längeren Ketten Fehler durch unerwünschte Proteolyse und zu starker Hintergrund durch vorherige Abbauprodukte - Lösung: Zerlegung der Proteine durch enzymatische Spaltung: Enzym Spaltung bei Trypsin Lys, Arg, C-Terminus Chymotrypsin Phe, Trp, Tyr, C-Terminus Pepsin Asp, Glu, Leu, Phe, Trp, Tyr, C-Terminus Thermolysin Leu, Ile, Val, N-Terminus - jetzt können die Bruchstücke im Idealfall komplett sequenziert werde - bei Verwendung mehrerer Spaltenzyme können Bruchstücke mit überlappender Sequenz erhalten werden -> Reihenfolge der Bruchstücke ist ermittelbar - z.B. Spaltung von Insulin mit Chymotrypsin und Thermolysin: Spaltung von Insulin mit Thermolysin ergibt folgende Bruchstücke: Gly Ile Val-Glu-Gln-Cys-Cys-Thr-Ser Ile-Cys-Ser Leu-Tyr-Gln Leu-Glu-Asn-Tyr-Cys-Asn Phe Val-Asn-Glu-His Leu-Cys-Gly-Ser-His Leu Val-Glu-Ala Leu-Tyr Leu-Val-Cys-Gly-Glu-Arg-Gly-Phe-Phe-Tyr-Thr-Pro-Cys-Thr Spaltung von Insulin mit Chymotrypsin ergibt folgende Bruchstücke: Gly-Ile-Val-Glu-Gln-Cys-Cys-Thr-Ser-Ile-Cys-Ser-Leu-Tyr Gln-Leu-Glu-Asn-Tyr Cys-Asn Phe Val-Asn-Gln-His-Leu-Cys-Gly-Ser-His-Leu-Val-Glu-Ala-Leu-Tyr Leu-Val-Cys-Gly-Glu-Arg-Gly-Phe-Phe-Tyr Thr-Pro-Lys-Thr Der erste Schritt beim Edmann-Abbau von Insulin liefert 2 Thiohydantoine, nämlich das von Glycin und das von Phenylalanin. Also besteht Insulin aus 2 miteinander verknüpften Aminosäureketten. Durch Überlagern der einzelnen Bruchstücke erhält man schließlich die Primärstruktur von Insulin. Primärstruktur von Insulin Der erste Schritt beim Edmann-Abbau von Insulin liefert 2 Thiohydantoine, nämlich das von Glycin und das von Phenylalanin. Also besteht Insulin aus 2 miteinander verknüpften Aminosäureketten. Durch Überlagern der einzelnen Bruchstücke erhält man schließlich die Primärstruktur von Insulin: b) Sekundärstruktur von Proteinen - generell bezeichnet man als Sekundärstrukturen regelmäßige lokale Strukturelemente - die Sekundärstruktur von Proteinen ist durch die Primärstruktur vorgegeben (-> theoretisch aus Aminosäuresequenz vorhersagbar) - im Wesentlichen werden Sekundärstrukturen von Proteinen durch Wasserstoffbrücken zwischen CO- und NH-Gruppen des Peptidrückgrates gebildet, die die Konformation maßgeblich beeinflussen: (anziehende Wechselwirkung zwischen dem kovalent gebundenen H der NH-Gruppe und einem freien Elekronenpaar der CO-Gruppe) Sekundäre Strukturelemente von Proteinen: - α-Helix (häufig, s.u.) - π-Helix (sehr selten, 4,4 Aminosäuren pro Windung) - 310-Helix (3 Aminosäuren pro Drehung, Ring aus 10 Bindungen inkl. HBrücken, oft an Enden von α-Helices) - linksgängige α-Kette (Kollagene, <-> rechtsgängige α-Helix ) - β-Faltblatt (häufig, s.u.) - β-Schleife (häufig, s.u.) - Random Coil (keine definierte Sekundärstruktur) Jede Sekundärstruktur weist ihre typischen H-Brücken auf, z.B. diverse Helices: i) α-Helix - häufige, sehr stabile Sekundärstruktur bei Proteinen - rechtsgängig gedrehte Spirale - im Schnitt 3,6 Aminosäuren pro Windung - H-Brückenbindung zwischen CO-Sauerstoff der n. und dem Amidproton der n+4. Aminosäure: - sehr dehnbar - typisch für Faserproteine (Haare, Muskelfasern) - aber auch bei löslichen, globulären Proteinen ii) -Faltblatt -Faltblätter bestehen aus parallelen bzw. antiparallelen Peptidketten - diese sind über H-Brücken miteinander verknüpft - die 3-dimensionale Struktur erinnert an ein gefaltetes Blatt: 2 antiparallele Peptidketten im Faltblatt -die Seitenketten sorgen für eine Zieharmonika-ähnlichen Aufbau der Peptidketten -sehr starre, stabile Struktur - z.B. bei Naturseide (Glanz durch Lichtreflexion an den mehrfachen Schichten) iii) -Schleifen - β-Schleifen bestehen aus 4 Aminosäuren - H-Brücke zwischen 1. und 4. Aminosäure der Schleife - ermöglicht Richtungswechsel in Peptidketten - häufig als Verbindung der parrallelen/antiparallelen Ketten in β-Faltblatt-Strukturen Typ 2: meist R3 = H -> AS 3 = Glycin Vorhersage und Bestimmung von Sekundärstrukturen (Proteine, Peptide) - insbesondere α-Helices lassen sich recht gut vorhersagen - aus Strukturanalysen ist bekannt, welche Aminosäuren eher in α-Helices vorkommen und welche diese verhindern (sterische Gründe, hydrophobe oder hydrophile Wechselwirkungen der Reste) - Bestimmung von Sekundärstrukturen z.B. durch Röntgenstrukturanalyse von kristallisierten Proteinen c) Tertiärstruktur von Proteinen - die Tertiärstruktur eines Proteins spiegelt den übergeordneten räumlichen Aufbau in der Regel einer Peptidkette wider - die Tertiärstruktur beschreibt die Abfolge von Elementen der Sekundärstruktur: Tertiärstruktur von Insulin - die räumliche Struktur ist für die Funktion von Proteinen wesentlich - die Struktur wird v.a. durch hydrophobe / hydrophile Wechselwirkungen bestimmt - stabilisiert wird die Tertiärstruktur zusätzlich durch Disulfidbrücken, Ionenbindungen und H-Brückenbindungen d) Quartärstruktur von Proteinen - mehrere Polypeptidketten oder Proteine können sich zu Komplexen zusammenlagern - man spricht dann von der Quartärstruktur der Proteine: - der Komplex setzt sich aus den Untereinheiten zusammen - die Untereinheiten können durch hydrophobe Wechselwirkungen, Ionenbindungen, H-Brücken, Disulfidbrücken, ... zusammengehalten werden Struktur von Proteinen: Überblick Primärstruktur Aminosäuresequenz Sekundärstruktur hier: links β-Faltblatt, recht α-Helix Tertiärstruktur räumlicher Aufbau eines Proteins / einer Polypeptidkette Quartärstruktur mehrere Proteine / Polypeptidketten bilden einen Komplex aus Untereinheiten 4.3 Einteilung von Proteinen a) nach der äußeren Form: i) Globuläre Proteine - annähernd kugelförmige Tertiär- bzw. Quartärstruktur - in der Regel gut löslich in Wasser oder Salzlösungen (polare Gruppen nach innen gerichtet, unpolare nach außen) Albumin (globulär, vermittelt Löslichkeit, z.B. bei Fettsäuren) - z.B. Transportproteinen, Albumin ii) Fibrilläre Proteine - fadenförmige oder faserige Struktur - oft unlöslich - meist mit Stütz- oder Gerüstfunktion - z.B. Keratine (Haare, Nägel); Kollagen (Bindegewebe) Kollagen (fibrillär, stabilisiert Bindegewebe, zugfest wg. helicaler Struktur) b) nach der Funktion: i) Schutzfunktion - Toxine, z.B. zur Lähmung von Beutetieren (Schlangengifte, Botox...) - Antikörper zur Abwehr von Mikroorganismen und Fremdstoffen ii) Struktur und Bewegung - hier viele fibrilläre Proteine - Kollagene stabilisieren Zellen bzw. Gewebe - Myosine und Aktine unterstützen die Muskelfunktion - Keratine ermöglicht Strukturen wie Haare, Nägel, Hufe, Federn,... - Seidenfäden bei Spinnen und Insekten iii) Metabolismus (Stoffwechsel), Transport, Signale - Enzyme als Biokatalysatoren - Ionenkanäle ermöglichen den Ionentransport durch Membranen - Transportproteine zum Transport wichtiger Stoffe (Hämoglobin für O2, Transferrin für Fe) zu iii) Metabolismus (Stoffwechsel), Transport, Signale - Membranrezeptoren, wichtig für Signalleitung (z.B. an Synapsen) - Hormone, steuern Prozesse im Organismus über gewisse Entfernungen - Blutgerinnungsfaktoren, verhindern größeren Blutverlust bei Verletzung iv) Reservestoff - Proteine dienen als Energielieferanten im Hungerzustand Beispiele: Botulinum-Toxin des Bakteriums Clostridium botulinum verhindert die Freisetzung von Neurotransmittern an Synapsen von Nervenzellen Beispiele: Hämoglobin dient dem Sauerstofftransport im Blut Prosthetische Gruppe Häm Insulin, Proteohormon der Bauchspeicheldrüse (links als Monomer, rechts als Hexamer) 4.4 Enzyme • Enzyme sind Biokatalysatoren, die die Reaktionsgeschwindigkeit von vielen Reaktionen im Körper auf das 1015-fache im Vergleich zu nicht katalysierten Reaktionen erhöhen. - Problematik: im Organismus muss eine Vielzahl von chem. Reaktionen bei bestimmter Temperatur (Mensch i.d.R. 37 °C) und bestimmten pH-Wert (Blut: 7,4) ablaufen -> Notwendigkeit von Katalysatoren Katalysator: - Stoff, der die Reaktionsgeschwindigkeit einer chemischen Reaktion durch Absenkung der Aktivierungsenergie erhöht, selbst aber nach der Reaktion unverändert bleibt. - Enzyme sind in der Regel Proteine, sehr selten RNA - Enzyme beschleunigen biochemische Reaktionen durch Herabsetzen der Aktivierungsenergie - dies geschieht durch Stabilisierung von Übergangszuständen durch nichtkovalente Wechselwirkungen - biochemische Reaktionen laufen ohne Katalyse i.d.R. spontan (ΔG<0), aber vernachlässigbar langsam ab Nomenklatur von Enzymen - Enzymnamen enden auf –ase (Ausnahmen: Enzymkomplexe) - der Enzymname soll die katalysierte Reaktion beschreiben - der Enzymname soll die Enzymklasse enthalten z.B. Acetylcholinesterase (katalysiert die Hydrolyse einer Esterbindung des Acetylcholins) Acetylcholin Klassifikation von Enzymen i) Oxidoreduktasen Redoxreaktionen ii) Transferasen Übertragung funktioneller Gruppen iii) Hydrolasen Bindungsspaltung mittels Wasser iv) Lyasen Spaltung oder Synthese komplexer Verbindungen ohne ATP v) Isomerasen vi) Ligasen/Synthetasen Umwandlung chemischer Isomere Additionsreaktionen mit i.d.R. ATP zu i) Oxidoreduktasen - katalysierter Reaktionstyp: A- + B → A + B- (Redoxreaktionen) - ‚Oxidasen‘; ‚Reduktasen‘, ‚Dehydrogenasen‘ - z.B. Alkohol-Metabolismus: R – CH2OH Alkohol-Dehydrogenase R – CHO NAD+ NADH + H+ Aldehyd-Dehydrogenase H2O + NAD+ R – COOH NADH + H+ - NAD+: Nicotinamidadenindinukleotid - an vielen Redox-Reaktionen beteiligtes Coenzym - überträgt formal Hydrid-Ionen NAD+ zu ii) Transferasen - katalysierter Reaktionstyp: A-X + B → A + B-X (Übertragung funktioneller Gruppen) - Spezialfall Kinasen: übertragen Phosphatreste (bedeutend u.a. für Energiestoffwechsel) - z.B. Hexokinasen: phosphorylieren Zucker mit 6 C-Atomen (z.B. Glucose im Zuge der Glykolyse) zu iii) Hydrolasen - katalysierter Reaktionstyp: A-B + H2O ⇌ A-H + B-OH (Bindungsspaltung mittels Wasser) - spalten z.B. Ester, Ether, Peptide, Glycoside, Säureanhydride reversibel - entsprechend gibt es Esterasen, Peptidasen, Nukleasen, Glycosidasen,.... - z.B. Aminopeptidasen: (spalten einzelne Aminosäuren vom N-Terminus eines Proteins / Peptids ab) Val – Gly – Ser - Ala L-Valin + Gly – Ser - Ala zu iv) Lyasen - katalysierter Reaktionstyp: A-B → A + B (Nicht-hydrolytische Spaltung ohne ATP) - Spaltung meist unter Bildung von Doppelbindungen oder Lösen einer Ringstruktur - Synthasen sind Lyasen, die Kondensationsreaktionen katalysieren (nicht mit Synthetasen verwechseln, die zu den Ligasen gehören!) - z.B. Fructose-1,6-bisphosphataldolase (Aldolase, ALD): (wichtiges Enzym der Glykolyse) Dihydroxyacetonphosphat Fructose-1,6-bisphosphat Glycerinaldehyd-3-phosphat zu v) Isomerasen - Umwandlung chemischer Isomere ineinander - Isomere: unterschiedliche Konfiguration oder Strukturformel bei gleicher Summenformel - z.B. Glucose-6-phosphat-Isomerase: (wichtiges Enzym der Glykolyse) ⇌ Glucose-6-phosphat-Isomerase Glucose-6-phosphat Fructose-6-phosphat zu vi) Ligasen/Synthetasen - katalysierter Reaktionstyp: oder M1 + M2 + NTP → M1-M2 + NDP + P M1 + M2 + NTP → M1-M2 + NMP + 2P - NTP = Nukleosidtriphosphat (z.B. ATP) als Energielieferant - kovalente Verknüpfung zweier Moleküle unter NTP-Verbrauch - Synthetasen: Untergruppe, die stets ATP verbraucht - besondere Bedeutung: DNA-Ligasen (verknüpfen DNA-Stränge) - z.B. Aminoacyl-tRNA-Synthetase: (bindet Aminosäuren an tRNA für die anschließende Translation) tRNA + Aminosäure + ATP → tRNA-Aminosäure + AMP + Pyrophosphat Aufbau von Enzymen - fast immer Proteine aus einer (Monomere) oder mehreren (Oligomere) Untereinheiten (selten RNA-Enzyme = Ribozyme) - Enzyme können sich zu Enzymkomplexen zusammenlagern - die eigentliche Katalyse findet am aktiven Zentrum statt a) Reine Protein-Enzyme - bestehen ausschließlich aus Protein b) Holoenzyme - bestehen aus einem Proteinanteil (Apoenzym) und einem Cofaktor (niedermolekular; kein Protein) - Cofaktoren sind für die Enzymfunktion unerlässlich - organische Cofaktoren nennt man Coenzyme - Cofaktor kovalent an Enzym gebunden: prosthetische Gruppe (z.B. Häm) - Cofaktor nicht kovalent an Enzym gebunden: Cosubstrat (z.B. ATP, NAD+) Aufbau von Enzymen Hämoglobin dient dem Sauerstofftransport im Blut Prosthetische Gruppe Häm, kovalent an Hämoglobin gebunden Cosubstrat NAD+/NADH (Nicotinamidadenindinukleotid) - nicht kovalent an Enzym gebunden - häufig an Redox-Reaktionen mit [H]-Übertragung beteiligt Funktionsweise von Enzymen - Enzyme fungieren als Biokatalysatoren → biochemische Reaktionen werden durch Herabsetzen der Aktivierungsenergie beschleunigt - die Edukte (Substrate) werden am aktiven Zentrum des Enzyms gebunden - es bildet sich ein Enzym-Substrat-Komplex - nach Umwandlung des Substrates werden die Produkte freigesetzt - das Enzym liegt nach der Reaktion wieder in der ursprünglichen Form vor - Enzyme besitzen eine hohe Substrat- und Reaktionsspezifität Enzyme stabilisieren durch nicht-kovalente Wechselwirkungen Übergangszustände und senken dadurch die Aktivierungsenergie. Die Lage des Chemischen Gleichgewichts wird nicht verändert, es stellt sich lediglich schneller ein. Das aktive Zentrum - die Substrat-Spezifität des aktiven Zentrums beruht auf dem Schlüssel-Schloss-Prinzip: - räumliche Struktur von katalytischem Zentrum und Substrat sowie oberflächlich mögliche Wechselwirkungen führen zur Ausbildung eines Enzym-SubstratKomplexes - Wechselwirkungen: elektrostatisch; Hydrophobe WW; H-Brücken; auch vorübergehende kovalente Bindungen • Funktionsweise von Enzymen am Beispiel von Chymotrypsin (Peptidase) nukleophiler Angriff Chymotrypson schneidet bevorzugt an Carboxyl-Gruppen aromatischer AS stabilisierende H-Brücke Histidin als Base nukleophiler Angriff Enzymkinetik - zentrale Größe: Reaktionsgeschwindigkeit (umgesetzte Substratmenge / Zeit) - Reaktionsgeschwindigkeit hängt ab von Temperatur, Salzkonzentrationen, pH-Wert, Aktivatoren, Inhibitoren, Substrat-, Produkt- und Enzymkonzentration - Enzymaktivität: Maß dafür, wieviel aktives Enzym in einer Probe vorhanden ist: 1 U (Unit) = 1 μmol / min → Menge an Enzym, die unter definierten Bedingungen in einer Minute ein Mikromol Substrat umsetzt Michaelis-Menten-Theorie - liefert Zusammenhang zwischen Reaktionsgeschwindigkeit bei Enzymreaktionen und Substrat- und Enzymkonzentration - Überlegung: 1) Reaktion verläuft über Enzym-Substrat-Komplex, der in Enzym + Produkt oder in Enzym + Substrat zerfällt 2) mit Substratkonzentration steigt auch Reaktionsgeschwindigkeit zur Michaelis-Menten-Theorie - Steigerung der Reaktionsgeschwindigkeit anfangs linear, später abgeflacht bis Steigerung der Substratkonzentration keinen Effekt mehr hat: hyperbolische Sättigungskurve wichtige Kenngrößen für Enzyme: Km: Michaelis-Konstante; Substratkonzentration, bei der ½ vmax erreicht wird kcat: Wechselzahl, Anzahl der Formelumsätze pro Zeiteinheit bei einem Katalysator [s-1] zur Michaelis-Menten-Theorie vereinfachte Reaktionsgleichung: k1 E + S k2 ES E + P k1‘ Bei der Michaelis-Menten-Theorie geht man von einem Fließgleichgewicht aus, das dafür sorgt, dass die Konzentration an Enzym-Substrat-Komplex (ES) konstant bleibt (die Enzymaktivität passt sich sehr schnell sich ändernden Enzymkonzentrationen an). Bei katalysierten Reaktionen erhält man eine Sättigung, d.h. die Reaktionsgeschwindigkeit strebt einem Maximum (vmax) entgegen. Es gilt: mit kcat= vmax / ([E]+[ES]) Abweichungen von der Michaelis-Menten-Theorie - einige Enzyme zeigen Abweichungen von der hyperbolischen Sättigungskurve - mögliche Ursachen z.B. a) Kooperativität - die Bindung von Substrat fördert die Bindung weiteren Substrats, bekannt vom Transportprotein Hämoglobin (Tetramer) und der Bindung von O2: sigmoide Sättigungskurve Abweichungen von der Michaelis-Menten-Theorie b) Alosterie - Bindung von Effektoren an Bindestellen außerhalb des aktiven Zentrums beeinflusst Aktivität Änderung der räumlichen Struktur von Hämoglobin durch Wechselwirkung mit Stoffwechselprodukten -> Änderung der Affinität zu O2 Enzymhemmung a) Kompetitive Hemmung: Der Hemmstoff (Inhibitor) konkurriert mit dem Substrat um das aktive Zentrum. Hohe Inhibitorkonzentrationen bewirken eine Blockierung des Enzyms, weil das aktive Zentrum eher von Inhibitor-Molekülen besetzt wird als von Substraten. Enzym-SubstratKomplex Enzym-InhibitorKomplex • Wichtig: Kompetitive Hemmungen sind reversibel! • b) Nicht-Kompetitive Hemmung: Der Nicht-Kompetitive Inhibitor bindet außerhalb des Aktiven Zentrums an das Enzym und verformt es dadurch, so dass das Substrat nicht mehr bis zum aktiven Zentrum vordringen kann. Inhibitor • c) Unkompetitive Hemmung: Der unkompetitive Hemmer bindet an den Enzym-Substrat-Komplex und verhindert dadurch, dass das Substrat umgesetzt wird. Inhibitor Enzym-SubstratInhibitor-Komplex • Wichtig: Nicht-Kompetitive Hemmungen und Unkompetitive Hemmungen sind ebenfalls reversibel! d) Irreversible Hemmung: Bei der Irreversiblen Hemmung wird das aktive Zentrum des Enzyms durch den Inhibitor chemisch irreversibel modifiziert, so dass das Substrat nicht mehr im aktiven Zentrum gebunden werden kann. Inhibitor • Beispiel für eine irreversible Hemmung: Aspirin hemmt die ProstaglandinH2-Synthase (Cyclooxygenase 1) durch Acetylierung der OH-Gruppe eines Serins. Dadurch kann das Substrat Arachidonsäure nicht mehr gebunden werden, es wird kein Prostaglandin H2 mehr gebildet (auch keine anderen Prostaglandine mehr und auch keine Thromboxane) und Schmerzen und Entzündungen klingen ab. 5. Kohlenhydrate (Saccharide) • Der Begriff „Kohlenhydrate“ leitet sich von der Summenformel vieler Verbindungen dieser Gruppe ab, die sich als CnH2nOn = Cn(H2O)n formulieren lässt. Formal ist also Kohlenstoff „hydratisiert“, es liegt aber kein freies Wasser vor. Kohlenhydrate sind Polyalkohole, die neben –OH meist weitere funktionelle Gruppen besitzen. • Das Wort Saccharide leitet sich vom griechischen sakcharon für Zucker ab. • Kohlenhydrate dienen als Nahrung für Pflanzen und Tiere. Der Kohlenhydratstoffwechsel ist die wichtigste Energiequelle der Tiere und Pflanzen. Kohlenhydrate spielen eine wichtige Rolle als Zellbausteine bei Pflanzen (Zellwand) und bei Tieren (Zellmembran enthält Oligosaccharide) und übernehmen als Glycoproteine und Glycolipide wichtige Aufgaben in der Signalübermittlung zwischen Zellen. • Bei Kohlenhydraten trifft man ähnliche Verhältnisse wie bei Proteinen und Aminosäuren an. Es gibt einzelne Bausteine, die man als Monosaccharide bezeichnet. Durch Verknüpfung von bis zu 6 Monosacchariden kommt man zu den Oligosacchariden. Bei mehr als 6 miteinander verknüpften Monosachharidbausteinen spricht man von Polysacchariden. Wichtige Vertreter der Polysaccharide sind Stärke, Glycogen und Cellulose. 5.1 Monosaccharide • Die Strukturen der Monosaccharide wurden von Emil Fischer aufgeklärt. Er konnte zeigen, dass es sich dabei um Oxidationsprodukte mehrwertiger Alkohole handelt, in denen eine OH-Gruppe zur Aldehydgruppe oder zur Ketogruppe oxidiert wurde. Liegt eine Aldehydgruppe vor, dann spricht man von Aldosen (Aldehydzuckern), bei Vorliegen einer Ketogruppe spricht man von Ketosen oder Ketozuckern. • Die einfachste Aldose ist der Glycolaldehyd, der sich durch Oxidation von Glycol erzeugen läßt. HO H H H HO Ox OH H H Glycol • O Glycolaldehyd Die einfachste Ketose ist das Dihydroxyaceton, das sich durch Oxidation von Glycerin erzeugen läßt. Aus Glycerin lässt sich auch die nächst höhere Aldose Glycerinaldehyd erzeugen. H OH HO HH O H Glycerinaldehyd Ox H OH HO OH HH HH Glycerin Ox O HO OH HH HH Dihydroxyaceton • Glycerinaldehyd ist chiral, d.h. Bild und Spiegelbild sind nicht deckungsgleich. Von Glycerinaldehyd existieren zwei Enantiomere, DGlycerinaldehyd und L-Glycerinaldehyd. CHO H OH CH2OH CHO HO H CH2OH D(+)-Glycerinaldehyd L(-)-Glycerinaldehyd Zur Erinnerung: waagerechte Bindungen zeigen aus der Projektionsebene auf den Betrachter hin, senkrechte vom Betrachter weg • D bedeutet: OH-Gruppe am letzten asymmetrischen C-Atom der Kohlenstoffkette auf der rechten Seite der Fischer-Projektion. • L bedeutet: OH-Gruppe am letzten asymmetrischen C-Atom der Kohlenstoffkette auf der linken Seite der Fischer-Projektion. • (+) bedeutet: Die Substanz dreht die Ebene von linear polarisiertem Licht nach rechts. • (-) bedeutet: Die Substanz dreht die Ebene von linear polarisiertem Licht nach links. • Je nach Zahl der C-Atome unterscheidet man bei Monosacchariden: • Diosen (2 C) • Triosen (3 C) • Tetrosen (4 C) • Pentosen (5 C) • Hexosen (6 C) Glucose (Traubenzucker) = Hexose (Kohlenstoffgerüst mit 6 C-Atomen) Ta = Aldose Tü - in der Natur nur D-Glucose Ta Ta - in wässriger Lösung (so auch in der Zelle): Gleichgewicht zwischen offenkettiger Aldehydform D-Glucose L-Glucose und ringförmiger Pyranoseform (0,25: 99,75) Aldehydform (0,25 %) Pyranoseform (99,75 %) = cycl. Halbacetal Halbacetal: Alkoxy- bzw. Aryloxygruppe und OH-Gruppe am selben C-Atom Bedeutung (D-Glucose): - von fast allen Lebewesen als Energie- und Kohlenstofflieferant verwertbar - Synthese : - Pflanzen: Photosynthese aus Wasser und Kohlendioxid: 6 CO2 + 6 H2O → C6H12O6 + 6 O2 - Tiere: Gluconeogenese, nur im Hungerzustand; Synthese von D-Glucose aus kleinen organischen Verbindungen (Ähnlichkeit mit Gykolyse, nur in anderer Richtung) - Speicherung: - Pflanzen: in Form von Polysacchariden wie Stärke oder Cellulose - Tiere: v.a. in Form von Glykogen - Abbau von Glucose v.a. über Glykolyse und Citratcyclus - Energiegewinnung über oxidative Phosphorylierung / Atmungskette Nachweis von reduzierenden Zuckern: Fehling-Probe - dient u.a. dem Nachweis reduzierender Zucker, z.B. solchen mit freier Aldehyd-Gruppe - geeignet für den Nachweis von Aldosen wie Glucose, aber auch einigen Ketosen wie Fructose (z.B. Unterscheidung von echtem Honig mit Glucose und Fructose von Kunsthonig mit Saccharose) - Fehling I: Kupfer(II)-Sulfat-Lösung - Fehling II: Kalium-Natrium-Tratrat-Tetrahydrat-Lösung blauer Cu-Tartrat-Komplex mit reduzierendem Zucker zu rotem Cu2O Fructose (Fruchtzucker) = Hexose = Ketose - in der Natur nur D-Fructose - isomer zu D-Glucose - in wässriger Lösung (so auch in der Zelle): überwiegend in Ringform, v.a. als β-D-Fructopyranose (ca. 76 %) D-Fructose β-D-Fructofuranose (ca. 20 %) L-Fructose Ribose = Pentose = Aldose - in der Natur überwiegend D-Ribose - wichtiger Bestandteil von RNA (Erbmaterial) - wichtiger Bestandteil des Energieträgers ATP - in wässriger Lösung überwiegend in Pyranose-Form: β-D-Ribopyranose (58 %) α-D-Ribofuranose (6 %) α-D-Ribopyranose (22 %) β-D-Ribofuranose (14 %) Ribose als Bestandteil von RNA und ATP Adenosintriphosphat (ATP) Ribonukleinsäure (RNA) - mit Ribose in Furanose-Form Desoxyribose = Pentose = Aldose - in der Natur überwiegend D-Desoxyribose - im Vergleich zu Ribose: OH-Gruppe am 2. C-Atom substituiert durch H-Atom - wichtiger Bestandteil von DNA (Erbmaterial) - in wässriger Lösung überwiegend in Pyranose-Form: Desoxyribonukleinsäure (DNA) - mit Desoxyribose in Furanose-Form 5.2 Oligosaccharide • Durch Aneinanderhängen von einzelnen Monosacchariden gelangt man zu den Oligosacchariden (Disaccharide, Trisaccharide usw.). Glykosidische Bindung: - entsteht zwischen der in Form eines Halbacetals vorliegenden OH-Gruppe eines Saccharids und der OH-Gruppe (O-glykosidische Bindung) oder NH2-Gruppe (N-glykosidische Bindung) eines anderen Moleküls - Oligosaccharid: das ‚andere‘ Molekül ist wieder ein Saccharid - je nach Konfiguration: α- bzw. β-glykosidische Bindung (hier nicht näher erläutert) - je nach Lage der beteiligten funktionellen Gruppen an den C-Atomen: z.B. 1,4-glykosidische Bindung (am 1. C-Atom des Zuckers und am 4. C-Atoms des ‚anderen‘ Moleküls) 5.2 Oligosaccharide zur glykosidischen Bindung: - unter recht geringem Energieaufwand durch Kondensation, dennoch sehr stabil - Lösen der Bindung durch Hydrolyse Halbacetal z.B. Saccharose: - O-glykosidische Bindung - 1,2-glykosidische Bindung zwischen D-Glucose (Halbacetal) und D-Fructose -das Produkt ist ein (Voll-) Acetal (besitzt C-Atom mit 2 O-R-Gruppen) ATP: Beispiel für eine N-glykosidische Bindung (hier zwischen Ribose und Adenin) • Wichtige Disaccharide: Saccharose (Rohrzucker), Lactose (Milchzucker), Maltose (Malzzucker) HO HO HO HO HO O OH OH O H OH -D-Glucose H O O O HO OH H OH HO OH OH O HO OH -D-Fructose -D-Galactose -D-Glucose Saccharose (,-1,2-Verknüpfung) Lactose (-1,4-Verknüpfung) (Zuckerrüben, Zuckerrohr,… (in Milch von Säugetieren) HO HO HO O OH HO O HO O OH OH -D-Glucose -D-Glucose Maltose (-1,4-Verknüpfung) (Abbauprodukt der Stärke; z.B. in Gerstekeimen (-> Bierherstellung!) 5.3 Polysaccharide • Wichtige Polysaccharide: Stärke, Glycogen, Chitin und Cellulose HO O O α-1,4-glykosidische Bindungen HO OH HO O O HO OH O Stärke - besteht aus DGlucose -Einheiten - idR 20-30 % Amylose, 70-80 % Amylopektin OH O HO löslich O HO HO O OH O HO O HO Amylose 100-1400 Glucoseeinheiten, unverzweigt O HO OH HO O O HO HO O OH O HO OH O O O HO α-1,4-glykosidische Bindungen und α-1,6-glykosidische Bindungen - Stärke dient im Wesentlichen als Reservestoff bei Pflanzen (Glucose- und damit Energie- und Kohlenstoffspeicher) OH O Amylopektin 1200-6000 Glucoseeinheiten, verzweigt unlöslich Glycogen - wie Amylopektin aufgebaut, nur sehr viel häufiger verzweigt (zwischen 600 bis 60 000 Glucoseeinheiten) - Reservestoff bei Tieren, entsprechend der Stärke (‚Tierische Stärke‘) - v.a. in Muskeln α-1,6-glykosidische Bindungen Glycogen aus D-Glucose-Bausteinen α-1,4-glykosidische Bindungen Cellulose und Chitin als strukturbildende Polysascharide Cellulose (unverzweigt, 1000-5000 Glucoseeinheiten): - Hauptbestandteil von Zellwänden bei Pflanzen - β-1,4-glykosidische Bindung Baumwolle aus fast reiner Cellulose Chitin (unverzweigt, aus N-Acetylglucosamin-Einheiten) - Zellwandbestandteil bei Pilzen - Exoskelett-Bestandteil bei Insekten, Spinnen... - β-1,4-glykosidische Bindung Deckflügel beim Maikäfer enthalten Chitin 5.4 Zuckerstoffwechsel 1) Glykolyse - Der zentrale Abbauprozess aller Kohlenhydrate bei Tieren, Pflanzen und Pilzen - bedeutender Vorgang im Energiestoffwechsel - aus 1 Molekül D-Glucose werden 2 Moleküle Pyruvat gebildet - dabei werden je 2 Moleküle ATP und NADH gebildet Startpunkt der Glykolyse: D-Glucose -> andere Zucker bzw. Polysaccharide müssen zunächst in Glucose umgewandelt werden z.B. Stärke-Abbau: Amylose Amylopektin -Amylase kleinere Bruchstücke Glycosidase Glucose zur Glykolyse - Vorteile - Energiegewinnung auch ohne Sauerstoff (begrenzt) - sehr schnelle Bereitstellung von Energie in Form von ATP - Glykolyse bereitet oxidative Energiegewinnung in der Atmungskette vor - Glykolyse liefert Ausgangsstoffe für viele Biosynthesen - Ort: im Cytoplasma aller Zelltypen; bei Pflanzen auch in Plastiden 1x C6 Übersicht: Gesamtbilanz Glucose + 2 ADP + 2 Pi + 2 NAD+ → 2 Pyruvat + 2 ATP + 2 NADH + 2 H+ + 2 H2O Pyruvat 2x C3 zur Glykolyse Schritt 1: Glucose → Glucose-6-phosphat Hexokinase / Glucokinase ATP ADP Zweck: - Glucose-6-phosphat verbleibt eher in Zelle - Glucose-Konz. in Zelle wird verringert -> weitere Glucose strömt nach Schritt 2: Glucose-6-phosphat → Fructose-6-phosphat Glucose-6-phosphatIsomerase zur Glykolyse Schritt 3: Fructose-6-phosphat → Fructose-1,6-bisphosphat Phosphofructokinase ATP ADP Zweck: - die Glykolyse wird an dieser Stelle unumkehrbar - der Schritt ermöglicht die Spaltung in 2 phosphorylierte Triosen zur Glykolyse Schritt 4: Spaltung von Fructose-1,6-phosphat in Glycerinaldehyd-3-phosphat und Dihydroxyacetonphosphat Triosephosphatisomerase Aldolase DHAP → Übergang von der Hexose zur Triose GAP GAP zur Glykolyse Schritt 5: Glycerinaldehyd-3-phosphat → 1,3-Bisphosphoglycerat GAPdehydrogenase GAP NAD+ + Pi NADH + H+ 1,3-bPG → Bildung von NADH - NADH kann zur weiteren Energiegewinnung genutzt werden (Atmungskette) - NADH kann für Reduktionsreaktionen genutzt werden - Phosphorylierung der Carboxygruppe zur Glykolyse Schritt 6: 1,3-Bisphosphoglycerat → 3-Phosphoglycerat Phosphoglyceratkinase ADP ATP 1,3-bPG 3-PG → Bildung von ATP - die bei der vorherigen Oxidation freigewordene Energie wird in Form von ATP gespeichert → Substratkettenphosphorylierung = Gewinnung des Energieträgers ATP (oder GTP) außerhalb der Atmungskette durch oxidativen Abbau von organischen Verbindungen zur Glykolyse Feedbackregulation Phosphoglyceratkinase ADP 1,3-bPG ATP 3-PG Zelle hat genügend Energie in Form von ATP → Glykolyse wird an dieser Stelle gehemmt zur Glykolyse Schritte 7-9: Phosphoglyceratmutase 3-Phosphoglycerat 2-Phosphoglycerat Enolase - H2O 3. irreversibler Schritt der Glykolyse Pyruvatkinase ATP ADP Pyruvat = Anion der Brenztraubensäure 2-Phosphoenolpyruvat → hohes Gruppenübertragungspotential wird zu weiterer Substratkettenphosphorylierung genutzt Einschub: ATP als Energieträger ATP = Adenosintriphosphat → bedeutende Energiequelle für biochemische Prozesse hydrolytische Spaltung zu ADP liefert 32,3 kJ / mol hydrolytische Spaltung zu AMP liefert weitere 32,3 kJ / mol 2) Oxidative Decarboxylierung - zur weiteren Metabolisierung im Citratzyklus wird Pyruvat bzw. Brenztraubensäure umgewandelt zu Acetyl-CoA: Pyruvat-Dehydrogenase-Kopmplex + CoA-SH + CO2 NAD+ NADH + H+ Brenztraubensäure Acetyl-CoA Ort: Matrix der Mitochondrien (dort auch Citratzyklus) Glykolyse im Cytoplasma 3) Citratzyklus - zentrales Abbauprodukt Acetyl-CoA als Ausgangspunkt - Abbau von Acetyl-CoA zu CO2 und H2O - Zweck: - Energiegewinnung (GTP, NADH, FADH) - Gewinnung von Ausgangsstoffen für Biosynthesen - Ort: Matrix der Mitochondrien Nettobilanz: CH3CO-S-CoA + 3 NAD+ + FAD + GDP + Pi + 2 H2O → 2 CO2 + 3 NADH + 3 H+ + FADH2 + GTP + CoA-SH zum Citratzyklus Guanosintriphosphat (GTP) - biochemischer Energiespeicher, ähnlich dem ATP FAD FADH2 Flavin-Adenin-Dinukleotid (FAD) speichert ähnlich dem NAD+ Wasserstoff für Atmungskette zum Citratzyklus Enzyme: 1 8 1) Citrat-Synthase 2 2) Aconitase 3) Isocitrat-Dehydrogenase 7 3 4) α-Ketoglutarat Dehydrogenase-Komplex 5) Succinyl-CoA-Synthetase 6) Succinat-Dehydrogenase 7) Fumarase 8) Malat-Dehydrogenase 6 5 4 zum Citratzyklus als Stoffwechsel-Drehscheibe Aminosäure-Biosynthese Glykolyse, Fettsäureoxidation... Aminosäureoxidation Fettsäure-Biosynthese Cholesterin-Biosynthese Gluconeogenese (D-Glucose) Asparaginsäure, Phenylalanin, Tyrosin Aminosäureoxidation Valin, Isoleucin, Methionin Oxidation ungerader Fettsäuren Porphyrin-Biosynthese (z.B. für Häm in Hämoglobin) Zwischenbilanz des Glucose-Abbaus • Glykolyse und Citratzyklus verlaufen anaerob, d.h. sie brauchen (verbrauchen) keinen Sauerstoff. (↔ aeorb: unter Sauerstof-Verbrauch) • Von der Energiebilanz entsteht bis dahin relativ wenig Energie (2 ATP + 2 GTP pro Molekül Glucose). • Zusätzlich entsteht aber viel Wasserstoff in Form von NADH und FADH2. • (10 NADH und 2 FADH2 pro Molekül Glucose) • Der Wasserstoff kann nun weitere Energie liefern, wenn er mit Sauerstoff zu Wasser reagiert („verbrannt“ wird). Dies ist letztlich eine „Knallgasreaktion“ ohne Explosion. • Dies passiert in der Atmungskette, die in den Mitochondrien („Kraftwerke der Zelle“) abläuft. 4) Atmungskette (Oxidative Phosphorylierung) Atmungskette bezeichnet a) einen Stoffwechselweg zur Energiegewinnung b) die Gesamtheit der an diesem Stoffwechselweg beteiligten Enzymkomplexe • Die Netto-Reaktion der Atmungskette ist die Reduktion von molekularem Sauerstoff O2 zu Wasser bzw. die Oxidation von Wasserstoff zu Wasser. 4 H +O2 2 H2O • Der Wasserstoff (z.B. in Form von NADH) stammt aus vorangehenden Stoffwechselprozessen wie Glykolyse, Ox. Decarboxylierung, Citratzyklus. • Der Sauerstoff stammt aus der Atmung • Die bei dieser Reaktion freiwerdende Energie wird in Form von ATP gespeichert. zur Atmungskette Lokalisierung: innere Membran der Mitochondrien - Beispiel für eine Elektronentransportkette - Beispiel für Chemiosmose Die Atmungskette als Elektronentransportkette - die Atmungskette besteht aus einer Reihe hintereinandergeschalteter Redoxmoleküle mit Schritt für Schritt fallendem Energieniveau - Elektronen werden von Wasserstoff (in Form von NADH oder FADH2) auf Sauerstoff übertragen - beteiligte Komponenten: - Enzymkomplexe I – IV, sowie Ubichinon und Cytochrom c als Elektronenüberträger Prinzip: - durch die Anordnung der beteiligten Komponenten werden Protonen in den Intermembranraum der Mitochondrien transportiert - der resultierende H+-Gradient wird zur ATP-Synthese genutzt → Chemiosmose Die Atmungskette als Elektronentransportkette Intermembranraum Ubichinon Matrix Komplex I: NADH-Dehydrogenase Ubihydrochinon - reduziert mittels NADH Ubichinon zu Ubihydrochinon - Nettobilanz: 3-4 H+ werden pro NADH in den Intermembranraum gepumpt Die Atmungskette als Elektronentransportkette Intermembranraum Matrix Komplex II: Succinat-Dehydrogenase - oxidiert Succinat zu Fumarat im Rahmen des Citrat-Zyklus - FAD als Elektronnen- bzw. [H]-Überträger - hier werden keine Protonen in den Intermembranraum gepumpt - Kopplung von Citrat-Zyklus und Atmungskette Die Atmungskette als Elektronentransportkette Cytochrom c Komplex III: Cytochrom-c-Reduktase - Rückoxidation von Ubihydrochinon zu Ubichinon - Reduktion von Cytochrom c - Transport von 4 H+ in Intermembranraum Cytochrom c Die Atmungskette als Elektronentransportkette Cytochrom c (104 Aminosäuren) prosthetische Gruppe Häm c (Porphyrin-Ringsystem) Die Atmungskette als Elektronentransportkette CN - Komplex IV: Cytochrom-c-Oxidase - Rückoxidation von Cytochrom c - Übertragung der Elektronen auf O2, Reduktion zu H2O - die benötigten Protonen stammen aus Matrix - pro O2-Molekül werde 4 H+ in den Intermembranraum transportiert - Komplex IV ist der wesentliche O2-Verbraucher fast aller Lebewesen Die Atmungskette als Elektronentransportkette „Komplex V“: ATP-Synthase Die Atmungskette als Elektronentransportkette „Komplex V“: ATP-Synthase Intermembranraum - Hauptaufgabe: Katalyse der ATP-Synthese ADP + Pi → ATP ΔH = ca. 30 kJ / mol → oxidative Phosphorylierung - Energiequelle: der im Zuge der Atmungskette aufgebaute Protonengradient zwischen Matrix und Intermembranraum - Matrix: niedrige H+-Konzentration (rel. hoher pH-Wert) - Intermembranraum: hohe H+-Konzentration → Chemiosmose (Kopplung von Transportvorgängen an Biomembranen mit Stoffwechselprozessen) Matrix 5) Bilanz der aeroben Atmung - Gesamtreaktion: C6H12O6 + 6 O2 → 6 CO2 + 6 H2O 1) Glykolyse Glucose + 2 NAD+ + 2 ADP + 2 Pi → 2 Pyruvat + 2 NADH + 2 H+ + 2 ATP + 2 H2O 2) Oxidative Decarboxylierung 2 Pyruvat + 2 NAD+ + 2 CoA → 2 Acetyl-CoA + 2 CO2 + 2 NADH + 2 H+ 3) Citratzyklus 2 Acetyl-CoA + 6 NAD+ + 2 FAD + 2 GDP + 2 Pi + 4 H2O → 4 CO2 + 6 NADH + 6 H+ + 2 FADH2 + 2 GTP + 2 CoA 4) Endoxidation in der Atmungskette 10 NADH + 10 H+ + 2 FADH2 + 28 ADP + 28 Pi + 6 O2 → 10 NAD+ + 2 FAD + 12 H2O + 28 ATP 5) Bilanz der aeroben Atmung Energiebilanz: Schritt Coenzym- ATPAusbeute Ausbeute Glykolyse ATPQuelle 2 ATP 3-5 ATP Substratkettenphosphorylier. Oxidative Phosphorylierung Oxidative 2 NADH Decarboxylier. 5 ATP Oxidative Phosphorylierung Citrat-Zyklus 2 ATP 15 ATP 3 ATP Substratkettenphosphorylier. Oxidative Phosphorylierung Oxidative Phosphorylierung 2 NADH 6 NADH 2 FADH2 Gesamt 30-32 ATP pro Molekül Glucose Hier ist einberechnet, dass an diversen Stellen Energie (ATP) für Transportvorgänge verbraucht wird. 5.5 Photosynthese = natürliche Erzeugung energiereicher Stoffe aus energieärmeren Stoffen mithilfe von Lichtenergie - Vorkommen: Pflanzen, Algen, einige Bakterien - Bedeutung: - einer der wichtigsten biochemischen Prozesse - O2-Quelle für aerob atmende Lebewesen (oxygene Photosynthese) - ermöglicht Erschließung von CO2 als C-Quelle, damit Verbrauch von CO2 (Klima!) - Gesamtgleichung der oxygenen Photosynthese: h 6 CO2 +6 H2O C6H12O6 +6 O2 Chloroplasten als Ort der Photosynthese 1) Drei Schritte der Photosynthese I) Lichtreaktion a) Absorbtion von Licht mit Hilfe von Farbstoffen b) Umwandlung der Lichtenergie in chemische Energie durch Elektronenübertragung II) Dunkelreaktion Verwendung der chemischen Energie zur Synthese energiereicher organischer Verbindungen - Reduktion von CO2 unter Verwendung geeigneter Reduktionsmittel, bei Pflanzen i.d.R. H2O 2) Lichtabsorbtion - die für die Photosynthese benötigte Energie in Form von Licht wird durch Farbstoffe (Chromophore) absorbiert - bei Pflanzen sind das vor allem Chlorophylle - Chromophore bilden mit umgebenden Proteinen Komplexe, die Pigmente - trifft Licht geeigneter Wellenlänge auf Pigmente, so gehen die Chromophore in einen angeregten Zustand über - je nach chemischer Struktur des Chromophors weisen Pigmente unterschiedliche Absorbtionsspektren auf - Chlorophyll a und b bei Pflanzen absorbieren v.a. im blauen und roten Bereich, weniger im grünen Bereich → grünes Erscheinungsbild der Pflanzen zu 2) Lichtabsorbtion Absorbtionsspektrum für Chlorophyll a und b Grünlücke, verantwortlich für grünes Erscheinungsbild der Pflanzen Engelmann‘scher Bakterienversuch: Sauerstoff-liebende Bakterien sammeln sich an Algenfäden bevorzugt in Bereichen an, wo mit blauem bzw. rotem Licht bestrahlt wird, weniger dagegen im grünen Bereich. zu 2) Lichtabsorbtion - durch Licht angeregtes Chlorophyll kann angeregte Elektronen auf einen Elektronenakzeptor übertragen - damit startet einen Elektronentransportkette - beim Elektronentransport werden Protonen über eine Membran transloziert - die Lichtenergie wird somit in elektrisches bzw. osmotisches Potential umgesetzt (Chemieosmose) Lichtsammelkomplex: (eine Art ‚Antenne‘) - Ansammlung von Farbpigmenten, v.a. Chlorophylle - dient der Vergrößerung der Absorbtionsfläche - Ergänzung des Absorbtionsspektrums durch Carotine und Xanthophylle - Weiterleitung der Lichtenergie an ein gemeinsames Reaktionszentrum (Chlorophyll a) zu 2) Lichtabsorbtion Chlorophyll mit derivatisiertem Porphyrinring und Mg2+ als Zentralion Die Grünlücke des Chlorophylls kann z.B. durch Carotine teilweise geschlossen werden β-Carotin, ein Tetraterpen mit konjugierten Doppelbindungen 3) Elektronenübertragung - im 2. Teil der Photosynthese wird durch Elektronenübertragung die Energie des absorbierten Lichts in chemische Energie (ATP) umgewandelt - zudem wird NADPH als Reduktionsmittel gewonnen (notwendig v.a. für die Reduktion von CO2 zu Kohlehydraten in der Dunkelreaktion) - bei der oxygenen (sauerstoffbildenden) Photosynthese von Pflanzen wird als Elektronenquelle H2O verwendet, das zu O2 oxidiert wird zu 3) Elektronenübertragung Lokalisierung: Chloroplasten in Pflanzenzellen Elektronentransportkette in der Thylakoidmembran zu 3) Elektronenübertragung Z-Schema der Lichtreaktion: Photosystem I Photosystem II Redoxpotential [V] NADP+ als Endpunkt der Elektronentransportkette Elektronenquelle H2O ATP-Synthese über H+-Gradient (Chemiosmose, Photophosphorylierung) O2 als Produkt der H2O-Oxidation zu 3) Elektronenübertragung Z-Schema der Lichtreaktion: primärer e--Akzeptor Phäophytin Photosystem II Redoxpotential [V] Cytochrom b6f-Komplex Plastochinon QA Plastochinon QB Ein Mn-haltiger wasserspaltender Komplex regeneriert das Reaktionszentrum II Plastocyanin (Cu-haltig) Chemiosmose Licht der Wellenlänge 680 nm regt Chlorophyll a-Elektronen im Reaktionszentrum an zu 3) Elektronenübertragung Z-Schema der Lichtreaktion: Photosystem I Redoxpotential [V] primärer e--Akzeptor A0, wohl Chlorophyll-Variante Phyllochinon Fe-S-Cluster Ferredoxin FerredoxinNAPD-Reduktase Plastocyanin regeneriert das Reaktionszentrum I Licht der Wellenlänge 700 nm regt Chlorophyll a-Elektronen im Reaktionszentrum an zu 3) Elektronenübertragung Bilanz der Lichtreaktion: - pro Elektron werden beim linearen Elektronentransport drei Protonen über die Thylakoid-Membranen der Chloroplasten transportiert - der resultierende Protonengradient wird zur ATP-Synthese durch einen ATPSynthase-Komplex genutzt (Chemiosmose, Photophoshporylierung) - ca. 12 Protonen liefern dabei 3 ATP und 6 NADPH/H+ Gesamtgleichung der Lichtreaktion: 6 H2O + 6 NADP+ + 3 ADP + 3 Pi → 3 O2 + 6 NADPH/H+ + 3 ATP zu 3) Elektronenübertragung ATP-Synthese ATP-Synthase + Pi ADP ATP NADP+ Nicotinamidadenindinukleotidphosphat = phosphoryliertes Derivat des Coenzyms NAD NADPH wird im Unterschied zu NADH im Wesentlichen als Reduktionsmittel bei Biosynthesen (z.B. Dunkelreaktion) verwendet 4) Dunkelreaktion / Calvin-Zyklus - die in der Lichtreaktion gewonnene chemische Energie (ATP) und die gewonnenen Reduktionsäquivalente (NADPH) werden in der Dunkelreaktion zur Kohlenhydratsynthese verwendet - die Dunkelreaktion ist nicht direkt von Licht abhängig, indirekt aber sehr wohl (findet daher ebenfalls bei Licht statt) - die Kohlenhydratsynthese erfolgt bei der oxygenen Photosynthese i.d.R. durch CO2-Fixierung über den Calvin-Zyklus - Ort: Stroma der Chloroplasten zu 4) Dunkelreaktion / Calvin-Zyklus 1. CO2-Fixierung 2. Reduktion des Fixierungsprodukts 3. Regeneration des Fixierungsprodukts Gesamtgleichung des Calvin-Zyklus: 3 CO2 + 6 NADPH + 5 H2O + 3 ATP + 6 H+ → Glycerinaldehydphosphat + 6 NADP+ + 9 ADP + 8 Pi 6. Nukleinsäuren 1) Nukleotide - Nukleotide sind die Grundbausteine der Nukleinsäuren (DNA, RNA), Energiespeicher (ATP, GTP), Signalmoleküle (cAMP) - sie bestehen aus einem Phosphat-, einem Zucker- und einem Basenbestandteil Nukleobase + Zucker = Nukleosid - als Zuckerbestandteil kommen Ribose und Desoxyribose vor - die wichtigsten Nukleobasen sind Adenin, Cytosin, Guanin, Thymin und Uracil 6. Nukleinsäuren zu 1) Nukleotide Nukleinbasen: a) Purinbasen Guanin Adenin Purin b) Pyrimidinbasen Cytosin Thymin Uracil Pyrimidin 6. Nukleinsäuren zu 1) Nukleotide Nukleoside: Bausteine aus Nukleobase + Zucker (Ribose oder Desoxyribose) Adenin + Ribose: Adenosin (A) Cytosin + Ribose: Cytidin (C) Guanin + Ribose: Guanosin (G) Uracil + Ribose: Uridin (U) Adenin + Desoxyribose Desoxyadenosin (dA) Cytosin + Desoxyribose Desoxycytidin (dC) Guanin + Desoxyribose Desoxyguanosin (dG) Thymin + Desoxyribose Desoxythymidin (dT) 6. Nukleinsäuren zu 1) Nukleotide Nukleotide als Bausteine der Nukleinsäuren a) RNA: Ribose als Zuckerbaustein; Basen A, C, G, U AMP CMP GMP UMP b) DNA: Desoxyribose als Zuckerbaustein; Basen A, C, G, T dAMP dCMP dGMP dTMP 6. Nukleinsäuren 2) Desoxyribonukleinsäure (DNA) - Polymer aus den vier Nukleotiden dAMP, dCMP, dGMP und dTMP - Vorkommen: in allen Lebewesen - Eukaryoten: v.a. im Zellkern, aber auch in den Mitochondrien (bei Pflanzen auch in Chloroplasten) - enthält die Erbinformation in Form der Nukleotid-Abfolge (Basenabfolge) - Gen: Abschnitt einer DNA, der die Information für die Herstellung einer RNA enthält 6. Nukleinsäuren zu 2) Desoxyribonukleinsäure Struktur: - doppelsträngiges Molekül - Rückgrat jeweils aus Phosphat + Desoxyribose - in wässriger Lösung Phosphat deprotoniert → negative Gesamtladung - die 4 Nukleobasen sind über glykosidische Bindungen an Desoxyribose gebunden (DNA) 6. Nukleinsäuren zu 2) Desoxyribonukleinsäure zur Struktur: - es treten 2 Basenpaare auf: Adenin – Thymin (A-T) (über 2 H-Brückenbindungen) Cytosin – Guanin (C-G) (über 3 H-Brückenbindungen) - jeder der beiden DNA-Stränge verfügt über ein 3‘- Ende mit einer OH-Gruppe und ein 5‘-Ende mit einer Phosphat-Gruppe (DNA) 6. Nukleinsäuren zu 2) Desoxyribonukleinsäure (DNA) zur Struktur: 5‘ 3‘ 6. Nukleinsäuren zu 2) Desoxyribonukleinsäure (DNA) zur Struktur: - die Wasserstoffbrückenbindungen erzeugen eine Doppelhelix-Sruktur Rückgrat: Zucker + Phosphat Basenpaare 6. Nukleinsäuren zu 2) Desoxyribonukleinsäure (DNA) Chromosomen - Die DNA des Zellkerns ist in Makromolekülkomplexen aus DNA und Proteinen organisiert, den Chromosomen - Der Mensch besitzt in einer normalen Körperzelle 2x 23 Chromosomen - 2 der 46 Chromosomen sind Geschlechtschromosomen (Frau: XX; Mann: XY) Chromosomen aus weiblicher Zelle; mit FluoreszenzFarbstoffen markiert (Metaphase; vor Zellteilung) Metaphasenchromosom zum Vergleich: ohne ‚Verpacken‘ wäre Chromosom mehrere cm lang 6. Nukleinsäuren 3)Ribonukleinsäure (RNA) - Polymer aus den vier Nukleotiden AMP, CMP, GMP und UMP - Hauptfunktion: - maßgebliche Beteiligung an der Umsetzung genetischer Informationen in Proteine - wichtige RNA-Typen: -z.B. a)mRNA (messenger RNA) b)rRNA (ribosomale RNA) c)tRNA (Transfer-RNA) 6. Nukleinsäuren zu 3) Ribonukleinsäure (RNA) Struktur: - i.d.R. einzelsträngiges Molekül - Rückgrat jeweils aus Phosphat + Ribose - RNA deutlich instabiler als DNA (hydrolysierbare OH-Gruppe der Ribose) - RNA neigt stark zur Ausbildung dreidimensionaler Strukturen (z.B. t RNA) - statt Thymin wird die Base Uracil verwendet (wohl wegen der weniger aufwendigen Synthese) 6. Nukleinsäuren zu 3) Ribonukleinsäure (RNA) mRNA: - messenger RNA / Boten-RNA - codierende RNA; enthält Information über Aminosäuresequenz eines Proteins - mRNA wird im Zuge der Transkription im Zellkern synthetisiert - DNA-Abschnitte (Gene) enthalten die Information zum Aufbau der mRNA mRNA-Transkription im Zellkern 6. Nukleinsäuren zu 3) Ribonukleinsäure (RNA) rRNA: - ribosomale RNA - nicht-codierende RNA; ist Bestandteil der Ribosomen - Ribosomen: makromolekulare Komplexe aus Proteinen und rRNA, an denen Proteine synthetisiert werden Ribosom typische Schleifenbildung bei rRNA Proteinsynthese an Ribosomen 6. Nukleinsäuren zu 3) Ribonukleinsäure (RNA) tRNA: - Transfer-RNA - nicht-codierende RNA; ist an Proteinsynthese beteiligt Bindestelle für Aminosäure - überträgt Aminosäuren auf wachsende Peptidkette doppelsträngige Abschnitte 3-dimensionale Darstellung einer tRNA tRNA erkennt den Code der mRNA Sekundärstruktur einer tRNA 2.4. Nukleinsäuren 4) DNA-Replikation = Vervielfältigung des Erbmaterials DNA in der Zelle nach dem semikonservativen Prinzip - semikonservativ bedeutet, dass ein DNA-Strang von einem bestehenden DNA-Doppelstrang erhalten bleibt und daran ein neuer Strang synthetisiert wird - die DNA-Replikation ist stark an die Zellteilung gekoppelt, da diese eine vorherige Verdoppelung des Erbmaterials voraussetzt - bei der Replikation wird die DNA i.d.R. nicht verändert Die DNA-Replikatiom geht der Zellteilung voraus 6. Nukleinsäuren zu 4) DNA-Replikation 1. Helicase bricht an bestimmter Stelle DNA-Doppelstrang auf 2. DNA-Polymerase lagert sich an die beiden entstandenen DNA-Einzelstrang an Topoisomerase vermindert Verdrillungen 6. Nukleinsäuren zu 4) DNA-Replikation 3. DNA-Synthese b) Leitstrang: durchgehende Synthese nach einmaligem Start a) Elternstränge d) Replikationsgabel e) RNA-Primer zum Start der DNA-Synthese f) Okazaki- Fragmente als Produkte der DNA-Synthese am Folgestrang c) Folgestrang: Synthese erfolgt hier Stück für Stück, da DNA-Synthese nur von 5‘ nach 3‘ möglich 6. Nukleinsäuren 4) Proteinbiosynthese = Neubildung von Proteinen in Zellen nach Vorlage der aufgearbeiteten Kopie (mRNA) eines bestimmten Gen-Abschnitts (DNA) - die Proteinbiosynthese ist DER zentrale Prozess der Genexpression 1. wesentlicher Teilprozess: Transkription - Umschreiben eines DNA-Codes in einen mRNA-Code - Ort: Zellkern 2. wesentlicher Teilprozess: Translation - Übersetzen des mRNA-Codes in eine Aminosäuresequenz - Ort: Cytoplasma 6. Nukleinsäuren zu 4) Proteinbiosynthese 4.1 Transkription = Synthese von RNA anhand einer DNA-Vorlage - bei der Proteinbiosynthese wird hier eine mRNA synthetisiert - bei der Transkription wird der Basen-Code der DNA in einen entsprechenden Basen-Code der RNA umgeschrieben, wobei statt der Base Thymin (T) die Base Uracil (U) verwendet wird - aus A auf DNA-Ebene wird U auf RNA-Ebene, entsprechend wird aus T ein A, aus C ein G und aus G ein C: 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.1 Transkription 1. Initiation: - der RNA-Polymerase-Proteinkomplex erkennt eine bestimmte Region auf der DNA, den Promotor 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.1 Transkription 2. Elongation - die DNA wird auf 10-20 Basen durch die RNA-Polymerase entspiralisiert - am antisense-Strang der DNA (= codogener Strang) lagern sich durch Basenpaarung komplementäre Ribonukleotide (Nukleosidtriphosphate) an 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.1 Transkription zu 2. Elongation - unter Eliminierung von Pyrophosphat wird eine Esterbindung zwischen der Ribose am 3‘-Ende der neuen RNA und der verbleibenden Phosphatgruppe des Ribonukleotids gebildet - die DNA wird am codogenen Strang von 3‘ nach 5‘ abgelesen - die Synthese der RNA erfolgt von 5‘ nach 3‘ +PPi + Ribose am 3‘-Ende der neuen RNA UTP Pyrophosphat 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.1 Transkription 3. Termination - am sogenannten Terminator wird die RNA-Synthese beendet Übung: Übersetze die folgenden DNA-Sequenzen in mRNA-Sequenzen: antisense 3‘ AACGGTAAACGAT 5‘ sense 5‘ TTGCCATTTGCTA 3‘ Lösung: 5‘ UUGCCAUUUGCUA 3‘ 3‘ CCATGGAAAT 5‘ 5‘ GGTACCTTTA 3‘ ???????? -> die Lösung ist am sense-Strang ablesbar (T durch U ersetzen!), daher der Name 6. Nukleinsäuren zu 4) Proteinbiosynthese 4.2 Posttranskriptionale Modifikation der mRNA - bei Eukaryoten wird die synthetisierte Vorläufer-mRNA (prä-mRNA) in der Regel nach der Transkription modifiziert: a) Spleißen - die prä-mRNA besteht aus Exons und Introns; die Introns werden durch das Spleißen entfernt: 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.2 Posttranskriptionale Modifikation der mRNA b) Capping - die mRNA erhält vor ihrem Transport in das Cytoplasma eine „Schutzkappe“ am 5‘-Ende - diese Kappe schützt die RNA vor enzymatischem Abbau und unterstützt den Transport aus dem Kern sowie die Translation häufige Cap-Struktur: ein Nukleotid mit einem methylierten Guanin wird ‚rückwärts‘ (5‘-5‘) an das mRNA-Ende angehängt 6. Nukleinsäuren zu 4) Proteinbiosynthese zu 4.2 Posttranskriptionale Modifikation der mRNA c) Polyadenylierung - die mRNA erhält vor ihrem Transport in das Cytoplasma eine Poly(A)-Kette am 3‘-Ende (bis zu 250 Nukleotide) - auch diese schützt die RNA vor Abbau und unterstützt den Kernexport d) RNA-Editing - durch RNA-Editing können einzelne RNA-Basen nach der Transkription modifiziert werden - dadurch besteht die Möglichkeit, aus einem Gen mehrere Proteine zu erhalten 6. Nukleinsäuren 4) Proteinbiosynthese 4.3 Translation = Übersetzung der Basensequenz einer mRNA in die Aminosäuresequenz eines Proteins - die Translation geschieht an den Ribosomen im Cytosol - je drei aufeinander folgende Basen der mRNA (Basentriplett) bilden ein Codon, das den Code für eine bestimmte Aminosäure darstellt - zu jedem Codon existiert eine tRNA mit einem entsprechenden Anticodon, die die passende Aminosäure binden kann - am Ribosom wird die tRNA mit der nächsten passenden Aminosäure an die wachsende Peptidkette herangeführt 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.3 Translation Ribosomen: makromolekulare Komplexe aus Proteinen und rRNA, die in Cytosol, Mitochondrien und Chloroplasten vorkommen. Sie bestehen aus einer großen und einer kleinen Untereinheit. An ihnen werden Proteine gemäß dem durch mRNA vermittelten genetischen Code synthetisiert 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.3 Translation Initialphase: Erreicht eine mRNA ein Ribosom, so läuft die kleine Untereinheit solange an der mRNA entlang, bis sie ein Startcodon (AUG) findet. Die dazu passende Methionin-tRNA mit dem Anticodon UAC heftet sich an das Codon; es bildet sich der Initiationskomplex. 6. Nukleinsäuren Elongationsphase: 4) Proteinbiosynthese Nach Abschluss der Initiationsphase zu 4.3 Translation lagert sich auch die große Untereinheit an. Die Methionin-tRNA befindet sich zunächst an der P-Bindestelle. Die nächste tRNA lagert sich nun an der ABindestelle an. Das Enzym Peptidyltransferase verknüpft die beiden Aminosäuren an der ABindestelle zu einem Dipeptid. Das Ribosom wandert um ein Triplett weiter. Die leere tRNA steht jetzt an der E: Ausgang (Exit) P: Bindungsort (Peptidyl-) A: Erkennungsort (Aminoacyl-) E-Bindestelle, die tRNA mit dem Dipeptid an der P-Bindestelle, die nächste tRNA kann an die ABindestelle. 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.3 Translation Termination: Bei Erreichen eines Stoppcodons, das für keine Aminosäurecodiert, wird die Translation beendet. Terminationsfaktoren release factors, RF) binden an das Stoppcodon und unterstützen die Termination. Stoppcodons: UAG UAA UGA 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.3 Translation Codon-Sonne, liefert zu einem Codon die passende Aminosäure bzw. das Stopp-Signal (zu lesen von innen nach außen) z.B. UAC → Phenylalanin AUG → Start / Methionin UAG → Stopp 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.3 Translation - die mRNA muss beim Übersetzen von 5‘ nach 3‘ und im richtigen Leseraster (also nicht um ein oder 2 Basen versetzt) abgelesen werden Übung: Übersetze die folgenden mRNA-Sequenzen in Aminosäuresequenzen: (Startcodon unterstrichen) 5‘ AUGUUUGGUAAGUAA 3‘ 5‘ CCAUGAAAGGGGGAGGC 3‘ Lösung: Met – Phe – Gly – Lys (stop) ????? 6. Nukleinsäuren 4) Proteinbiosynthese 4.4 Protein-Modifikation - viele Proteine werden während (cotranslational) oder nach (posttranslational) der Translation modifiziert - mögliche Modifikationen: - Abspaltung von Teilen der Polypeptidkette (z.B. Abspaltung einer N-terminalen Signalsequenz) - Hydroxylierung, Decarboxylierung oder Oxidation einzelner Aminosäuren - Glykosylierungen (Anbau von Zuckerbausteinen) - Chaperon-unterstützte Formgebung 6. Nukleinsäuren 4) Proteinbiosynthese zu 4.4 Protein-Modifikation Beispiel: Abspaltung von Signalsequenzen - Proteine, deren Bestimmungsort außerhalb der Zelle oder in Zellkompartimenten wie Endoplasmatischem Retikulum oder Zellkern liegt, besitzen häufig N-terminale Signalsequenzen (gleichsam einer Postleitzahl), die während oder nach der Translation abgespalten werden: 6. Nukleinsäuren 5) Genmutation = dauerhafte Veränderung des Erbgutes (i.d.R. DNA) in einem Gen, die an die Tochterzellen weitergegeben wird Beispiele: a) Substitution: eine DNA-Base wird durch eine andere ersetzt b) Deletion: Verlust einer DNA-Base c) Insertion: Einfügen einer DNA-Base 6. Nukleinsäuren zu 5) Genmutation a) Substitution – Ersatz einer DNA-Base durch eine andere Leserichtung Mutation 3‘-ACGTCGGGCACG-5‘ 5‘-TGCAGCCCGTGC-3‘ DNA 3‘-AGGTCGGGCACG-5‘ 5‘-TCCAGCCCGTGC-3‘ Transkription mRNA 5‘-UCCAGCCCGUGC-3‘ Protein Ser-Ser-Pro-Cys 5‘-UGCAGCCCGUGC-3‘ Translation Cys-Ser-Pro-Cys 6. Nukleinsäuren zu 5) Genmutation zu a) Substitution – Ersatz einer DNA-Base durch eine andere -> eine Basenpaarsubstitution kann zum Austausch von Aminosäuren im Protein führen -> Ersatz durch ähnliche Aminosäure i.d.R weniger folgenreich als Ersatz durch ganz andersartige AS Wildtyp Cys-Ser-Pro-Cys Mutante Ser-Ser-Pro-Cys 6. Nukleinsäuren zu 5) Genmutation b) + c) Deletion bzw. Insertion – Entfernen bzw. Einfügen einzelner Basenpaare -> hier kommt es i.d.R. zur Verschiebung des gesamten Leserasters Leserichtung Insertion 3‘-ACGTCGGGCACG-5‘ 5‘-TGCAGCCCGTGC-3‘ DNA 3‘-AACGTCGGGCACG-5‘ 5‘-TTGCAGCCCGTGC-3‘ Transkription mRNA 5‘-UUCCAGCCCGUGC-3‘ Protein Phe-Gln-Pro-Val 5‘-UGCAGCCCGUGC-3‘ Translation Cys-Ser-Pro-Cys 7. Lipide 7.1 Chemie der Lipide Lipide = Naturstoffe, die v.a. aufgrund langer Kohlenwasserstoffreste keine oder eine geringe Wasserlöslichkeit aufweise. Sie sind aufgrund geringer Polarität hydrophob (lipophil) Einteilung der Lipide: 1.Fettsäuren 2.Fette und Öle (Triacylglycerine) 3.Phospholipide 4.Sphingolipide 5.Lipopolysaccharide 6.Isoprenoide 7.Wachse 7. Lipide 7.1 Chemie der Lipide 1.Fettsäuren = meist unverzweigte Monocarbonsäuren mit endständiger Carboxy-Gruppe - Bestandteil der Fette und Öle - gesättigte Fettsäuren: keine Doppelbindungen - ungesättigte Fettsäuren: enthalten Doppelbindungen Stearinsäure, gesättigt Octadecansäure) Ölsäure, ungesättigt (cis-Octadecensäure) 7. Lipide 7.1 Chemie der Lipide 2. Fette und Öle (Triacylglycerine) = Ester des dreiwertigen Alkohols Glycerin mit drei, meist verschiedenen Fettsäuren - Fette: bei Raumtemperatur fest - (fette) Öle: bei Raumtemperatur flüssig Allgemeine Struktur von Fetten und Ölen; blau: 3-fach acyliertes Glycerin Veresterung zwischen Glycerin und Stearinsäure unter Wasserabspaltung (Umkehrreaktion: Verseifung) 7. Lipide 7.1 Chemie der Lipide 3. Phospholipide = phosphorhaltige, amphiphile Lipide - amphiphil: sowohl hydrophil als auch lipophil (natürliche Tenside) - wichtigster Bestandteil von Biomembranen a) Phosphoglyceride b) Sphingomyeline z.B. Cholinrest Amidbindung zwischen Sphingosin und Fettsäure Aufbau der Phosphoglyceride: Veresterung von Glycerin mit zwei Fettsäuren und einem Phosphorsäurederivat 7. Lipide 7.1 Chemie der Lipide zu 3. Phospholipide - amphiphile Lipide ermöglichen den Aufbau von Lipiddoppelschichten: hydrophile PhosphatKöpfchen hydrophobe FettsäureSchwänze -> Bauprinzip von Biomembranen zur Trennung zellulärer Kompartimente: nahezu undurchlässig für Wasser, Ionen und wasserlösl. Moleküle, dabei aber sehr stabil 7. Lipide 7.1 Chemie der Lipide 4. Sphingolipide = von Sphingosin abgeleitete, amphiphile Lipide - wie Phospholipide wichtiger Bestandteil von Biomembranen - Vertreter: z.B. Sphingomyeline, die auch zu Phospholipiden gehören Aufbau der Sphingolipide: amidische Verknüpfung zwischen dem Aminoalkohol Sphingosin und einer Acylgruppe, z.B. einer Fettsäure 7. Lipide 7.1 Chemie der Lipide 5. Lipopolysaccharide = Verbindungen aus fettähnlichen Bestandteilen und Zuckerbestandteilen, z.B. in Bakterienmembranen Lipopolysaccharid 6.Isoprenoide = von Isopren abgeleitete Naturstoffe - z.B. Steroide wie Cholesterin; Terpene in Harz von Nadelbäumen Isopren Cholesterin (stabilisiert u.a. Biomembranen) 7. Lipide 7.1 Chemie der Lipide 7. Wachse = Einfach-Ester von Fettsäuren mit langen, gesättigten Alkylresten im Fettsäure- und im Alkoholteil - meist knetbar bei Raumtemperatur, fest bis brüchig hart Bienenwachsbestandteil Palmitinsäuremyricylester Waben aus Bienenwachs 7. Lipide 7.2 Funktion der Lipide a)Brennstoff im Energiestoffwechsel (β-Oxidation von Fettsäuren) b)Energiespeicher (Fette, Öle) c)Membranbausteine (v.a. Phospholipide) d)Signalmoleküle e)Hormone (z.B. Steroide wie Testosteron oder Cortisol) f)fettlösliche Vitamine A, D, E, K g)Cofaktoren von Enzymen h)Pigmente (z.B. Carotinoide = Isoprenoide) 7. Lipide 7.2 Funktion der Lipide zu a) Lipide als Brennstoff: β-Oxidation von Fettsäuren Beispiel: β-Oxidation ungesättigter Fettsäuren mit gerader C-Atom-Zahl Vorbereitung: Aktivierung der Fettsäure + ATP + CoA-SH + AMP + PPi Fettsäure-CoA-Ligase - danach: Transport in die Mitochondrien-Matrix - Aktivierung ist irreversibel: aktivierte Fettsäuren werden abgebaut 7. Lipide 7.2 Funktion der Lipide Beispiel: β-Oxidation ungesättigter Fettsäuren mit gerader C-Atom-Zahl Schritt1: FAD-abhängige Oxidation → Knüpfen einer trans-Doppelbindung zwischen 2. und 3. C-Atom → Gewinnung von FADH2 für die Atmungskette 7. Lipide 7.2 Funktion der Lipide Beispiel: β-Oxidation ungesättigter Fettsäuren mit gerader C-Atom-Zahl Schritt 2: Hydratisierung → Addition von H2O an die neue Doppelbindung Schritt 3: NAD+-abhängige Oxidation (die eigentliche β-Oxidation) → Oxidation am β-C-Atom; Gewinnung von NADH für die Atmungskette 7. Lipide 7.2 Funktion der Lipide Beispiel: β-Oxidation ungesättigter Fettsäuren mit gerader C-Atom-Zahl Schritt 4: Thiolyse weiter in Schritt 1, bis 2 Acetyl-CoA übrig z.B. Citratzyklus - pro Runde: ca. 1,5 – 2,5 ATP aus FADH2 und NADH - zusätzlich ca. 10 ATP aus Acetyl-CoA-Abbau über Citrat-Zyklus - z.B. Palmitinsäure (C16H32O2): ca. 106 ATP bei vollständigem Abbau