5b. Epigenetsche Regulierung.word
Werbung

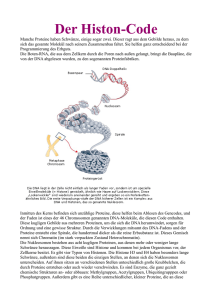
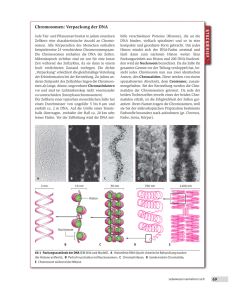
5b. Epigenetische Regulierung Epigenetische Regulierung 1. Die epigenetische Regulierung der Transkription Dia 1. Die Struktur des Chromatins Nach der S-Phase des Zellzyklus gibt es in den Zellen ca. 4x1 Meter langen DNA, in 4x23 Stücke geschnitten (4N-Chromosomensatz gibt es im Zellkern in dieser Phase). Diese lange DNA Fäden müssen kompaktiert werden, damit die Trennung der Chromosomen in die Tochterzellen einwandfrei ist. Dies ist eine schwierige Aufgabe, und die Histone sorgen für die Durchführung. Neben der Chromosomenbildung die andere Funktion der Histone ist die Regulierung der Transkription. Das Chromatin besteht aus DNA, Histonen und Nicht-HistonProteinen. Die Grundeinheit des Chromatins sind die Nukleosomen die aus 146 Basenpaare langen DNA und einem Histonoktamer bestehen. Die Nukleosomen sind durch ein LinkerDNA verbunden. Die Histonoktamere bestehen aus zwei H2A, H2B, H3 und H4 Histonmolekülen, so gibt es insgesamt acht Histon-Moleküle (oktamer) in einem Nucleosom. Die Rolle von H1-Moleküle ist die Verbindung der Nukleosomen. Die Histon-Proteine sind sehr konservativ, zum Beispiel die H4-Untereinheit des Menschen und der Erbse unterscheiden sich von einander nur in zwei Aminosäuren. Die sogenannte konstitutive Heterochromatin Region der DNA (vor allem die Enden und die Centromer-Region der Chromosomen) ist ein hoch gewickelten Zustand, und wird nie transkribiert. Die fakultative Heterochromatin kann je nach Zelltyp transkriptionell aktiv sein. Die eukromatinische Regionen der DNA sind in der Regel reich an Genen, sind leicht mit Histonen bedeckt, und sind entweder ständig oder auf Induktionswirkung transkribiert. Methylierung der Histone beeinflusst die Transkription meistens (nicht immer) negativ, Acethylierung der Histone beeinflusst die Transkription meistens (nicht immer) positiv. Histon-Moleküle können noch mit Phosphat-Gruppen, mit Ubiquitin oder mit einem SUMO-Peptid modifiziert werden, die alle die Histon-DNA-Bindung und damit die Transkription beeinflussen. Nicht nur Histone, sondern auch die DNA methyliert werden. Stark methylierte DNA-Abschnitte werden nicht transkribiert. Dia 2. Chromatinmodifikation Regulation des Chromatin (Slide:7) Regulation der Histone – DNA Bindung ermöglicht verschiedene Chromatinzustände, die zu ganz spezifischen ‘readouts’ (Auslese) der genetischen Information führen, wie z.B. Genaktivierung oder Gensilencing. Zielpunkt der Chromatin-Modifikation kann die DNA sein, die methyliert werden kann; bzw. das Histon, welches methylierungs-, acethylierungs- und andere chemische Modifikationen erleben kann. Die chemischen Modifikationen werden von unterschiedlichen Enzymen (Methylase, Acethylase, usw.) durchgeführt, deren Expression und Wirkung von nicht-kodierenden RNS reguliert werden. Die Acetylierung von Histon-Proteinen entfernt positive Ladungen, womit die Affinität zwischen Histonen und DNA reduziert wird, was letztendlich das Binden von RNA Polymerase und Transkriptionsfaktoren an die Regulatorsequenzen erleichtert und die Transkription ermöglicht. Die Histon-Acetyl-Transferase (HAT)-Enzym verknüpfen LysinAminosäuren der einzelnen Histone mit einer Acetyl-Gruppe (-COCH3), während Deacetylasen entfernen die Acetylreste. Methylierung von Histonen und DNA Sequenzen führt zum gegenteiligen Effekt wie die Acetylierung. Methylierung von Histonen bedeutet das Verknüpfen einer CH3-Gruppe an die N-terminalen Lysin oder Arginin Aminosäuren der Histone und resultiert in einer stärkeren Bindung der Histone zur DNA. DNA kann methyliert (aber nie acethyliert) werden. Hier werden an einzelne Cytosine CH3-Gruppen durch DNAMethyltransferasen angehängt. DNA-Demethylasen können diese CH3-Gruppen entfernen. Grundanforderung Vorlesung 7. Boldogkői Zsolt © 1 5b. Epigenetische Regulierung Bei der Methylierung von DNA-Sequenzen stellte sich heraus, dass dadurch die Genexpression in diversen Zellen stillgelegt wird. All diese Typen von Chromatinmodifikationen regulieren die Zugänglichkeit der Transkriptionsfaktoren an die DNA. Dia 2 Funktionelle und strukturelle Domäne des Chromatins Einige Gene und Gencluster werden durch sogenannte Nukleosome-abstoßende-Sequences (nucleosome repulsing sequences) umklammert, z.B. die Matrix attachment region (MAR), die Locus control region (LCR) und die Scaffold-associated region (SAR) Sequenzen. Diese Sequenzen zeigen eine Hemmung der Chromatinkondensation über ihre abstoßenden Effekte auf Nukleosome, sodass sie eine Transkription von DNAAbschnitten, die sie umklammern, erlauben. Dia 3 Die verschiedenen chemischen Reaktionen (Acetylierung, Methylierung, etc) können die Chromatinstruktur auf drei Arten ändern: (1) Stärkung oder Lockerung der Struktur der Nukleosomen (2) Verschiebung oder (3) Entfernung der Nukleosomen. Das wird ChromatinRemodeling genannt. Die letzten 20-Aminosäuren an der N-terminalen Ende der Histone erstrecken sich auf der Oberfläche der Nukleosomen. Dieser Teil der Histon ist ein Target einer Vielzahl von modifizierenden Enzymen, die die Histon-DNA-Bindungsstärke beeinflussen. Die DNA ist negativ, die Histone positiv geladenen. Die positive Ladung der Histone entsteht in erster Linie von den Lysin-Aminosäuren. Die N-terminalen Histoneenden enthalten viele Lysine und diese Aminosäure ist das Target der Histon-modifizierenden Enzyme. Dia 4 Acetylierung der Histone bedeutet die chemische Veränderung der N-terminalen Lysin-Aminosären, wo ein H-Atom durch ein Acetylrest (COCH3) ersetzt wird. Durch diesen Prozess nimmt die positive Ladung des Histons ab, und damit wird seine Affinität zur DNA reduziert. Diese chemische Reaktion wird von Histon-Acetyltransferasen (HAT) Enzyme katalysiert. Es gibt mehrere verschiedene HATs. Die Histon-Deacetylase (HDAC)-Enzyme, haben eine entgegengesetzte Wirkung. Sie entfernen Sie die Acetylreste von den Histonen und stabilisieren damit die Histon-DNA-Wechselwirkungen, und hemmen dadurch die Transkription. Sowohl die HAT als auch die HDAC-Enzyme, bestehen aus mehreren Untereinheiten und werden Multi-Protein-Komplexen genannt. Die hyperacethylierten Histone kommen hauptsächlich in transkriptionell aktiven DNA-Segmenten, während die hyperacethylierten Histonen in den nicht-transkribierten DNA-Regionen vor. Bestimmte aktivierende oder hemmende Transkriptionsfaktoren regulieren direkt die HistonAcetylierung. Andere Transkriptionsfaktoren beeinflussen die HAT- oder die HDAC-Enzyme (zB durch Phosphorylierung). Die Acetylierung beeinflusst die Chromatinstruktur: (1) reduziert die positive Ladung des Histons und damit seine Bindung an die DNA, was den Zugang der Transkriptionsfaktoren an die DNA fördert, (2) verändert den Kontakt zwischen dem Histon und dem benachbarten Histon oder verändert die Affinität zwischen regulatorischen Proteinen und Histonen. Bestimmte Krankheiten (zB Krebs) werden durch eine Dominanz der Deacethylierung der Histone gekennzeichnet. So werden Gene, die den Zellzyklus hemmen, inaktiv. Einige Möglichkeiten zur Tumortherapie basieren auf Blockierung der Deacetylasen mit verschiedenen Medikamenten. Es gibt zwei versciedene HAT-Enzyme, die an verschiedenen Phasen der Transkription wirken. Das eine wirkt in der Initiationsphase, eine ander wirkt in der Elongationsphase. Grundanforderung Vorlesung 7. Boldogkői Zsolt © 2 5b. Epigenetische Regulierung Dia 5 Die Histonmethylierung (5a) bedeutet das Ersetzen von 1, 2 oder 3 H-Atome von Lysin Aminosären der Histone durch 1, 2 oder 3 Methylgruppen –(CH3), wodurch MethylLysin, Dimethyl-Lysin oder Trimethyl-Lysin entsteht. Manchmal kann die ebenfalls positiv geladene Aminosäure Arginin methyliert werden. Eine gleichzeitige Lysin-Acetylierung und Methylierung am gleichen Histonmolekül schließen sich gegenseitig aus. Die Methylierung von Histone beeinflusst nicht ihre Ladung, sondern verändert die Wechselwirkung zwischen Histonen miteinander und mit regulatorischen Proteinen. Die Methylierung in der Regel verstärkt die Histon-DNA-bindung, wodurch die Transkriptionsaktivität eines DNA-Segments gehemmt oder sogar blockiert wird. (In manchen Fällen kann Methylierung, besonders die Arganinmethylierung, eine offene Chromatinstruktur zur Folge haben, und die Transkription positiv beeinflussen.) Die Histonmethylierung wird durch spezifische HistonMethyltransferase (HMT) Enzymen durchgeführt. Wie die Acethylierung, ist auch die Methylierung reversibel (umkehrbar). Die CH3-Gruppe der Arginin und Lysin-Resten der Histone wird durch verschiedene Histon-Demethylase Enzymen entfernt. (5b) Die HP1 (Heterochromatin Protein 1) Protein erkennt in der H3 Untereinheit ein methyliertes Lys9 (Lysin in der neunten Position in der Histon H3 Untereinheit). Danach bindet HP1 ein Histon-Methyltransferase Enzym, das das H3-Lys9 der benachbarten Nukleosomen methyliert. Also durch eine positive Rückkopplung entsteht ein stark kondensiertes Chromatin. (5c) Während der Zellteilung wird die Methylierungsmuster an die Tochterzellen vererbt. Die neuen Zellen enthalten die halbe Anzahl von methylierter Nukleosomen, aber sie ziehen Histon-Methyltransferase (HMT) Enzymen durch den oben genannten Mechanismus an, und dadurch werden auch die neuen Nukleosomen in der gleichen DNA-Region methyliert. Da die Muster der Nukleosomen den Zelltyp beeinflusst, dieser Mechanismus bietet eine Möglichkeit, wodurch aus einer Mutterzelle, zwei Tochterzellen des gleichen Typs entstehen. Ubiquitinirung der Histone. Das Ubiquitin-Peptid bestehet aus 76 Aminosären. Das Ubiquitin, ähnlich wie die Acetylreste reduziert die positive Ladung der Histone durch das Auslöschen der positiven Ladung der Lysine. Darüber hinaus Ubiquitin selbst enthält eher negativ geladene Aminosäuren. Im Gegensatz zu den Methyl-und Acetylgruppen, wird Ubiquitin, nur an Lysine in der Position 119 in H2A und in der Position 120 in H2B angehängt. Die H2B-Ubiquitinirung stimuliert, die H2A-Ubiquitinirung hemmt die Transkription der DNA. Die Histon-Sumoylierung bedeutet das Anhängen eines SUMO- Peptids (kleine, Ubiquitin-verwandte modifier = small ubiquitin-related modifier) an die Lysine der Histone. Das sumoylierte Lysin zieht die HDAC (Histone Deacetylase) Enzyme an, und verursacht damit den Verlust der Acetyl-Gruppen des sumoylierte Histonmoleküls. Die Phosphorylierung von Histonen bedeutet das kovalente Anhängen Phosphat- (PO4) Gruppe an einem Histonmolekül durch Kinase-Enzyme. Phosphatasen entfernt die Phosphat-Gruppen von den Histonen.Im Gegensatz zu anderen chemischen Modifikationen der Histonproteine, die Phosphorylierung findet nicht an Lysinen, sondern an die N-terminalen Serin und Threonin Aminosäuren statt. Auch die H1 Histone werden phosphoryliert. Die Phosphat-Gruppe ist negativ geladen, wodurch die positive Nettoladung von Histonen verringert wird, wodurch ein offener Chromatinentsteht, was die Transkription fördert. Dia 6 DNA-Methylierung. Nicht nur die Histone, sondern auch die DNA methyliert werden. Dadurch entsteht eine kompakte Chromosomenstruktur und die Transkription wird gehemmt. Diese chemische Reaktion wird durch die DNA-Methyltransferase-Enzyme durchgeführt, in der Regel an der Cytosin-Komponente der CpG-Dinukleotiden ( "p" bedeutet, dass die zwei Nukleotiden durch Phosphodiesterbindung miteinander verbunden sind). Die eine Funktion der DNA-Methylierung ist die Regulierung der Genexpression. Die stark methylierte DNA (z. B. das Heterochromatin) ist transkriptionell inaktiv. Die Methylierung von DNA durch zwei verschiedene Mechanismen durchgeführt: (1) Durch Aufrechterhaltende-Methylierung wird sichergestellt, das die beiden Tochterzellen nach der Zellteilung, die gleiche DNAGrundanforderung Vorlesung 7. Boldogkői Zsolt © 3 5b. Epigenetische Regulierung Methylierung haben, wie die Mutterzelle. Die Dnmt1 (DNA-Methyltransferase1) erkennt die Methylgruppe am Cytosin im alten DNA-Strang, und hängt eine andere Methylgruppe auf das Cytosin des neuen DNA-Strangs an. So ist Dnmt1 dafür verantwortlich dass die DNAMethylierungsmuster während der Zellteilung geregelt vererbt wird. Da die DNAMethylierungsmuster den Zelltyp beeinflusst, dieser Mechanismus bietet eine Möglichkeit, wodurch aus einer Mutterzelle, zwei Tochterzellen des gleichen Typs entstehen. (z. B. zwei Epithelzellen aus einer Epithelzelle). (2) Die de novo Methylierung von DNA führt zum Anhängen neuer Methylgruppen. Die wichtigsten Enzyme sind Dnmt 3a und Dnmt-3bMethyl-Transferasen. DNA-Methylierung wirkt negativ auf die Transkription. Das MeCP2-Protein ist für die Bildung stark methylierten DNA-Abschnitte (Heterochromatin) zuständig. Dieses Protein erkennt methylierte Cytosine und baut um sich ein grosses Proteinkomplex auf. Ein Bestandteil dieser Proteinkomplexes ist einer Histon-Deacetylase (HDAC), dass die Acetyl-Reste von den Histonen entfernt und so zu einer kompakteren Chromatin führt. Die Mutation des MeCP2-Protein im Menschen verursachen RettSyndrom. Das Muster der Methylierung ist unter bestimmten Bedingungen reversibel, Demethylierungsenzyme entfernen die Methyl-Gruppen. Dia 7 Human Epigenom Projekt (HEP) ist ein Projekt bei welchem mehrere Laboratorien zusammenwirken. Zielsetzung des Projekts ist die Erkundung des Methylation-Musters der DNA. Deshalb nur der DNA, weil diese viel einfacher zu untersuchen ist, wie die Methylierung der Histone. Vor Kurzem (Oktober 2009) wurde das erste Ergebnis veröffentlicht: das Epigenom der menschlichen Stammzellen und Fibroblasten. Dia 8 Manche aktivierende Chromatin-Proteine sind in der Lage Chromatin durch direkte Entfernung Histonen zu modifizieren. Zum Beispiel die Bindungsstelle (HSE) des Hitzeschock-Faktors (HSF) wird durch Histone abgedeckt. Der sogenannte GAGA-Faktor bindet den entsprechenden DNA-Abschnitt (GAGA), bewegt die Histone (das Nukleosom zerfällt) und so kann das HSF-Protein das HSE-DNA-Abschnitt binden und die Transkription starten.. 2. Embryogenese Dia 9 Zelltypbestimmung Im Laufe der Ontogenese, oder als Ergebnis einer Geweberegeneration aus den Stammzellen, entwickeln sich differenzierte Zellen, die sich selbst gleichende Zellen produzieren. Dieser Mechanismus wird durch das Aufrechterhalten des Methylationsmusters des Histons und der DNA gesichert (siehe Vorlesung „Genetische Regelung“). Obwohl also jeder Zellentyp die gleiche genetische Information enthält wird eine Epithelzelle immer Epithelzellen herstellen und nie Nervenzellen. Im Laufe der Differenzierung bekommen die nicht-differenzierten Zellen unterschiedliche, einander folgende Anweisungen, das sieht ungefähr so aus: sei solch eine Zelle, aus der später irgendeine Nervenzelle oder ein Glia entstehen kann. Die darauffolgende Anweisung bezieht sich darauf, dass es keine Gehirnzelle sondern eine Zelle im Rückenmark sein soll. Danach bekommt die Zelle Anweisungen Axone zu bilden, dann neuronale Kontakte zu knüpfen, und fertig ist der Motoneuron des Rückenmarkes. Man kann sehen, dass die Potenz der Zelle sich in jegliche Richtungen zu entwickeln im Laufe der Differenzierung immer geringer wird. Dia 10 Klonierung Da die Differenzierung der Zellen ein epigenetischer Prozess ist (keine Änderung in der DNS Sequenz), die Körperzellen sind theoretisch neu programmierbar und aus ihnen können der Zygote ähnliche totipotente Zellen hergestellt werden. Die Klonierung basiert auch auf dieser These (das Thema wird detaillierter im 2. Semester behandelt). Wenn man den Kern einer Körperzelle (z.B. Epithelzelle) in den einer von ihrem Grundanforderung Vorlesung 7. Boldogkői Zsolt © 4 5b. Epigenetische Regulierung Kern befreiten Eizelle tut vergisst der Zellkern, dass er i Wirklichkeit eine Epithelzelle war, wird totipotent und so kann aus der auf diese Weise hergestellten Zygote sogar ein Neugeborenes entstehen. Momentan ist das Interesse an Möglichkeiten durch epigenetische Modifikationen in den Nachwuchs zu bringen groß. Dia 11 Der Histon-Kode Hypothese nach läuft während der Ontogenese die Differenzierung der Zellen anhand eines vorher festgelegten Planes ab. Hauptsache des Planes ist die zeitlich programmierte Änderung des Ankoppelungsmusters von den Histon-Molekülen an die DNS. Dieser Prozess ergibt unterschiedliche Zellenarten in welchen sich für die Zellenart typische Gene exprimiert werden. Wie bekannt, regelt die Affinität der Histon Molekülen der DNS gegenüber die Methylation und die Acethylation (auf dem Bild ist nur die Methylation abgebildet), und deshalb ist das Programm eigentlich das Programm der chemischen Modifikation der Histons. Alle diese Informationen sind letztendlich genetisch determiniert, was so der Definition wiederspricht (epigenetische Vererbung: von der DNS Basis Reihenfolge unabhängige Informationübertragung). Viele halten die epigenetische Vererbung nur als Erscheinungsmechanismus von genetischen Informationen und mögen deshalb den Ausdruck „Epigenetik“ nicht. Frage ist, ob das epigenetische Programm im Stande ist an derselben DNS Alternativen zu produzieren. Dia 12 Epigenetischer Kode im Gegensatz zum Histon Code wird hier nicht nur die Histonmethiliertheit sondern auch jeder epigenetischer Prozess beachtet. Dia 13 Epigenetisches Programm ist nur der Expressionsmechanismus der genetischen Information, jedenfalls nach einigen Meinungen. Anderen nach laufen das epigenetische und das genetische Programm parallel, das heißt, die epigenetischen Prozesse sind jedenfalls teilweise unabhängig vom genetischen Programm. Die großen Fragen sind: was ist die Rolle der folgenden Faktoren beim Verwirklichen des genetischen Programms?: eigene DNS, Ausgangsvoraussetzungen, Umgebung. Dia 14 Kanalisierung. Wie das epigenetische Programm, das die Ontogenese reguliert, durch die in der DNA kodierte genetische Information bestimmt? Wie werden diese Prozesse durch die Umwelt beeinflusst? Conrad Hall Waddinton das Konzept der Kanalisation eingeführt, um die Ontogenese zu erklären. Er sagte, dass die Entwicklung einem angegeben Weg folgte und innerhalb eines bestimmten Wegs gegen Störungen (Mutation und Umwelt) resistent wäre. An kritischen Zeitpunkten aber starke Wirkungen (die genetisch programmiert sein können) können den Entwicklungsweg in eine neue Richtung ablenken Heute nennen wir den Widerstand gegen solche Perturbationen Robustheit. Ein Beispiel: Bie Männern lenkt TDF (testis bestimmender Faktor = testis determining factor) die Entwicklung von der weiblichen Richtung in die männliche. Dia 15 Die DNA und die Umgebung haben Einfluss auf das genetische Programm. Die Verwirklichung des epigenetischen Programms wird grundsätzlich durch die DNA gesteuert indem sie zur rechten Zeit und in richtiger Menge die Entstehung der die epigenetischen Prozesse direkt kontrollierenden Faktoren in den einzelnen Zellenarten reguliert. Grundanforderung Vorlesung 7. Boldogkői Zsolt © 5 5b. Epigenetische Regulierung 11 3. Die Inaktivierung der X Chromosoms Dia 16 Die Inaktivierung des X Chromosoms bedeutet das eine Exemplar der X Chromosomen im weiblichen Säugetier nicht mehr funktioniert und nur einige Gene am X Chromosom aktiv sind. Das eine X Chromosom gerät durch die starke Methylierung der DNA in einen inaktiven Zustand. In Folge des Prozesses gibt es in den Zellen der Frauen nicht die doppelten Chromosom X Produkte (Proteine), wie in denen, der nur über ein X Chromosom verfügenden Männer; dies ist die Dosiskompensation. Das Maß der Genexpression ist nämlich optimalisiert, zweifache Dosis-Änderungen konnte man nicht überleben. Bei den höheren Säugetieren wird zufällig entschieden welches X Chromosom (mütterliches oder väterliches) inaktiviert wird, aber bei den Nachwuchszellen bleibt diese Entscheidung später auch aufrechterhalten. Bei den Beuteltieren (z.B. Kenguru) werden immer die väterlichen X Chromosomen inaktiviert. Den Inaktivierungsprozess startet eine am X Chromosom kodierte große, nicht kodierende RNA (XIST; X-inactive specific transcript). Die XIST RNA bezieht das inaktive X Chromosom, welches so eine Heterochromatisation erlebt, so wird deren Aktivität verhindert. Am aktiven X Chromosom wird das Xist Gen ebenfalls durch eine Heterochromatisation gehemmt. Die X Chromosome, welche nicht fähig sind das Xist Gen zu exprimieren, sind auch nicht fähig inaktiv zu werden. Interessant ist, dass der Hauptregulator der Xist Gens eine vom komplementerem Strang des Gens tanskribierte antisens RNA, namens Tsix ist. Notizen: Grundanforderung Vorlesung 7. Boldogkői Zsolt © 6