Zusammenfassung Stunde 1
Werbung

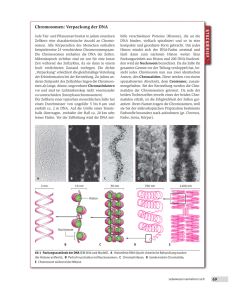
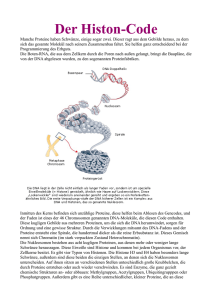
Zusammenfassung Stunde 1 Die Bedeutung der Chromatinstruktur für Chromosomensegregation und Regulation der Genexpression. Nukleosomen haben 147 bp ds DNA um ein Oktamer aus Core Histonen (2 H2A/H2B Dimeren und ein H3/H4Tetramer) als Superhelix gewunden. zusätzlich findet man Linker Histone (z.B. H1) die an der Kondensation beteiligt sind. spezielle Histonvatianten könnengegen die 4 Core Histone ausgetauscht werden um spezielle Chromatinbereiche mit speziellen Funktionen zu definieren. H2AX z.B. für Recombiniation oder CENPA für die Kinetochorefunktion. Nukleosomen sind dynamische Strukturen und werden dadurch für nicht Histonproteine zugänglich. Histon Chaperone vermitteln die Beladung der DNA mit Histonen bei der Replikation und den Austausch von Histonen in der Interphase. Histone sind konservierte Proteine mit N-terminalen, ungeordneten Regionen, (talis) wo die meisten kovalenten Modifikationen zu finden sind. 29 Beginn Stunde 3 Klausur zum Kernmodul: Fr. 13.2.09 10 Uhr, Hörsaal G. 30 genetische Identifizierung von Chromatinkomponenten 31 ade2- Chromosomenverlust ADE2+ ade2- 32 SMC1 / SMC3 / SCC1 / SCC3 sind Komponenten eines Multiproteinkomplexes der Schwesterchromatiden zusammenhält: Cohesine SMC1 /SMC3 „Cohesine“ SCC1 /SCC3 Cohesine bilden höchstewahrscheinlich einen Ring um die Schwesterchromatiden 33 Cohesion von Schwesterchromatiden wird bei der Replikation etabliert. 34 Eines der Modelle für die ATP- abhängige Öffnung des Proteinrings bei Ausbildung der Cohesion zusätzliche Faktoren, sogenannte Adherine (SCC2; nibbed-B) werden benötigt, um Cohesin Moleküle an DNA zu binden. Shintom and Hirano, (2007) TIBS, 32, pp154ff Cohesine binden entlang der Chromosomenarme und an den Centromerregionen Wie wird die Cohesion aufgehoben? 36 SCC1 (Kleisin) wird proteolytisch durch das Separin Esp1 gespalten SMC1 /SMC3 Separin Securin (Pds1) SCC1 /SCC3 Securin-Degradation Ubiquitinylierung Die Aktivität von Securin und damit der Beginn der Anaphase wird durch die Ubiquitin-Protein-Ligase (E3) APC ( Anaphase-promoting factor) 37 reguliert. Cohesine beeinflussen auch die Genexpression Cohesine findet man auch an Insulator Elementen die Gene von der Wirkung von Enhancern ander Genloci isolieren. Der Verlust von Cohesinen kann zum Überlesen solcher Insulator Elemente führen. 38 Proteine in Heterochromatin 39 Proteine in Heterochromatin wurden biochemisch und genetisch identifiziert: Das HP1 Protein (Sarah Elgin 1986) Ein „non-histon“ Protein, dass aus Chromatin von Drosophila isoliert wurde. Der Su(var) 2-5 Lokus codiert für HP1. Polycomb Gruppe von Proteinen (PcG) Ein holologer Bereiche in diesen Proteinen wird als Chromodomäne bezeichnet („chromatin organisation modifier“ erkennt Histonmodifikationen s. später) 40 Ist die Struktur von Chromatin wichtig für die Regulation der Gen-Expression? Es gibt lokale und globale Unterschiede in der Chromatinstruktur. Die Modifikation von Histonen ist dabei von großer Bedeutung. Histon Chaperone (beladen DNA mit Histonen) Chromatin Remodelling Komplexe (verschieben Nukleosomen auf der DNA) Histon – Acetyltransferasen Histon – Deacetylasen Histon – Methylasen Histon – Demethylasen Acetylierung von Histonen entfernt positive Ladung, schwächt die Histon-DNA Wechselwirkung – DNA wird zugänglicher. 41 Histon – Acetyltransferasen und Deacetylen (HAT und HDACs) Die Bedeutung der Histonmodifikation für die Genregulation wurde früh postuliert, aber erst 30 Jahre später bewiesen. Histonacetylierung: DM Philips (1963) Biochem J. Vol. 87, 258ff Allfrey et al. (1964), PNAS Vol.51, 786ff 42 Die Bedeutung der Histonacetylasen und Deacetaylasen Histonacetylasen : HATs Histondeactylasen: HDACs wichtige Entdeckungen: 1. Es stellte sich heraus, dass Gene für biochemisch charakterisierte Histon modifizierende Enzyme homologie zu Genregulatoren der Bäckerhefe haben. 2. Es stellte sich heraus, dass biochemisch als „Coaktivatoren“ identifzierte Protein ( CBP) Histonactetyltransferaseaktivität hatten. 43 Isolierung des ersten Gens für eine Histonacetyltransferase 1995 R. Sternglanz: HAT1 in Hefe. 250 ts-Mutanten – Ganzzellextrakte – Ionenaustauscher – Acetylierungsassay mit H3 - AcetylCoA und Histone H4 Peptid (Aminosäuren 1-28 ) eine Mutante hatte nur 50% Aktivität. (die ts- Mutation war allerdings nicht mit der Mutation in HAT1 gekoppelt.) 44 Ja das hat funktioniert ! D. Allis und Mitarbeiter , (1996) Cell, Vol. 84, p. 843 ff Tetrahymena Histone Acetyltransferase A: A Homolog to Yeast Gcn5p Linking Histone Acetylation to Gene Activation Gcn5 : Ein Genregulator des Stickstoffhaushalts des Hefe 45 A mammalian histone deacetylase related to the yeast transcriptional regulator Rpd3p. Science 19 April 1996: 408 Taunton J, Hassig CA, Schreiber Taunton J, Hassig CA, Schreiber SL.Howard Hughes Medical Institute, Harvard University, Cambridge, MA 02138, USA.Trapoxin is a microbially derived cyclotetrapeptide that inhibits histone deacetylation in vivo and causes mammalian cells to arrest in the cell cycle. A trapoxin affinity matrix was used to isolate two nuclear proteins that copurified with histone deacetylase activity. Both proteins were identified by peptide microsequencing, and a complementary DNA encoding the histone deacetylase catalytic subunit (HD1) was cloned from a human Jurkat T cell library. As the predicted protein is very similar to the yeast transcriptional regulator Rpd3p, these results support a role for histone deacetylase as a key regulator of eukaryotic transcription. 46 Die Entdeckung, dass Transktiptionsfaktoren an Histonmodifizierende Enzyme binden Das Konzept der Co-Aktivatoren /Co-Repressoren Bindung von Co-Activatoren (Co-Repressoren) POL II + gen. TF Coaktivator lokale Veränderung der Chromatin-Struktur? Identifizierung: Identifizierug von Proteinen die and den Thyroid Hormon Rezeptor (DNA bindendes Protein; Tx-Faktor) binden oder die an den Regulator CREB (cAMP) binden (s. L. Nitschke) 47 Nature, 1996, Vol 384 pp 641 ff 48 Histon Acetyl-Transferasen (HATs) oft Multiproteinkomplexe 49 HATs und HDACs werden durch genspezifische Tx- Faktoren an DNA rekrutiert. Aktivierung durch lokale Histon Acetylierung Repression durch lokale Histon Deacetylierung 50 Histon Chaperone (Beladen DNA mit Histonen) Chromatin Remodelling Komplexe (verschieben Nukleosomen auf der DNA) Histon – Acetyltransferasen Histon – Deacetylasen Histon – Methylasen Histon – Demethylasen Chromatin Remodelling Faktoren wurden als Genregulatoren entdeckt SWI/ SNF Gene des Hefe. (SNF: Sucrose non fermeting) (SWI: mating type switching) 51 ATP – abhängig werden Nukleosomen lokal auf der DNA verschoben und von einer DNA auf eine andere transferieren. 52 3d- Reconstruktion eines Nukleosomen /RSC Komplexes ( RSC: Remodelling Structure Chromatin) Leschziner A. E. et.al. PNAS 2007;104:4913-4918 ©2007 by National Academy of Sciences Fitting of a nucleosome into the open and closed conformations of RSC Leschziner A. E. et.al. PNAS 2007;104:4913-4918 ©2007 by National Academy of Sciences Remodelling Komplexe helfen Histon-Chaperonen beim Austauch von Histonen 55 ATP-abhängige "Chromatin-Remodelling" Komplexe sind aus vielen Untereinheiten aufgebaut Pionier: SWI/SNF Komplex) 56 Wie werden die Nukleosomen erkannt, die verschoben werden sollen? Remodelling Komplexe erkennen Histonmodifikationen (Acetylierungen ) Chromatin remodelling Faktoren können mit diesen Domänen spezifische Histonmodifikationen erkennen. Bromodomäne: acetylierte Histone SANT Domäne: unmodifizierte Histone Chromo- Tudordomänen und PHD- Finger: unterschiedliche Methylierungen von Histonen 57 Histon-Modifikationen können gelesen werden. Erkennung unterchiedlicher Methylierungen durch Reader Domänen 58 Der „Histoncode“ Die Kombination spezieller Modifikationen haben unterschiedliche Bedeutung für die Zelle. Histon H3 Modifizierende Enzyme selbst, Remodelling Faktoren und andere „nicht-Histon“ Proteine lesen und schreiben zusammen diesen Code der Histone. 59 Lesen und Schreiben des Codes: Modifikationsmuster können sich so ausbreiten und an die Tocherzellen weitergegeben werden. 60 Analyse der Nukleosomendynamik durch FRET Li and Widom (2004) Nature, vol. 8; pp 763 Keq = 0,1 i.e. teilweise unverpackt 10% der Zeit. 61 Zusammenfassung Stunde 2 +3 Neben Histonen findet man in Chromatin weitere wichtige Komponenten: Topoisomerasen; Condensine ; Cohesine u.a. Condensine und Cohesine sind konservierte Strukturproteine mit ATPase Domänen Cohesine vermitteln Schwesterchromatid Cohesion durch Ausbildung eines Rings um die Chromatiden. Spaltung der SCC1 Komponente durch Separin leitet die Anaphase ein. Ubiquitin-abhängiger Proteinabbau eines Separin Inhibitors reguliert die Auflösung der Cohesion. Entdeckung von Histonacetylasen und Histondeacetylasen Funktion von Chromatin Remodelling Faktoren ( SWI/SNF – Komplex) Der Histoncode: Histonmodifikation werden über konservierte Proteindomänen gelesen und von Chromatinmodifizierenden Enzymen gechrieben. 62