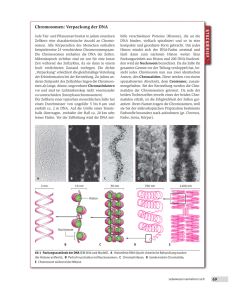
Frage: Sind die Basen in der "Watson
Werbung

Frage: Sind die Basen in der "Watson-Crick" Basenpaarung in der Keto- oder der Enolform? Antwort: In der Keto-Form. Frage: Was ist stabiler, ein A-T oder ein G-C-Basenpaar? Antwort: Ein G-C-Basenpaar (weil es 3 Wasserstoffbrücken bildet). Frage: Gibt es andere als die "Watson-Crick" Basenpaarung? Antwort: Ja. Frage: Wie schnell muss die DNA-Replikation ablaufen, damit sich E. coli Zellen in 20 Min teilen können? Antwort: Das E.coli Genom hat ca. 4,8 x 10 hoch 6 Basenpaare. Bei bidirektionaler Replikation müssen zwei DNA-Polymerase-Komplexe je 2,4 x 10 hoch 6 Basenpaare in 1200 Sekunden replizieren, also ca. 2000 Basenpaare pro Sekunde. Frage: Aneinandergereihte Nucleosomen nennen wir Chromatin. Wie unterscheidet sich Hetero- und Euchromatin? Aufgrund der Histone? Antwort: Unter "Chromation" verstehen wir (durch weitere Proteine) gepackte Nukleosomen-Ketten. Euchromatin ist lose gepackt, Heterochromatin ist fester gepackt. Genexpression findet wahrscheinlich ueberwiegend im Euchromatin statt. Frage: Auf dem diskontinuierlichen Strang werden die Primer von der DNA-Polymerase durch DNA ersetzt. Auf dem kontinuierlichen Strang gibt es zwar nur einen Primer, aber es ist mir nicht klar wie dieser durch DNA ersetzt werden kann. Antwort: Bei der Replikation werden der kontinuierliche und der diskontinuierliche DNAStrang durch die DNA-Polymerase III synthetisiert. Die RNA-Primer werden durch die DNAPolymerase I verdaut und durch DNA ersetzt. Dies gilt sowohl fuer den einen RNA-Primer auf dem kontinuierlichen als auch fuer die vielen RNA-Primer auf dem diskontinuierlichen Strang. Frage/Behauptung: Die Rekombinationsvorgänge in der Meiose selektionieren für optimale genetische Eigenschaften! Antwort: Wir gehen bis heute davon aus, dass Mutationen und Veraenderungen der genetischen Information durch Rekombination zufaellige Prozesse sind. Allerdings gibt es Regionen in Genomen, wo diese Prozesse haeufiger vorkommen als im Rest des Genoms. Wir gehen weiter davon aus, dass es dann die Umweltbedingungen sind, welche entscheiden, ob eine neue genetisch fixierte Eigenschaft in einer Population ueberlebt oder sich gar ausbreitet. Also: zufaellige Mutation und dann Selektion durch die Umwelt, wie zuerst von Darwin postuliert. Es gibt ab und zu Diskussionen darueber, ob diese einfache Ansicht wirklich befriedigend sei. Bisher erklaert sie aber die Beobachtungen der Genetiker und bleibt damit die akzeptierte "Modell-Vorstellung". Frage: Was versteht man unter "hyperchromer Effekt"? Antwort: Wenn die Basenstapelung aufgehoben wird, absorbieren die Basen bei 260nm staerker, das nennt man den hyperchromen Effekt. Frage: Warum haengt der Schmelzpunkt einer DNA-Doppelhelix von der Salzkonzentration der Loesung ab? Antwort: Weil Kationen durch Binden an die Phosphate (Anionen) negative Ladungen und damit abstossende Kraefte zwischen den beiden Ketten der Doppelhelix neutralisieren. Frage: Das Histon H1 sitzt zwischen den Histon-Oktameren auf der DNA. Was macht es dort? Antwort: Da das Histon H1 zwischen den Nukleosomen sitzt, kann es benachbarte Nukleosomen binden, bringt sie sehr nahe zusammen und packt dadurch die DNA dichter. Frage: Aufgrund der Phosphatgruppe ist DNA negativ geladen. Ich habe gelesen, dass Kationen die DNA neutralisieren; doch Histone kompensieren ebenfalls neg. Ladungen der DNA. Was ist wann beteiligt? Antwort: Im Chromatin passiert beides. Phosphatgruppen, die zum Histon gerichtet sind, koennen durch positive Ladungen auf dem Eiweiss (protonierte Aminogruppen) neutralisiert werden. Phosphatgruppen, welche nach aussen stehen (von den Histonen weg), koennen durch andere posiv geladene Gruppen (vorwiegend K+) neutralisiert werden. Frage: Solenoid = Verknüpfung von Nukleosomen durch Histon H1. Heisst das, dass Solenoide Chromatide sind? Was sind Solenoide genau, und wo findet man sie? Antwort: Solenoid ist DNA, die um Histon-Oktamere gewickelt ist. Die Histon-Oktamere sind durch H1 miteinander verbunden. Solenoid ist also eine kondensierte Form der DNA. In dieser Form liegt ein Teil der DNA unserer Chromosomen in der Interphase vor. Begriffe: Chromatin=DNA-Eiweiss-Komplexe im Interphasenzellkern. Chromatid=Hälfte eines replizierten Chromosoms vor der Verteilung in die Tochterzellen. Frage: Was ist der Unterschied zwischen der DNA-Polymerase I und III? Sie erwähnen diese auf der CD, aber ich habe nirgends eine Unterscheidung gefunden. Antwort: Die DNA-Polymerase III repliziert die DNA, den kontinuierlichen und den diskontinuierlichen Strang gleichzeitig. Sie ist also das eigentliche Replikations-Enzym. Die Polymerase I entfernt die RNA-Primer und ersetzt sie durch DNA. Frage: Wie findet die DNA-Glykosylase welche Base verändert ist? Antwort: Die Glycosylase erkennt Uracil, welches durch Desaminierung von Cytosin entsteht. Das resultierende G-U Basenpaar (das in der DNA wegen der Geometrie der Doppelhelix keine H-Brücken bilden kann) wird vom Enzym erkannt und Uracil vom Zucker weggeschnitten; es entsteht ein "Loch" (Phosphat-Zucker-Rückgrat ist intakt, aber eine Base fehlt!). Dieses Loch wird von einem Reparatur-Enzymkomplex erkannt. Dieser depolymerisiert den defekten Einzel-Strang der Doppelhelix in der ganzen Region (welche den Defekt umgibt) und synthetisert den in dieser Region Strang neu. Frage: Welche Rolle spielt die Position von Operatoren in prokaryotischen Genen? Antwort: Ein Operator ist eine Bindungsstelle für ein Protein, welches die Transkription aktiviert oder reprimiert. Die Lage des Operators ist damit vom Protein und dessen Funktion abhängig. Beim Lac-Operon bindet der Operator II den Repressor. Der liegt nach der Polymerase-Bindungsstelle und kann damit die Wanderung der RNA- Polymerase hemmen. Der Operator I bindet den Aktivator. Die Bindung dieses Proteins oberhalb der Bindungsstelle der RNA- Polymerase stimuliert die Bindung der RNA- Polymerase. Frage: Was bezeichnen die Buchstaben G, E, S und I in Figur 19 (Regulation der Transkription durch ein Hormon)? Antwort: Da fehlt tatsächlich die Legende bei dieser Figur! G=G-Protein (GTP-bindendes Protein, welches nach Kontakt mit dem aktivierten Rezeptor Enzyme aktiviert). E=Enzym. S=second messenger, kleines Signal-Molekuel, z.B. cAMP (cyklisches AdenosinMonophosphat). I=Inhibitor, Protein, welches einen Transkriptionsfaktor bindet und so inaktiv hält. Frage: In einer normalen (sich nicht in der Zellteilung befindend) diploiden Zelle befinden sich beim Menschen 46 Chromosomen , d.h. 23 Chromosomenpaare. Ein Chromosomenpaar besteht doch aus 2Schwesterchromatiden, eine Chromatide vom Vater, die andere von der Mutter (?). Bisher habe ich gedacht, dass diese zwei Chromatiden durch ein Centromer miteinander verbunden sind. Kommt das Centromer nur bei in der S-Phase bei der Zellteilung vor? Antwort: Sie sind jetzt bei diesem wichtigen und vielleicht etwas verwirrenden Punkt "Chromosom". Gehen wir es so an: In der Interphase haben sie 46 DNA-Straenge, jeweils zwei "identische" (je eine Kopie urspruenglich vom Vater bzw. der Mutter, also nicht voellig identisch!). Jeder der 46 DNA-Straenge ist eine Doppelhelix, bezeichnen wir sie als "Interphasen-Chromosomen". Vor der Mitose werden alle 46 DNA-Straenge verdoppelt, bleiben aber zusammen (verbunden ueber die Centromeren). Sie haben dann 46 Chromosomen, jeweils aus 2 Doppelhelices bestehend, nennen wir diese Gebilde "MitoseChromosomen". Jede der Doppelhelices im "Mitose-Chromosom" wird als "Chromatid" bezeichnet. Wenn sich das "Mitose-Chromosom" nun waehrend der Mitose stark verkuerzt wird es im Mikroskop sichtbar und von Cytologen als "Chromosom" bezeichnet. Sie sehen, dieser Begriff ist nicht genuegend gut definiert, deshalb habe ich die Begriffe "InterphasenChromosomen" und "Mitose-Chromosomen" verwendet. Frage: Wie erkennt die RNA-Polymerase von E. coli den zu transcribierenden Strang? Antwort: Der assoziierte Sigma-Faktor erkennt die Basen-Sequenz der Pribnow-Box: Figur (pdf-Format) Frage: Warum wäre eine 3'-5' Verlängerung einer Polynukleotidkette bei Korrekturlesen durch die Polymerase nicht möglich? Antwort: Frage: Beim Studium über das DNA-Fehlpaarungs-Korrektursystem habe ich gelesen, dass ohne Mismatch-Korrektur eine Fehlerquote von 10 hoch -7 , d.h. jedes 10'000'000ste Basenpaar ist fehlerhaft, auftritt. Auf das ganze Genom gesehen (3x10 hoch9 Basenpaare) wären das dann 300 Fehler pro Genom. Dank des zusätzlichen Mismatch-Korrektur-Systems, das zu 99% die Fehler beheben kann, sind das dann noch 300 x 99% = 3 Fehler pro Genom. Das heisst also bei jeder Zellteilung, wo die gesamte Erbinformation auf die Tocherzelle übertragen wird, geschehen im Durchschnitt stets 3 Fehler! Aber wenn man bedenkt das ein einziger Fehler z.B. zu einer Sichelzellanämie führen kann, erscheint mir 3 Fehler pro Zellteilung ungeheuerlich viel!! Wie kann es sein, dass diese 3 Fehler die sich in einem Menschenleben wohl auf das x-fache vervielfachen werden, bei den meisten keine gesundheitlichen oder andere Auswirkungen haben? Antwort: Zum Glück fuehren die allermeisten Mutationen nicht zu einer Veränderung des Phänotyps, d.h. sie haben keinen sichtbaren Effekt. Gründe sind: 1. Nur etwa 5% unserer DNA sind Gene, d.h. kodieren für Proteine; 2. nur Veränderungen jeder 3. Base in einem Gen führen zu Veränderungen der Aminosäuresequenz des Proteins (ich werde das in der Konzeptvorlesung streifen); 3. nicht jede Aenderung der Aminosäuresequenz eines Proteins verändert dessen Aktivität; 4. veränderte Aktivität eines Proteins kann toleriert werden, wenn die gesunde Kopie des Gens (wir sind ja diploid) genügend aktives Protein liefert. Es müssen also viele Bedingungen erfüllt sein, bis eine Mutation (Aenderung des Genotyps) zu einer feststellbaren Veränderung (Aenderung des Phaenotyps) führt. Frage: Wieso renaturieren die häufigen Sequenzen vor den weniger häufigen? Antwort: Renaturierung ist eine Reaktion 2. Ordnung, d.h. abhängig von der Konzentration der Reaktionspartner (die sich finden müssen, um zu renaturieren). Repetierte Sequenzen hybridisiesen deshalb schneller: Frage: Meiner Meinung nach ist die Antwort, die zur Frage der Transkriptionsrichtung im EM Bild (http://www.aum.iawf.unibe.ch/VLZ/BWL/Gen_Kurs/Gen_Kurs/transla/transl01.htm) gegeben wird, falsch. Wenn die kleinen Fäden den Anfang darstellen, müsste die Laufrichtung nicht von rechts nach links wie angegeben, sondern genau umgekehrt sein! Antwort: Die Richtung ist von links nach rechts, wie Sie richtig bemerken. Ich werde versuchen, den Fehler in der on-line Version sobald wie möglich zu korrigieren. Frage: Wie werden Exon-Intron-Grenzen erkannt? Antwort: Splicing-Komplexe erkennen die Exon-Intron-Grenzen aufgrund der Nukleotidsequenzen und einer Intron-Sequenz (mit der Base A, siehe Figur): Frage: In der PCR-Reaktion synthetisiert die tac DNA-Polymerase von den DNA-Primern ausgehend den komplementären DNA-Strang. Wieso wird nur das gewünschte Gen repliziert? Warum hört die Polymerase auf? Antwort: Beim ersten Zyklus "weiss" die Polymerase in der Tat nicht, wo sie aufhören soll, sie bricht spontan ab. In späteren Zyklen definieren die Primer Anfang und Ende. Das ersieht man aus der folgenden Darstellung: Die zu langen Produkte fallen mengenmässig nicht ins Gewicht, weil sie linear vermehrt werden, während die Produkte mit der richtigen Länge exponentiell vermehrt werden. Frage: Sedimentationskoeffizient der Ribosomen: ein 70S Ribosom besteht aus einer Untereinheit 30S und einer zweiten Untereinheit 50S. Warum ist dann das ganze Ribosom nicht 80S, da ja bekanntlich 50+30=80? Geht ein Teil verloren, wenn sich die beiden Untereinheiten zusammenlagern? Antwort: Es geht nichts verloren! "S" steht für "Svedberg" Einheit. S ist ein Mass für die Sedimentationsgeschwindigkeit eines Moleküls in einer Ultrazentrifuge und ist abhängig von der Masse und der Form des Moleküls. Da der Komplex aus 50S und 30S eine etwas "lockerere" Form hat als die einzelnen Untereinheiten ist sein S-Wert kleiner als die Summe der S-Werte der Untereinheiten.