REPLIKATION
Werbung

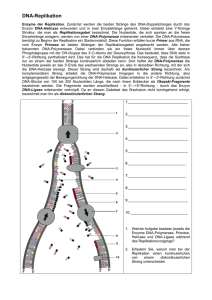
REPLIKATION EM-Aufnahmen von sich verdoppelnder Eukaryoten-DNA zeigen zahlreiche Replikationsblasen. Daraus folgt, dass die DNA-Neusynthese an vielen Stellen des Doppelstranges gleichzeitig abläuft. Molekularer Mechanismus. Die Replikation beginnt mit einer Entwindung des DNA-Doppelstranges durch eine DNA Helicase. DNA-Helicasen sind Enzyme, die unter ATP-Verbrauch die Wasserstoffbrückenbindungen der DNA-Doppelhelix spalten. Die entstehenden Einzelstränge werden durch einzelstrangbindende Proteine vorübergehend stabilisiert, d.h., sie verhindern, dass sich die beiden Einzelstränge sofort wieder verbinden. Auf diese Weise entsteht eine Y-förmige Struktur, eine Replikationsgabel. An beiden Einzelsträngen wird nun mit Hilfe einer Primase (eine RNA-Polymerase), ein RNA-Primer synthetisiert. Der RNA-Primer ist ein kurzer Abschnitt aus Ribonucleotiden, der als Starter für die eigentliche DNA-Replikation durch die DNA-Polymerase notwendig ist. Die DNA-Polymerase kommt in verschiedenen Formen vor. Für die DNA-Replikation sind die DNA -Polymerase 1 und III von Bedeutung. Die DNA Polymerase I entfernt die Ribonucleotide des RNA Primers und ersetzt sie durch Desoxyribonucleotide. Die DNA-Polymerase III ist für die Verknüpfung (= Polymerisation) von Desoxyribonucleotiden zu ständig. Allerdings kann sie die Nucleotide an der Matrize nur in 5‘ —> 3‘-Richtung verknüpfen, d. h., sie kann nur 3‘-Enden verlängern. Die DNA-Neusynthese verläuft daher am so genannten Leitstrang, kontinuierlich: Die DNA Polymerase III katalysiert hier die Bindung von Desoxyribonucleotiden an das 3‘-OH-Ende des wachsenden neuen Stranges.. Der zweite elterliche DNA-Strang, der so genannte Folgestrang, kann jedoch nicht kontinuierlich kopiert werden. Dieser Strang wird diskontinuierlich synthetisiert: Die DNA-Polymerase III kann nur in 5‘ — 3‘-Richtung von der Replikationsgabel weg verknüpfen. Hierzu stellt die Primase zunächst an verschiedenen Stellen kurze RNA-Primer her, die durch DNA-Polymerase III zu Fragmenten, den OKAZAKI Stücken, verlängert werden. Anschließend werden die RNA-Primer von der DNA-Polymerase 1 entfernt und durch Desoxyribonucleotide ersetzt. Die OKAZAKI-Stücke sind allerdings noch nicht mit einander verbunden. Die Herstellung der Phosphodiesterbindung zwischen direkt aufeinander folgenden OKAZAKI-Stücken wird von dem Enzym DNA Ligase bewirkt. Die OKAZAKI-Stücke wurden 1967 von dem japanischen Biochemiker OKAZAKI entdeckt.