30 - Medi
Werbung

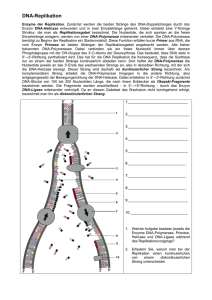
1 Speicherung, Übertragung und Expression genetischer Informationen 2. Helicasen trennen unter ATP-Verbrauch die DNA-Doppelstränge und erzeugen damit die Replikationsgabel, 3. eine Primase (RNA-Polymerase) synthetisiert die Primer-RNA, um ein freies 3´-OHEnde und somit einen Anknüpfungspunkt für die DNA-Polymerase zu schaffen. Das Wachstum des neu synthetisierten DNAEinzelstrangs erfolgt vom 5´ zum 3´- Ende, die Ableserichtung vom elterlichen DNA-Einzelstrang daher vom 3´ zum 5´-Ende. Durch die entgegengesetzte Polarität im DNA-Doppelstrang ist es notwendig, einen Strang um 180° einzudrehen, um so eine identische Syntheserichtung für die DNA-Polymerase III zu schaffen. Hierdurch wird ein Leitstrang von einem Folgestrang unterschieden. 4. Die kontinuierliche DNA-Synthese des Leit­ strangs wird durch die schnell arbeitende DNA-Polymerase III in Richtung auf die Repli­kations­gabel zu ausgeführt. Zur Synthese des Leitstrangs ist pro Replikationsgabel daher nur einmal ein einziger Primer (Synthese durch Primase) zur Lieferung eines freien 3´-OH-Endes notwendig. 5. Der unterbrochene Folgestrang kann durch die Eindrehung um 180° diskontinuierlich (abschnittsweise) nun ebenfalls von der gleichen DNA-Polymerase III in Richtung auf die Repli­kationsgabel synthetisiert werden. Durch das Ein- und Ausdrehen immer neuer DNA-Abschnitte bilden sich die Okazaki-Fragmente, die aus einem RNA-Primer und der neu synthetisierten DNA bestehen. 6. Am Folgestrang (und einmal auch am Leit­ strang) müssen jetzt noch die RNA-Primer durch eine 5´-3´-Exonuklease, die DNA-Polymerase I, herausgeschnitten und durch DNA ersetzt werden. Hierzu besitzt die DNA-Polymerase I eine Ribonuklease- und eine DNAPolymerasefunktion. 7. Im letzten Schritt der Replikation muss das Enzym DNA-Ligase den Kettenschluss zwischen den in Schritt 6 synthetisierten Nukleotid­abschnitten durchführen und die DNA-Teilstücke unter ATP-Verbrauch miteinander verbinden. Hierzu aktiviert die DNA- 30 Ligase das jeweilige 5´-Phosphat-Ende durch Anheften eines AMP-Rests. Die Abspaltung dieses AMP-Rests liefert die Energie für den DNA-Kettenschluss. 5’-Phosphat-Ende O -O P O CH2 Base O O O -O P O CH2 Base O O O -O P O CH2 Base O O OH freies 3’-OH-Ende Abb. 27: Freies 3´-OH-Ende medi-learn.de/6-bc4-27­ Als Cofaktoren für die Replikation der DNA werden die Nukleotide dATP, dTTP, dCTP und dGTP benötigt. –– Die Energie für die Verknüpfung der Nukleotide kommt aus der Hydrolyse des gespaltenen Pyrophosphats (PPi), katalysiert durch Pyrophosphatasen. –– Die DNA-Polymerase III knüpft Phosphodiesterbindungen (1000 Nukleotide/Sec) und besitzt eine Korrekturlesefunktion. –– Die DNA-Polymerase I knüpft Phosphodiesterbindungen (10 Nukleotide/sec), besitzt eine Korrekturlesefunktion sowie eine