als nur Überbleibsel einer vergessenen RNA-Welt - Wiley-VCH
Werbung

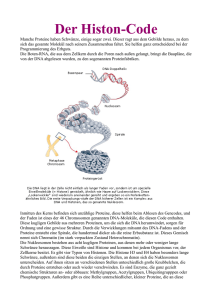
| G E N O M FO R S C H U N G | npcRNAs: Mehr als nur Überbleibsel einer vergessenen RNA-Welt In den vergangenen Jahren rückte eine Gruppe von Molekülen ins Rampenlicht, die zuvor – im Vergleich zur DNA und den Proteinen - weitgehend vernachlässigt wurde: die so genannten nicht-proteincodierenden RNAs, also RNA-Moleküle, die keine Bauanleitung für Proteine enthalten (engl.: „non-protein-coding RNA“, abgekürzt npcRNA). In der Biologie gibt es eine Variante des Henne-Ei-Problems: Was war zuerst da? Gab es zuerst die DNA, die ja die genetische Information für den Zusammenbau von Proteinen enthält? Oder sind zuerst die Proteine entstanden, ohne die wiederum Nukleinsäuren wie die DNA gar nicht synthetisiert werden können? Neue Erkenntnisse deuten darauf hin, dass weder Proteine noch die DNA zuerst existierten, sondern eine andere Molekül-Gruppe: die RNA. Sie kann - anders als die DNA oder Proteine - zwei Funktionen wahrnehmen: RNA kann zum einen genetische Information speichern und vervielfältigen, zum anderen kann sie auch die katalytische Aktivität eines Enzyms haben. Möglicherweise ist am Anfang der Evolution in einer bestimmten physikalischen und chemischen Umgebung zufällig einmal ein RNA-(ähnliches) Molekül entstanden, das in der Lage war, zuerst sich selbst zu reproduzieren und später auch andere RNA-Moleküle nachzubilden. Auf diese Weise entstanden nach und nach immer mehr RNA-Moleküle, die bestimmte Enzym-Funktionen hatten. Gleichzeitig entstand zufällig eine Membran, die die RNAs zusammenhielt: das war die Geburt der ersten primitiven Zelle [1, 3, 7]. Dann entwickelte sich in dieser Zelle aus einem der RNA-Moleküle ein primitives Ribosom, das die Synthese von kleinen Protein-Fragmenten ermöglichte. Die Proteine bildeten also wahrscheinlich die zweite große Gruppe von Molekülen, die auf dem Schauplatz der Zellentwicklung erschien. Nach und nach übernahmen die Proteine die katalytischen Funktionen der RNAs und entwickelten sich zu schnellen und effizienten Enzymen, die den Grundstein für einen weiteren wichtigen Evolutionsschritt legten. Die Proteine machten es möglich, die in der RNA verschlüsselte genetische Information in ein DNA-Molekül umzuschreiben (Reverse Transkription) und umgekehrt konnten sie die DNA als Matrize nutzen, um die © 2006 Wiley-VCH Verlag GmbH & Co. KGaA, Weinheim T R E F F P U N K T FO R SC H U N G genetische Information der DNA in ein RNA-Molekül umzukopieren (Transkription). Auf diese Weise ging die Funktion, genetische Information zu speichern, allmählich von der RNA auf die DNA über. Der Vorgang der Reversen Transkription findet heute noch bei vielen Viren statt, beispielsweise beim HIV-Virus. Diese Viren schreiben mit Hilfe bestimmter Enzyme ihr RNA-Erbgut in ein DNA-Molekül um und sind so in der Lage, ihre Virus-Erbinformation in das SäugetierGenom einzuschleusen. Obwohl bereits mehrere Milliarden Jahre vergangen sind, seit die DNA anstatt der RNA zum Träger der genetischen Information wurde, nutzen auch heute noch viele Organismen die zelluläre RNA als Nachbildungsmatrize, und zwar nicht nur die RNA-Viren, sondern auch Lebewesen mit Zellkern (Eukaryoten). So besteht mit insgesamt 42 Prozent ein beeindruckend großer Anteil des menschlichen Erbgutes aus Elementen, die ursprünglich einmal RNA-Moleküle waren, dann in DNA umgeschrieben wurden und sich schließlich in das menschliche Genom integrierten. Dieser Prozess ähnelt der Reversen Transkription der Viren und wird „Retroposition“ genannt. Eine Gruppe dieser Retropositions-Sequenzen sind die SINEs. Die Abkürzung steht für den englischen Ausdruck Short Interspersed Elements (= kurze, verstreute Elemente). Etwa 13 Prozent des menschlichen Erbguts www.biuz.de 2/2006 (36) | Biol. Unserer Zeit | 77 T R E F F P U N K T FO R SC H U N G | stammen von den SINES ab, weitere 29 Prozent leiten sich von Long Interspersed Elements (LINEs) und anderen Retropositions-Elementen ab. Man schätzt, dass etwa 15 Prozent aller menschlichen proteincodierenden Gene durch Retroposition von mRNA entstanden sein könnten. Man könnte erwarten, dass ein ähnlich großer Anteil von nicht proteincodierenden RNA-Genen (npcRNA) durch Retroposition entstanden ist. A B B . Wie aus Alu-Elementen neue proteincodierende Exons entstehen können: Exons sind als blaue, Spleiß-Erkennungsstellen sind als rote Kästchen dargestellt. Weiße Linien repräsentieren DNA-Bereiche, die keine Bauinformation für Proteine oder npcRNA enthalten; grüne Linien kennzeichnen RNA-Moleküle. Bereiche dieser RNA-Moleküle, die durch das Spleißen herausgeschnitten werden, sind gepunktet dargestellt. P = Promoter (Sequenz, die die Genaktivität steuert). Im Laufe der Evolution wird eine Alu-RNA in DNA (gelbes Kästchen) umgeschrieben und integriert sich dabei in das menschliche Genom. Dieser Vorgang wird Retroposition genannt. Das Alu-Element enthält mindestens zwei Sequenzen, die Spleißstellen ähneln, so dass die Spleißwerkzeuge der Zelle an diesen Stellen gelegentlich schneiden. Das translatierte Protein enthält somit eine zusätzliche Domäne, die von einem Stück eines Alu-Elements codiert wird. Auf diese Weise entsteht zusätzlich zum ursprünglichen Genprodukt eine neue mRNA-Form, die ein neues zusätzliches Exon enthält, das von der Alu-Sequenz stammt. 78 | Biol. Unserer Zeit | 2/2006 (36) www.biuz.de Retroposition – ein wichtiges Element der Evolution Die Retroposition ist äußerst wichtig für die Evolution, denn dieser Vorgang ermöglicht genetische Diversität. Vergleicht man das Erbgut der Maus und des Menschen miteinander, so findet man fast 25.000 proteincodierende Sequenzen, die in beiden Genomen vorkommen, aber nur 300 Gene, die entweder nur im Erbgut der Maus oder nur im Genom des Menschen existieren. Das ist erstaunlich, wenn man bedenkt, dass sich die evolutionären Wege von Mensch und Maus vor über 80 Millionen Jahren trennten. Was also ist der genetische Grund für die Unterschiede zwischen Mensch, Maus und anderen Säugetieren? Die Antwort liegt zum einen darin, dass sich die Aktivitäten von identischen oder ähnlichen Genen in Mensch und Maus in Bezug auf den Ort und den Zeitpunkt ihrer Expression unterscheiden. Ein anderer Grund ist, dass die Evolution zufällig und blind, aber kontinuierlich an der RNA- und Protein-Bauanleitung „herumbastelt“ und mal ein Stück einfügt, ein anderes Mal ein Stück herausnimmt, so dass Proteine mit teilweise neuen Domänen entstehen können. Diese beiden Vorgänge der Evolution werden nicht nur durch Mutationen (d.h. Nukleotidaustausch oder Einfügen/Verlust eines Teils der Gensequenz) verursacht, sondern sie werden auch maßgeblich durch die Retroposition beeinflusst, die neue Module in Gene oder in die Nähe von Genen bringt. Dabei stellen RNASequenzen, die eigenständig keine Bauinformation für Proteine enthalten und die deshalb häufig unzutreffend als “Junk-DNA” bezeichnet werden, ein Repertoire dar, aus dem neue Gen-Module rekrutiert werden können: Die Protein-Bauanleitung der Gene liegt meist gestückelt vor. Zwischen Bereichen mit wichtiger Information für die Herstellung eines Proteins (Exons) liegen immer wieder Abschnitte ohne Bauinformation, deren Funktion noch weitgehend un- © 2006 Wiley-VCH Verlag GmbH & Co. KGaA, Weinheim | bekannt ist (Introns). Zur Proteinherstellung wird zunächst eine durchgängige Kopie des gesamten Gens erstellt. Aus dieser Kopie werden dann alle Bereiche ohne Information herausgeschnitten. Dieser Mechanismus wird in der Fachsprache als „Spleißen“ bezeichnet. An den Nahtstellen von Exons und Introns gibt es SpleißErkennungsstellen, die markieren, welche Teile der mRNA herausgeschnitten werden sollen. Unsere Arbeitsgruppe vom Institut für Experimentelle Pathologie der Universität Münster hat 153 Exons im Genom untersucht, an deren Entstehung wahrscheinlich Alu-Elemente beteiligt waren. Alu-Elemente sind etwa 300 Basen lang, gehören zur Familie der SINEs und sind spezifisch für Primaten. Alu-Elemente stammen von einer npcRNA ab, die wiederum der 7SL-RNA ähnlich ist. Letztere ist in der Zelle für den Transport von Proteinen zuständig. Alu-Elemente enthalten mehr als ein Dutzend Sequenzen, die Spleißstellen so stark ähneln, dass sie unter Umständen das Spleißen einleiten können. So wurden zusätzliche codierende Domänen in die Proteinbauanleitung eingefügt. Unsere Arbeitsgruppe hat vier ausgewählte Beispiele zwischen Primatenarten (Halbaffen, Neuweltaffen, Altweltaffen und Menschenaffen) verglichen. Wir konnten auf diese Weise die evolutionären Schritte rekonstruieren, die bei den unterschiedlichen Spezies abgelaufen sind. Die Untersuchungen zeigen, dass die Entstehung neuer Exons mithilfe der Alu-Sequenzen ein wichtiger Mechanismus der Evolution ist. Der evolutive Vorteil liegt darin, dass das Spleißen zunächst ineffektiv ist. Deshalb wird zunächst hauptsächlich noch das ursprüngliche Genprodukt hergestellt, während die neue Form, die beispielsweise ein neues zusätzliches Exon enthält, nur einen kleinen Anteil des Genproduktes ausmacht (siehe Abbildung). So kann das neue Genprodukt „getestet“ werden, während das alte weiterhin eingesetzt wird. Bietet die neue Spleißvariante dem Organismus keinen Vorteil, geht sie nach Generationen wahrscheinlich verloren. Wenn aber die veränderte Genproduktvariante nützlich ist, dann setzt sie sich im weiteren Verlauf der Evolution durch, indem die ineffektive Spleiß-Erkennungsstelle in dem Alu-Element über Mutationen (Nukleotid-Austausche) in eine effektive Spleiß-Erkennungsstelle verwandelt wird. Oft sind beide Protein-Varianten für die Zelle nützlich. In diesem Fall werden beide Protein-Bauanleitungen hergestellt, eine Situation, die in der Fachsprache „Alternatives Spleißen“ genannt wird. Aber nicht nur neue Protein-Module können aus dieser Verfügungsmasse der Genome hergestellt werden, sondern es werden auch immer wieder ganze Gene erzeugt und getestet, die in nicht-proteincodierende RNAs übersetzt werden. Welche Funktionen haben die npcRNAs in der Zelle? In einem Explorativen Projekt, das Teil des Nationalen Genomforschungsnetzes ist, suchen wir mit experimentellen und bioinformatischen Verfahren nach potenziellen Genen, die für npcRNAs codieren. Das Ergebnis dieser Computeranalyse lässt darauf schließen, dass im SäugetierGenom eine enorm große Anzahl von RNA-Transkripten vorhanden ist, nämlich schätzungsweise über T R E F F P U N K T FO R SC H U N G 50.000. Mit Hilfe experimenteller Gen-Chip-Daten [4] lässt sich überprüfen, ob die Gene auch tatsächlich aktiv sind. Dabei zeigt sich, dass mindestens zehn Prozent der von uns vorhergesagten npcRNA-Gene mit verlässlichen Methoden detektierbar sind. Das bedeutet, dass es im Säugetier-Genom mehr Gene gibt, die für npcRNAs codieren, als man sich jemals vorstellen konnte. In weiteren Experimenten untersuchen wir, in welchen Geweben bestimmte npcRNA-Gene aktiv sind, in welchen Zellkompartimenten die npcRNA-Gene lokalisiert sind und ob die npcRNAs mit Proteinen wechselwirken. Mit diesen Daten können wir dann die Frage beantworten, wie viele der npcRNAs eine biologische Funktion innerhalb der Zelle haben. Einige Wissenschaftler glauben, dass viele der npcRNAs überhaupt keine Funktion haben. Aber ganz gleich, wieviele der RNAs funktionstüchtig oder Nebenprodukte der Transkription sind, schon jetzt ist klar, dass die Anzahl der funktionell aktiven npcRNAs verblüffend groß ist. Seit den 1990er Jahren wird immer deutlicher, dass das Funktionsspektrum der RNAs deutlich über ihre Rolle bei der Transkription und Translation hinausgeht. Insbesondere die sehr kurzen npcRNA-Moleküle, die so genannten siRNAs (21-25 Nukleotide) und microRNAs (21-23 Nukleotide), sind PROJ E K T E D E S N G F N : E X PLO R AT I V E PROJ E K T E Einen weiteren Artikel zum Thema „Junk-DNA“ finden Sie in BIUZ 3/2006, die Anfang Juni 2006 erscheint. | Ein wichtiges Strukturelement des Nationalen Genomforschungsnetzes (NGFN) sind die Explorativen Projekte. In einem dieser Projekte durchforsten NGFN-Wissenschaftler systematisch das Genom hochbetagter Menschen, um nach den „Langlebigkeits-Genen“ zu suchen. Die Ergebnisse dieses Projektes könnten zu neuen Ansätzen für die Behandlung altersbedingter Krankheiten führen. In einem anderen Explorativen Projekt werden Stammzellen aus dem menschlichen Knochenmark untersucht. Die NGFN-Forscher wollen herausfinden, welche Faktoren im genetischen Programm diejenigen Prozesse steuern, die bewirken, dass sich die Stammzellen nach einigen Zellteilungen zu spezialisierten Zellen, beispielsweise Knochen-, Nerven- oder Blutzellen entwickeln. Der nebenstehende Artikel gibt einen ausführlichen Einblick in ein weiteres Exploratives Projekt: Wissenschaftler um den Münsteraner Professor Jürgen Brosius wollen das Geheimnis lüften, welche Aufgabe die vielen tausend RNA-Transkripte haben, die nicht für Proteine codieren. Informationen zu den Forschungsarbeiten über npcRNAs am Institut für Experimentelle Pathologie, Universität Münster: http://zmbe2.uni-muenster.de/expath/frames.htm Informationen zum Nationalen Genomforschungsnetz unter www.ngfn.de © 2006 Wiley-VCH Verlag GmbH & Co. KGaA, Weinheim www.biuz.de 2/2006 (36) | Biol. Unserer Zeit | 79 T R E F F P U N K T FO R SC H U N G | für viele molekularbiologische Vorgänge in der Zelle enorm wichtig, vor allem bei Entwicklungsprozessen. Sie sind an der Regulation der Fettspeicherung, der Insulin-Sekretion, des programmierten Zelltodes, an der Herzentwicklung und vielen anderen Prozessen beteiligt. Auch bei der Entstehung genetischer Krankheiten spielen die npcRNAs eine Rolle, wie folgendes Beispiel zeigt: Das Fehlen eines bestimmten Bereiches von Chromosoms 15 verursacht das Prader-WilliSyndrom, eine Erkrankung des Nervensystems. Wenn man bei Mäusen durch gentechnische Methoden jedes einzelne in dieser Region liegende 80 | Biol. Unserer Zeit | 2/2006 (36) proteincodierende Gen ausschaltet, so kommt es trotzdem nicht zu der Krankheit. Also kann ausgeschlossen werden, dass proteincodierende Gene am Prader-Willi-Syndrom beteiligt sind. In dem betreffenden Bereich befindet sich allerdings auch eine Gruppe von Genen, die für gehirnspezifische npcRNAs codieren. Die laufenden Forschungsarbeiten konzentrieren sich deshalb auf diese npcRNAs. In den nächsten Jahren und Jahrzehnten warten noch zahlreiche Überraschungen auf uns, die die Vielseitigkeit der npcRNAs vorführen werden. Die RNA-Welt ist auch noch drei bis viereinhalb Milliarden www.biuz.de Jahre nach ihrer Entstehung sehr lebendig. [1] D. P. Bartel, P. J. Unrau, Trends in Cell Biology 1999, 9, M9–M13. [2] J. Brosius, Trends in Biochemical Sciences 2001, 26, 653–656. [3] J. Brosius, Journal of Structural and Functional Genomics 2003, 3, 1–17. [4] J. Cheng, P. Kapranov, J. Drenkow et al., Science 2005, 308(5725), 1149-54.. [5] C. Guerrier-Takada, K. Gardiner, T. Marsh, N. Pace, S. Altman, Cell 1983, 35, 849–857. [6] K. Kruger, P. J. Grabowski, A. J. Zaug, J. Sands, D. E. Gottschling, T. R. Cech, Cell 1982, 31, 147–157. [7] J. W. Szostak, D. P. Bartel, P. L. Luisi, Nature 2001, 409(Suppl.), 387–390. [8] C. R. Woese, RNA 2001, 7, 1055–1067. Jürgen Brosius, Münster © 2006 Wiley-VCH Verlag GmbH & Co. KGaA, Weinheim