Eukaryotische messenger-RNA
Werbung

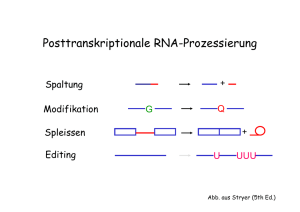
Eukaryotische messenger-RNA • Cap-Nukleotid am 5’-Ende • Polyadenylierung am 3’-Ende • u.U. nicht-codierende Bereiche (Introns) Abb. aus Stryer (5th Ed.) Spleißen von prä-mRNA Viele Protein-codierende Gene in Eukaryoten sind durch nicht-codierende Abschnitte (Introns) unterbrochen. Beim Spleißen werden die Introns entfernt und die codiernde Abschnitte (Exons) verknüpft. Transkription Addition vom Cap und Poly(A)-Schwanz Primär-Transkript (prä-mRNA) Spleißen Abb. aus Stryer (5th Ed.) Spleißen von prä-mRNA • Im Kern von eukaryotischen Zellen • Spleißosom erkennt die Introns und katalysiert die Spleißreaktion • Das Spleißosom besteht aus ca. 100 verschiedenen Proteinen und 5 snRNAs Abb. aus Stryer (5th Ed.) Das Spleißosom • Die snRNAs und die meisten Proteine bilden stabile RibonukleoproteinKomplexe, die snRNPs (small nuclear ribonucleoprotein particle) • Für jede Spleißreaktion muss das Spleißosom neu gebildet werden Abb. aus Stryer (5th Ed.) Das Spleißosom U4/U6.U5 snRNP U2 snRNP U2 snRNP Abb. aus Stryer (5th Ed.) Erkennung der 5’-SpleißStelle durch das U1 snRNP Abb. aus Stryer (5th Ed.) Das Spleißen von prä-mRNA erfolgt in zwei Schritten Abb. aus Stryer (5th Ed.) Alternatives Spleißen erhöht die Zahl möglicher Proteinprodukte Abb. aus Stryer (5th Ed.) Selbst-spleißende Introns Erstmals entdeckt für eine prä-rRNA in Tetrahymena Abb. aus Stryer (5th Ed.) Struktur einer selbst-spleißenden RNA Abb. aus Stryer (5th Ed.) Mechanismus des Spleißens in einer selbst-spleißenden RNA 1. Bindung eines Guanosin-Nukleosids Abb. aus Stryer (5th Ed.) Mechanismus des Spleißens in einer selbst-spleißenden RNA 2. Durch nukleophilen Angriff der 3’-OH des Guanosins wird die Phosphodiester-Bindung an der 5’-Spleißstelle gespalten Abb. aus Stryer (5th Ed.) Mechanismus des Spleißens in einer selbst-spleißenden RNA 3. Die nun freie 3’-OH Gruppe des 5’-Exons kann die Phosphodiester-Bindung an der 3’-Spleißstelle nukleophil angreifen, spalten und somit die beiden Exons verbinden Abb. aus Stryer (5th Ed.) Selbst-spleißende RNAs • Introns der Gruppe I im Zellkern, sowie in den Mitochondrien und Chloroplasten verschiedener Eukaryoten (aber nicht in Wirbeltieren) • Introns der Gruppe II in den Mitochondrien und Chloroplasten von Pilzen und Pflanzen Abb. aus Stryer (5th Ed.) Vergleich der Spleiß-Mechanismen Abb. aus Stryer (5th Ed.) RNA - Welt • RNA kann komplizierte 3D-Strukturen ausbilden (RNA-Faltung) und spezifisch kleine Moleküle binden • RNA besitzt katalytische Aktivität → RNAs waren die ursprünglichen Katalysatoren in der prä-zellulären Zeit Abb. aus Stryer (5th Ed.) Translation (Protein-Synthese) Basen-Sequenz der mRNA AS1 AS2 AS3 AS4 Aminosäure-Sequenz des Proteins Abb. aus Stryer (5th Ed.) Genetische Code • Basen-Triplett codiert für eine Aminosäure oder Stop • 64 Codons 60 für Aminosäuren 3 für Stop 1 Methionin-Codon ist immer das Start-Codon Abb. aus Stryer (5th Ed.) Abb. aus Stryer (5th Ed.) Für die Protein-Biosynthese werden die Aminosäuren an tRNAs gebunden Die mRNA Codons werden durch komplementäre tRNA Anticodons erkannt Abb. aus Stryer (5th Ed.) Manche tRNAs erkennen mehr als ein Codon (Wobble-Basenpaarung) Abb. aus Stryer (5th Ed.) Bei der Protein-Biosynthese ist die wachsende Polypeptidkette immer an eine tRNA gebunden Abb. aus Stryer (5th Ed.) Struktur der tRNAs Abb. aus Stryer (5th Ed.) Abb. aus Stryer (5th Ed.) Aminoacyl-tRNA Synthetasen aktivieren Aminosäuren durch Bindung an tRNA Aminosäure + tRNA + ATP Aminoacyl-Adenylat + tRNA + PP Aminoacyl-tRNA + AMP + PP Abb. aus Stryer (5th Ed.) Schritte der tRNAAminoacylierung Abb. aus Stryer (5th Ed.) Es gibt 20 verschiedene Aminoacyl-tRNA Synthetasen GlutaminyltRNA-Synthetase Abb. aus Stryer (5th Ed.) Die meisten Aminoacyl-tRNA Synthetasen haben einen Korrekturlese-Mechanismus (proofreading) Abb. aus Stryer (5th Ed.) Translation und Polypeptid-Synthese finden am Ribosom statt. Mehrere Ribosomen Können gleichzeitig eine mRNA translatieren: POLYRIBOSOM bzw. POLYSOM Abb. aus Stryer (5th Ed.) Ribosomen bestehen aus einer großen und einer kleinen Untereinheit Abb. aus Stryer Figure (5th4-32 Ed.) Die Übersetzung des genetischen Codes Figure 4-20 Abb. aus Stryer (5th Ed.) Die drei Phasen der Translation • Initiation Erkennung des Start-Codons durch den Initiationskomplex • Elongation pro Zyklus wird die wachsende Polypeptidkette um eine Aminosäure verlängert • Termination Erkennung des Stop-Codons, Dissoziation des Komplexes Abb. aus Stryer (5th Ed.) Wo ist der Startpunkt für die Translation auf der mRNA ? Abb. aus Stryer (5th Ed.) In Prokaryoten befindet sich vor dem Start-Codon eine konservierte RibosomenBindungstelle (Shine-Dalgarno Sequenz) Abb. aus Stryer (5th Ed.) In Prokaryoten ist die Start-tRNA mit einem modifizierten Methionin beladen N-FormylMethionin Abb. aus Stryer (5th Ed.) Bei der INITIATION bindet die kleine RibosomenUntereinheit mit Hilfe von drei Initiationsfaktoren und der fMet-tRNA an die mRNA. Anschliessend bindet die große Untereinheit an den Initiationskomplex und bildet das aktive Ribosom. Abb. aus Stryer (5th Ed.) Beim ersten Schritt der ELONGATION befindet sich die tRNA mit der ersten Aminosäure bzw. die tRNA mit der Polypeptidkette in der P-Stelle. Die nächste Aminoacyl-tRNA bindet mit Hilfe von Elongationsfaktor-Tu (EF-Tu) in der A-Stelle, wobei ein GTP verbraucht wird. EF-Tu GTP Es gibt 3 tRNA-Bindungstellen: P = Peptidyl-Stelle, A = Akzeptor-Stelle, E = Ausgang-Stelle Abb. aus Stryer (5th Ed.) Beim zweiten Schritt der ELONGATION wird die Peptidbindung gebildet, wobei die Aminosäure von der tRNA in der P-Stelle auf die Aminoacyl-tRNA in der A-Stelle übertragen wird. E P A P = Peptidyl-Stelle, A = Akzeptor-Stelle, E = Ausgang-Stelle Abb. aus Stryer (5th Ed.) Beim dritten Schritt der ELONGATION, der sog. Translokation, wandert das Ribosom ein Codon auf der mRNA weiter, wobei die tRNA von der P-Stelle in Die E-Stelle, und die Aminoacyl-tRNA Von der A-Stelle in die P-Stelle Verschoben wird. Die Translokation erfordet Energie, die der Elongationsfaktor G durch Spaltung von GTP bereit stellt. Abb. aus Stryer (5th Ed.) Im letzten Schritt der ELONGATION verläßt die tRNA die E-Stelle und das Ribosom kann die nächste Aminoacyl-tRNA aufnehmen. Abb. aus Stryer (5th Ed.) Pro ELONGATION-Zyklus werden 2 GTP zu GDP hydrolisiert EF-Tu GTP EF-Tu GDP Abb. aus Stryer (5th Ed.) Die EF-G Struktur ähnelt dem EF-Tu – tRNA Komplex Abb. aus Stryer (5th Ed.) Mechanismus der Translokation Abb. aus Stryer (5th Ed.) Termination Es gibt keine komplementäre tRNAs zu den Stop Codons (UAA, UGA, UAG). Erreicht das Ribosom ein Stop Codon binden mehrere Release Faktoren (RF), das Ribosom dissoziert und das neu-synthetisierte Protein wird nach Abspaltung von der tRNA freigesetzt. Bei der Termination wird ein GTP zu GDP hydrolisiert. Abb. aus Stryer (5th Ed.)