Kontrolle der Genexpression auf mRNA
Werbung

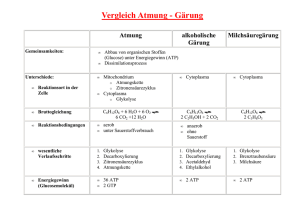
Kontrolle der Genexpression auf mRNA-Ebene Abb. aus Stryer (5th Ed.) RNA interference (RNAi) • siRNA • (small interfering RNA) • miRNA (micro RNA) Abb. aus Stryer (5th Ed.) Transcriptional silencing Inhibition of translation mRNA degradation Abb. aus Stryer (5th Ed.) Antibiotika sind oft Inhibitoren der Genexpression • Inhibitoren der Transkription Rifampicin, Actinomycin α-Amanitin • Inhibitoren der Translation Puromycin, Streptomycin, Tetracycline, Chloramphenicol Diphterie-Toxin Abb. aus Stryer (5th Ed.) Rifamycin & Rifampicin inhibieren prokaryotische, aber nicht eukaryotische RNA-Polymerasen Rifampicin ist ein halbsynthetisches Derivat von Rifamycin B, das von Streptomyces mediterranei produziert wird. Abb. aus Stryer (5th Ed.) Actinomycin D • aus Streptomyces antibioticus • inhibiert DNA- und RNA-Polymerasen • Interkaliert zwischen Basenpaare der dsDNA Abb. aus Stryer (5th Ed.) α-Amanitin (Amatoxine) • aus Amanita phalloides (Grüner Knollenblätterpilz) • Inhibiert v.a. eukaryotische RNA-Polymerase II u. III, aber nicht I, und nicht prokaryot. RNA-Polymerasen Abb. aus Stryer (5th Ed.) Puromycin (inhibiert die Translation) Bindet ohne EF-Tu die A-Stelle im Ribosom und es wird ein Peptidylpuromycin gebildet. Eine weiter Transpeptidierung kann nicht stattfinden. Abb. aus Stryer (5th Ed.) Chloramphenicol Bindet an die grosse Untereinheit und inhibiert die Peptidyltransferase Aktivität von prokaryotischen Ribosomen Abb. aus Stryer (5th Ed.) Tetracycline Binden an die kleine Untereinheit prokaryotischer Ribosomen und verhindert die Bindung von AminoacyltRNAs. Abb. aus Stryer (5th Ed.) Streptomycin Streptomycin und andere Aminoglycoside inhibieren die Initiation (bei hoher Konzentration) und verursachen bereits bei niedriger Konzentration Fehleinbau Abb. aus Stryer (5th Ed.) Diphterie-Toxin (inhibiert die Translation) Diptherie-Toxin ist ein Protein aus Corynebacterium diphteriae, das in der infizierten Zelle in zwei Fragmente gespalten wird. Ein Fragment ist ein Enzym, das ADP-Ribose auf ein modifiziertes Histidin im EF-2 überträgt und dadurch EF-2 inaktiviert. Abb. aus Stryer (5th Ed.) Bioenergetik & Stoffwechsel Thermodynamik • Erster Hauptsatz (Energie bleibt erhalten) • Zweiter Hauptsatz (Entropie nimmt zu) Exergone / Endergone Reaktionen Freie Enthalpie, Enthalpie und Entropie ΔG = ΔH – T·ΔS ‹0 ›0 (exergone Reaktion) (endergone Reaktion) ΔG ist die Änderung der freien Enthalpie bis zum Erreichen des Gleichgewichts, wenn die Konzentrationen der Reaktanten und Produkte zu Beginn der Reaktion gleich 1 mol/l sind (bei Gasen: 101,3 kPa) Kriterium für die Spontanietät einer chemischen Reaktion Biochemische Standardzustand ΔGº Standardzustand (T=298K, P=101.3kPA, Konzentrationen = 1 mol/l, pH=0) ΔG’º Biochemische Standardzustand (pH 7.0, [H+]=10-7 mol/l ) Spontanietät einer Reaktion ΔH ΔS ΔG = ΔH - TΔS - + Die Reaktion ist sowohl seitens der Enthalpie (exotherm) als auch seitens der Entropie begünstigt. Sie läuft bei allen Temperaturen spontan (exergon) ab. - - Seitens der Enthalpie ist die Reaktion begünstigt, die Entropie wirkt aber dem entgegen. Sie läuft nur bei Temperaturen niedriger als T = ΔH/ΔS spontan ab. + + Die Reaktion ist seitens der Enthalpie gehindert (endotherm), aber entropische beguenstigt. Sie läuft nur bei Temperaturen oberhalb T = ΔH/ΔS spontan ab. + - Die Reaktion ist sowohl seitens der Enthalpie als auch Entropie gehindert (endergon). Sie läuft bei keiner Temperatur spontan ab. Gleichgewichtskonstante und ΔG Die Freie Enthalpie ΔG einer chemischen Reaktion hängt von den Konzentrationen ihrer Reaktanden und Produkte ab: a·A + b·B ΔG R ist die Gaskonstante (8,315 J mol-1 K-1) c·C + d·D = ΔG’º + R·T·ln ΔG’º [C]c [D]d [A]a [B]b = - R·T·ln K’eq tatsächlichen Konzentrationen im Gleichgewicht Beziehung zwischen K‘eq und ΔG’º S P ΔG’º = - R·T·ln K’eq [P] = - R·T·ln [S] K‘eq ΔG’º (kJ / mol) 10-6 34.2 10-4 22.8 10-2 11.4 10-1 5.7 1 0.0 101 -5.7 102 -11.4 103 -17.1 Änderungen der freien Standardenthalpie sind additiv ΔG Glucose + Phosphat Glucose-6-Phosphat + H20 (kJ·mol-1) 13.8 ATP + H20 ADP + Phosphat -30.5 _____________________________________________________ Glucose + ATP Glucose-6-Phosphat + ADP -16.7 Gleichgewichtskonstanten gekoppelter Reaktionen Keq(1) = Keq(2) = Keq(3) = [Glucose-6-P] [Glucose][P] [ADP] [P] [ATP] = 3.9 · 10-3 mol-1 l = 2.0 · 105 mol l-1 [Glucose-6-P] [ADP] [P] [Glucose] [P] [ ATP] = 7.8 · 102 mol-1 l Der Stoffwechsel (Metabolismus) Übertragung und Speicherung von Energie Der Metabolismus besteht aus vielen gekoppelten Reaktionen Katabolismus Anabolismus ATP – ADP Zyklus Biosynthesen Bewegung Aktiver Transport Signalverstärkung Oxidation von Brennstoffmolekülen oder Photosynthese Die Freie Enthalpie der ATP-Hydrolyse in Zellen ΔG = ΔG’º + [ADP] [P] R·T·ln [ATP] Beispiel: in Erythrocyten beträgt [ATP] = 2,25 mmol l-1 [ADP] = 0,25 mmol l-1 [P] = 1,65 mmol l-1 ΔG = -30.5 J mol-1 + 8.315 J mol-1 K-1 ·298 K · ln ΔG (2.5 10-4)(1.65 10-3) 2.25 10-3 = -51800 J mol-1 =-51.8 kJ mol-1 Mg2+ Adenosintriphosphat (ATP) Adenosindiphosphat (ADP) Adenosinmonophosphat (AMP) Biologische Redoxreaktionen Messung des StandardReduktionspotentials - X + e- → X + H + e- → ½ H2 Negatives ReduktionsPotential: X besitzt eine geringere Elektronenaffinität als H2 Bedeutung der Standard-Reduktionspotentiale Oxidierte Form (Oxidationsmittel) Reduzierte Form (Reduktionsmittel) Ein starkes ReduktionsMittel (zB NADH) gibt leicht Elektronen ab und besitzt daher ein negatives Redoxpotental. Sauerstoff (O2) ist ein starkes Oxidationsmittel, d.h. es nimmt bereitwillig Elektronen auf und besitzt daher ein positves Redoxpotential. Änderung der freien Enthalpie: ΔG’º = -nFΔE’º Nicotinamid-adenin-dinucleotid (NAD+) reaktive Stelle Bei der Oxidation eines Substrates nimmt das NAD+ zwei Elektronen und ein Proton auf (= ein Hydridion) R = H : NAD+ R = Phosphat: NADP+ Flavin-adenin-dinucleotid (FAD) Reaktive Stellen Glucose Stoffwechsel Glykolyse, Milchsäuregärung und alkoholische Gärung Gärung Glykolyse vollständige Oxidation Glykolyse 1: Phosphorylierung von Glucose Mg2+ Glykolyse 2: Isomerisierung von Glucose-6-phosphat Glucosephosphat-Isomerase Glucose-6-phosphat Glucose-6-phosphat Fructose-6-phosphat Fructose-6-phosphat Glykolyse 3: Phosporylierung von Fructose-6-phosphat PFK ist ein allosterisches Enzym, das die Geschwindigkeit der Glykolyse bestimmt. Glykolyse 4: Spaltung von Fructose-1,6-bisphosphat - Glycerinaldehyd3-phosphat (GAP) Glykolyse 5: Isomerisierung von Dihydroxyacetonphosphat TriosephosphatIsomerase - Glycerinaldehyd3-phosphat (GAP) Struktur und Funktion der Triosephosphat-Isomerase (αβ)8-Barrel Katalytische Mechanismus der Triosephosphat-Isomerase (1) Katalytische Mechanismus der TriosephosphatIsomerase (2) Katalytische Mechanismus der Triosephosphat-Isomerase (3) Zusammenfassung “Vorbereitende Stufe” der Glykolyse