MOLEKULARE GRUNDLAGEN DER VERERBUNG
Werbung

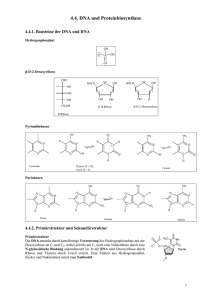
MOLEKULARE GRUNDLAGEN DER VERERBUNG 1. Bakterien und Viren als genetische Forschungsobjekte Bakterium: Plasmide (kleine Zusatzchromosömchen) ringförmige DNA, das s. g. „Bakteriumchromosom“ Versuch von Griffith 1928 Pneumokokken: Erreger der Lungenentzündung S-Stamm: smooth, Kapsel aus Schleim vorhanden, virulent R-Stamm: rough, Kapsel aus Schleim nicht vorhanden, nicht virulent Abkochen zum Abtöten der Bakterien R-Stamm: keine schützende Schleimhülle, daher nicht virulent keine Erkrankung R-Stamm: lebendig Aber ungefährlich !! Versuch von Avery 1944 (in vitro) S-Stamm Pathogen R-Stamm nicht pathogen Pneumokokken – Transformationsexperiment Seite 58 © Florian Zeller 07/08 Die DNA des S-Stamms muss in den lebenden R-Stamm übergegangen sein, dort eingebaut & realisiert worden sein. Transformation = Übertragung von Erbanlagen durch reine blanke DNA Bei solchen Experimenten arbeitet man oft mit s.g. Mangelmutanten; das sind Mutanten, die die Fähigkeit zur Herstellung oder Verwertung eines bestimmten Stoffes verloren haben. Sie sind auxotroph in Bezug auf diesen Stoff. Gewinnung von Mangelmutanten Seite 59 © Florian Zeller 07/08 Schema der Stempeltechnik zur Suche nach Mangelmutanten ohne Lysin lys- ohne Leucin leu- ohne Valin val- Transformation bei Bacillus subtilis Die Zellen einer Wildtyp-Kultur werden künstliche lysiert. Mit Phenol werden die Proteine denaturiert. Bakterienlysat Das Lysat wird mit Chloroform geschüttelt. Zentrifugation wässrige Phase mit DNA Die DNA wird aus der wässrigen Phase gewonnen !!! Chloroform-Phase mit Protein Bacillus subtilis Bacillus subtilis auxotropher Stamm im Vollmedium auxotropher Stamm im Vollmedium + DNS aus Wildtypzellen Keine Kolonien auf einer MinimalmediumAgarplatte Koloniebildung auf einer MinimalmediumAgarplatte Seite 60 © Florian Zeller 07/08 Ablauf der Transformation Voraussetzung für die Aufnahme reiner DNA einer Spenderzelle ist ein bestimmter Stoffwechselzustand der Empfängerzelle, d.h. sie ist kompetent (empfangsbereit). In diesem Zustand werden Rezeptoren auf der Zellwand gebildet oder aktiviert. Die Spender-DNA muss doppelsträngig vorliegen und eine bestimmte Molekülgröße aufweisen, damit die Aufnahme durch Endozytose (winzige fingerförmige Einstülpungen der Zellmembran schnüren kleine mit der aufzunehmenden Substanz gefüllte Bläschen ab, die ins Zellinnere wandern) in die Empfängerzelle möglich ist. 1. Endozytose Ein Einzelstrang der Spender-DNA wird mit dem homologen Einzelstrangabschnitt der Empfänger-DNA verbunden. Bei der anschließenden Integration verdrängt dieser Einzelstrang der Spender-DNA den entsprechenden Abschnitt der Empfänger-DNA, der dann ausgeschnitten und abgebaut wird. 2. Integration Bei der folgenden Replikation wird der eingebaute Spender-DNA-Einzelstrang verdoppelt und bildet eine durchgehende Doppelhelix, während der nun ungepaarte Empfänger-DNA-Einzelstrang abgebaut wird. 3. Replikation Vorteile von Bakterien für genetische Untersuchungen: - Einfach strukturiert, 1 ringförmiges Chromosom (DNA) + Plasmide Mutation erscheint meist sofort im Phänotyp - Geringer Aufwand für Kultivierung - Rasche Vermehrung - Viele nachkommen statistisch haltbare Ergebnisse - Keine ethisch-moralischen Bedenken Seite 61 © Florian Zeller 07/08 Vergleich von VIREN und ZELLEN VIREN Nucleinsäuren DNA oder RNA Fähigkeit zur Mutation ZELLEN DNA RNA vorhanden vorhanden Stoffwechsel ---- Vermehrung lässt sich vermehren, nur in Wirtszellen möglich vorhanden vermehrt sich selbst mitotisch oder meiotisch; Spaltung bei Bakterien Vorhanden Begrenzende Membran fehlt Genübertragung und Genaustausch (= Rekombination) bei Bakterien Bei höheren Lebewesen erfolgt die Rekombination durch Meiose und Befruchtung. Bei Bakterien gibt es hierfür andere Vorgänge, man nennt sie parasexuell. 1. GENAUSTAUSCH = REKOMBINATION Versuch: 1946 mit 2 Doppelmutanten von E. coli: 1. A-B- , d.h. Synthese von Aminosäuren A und B nicht möglich 2. C-D- , d.h. Synthese von Aminosäuren C und D nicht möglich Minimalmedium ohne A,B,C,D Rekombination hat stattgefunden !!! „Geheilte“ Mangelmutanten (Rekombinanten) erkennt man im Experiment daran, dass sie Auf Minimalagar wieder Kolonien bilden können!! Problem: Wie gelangen die Gene von einer Bakterienzelle in die andere, d.h. wie erfolgt der Gentransfer, die Genübertragung? 2. GENTRANSFER = GENÜBERTRAGUNG a) Konjugation Bakterien, die auf einem Plasmid den sog. F+-Faktor (= Fertilitätsfaktor) haben, sind in der Lage, dünne Röhren auszubilden und damit Kontakt zu F--Zellen aufzunehmen. Es kann zur Ausbildung von Plasmabrücken kommen, über diese können Gene transferiert werden! Seite 62 © Florian Zeller 07/08 Transferiert werden können Kopien eines ganzen Plasmids (z.B. mit F+-Faktor) oder Kopien von Stücken der Spender-DNS !!! Der dauerhafte Einbau von Spender-Genen in die Empfänger-DNS erfolgt über Crossing over Prozesse und kann z.B. so erfolgen: Empfängerbakterium: a-b-c- (Genotyp) Dreifachmutante!! Einbaumöglichkeiten (Rekombinationsmöglichkeiten) Rekombinante: A+B+c- Aufgabe: 1. Kann auch der „Wildtyp“ wieder entstehen? Ja, wenn alle 3 eingebaut werden A+B+C+ ; aber zwar Rekombinante 2. Gibt es weitere Rekombinanten? Nennen Sie alle möglichen neuen Genotypen!! A+b-c- ; a-B+c- , a-b-C+ , A+b-C+ , a-B+C+ Konjugation = Übertragung von genetischem Material mit Ausbildung einer Plasmabrücke b) Transduktion Transduktion = Übertragung von genetischem Material durch Vieren Vieren werden als „Gentaxis“ oder „Genfähren“ benutzt! Sie nehmen z.B. bakterielle Gene wie Passagiere auf und laden sie irgendwo wieder ab, dies kann auf zwei unterschiedliche Weise geschehen! 1. Allgemeine Transduktion Lytischer Vermehrungszyklus Seite 63 © Florian Zeller 07/08 Wenn in einem phagenbefallenen Wildtypbakterium die vermehrte Phagen-DNS in die Kopfhüllen verpackt wird, kann versehentlich ein Stück Bakterien-DNS in den Kopf gelangen. Injiziert ein solcher Phage die DNS in eine Mangelmutante, kann er zufällig das Gen A mitbringen, das bei der Mangelmutante zu a mutiert ist. Durch einen Paarungs- und Rekombinationsvorgang (crossing over) kann das defekte Gen a gegen das intakte Gen A ausgetauscht werden. Gen a (defekt) Gen a (defekt) ausgebaut Mangelmutante ist genetisch geheilt! 2. Spezielle Transduktion (Modell für gezielte Gentransplantation) Gefesselter Prophage nimmt bei seinem Austritt aus dem Bakterienchromosom benachbarte Gene mit und wird vermehrt. In alle Phagenköpfe, die im Bakterium hergestellt werden, gelangt eine Kopie desselben bakteriellen Gens! Befallen diese transduzierten Phagen einen bezüglich dieses Gens mutierten Bakterienstamm, erfolgt mit hoher Erfolgsrate dessen Heilung durch Austausch des defekten Gens. T4 – Bakteriophage HIV-Virus Seite 64 © Florian Zeller 07/08 Bakterien und Viren als Forschungsobjekte 2. Vorkommen, Struktur und Replikation von Nucleinsäuren Vorkommen: Struktur: 1. DNA: chromosomal Bestandteil der Chromosomen extrachromosomal Platiden und Mitochondiren RNA: in und außerhalb des Zellkerns Cytoplasma Ribosomen Mitochondrien und Plastiden Wenn man DNA durch Kochen mit Säure hydrolisiert, so kann man im Hydrolysat stets folgende Bestandteile nachweisen: BESTANDTEILE: Phosphorsäure P Zucker (Desoxyribose, Ribose) Z Organische Basen B Purinbasen Adenin A Guanin G Basenpaarung ------------- Pyrimidinbasen T Thymin (U = Uracil bei RNA) C Cytosin Seite 65 © Florian Zeller 07/08 2. MONOMERE (= Bausteine) der Nucleinsäuren Nucleotid: 3. POLYNUCLEOTID = Nucleinsäuren 5‘-Ende einsträngig (bezogen auf das Zuckermolekül) Reihenfolge der Basen im Molekül Der Faden hat einen Richtungssinn (Polarität) !! = Basensequenz 3‘-Ende doppelsträngig 2 Stränge, paralleler und antiparalleler Polynucleotidstrang mit komplementärer Basenpaarung Komplementäre Polynucleotidstränge! zwischen den Basen: Wasserstoffbrücken! aus 1 – 3 4. WATSON-CRICK-STRUKTURMODELL DER DNA Strickleiterprinzip: Basenpaare = Sprossen der Leiter Zucker-Phosphatketten = Holme der Leiter Komplementarität der Stränge ! Raumstruktur: (= Basen ergänzen sich gegenseitig) Die beiden Stränge sind um eine gemeinsame (gedachte) Achse gewunden und bilden so eine sog. Doppelhelix (Doppelschraube). Nach je 10 Basenpaaren ist eine vollständige Schraubenwindung durchlaufen. Seite 66 © Florian Zeller 07/08 Tertiärstruktur der DNA Doppelhelix Raumstruktur der DNA = WATSON-CRICK-Modell Kurzschreibweisen Nukleinsäuren 1. (Nicht für die Prüfungen besonders relevant) Bestandteile a) Phosphorsäure b) Zucker: c) Desoxyribose Ribose Organ Basen Seite 67 © Florian Zeller 07/08 2. Monomere der Nukleinsäuren a) Bildung eines Nucleosids: 𝜶-Thymidin (Nukleosid) Thymin N-glykosidische Bindung b) Bildung eines Nucleotids: Esterbindung 𝛼-Thymidin-5‘-monophosphat = 𝜶TMP 3. Polynukleotide = Nukleinsäuren Primärstruktur der Desoxiribonukleinsäure (DNS = DNA) einsträngig: Richtungssinn des Fadens (Polarität) Sekundärstruktur der DANN: doppelsträngig Anordnung der beiden Polynukleotidfäden „antiparallel“ 5‘ Ende 3‘ Ende Guanin Adenin Cytosin 3‘ Ende Cytosin Thymin Guanin 5‘ Ende Seite 68 © Florian Zeller 07/08 4. Wasserstoffbrückenbindung, Basenpaarung „Komplementäre“ Polynukleotidstränge Identische Verdoppelung der DNS Denkmöglichkeiten: Wenn von Zellteilung zu Zellteilung keine genetische Information verlorengeht, muss vor jeder Teilung eine identische Verdoppelung der Erbsubstanz erfolgen. Das Strukturmodell der DNS von Watson und Crick bietet hierfür durch das Prinzip der komplementären Basenpaarung eine verblüffend einfache Modellvorstellung an. Der Doppelstrang öffnet sich wie ein Reißverschluss. An die frei werdenden Basen lagern sich in jedem Strang einzelne Nucleotide mit den jeweils komplementären Basen an, die miteinander verknüpft werden. Die angedockten Nucleotide werden über Phosphor-Esterbindungen miteinander verknüpft. Dadurch entstehen zwei neue Doppelstränge von DNS mit genau derselben Aufeinanderfolge von Basenpaaren. Nach dieser Modellvorstellung besteht jeder Doppelstrang zur Hälfte aus altem, zur anderen Hälfte aus neuem Material. Den experimentellen Nachweis für die semikonservative Replikation der DNA lieferten MESELSON und STAHL 1958. Einfachste Schema der identischen VerDoppelung der DNS Seite 69 © Florian Zeller 07/08 Markierungsexperiment von Meselson und Stahl 1958 15 N = schwerer Stickstoff Eichungsschritt 1. Replikationszyklus auf Nährboden mit 14N 1 halbschwere Bande 2. Replikationszyklus auf Nährboden mit 14N Ausschluss des Konservativen Mechanismus 1 halbschwere und 1 leichte Bande Ausschluss des Dispersiven Mechanismus Kontrollversuch Nach 3 Replikationszyklen auf Nährboden mit 14N SEMIKONSERVATIVER MECHANISMUS !!! Diese sogenannte semikonservative Art der DNS-Verdoppelung oder DNSReplikation konnte durch Isotopen-Markierungsversuche bestätigt werden. Escherichia coli-Bakterien werden während mehrerer Replikations- und Teilungszyklen in einem Nährmedium gehalten, das in seinen Stickstoffverbindungen das „schwere“ Isotop 15N enthält. Dabei wird 15N über die Purinund Pyrimidinbasen schließlich in beide Stränge der DNS eingebaut. In einer sogenannten analytischen Ultrazentrifuge ist es möglich, 15N-haltige, „schwere“ DNS von 14N-haltiger, „leichter“ DNS, sowie „halbschwerer“ 15 14 N/ N-haltiger DNS als Banden optisch zu unterscheiden. Bakterien mit 15N-haltiger DNS werden in 14N-haltiges Medium überführt und dort für die Zeit eines Replikationszyklus belassen. Isoliert man dann die DNS aus diesen Bakterien und untersucht sie in der Ultrazentrifuge, so erweist sie sich als „halbschwer“. Untersucht man die Bakterien-DNS nach zwei Replikationen in normalem Medium, so findet man leichte und halbschwere DNS im Verhältnis 1:1, in Übereinstimmung mit der Modellvorstellung der semikonservativen Art der Replikation. Seite 70 © Florian Zeller 07/08 Seite 71 © Florian Zeller 07/08 Ablauf der DNS-Verdoppelung DNS-Doppelhelix wird enzymatisch entdrillt und geöffnet. Komplementäre Nucleotide lagern sich an (liegen als energiereiche Nucleosidtriphosphate vor: PPP-Z-B, Abspaltung von PP liefert Energie nur Nucleotidverknüpfung) DNS-Polymerase kann Nucleotide nur in 3‘ 5‘-Richtung des Mutterstranges verknüpfen, deshalb erfolgt die Ergänzung zum Doppelstrang an beiden Ästen der Replikationsgabel unterschiedlich: an einem Strang kontinuierlich von der Gabelungsstelle weg, am anderen Ast diskontinuierlich (nach und nach) in kleinen Stücken (Okazaki-Fragmente), die dann von DNSLigase verbunden werden. DNS-Reparatur-Polymerase korrigiert fehlerhafte Basenpaarungen. DNS-Polymerase kann DNS-Synthese nur fortsetzen, nicht beginnen; ein Primer (ein kurzes RNS-Stück), von Primase aufgebaut, startete den Vorgang. DNS-Polymerase braucht am Leitstrang nur 1 Primer, für die Synthese jedes Okazaki-Fragmentes einen eigenen Primer. (Okazaki-Fragmente bei Eukaryonten: 100 – 200 Nucleotide, bei Prokaryonten: 1000 – 2000 Nucleotide). Die Replikation der Bakterien-DNS erfolgt von einem Startpunkt aus. Zur Anlagerung der Nucleotide ist Entspiralisierung notwendig. Die Bakterienzelle besitzt 3 × 105 Windungen. Verdoppelung ca. alle 40 Minuten 3 × 105 : 40 = 7500 Umdrehungen pro Minute = mittelschwere Zentrifuge! Problem weitgehend ungeklärt, müsste eigentlich für die Zelle verheerende Folgen haben. Heute weiß man, dass DNS-Topoisomerasen DNS entwinden können, ohne die Doppelhelix dabei zu drehen: die „Holme“ der verdrillen „Leiter“ werden an vielen Stellen durchtrennt, entflochten und anschließend wieder repariert. Die Verdoppelung der DNS erfolgt bei Eukaryonten an mehreren Startpunkten gleichzeitig. Startstelle = Replikationsursprung (spezifische Nucleotidsequenz), wird von bestimmtem Enzym erkannt und leitet Replikation ein. Seite 72 © Florian Zeller 07/08 ATP – Adenosintriphosphat Adenin + Desoxyribose + P P P GTP – Guanosintriphosphat CTP – Cytidintriphosphat TTP – Thymidintriphosphat Vergleich DNA und RNA DNA RNA Aufbau Zucker: Desoxiribose Phosphat Basen: A , T , C , G Zucker: Ribose Phosphat Basen: A , U , C , G Form Doppelstrang Helix (spiralig gewunden) Einzelstrang nicht gewunden Arten Kern – DNA Mitochondriale DNA Plastiden – DNA messenger-RNA: langgestreckt ribosomale RNA: Kleeblattstruktur transfer-RNA: Kleeblattstruktur Kern Mitochondrien Plastiden (Chloro-, Chromo-, Leukoplasten) Kern Plasma Mitochondrien Plastiden Ribosomen Vorkommen Seite 73 © Florian Zeller 07/08 Eiweißverbindungen (Proteine und Peptide) Bedeutung: Baustoffe und Wirkstoffe 1. Strukturproteine: Substanzen, die den Körper aufbauen (Faserprotein Kollagen, Muskelproteine Aktin und Myosin). 2. Enzyme: Biokatalysatoren, ermöglichen chemische Reaktionen im Körper 3. Membranproteine: Diese Proteine sorgen für den selektiven Stofftransport durch die Biomembran, sie dienen der Erkennung von zellen und sind als Rezeptormoleküle in Membranen von Nervenzellen an der Informationsübermittlung beteiligt. 4. Transportproteine: Hämoglobin transportiert Sauerstoff. 5. Immunproteine: Antikörper binden Krankheitserreger. 6. Regulatorproteine: Hormone sind an der Regulation von Stoffwechselreaktionen beteiligt. Im menschlichen Körper kommen ca. 50.000 verschiedene Proteine vor. (+ Immunsystem Millionen) Aufbau: Die Bausteine der Proteine und Peptide sind die Aminosäuren: H Aminogruppe H2 N C COOH Säuregruppe (= Carboxylgruppe) R In Proteinen können 20 verschiedene (= proteinogene) Aminosäuren vorkommen, die sich in den Resten R unterscheiden. Es gibt unpolare lipophile Reste und polare hydrophile Reste. H H2N C H COOH H2N C COOH H2C – OH CH3 Alanin (unpolar) Serin (polar) DIPEPTID Aminosäure 1 Aminosäure 2 Von 1 bis 99 Aminosäuren spricht man von Peptide, ab 100 Aminosäuren spricht man von Proteinen (Primärstruktur). Seite 74 © Florian Zeller 07/08 Verknüpfung von Aminosäuren durch Peptidbdindungen Prinzip: Aminosäure 1 Dipeptid Aminosäure 2 Aminosäure 3 DIPEPTID TRIPEPTID Seite 75 © Florian Zeller 07/08 Allgemeine Schreibweise: H2N – AS1 – AS2 – AS3 – COOH Aminoende Carboxylende R1 , R2 , R3 = Aminosäurereste Bauprinzip der Proteine Primärstruktur (Verknüpfungsprinzip der Aminosäuren) Der räumliche Bau und das chemische Verhalten der Proteine hängt ab von: - Der Art der Aminosäure - Der Anzahl der Aminosäuren - Vor allem von der Reihenfolge der Aminosäuren Die Aufeinanderfolge der Aminosäuren in einem Protein wird als dessen Aminosäuresequenz bzw. Primärstruktur bezeichnet. z.B. Val – His – Leu – Ser – Ala – Glu – Lys – … Bei einem Polypeptid mit 100 Aminosäuren gibt es 20100 Möglichkeiten !!!! Ursache für die Mannigfaltigkeit der Proteine !!! (Im menschlichen Körper nur 5 Mio. verschiedene Proteine) Sekundärstruktur (Raumstruktur der Peptidketten) Verursacht durch Wasserstoffbrückenbindungen zwischen C O und N H - Schrauben- oder Helixstruktur: innermolekulare Wasserstoffbrückenbindungen - Faltblattstruktur: zwischenmolekulare Wasserstoffbrückenbindungen Ob Helix- oder Faltblattstruktur, hängt von der Primärstruktur, also von der Aminosäuresequenz des Proteins ab! Tertiärstruktur (spezielle Raumgestalt globulärer Proteinmoleküle) Verursacht durch spezielle kovalente Bindungen oder Kohäsionskräfte innerhalb des Makromoleküls: Kovalente Bindung: Disulfidbrücke Kohäsionsbindung: Ionenbindung Wasserstoffbrückenbindung Van der Waals-Kräfte Seite 76 © Florian Zeller 07/08 Myoglobin Häm Quartärstruktur Ist ein Eiweißmolekül aus mehreren einzelnen Polypeptidketten, die durch Wechselwirkung (nicht durch Peptidbindungen) zusammengehalten werden, aufgebaut, so besitzt es eine Quartiärstruktur. Bsp.: Hämoglobin Viele Enzyme scheinen nach diesem Prinzip aus verschiedenen Untereinheiten bestimmter Tertiärstruktur zu Molekülverbänden mit Quartärstruktur zusammengesetzt zu sein, wobei stets die charakteristische Quartärstruktur erst die typische Wirkung ermöglicht. Hämoglobin Seite 77 © Florian Zeller 07/08 Molekulare Wirkungsweise der Gene Was tun Gene nun eigentlich genau? Gene machen Enzyme ( Merkmale) Beadle-Tatum Versuch Neurospora gedeiht auf einem „Minimal-Nährmedium“ UV-Strahlen, können Mutationen in den Sporen bewirken (Zucker und Nährsalz) Watte Entwicklung einer Kolonie nur auf Vollnährmedium (Bestandteile des Minimalnährmediums + Vitamine + alle Aminosäuren) Kein Wachstum auf Minimalnährmedium + Vitamine (Beweis dafür, dass durch die Mutation die Fähigkeit zur Bildung von Aminosäuren gestört ist) Kolonie auf Minimalmedium + Aminosäuren Kein Wachstum auf Minimalmedium + Aminosäure Lysin Kolonie auf Minimalmedium + Aminosäure Tryptophan Kolonie auf Minimalmedium + Indol (Vorstufe des Tryptophans Experimentelle Untersuchung des Zusammenhangs zwischen Gen und Merkmal beim roten Schimmelpilz Neurospora, durch UV-Strahlen erzeugt. Eine Mutante kann eine Vorstufe des Tryptophans nicht mehr bilden. Neurospora-Myzel ist haploid, man kann von Phänotyp direkt auf den Genotyp schließen. a Gefäß mit Minimal-Nährmedium. Nicht mutierte Sporen keimen, das auswachsende Myzal wird durch Gift abgetötet. Mutierte Sporen keimen nicht, bleiben somit vom Gift unbeeinflusst und kommen ins Vollnährmedium. Seite 78 © Florian Zeller 07/08 z.B. Tryptophan als Endprodukt Wenn Gen 3 ausfällt, kann das Enzym 3 nicht produziert werden, was wiederum nicht zu dem Produkt Idol führt. Somit kann die Kette nicht weiter fortgesetzt werden und es kommt nicht zu dem Endprodukt Tryptophan. Seite 79 © Florian Zeller 07/08 Genetischer Code und dessen Verwirklichung (= Proteinbiosynthese) Überlegungen zum Dreiercode auf der DNS 1. Zucker-Phosphat-Homen der DNS Z P Z = Aminosäure 1 P Z P = Aminosäure 2 Die Information für die Aminosäuren kann nicht durch die Zucker-Phosphat-Holmen verschlüsselt sein, da nur 2 der 20 Aminosäuren codiert werden können. 2. Eine Base bedeutet eine Aminosäure Code reicht nur für 4 Aminosäuren aus 3. Ein Basendoublett bedeutet eine Aminosäure AA CA GA TA 4. AC CC GC TC AG CG GG TG AT CT GT TT für zusammen 16 Aminosäuren Ein Basentriplett bedeutet eine Aminosäure AAA AGA ACA ATA AAG AGG ACG ATG AAC AGC ACC ATC AAT AGT ACT ATT GCA CGA CCA CTA GCG CGG CCG CTG GCC CGC CCC CTC GCT CGT CCT CTT GAA GGA GTA CAA GAG GGG GTG CAG GAC GGC GTC CAC GAT GGT GTT CAT TAA TGA TCA TTA TAG TGG TCG TTG TAC TGC TCC TTC TAT TGT TCT TTT Codewort auf der DNA = Codogen 64 Möglichkeiten Basentriplett der DNA = Codogen z.B. AAA AAC AAG TAG lückenloset, kommafreier Code Basentripplet der RNA = Codons z.B. UUU UUG UUC AUC STP = Terminator-Codon = Abbruch-Codon Code-Sonne (Code-Lexikon, genetisches Wörterbuch) Start Start = Starter-Codon, die am Anfang der Translation stehen. Aminosäuren: Arg Arginin Asn Asparagin Asp Asparaginsäure Ala Alanin Cys Cystein Gln Glutamin Glu Glutaminsäure Gly Glycin His Histidin Ile Isoleucin Leu Lys Met Phe Pro Ser Thr Trp Tyr Val Leucin Lysin Methionin Phenylalanin Prolin Serin Rhreorin Tryptophan Tyrosin Valin Seite 80 © Florian Zeller 07/08 Der genetische Code ist UNIVERSELL !!! d.h. ein bestimmtes Codon bedeutet bei allen bisher untersuchten Organismen die selbe Aminosäure. 64 Codons: 61 für die Bezeichnung der unterschiedlichen Aminosäuren (20) degeneriertes Codesystem UAG UAA UGA = Abbruch Codons (= Stopp Codons) Wenn diese Tripletts in der m-RNA (mesenger-RNA) vorkommen, erfolgt KEINE Synthese mehr bzw. eine begonnene Polypeptidkette wird abgebrochen. AUG GUG = Start Condons Sie signalisieren den Beginn einer Polypeptidsynthese, aber nur wenn sie auf ein oder eine kleine Serie von Stopp-Codons folgen. Ansonsten, d.h. innerhalb eines Gens, bedeuten diese Triplets normale Aminosäuren. Entzifferung des genetischen Codes Nirenberg, Matthei 1961 künstlich synthetisierte m-RNS m-RNS: U-U-U-U-U-U-RNS 20 Ansätze des zellfreien Systems von E.coli mit je einer anderen 14C markierten Aminosäure Zellfreies System von E.coli (Ribosomen, energieReiche Phosphate, Enzyme) (Ausflockung) Phe Phe Phe Filter Trichter Der Ansatz mit der Aminosäure Phenylalanin zeigte gegenüber den anderen Ansätzen eine 1000-fach gesteigerte Aktivität. Folgerung: ein Polypeptid aus Phe* ist entstanden Codewort UUU bedeutet Phenylalanin Nirenberg 1965 : Khorana 1965: Synthese von m-RNS-Trinucleotiden bekannter Basenfolge Entzifferung fast aller Codeworte Synthese von DNS-Oligonucleotiden definierter Zusammensetzung Endgültige Klärung des genetischen Codes Proteinsynthese Transkription = die Phase des Umschreibens einer bestimmten Basensequenz von der DNS in die mesenger-RNS Information Gen = DNA-Abschnitt Speicher der genet. Information m-RNA Transportform der Information Protein Bausteine Aminosäure t-RNA Baustein der Proteine transportiert und aktiviert die AS Aminosäure-tRNA sorgt zusamen mit der mRNA für die Reihenfolge Translation = ist die Phase des Übersetzens der Basensequenz der m-RNS in die Aminosäuresequenz eines bestimmten Proteins mit Hilfe der Kleeblattförmigen t-RNS (transfere-RNS) Seite 81 © Florian Zeller 07/08 Modellvorstellung vom Transkriptionsvorgang Der Enzymkomplex der Transkriptase erkennt Startstellen auf der DNS an einer bestimmten Basensequenz und setzt sich darauf fest. Anhand der Basensequenz erkennt er, welcher der beiden Stränge in welcher Richtung abgeschrieben werden soll. Er öffnet den Doppelstrang und ermöglicht Basenpaarung zwischen dem sog. codogenen Strang und den Basen der viererlei Nucleotide, die er zu m-RNS verknüpft. Während der Transkription wandert der hohlzylinderförmige Enzymkomplex an der DNS entlang, bzw. diese durch ihn hindurch, wobei ein m-RNS-Strang aus ihm herauswächst. Er enthält die Basensequenz des sog. Code-Stranges. Bestimmte Stellen erkennt der Komplex schließlich als Zielstellen, wo er sich und die m-RNS ablöst. Ein Transkriptionsabschnitt auf der DNS und damit auch eine m-RNS enthält die Information von wenigstens einem Gen, meist von mehreren. Über einen Transkriptionsabschnitt wandern (bei E. coli) gleichzeitig mehrere Transkriptase-Moleküle. Die wachsenden m-RNS-Stränge werden sofort von Ribosomen besetzt. Auf m-RNS aufgefädelte Ribosomen nett man Poly-Ribosomen. Translation – ein Protein wird „montiert“ „Vorbereitung“: Die t-RNS wirkt als Vermittler zwischen m-RNS und Aminosäuren! Für jede Aminosäure gibt es eine spezifische t-RNS, die sie zu den Ribosomen transportiert. Die Aktivierung der Aminosäure und deren Anbindung an „ihre“ t-RNS erfolgen unter Energieverbrauch durch sog. Synthetasen (Enzyme). t-RNS-Molekül: Kleeblattstruktur Anticodon: komplementär zu 1 bestimmten Basentriplett der m-RNA, also zu einem codon! Die Zuordnung der passenden Aminosäure erfolgt über die Synthetasen welche die spezifische Raumstruktur der t-RNS Moleküle abgreifen und jeweils die richtige Aminosäure auswählen und anheften. Aminosäureanheftestelle: immer gleich; kann daher nur anheften, aber nicht auswählen! (immer CCA) Synthese des Proteins Zunächst lagern sich die beiden Untereinheiten des Ribosoms an der Starsequenz der m-RNS zu einem funktionsfähigen Ribosom zusammen. Im Ribosom paart an jedes Codon (3 Nucleotide = Triplett) der m-RNS eine t-RNS (beladen mit ihrer spezifischen Aminosäure) mit „passendem“ Anticodon (zum Codon komplementäres Triplett). Das Ribosom besitz zwei t-RNS-Bindestellen. Dadurch wird der Kontakt zwischen den an den beiden t-RNS hängenden Aminosäuren hergestellt – die Peptidbindung kann geknüpft werden. Die „vordere“, nun freie t-RNS löst sich aus dem Ribosom und macht Platz für die nächste Ankopplung einer „neuen“ beladenen t-RNS an die um ein Triplett weitergerückte m-RNS. Seite 82 © Florian Zeller 07/08 Durch die Wiederholung dieses Vorgangs wird die angefangene Aminosäure-Kette um eine Aminosäure nach der anderen verlängert, und zwar exakt so, wie es die Codons der m-RNS vorschreiben. Kommt das Ribosom an ein Stop-codon der m-RNS, so endet der Translationsprozess. Das gebildete Protein wird frei und nimmt seine funktionsfähige Raumstruktur ein. Seite 83 © Florian Zeller 07/08 Analogen aus der menschlichen Sprache 4 Buchstaben des genetischen Codes A,C,G,T bzw. U entsprechen 4 anderen Buchstaben des Alphabets E, I, N, S. Eine kurze Sequenz von Tripletts wird in die menschliche Sprache übertragen. Die Wortfolge ISS NIE EIN EIS soll als Modell für ein Protein aus 4 Aminosäuren dienen. Dieser Satz soll nun verändert werden: a) Austausch eine Buchstaben (= Base) in einem Wort (=Triplett) ISS NIE EIN SIS Sie kann, muss aber nicht entstellt sein. b) Einschub eines Buchstaben (= zusätzliche Base oder Nucleotid) INS SNI EEI NEI S Leseraster ist verschoben; kein Sinn mehr! c) Leseraster ebenfalls verschoben; kein sinn! Ausfall eines Buchstaben ISS …IEE INE IS d) Ausfall eines ganzen Wortes (= Triplett) … NIE EIN EIS ISS NIE … EIS Takt bleibt erhalten; Sinn entstelt Sinn bleibt Zellverhältnisse Codogener DNA-Strang: m-RNA-Original: codierte Aminosäure: a) Basenaustausch (m-RNA): [A gegen G in der DNA] GAC CUG Leu CCT GGA Gly ATG UAC Tyr CGT GCA Ala CAC GUG Val AAG UUC Phe TCG AGC Ser CUG Leu GGA Gly CAC His GCA Ala GUG Val UUC Phe AGC Ser liefert Fehlsinn: ein Enzym mit einer falschen Aminosäure und dadurch mehr oder weniger Verminderte Aktivität. b) Baseneinschub (m-RNA): [zusätzlich G in der DNA] CUG Leu GGA Gly CUA Leu CGC Arg AGU Ser GUU Val CAG C Gln liefert Unsinn: ein Protein mit völlig veränderter Aminosäuresequenz und Tertiärstruktur. Ursprüngliche Enzymfunktion verloren. c) Basenverlust (m-RNA) [Wegfall von A in der DNA] CUG Leu GGA Gly ACG Thr CAG Gln UGU Cys UCA Ser GC. ähnlich wie bei b. d) Verlust eines Tripletts CUG GGA GCA GUG UUC AGC [Wegfall von ATG in der DNA] Leu Gly Ala Val Phe Ser liefert mehr oder weniger Sinn: ein Protein, dem 1 Aminosäure fehlt und das, je nach der Bedeutung dieser Aminosäure für die Tertiärstruktur, eine mehr oder weniger gestörte Enzymaktivität. Durch energiereiche Strahlung mögliche Veränderungen der DNS: 1. Doppelstrangbruch 2. Zweistrangbruch 3. Basenveränderung (=Punktmutation) Seite 84 © Florian Zeller 07/08 Allgemeine Fehlerquellen: - Gelegentlich bei normaler Replikation - Mutagene (Strahlung, Chemikalien) Ohne Mutation keine Evolution !!! - Veränderungen im Erbgut einer angepassten Art sind meist negativ Wenn sie in seltenen Falle positiv sind, bringen sie einen Selektionsvorteil, die Veränderungen im Erbgut führen auf lange Sicht zur Entstehung neuer Arten; z. B. fliegender Fisch, Schneearten Reparaturenzyme beseitigen Schäden: - Bei der Replikation meist sofort - Durch Mutagene in kurzer Zeit, sofern nur einer der beiden DNA-Stränge betroffen ist Reparaturmechanismus: Das beschädigte Strangstück wird durch ein Enzym herausgeschnitten und abgebaut. Das fehlende Stück wird neu gebildet, wobei der Komplementäre unbeschädigte Strang als Martritze dient. Anmerkung: Wenn beide Stränge der DNA geschädigt wurden Mutationsmechanismen 1. bleibende Veränderung !!! (nur zur Anschauung! / erklären können!) Mutagen salpetrige Säure HNO2 bzw. chemische Veränderung einzelner Basen HNO2 setzt aus der Aminogruppe der Basen (z.B. Cytosin) Stickstoff frei. Beispiel: Aus Cytosin entsteht Uracil Cytosin Uracil Folgen einer solchen Basenveränderung HNO2 hat letztlich den Austausch des Basenpaares Mutierte Form !!! gegen das Basenpaar T=A bewirkt. Es wurde ein REPLIKATIONSFEHLER ausgelöst. Seite 85 © Florian Zeller 07/08 2. Einbau von Basenanaloga z.B. 5-Bromuracil (BU), eine dem Thymin analoge Base Es kann nur während der DNS-Replikation anstelle von Thymin eingebaut werden und dann unter Umständen einen weiteren Basenaustausch bewirken. Keine Folgen, wenn weiterhin Adenin als Paarungspartner verwendet wird: Folgen, wenn Ketoform des BU in die Enolform übergeht! Diese paart mit Guanin !!! Wurde einmal Guanin eingebaut, dann wird es stets Cytosin als Paarungspartner suchen und die Mutation ist fixiert! Aber: Die Enolform kann wieder in die Ketoform übergehen, dann stellt sich in den nächsten Generation wieder die ursprüngliche Basensequenz ein ! EINE IN DIE DNS EINGEBAUTE BU-BASE WIRKT WIE EIN ZUFALLSGENERATOR, DER ÜBER GENERATIONEN IMMER WIEDER BASENAUSTAUSCHE UND DAMIT GENMUTATIONEN BEWIRKT !!! 3. Rastermutationen: EINBAU oder VERLUST von NUCLEOTIDEN Acridinmoleküle sind so groß wie Nucleotide. Sie können sich in die DNS einschieben und ebenso wieder ausgebaut werden. Möglichkeit 1: Acridineinschub in ruhende DNS, d.h. Einbau vor der Replikation Seite 86 © Florian Zeller 07/08 Einbau vor der Replikation X‘ = beliebiges zusätzliches Nucleotid Ausbau vor nächster Replikation Ausgangsform Einbau ! DNS-Molekül mit zusätzlichem Basenpaar !! Folgen bei Manifestation im fertigen Protein: Rastermutation, Verschiebung des Leserasters auf DNS & RNS Aminosäuresequenz wird verändert !! Raumstruktur (Tertiär- & Quartiärstruktur) es Proteins verändert !! Möglichkeit 2: Acridineinbau während der Replikation, d.h. anstelle eines Nucleotids Einbau während der Replikation Ausstoß vor der nächsten Replikation Ausgangsform Verlust !! DNS-Molekül, dem 1 Basenpaar fehlt !! Folgen bei Manifestation im fertigen Protein: Rastermutation, Verschiebung des Leserasters auf DNS & RNS Aminosäuresequenz wird verändert !! Raumstruktur (Tertiär- & Quartiärstruktur) es Proteins verändert !! Seite 87 © Florian Zeller 07/08 Wirkungsmechanismen von Mutagenen Mutagene können auf vielerlei Art und Weise wirken. Dies hängt beispielsweise von der Struktur und der Reaktion eines chemischen Mutagens mit den Basen der DNA ab. Einige Beispiele für die Wirkungsweise von Mutagenen sollen hier dargestellt werden. 1. Chemische Änderung normaler Basen z.B. durch salpetrige Säure HNO2: sie bewirkt den Umbau von Cytosin in Uracil: während sich Cytosin mit Guanin paart, verbindet sich Uracil bei der Replikation mit Adenin. Nach zwei Replikationen wurde wurde letztlich das Basenpaar C-G durch das Basenpaar T-A ausgetauscht. [vgl. Daumer „Genetik“, Abb. 85.1] 2. Einbau von instabilen „Basenanaloga“ anstatt der natürlichen Basen (Basenanaloga sind basenähnliche Stoffe, die wie normale Basen, z.B. bei der Replikation, in die DNA eingebaut werden. Durch spontane Umlagerung können sie ihre Molekülstruktur und damit ihre Paarungseigenschaften ändern) z.B. von 5-Bromuracil (BU): die Normalform ist Thymin analog, die Sonderform paart sich aber wie Cytosin mit Guanin. Damit kommt es nach zwei Replikationen zum austausch A-T gegen G-C. 3. Veränderung der Nukleotidzahl durch zeitweiligen Einschub nukleotidähnlicher Moleküle z.B. von Acridin (oder Teerstoffe des Zigarettenrauchs): Es sind Moleküle mit Ringsystemen, die sich zwischen benachbarte Basenpaare der DNA schieben und eine Base zuviel vortäuschen. Bei einer Replikation wird an diese vermeintliche Base eine beliebige andere angelagert. Je nach dem Ort des zeitweiligen Einschubs bzw. Einbaus kommt es zum Einschub (Insertion) oder zum Verlust (Deletion) von einem Nukleotid. [vgl. Daumer „Genetik“, Abb. 86.1] 4. Vernetzung zweier benachbarter oder gegenüberliegender Basen der DNA z.B. durch kurzwellige UV-Strahlung: Am häufigsten ist die Vernetzung zweier benachbarter Thymin-Moleküle in demselben Einzelstrang, wodurch ihre komplementäre Basenpaarung unmöglich gemacht wird. Als Folge wird die DNA nicht mehr richtig transkribiert und repliziert. Die hier beschriebenen Mutagene werden gewöhnlich in Laboratorien benutzt. Eine große Zahl anderer Chemikalien ist auch Mutagen. Da wir ständig mit Stoffen in Berührung kommen, die potentielle Mutagene sein können, ist die Erforschung mutagener Einflüsse und ihrer Wirkungen wichtig, um die nötigen Schutzmaßnahmen für Bevölkerung und Umwelt treffen zu können. Dazu werden in zunehmendem Umfang Medikamente, Kosmetika, Nahrungsmittelzusätze, Konservierungs- und Düngemittel, Insektizide, usw. durch geeignete Verfahren einem Mutagenitätstest unterzogen. Regulation der Genaktivität Wären alle Strukturgene (ca. 1 Million) einer Zelle gleichzeitig aktiv, so gäbe das ein fürchterliches Chaos in der Zelle. Das ist nicht der Fall. Offenbar unterliegt die Genaktivität einer Kontrolle, die dazu führt, dass jeweils nur das produziert wird, was gerade nötig ist! Regulation der Enzymaktivität Das für die Regulation verantwortliche Enzym ist ein besonderes. Normale Enzyme haben 1 aktives Zentrum, also einen Bindungsort für das Substrat. Allosterisches Enzym ist ein regulierbares Enzym mit zwei verschiedenen, hochgradig spezifischen Bindungsorten Seite 88 © Florian Zeller 07/08 Zeichenerklärung: = ungleichsinniger Zusammenhang (je weniger, desto mehr / je mehr, desto weniger) + = gleichsinniger Zusammenhang (je mehr, desto mehr / je weniger, desto weniger) durch das Endprodukt gehemmter Zustand des Enzyms Aktiver Zustand des Enzyms Endprodukt allosterische Konformationsänderung (= Raumstrukturänderung) des Enzyms E1 Bindung für das Endprodukt = allosterisches Zentrum Bindungsort für das Ausgangssubstrat = aktives Zentrum (findet Umsetzung statt) Bindungsort blockiert Ausgangssubstrat Ausgangssubstrat Bei hohem Vorkommen des Endprodukt D erfolgt durch das Andocken die allosterische Konformationsänderung und blockiert die Andockung des Stoffes A und dessen Umsetzung. Bei mangel am Endprodukt D erfolgt Abkopplung vom Enzym und die Umsetzung kann von statten gehen. Nun gibt es auch Stoffe, welche die Aktivität eines Enzyms beeinflussen, man nennt sie Effektoren: Hat der Effektor hemmende Wirkung, wirkt er als Inhabitor, hat der Effektor fördernde Wirkung, wirkt er als Aktivator. Allosterische Enzyme können also fördernde und hemmende Einflüsse empfangen und darauf reagieren: Feinregulierung des Stoffwechsels Rolle beim An- und Abschalten von Genen, d.h. Regulation bei der Proteinbiosynthese Seite 89 © Florian Zeller 07/08 Regulation der Genaktivität bei Bakterien Jacob-Monod-Modell 1. Aufbauender Stoffwechsel Abschalten er Enzymproduktion durch ein Endprodukt Die Produktion der gesamten Enzymserie, die an einer Synthesekette beteiligt ist, wird abgestellt. Begriffe: Strukturgene S: Gene für die Synthesekette sind auf dem Chromosom unmittelbar benachbart. Operator O: Ein Gen, das die Tätigkeit (Operation) einer nachfolgenden Gruppe von Strukturgenen kontrolliert, nennt man Operator. (DNS-Abschnitt unmittelbar vor dem 1. Strukturgen der Synthesekette) Die Steuereinheit Operatorgen + Gruppe der Strukturgene = Operon Promoter P: Im vorderen Abschnitt des Operatorgens liegt der Startplatz für die Transkriptase Abschalten des tätigen Operons: Vermutung: das Endprodukt verändert irgendwie den Zustand des Operatorgens, so dass die Transkriptase die nachfolgenden Gene nicht mehr ablesen kann. Hierzu ist ein Vermittler notwendig, das Regulatorgen. Regulatorgen R: - reguliert die Tätigkeit des Operons - ist ein DNA-Abschnitt, der nicht in unmittelbarer Nähe des Operons liegt - sein Genprodukt ist ein allosterisches Enzym!!! Es kann in einem bestimmten Konformationszustand an das Operatorgen binden und die Transkription unterbinden (= Repressor) Der Repressor ist aber zunächst inaktiv!! Das Endprodukt kann aber als Effektor an den allosterischen Repressor binden Konformation des Enzyms ändert sich Repressor wird aktiv, d.h. er kann jetzt an das Operatorgen binden Seite 90 © Florian Zeller 07/08 Was geschieht, wenn nun die Endproduktkonzentration durch Verbrauch sinkt? Repressor und Corepressor trennen sich, die Blockierung des Operons wird aufgehoben, Transkription kann erneut durchgeführt werden (bis eben wieder genug EP vorhanden ist…) 2. Abbauender Stoffwechsel Anschalten der Enzymproduktion durch ein abzubauendes Substrat Kommt E.coli z.B. plötzlich in Kontakt mit Lactose, so stellt es daraufhin die nötigen Enzyme für den Abbau des Substrates Lactose her. Die Lactose löst also die Bildung der zu ihrem Abbau nötigen Enzyme selbst aus! Auslösevorgang = Induktion der Enzymsynthese Substrat, das die Induktion bewirkt = Induktor Lactose – Operon: enthält 3 Strukturgene Wenn keine Lactose im Medium: Operon liegt „ausgeknipst“ vor. Lac-Repressor (Produkt des Regulatorgens) ist aktiv, er bindet an den Operator und blockiert so die Transkription. Da der Repressor ein allosterisches Protein ist, hat er eine zweite Bindungsstelle für das Substrat Lactose. Wenn Lactose in die Zelle gelangt: Operon wird „eingeschaltet“ Substrat bindet an Repressor, dieser ändert dadurch seine Konformation, löst sich vom Operator ab und ist inaktiv. Folge: Transkriptese kann ablesen, Transkription, Translation, Enzyme zum Abbau der Induktion der Genaktivität. Die Lactose verschwindet durch den Abbau, das Lac-Operon wird wieder geschlossen, da der Repressor nach Austritt der lactose wieder den aktiven Konformationszustand annimmt und an den Operator bindet. (alle Abbildungen von Seite 89 bis 91 skizzieren können !!!) Seite 91 © Florian Zeller 07/08