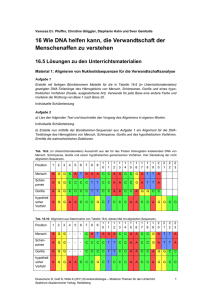
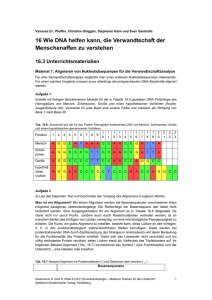
Kartierung beta-Globin Mensch vs. Tiere Anleitung
Werbung

Muster: Schnittstellen von Restriktionsenzymen Projektthema: Ähnlichkeitsanalyse anhand von Restriktionsmustern Dieses Unterrichtsprojekt hat eine einfache Verwandschaftsanalyse zum Inhalt. Sie basiert auf einem Vergleich von Fragmentmustern (Anzahl und Länge), die bei einer Restriktion von Globin-Sequenzen verschiedener Organismen erhalten werden. Diese Art des Vergleichs kann dann als Kriterium herangezogen werden, wenn Sequenzen eine hohe Sequenzübereinstimmung zeigen. Das Projekt verwendet das für verschiedene Rechner-Platformen frei erhältliche Programm ApE (http://www.biology.utah.edu/jorgensen/wayned/ape/ )und ein beliebiges Tabellenkalkulationsprogramm. Neben dem Lehrplaninhalt „Alignment“, der lediglich auf der Ebene der Anwendung integriert ist, wird auch der Lehrplaninhalt Dotplot berücksichtigt. Dies geschieht auf einer Ebene, die es erlaubt unter die Ebene der reinen Anwendung zu gehen, wenn die eingesetzte Rechenblatt-Datei mit den Schülern selber erstellt. Das Projekt besteht aus zwei Teilen: A. In-silico-Restriktionsverdau mit mehreren Enzymen B. Analyse der Restriktionsmuster Mensch-Tier mit Dot-Plots A. In-silico-Restriktionsverdau mit verschiedenen Enzymen Schritt 1: Laden einer DNA-Sequenzdatei am Beispiel der -Globin-DNA des Menschen. Öffnen Sie die ApE-Software. Wählen Sie unter FILE die Option OPEN. Wählen Sie die FASTA-Datei Homo sapiens beta hemoglobin NM_000518 aus. Wählen Sie die Schaltfläche ÖFFNEN. Die DNA-Sequenz erscheint im ApE-Fenster. Schritt 2: Verdau einer gewählten DNA-Sequenz mit ausgewählten Restriktionsenzymen Wählen Sie unter ENZYMES die Option ENZYME SELECTOR aus. Wählen Sie folgende Restriktionsenzyme aus: AccI , BamHI , BseRI , BstXI , DraIII , EcoRI Klicken Sie auf das Kästchen SELECTION ONLY in der oberen rechten Ecke. Klicken Sie auf DIGEST. Es erscheint das sog. „Digestion Window“. Es zeigt die Darstellung eines DNARestriktionsfragmentmusters wie es nach einer Gelelektrophorese erscheinen würde. Fahren Sie mit dem Cursor über die dargestellten Restriktionsfragmente und überprüfen Sie die Position der einzelnen Fragmente innerhalb der vertikal dargestellten Gesamtsequenz. Die übereinstimmenden Bereiche werden rot hervorgehoben. Die Tabelle zeigt in der linken Spalte die Längen der einzelnen Fragment in der Einheit Basenpaare (Bp) an. Schritt 3: Graphische Darstellung (Kartierung) der gefundenen Restriktionsorte Wählen Sie unter ENZYMES die Option GRAPHIC MAP aus. Das erscheinende Fenster – die sog. GRAPHIC MAP („Gen-Karte“) – zeigt die Positionen der Restriktionsorte für jedes verwendete Enzym an. Hinter jedem Enzymnamen steht in Klammern die Anzahl der Restriktionsorte dieses Enzyms für die analysierte DNA-Sequenz. Schieben Sie das Fenster mit dem Cursor von der unteren Kante aus möglichst klein nach oben zusammen (siehe unten) Schritt 4 bis 6: Wiederholung mit verschiedenen tierischen Sequenzen Wiederholen Sie diesen Vorgang mit den drei folgenden -Globin-DNA-Sequenzen von Schimpanse, Gorilla und Krallenfrosch: Schimpanse Gorilla Krallenfrosch Pan troglodytes beta hemoglobin X02345 Gorilla gorilla beta hemoglobin X61109 Xenopus tropicalis beta hemoglobin M32457 (FASTA-Datei) (FASTA-Datei) (FASTA-Datei) Ordnen Sie die vier Fenster der Graphic Maps auf dem Desktop untereinander an. Vergleichen Sie die Anzahl und die Positionen der angezeigten Restriktionsorte ! Wieviele verschiedene Enzyme sind angezeigt ? Wie oft schneiden die Enzyme in jeder DNA-Sequenz ? Welche DNA-Sequenzen weisen identische bzw. nicht-identische Positionen für vorhandene Restriktionsorte auf ? Ergänzen Sie folgendeTabelle ! Anzahl der Restriktionsschnittstellen AccI BamHI BseRI BstXI DraIII EcoRI Mensch Schimpanse Gorilla Krallenfrosch Aufgabe: Analyse der Restriktionsmuster Mensch vs. Tier mit Dot-Plots Schritt 1: Dot-Plot: Restriktionsorte Öffnen Sie folgende Excel-Datei: Kartierung beta-Globin Mensch Tiere AB 230306.xls Wählen Sie das Tabellenblatt Dot-Plot aus. Bearbeiten Sie die drei Dot-Plots unter der Rubrik a) Dot-Plot: Restriktionsorte. Ergänzen Sie die fehlenden Werte mit den Angaben zu den vorhandenen Restriktionsorten aus den „Graphic Maps“ auf dem Desktop. Folgen Sie dabei den Anweisungen im Tabellenblatt Einführung. Schritt 2: Bearbeiten Sie die Rubrik b) Dot-Plot: Positionen der Restriktionsorte. Ergänzen Sie die fehlenden Werte mit den Angaben zu den genauen Positionen der Restriktionsorte aus den „Graphic Maps“ auf dem Desktop. Bewerten Sie den jeden Dot-Plot. Schritt 3: Bearbeiten Sie die Rubrik c) Dot-Plot: Differenzen zwischen Restriktionsorten. Errechnen Sie die Differenzwerte zwischen zwei aufeinanderfolgenden Restriktionsorten. Schritt 4: Endbewertung Ermitteln Sie die Summe der Bewertungen für die einzelnen Tierarten. Welche -Globin-DNA der drei verschiedenen Tierarten weist mit der -Globin-DNA des Menschen die meisten Ähnlichkeiten im Restriktionsmuster auf ? ----------------------------------------------------------------Welche -Globin-DNA der drei verschiedenen Tierarten weist mit der -Globin-DNA des Menschen kaum Ähnlichkeiten im Restriktionsmuster auf ? ----------------------------------------------------------------Führen Sie je ein paarweises Alignment mit den Sequenzen von Mensch und Schimpanse bzw. Mensch und Gorilla durch ? Vergleichen Sie die ApE-Bewertung mit Ihrer Bewertung, die auf einer Restriktionskartierung beruht. -----------------------------------------------------------------