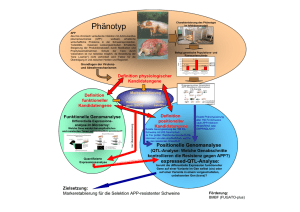
Datenanalyse von Affymetrix-Microarrays
Werbung

Vorlesung: Vorverarbeitung von Affymetrix-Daten 28.10.2003 Vorverarbeitung von Microarray Daten: Beispiel: Affymetrix Biologie Diagnostik Therapie ... ? ExperimentDesign Experiment (Microarray) ! Bildverarbeitung Biologische Verifikation Rohe Intensitätswerte Vom “Tiff” zum Expressions Level Analyse: Clustering; Class Discovery; Klassifikation; Differentielle Gene; .... Normalisierung Expressions Level Vorverarbeitung von Microarray Daten: Beispiel: Affymetrix * GeneChip Probe Array Single stranded, labeled RNA target * * * * Oligonucleotide probe 18µm 1.28cm 106-107 copies of a specific oligonucleotide probe per feature >450,000 different probes Bild eines hybridisierten Arrays Compliments of D. Gerhold Extraktion der Poly-A RNA Zellpool aus Gewebeproben oder Zellkulturen Amplifikation und Markierung der RNA Chip-hybridisierung Oligonukleotid Auslesen des Fluoreszenzsignals Chipzelle Vorverarbeitung von Microarray Daten: Beispiel: Affymetrix ... TGTGATGGTGGGAATGGGTCAGAAGGACTCCTATGTGGGTGACGAGGCC TTACCCAGTCTTCCTGAGGATACACCCAC TTACCCAGTCTTGCTGAGGATACACCCAC probe cell 1 2 PM MM 3 4 ... ... ... probe pair 17 18 19 20 probe set Oligopaar PM (A) MM (B) Bildzelle Saturierte Zelle Vorverarbeitung von Microarray Daten: Beispiel: Affymetrix 3331.04 1178.82 128 206.46 74.32 357713 2939.71 1053.74 270.6 167.4 78 207.5 76.19 14696 108.74 68.25 3331.04 1178.82 122 128183.2 206.4678.4174.32 357713 2939.71 1053.74 4009.59 1450.57 452714 3918.8 1434.41 108.7487.1968.25 351.81270.6 153.39167.4 98 78166.4207.5 69.176.191983814696 176.04 3331.04 1178.82 128183.2 206.46 74.32 357713 2939.71 1053.74 4009.59 1450.57 452714 3918.8 1434.41 323.52 152.39 102 122 159.41 67.8578.4110792 74.37 45.75 270.6 78166.4207.5 76.1919838 14696 108.7487.1968.25 351.81 153.39167.4 176.04 2533 789.54 95 98 161.26 68.04 69.1 209832 2216.25 556.29 3331.04 1178.82 128183.2 206.46 74.32 357713 2939.71 1053.74 4009.59 1450.57 122 78.4110792 452714 3918.8 1434.41 323.52 152.39 102 159.41 67.85 74.37 45.75 6614.93 2105.24 123 152.99 63.89 1086014 9425.24 2987.65 270.6 78166.4 76.19 14696 108.74 351.81 153.39 98 69.1 19838 176.04 789.54 95 161.26 68.0428666 209832 2216.25 556.2987.1968.25 449.05 2533 156.78 83167.4 158.35 69.38207.5 340.77 142.2 3331.04 1178.82 12863.89 206.46 74.32 357713 2939.71 1053.74 4009.59 1450.57 122 78.41 452714 3918.8 1434.41 323.52 152.39 102 159.41 67.85 10792 74.37 45.75 6614.93 2105.24 152.99 1086014 9425.24 2987.65 1011.52 296.56 96 123 164.46 70.44183.2 55635 605.57 196.27 270.6 167.4 7869.38207.5 76.19 14696 108.7487.1968.25 351.81 153.39 98 69.1 19838 176.04 2533 789.54 95 161.26 68.0428666 209832 2216.25 556.29 449.05896.3 156.78 83 158.35 340.77 142.2 3136.51 121 168.92 69.6166.4 262667 2281.34 727.88 4009.59 1450.57 122 183.2 78.41 452714 3918.845.75 1434.41 323.52 152.39 102 159.41 67.85 10792 74.37 6614.93 2105.24 123 152.99 63.8955635 1086014 9425.24 2987.65 1011.52 296.56 96 164.46 70.4480968 605.57 196.27 1591.61 547.79 101 193.76 80.3 762.04 232.46 351.81 153.39 98 166.4 69.1 19838 176.04 2533 789.54 95 161.26 68.04 209832 2216.25 556.2987.19 449.05896.3 156.78 83 158.35 69.38 28666 340.77 142.2 3136.51 121 168.92 69.668256 262667 2281.34 727.88 879.87 388.18 99 190.72 75.02 643.04 288.53 323.52 152.39 102 159.41 67.85 10792 74.37 6614.93 2105.24 123 152.99 63.89 1086014 9425.24 2987.6545.75 1011.52 296.56 96 164.46 70.4480968 55635 605.57 196.27 1591.61 547.79 101 193.76 80.3 762.04 232.46 6840.42 2674.61 131 222.09 83.66 766497 6212.39 2297.01 2533 789.54 95 161.26 68.04 209832 2216.25 556.29 449.05 156.78 83 158.35 69.38 28666 340.77 3136.51 121 168.92 69.668256 262667 2281.34 727.88142.2 879.87 388.18896.3 99 190.72 75.02 643.04 288.53 1415.85 497.36 93 202.82 73.02 119114 1274.41 388.28 6614.93 2105.24 123 152.99 63.89 1086014 9425.24 2987.65 1011.52 296.56 96 164.46 70.44 55635 605.57 196.27 1591.61 547.79 101 193.76 80.3 80968 762.04 232.46 6840.42 2674.61 131 222.09 83.66 766497 6212.39 2297.01 2121.88 663.31 104 176.59 70.42 182108 1818.55 636.71 449.05896.3 156.78 83 158.35 69.38 28666 340.77 3136.51 121 168.92 69.6 262667 2281.34 727.88142.2 879.87 388.18 99 190.72 75.02 68256 643.04 288.53 1415.85 497.36 202.82 73.02 119114 1274.41 388.28 929.93 351.67 103 93 180.81 77.91 66478 628.77 219.12 1011.52 296.56 96 164.46 70.44 55635 605.57 196.27 1591.61 547.79 101 193.76 80.3 80968 762.04 232.46 6840.42 2674.61 131 222.09 83.66 766497 6212.39 2297.01 2121.88 663.31 104 176.59 70.42 182108 1818.55 636.71 652.06 248.54 110 158.69 68.76 75527 703.35 291.83 3136.51 896.3 121 168.92 69.6 262667 2281.34 727.88 879.87 388.18 99 190.72 75.02 68256 643.04 288.53 1415.85 497.36 93 202.82 73.02 119114 1274.41 388.28 929.93 351.67 103 180.81 77.91 628.77 219.12 212.99 135.17 16 163.35 68.26 135266478 58.06 23.88 1591.61 547.79 101 193.76 80.3 80968 762.04 232.46 6840.42 2674.61 131 222.09 83.66 766497 6212.39 2297.01 2121.88 663.31 104 176.59 70.4275527 182108 1818.55 636.71 652.06 248.54 158.69 68.76 703.35 291.83 1089.73 368.88 90 110 157.89 65.12 80666 952.99 312.51 879.87 388.18 99 190.72 75.02 68256 643.04 288.53 1415.85 497.36 93 202.82 73.02 119114 1274.41 388.28 929.93 351.67 103 180.81 77.91 66478 628.77 219.12 212.99 135.17 16 163.35 68.26 1352 58.06 23.88 282.58 142.43 55 158.36 67.76 9296 122.18 53.3 6840.42 2674.61 131 222.09 83.66 766497 6212.39 2297.01 2121.88 663.31 104 176.59 70.4275527 182108 1818.55 636.71 652.06 248.54 110 158.69 68.7680666 703.35 291.83 1089.73 368.88 90 157.89 65.12 952.99 312.51 468.83 195.36 79 164.54 71.02 22005 270.1 129.46 1415.85 497.36 93 202.82 73.02 119114 1274.41 388.28 929.93 351.67 103 180.81 66478 628.77 219.12 212.99 135.17 16 163.35 68.2677.91 1352 58.06 23.88 282.58 142.43 55 158.36 67.76 9296 122.18 53.3 6945.94 2500.55 102 175.1 73.52 607270 6369.08 2275.77 2121.88 663.31 104 176.59 70.4275527 182108 1818.55 636.71 652.06 248.54 110 158.69 68.76 703.35 291.83 1089.73 368.88 90 157.89 65.1222005 80666 952.99 312.51 468.83 195.36 79 164.54 71.02 270.1 129.46 299.35 156.95 41 183.86 72.1 10433 153.31 89.69 929.93 351.67 103 180.81 77.91 66478 628.77 219.12 212.99 135.17 16 163.35 68.26 1352 58.06 282.58 142.43 55175.1 158.36 67.76 9296 122.18 53.323.88 6945.94 2500.55 102 73.52 607270 6369.08 2275.77 1443.79 540.98 88 191.7 81.41 122311 1485.36 515.21 652.06 248.54 110 158.69 68.76 75527 703.35 291.83 1089.73 368.88 90 157.89 65.1222005 80666 952.99 312.51 468.83 195.36 79 164.54 71.0210433 270.1 129.46 299.35 156.95 41 183.86 72.1 153.31 89.69 862.38 274.78 123 236.58 90.37 72666 553.8 192.45 212.99 135.17 16 163.35 68.26 1352 58.06 53.323.88 282.58 142.43 55175.1 158.36 67.76 9296 122.18 6945.94 2500.55 102191.7 73.52 607270 6369.08 2275.77 1443.79 540.98 88 81.41 122311 1485.36 515.21 1089.73 368.88 90 157.89 65.1222005 80666 952.99 312.51 468.83 195.36 79 164.54 71.0210433 270.1 129.46 299.35 156.95 41 183.86 72.172666 153.31 89.69 862.38 274.78 123 236.58 90.37 553.8 192.45 282.58 142.43 55175.1 158.36 67.76 9296 122.18 6945.94 2500.55 607270 6369.08 2275.77 53.3 1443.79 540.98 88 102191.7 81.4173.52 122311 1485.36 515.21 468.83 195.36 79 164.54 71.021043322005 270.189.69 129.46 299.35 156.95 183.86 153.31 862.38 274.78 123 41 236.58 90.37 72.172666 553.8 192.45 6945.94 2500.55 88 102191.7175.181.4173.52 607270 6369.08 2275.77 1443.79 540.98 122311 1485.36 515.21 299.35 156.95 123 41 183.8690.37 72.17266610433553.8 153.31 862.38 274.78 236.58 192.4589.69 1443.79 540.98 88 191.7 81.41 122311 1485.36 515.21 862.38 274.78 123 236.58 90.37 72666 553.8 192.45 Lokalisation Intensität Annotation PM-MM Sequenz ... Vorverarbeitung von Microarray Daten: Probleme (1) Hintergrund (2) PM / MM (perfect match / mismatch) (3) “Summary statistics” ? PM MM 1.5 2.4 10.4 0.1 ... 1.3 3.4 ... ... Vorverarbeitung von Microarray Daten: Beispiele: MAS 5.0 (Affymetrix Microarray Suite, Affymetrix Software) Li/Wong: PNAS 2001 vol 98 (1), pp31-36 RMA: Robust Multiarray Analysis, Irizarry/Bolstad/Speed (NAR, 2003 31(4), e15) Vorab: Was ist Tukeys Biweight ? • Angabe der Tendenz • Maß für den Mittelwert • sehr robust gegenüber Ausreißern Vektor: X = (x1,...,xn) Berechne Tukey´s Biweight von X: T(X) (1) (2) (3) (4) (5) Berechne die absolute Distanz von jedem Datenpunkt zum Median Berechne S = Median der absoluten Abweichungen (MAD) Definiere u = (Datenpunkt-Median(Datenpunkte) )/(Konstante*S + ) Konstante = 5; = 0.00001 Gewichtsfunktion: (1 - u2)2 for |u| <= 1 w(u) = 0 else T(x) = i w(u) xi / i w(u) Vorab: Was ist Tukeys Biweight ? 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 xxx xx x X = 1,2,3,2,4,20 Median 2.5 Mittelwert 5.3 Tukeys Biweight 2.3 Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Array: k = 16 (zB) Kontrollzellen und leere Spots werden nicht weiter beachtet Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Alle Zellen eines Sub-Arrays (=Zone) werden der Größe nach geordnet Jeder Zelle wird ein Rang zugeordnet C1 C2 C3 C4 … Cn Definition von Hintergrund eines SubArrays: . Zbg = niedrigsten 2% des jeweiligen Subarrays Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Problem: Bei dieser Definition von Hintergrund (Zbg) gibt es “scharfe” Grenzen zwischen den einzelnen “Subarrays” Lösung: Glättung der Übergänge Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Glättung der Übergänge . . dk(x,y) = Distanz vom Mittelpunkt (.) des k-ten Segments zu einem Punkt mit den Koordinaten (x,y) Gewichtung: wk(x,y)=1/(dk2 + s) (default s=100) Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Neuer Hintergrund: b(x,y) = w (x,y) Z / w (x,y) k k bg k . . k Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Perfect match und Mismatch (PM MM) PM MM ... ... Definitionen: Adjustierte Intensität: A(x,y) = maxInt(x,y) – b(x,y) | NoiseFrac * n(x,y) NoiseFrac = 0.5 default n(x,y) = 1 / w(x,y) * (w(x,y) n Zk) n Zk = Standardabweichung (niedrigste 2% Intensitäten) Int(x,y) = max Int(x,y) , 0.5 Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Perfect match und Mismatch (PM MM) ... ... PM MM Definitionen: a = / (1+(( -Sbi)/ ’))) Idealer Mismatch: IM i,j = MM i,j MM i,j < PM i,j PM i,j / 2 Sbi MM i,j > PM i,j ; Sbi > PM i,j / 2 a MM i,j > PM i,j ; Sbi <= = 0.03 ’ = 10 Sbi = biweight specific background Vorverarbeitung von Microarray Daten: Beispiel 1: MAS 5.0 Perfect match und Mismatch (PM MM) PM MM ... ... Definitionen: Neuer Signalwert (Intensität): i = 1,…,n j = 1,…,m probe pair array probe set V i,j = max(PM i,j - IM i,j , ) = 2 –20 PV i,j = log(V i,j) für alle j Neuer Signalwert = Tbi (PV i1, … , PV in ) Vorverarbeitung von Microarray Daten: Beispiel 2: Li/Wong Li/Wong (PNAS 2001 vol 98 (1), pp31-36) Modell: MMij = j + i j + PMij = j + i j + i j + j i j j Baseline Expression eines Gens in der i ten Probe Anstiegsrate: MM im j ten “probe pair” zuätzliche Anstiegsrate im korrespondierenden PM Wert Zufälliger Fehler Vorverarbeitung von Microarray Daten: Beispiel 2: Li/Wong Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) 7 6 5 4 3 2 1 0 0,00 0,50 1,00 1,50 2,00 2,50 Vorverarbeitung von Microarray Daten: Beispiel 2: Li/Wong Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) 7 6 5 4 Summe der Fehlerquadrate ist minimal 3 2 1 0 0,00 0,50 1,00 1,50 2,00 2,50 Vorverarbeitung von Microarray Daten: Beispiel 2: Li/Wong Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) 4,5 4 3,5 3 2,5 2 1,5 1 0,5 0 0,00 X=(1,2,3,4,5) Y=(1,1,2,2,4) 2,00 4,00 6,00 Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) 4,5 4 3,5 3 2,5 2 1,5 1 0,5 0 0,00 y = 0,7x - 0,1 X=(1,2,3,4,5); Y=(1,1,2,2,4) 2,00 4,00 6,00 Y= ß0+ ß1*x Es werden ß1 und ß0 so geschätzt, daß die Summe der Quadrate der Residuen minimal werden: Min Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) i xi yi 12 n X (xi) X i n i 1 1 2 … x1 x2 … y1 y2 … y1 12 n y22 Yi n i 1 … Y xi * yi x1y1 x2y2 … n n xn yn xi yi 1 n X Xi n i 1 1 n Y Yi n i 1 n 1 n SQxx X ( X i ) 2 n i 1 i 1 2 i n n 1 n SQxy X iYi ( X i )( Yi ) n i 1 i 1 i 1 n 1 SQxxyn2 X xnyn ( X i ) 2 n i 1 2 i 1 2 i (xi) n xi * yi n 1 n SQxy X iYi ( X i )( Yi ) n i 1 i 1 i 1 SQxy 1 SQxx 0 Y 1 X y 0 1 x Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) i xi yi (xi)2 xi * yi 1 2 3 4 5 1 2 3 4 5 1 1 2 2 4 1 4 9 16 25 1 2 6 8 20 xi = 15 yi = 10 (xi)2 = 55 xi * yi = 37 Vorab: Was ist “Least Square Fit” (= Methode kleinster Fehlerquadrate) Vorverarbeitung von Microarray Daten: Beispiel 2: Li/Wong Li/Wong: Modell: MMij = j + i j + PMij = j + i j + i j + => PMij - MMij = i j + ij Angenommen: ij ~ N(0,2) Least Square Fit von PMij - MMij = i j + ij Vorverarbeitung von Microarray Daten: Beispiel 3: RMA RMA: Irizarry/Bolstad/Speed (NAR, 2003 31(4), e15) Modellannahme: Signal PM = Hintergrund + Signal = hg + s = Optisches Rauschen + unspezifische Bindung + = Hintergrund Korrektur: B(PM) = E(s|PM) s ~ exponential hg ~ normal Vorverarbeitung von Microarray Daten: Beispiel 3: RMA RMA PM, MM: “Forget about MM” Grund: was immer da auch gemessen wird; momentan laesst sich das nicht sinnvoll in biologische Interpretationen fassen ev. kann man in der Zukunft die Hintergrundkorrektur etwas besser durchführen, indem man die MM-Werte benutzt. Vorverarbeitung von Microarray Daten: Beispiel 3: RMA RMA Summary Statistic: Yijn = jn + jn + ijn i=1,...,I j=1,...,J n=1,...,n (chips) (probes) (probe set) jn “probe affinity effect” jn “log scale expression level” ijn error iid N(0, 2) j j= 0 n -> median polish Vorverarbeitung von Microarray Daten: Beispiel 3: RMA RMA Was ist “Median Polish”: An eine Matrix M wird ein additives Modell gefittet: Konstante + Spalten + Zeilen. Im Algorithmus werden abwechselnd Zeilen- bzw Spalten Mediane entfernt und wird solange durchgefuehrt, bis die proportionale Reduktion in der Summe der absoluten Residuen kleiner epsilon ist oder bis zu einem Max von Iterationsschritten. Macht es etwas aus, welche Methode ich wähle? all spots bgMAS+Av Diff pm only Av Diff pm only Av Diff pm - mm “MAS 5.0” Li/Wong pm only Li/Wong pm-mm RMA Macht es etwas aus, welche Methode ich wähle? Reference distribution is normal for the log fold change from: Terry Speed, Summarizing and comparing GeneChip data Vergleich von mehreren Proben cDNA Arrays Oligonucleotide Arrays Vergleich von mehreren Proben Patient Patient CC Kontrolle G A CC G A CC G A Kontrolle CC G A Vergleich von mehreren Proben Affymetrix in MAS5.0: - nicht einzeln auswerten der Chips sondern direkter paarweiser Vergleich: q (q1,...qn) z ( z1,..., zn) qi PM MM Zi PM b “Balancing factors” Wilcoxon Ranksummen Test Software • Open source-open development software Projekt • seit 2001 • erste Bioconductor software release, May 2002 • R basiert • http://www.bioconductor.org Software library(affy) x = data.rma = ReadAffy(celfile.path="/project/gene_expression/spikein/") express ( x, subset = NULL , bg.correct = bg.correct.rma , pmcorrect.method="pmonly" , summary.stat = medianpolish , normalize=F , verbose = TRUE )