Mikrobielle Jäger und Parasiten - Naturwissenschaftliche Rundschau
Werbung

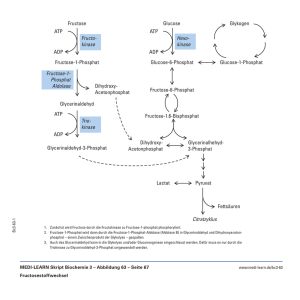
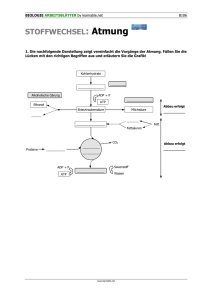
Forschung Stephan C. Schuster und Snjezana Rendulic, University Park, Pennsylvania, USA Mikrobielle Jäger und Parasiten Wie räuberische Bakterien Bakterien fressen Seit den frühen 1960er Jahren ist bekannt, dass Bakterien in der Lage sind, ihresgleichen als ­Nahrungsquelle zu nutzen. Zu diesem Zweck haben räuberische Bakterien unterschiedliche ­Strategien des Jagdverhaltens und des Jagdmechanismus entwickelt. Unter allen bisher untersuchten mikrobiellen Räubern nimmt das Bacterium Bdellovibrio bacteriovorus eine einzigartige Stellung ein. Einen Teil seines raffinierten Lebenszyklus verbringt es als Parasit in einem Wirtsbacterium. Obwohl B. bacteriovorus bereits vor 40 Jahren von dem deutschen Mikrobiologen Heinz Stolp entdeckt wurde, blieben viele Aspekte seines Lebenszyklus bis heute im Verborgenen. Durch eine im vergangenen Jahr veröffentlichte Genomanalyse, die den molekularen Bauplan ­dieses Organismus zugänglich machte, wurde dieses Forschungsgebiet neu belebt. Dieser Artikel gibt einen Überblick über die derzeit bekannten bakteriellen Räuber und ihre Strategien und fasst den derzeitigen Kenntnisstand der Genomanalyse zusammen. Zudem werden mögliche ­Anwendungen des bakteriellen Räubers bei der Bekämpfung von schädlichen Bakterien in der Umwelt sowie im medizinischen Bereich aufgezeigt. R äuber-Beute-Beziehungen sind auf allen Ebenen des Lebens beschrieben, in der makroskopischen und mikroskopischen Welt der Zoologie und der Mikrobiologie. Erstaunlicherweise ist wenig bekannt, dass auch bestimmte Bakterien selbst eine räuberische Lebensweise verfolgen [1]. Bisher ging man hauptsächlich davon aus, dass Bakterien nur als Nahrungsgrundlage für mikrobielle Räuber wie Amöben oder tierische Räuber wie Nematoden dienen. Angesichts der Tatsache, dass Bakterien den größten Teil der Biomasse dieses Planeten ausmachen, erscheint es mehr als unwahrscheinlich, dass Bakterien selbst diese außerordentlich große Nahrungsressource ungenutzt lassen. Nach heutigem Kenntnisstand ist es daher wenig überraschend, dass räuberische Bakterien in allen terrestrischen und aquatischen Biotopen gefunden werden, inklusive menschlicher und tierischer Därme [2]. Wolfsrudel oder Einzelkämpfer Ein Großteil des Stoffwechsels im Boden basiert auf der Zersetzung von organischem Material durch Bodenbakterien, die als Destruenten hochmolekulare Biopolymere zu organischen, niedermolekularen Substanzen zersetzen. Diese Bakterien können ihrerseits als Nahrungsgrundlage für andere räuberische Bodenbewohner wie Amöben und Nematoden dienen. Ähnlich wie Amöben haben auch Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005 bestimmte Bakterien, die Myxobakterien, gelernt, im Team zu jagen. Sie jagen im „Wolfsrudel“, d. h. sie schwärmen als Gemeinschaft aus und sondern dabei lytische Enzyme ab, die die Zellwand anderer Bakterien zersetzen und letztere abtöten [1]. Die freigesetzten Nährstoffe werden von den Myxobakterien aufgenommen, denen die lytischen Substanzen nichts anhaben. Für diese Art der Bejagung war es in der Evolution notwendig, eine soziale Interaktion mit Individuen derselben Art einzugehen, von der alle Beteiligten profitieren. Im Gegensatz zu diesem gemeinschaftlichen Vorgehen wurde für mehrere bakterielle Spezies ein direkter Angriff eines einzelnen Bacteriums auf ein anderes beschrieben. Vertreter hierfür gehören den Gattungen Vampirococcus, Daptobacter, Bacteriovorax oder Bdellovibrio an. Während die Vertreter der ersten beiden Gattungen vor allem in limnischen Systemen leben [3], kommen die Arten der anderen beiden Gattungen sowohl im Boden als auch in limnischen Systemen und in marinen Gewässern vor. Alle diese Vertreter unterscheiden sich grundlegend im Mechanismus ihres Angriffs und lassen sich den Kategorien Endo- und Ektoparasit zuordnen. Ektoparasiten wie Vampirococcus heften sich von außen an die Beute an und verweilen dort, während sie das Innere ihrer Beute aussaugen. Im Gegensatz hierzu durchdringen Endoparasiten wie Daptobacter beide 469 Forschung Bdellovibrio bacteriovorus Gram-negative Wirtszelle VIII. Freisetzung I. Beutelokalisierung freischwimmende Phase VII. Ausdifferenzierung Äußere Membran Periplasma Innere Membran Cytoplasma II. Anheftung Abb. 1. Der zweiphasige Lebenszyklus von Bdellovibrio bacteriovorus. Während der Angriffsphase ist Bdellovibrio hochmotil und kann chemotaktisch Beutekonzentrationen ausfindig machen. Hat es ein adäquates Beutebacterium aufgefunden, dringt es in dieses ein und geht in die Reproduktionsphase über. Diese findet im periplasmatischen Raum innerhalb der Wirtszelle statt, wobei die innere Membran, die das Cytoplasma abgrenzt, nicht durchbrochen wird. Reproduktionsphase III. Eindringen VI. Teilung V. Bdelloplast IV. Etablierung Membransysteme ihrer Beute, um nach dem Eindringen in die Wirtszelle ihre Wachstums- und Vermehrungsphase innerhalb des Cytoplasmas der Beute zu beginnen. Die Familie Bdellovibrionaceae nimmt eine Sonderstellung ein [1], da sie auf Grund ihres Jagdverhaltens und Lebenszyklus sowohl als Jäger als auch als Parasitoide klassifiziert werden können. Bdellovibrio kann mit „der zitternde Sauger“ übersetzt werden (von griech. bdallein, saugen und lat. vibrare, zittern), wobei der Namensteil -vibrio auf Ähnlichkeit in Form und Gestalt mit Bakterien der Gattung Vibrio zurückzuführen ist [4]. Zum Wirtsspektrum von ­Bdellovibrio gehören Gram-negative Bakterien [4]. Es durchdringt nur die äußere Membran seiner Beute, verbleibt aber außerhalb des Cytosols im sog. Periplasma der Zelle, wo es von den Ressourcen der Wirtszelle zehrt (Abb. 1). Reproduktion unter Ausschluss der Öffentlichkeit Bdellovibrio bacteriovorus als Modellorganismus der Familie der Bdellovibrionaceae durchläuft in seinem Lebenszyklus zwei Phasen (Abb. 1). In der ersten Phase schwimmt das Bacterium durch Geißelantrieb (Abb. 2a) mit den bislang schnellsten bei Bakterien gemessenen Geschwindigkeiten (> 100 µm/s [5]) und verwendet seine Chemosensoren, um hohe Beutekonzentrationen auszumachen. Nach Zusammentreffen mit einer Beutezelle überprüft der Räuber während einer ersten reversiblen Anheftung die Eignung des Objekts als Nahrungsquelle (Abb. 2b). Stellt sich die Beute als geeignet heraus, wird die Anheftung unumkehrbar und der Räuber beginnt, in das Periplasma der Wirtszelle einzudringen, indem er mit Hilfe eines lytischen Cocktails aus Proteasen, Peptidasen, Glycanasen und Lipasen eine Eintrittsöffnung in die äußere Membran schneidet. Durch diese Öffnung, die enger ist als das eindringende Bacterium selbst, zwängt sich Bdellovibrio durch die äußere Zellwand, nachdem es seine Geißel abgeworfen hat, und nistet sich im Periplasma ein. Im Anschluss an das Eindringen verändert sich die Gestalt des Wirtsbacteriums dahingehend, dass die stäbchenförmi­ 470 ge Form in eine sphärische übergeht. Man nimmt an, dass für diesen Prozess ein vom Räuber bedingter Umbau des Peptidoglycangerüsts, einer netzartigen Membranstruktur des Wirtsbacteriums, verantwortlich ist. Diese Veränderung verhindert gleichzeitig eine zusätzliche Infektion durch weitere Bdellovibrio-Zellen. Das Verhindern einer solchen Superinfektion dient der ausschließlichen Nutzung der Wirtsressourcen durch den erfolgreichen Räuber und stellt eine Analogie zum Befruchtungsprozess der Eizelle mit Spermium dar. Jetzt, da sich die parasitische Zelle ihrer Beute sicher sein kann, beginnt eine Umprogrammierung der Genexpression a. b. c. d. Abb. 2. Lebensstadien von Bdellovibrio. Nachkolorierte transmissionselektronenmikroskopische Aufnahmen einzelner Lebensstadien von Bdellovibrio. – a. Begeißelter Schwärmer auf der Suche nach Beutebakterien, – b. Anheften an eine potentielle Wirtszelle, – c. Abgerundete Wirtszelle mit aufgenommenem Bdellovibrio (sog. Bdelloplast), in der Bdellovibrio zunächst in die Länge wächst, um sich in mehrere Tochterzellen zu teilen, – d. Freisetzung von ausdifferenzierten Bdellovibrio-Zellen aus der Hülle des Wirtsbacteriums. Vergrößerung ca. 15500-fach, Balken 1µm. Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005 Schuster, Rendulic: Mikrobielle Jäger und Parasiten des Räubers und damit ein Übergang von der Angriffsphase in die Fortpflanzungsphase. Hierzu wird der Zellzyklusstop, der während der Angriffsphase besteht, aufgehoben. Obwohl es sich bei der Familie der Bdellovibrionaceae ebenso wie bei ihrer Beute um Gram-negative Bakterien handelt, weicht der Zellteilungsprozess von Bdellovibrio dramatisch von dem üblichen Teilungsprozess der Gramnegativen Bakterien ab. Zunächst wächst die eingedrungene Zelle durch Verlängerung um ein Vielfaches ihrer ursprünglichen Größe. In einem synchronen Prozess wächst diese filamentöse Zelle in dem Maße, wie das Cytoplasma der Beutezelle schrumpft. Die Zahl der Tochterzellen wird durch die Größe der Wirtszelle bestimmt, da der zur Verfügung stehende Platz innerhalb der Beutezelle durch das starre Peptidoglycangerüst begrenzt ist. Die Tochterzellen entstehen gleichzeitig durch einen Abschnürungsprozess aus der filamentösen Mutterzelle, wobei häufig eine ungerade Anzahl von Nachkommen beobachtet werden kann (Abb. 2c). Nun erfolgt zum zweiten Mal eine genetische Umprogrammierung des Parasiten, da alle Tochterzellen sich in Angriffszellen verwandeln müssen. Dieser Prozess ist beendet, wenn alle Tochterzellen über einen Motilitätspol mit einer Flagelle sowie über den notwendigen Angriffspol am gegenüberliegenden Ende verfügen. Der letzte Schritt für die Beendigung des Zellzyklus besteht in der Freisetzung der Tochterzellen aus dem Überbleibsel der Wirtszelle. Dieser Vorgang ­erinnert stark an die Freisetzung von Bakteriophagen, da nun die noch verbleibende Zellhülle des Wirts, bestehend aus äußerer Membran und Peptidoglycan, mit Hilfe von ­lytischen Enzymen aufgerissen wird. Die so freigesetzten Tochterzellen (Abb. 2d) sind augenblicklich in der Lage, weitere Wirtszellen aufzuspüren und zu befallen. Genomanalyse liefert Bauplan eines Organismus In den 40 Jahren seit der Entdeckung von Bdellovibrio konzentrierten sich die Forschungsvorhaben hauptsächlich auf mikrobiologische, morphologische und biochemische Aspekte dieses Organismus. Durch diese Arbeiten gelang es, den Lebenszyklus von Bdellovibrio darzustellen sowie Einzeluntersuchungen zu den Transportphänomenen durch­ zuführen, die bei der Aufnahme von Nährstoffen aus dem Wirt eine Rolle spielen. Erst in den letzten beiden Jahren gelang es, ein genetisches System zu etablieren, das Veränderungen im Erbgut des Bacteriums zulässt [6]. Im vergangenen Jahr konnte von unserer Forschergruppe erstmals die komplette Genomsequenz sowie eine Analyse derselben veröffentlicht werden [7]. Es zeigte sich, dass Bdellovibrio keine nähere Verwandtschaft zu den 150 Bakterien hat, deren Genome zu diesem Zeitpunkt bekannt waren, und dass durch Sequenzvergleiche lediglich 50% der vorhergesagten Gene eine Genfunktion zugeordnet werden konnte. Dies ist zum einen dadurch begründet, dass das Bdellovibrio-Genom das erste vollständig analysierte Genom der Klasse der räuberischen d-Proteobakterien ist, zum anderen dadurch, dass es keine Neigung zeigt, sich genetische Information aus der bio­ Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005 K a s te n : F u n kt i o n s v o r h e r s a g e v o n Ge n e n Genomsequenzierung Die gesamte genetische Information von Bdellovibrio bacteriovorus wurde durch Sequenzanalyse von Nucleinsäurefragmenten ermittelt. Dabei wurde jeder Bereich des Genoms durchschnittlich achtmal sequenziert. Durch überlappende Bereiche in den Sequenzen der sequenzierten Fragmente konnten diese zu einem ringförmigen Bakterienchromosom zusammengesetzt werden. Identifizierung von Genen Die Nucleinsäuresequenz wird durch bioinformatische Methoden analysiert. Anhand von bekannten Mustern und Genstrukturen werden so genannte offene Leserahmen definiert, das sind die ­Sequenzbereiche, die in die entsprechenden Aminosäuresequenzen der Genprodukte übersetzt werden. Da die Aminosäuren der Proteine durch Nucleotid-Tripletts codiert werden und die Nucleotidsequenz aus zwei Richtungen abgelesen werden kann, müssen dabei sechs mögliche Leserahmen berücksichtigt werden. Funktionsvorhersage Die vorhergesagten Gensequenzen werden mit bekannten Genen in Datenbanken verglichen, die entweder alle bisher bekannten Nucleinsäure- und Aminosäuresequenzen enthalten [genbank, http://www.ncbi.nlm.nih.gov/Genbank/] oder nur jene Sequenzen, deren Funktion durch experimentelle Untersuchungen nachgewiesen worden sind [swissprot, http://www.ebi.ac.uk/swissprot/]. Dabei werden auch Gene von Organismen, die phylogenetisch weit entfernt sind, berücksichtigt, da diese essentiell und somit nicht beliebig veränderbar sind. Die Analyse der ca. 200 bisher entschlüsselten Bakteriengenome zeigt, dass selbst nahe verwandte Bakterienstämme sich in bis zu 40% ihres Erbmaterials voneinander unterscheiden. Die Zahl der bisher unentdeckten Gene ist in Organismen ohne enge Verwandtschaft noch um ein Vielfaches höher und lässt den Anteil der uns unbekannten Gene des globalen mikrobiellen Genpools nur erahnen. Stoffwechselmodell Die Funktion der vorhergesagten Gensequenzen eines Genoms kann auf Karten der Stoffwechselwege eines Organismus im Computer projiziert werden. Die Zuordnung erfolgt dabei über einen Sequenzvergleich zu der Datenbank „Kyoto Encyclopedia of Genes and Genomes“ [KEGG, http://www.genome.jp/kegg/]. Aus der Summe aller Funktionsvorhersagen können Aussagen über das metabolische Potential eines Organismus gemacht werden und somit zumindest teilweise seine Physiologie abgeleitet werden. Wegen des Anteils an Genen mit unbekannter Funktion im Genom eines Organismus können komplementäre und bisher unbekannte Stoffwechselwege mit diesem Verfahren nicht ermittelt werden. tischen Umwelt oder seiner Beute anzueignen (horizontaler Gentransfer). Aus der Klassifizierung der vorhergesagten Gene lässt sich schließen, dass es sich um einen Organismus handelt, der obligatorisch auf Wirtsbakterien angewiesen ist, da nur wenige Gene für Stoffwechsel und Energiegewinnung vorhanden sind. Bdellovibrio besitzt die höchste jemals in einem Bacterium festgestellte Anzahl an Genen für hydrolytische Enzyme (Anzahl Gene/Megabasen), die sehr wahrscheinlich am Abbau der Wirtsbiopolymere beteiligt sind 471 Forschung Tab. 1. Gene, die für hydrolytische Enzyme codieren [7]. (Tab. 1). Diese ca. 290 verschiedenen Enzyme zerlegen Proteine, Polysaccharide, DNA, RNA sowie Lipide in ihre niedermolekularen Bestandteile. Interessanterweise spiegelt sich diese Ansammlung an degradierenden Enzymen in einer großen Anzahl an Energie verbrauchenden Transportern (ABC-Transportern) sowie diffusionskontrollierten Membrandurchgängen (Permeasen) wider (Abb. 3). Somit scheint klar zu sein, dass Bdellovibrio eine generelle Tendenz hat, vorhandene Biopolymere abzubauen, anstatt sie als Ganzes in seinem eigenen Stoffwechsel zu verwenden. Diese anhand der Genomanalyse gemachte Vorhersage stimmt gut mit experimentellen Beobachtungen der vergangenen 40 Jahre überein. Mit Hilfe bioinformatischer Programme konnte für Bdellovibrio ein Stoffwechselschema konstruiert werden, das viele experimentelle Befunde erklärt. So lässt sich feststellen, dass der sequenzierte Stamm Bdellovibrio bacteriovorus HD100 sehr wohl in der Lage ist, ihm zur Verfügung stehende Nährstoffe umzusetzen und mit der gewonnenen Energie ein Membranpotential aufzubauen, das alle physiologisch wichtigen Vorgänge der Zelle betreibt. Jedoch ist dieser Bdellovibrio-Stamm nicht in der Lage, sich außerhalb seines Wirts zu reproduzieren. Der Stoffwechsel dieses Bacteriums enthält alle notwendigen Enzyme zur Durchführung von Glycolyse, oxidativer Phosphorylierung, Fettsäureabbau sowie einen kompletten Citratzyklus (Abb. 3). Ebenso ist es in der Lage, seine eigenen Bausteine zur DNA-Synthese zu produzieren. Eine wesentliche Erkenntnis der Genomanalyse besteht darin, dass Bdellovibrio nur elf der 20 essentiellen Aminosäuren selbst synthetisieren und nur zehn derselben abbauen kann. Dem räuberischen Bacterium stehen also neun essentielle Aminosäuren nur mittels Abbau von Wirts- Peptidexporter Art des hydrolytischen Enzyms Lipasen Glycasen Proteasen und Peptidasen DNasen RNasen andere Hydrolasen proteinen und Transport aus dem Wirtsbacterium zur Verfügung. Hieraus erklärt sich zugleich die große Anzahl an ABC-Transportsystemen und Permeasen. Ebenso fehlen Bdellovibrio alle benötigten Enzyme zur Speicherung von organischem Kohlenstoff. In diesem Zusammenhang ist zu erwähnen, dass das Bacterium in der Lage ist, Glycolyse durchzuführen, jedoch weder die Neusynthese von Glucose (Gluconeogenese) noch den Aufbau von polymeren Zuckermolekülen betreibt. Da Bdellovibrio nicht in der Lage ist, sich außerhalb seines Wirtes zu vermehren, jedoch durch die Verstoffwechselung von Nährstoffen aus seiner Umwelt Energie zu gewinnen vermag, liegt der Schluss nahe, dass anhand von Bdellovibrio die Lebensdauer einer Bakterienzelle untersucht werden kann, ohne dass eine Verjüngung der Kultur durch Zellteilung auftritt. Diesbezüglich wurde beobachtet, dass Bdellovibrio nicht wie viele seiner Artverwandten in eine stationäre Phase mit reduziertem Wachstum eintritt, sondern über einen längeren Zeitraum metabolisch aktiv bleibt, ein Sachverhalt, der durch die anhaltend hohe Motilität dokumentiert ist. Peptidimporter Peptidpermease Aminosäurenimporter Aminosäurenpermeasen Kohlenhydrattransporter und -permeasen Polyamintransporter Zuckertransporter Arginin Cl - H+ 2+ Ca Na+ K+ K+ Ionentransporter und -permeasen K+ ADP ATP ADP Na+ cat i on Cl - ADP H+ ATP ATP AD P P AD Na+ Mg2+ Co2+ Cl - Mg2+ Co2+ P AD P AT ADPTP A ADP ATP AD P ATP AD AT P P AD AT P P ADP AT P Ornithin Succinyl-CoA Phosphattransporter Multidrug Resistenz Exporter Glutamat 2-Oxoglutarat Succinat PO34- Fettsäuresynthese A AT DP P AT P Ethanol TTP TMP vorhergesagte Transporterkomponenten ATP Orotat Prolin P AD ATP Acetaldehyd H+ P AD P AT H+ Isocitrat Fumarat Acetyl-CoA CO2 Malonyl-CoA CO2 Acetat H+ Glutamin Citratzyklus H H+ UMP vorhergesagte Permeasenkomponenten ATP ADP Citrat Malat Pyruvat Isoleucin + Nukleotidsynthese UTP ATP α-Ketoisovalerate Leucin GTP Acetyl-CoA Oxaloacetat Phosphoenolpyruvat ? P AD P AT Valin CTP 2-P-Glycerat Cystein ADP GMP Asparagin Aspartat Alanin ADP P AD ATP CMP 3-P-Glycerat Mg2+ Co2+ Threonin ATP ATP Lysin Aspartat semialdehyd Serin AMP IM Methionin 1,3-Di-P-Glycerat Glycin Phosphoribosyl Pyrophosphat ATP AT P AD AT P P P AD Histidin Homoserin AD P Sonstige Transporter P AT Glyceraldehyd-3-P Glycolyse 3x 472 AT P Fe2+ Zn2+ Tryptophan Tyrosin Phenylalanin Ribulose-5-P Gluconeogenese 2x ATP Xylulose-5-P DNAimporter AD P Glucose-1-P Fructose-1,6-di-P Fe2+ Fe2+ Zn2+ Multidrug Effluxpumpen Ribose-5-P Glucose-6-P Fructose-6-P Fe2+ ATP Sedoheptulose-7-P TonB/ExbD/ExbB 16 Proteine Nukleotidpermeasen Chorismat Pentosephosphatweg Glucose Eisentransporter ADP ATP ADP ATP ADP ATP ADP ADP ATP ADP ATP ATP ATP ADP ADP ADP ATP Shikimat Erythrose-4-P Na + ADP TEM Anzahl der vorhergesagten Gene aus dieser Grupppe 15 10 150 20 9 89 Co2+ Zn2+ Co2+ Cd2+ Zn2+ Cd2+ Ag2+ AD P Ni2+ CrO24 Metalltransporter und -permeasen Abb. 3. Schematische Darstellung der vorhergesagten Stoffwechselwege von Bdellovibrio bacteriovorus. Die Stoffwechselwege wurden durch Sequenzvergleich der vorhergesagten Gene von B. bacteriovorus mit bekannten Genen, die für Enzyme codieren, zusammengestellt. Die Biosynthesewege der essentiellen Aminosäuren, die B. bacteriovorus selbst herstellen kann, sind schwarz dargestellt, nicht vollständige Synthesewege rot. Die dazugehörigen Degradationswege sind durch Unterstreichung der jeweiligen Aminosäure gekennzeichnet: schwarz bei kompletter Enzymausstattung und rot bei unvollständigem Degradationsweg. Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005 Schuster, Rendulic: Mikrobielle Jäger und Parasiten Wie oben gezeigt, kann das wesentliche Ergebnis einer Genomanalyse nicht nur im Auffinden spezifischer Gene und ihrer Genprodukte, sondern vor allem im Nachweis ihrer Abwesenheit bestehen. Diese Feststellung ist insofern besonders wichtig, als im Vorhandensein eines Gens keine Garantie für die Expression und Nutzung des Genproduktes gegeben ist. Im Gegensatz hierzu kann bei Fehlen eines Gens die physiologische Befähigung, die durch das Genprodukt vermittelt würde, ausgeschlossen werden. In dieser Hinsicht ist beispielsweise das Fehlen der beiden Protein­ exportsysteme Typ III und Typ IV aufschlussreich. Diese Proteinexportsysteme finden sich in all jenen Bakterien, die ihre eigenen Proteine und Enzyme in Wirtszellen (v. a. Säugetierzellen) einschleusen, um ihre Aufnahme in die jeweilige Zelle zu erleichtern. Bdellovibrio scheint diese Befähigung zu fehlen, eine Tatsache, die einen experimentellen Befund aus den 1970er Jahren erklärt, bei dem es nicht gelungen war, Kanincheneizellen und Erythrocyten mit Bdellovibrio zu infizieren [8]. Es ist daher nach jetzigem Kenntnisstand auszuschließen, dass Bdellovibrio in der Lage ist, eine tierische Zelle zu infizieren. Diese Tatsache zusammen mit der Beobachtung, dass Bdellovibrio über besonders schwach immunogene Lipide verfügt [9], lässt es möglich erscheinen, Bdellovibrio im tierischen Organismus zur Infektionsbekämpfung einzusetzen, ohne dabei einen anaphylaktischen Schock auszulösen. Bdellovibrio als lebendes Antibiotikum? Die Summe der oben genannten Eigenschaften, die mittels der Genomanalyse sowie experimenteller Befunde festgestellt wurden, legt den Schluss nahe, dass Bdellovibrio ein geeignetes Agens sein könnte, um große Ansammlungen von Gram-negativen Bakterien zu bekämpfen. Inzwischen sind viele verschiedene Isolate von Bdellovibrio bekannt, manche davon mit einem sehr breiten Wirtsspektrum, während andere Stämme auf eine einzige Bakterienart als Wirt spezialisiert sind [10]. Praktische Anwendungen sind daher sowohl im ökologischen als auch im medizinischen Bereich vorstellbar. Im ökologischen Bereich eignet sich Bdellovibrio beispielsweise für den Einsatz bei der Abwasserreinigung in der biologischen Reinigungsstufe, im Pflanzenschutz, sowie generell gegen Biofilme, die auf nahezu allen Oberflächen in der Umwelt zu finden sind. Um Rückstände aus Klärwerken (Klärschlamm) zu desinfizieren, die eine völlig unbekannte Zusammensetzung von Mikroorganismen enthalten, wären Bdellovibrio-Stämme mit breitem Wirtsspektrum geeignet. Im Bereich des Pflanzenschutzes fallen zwei wichtige Krankheitserreger in das Beutespektrum von Bdellovibrio: Pseudomonas glycinea, der Erreger von Mehltau, sowie Erwinia amylovora, der Erreger von Feuerbrand. Besonders bei dem Erreger des Feuerbrandes, der ein zunehmendes Problem im Bereich des Obstbaus darstellt, erscheint der großflächige Einsatz von Antibiotika aus Umweltschutzgründen bedenklich. Da Bdellovibrio weltweit in allen bisher untersuchten Böden nachgewiesen werden konnte, scheint es Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005 Bestandteil der natürlichen Nahrungskette im Boden zu sein. Sein Einsatz im landwirtschaftlichen Bereich in Form eines wässrigen Aerosols könnte demnach eine umweltverträgliche Maßnahme gegen die genannten Erreger darstellen. Biofilme sind komplexe mikrobielle Gemeinschaften, die sich aus einer definierten Ansammlung von verschiedenen bakteriellen Spezies zusammensetzen. Diese Gemeinschaften produzieren eine zuckerhaltige Matrix, die die Diffusion von bestimmten Substanzen verhindern kann und die Bekämpfungsmöglichkeiten mittels Antibiotika einschränkt. Interessanterweise scheint Bdellovibrio in diese komplexen Biofilmstrukturen ebenso eindringen zu können wie in Bakterien, die sich mittels einer sehr umfassenden Oberflächenzuckerstruktur vor Bakteriophagen und anderen negativen Umwelteinflüssen schützen können [11]. Es ist daher offensichtlich, dass der Angriff von Bdellovibrio auf seine Beute einen äußerst robusten Mechanismus darstellt, der nicht durch kurzfristige Änderungen (Mutationen) in der genetischen Ausstattung der Wirtszelle zu einer Resistenz gegen den Fressfeind führt. Obwohl die Wirtsbakterien in der Lage sind, sich den ständigen Änderungen in der Umwelt anzupassen, ist es ihnen offensichtlich nicht gelungen, sich dem Befall von Bdellovibrio zu entziehen. Dies ist umso erstaunlicher, als bei der „interbakteriellen Kriegsführung“ ein ganzes Arsenal an chemischen Substanzen zum Einsatz kommt, von denen wir viele in Form von Antibiotika für unsere Zwecke einsetzen. Die Neigung der Wirtsbakterien, sich Resistenzen gegen Antibiotika anzueignen, stellt jedoch in keinem Fall einen Schutz gegen die Bejagung durch Bdellovibrio dar. Durch die Einzeluntersuchungen der Wirkungsweise der hydrolytischen Enzyme von Bdellovibrio können möglicherweise neue Klassen von peptidbasierten antimikrobiellen Substanzen charakterisiert werden, die in neuen Pharmazeutika resultieren könnten. Diese rekombinanten Polypeptide könnten wie die natürlichen Hydrolasen wirken. Außerdem von Bedeutung für den medizinischen Einsatzbereich ist die Tatsache, dass die in der Genomanalyse identifizierten Enzyme an zellulären Mechanismen der Wirtszelle ansetzen, die sich von jenen wenigen unterscheiden, gegen die heute eingesetzte Antibiotika wirken. Daher lassen sich durch das Studium von Bdellovibrio neue Angriffspunkte für weitere Pharmazeutika ausmachen. Denkbar erscheint auch der gezielte Einsatz von Bdellovibrio als lebender Organismus, da sein Wirtsspektrum eine Vielzahl von medizinisch relevanten Pathogenen umfasst. Beispiel hierfür sind Legionellen, die die Legionärskrankheit verursachen, Salmonellen, die Erreger bakteriellen Durchfalls, sowie die uropathogenen Escherichia coli. Weitere Anwendungen könnten die Behandlung von Patienten mit Cystischer Fibrose umfassen, die unter einer Infektion der Lunge mit Burkholderia- und PseudomonasBakterien leiden. Ebenso erscheint der Einsatz im Harntrakt von Patienten möglich, die mit dem Bacterium Proteus infiziert sind. Im chirurgischen Bereich lässt sich an eine 473 Forschung Anwendung denken, um chirurgische Implantate frei von Biofilmen zu halten, die beispielsweise Katheter verstopfen und häufig zusätzliche Eingriffe nötig machen. Während eine systemische Anwendung verwegen er­scheinen mag, da eine außerordentlich große Anzahl von Faktoren berücksichtigt werden muss, erscheint derzeit eine äußere Anwendung vertretbar. Ein aussichtsreiches Beispiel hierfür sind Studien, die im Labor der Autoren begonnen wurden, um Infektionen von Brandwunden zu bekämpfen, die durch das Bacterium Pseudomonas aeruginosa ver­ ursacht werden. Zukunft der Genomanalyse Am Beispiel des räuberischen Bacteriums Bdellovibrio bacteriovorus lässt sich zeigen, welche umfassenden Möglichkeiten die Aufklärung und Analyse kompletter Genomsequenzen von Bakterien bieten. Erst durch diese Technik ist es gelungen, die noch vor zehn Jahren sehr geringe Breite an mikrobiellen Modellorganismen dramatisch zu erweitern. Somit lassen sich biologische Systeme, denen bisher nur ein Nischendasein im wissenschaftlichen Umfeld zugestanden wurde, auf Augenhöhe mit etablierten Systemen wie E. coli bringen. Viele molekularbiologische Methoden können erst mithilfe vollständig analysierter Genomsequenzen der zu untersuchenden Organismen zur Anwendung kommen. Gleichzeitig erlaubt dieser Forschungsansatz, testbare Hypothesen zu formulieren und gezielt nur die erfolgversprechendsten experimentellen Studien durchzuführen. 474 Literatur: [1] M. O. Martin, J. Mol. Microbiol. Biotechnol. 4, 467 (2002). – [2] L. Hu, B.Y. Liu, Chinese J. Microbiol. and Immunol. 10, 95 (1990). – [3] R. Guerrero et al., Proc. Natl. Acad. Sci. USA 83, 2138 (1986). – [4] H. Stolp, M. P. Starr, Antonie van Leeuwenhoek J. Microbiol. Serol. 29, 217 (1963). – [5] M. P. Starr, R. J. Seidler, Ann. Rev. Microbiol. 25, 649 (1971). – [6] C. Lambert et al., Environ. Microbiol. 5, 127 (2003). – [7] S. Rendulic et al., Science 303, 689 (2004). – [8] R. W. Lenz, R. B. Hespell, Arch. Microbiol. 119, 245 (1978). – [9] D. Schwudke et al., J. Biol. Chem. 278, 27502 (2003). – [10] E. Jurkevitch et al., Appl. Environ. Microbiol. 66, 2365 (2000). – [11] S. F. Koval, M. E. Bayer, Microbiology 143, 749 (1997). Prof. Dr. Stephan C. Schuster studierte Chemie (Diplom) in München und Konstanz. Promotion am Max-Planck-Institut für Biochemie, Martinsried, mit einer Arbeit über den bakteriellen Flagellenmotor. Arbeiten zur bakteriellen Motilität und Chemotaxis als Postdoktorand am California Institute of Technology, Pasadena, USA. Forschungsgruppenleiter am Max-Planck-Institut für Biochemie, Martinsried (1994–2000) und am Max-Planck-Institut für Entwicklungsbiologie, Tübingen, (2000–2004). Seit 2005 Associate Professor for Molecular Microbial Ecology und Mitglied des Center for Comparative Genomics and Bioinformatics an der Pennsylvania State University (USA). Fachgutacher für Genprojekte und Editor von Fachzeitschriften. Department of Biochemistry and Molecular Biology, Center for Comparative Genomics and Bioinformatics, 310 Wartik Laboratory, Penn State University, University Park, PA 16802, USA Dipl.-Biol. Snjezana Rendulic studierte Mathematik und Biologie in Tübingen und Zagreb. Doktorandin in der Arbeitsgruppe von Prof. Dr. Stephan C. Schuster am Max-Planck-Institut für Entwicklungsbiologie in Tübingen und zurzeit an der Penn State University in Pennsylvania (USA). Department of Biochemistry and Molecular Biology, Center for Comparative Genomics and Bioinformatics, 310 Wartik Laboratory, Penn State University, University Park, PA 16802, USA Naturwissenschaftliche Rundschau | 58. Jahrgang, Heft 9, 2005