Beeinflusst die Darmflora Gesundheit und Krankheit
Werbung
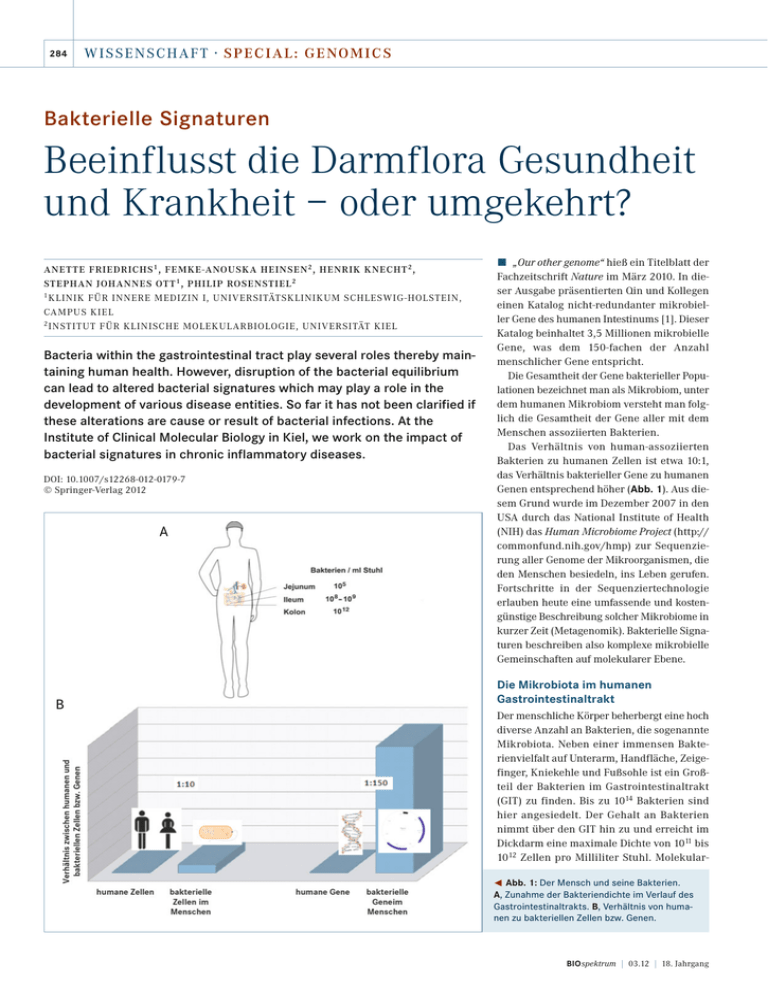
284 W I S S ENS CHAFT · S PECIA L : G ENO MI CS Bakterielle Signaturen Beeinflusst die Darmflora Gesundheit und Krankheit – oder umgekehrt? ANETTE FRIEDRICHS 1 , FEMKE-ANOUSKA HEINSEN 2 , HENRIK KNECHT 2 , STEPHAN JOHANNES OTT 1 , PHILIP ROSENSTIEL 2 1 KLINIK FÜR INNERE MEDIZIN I, UNIVERSITÄTSKLINIKUM SCHLESWIG-HOLSTEIN, CAMPUS KIEL 2 INSTITUT FÜR KLINISCHE MOLEKULARBIOLOGIE, UNIVERSITÄT KIEL Bacteria within the gastrointestinal tract play several roles thereby maintaining human health. However, disruption of the bacterial equilibrium can lead to altered bacterial signatures which may play a role in the development of various disease entities. So far it has not been clarified if these alterations are cause or result of bacterial infections. At the Institute of Clinical Molecular Biology in Kiel, we work on the impact of bacterial signatures in chronic inflammatory diseases. DOI: 10.1007/s12268-012-0179-7 © Springer-Verlag 2012 A B ó „Our other genome“ hieß ein Titelblatt der Fachzeitschrift Nature im März 2010. In dieser Ausgabe präsentierten Qin und Kollegen einen Katalog nicht-redundanter mikrobieller Gene des humanen Intestinums [1]. Dieser Katalog beinhaltet 3,5 Millionen mikrobielle Gene, was dem 150-fachen der Anzahl menschlicher Gene entspricht. Die Gesamtheit der Gene bakterieller Populationen bezeichnet man als Mikrobiom, unter dem humanen Mikrobiom versteht man folglich die Gesamtheit der Gene aller mit dem Menschen assoziierten Bakterien. Das Verhältnis von human-assoziierten Bakterien zu humanen Zellen ist etwa 10:1, das Verhältnis bakterieller Gene zu humanen Genen entsprechend höher (Abb. 1). Aus diesem Grund wurde im Dezember 2007 in den USA durch das National Institute of Health (NIH) das Human Microbiome Project (http:// commonfund.nih.gov/hmp) zur Sequenzierung aller Genome der Mikroorganismen, die den Menschen besiedeln, ins Leben gerufen. Fortschritte in der Sequenziertechnologie erlauben heute eine umfassende und kostengünstige Beschreibung solcher Mikrobiome in kurzer Zeit (Metagenomik). Bakterielle Signaturen beschreiben also komplexe mikrobielle Gemeinschaften auf molekularer Ebene. Die Mikrobiota im humanen Gastrointestinaltrakt Der menschliche Körper beherbergt eine hoch diverse Anzahl an Bakterien, die sogenannte Mikrobiota. Neben einer immensen Bakterienvielfalt auf Unterarm, Handfläche, Zeigefinger, Kniekehle und Fußsohle ist ein Großteil der Bakterien im Gastrointestinaltrakt (GIT) zu finden. Bis zu 1014 Bakterien sind hier angesiedelt. Der Gehalt an Bakterien nimmt über den GIT hin zu und erreicht im Dickdarm eine maximale Dichte von 1011 bis 1012 Zellen pro Milliliter Stuhl. Molekular¯ Abb. 1: Der Mensch und seine Bakterien. A, Zunahme der Bakteriendichte im Verlauf des Gastrointestinaltrakts. B, Verhältnis von humanen zu bakteriellen Zellen bzw. Genen. BIOspektrum | 03.12 | 18. Jahrgang 285 biologisch werden die Bakterien in 22 verschiedene Phyla eingeteilt, wobei die vier im GIT dominierenden einen Anteil von über 92 Prozent der Mikrobiota des menschlichen GITs darstellen. In absteigender Dominanz sind dies die Firmikuten, Bakteroideten, Proteobakterien und die Aktinobakterien. Die vorhandenen Phyla können in 800 bis 1.000 verschiedene Spezies und mehr als 7.000 verschiedene Stämme untergliedert werden. Innerhalb des GITs kann zwischen der luminalen und der Mukosa-assoziierten Mikrobiota unterschieden werden. Die Mukosa-assoziierte Mikrobiota, die durch Biopsieproben gewonnen wird, hat eine Zusammensetzung, die sich über den gesamten Dickdarm (aufsteigendes, transverses, absteigendes und sigmoidales Kolon) sehr ähnelt, sich aber von der luminalen Mikrobiota, welche durch Stuhlproben erfasst werden kann, unterscheidet. Die einzelnen Aufgaben der (hauptsächlich anaeroben) Bakterien sind vielfältig. Zusammengenommen stimulieren sie die Proliferation, Angiogenese und die epitheliale Restitution des GITs, sodass die Mikrobiota an der gastrointestinalen Homöostase und damit letztendlich am Erhalt der Gesundheit beteiligt ist. Eine Störung dieser Homöostase kann zu Veränderungen der Mikrobiota führen, die wiederum in der Pathogenese von einer Vielzahl entzündlicher Darmerkrankungen wie Colitis ulcerosa und Morbus Crohn implementiert sind. Unser Wissen über die Mechanismen, die zu Verschiebungen in der Zusammensetzung des Mikrobioms führen, ist jedoch noch begrenzt. Eine abnormale Veränderung der gastrointestinalen Mikrobiota sowie das Wechselspiel zwischen dem aktiven und inaktiven (dormanten) Zustand bakterieller Stämme müssen entgegen früherer Annahmen nicht zwangsläufig der Grund, sondern können auch das Resultat einer bakteriellen Infektion sein. Es konnte mehrfach gezeigt werden, dass sowohl chronisch-entzündliche Darmerkrankungen als auch die Veränderung der entsprechenden Mikrobiota durch den Genotyp des Menschen, die Ernährungsweise und andere exogene Umweltfaktoren ausgelöst bzw. beeinflusst werden [2]. Vom klinischen Probenmaterial zur Bakteriensignatur Die molekularbiologische Analyse der komplexen gastrointestinalen Mikrobiota im Zusammenhang mit entzündlichen Darmerkrankungen sowie die Identifizierung infektionsbeeinflussender bakterieller Familien und Gattungen steht daher im Mittelpunkt unserer Arbeit. Da 70 bis 90 Prozent der Bakterien unkultivierbar sind, werden Kulturunabhängige Verfahren wie die Hochdurchsatz-Sequenzierung des 16S-rRNA-Gens durchgeführt. Das 16S-rRNA-Gen codiert für eine 16SrRNA, die in der kleinen Untereinheit des bakteriellen Ribosoms zu finden ist. Es wird in neun variable und zehn konservierte Bereiche gegliedert. Primer binden an konservierte Regionen und umspannen die variablen Regionen, anhand derer bakterielle Organismen unterschieden werden können [5]. Als Probenmaterial werden im Institut für Klinische Molekularbiologie (IKMB) Stuhlproben und gastrointestinale Biopsien, die mittels Endoskopie gewonnen werden, verwendet. Aus diesen Proben wird zunächst die gesamte genomische DNA und/oder RNA extrahiert, wobei die RNA in einem zweiten Schritt mittels reverser Transkription in cDNA umgeschrieben wird. Die umgeschriebene RNA gibt später einen Hinweis auf die Zusammensetzung der aktiven Bakterien, während die DNA die der präsenten Bakterien beschreibt. Ausgehend von der DNA bzw. cDNA wird in einer PCR-Reaktion mittels Barcode-codierter Primer ein etwa 420 Basenpaare großes PCR-Fragment amplifiziert, welches die variablen Regionen 1 und 2 des 16S-rRNA-Gens umspannt (Abb. 2). Nach BIOspektrum | 03.12 | 18. Jahrgang ˚ Abb. 2: Vom Bakterium zur Bakteriensignatur. Jedes Bakterium enthält Ribosomen und in diesen 16S-rRNA. Das codierende 16S-rRNAGen besteht aus konservierten sowie variablen Regionen [5], anhand derer zwischen den unterschiedlichen Bakterien differenziert werden kann. einer Überprüfung der korrekten Größe des Amplifikats durch eine Gelelektrophorese wird die entsprechende Gelbande extrahiert und mittels FLX-Hochdurchsatz-Technologie sequenziert. Aufgrund der Barcode-codierten Primer können mehrere Amplifikate parallel sequenziert werden. Die resultierenden Sequenzen werden anhand ihrer Barcodes den entsprechenden Proben zugeordnet und zunächst gereinigt. Sequenzen, die eine bestimmte Länge unterschreiten, die Anzahl an Homopolymeren oder mehrdeutigen Basen überschreiten oder Chimäre sind, werden eliminiert. Über die Berechnung von Dis- 286 W I S S ENS CHAFT · S PECIA L : G ENO MI CS A B Aufrechterhaltung einer gesunden Flora und Unterdrückung pathogener Stämme denkbar. ó Literatur ˚ Abb. 3: Einfluss von Breitbandantibiotika auf die Mikrobiota des humanen Gastrointestinaltrakts (ermittelt in 15 Patienten). A, Mikrobiota vor der Antibiotikatherapie. B, Mikrobiota während der Antibiotikatherapie. tanzmatrizen werden die gereinigten Sequenzen in sogenannten operational taxonomic units (OTUs) gruppiert. Anhand der Sequenzverteilung zwischen den OTUs können Rückschlüsse auf das bakterielle Profil einer Probe und letztendlich auf Bakteriensignaturen von verschiedenen Probenarten (z. B. Kontrolle/Erkrankung) gezogen werden. Krankheitsbezogene Bakteriensignaturen Unsere Arbeitsgruppe konnte zeigen, dass das Protein NOD2/CAD15 eine essenzielle Rolle bei der Entwicklung der intestinalen Mikrobiota und ihrer Veränderung über die Zeit spielt [3]. In einer weiteren Studie am IKMB wurde deutlich, dass Patienten mit Morbus Crohn und Colitis ulcerosa im Vergleich zu gesunden Menschen eine geringere bakterielle Vielfalt im GIT besitzen [4]. Derzeitig beschäftigen uns die Veränderung des gastrointestinalen Mikrobioms durch den Einsatz von Breitbandantibiotika und der Bezug zu darauffolgenden Entwicklungen von Infektionen wie beispielsweise der Antibiotika-assoziierten Diarrhoe. Breitspektrumantibiotika bewirken eine Reduktion von Firmikuten und eine Zunahme von Bakteroideten im GIT. Dies schließt bei den Firmikuten insbesondere die Familien der Ruminoccoaceae (hier insbesondere die Gattung Faecalibacterium) und Clostridiaceae sowie die Bacteroidaceae der Bakteroideten ein (Abb. 3). Einige Patienten entwickeln unter antibiotischer Behandlung eine Antibiotika-assoziierte Diarrhoe. Sowohl die Zusammensetzung der Mikrobiota als auch potenziell vorhandene toxinbildende Stämme von Clostridium difficile können dafür ursächlich sein. Basierend auf der FLX-Hochdurchsatz-Technologie konnten wir zeigen, dass eine charakteristische Reduzierung der Mikrobiota zum Ausbruch der Antibiotika-assoziierten Diarrhoe führen kann. Ausblick Die Aufklärung des Zusammenspiels zwischen der Mikrobiota und dem Gesundheitszustand des Menschen wird auch in den nächsten Jahren eine wichtige Rolle spielen. Noch gibt es zu wenige Informationen, wie und warum die Zusammensetzung der Mikrobiota einerseits krankmachend und andererseits gesundheitsstabilisierend wirken kann. Die Identifizierung krankheitsbeeinflussender, aber auch gesundheitsstabilisierender Kompositionen der Mikrobiota und deren Rolle bei Entzündungsprozessen im Darm besitzt eine enorme klinische Relevanz. Basierend auf erworbenen Kenntnissen ist unsere Arbeitsgruppe daran interessiert, neue Biomarker zu finden und gezielt Präventivstrategien zu entwickeln. Dabei ist der therapeutische Einsatz von bakteriellen Stämmen zur [1] Qin j, Li R, Raes J et al. (2010) A human gut microbial gene catalogue established by metagenomic sequencing. Nature 464:59–67 [2] McGovern DP, Gardet A, Törkvist L et al. (2010) Genome wide association identifies multiple ulcerative colitis susceptibility loci. Nat Genet 42:332–337 [3] Rehman A, Sina C, Gavrilova O et al. (2011) Nod2 is essential for temporal development of intestinal microbial communities. Gut 60:1354–1362 [4] Ott SJ, Schreiber S (2006) Reduced microbial diversity in inflammatory bowel disease. Gut 55:1207 [5] Baker GC, Smith JJ, Cowan DA (2003) Review and re-analysis of domain specific 16 S primers. J Microbiol Methods 55:541–555 Korrespondenzadresse: Dr. med. Anette Friedrichs Klinik für Innere Medizin I Uniklinikum Schleswig-Holstein, Campus Kiel Schittenhelmstraße 12 D-24105 Kiel Tel.: 0431-597-1393 Fax: 0431-597-1842 [email protected] www.uksh.de www.ikmb.uni-kiel.de AUTOREN Femke-Anouska Heinsen, Henrik Knecht, Anette Friedrichs, Stephan Johannes Ott und Philip Rosenstiel (v. l. n. r.) Die Arbeitsgruppe „Bakterielle Metagenomik“ wurde von PD Dr. Stephan Ott gegründet und existiert seit 2001 am Institut für Klinische Molekularbiologie, Universität Kiel. In den letzten Jahren konnten entscheidende Arbeiten zu bakteriellen Signaturen insbesondere bei entzündlichen Darmerkrankungen, aber auch koronarer Atherosklerose und Periimplantitis veröffentlicht werden. BIOspektrum | 03.12 | 18. Jahrgang