HLA Fusion™ Version 3.0
Werbung

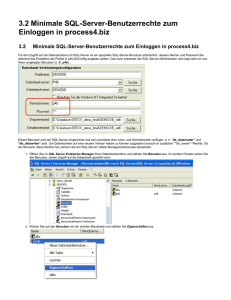
SOFTWARE VERSIONSHINWEISE HLA Fusion™ Version 3.0 FUSPGR Nur zur In-vitro-Diagnostik. ZIELGRUPPE ® ® ® Alle Benutzer von HLA Fusion™. Diese Software kann für LABScreen , LAT™, LCT™, FlowPRA , LABType und Micro SSP™ verwendet werden. INHALT DER VERSION Das HLA Fusion™ 3.0-Paket setzt sich aus den folgenden Komponenten zusammen: 1. HLA Fusion™ 3.0-CD 2. HLA Fusion™ 3.0-Benutzerhandbuch 3. HLA Fusion™ 3.0-Installationshandbuch 4. HLA Fusion™ 3.0-Versionshinweise 5. HLA Fusion™ 3.0 Database Utility-Benutzerhandbuch MINDESTVORAUSSETZUNGEN – SOFTWARE • Eines der folgenden Betriebssysteme: − − Microsoft® Windows® 7 Microsoft® Windows® XP (mindestens Service Pack 2 oder 3) (nur 32 Bit) • Bei Windows XP-Systemen Microsoft® Windows® Installer 3.1 • Microsoft® .NET Framework Version 3.5 (Service Pack 1)* • Visual JSharp (Version muss zur verwendeten .NET Framework-Version passen)* • Microsoft® SQL Server 2005 Express*, Microsoft® SQL Server 2005 Enterprise, Microsoft® SQL Server 2008 Express, Microsoft® SQL Server 2008 Enterprise Hinweis: Bevor Sie ein Upgrade auf ein neues Service Pack eines Drittanbieters wie Microsoft durchführen, erkundigen Sie sich bei Ihrem One Lambda-Vertreter, ob HLA Fusion dieses Service Pack unterstützt. Wenn die genannten Microsoft-Voraussetzungen bei Ihnen nicht erfüllt sind, lesen Sie auf der Website Microsoft.com nach. MINDESTVORAUSSETZUNGEN – HARDWARE • Pentium-Prozessor mit 1 GHz • 32-Bit (x86)- oder 64-Bit (x64)-Mikroprozessor • 1 GB Festplattenspeicherplatz (bei großen Datenbanken möglicherweise mehr) Hinweis: Für das Installieren von HLA Fusion werden, unabhängig vom Installationsort, für temporäre Installationsdateien und andere verwendete Programme bis zu 400 MB freier Speicherplatz auf Ihrer lokalen Festplatte benötigt. • 512 MB Hauptspeicher (RAM) • 8-Bit-Grafikadapter und -Display (für das gleichzeitige Anzeigen von 256 Farben) • VGA-Monitor mit einer Mindestauflösung von 1280 × 768 Pixel • Maus oder ein anderes Windows®-kompatibles Zeigegerät; bei einigen Produkten ist eine Maus mit Rad erforderlich www.onelambda.com One Lambda, Inc. 21001 Kittridge Street, Canoga Park, CA 91303, USA T: +1.818.702.0042 F: +1.818. 702.6904 HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 1 von 13 One Lambda, Inc. • | Software-Versionshinweise: HLA Fusion™ Version 3.0 Windows®-kompatibler Druckertreiber (PDF Distiller oder der Microsoft Document Image Writer-Treiber sind kostenlos verfügbar) * Im Installationsumfang von HLA Fusion enthalten ÄNDERUNGEN Seit Version 2.0 und deren Service Packs sind an HLA Fusion™ folgende Änderungen vorgenommen worden: Erweiterungen und Verbesserungen • Das System unterstützt jetzt den Algorithmus zur Separation überlappender Perlen für alle LABType HDProdukte und verwendet dazu die vorhandene RSSOH1C-Logik. • Das System unterstützt jetzt die FJ-Korrektur für Einzelantigene der Klasse II, ähnlich W632 für die Klasse I. • Benutzer können jetzt selbst auswählen, welche Startseite beim Anmelden bei HLA Fusion angezeigt werden soll. • Das System erlaubt es Benutzern jetzt, den Pfad für die LABType HD-Konvertierungsausgabe festzulegen oder anzuzeigen. Dieser Pfad wird zum Speichern konvertierter CSV-Dateien für LABType HD-Kits verwendet. • Das System bietet jetzt auf dem Patientenformular die Auswahl zwischen den Blutgruppen A1, B2, A1B und A2B. • Das System verfügt über ein zusätzliches Tutorial und Funktionen zur Anzeige von Videos in der Onlinehilfe. • Das System enthält jetzt ein Feld für den Pfad der LABType HD-Ausgabedatei. Der Pfad wird zum Speichern konvertierter CSV-Dateien für LABType HD-Kits verwendet. • Die Bw4/Bw6-Farbcodierung in LABScreen wurde verbessert, um die Spezifität besser darstellen zu können. • In der Reaktionstabelle in LABType Analysis werden die Sonden zur besseren Unterscheidung jetzt dem Exon-Bereich entsprechend farbig dargestellt. • Das System verwendet jetzt eine neue Konvention zur Perlenbenennung (Analyt). Das System enthält jetzt im Import- und im Stapelzusammenfassungsbildschirm sowie in den benutzerdefinierten Berichten ein Feld „Luminex Device“ (Luminex-Gerät) und ein Feld „Software Version“ (Softwareversion). Diese Felder erscheinen nur für LABType- und LABScreen-Produkte. • Benutzer können jetzt jede HLA Fusion-Datenbank umkonfigurieren und damit die maximale Datenbankgröße heraufsetzen. • Benutzer können jetzt HLA Fusion-Verlaufsprotokoll-Datenbanken zusammenführen. • Benutzer können jetzt direkt aus HLA Fusion heraus per E-Mail Softwarefeedback und Berichte senden. • Die HLA Fusion-Datenbank wurde hinsichtlich Speicherplatz und Indexierung optimiert. • Benutzer können jetzt nach allen Proben innerhalb einer Spezifität suchen und diese sortieren. • Typisierungsergebnisse werden jetzt in Form einer Quickinfo (Tooltip) angezeigt, sobald der Benutzer mit der Maus im QC-Histogramm im ersten Quadranten der LABType-Analyse auf eine Probe zeigt. • Für LABScreen PRA-Produkte steht jetzt eine benutzerseitig anpassbare Cut-off-Funktion zur Verfügung. • In der „Data Export“-Konfiguration (Datenexport) sind jetzt die Perlen-ID, Rohdaten und Spezifitäten verfügbar. • Benutzer können in benutzerdefinierte Berichte jetzt mehr Laborinformationen aufnehmen. • Im Modul „Patient Ab Tracking“ (Patientenantikörpererfassung) können Benutzer jetzt festlegen, dass nur die Antikörper verfolgt werden, die einem benutzerdefinierten Cut-off-Wert entsprechen. • Exporte und Ausdrucke des Diagramms „Column (Spec)“ (Säulendiagramm – Spez.) enthalten jetzt eine Legende mit den Proben-IDs für jeden einzelnen Probenindex. Auf diese Weise können Benutzer zuordnen, welcher Balken zu welcher Proben-ID gehört. ® HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 2 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Auf der Registerkarte „Haplo“ in LABType erfolgt die Gruppierung jetzt nach der eigentlichen Organisation des Haplotyps. • Die LABScreen-Analyse zeigt jetzt die Übereinstimmungs-/Nichtübereinstimmungsinformationen für spenderspezifische Antikörper (Donor Specific Antibodies, DSA) an. • Der Bericht „Antibody Custom“ (Antikörper benutzerdefiniert) enthält jetzt die Übereinstimmungs/Nichtübereinstimmungsinformationen für DSA. Die Informationen werden als Tabelle angezeigt, die die Spender-IDs, DSA und Rohdaten für den Test enthält und die Übereinstimmungen/Nichtübereinstimmungen hervorhebt oder nach Übereinstimmung/Nichtübereinstimmung gruppiert ist. • Das System erlaubt es dem Benutzer jetzt, eine endgültige Einzelallelzuordnung vorzunehmen Außerdem kann der Benutzer bei Bestätigung eines homozygoten Ergebnisses durch eine Familienstudie zu Berichtszwecken zusätzlich zum Anhang der entsprechenden Berichte eine Anmerkung „Homozygous Assignment Confirmed“ (Homozygote Zuordnung bestätigt) aktivieren. • Der Bericht „Custom Antibody“ (Antikörper benutzerdefiniert) enthält jetzt ein Diagramm, das die Spezifität (mindestens serologisch) und die Perlen-ID gegen die normalisierten Werte für die mittlere Fluoreszenzintensität (MFI) darstellt. • In der Datentabelle werden jetzt für jede Probe im Modul „Antibody Tracking“ (Antikörpererfassung) Kontrollwerte (negativ und positiv) angezeigt. • Es ist jetzt ein einem Microsoft® Excel-Arbeitsblatt ähnelnder Bericht/Export verfügbar, dessen erste Spalte alle Probennamen enthält, wobei jede Zeile für eine andere Probe steht, und dessen übrige Spalten von links nach rechts die Perlen-IDs mit den Rohdaten der jeweiligen Probe enthalten. • Das Feld „%SA“ ist jetzt Teil von „Field Chooser“ (Feldauswahl), sodass Benutzer das Feld aus ihren Zusammenfassungsberichten entfernen können. • In den Ergebnissen der Epitopanalyse in der LABScreen-Analyse und den zugehörigen Berichten wurde der Rohmittelwert der Positivwerte durch den Normalmittelwert der Positivwerte ersetzt. • Die MFI und die normalisierten Werte werden jetzt als ganze Zahlen angezeigt. • Benutzer können jetzt im Abschnitt „HLA Assignments Serology“ (HLA-Zuordnungen Serologie) des Moduls „Manage Patient“ (Patient verwalten) Bw4/6 zuweisen und dann im Modul „Ab Tracking“ (Antikörpererfassung) Bw4/6 verfolgen. • Benutzer können jetzt LABType-Produktinformationen exportieren. Diese ähneln den temporären Dateien, die zum Importieren von Produktinformationen in HLA Visual verwendet wurden. • Die Funktion „Find Antigen“ (Antigen suchen) in der LABScreen Mixed-Analyse arbeitet jetzt genauso wie ihr Gegenstück in der LABScreen PRA/SA-Analyse, mit der nach breiten Antigenen gesucht werden kann und bei der die gespaltenen Antigene eingekreist werden. Wird beispielsweise nach einem breiten Antigen, A9, gesucht, werden die gespaltenen Antigene, A23 und A24, eingekreist. • Das System besitzt jetzt eine „Donor PRA“-Funktion (Spender-PRA-Funktion) ähnlich der in der LABScreenAnalyse für „Patient Ab Tracking“ (Patientenantikörpererfassung) verwendeten Funktion, mit der der Prozentsatz für Klasse I und II eines gewählten Serums berechnet wird. • Die Spezifitäten im Abschnitt mit den Testdetails des Berichts „Custom Antibody“ (Antikörper benutzerdefiniert) sind jetzt alphanumerisch sortiert, wobei der Locus als alphabetisches Zeichen und der Nummernteil als numerische Zeichen behandelt werden. • Bei der Schwanzanalyse und der Epitopanalyse für das Klasse-II-Einzelantigen werden die Allele DQA und DPA berücksichtigt. • Die Liste unterhalb des Tablettrasters im LAT-Mixed Raw Data Report (LAT-Mixed-Rohdatenbericht) enthält jetzt Cut-off-Werte für die einzelnen Tests. Außerdem wurde für jede Cut-off-Stelle eine eigene Spalte hinzugefügt. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 3 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Die LABType-Analyseseite wurde verbessert und enthält jetzt Details zu allen Ergebnissen für jeden einzelnen Locus. Da für jeden Locus die Ergebnisse gespeichert werden, erscheint in diesem Abschnitt zu allen Loci entweder das Serologie-Ergebnis oder der Allelcode des jeweiligen Locus. • Titel in benutzerdefinierten Berichten (wie z. B. im Bericht „Molecular Custom“ (Molekular benutzerdefiniert)) dürfen jetzt bis zu 50 Zeichen lang sein. • Suchvorgänge anhand der Sitzungs-ID und Proben-ID in Modulen wie „Manage Data“ (Daten verwalten) und „Reporter“ (Berichterstellung) unterstützen jetzt chinesische Zeichen. • Das System erkennt das ELISA-Lesegerät jetzt auch an anderen COM-Ports als COM 1. • Die Micro SSP-Analyse ignoriert jetzt bei der Entscheidung, ob Wells ohne Amplifikation mit einem positiven und negativen Score analysiert werden sollten, das erste Well (1H). • Die Spezifitäten im Abschnitt „Reaction Score“ (Reaktionsergebnis) des Berichts „Custom Antibody“ (Antikörper benutzerdefiniert) sind jetzt so geordnet, wie UNOS die Spezifitäten sortiert, d. h. nach Locus (A, B, BW, DR, DQAB) und innerhalb der Loci numerisch. Innerhalb der einzelnen Reaktionsgruppen werden die einzelnen Spezifitäten außerdem immer nur einmal aufgeführt, während sie für jede Perle erneut aufgelistet werden. • Der Bericht „Combined Sample“ (Kombinierte Probe) im Modul „Reporter“ (Berichterstellung) enthält jetzt eine Option zum Anzeigen der zugeordneten Allelpaare im Feld „Corrected Typing“ (Korrigierte Typisierung), statt sie im Feld „Assigned Allele Codes“ (Zugeordnete Allelcodes) auszugeben. • Das System macht jetzt deutlich, dass LAT-Analyse-Konfigurationseinstellungen nicht für die LATM-Analyse gelten. • Das System erlaubt es Benutzern jetzt, vorhandene NMDP-Codes zu aktualisieren. Bei jeder Aktualisierung überschreibt das System vorhandene NMDP-Codes. • Das Modul „Manage Data“ (Daten verwalten) enthält jetzt einen Patientenfilter. • Beim Filter „Sample ID“ (Proben-ID) im Modul „Manage Data“ (Daten verwalten) sind jetzt Suchvorgänge mit Platzhalterzeichen möglich. • Das Feld „Test Date“ (Testdatum) im Bericht „Custom SSP“ (SSP benutzerdefiniert) zeigt jetzt das Datum der Probenanalyse und nicht das Sitzungsdatum an. • Im Abschnitt „Test Details“ (Testdetails) des Berichts „Custom SSP“ (SSP benutzerdefiniert) werden Allelspezifitäten nicht mehr abgekürzt. • Die Funktion „User defined Cutoffs“ (Benutzerdefinierte Cut-offs) wurde auf die Verwendung mit LABScreen PRA-Produkten erweitert. • Benutzer können zur Eingabe von Reaktionen bei der SSP-Analyse jetzt die Zifferntasten auf der Tastatur verwenden. • Das Textfeld für LABType-Zuordnungen ist jetzt größer, sodass alle Zuordnungen auch ohne Nutzung der Bildlaufleiste zu sehen sind. • Benutzer können jetzt eine endgültige Einzelallelzuordnung vornehmen Außerdem können Benutzer nach der Bestätigung eines homozygoten Ergebnisses durch eine Familienstudie zu Berichtszwecken die Anmerkung „homozygous assignment confirmed“ (homozygote Zuordnung bestätigt) aktivieren. • Die Spezifitäten in der Liste „Final Assignment“ (Endgültige Zuordnung) sind jetzt farbcodiert, um die Spezifitäten, die in den Tail Analysis (Schwanzanalyse)- und Epitope Analysis (Epitopanalyse)-Ergebnissen zugeordnet wurden, und die manuellen Zuordnungen unterscheiden zu können. • Der Stapelzusammenfassung wurde „Donor PRA“ (Spender-PRA) hinzugefügt. • Das System besitzt jetzt im Navigator eine Option, mit der Sitzungen nicht nach Sitzungsdatum, sondern nach Testdatum gruppiert werden können. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 4 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Benutzer können jetzt Spender zu Gruppen zuordnen und den Spender-PRA auf der Basis der Spender in der ausgewählten Gruppe berechnen, statt alle Spender in der Datenbank verwenden zu müssen. • Das System bietet jetzt eine Konfigurationseinstellung, über die der Benutzer festlegen kann, dass die Warnmeldung bezüglich einer bestimmten erforderlichen Bildschirmauflösung nicht angezeigt wird und er nicht gezwungen ist, die Bildschirmauflösung zu ändern. • Die CREG-Leiste in der Analyse lässt sich jetzt über die Konfigurationseinstellungen für die Antikörperanalyse ausblenden. Auf diese Weise kann bei Computern mit kleinerem Bildschirm verhindert werden, dass Teile des Diagramms und der Spezifitäten durch die CREG-Leiste verdeckt werden. • Die Spender-PRA-Berechnung enthält jetzt DQA1*. Das bedeutet, dass die Software die Molekülzuordnungen berücksichtigt, die zur Berechnung des Spender-PRA an der Patientenkarte vorgenommen wurden. • Das System zeigt jetzt an, ob der aktive NMDP-Code v2 oder v3 ist. • Benutzer können jetzt nach dem Wählen von „More Test“ (Weitere Prüfung) angeben, welche Tests sie durchführen möchten. Proben, für die Tests ausgewählt werden, werden entsprechend den angegebenen Prüfungen einer kumulativen Proben-/Testliste hinzugefügt. • Die verschiedenen DSA-Stärken werden jetzt in „Pt Ab Tracking“ (Patientenantikörpererfassung) durch Farbcodierung der Antikörper-Spezifitäten in der Antigen-Tabelle besser kenntlich gemacht. • Das System erlaubt jetzt das Umschalten zwischen Benutzern, ohne dass dazu die Anwendung beendet und dann mit anderen Anmeldedaten neu gestartet werden muss. • Das System zeigt jetzt Serologie- und NMDP-Updates auf den Moleklarprodukte-Startseiten an. • Benutzer können jetzt durch Wählen von „Auto Accept All“ (Alle automatisch akzeptieren) alle vorgeschlagenen Ergebnisse für alle Proben einer LAT Mixed-Sitzung speichern. • Bei der LABType-Analyse können jetzt G- und P-Gruppen analysiert werden. • Die Spezifitäten, die im Abschnitt „Rxn Score“ (Reaktionswert) des Berichts „Custom Antibody“ (Antikörper benutzerdefiniert) aufgeführt werden, stimmen jetzt mit den Spezifitäten im Abschnitt „Test Details“ (Testdetails) überein. • Benutzer können jetzt für die LABType-Sitzungsregisterkarten „Summary“ (Zusammenfassung), „Control Value“ (Kontrollwert) und „Bead Analysis“ (Perlenanalyse) Kommentare auf Sitzungsebene bearbeiten. • Das System enthält jetzt eine Funktion, mit der verhindert werden kann, dass im Modul „Manage Patients“ (Patienten verwalten) vorhandene Patientendaten überschrieben werden. • Während der Analyse wird jetzt das Probendatum angezeigt. • Die Rohdatenberichte, die von den Analysefenstern aus generiert werden, enthalten jetzt die Sitzungs-ID, die Patienten-ID, die Well-ID und das Testdatum. • Benutzer können jetzt den Standardgrenzwert (z. B. „2X“) und andere Konfigurationsoptionen festlegen. • Im Abschnitt „Rxn Score“ (Reaktionswert) des Berichts „Antibody Custom“ (Antikörper benutzerdefiniert) werden die einzelnen Spezifitäten jetzt in separaten Zellen aufgeführt. • Es ist ein neuer Statistikbericht mit einer Auflistung aller LABType-Kontrollwerte und statistischen Informationen, wie Durchschnittsanzahl der Proben, Mindestwerte und Höchstwerte, verfügbar. • Das SSP-Gel-Bild im Bericht „Custom SSP“ (SSP benutzerdefiniert) ist jetzt größer als 700 Pixel. • Benutzer können jetzt Gel-Bilder, die der Analyse angehängt sind, drehen und diese zoomen. • Benutzer können jetzt in den Bericht „Custom Antibody“ (Antikörper benutzerdefiniert) ein Diagramm mit MFIWerten auf der y-Achse und Antigenen auf der x-Achse aufnehmen. Die Sortierung erfolgt absteigend nach dem MFI-Wert. • Die Sortierung der endgültigen Zuordnungen in der Antikörperanalyse erfolgt jetzt alphanumerisch. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 5 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Der Bericht „LABType (BMT)“ enthält jetzt eine Spalte „Comments“ (Kommentare) und eine Spalte „Other Assignment“ (Andere Zuordnung). • Das System hängt jetzt an das Ende des Berichts „BMT - LABScreen“ den Spender-PRA und dann die Allelspezifitäten (MFI) der endgültigen Zuordnungen an. Die Allelspezifitäten sind alphanumerisch sortiert. • Benutzer können jetzt nach dem Speichern oder Bestätigen bei der aktuellen Probe bleiben, statt sich zur nächsten Probe bewegen zu müssen. • Der zugeordnete Allelcode („Assigned Allele Code“) im BML-Bericht wurde durch zugeordnete Allelpaare („Assigned Allele Pairs“) ersetzt. • Das System bietet jetzt beim Importieren ein zusätzliches Feld, in das das verwendete Sekundärattribut eingegeben werden kann. Dies ermöglicht auch die Erfassung des Sekundärantikörpers. • Benutzer können die Proben bei der Antikörpererfassung jetzt nach den Sekundärattributen filtern. • Das System bietet jetzt separate Felder für Kommentare: ein Feld für Systemstatusmeldungen und ein Feld für vom Benutzer eingegebene Kommentare. • Wird eine globale Cut-off-Anpassung vorgenommen, zeichnet das System diese jetzt in den Kommentaren nur für die Proben auf, auf die sich der Cut-off auswirkt. Erfolgt beispielsweise eine globale Cut-offAnpassung und von 96 Proben sind nur 5 Proben betroffen, wird der Statuskommentar zum globalen Cut-off nur für diese 5 Proben aufgezeichnet. • Die Spalten im Bericht „UMC-Utrecht“ mit dem Titel „A1_1 Result“ (Ergebnis A1_1) und „A1_2 Result“ (Ergebnis A1_2) enthalten jetzt 4-stellige Ergebnisse ohne den Locus-Buchstaben (Beispiel: 03:01). • Als Anfangsdatum in „Ab Tracking“ (Antikörpererfassung) wird jetzt standardmäßig das Probendatum der Probe verwendet, die mit der Patienten-ID verknüpft ist. • Benutzer können jetzt einen Mehrfachstapel aus Patientenlisten erstellen, indem sie über das Tool „Plate Designer“ die einzelnen Patientenlisten aus einer Luminex®-Patientenliste anwenden. • Benutzer können jetzt Micro SSP-Sitzungen als Stapel importieren und haben dabei die Möglichkeit, die Dateien zu durchsuchen. • Benutzer können in „Ab Tracking“ (Antikörpererfassung) jetzt Diagrammen Kommentare hinzufügen und die Diagramme mit den Kommentaren ausdrucken. • Benutzer können jetzt von der Analyse aus zum Fenster „Ab Tracking“ (Antikörpererfassung) navigieren und nach dem Schließen des Fensters „Ab Tracking“ wieder zurück zur Analyse navigieren. • Die Zeitleisten für Diagramme in „Ab Tracking“ (Antikörpererfassung) können jetzt beispielsweise so konfiguriert werden, dass sie Gesamttage repräsentieren, die durch den Probensatz, die tatsächlichen Probendaten usw. dargestellt werden. • Diagramme aus „Ab Tracking“ (Antikörpererfassung) enthalten jetzt den Patientennamen (Vor- und Nachnamen). • Benutzer können jetzt anhand der Proben-ID, der lokalen ID und anderen Patientendaten nach einem Patienten suchen. • Benutzer können jetzt in „Ab Tracking“ (Antikörpererfassung) anstelle der Patienten-ID den Patientennamen verwenden. • Benutzer können jetzt in „Ab Tracking“ (Antikörpererfassung) einen Spender und Spenderdaten hinzufügen und diese mit dem aktuellen Patienten verknüpfen. • Das System teilt HLA-Spezifitäten nicht mehr auf mehrere Zeilen auf. Dies gilt insbesondere für das Feld „Antibody Assignment“ (Antikörperzuordnung) im Abschnitt „Test Results“ (Testergebnisse) der Berichte „Antibody Custom“ (Antikörper benutzerdefiniert) und „Antibody Screening/ID“ (Antikörper-Screening/-ID). • Das System zeigt jetzt im Modul „Ab Tracking“ (Antikörpererfassung) den Patientennamen an. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 6 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Benutzer können jetzt nach einer Patienten-ID suchen, indem sie die ID teilweise eingeben und dann aus der in „Ab Tracking“ (Antikörpererfassung) angezeigten Liste die entsprechende Patienten-ID auswählen. • Benutzer können in „Ab Tracking“ jetzt Kreatininwerte für verschiedene Tage eingeben, die zusammen mit den Antigenen grafisch dargestellt werden. • Die Diagramme in „Ab Tracking“ (Antikörpererfassung) enthalten jetzt eine Schaltfläche, über die sich die Diagramme ein- und ausblenden lassen. • Benutzer können jetzt anhand der Patienten-ID nach Datensätzen suchen und dann bei Bedarf die Datensätze des betreffenden Patienten verschieben, archivieren und löschen. • Benutzer können jetzt den Bericht „Catalog Information“ (Kataloginformationen) im alten Format generieren. • Benutzer können jetzt einen Mindestmaßstab für die Diagramme in „Ab Tracking“ (Antikörpererfassung) festlegen. • „Patient Ab Tracking“ (Patientenantikörpererfassung) enthält jetzt ein viertes Diagramm zur Verfolgung der Probenverdünnung. Die x-Achse gibt dabei den Verdünnungsfaktor und die y-Achse die Rohdaten an. • Benutzer können jetzt für „LABScreen Single Antigen“ die W632-Normalisierung als Standard festlegen. • Benutzer können die W6-32-Normalisierung jetzt auch auf importierte Sitzungen anwenden. • Benutzer können jetzt in der Tabelle „Raw Data“ (Rohdaten) W632-normalisierte Daten mit nicht durch W632 normalisierten Daten vergleichen. • Das System berechnet jetzt beim Speichern/Bestätigen einer Probenanalyse den Spender-PRA automatisch. • Für Proben, die bereits getestet wurden, steht in den verschiedenen LABScreen-Kits für den Vergleich verschiedener Antigene überall dasselbe Diagramm zur Verfügung. • Das System unterteilt jetzt in „Forced Rxn“ (Erzwungene Reaktion) die Allele in zwei Spalten, sodass diese separat sortierbar sind. • Das System unterstützt jetzt einen Perlenausschlussbericht, aus dem übersichtsmäßig hervorgeht, wie oft jede einzelne Perle für einen ausgewählten Katalog ausgeschlossen wurde. • Der erste Quadrant des LABType-Analysefensters enthält jetzt eine Registerkarte mit einem Histogramm der kumulativen Daten des Benutzers. Dieses Histogramm zeigt die normalisierten Werte für alle die Proben an, die dieser Benutzer jemals für das Produkt (dieselbe Charge) durchgeführt hat, und dient als benutzererstelltes QC-Diagramm. Das Diagramm wird im Zuge der weiteren Nutzung des Produkts durch den Benutzer fortlaufend aktualisiert. • Bei der Probensuche können Benutzer jetzt Allele und eine Katalog-ID eingeben. Das System zeigt dann im Fenster „Allele Pair“ (Allelpaar) oder im Fenster „Assigned Allele“ (Zugeordnetes Allel) alle analysierten Proben mit der eingegebenen Katalog-ID und den eingegebenen Allelen an. • Benutzer können jetzt den Bericht „Combined Sample“ (Kombinierte Probe) so anpassen, dass serologische Ergebnisse und andere Zuordnungen darin aufgenommen werden. • Das System verfügt jetzt über ein Feld mit einer Dropdownliste für die Art des Transplantats, das der Patient erhalten wird oder erhalten hat. Die Dropdownliste enthält beispielweise die folgenden Einträge: „SPK“ (für „Simultaneous Pancreas Kidney Transplantation“, Gleichzeitige Transplantation von Pankreas und Niere), „KTA“ (für „Kidney Transplant Alone“, Nur Nierentransplantation), „LDKT“ (für „Living Donor Kidney Transplantation“, Transplantation Lebendspenderniere), „SPLK“ (für „Simultaneous Pancreas Living-donor Kidney“, Gleichzeitige Transplantation von Pankreas/Lebendspenderniere) und „Other“ (Andere). Es gibt ein weiteres Dropdownfeld für den Patientenstatus mit u. a. folgenden Einträgen: „C0“ = in der Bewertung, „C1“ = aktive Listung, „C2“ = anstehend, „C3“ = transplantiert und „C9“ = verstorben. • Der Sitzungs-(Stapel-)Zusammenfassungsbildschirm unterstützt jetzt die Anzeige der Spender-PRA-Prozentsätze. Diese können von diesem Bildschirm in die Berichte übernommen werden. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 7 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Im dritten Quadranten des LABType-Analysefensters können Benutzer jetzt als Standardkonfiguration festlegen, dass immer zuerst die Abweichung angezeigt wird. • Benutzer können jetzt LAT-Testinformationen (Proben-IDs, Tablettinformationen usw.) eingeben und speichern und dann den Tablettlesevorgang mit der Software zu einem späteren Zeitpunkt durchführen. • Benutzer können jetzt zu exportierende „Ab Tracking“ (Antikörpererfassung)-Diagramme mit einem Namen versehen und den Zielort für den Export auswählen. • Wenn der Benutzer die Registerkarte „Bead Analysis“ (Perlenanalyse) für Sitzungen auswählt, zeigt das System jetzt dieselben Perleninformationen an, wie wenn der Benutzer während der Probenanalyse die Registerkarte „Bead Info“ (Perleninformationen) auswählt. • Benutzer können jetzt die Tabelle „Raw Data“ (Rohdaten) offen lassen und von der Tabelle aus Zuordnungen vornehmen. • Mithilfe der LABType HD-Produkte zeigt das System jetzt bei Falschreaktionen die Proben und deren Alleldarstellung an, die für die Falschreaktionen relevant sind. • Die Berichte „Thai Export“ und „Thai Export V3“ wurden geändert und enthalten jetzt auch die exportierten Ergebnisse für LABType und LABScreen. • Das System unterstützt jetzt die Stapelerfassung der Allelcodes mit XX-Code in einer korrekt formatierten Excel-Datei, sodass diese geprüft und an NMDP gesendet werden kann. • Benutzer können jetzt mithilfe des Moduls „Data“ (Daten) endgültige Allelzuordnungen von der V2- in die V3Nomenklatur übersetzen. • Benutzer können jetzt während der LAT-Analyse das Probendatum eingeben. • Das System enthält jetzt im Modul „Catalog Management“ (Katalogverwaltung) eine Spalte „Catalog Import Date“ (Katalogimportdatum). • Alle „Updates Available“-Links (Updates verfügbar) für Produktkataloge sind jetzt mit dem Nomenklaturdatum und Überarbeitungshinweisen versehen. • Im Bericht „LSM Detail“ werden jetzt oben die Patienten-ID, der Patientenname und die lokale ID angezeigt. • Es wurde ein neuer Bericht mit Angaben zur bisherigen Leistung eines LABScreen-Produkts hinzugefügt, der die MFI-Werte für jede Perle aller getesteten Proben enthält. • Der Bericht „Molecular Custom“ (Molekular benutzerdefiniert) enthält jetzt Verträglichkeitsreaktionen. • Benutzer können jetzt Packlisten importieren, die das neue Format aus NMDP verwenden. • Wenn der Benutzer auf der Seite „Auto Update“ (Automatisch aktualisieren) unter „Update References“ (Referenzen aktualisieren) neben „Get Docs“ (Dokumente abrufen) das Kontrollkästchen „Update Previously Downloaded Documents“ (Zuvor heruntergeladene Dokumente aktualisieren) markiert, wird die neueste Version der Dokumente importiert. Dies gilt auch, wenn zuvor ältere Versionen importiert wurden. • Es wurde ein neuer Bericht hinzugefügt, der dem Zusammenfassungsbericht „Cutoff Adjustment“ (Cut-offAnpassung) ähnelt. Dieser neue Bericht zeigt für einen konkreten Katalog die Anzahl der Proben an, die diesen Katalog verwendet haben, wie viel Prozent dieser Proben einen niedrigen Positivkontrollwert hatten und wie viel Prozent eine niedrige Perlenanzahl aufwiesen. • Für LABScreen-Kataloge wurde ein neuer Statistikbericht hinzugefügt, der u. a. die folgenden statistischen Angaben enthält: • Gesamtanzahl der Proben, die den Katalog verwendet haben • Anzahl der Proben mit niedriger Perlenanzahl • Anzahl der Proben mit hohen Negativkontrollwerten • Anzahl der Proben mit niedrigen Positivkontrollwerten HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 8 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Der Bericht „BmT LABType“ wurde um SSP-Tests erweitert. Er verwendet dasselbe Format wie der aktuelle Bericht „BmT LABType“, exportiert dabei aber SSP-Ergebnisse in dieses Format. • Das System erkennt jetzt automatisch, ob ein Softwareupdate/-patch vorhanden ist, und informiert die Benutzer bei verfügbaren Updates. Die Softwareupdatefunktion „Upgrade“ steht nur HLA Fusion-Benutzern mit der Rolle „Supervisor“ und MS Windows-Administratorrechten zur Verfügung. • Der Bericht „BmT LABScreen“ enthält jetzt ein Spender-PRA-Feld. • Die manuelle Eingabe bei der LCT-Analyse ähnelt jetzt der bei Micro SSP, wo mehrere Sitzungen mit unterschiedlichen Katalogen und/oder Proben eingegeben werden können. • Die Allelspezifität des Berichts „Antibody Custom“ (Antikörper benutzerdefiniert) wird jetzt für jeden Locus in einer eigenen Spalte angezeigt, wobei die Anzeige der einzelnen Allele in der jeweils zugehörigen Locus-Spalte erfolgt. • Der Bericht „Custom Antibody“ (Antikörper benutzerdefiniert) enthält jetzt ein Diagramm, das Antigene und Perlen-ID in Bezug zu den normalisierten MFI-Werten in absteigender Reihenfolge darstellt. Das Diagramm ähnelt dem LABScreen-Analysediagramm. • Die „LABScreen PRA/Single Antigen Summary“ (LABScreen PRA/Single Antigen-Zusammenfassung) ist jetzt ein Bericht, auf den über die Registerkarte „Report“ (Bericht) in der Sitzungszusammenfassung zugegriffen werden kann. Der Bericht enthält die Patienten-ID. • Benutzer können jetzt während der LABScreen SA-Analyse die DQA- und DPA-Spezifitäten im Histogramm sortieren. • Im Feld „Antibody Assignment“ (Antikörperzuordnung) im Abschnitt „Test Results“ (Testergebnisse) der Berichte „Antibody Custom“ (Antikörper benutzerdefiniert) und „Antibody Screening/ID“ (Antikörper-Sreening/-ID) können Benutzer jetzt festlegen, dass beim Erweitern über die Zeile hinaus die Spezifitäten nicht aufgeteilt werden. • Benutzer können jetzt das Feld „Patient ID“ (Patienten-ID) in „Manage Sample Info“ (Probendaten verwalten) bearbeiten und damit eine vorhandene Patienten-ID ändern oder einen Patienten hinzufügen. • Das System besitzt jetzt in der Micro SSP-Analyse eine vom Computer zugewiesene Serologiefunktion, die der Funktion ähnelt, die für die LABType-Analyse bereits vorhanden ist. • Beim Starten einer neuen Micro SSP-Sitzung können Benutzer jetzt einen Locus-Filter hinzufügen, um die Anzahl der in der Dropdownliste angezeigten SSP-Kataloge zu begrenzen und, wichtiger noch, um nur die Kataloge anzuzeigen, die den ausgewählten Locus testen. • Die LABType-Analyse erfolgt beim Wechseln von einer Probe zu einer anderen jetzt schneller. Erreicht wird dies, indem die Liste der vorgeschlagenen Allelpaare abgekürzt wird, statt sie jedes Mal vollständig anzuzeigen. • Die Proben-ID-Informationen werden im Bericht „Custom Molecular“ (Molekular benutzerdefiniert) jetzt für jedes Ergebnis angezeigt, auch wenn sich die Proben-ID innerhalb einer Sitzung wiederholt. • Es wurde ein Bericht hinzugefügt, der alle zugeordneten Allelcodes übersichtlich gruppiert und Serologiezuordnungen, Perlen-Reaktionen im Nahbereich des Grenzwertes und Kommentare enthält. • Der Bericht „Molecular Custom“ (Molekular benutzerdefiniert) wurde um einen Abschnitt ausschließlich für PC- und NC-Ergebnisse ergänzt. Dieser ist neben dem Abschnitt mit den Sitzungsinformationen zu finden. • Es wurde eine virtuelle Kreuzprobenfunktion hinzugefügt, mit der Benutzer Klasse-I- und Klasse-II-Antikörper des Patienten aufzeichnen und anhand der Spendertypisierung nach diesen suchen können. • Das System gibt jetzt im Modul „Manage Patient“ (Patient verwalten) an, ob ein Patient oder ein Spender aus einer anderen Einrichtung stammt und wie der Name dieser Einrichtung lautet. Diese Informationen erscheinen in Berichten. • Das System unterstützt jetzt die Transplantationsvorgeschichte des Patienten. • Das System unterstützt jetzt im Patientenverwaltungssystem CDC-, CDC/AHG- und VX-Crossmatches. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 9 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Benutzer können jetzt sowohl automatisch als auch manuell P- und G-Gruppen von der IMGT-Website importieren. • Das System unterstützt im SSP-Modul jetzt die automatische Serologiezuordnung. • Das System protokolliert jetzt die Datenbankwartungsprozesse, geordnet nach Benutzer. • Benutzer können jetzt von HLA Fusion aus Änderungen an der Datenbank vornehmen, ohne die Software schließen zu müssen. • Benutzer können jetzt EPITOPE-Tabellen anpassen. Benutzer können jetzt eine ihrer im HLA Fusion-System selbst erstellten EPITOPE-Tabellen verwenden. Das System lässt bei Antigennamen nur Großbuchstaben zu und erlaubt, beim Erstellen neuer EPITOPE-Tabellen vorhandene Tabellen als Vorlage zu verwenden. • Benutzer können auf dem Analysebildschirm jetzt auf der Grundlage der ausgewählten EPITOPE-Gruppe/Tabelle Antigene einkreisen. • Das Laborprofil (LAB Profile) müsste so aktualisiert worden sein, dass es jetzt die Namen und E-Mail-Adressen der Distributoren enthält. Die E-Mail-Adressen müssen durch Semikolon (;) oder Komma (,) voneinander getrennt sein. • Das Berichterstellungsmodul unterstützt jetzt die Übertragung ausgewählter Berichte per E-Mail. Die E-MailSchaltfläche wird nur aktiviert, wenn im Benutzerprofil die E-Mail-Adresse konfiguriert wurde. Es ist eine Internetverbindung erforderlich. • Das Benutzerprofil unterstützt jetzt die E-Mail-Konfiguration, sodass der E-Mail-Versand über Microsoft® Outlook, Hotmail, Yahoo!, AOL und Google möglich ist. • Zu beachten ist, dass HLA Fusion die bereitgestellte E-Mail-ID und den bereitgestellten E-Mail-Anbieter verwendet. HLA Fusion garantiert weder die Korrektheit der E-Mail-Adressen noch die Zustellung der E-Mail. Es ist Aufgabe des Benutzers zu kontrollieren und zu bestätigen, ob die E-Mail-Adressen korrekt sind und ob die E-Mail beim Empfänger eingetroffen ist. • HLA Fusion verwendet das Protokoll und die Konfiguration, die vom E-Mail-Anbieter mit Stand vom Dezember 2011 veröffentlicht waren. Diese können sich jedoch von Zeit zu Zeit ändern. Sollte es Änderungen an der Konfiguration oder am Protokoll des E-Mail-Anbieters geben, werden diese in der nächsten HLA Fusion-Version entsprechend berücksichtigt werden. • Das System unterstützt jetzt den NKR (National Kidney Registry)-Datenexport. Dazu werden Patientendaten und Datumsbereiche für LABScreen SA-Produkte verwendet. • Ab RSSOH2B1 (LABType HD DRB1 SSO DNA Typing Test))-Charge 7B unterscheidet und trennt das System die Signale von regulären von denen überlappender magnetischer Perlen. Die Notwendigkeit für diese Änderung ergibt sich erst ab DRB1 HD-Kit-Charge 07B. • Das System verwendet jetzt bei HD-Kits für die Warnmeldungen bei niedriger Perlenanzahl die LuminexOriginalperle. • Benutzer können jetzt im Rahmen der Molekularanalyse-Konfiguration festlegen, ob die berechnete HDPerlenanzahl in der Rohdatentabelle angezeigt werden soll. • Das System kann jetzt die Art der Uneindeutigkeit und deren Ursache hinsichtlich der „High Definition“Analyse angeben. • Benutzer können jetzt in einer Sitzung mithilfe verschiedener SSP-Produkte mehrere Proben analysieren. • Benutzer können jetzt für die Funktion „Schedule Backup“ (Sicherung planen) festlegen, dass keine Datenbank erstellt wird. • Benutzer können jetzt den Bericht „LAT-Mixed“ nach Wellposition sortieren. • Benutzer können jetzt in die Funktion „Export Data“ (Daten exportieren) mögliche Allelpaare aufnehmen. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 10 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 • Das System bietet jetzt einen Bericht, der die Anzahl der Proben verfolgt, die bei den einzelnen Perlen pro Katalog-ID ausgeschlossen wurden. • Benutzer können jetzt benutzerdefinierte Berichte für LAT-, LCT- und Flow-Tests erstellen. • Benutzer können jetzt für eine Probe „More Test“ (Weitere Prüfung) aktivieren und dann angeben, welche zusätzlichen Prüfungen durchgeführt werden sollen. • Benutzer können jetzt festlegen, dass die Wells in der Geldarstellung in der SSP-Analyse nummeriert werden sollen. • Benutzer können jetzt während der Analyse im Probenauswahlfenster das Probendatum sehen und so Vergleichsanalysen anstellen. • Das System zeigt jetzt in Berichten die vollständigen Informationen zur Softwareversion (einschließlich SPVersion) an. • Das System hat in HLA Fusion jetzt einen Australien-Bericht für LABType („LABType - Australia“). • Das System unterstützt jetzt einen Export, der dem Bericht aus älterer LAT-Software ähnelt. • Die Felder „Class I Antibody Specificity“ (Klasse-I-Antikörper-Spezifität), „Class II Antibody Specificity“ (KlasseII-Antikörper-Spezifität), „MIC Antibody Specificity“ (MIC-Antikörper-Spezifität), „Unacceptable Antigens“ (Nicht-akzeptable Antigene) und „Acceptable Antigens“ (Akzeptable Antigene) auf der Registerkarte „Antibody“ (Antikörper) in der Patientenprobenübersicht sind jetzt größer, sodass die Benutzer alle Angaben sehen können, ohne die Bildlaufleiste verwenden zu müssen. • In den benutzerdefinierten Berichten („Molecular Custom“ (Molekular benutzerdefiniert), „Antibody Custom“ (Antikörper benutzerdefiniert), „Patient Custom“ (Patient benutzerdefiniert), „Custom SSP“ (SSP benutzerdefiniert) usw.) können Benutzer jetzt beliebige/alle Patientendaten auswählen und aufnehmen, die im Fenster „Patient/Donor Information“ (Patienten-/Spenderdaten) vorhanden sind, wie das Geburtsdatum, die ethnische Zugehörigkeit, die Diagnose, die Blutgruppe usw. • Das System wurde hinsichtlich der kombinierten Exon 4+-Analyse verbessert. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 11 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 Behobene Fehler • Möglichkeit, Exon 3 aus der Analyse für SSP und RSSO-Proben auszuschließen, die auf DQ- und DP-Produkte verweisen • Bei einem Cut-off in einem DRB345-Kit werden verschiedene Ergebnisse, wie beispielsweise DR52 und DR53, nicht korrekt zugeordnet. • Die Reaktionsmustertabelle ist jetzt alphanumerisch geordnet. Die Registerkarte „Force“ (Erzwingen) listet die Allele jetzt konsistent in der richtigen Reihenfolge auf, das heißt, das niedrigere der beiden Allele wird auf der linken Seite aufgeführt. • Die Bw4/Bw6-Zuordnung wurde korrigiert. Sie erfolgt jetzt auf Allel-Ebene und nicht mehr auf der Ebene der serologischen Gruppe. Die Erkennungsstellensteuerung wurde aktualisiert. Sie erkennt jetzt Negativwerte an der Position der Erkennungsstelle. • Proben verbleiben jetzt auf der Plate, wenn zwei Testlisten mit derselben Proben-ID und demselben Test auf der Plate zugewiesen wurden. • Die Suchfunktionen wurden aktualisiert. • Installationsproblem auf 64-Bit-Plattform. Der Abschnitt zu den unterstützten Plattformen wurde aktualisiert. Nur die 32-Bit-Version von Windows XP und die 32- und 64-Bit-Version von Windows 7 werden unterstützt. • Nur Benutzer mit Systemadministratorrolle dürfen die Datenbank auf eine andere Version aktualisieren. Alle anderen Benutzer können keine datenbankbezogenen Aufgaben ausführen. • Die Ausgabe von Ausnahmefehlern beim Kombinieren von LABType HD-Proben mit Exon 4-7-Proben aufgrund niedriger Positivkontrolle wurde korrigiert. • Die Spalten „NC“ und „PC“ sind jetzt Teil des Datenexports. • Wenn die LAT-Analyse ergibt, dass alle Perlen negativ sind, erscheinen die Antikörper DR51/52/53 nicht mehr in den Testergebnissen. • Die Probleme mit Datenbanknamen, die dazu geführt haben, dass die geplante automatische Sicherung eine Ausnahme ausgibt, wurden behoben. • Der Filter „Show matches with all testing“ (Übereinstimmung mit allen Testergebnissen anzeigen) berücksichtigt jetzt auch C-Locus-Testergebnisse. • In ganz bestimmten Situationen war es möglich, dass der Benutzer einen Vorlagennamen mit mehreren Katalogen verknüpfen konnte. Dies ist jetzt nicht mehr möglich. • Die Sortierung der serologischen und/oder Allelspezifitäten im Feld mit den endgültigen Zuordnungen wurde geändert. • Die Sortierreihenfolge bei der Antikörperanalyse wird jetzt standardmäßig auf die Systemsortierung zurückgesetzt und behält nicht mehr die Sortierreihenfolge der vorherigen Probe bei. • Bei der Einrichtung des Berichts „Custom SSP“ (SSP benutzerdefiniert) werden jetzt das aktualisierte Nomenklaturdatum und der NMDP/lokale Code im Bericht angezeigt. HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 12 von 13 One Lambda, Inc. | Software-Versionshinweise: HLA Fusion™ Version 3.0 ANWEISUNGEN Aktualisieren von Datenbanken, die mit früheren Versionen von HLA Fusion™ erstellt wurden Aktualisieren einer Datenbank, die mit einer früheren Version von HLA Fusion erstellt wurde, nach einer Neuinstallation Benutzer früherer Versionen von HLA Fusion: 1. Nachdem Sie die im HLA Fusion-Installationshandbuch genannten Schritte zum Installieren von HLA Fusion Version 3.0 durchgeführt haben, doppelklicken Sie auf Ihrem Computerdesktop auf das Fusion Database Utility-Symbol, um das Database Utility zu öffnen. 2. Wählen Sie im Fusion Database Utility die Funktion Upgrade prior versions of HLA Fusion database to 3.0 (Upgrade älterer Datenbankversionen auf HLA Fusion Database 3.0 durchführen). 3. Wählen Sie die Datenbank aus, die Sie auf die neue Version aktualisieren möchten, und geben Sie den Speicherort an, an dem die Sicherungskopie gespeichert werden soll. 4. Klicken Sie auf die Schaltfläche Upgrade. 5. Wählen Sie im Fusion Database Utility die Funktion Select Database (Datenbank auswählen). 6. Wählen Sie die Datenbank aus, für die Sie in Schritt 3 ein Upgrade durchgeführt haben, und klicken Sie auf Set (Festlegen). HLAF-REL NOTE-v3.x.x-DE-00, Rev 0 Seite 13 von 13