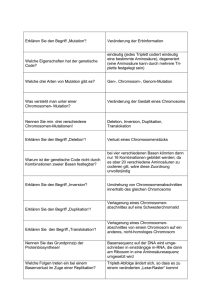
Biochemie_Breitling_Stryer_Kap28_1
Werbung

Vertretung durch Frank Breitling (Institut für Mikrostrukturtechnik (IMT), Campus Nord; www.imt.kit.edu/529.php) Vorlesungsdoppelstunde am 26.06.2014 Basis der Vorlesung: Stryer, Biochemie, 6. Auflage, Kapitel 28 Replikation, Rekombination und Reparatur von DNA Genauigkeit der DNA-Replikation Mittelalterliche Bücher mussten genau kopiert werden. DNA-Polymerase beim kopieren der beiden Gegenstränge (DNA Replisome image by Drew Berry, the Walter and Eliza Hall Institute). DNA-Replikation Jeder Strang einer Doppelhelix (blau) kann für die Synthese eines neuen komplementären Stranges (rot) als Matrize dienen (Semikonservative Replikation). Genauigkeit der Kopierrate: etwa ein Fehler pro 103 bis 104 kopierten Basen; Genauigkeit nach Korrekturlesefunktion: etwa ein Fehler pro 106 bis 107 kopierten Basen; Genauigkeit nach der Reparatur von fehlgepaarten Basen: etwa ein Fehler pro 109 bis 1010 kopierten Basen. Replikation, Schädigung und Reparatur von DNA Beim Replikationsvorgang können Fehler auftreten (schwarze Punkte). Weitere Defekte wie etwa Modifizierung von Basen, Quervernetzung sowie Einzel- und Doppelstrangbrüche (gelb) werden durch anschließende DNAschädigende Reaktionen in die DNA eingeführt. Viele dieser Fehler werden erkannt und in der Folge repariert. B-Form und A-Form der DNA DNA kann verschiedene Formen annehmen: Die Kalottenmodelle von jeweils zehn Basenpaaren der DNA in A- und B-Form lassen die rechtsgängige Helixstruktur erkennen. Die B-Form ist länger und schmaler als die A-Helix. Die Kohlenstoffatome des Molekülrückgrats sind weiß dargestellt. (Zeichnung nach 1BNA.pdb und 1DNZ.pdb.) Faltung der Zuckerringe In der A-DNA liegt das 3'-Kohlenstoffatom oberhalb der ungefähren Ebene, die durch die vier anderen NichtWasserstoffatome des Zuckermoleküls definiert wird. Diese Form heißt C-3'endo. In der B-DNA befinden sich die Riboseeinheiten in der C-2'-endoForm, bei der C-2' außerhalb der Ebene liegt. Die Seiten der großen und kleinen Furche Da die beiden glykosidischen Bindungen einander nicht diametral gegenüber stehen, hat jedes Basenpaar zwei Seiten: eine größere, welche die große Furche definiert, und eine kleinere auf der Seite der kleinen Furche. Die Furchen sind von potenziellen Donoren (blau) und Akzeptoren (rot) für Wasserstoffbrücken gesäumt. Große und kleine Furche in der BForm der DNA Man folge der großen Furche (orange) und der schmaleren kleinen Furche (gelb). Die Kohlenstoffatome im Molekülrückgrat sind weiß dargestellt. Die Grundstruktur der DNA zeigt sehr klar, dass z.B. DNA-bindende Proteine „messen“ können welche DNASequenz vorliegt. Die Propellerverdrehung Die 3D-Struktur von DNA ist überraschend vielfältig. Kristallstrukturdaten zeigten z.B., dass bei einigen kristallisierten DNA-Molekülen die Basen eines DNA-Basenpaares nicht in einer Ebene liegen (sind nicht coplanar angeordnet), sondern sind wie die Blätter eines Propellers gegeneinander verdreht sind. Diese Propellerverdrehung verstärkt die Basenstapelung in jedem Strang. Die Z-DNA DNA-Oligomere wie dCGCGCG nehmen unter bestimmten Bedingungen eine andere Konformation an. Diese wird als Z-DNA bezeichnet, weil die Phosphatgruppen im Molekülrückgrat zickzackförmig angeordnet sind. (Zeichnung nach 131D.pdb.) Vergleich der Struktureigenschaften von A-, B- und Z-DNA StrukturEigenschaften A B Helixtyp Z Gestalt am breitesten Höhe pro Basenpaar Helixdurchmesser Drehsinn Konformation der glykosidischen Bindung Anzahl der Basen pro Helixwindung Ganghöhe Neigung der Basenpaare zur Helixachse Große Furche Kleine Furche 0,23 nm 2,55 nm rechtsgängig anti zwischen Aund Z-Typ 0,34 nm 2,37 nm rechtsgängig anti 11 10,4 0,38 nm 1,84 nm linksgängig abwechselnd anti und syn 12 2,53 nm 11° 3,54 nm 1° 4,56 nm 9° eng und sehr tief breit und tief sehr breit und eng und flach ziemlich tief am schmalsten flach sehr eng und tief Die Verwindungszahl Das Schema zeigt die Zusammenhänge zwischen Verwindungszahl (linking number, Lk), Verdrehung (twist, Tw) und Windung (writhe, Wr) in einem ringförmigen DNA-Molekül. Topoisomere Entspannte und negativ superspiralisierte DNA in einer elektronenmikroskopischen Aufnahme. Struktur der Topoisomerase I Topoisomerasen entwinden DNA, oder – im Gegenteil – führen Superspiralen ein. Dargestellt ist die Struktur des Komplexes aus DNA und einem Fragment einer Typ-I-Topoisomerase des Menschen. Die DNA liegt in einem zentralen Hohlraum innerhalb des Enzyms. Mechanismus der Topoisomerase I Nachdem die Topoisomerase I an die DNA gebunden hat, greift ein Tyrosin (Y) ein Phosphat an, sodass ein Strang der DNA gespalten wird. Anschließend vollführt der geschnittene Strang kontrollierte Drehungen um den anderen Strang. Abgeschlossen wird die Reaktion durch Wiederverbinden des gespaltenen Stranges. Der Vorgang führt dazu, dass sich ein superspiralisiertes Plasmid ganz oder teilweise entspannt. Topoisomerasen vom Typ 1 entwinden die DNA. Bei Typ 1-Topoisomerasen wird ein Strang der Doppelhelix reversibel gespalten Die Hydroxylgruppe des Tyrosins 723 greift eine Phosphatgruppe in einem Strang des DNA-Rückgrats an und bildet eine Phosphodiesterbindung zwischen Enzym und DNA. Dabei wird die DNA gespalten und es entsteht eine freie 5‘-Hydroxylgruppe. Struktur der Topoisomerase II Dargestellt ist eine zusammengesetzte Struktur der Topoisomerase II aus der aminoterminalen, ATP-bindenden Domäne der Topoisomerase II aus E. coli (grün) und dem carboxyterminalen Abschnitt der Topoisomerease II aus Hefe (gelb). Wie man leicht erkennen kann, haben beide Einheiten eine Dimerstruktur. Man beachte den Hohlraum in der Mitte eines jeden Fragments des Dimers. (Zeichnung nach 1EI1.pdb und 1BJT.pdb.) Topoisomeren des Typs II führen Superspiralisierungen in die DNA ein. Dabei verbrauchen sie Energie in Form von ATP. Mechanismus der Topoisomerase II Die Topoisomerase II bindet zunächst an einen DNA-Doppelstrang, der als G-Segment (für gate = Tor) bezeichnet wird. Die beiden N-terminalen Domänen binden ATP und rücken dadurch näher zusammen. Diese Konformationsänderung hat zur Folge, dass beide Stränge des G-Segments gespalten werden, während gleichzeitig ein weiterer DNA-Doppelstrang, das T-Segment, gebunden wird. Das T-Segment wandert dann durch die Lücke im G-Segment und verlässt das Enzym an der Unterseite. Durch die Hydrolyse des ATP kehrt das Enzym mit dem immer noch gebundenen G-Segment in den Ausgangszustand zurück. Die Topoisomerase II der Bakterien dient als Angriffspunkt für mehrere Antibiotika Novobiocin blockiert die Bindung des ATP an die bakterielle Topoisomerase II (= Gyrase), Naldixinsäure und Ciprofloxacin hemmen die Spaltung und Wiederverbindung der DNA-Ketten. Die Struktur der DNAPolymerase Die erste DNA-Polymerase, deren Struktur man aufklärte, war das so genannte KlenowFragment der DNA-Polymerase I aus E. coli. Ihre Polymeraseeinheit ähnelt wie die anderer DNA-Polymerasen einer rechten Hand mit Fingern (blau), Handfläche (gelb) und Daumen (rot). Zum Klenow-Fragment gehört außerdem auch eine Exonucleasedomäne, die falsche Nucleotide entfernt. (Zeichnung nach 1DPI.pdb.) Der Wirkmechanismus der DNA-Polymerase An der Polymerasereaktion sind zwei Metallionen (in der Regel Mg2+) beteiligt. Das eine geht eine koordinative Bindung zur 3'-Hydroxylgruppe des Primers ein, während das andere nur mit dem dNTP wechselwirkt. Die Phosphatgruppe des Nucleosidtriphosphats bildet eine Brücke zwischen den beiden Metallionen. Die Hydroxylgruppe des Primers greift die Phosphatgruppe an, sodass eine neue O–P-Bindung entsteht. Primer: Das zu verlängernde Anfangssegment eines Polymers, von dem die Elongation abhängt. Die komplementären Formen der Basen bewirken die Spezifität der Replikation Das Basenanalogon rechts besitzt die gleiche Form wie Adenosin, aber chemische Gruppen, die in Basenpaaren Wasserstoffbrücken ausbilden, wurden durch bestimmte Gruppen (rot) ausgetauscht, die keine Wasserstoffbrücken bilden können. Dennoch zeigen die Untersuchungen, dass dieses Analogon, wenn es sich in der Matrize befindet, den Einbau von Thymidin bei der DNAReplikation bewirkt. Die Seiten der großen und kleinen Furche Da die beiden glykosidischen Bindungen einander nicht diametral gegenüber stehen, hat jedes Basenpaar zwei Seiten: eine größere, welche die große Furche definiert, und eine kleinere auf der Seite der kleinen Furche. Die Furchen sind von potenziellen Donoren (blau) und Akzeptoren (rot) für Wasserstoffbrücken gesäumt. Wechselwirkungen an der kleinen Furche dienen der DNAPolymerase als „Messlatte“ DNA-Polymerasen wirken gegenüber den Basenpaaren in der kleinen Furche als Donoren für zwei Wasserstoffbrücken. An den betreffenden Positionen stellen alle Watson-Crick-Basenpaare Wasserstoffbrückenakzeptoren bereit. Als Beispiel ist hier das AT-Basenpaar abgebildet. Selektive Form Die Bindung eines Desoxynucleosidtriphosphats (dNTP) an die DNA-Polymerase löst eine Konformationsänderung aus. Dabei entsteht eine enge Tasche für das Basenpaar, das aus dem dNTP und seinem Partner im Matrizenstrang besteht. Diese Konformationsänderung ist nur dann möglich, wenn es sich bei dem dNTP um den Watson-Crick-Partner der Base in der Matrize handelt. Warum gibt es ähnliche Konformationsänderungen bei allen Enzymen, die in irgendeiner Form NTPs oder dNTPs hydrolisieren? Die Funktion des Primers Als Primer der DNA-Replikation dient ein kurzes RNA-Stück, das von einer Primase, einer RNA-Polymerase, synthetisiert wird. Die Primer-RNA wird zu einem späteren Zeitpunkt der Replikation ausgeschnitten. Okazaki-Fragmente An der Replikationsgabel werden beide DNA-Stränge gleichzeitig in 5'→3‚ Richtung synthetisiert. Der Leitstrang wird kontinuierlich zusammengesetzt, der Folgestrang in Form kurzer Teile, die man Okazaki-Fragmente nennt. Die DNA-Ligase-Reaktion verknüpft die Okazaki-Fragmente miteinander Die DNA-Ligase katalysiert die Verknüpfung eines DNA-Stranges, der eine freie 3'-OH-Gruppe besitzt, mit der freien 5'-Phosphatgruppe eines anderen. Bei Eukaryoten und Archaea wird ATP dabei zu AMP und PPi gespalten, um die Reaktion anzutreiben. In Bakterien stammt die Energie aus der Spaltung von NAD+ zu AMP und Nicotinamidmononucleotid (NMN). Struktur einer Helikase Die bakterielle Helikase PcrA enthält vier Domänen: A1, A2, B1 und B2. Zur A1Domäne gehört eine P-Schleife-NTPase-Falte (violett unterlegt; P-Schleife grün dargestellt). Die B1Domäne hat insgesamt eine ähnliche Struktur, ihr fehlt aber die P-Schleife und sie bindet keine Nucleotide. Einzelsträngige DNA bindet an die Domänen A1 und B1 in der Nähe der Berührungsflächen zu A2 und B2. (Zeichnung nach 3PJR.pdb.) Die Trennung der DNA-Stränge erfordert spezifische Helikasen und die Hydrolyse von ATP Anfangs binden die Domänen A1 und B1 von PcrA einzelsträngige DNA. Nachdem ATP gebunden wurde, schließt sich die Spalte zwischen den beiden Domänen und die Domäne A1 gleitet an der DNA entlang. Durch die Hydrolyse des ATP öffnet sich die Spalte und zieht die DNA von Domäne B1 in Richtung A1. Der gleiche Vorgang wiederholt sich vielfach, sodass die DNA entwunden wird. Die Punkte auf dem roten Strang, die zwei Orte auf dem Strang markieren, bewegen sich, wenn die Doppelhelix entwunden wird. Das Sequenzmotiv P-Schleife-NTPase-Falte kommt auch in vielen anderen Proteinen vor, die eine starke Konformationsänderung nach der Bindung von einem Nucleotid durchmachen. Beispiele sind G-Proteine, NMP-Kinasen und Myosin. Konservierte Aminosäuren in Helikasen Beim Vergleich der Aminosäuresequenzen mehrerer Hundert Helikasen fand man mehrere Abschnitte mit stark konservierter Sequenz (farbig dargestellt). Auf die Struktur von PcrA übertragen, liegen diese konservierten Bereiche an der Grenzfläche zwischen den Domänen A1 und B1 sowie an der Oberfläche, die das ATP bindet. (Zeichnung nach 3PJR.pdb.) Dies unterstützt die Annahme, dass alle Helikasen ganz ähnliche Konformationsänderungen durchmachen. Struktur der gleitenden DNA-Klammer DNA-Polymerasen sind hochprozessiv: Die β2Dimeruntereinheit der DNA-Polymerase III bildet einen Ring, der den DNA-Doppelstrang umfasst. Man beachte den zentralen Hohlraum, durch den die DNA gleitet. Durch die Umklammerung der DNA in diesem Ring kann die Polymerase die DNA entlangwandern, ohne von ihrem Substrat abzufallen. Die Replikationsgabel Bei E. coli werden etwa 2.000 Basen pro Sekunde eingebaut! Schematische Darstellung der Anordnung der Polymerase III und der assoziierten Enzyme und Proteine, wie sie bei DNA vorkommen, die repliziert wird. Die Helikase trennt die beiden Stränge der ursprünglichen Doppelhelix, sodass die DNA-Polymerasen jeden Strang als Matrize für die DNA-Synthese verwenden können. Abkürzung: SSB, einzelstrangbindendes Protein. Das Holoenzym der DNAPolymerase III Jedes Holoenzym besteht aus zwei Kopien des Core-Enzyms der Polymerase, das die Untereinheiten α, ε und θ sowie zwei Kopien der β-Untereinheit umfasst und mit der zentralen Struktur verknüpft ist. Diese enthält den clamp loader-Komplex und die hexamere Helikase DnaB. Posaunenmodell Die Replikation des Leit- und des Folgestranges erfolgt koordiniert, indem die Matrize des Folgestranges eine Schleife bildet. Dadurch entsteht eine Struktur, die sich so ähnlich wie der Außenzug einer Posaune bewegt und sich mit der Vorwärtsbewegung der Replikationsgabel verlängert. Wenn die Polymerase des Folgestranges einen Abschnitt erreicht, der bereits repliziert ist, wird die gleitende Klammer freigesetzt und eine neue Schleife gebildet. Die DNA-Polymerase I entfernt den Primer und füllt die Lücken zwischen den neu gebildeten Okazaki-Fragmenten auf. Bei E. coli beginnt die DNA-Replikation an einer einzigen Stelle, dem Replikationsursprung OriC ist 245 Basenpaare lang und enthält eine Tandemanordnung von drei nahezu identischen Oligonucleotiden mit je 13 Nucleotiden (grün) sowie fünf Bindungsstellen (gelb) für das DnaAProtein. DnaA gehört zu der Familie der P-Schleifen-NTPasen. Der daraus gebildete Komplex zerfällt nach der Hydrolyse von ATP. Zusammenbau von DnaA Monomere von DnaA binden an ihre Bindungs-stellen (gelb) in der oriC-Region und bilden zusammen eine komplexe Struktur, möglicherweise ein ringförmiges Hexamer, wie es hier dargestellt ist. Diese Struktur markiert den Replikationsursprung und unterstützt die Trennung der DNA-Stränge in den ATreichen Abschnitten (grün). Auch DnaA hat das Sequenzmotiv P-SchleifeNTPase-Falte: Nach der Hydrolyse von ATP ändert sich seine Konformation stark und das DnaA Hexamer zerfällt. Einige Arten von DNA-Polymerasen Bezeichnung Funktion Prokaryotische Polymerasen DNA-Polymerase I entfernt den Primer und füllt die Lücken im Folgestrang DNA-Polymerase II DNA-Reparatur (fehleranfällige Polymerase) DNA-Polymerase III Hauptenzym der DNA-Synthese Eukaryotische Polymerasen DNA-Polymerase a Initiatorpolymerase Primaseuntereinheit synthetisiert den RNA-Primer DNA-Polymerasehängt etwa 20 Nucleotide an den Primer untereinheit DNA-Polymerase b DNA-Reparatur (fehleranfällige Polymerase) DNA-Polymerase d Hauptenzym der DNA-Synthese Bei Eukaryonten beginnt die DNA-Synthese an mehreren Stellen („Replikons“). Analog zu E. coli bilden sich zunächst Replikationsursprungskomplexe aus sechs verschiedenen Proteinen, die alle zu DnaA homolog sind. Anschließend wird eine Helikase mobilisiert. Für das kopieren eines eukaryotischen Replikons werden zwei verschiedene DNAPolymerasen benötigt. Der eukaryotische Zellzyklus Bei Eukaryoten müssen DNAReplikation und Zellteilung genau koordiniert werden. Die Mitose (M) findet immer erst nach der DNA-Synthese (S) statt. Die beiden Vorgänge sind durch die zwei Phasen G1 und G2 (G für gap = Lücke) getrennt. Bei Eukaryonten beginnt die DNA-Synthese an mehreren Stellen („Replikons“). Telomere sind besondere Strukturen an den Enden linearer Chromosomen Die DNA an den Telomeren enthält mehrere Hundert Tandemwiederholungen aus einer Sequenz aus sechs Nucleotiden. Aus dem Ende des Telomers ragt ein einzelsträngiger Abschnitt des Greichen Stranges. Nach einem Modell für den Telomermechanismus wandert dieser Strang in den Doppelstrang ein, sodass sich eine große doppelsträngige Schleife bildet. Bildung der Telomere Der Synthesemechanismus für den G-reichen Strang der Telomer-DNA. Die RNA-Matrize der Telomerase ist blau dargestellt, die an den Greichen Strang des Primers angefügten Nucleotide sind rot. (Nach Blackburn EH (1991) Nature 350: 569–573.) Der Proteinbestandteil der Telomerase ist mit den Reversen Transkriptasen verwandt. Telomerasen bringen ihre eigene Matrize mit. Bei der DNA-Replikation kann es zu Fehlern kommen: Vermehrung von Triplettwiederholungen Sequenzen mit Tandemwiederholungen von Triplettsequenzen können länger werden und weitere Tripletts aufnehmen, wenn einige Wiederholungseinheiten bei der Replikation eine Schleife bilden. Die mit dem roten Strang als Matrize gebildete Doppelhelix enthält dann zusätzliche Sequenzen, die der ausgestülpten Region entsprechen. Solche Fehler treten bei der Krankheit Chorea Huntington auf. Basen können durch oxidierende, alkylierende Agenzien und durch Licht beschädigt werden Wenn Guanin zu 8-Oxoguanin oxidiert wird, kann die so beschädigte Base über einen Randbereich des Moleküls, der sonst nicht an der Ausformung von Basenpaaren beteiligt ist, mit Adenin ein Basenpaar bilden. Desaminierung von Adenin kann zu Mutationen führen Die Base Adenin kann zu Hypoxanthin desaminiert werden. Hypoxanthin bildet Basenpaare mit Cytosin ähnlich wie Guanin, sodass die Desaminierungsreaktion zu einer Mutation führen kann. Manche mutagene Verbindungen entstehen erst durch Leberenzyme Aflatoxin, ein Produkt einzelliger Pilze, die auf Erdnüssen gedeihen, wird durch Cytochrom P450 aktiviert. In dieser hoch-reaktiven Form verändert sie das Guanin und andere Basen in der DNA, sodass es zu Mutationen kommt. Manche Mutationen entstehen durch (UV-) Licht Durch ultraviolettes Licht entstehen Querverbindungen zwischen benachbarten Pyrimidinen in einem Strang der DNA. Wenn diese Thymindimere nicht repariert werden entstehen Mutationen in der Erbsubstanz. Eine Verbindung, die Quervernetzungen erzeugt Die Verbindung Psoralen und seine Derivate können durch zwei reaktive Stellen, die mit Nucleotidbasen reagieren, Quervernetzungen zwischen DNA-Strängen erzeugen. DNA-Schäden können auf verschieden Weise erkannt und repariert werden: (1) Korrekturlesen Wenn eine falsche Base eingebaut wird, verlangsamt sich die DNA-Synthese aufgrund der Schwierigkeit, ein Nicht-Watson-Crick-Basenpaar durch die Polymerase zu fädeln. Gelegentlich verlässt die wachsende Polynucleotidkette das Polymerasezentrum der DNA-Polymerase und wandert zum Exonucleasezentrum. Dort werden ein oder mehrere Nucleotide aus der neu synthetisierten Kette herausgeschnitten und möglicherweise falsche Basen entfernt. (2) Die Mismatch-Reparatur kommt in allen Zellen vor Die Reparatur von Fehlpaarungen in der DNA von E. coli beginnt mit dem Wechselspiel der Proteine MutS, MutL und MutH. MutS erkennt eine GT-Fehlpaarung und MutH spaltet in der Nähe der Fehlpaarung das Rückgrat. Ein Teil des DNAStranges, der das fehlerhafte Thymin enthält, wird von der Exonuclease I ausgeschnitten und von der DNAPolymerase III neu synthetisiert. (Nach Service RF (1994) Science 263: 1559–1560.) (3) Die DNA-Photolyase nutzt Lichtquanten um Pyrimidindimere in die ursprünglichen Basen zu spalten. (4) Basenexcisionsrearatur Dargestellt ist der Komplex aus dem DNA-Reparaturenzym AlkA und dem Analogon eines DNAMoleküls, dem eine Purinbase fehlt (eine apurinische Stelle). Der Zucker im DNA-Rückgrat der apurinischen Stelle „springt“ zum Ausschneiden aus der DNA-Doppelhelix in das aktive Zentrum des Enzyms. (4) Excisionsreparatur Die Reparatur eines DNAAbschnitts mit einem Thymindimer durch die aufeinander folgende Tätigkeit (i) einer spezifischen Excinuclease, (ii) einer DNA-Polymerase und (iii) einer DNA-Ligase. Das Thymindimer ist blau dargestellt, der neue DNA-Abschnitt ist rot. (Nach Hanawalt PC (1982) Endeavour 31: 83.) Warum gibt es Thymin statt Uracil in der DNA? Durch spontane Desaminierung von Cytosin entstehen häufig Uracilreste in der DNA. Diese – und nicht die ThyminStellen – werden von dem Enzym Uracil-DNA-Glycosylase erkannt und ausgeschnitten. Danach wird der Bereich ohne Pyrimidin („AP“) durch eine Endonuclease geschnitten und die Fehlstelle durch Cytosin ersetzt. Die Methylstelle von Thymin ist die Markierung, die es von desaminiertem Cytosin unterscheidet! Viele Krebsarten entstehen durch fehlerhafte DNA-Reparatur Krebszellen sind häufig durch DNAschädigende Moleküle angreifbar: (1) Sie teilen sich häufig (2) Sie haben häufig Defekte in DNAReparatursystemen Cyclophosphamid und Cisplatin schädigen die DNA schnell wachsender Zellen mehr als die DNA normaler Zellen. Deswegen kann manchmal der Krebsteufel mit diesen Belzebuben ausgetrieben werden. Es gibt aber auch genetisch bedingte Schwächen in den Reparatursystemen, z.B. bei Xeroderma pigmentosum. Dabei sind Komponenten des Nucleotidexcisionsreparatursystems mutiert. Viele betroffene entwickeln bösartige Hauttumoren unter Einfluss von Sonnenlicht. Der Ames-Test A) Eine Petrischale mit etwa 109 Bakterien, die kein Histidin synthetisieren können. B) Zugabe eines Stückes Filterpapier mit einem Mutagen, das zu einer großen Zahl von Revertanten führt, die Histidin synthetisieren können. Diese Revertanten werden nach zwei Tagen als Ring von Kolonien um das Filterpapierstück sichtbar. Die wenigen sichtbaren Kolonien in Schale A sind spontane Revertanten. (Ames BN et al (1975) Mutat Res 31: 347–364.) Die DNA-Rekombination spielt bei der Replikation, Reparatur und anderen Reaktionen der DNA eine wichtige Rolle Zwei DNA-Moleküle können rekombinieren und neue Moleküle mit Abschnitten beider Ausgangsmoleküle bilden. Dies ist wichtig für: (1) Unterbrochene Replikation (2) Reparatur von Doppelstrangbrüchen (3) Meiose (4) Herstellung der Antikörpervielfalt (5) Für Viren, die so ihr genetisches Material in die Wirtszelle einbauen (6) Für die Gentechnik, um z.B. „Knockout-Mäuse“ herzustellen. Stranginvasion Dieser Vorgang, an dem Proteine wie RecA beteiligt sind, kann eine Rekombination initiieren. Eine Stranginvasion kann viele Prozesse in Gang setzen, beispielsweise die Reparatur von Doppelstrangbrüchen, wobei der Rekombinationspartner ein intaktes DNA-Molekül ist mit einer überlappenden Sequenz. Mechanismus der Rekombination Zu Beginn lagern sich zwei DNA-Moleküle zu einer Rekombinationssynapse zusammen. In jeder Doppelhelix wird ein Strang von der Rekombinase geschnitten, und die dabei freiwerdenden 3'- Enden werden jeweils mit einem Tyrosinrest (Y) des Enzyms verknüpft. Die neuen Phosphodiesterbindungen bilden sich, wenn ein 5'-Ende des anderen gespaltenen Stranges dieses Tyrosin-DNA-Addukt angreift. Nach der Isomerisierung wiederholen sich diese Schritte, sodass die rekombinierten Produkte entstehen. Rekombinasen und Topoisomerase I Der direkte Vergleich von CreRekombinase (blau) und Topoisomerase I (orange) zeigt, dass beide Enzyme eine gemeinsame Kernstruktur besitzen. Die Tyrosinreste, die an der DNA-Spaltung mitwirken, sind für beide Enzyme als rote Kugeln eingezeichnet. (Zeichnung nach 2CRX.pdb und 1A31.pdb.)