Methoden der künstlichen DNA-Rekombination
Werbung

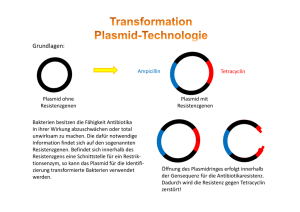
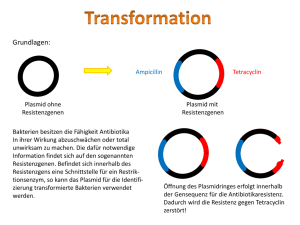
Gentechnik 1 Methoden der künstlichen DNA-Rekombination 106.1 Grundoperationen der Gentechnik 1973 gelang es den amerikanischen Wissenschaftlern COHEN und BOYER erstmals, DNAFragmente von verschiedenen Bakterien im Reagenzglas neu zu kombinieren und in das Bakterium Escherichia coli einzuschleusen. Das Bakterium erhielt so neue Antibiotikaresistenzen. Diese Experimente leiteten den Beginn der Gentechnik ein. Bakterium Zelle Plasmid wird isoliert Bakterien- Plasmid chromosom gereinigte DNA Gen X DNA mit dem Gen X Isolation rekombinante DNA (Plasmid) Gen X wird in Plasmid eingebaut Rekombination rekombinantes Bakterium Plasmid wird in Bakterienzelle übertragen Transformation Genkopien werden isoliert und auf andere Organismen übertragen Pflanzen werden resistent gegen Insekten Anwendung 106 Gentechnik Zellen mit Gen X werden kloniert produziertes Protein wird isoliert und wirkt als Enzym Chymosin stellt aus Milch Käse her Plasminogen Aktivator löst Blutgerinnsel nach einem Herzinfarkt auf Gentechnik beinhaltet die Isolierung von DNA, deren Rekombination im Reagenzglas und ihre Übertragung in vermehrungsfähige Zellen. Ein wichtiges Werkzeug der Gentechnik wurde bereits in den 60er Jahren entdeckt. Man beobachtete, dass E. coli eine Phageninfektion erfolgreich verhindert, indem die Phagen-DNA von Enzymen zerschnitten wird. Den Prozess bezeichnet man als Restriktion und die Enzyme als Restriktionsenzyme. Die eigene DNA wird nicht geschnitten, da diese durch Methylgruppen geschützt ist. Heute sind sehr viele Restriktionsenzyme bekannt. Sie werden nach der Bakterienart benannt, aus der sie stammen. So bedeutet der Name EcoRI, dass es als erstes Restriktionsenzym aus dem Stamm R des Bakteriums Escherichia coli isoliert wurde. HindIII wurde als drittes Restriktionsenzym aus dem Stamm d des Bakteriums Haemophilus influenzae gewonnen. Die unterschiedlichen Restriktionsenzyme finden die spezifische Schnittstelle durch eine ganz bestimmte Basensequenz aus zumeist vier bis acht Nucleotiden. Diese Stelle nennt man deshalb Erkennungssequenz. Bei den gentechnisch wichtigen Enzymen liegt die Schnittstelle in der Erkennungssequenz. Für EcoRI heißt diese Sequenz GAATTC, die Schnittstelle liegt zwischen G und A. Häufig ist diese Basensequenz am Komplementärstrang der DNA entgegengesetzt gelesen identisch. So ein Phänomen heißt Palindrom. Dafür gibt es auch sprachliche Beispiele: ERIKA FEUERT NUR UNTREUE FAKIRE. Da der DNA-Doppelstrang versetzt geschnitten wird, entstehen überstehende Einzelstrangenden. Sie sind zu den Enden der DNA-Fragmente, die mit dem gleichen Restriktionsenzym geschnitten wurden, komplementär. Man nennt diese Enden „klebrige Enden“ (sticky ends), da sie sich leicht über Wasserstoffbrücken verbinden. Das hinzugefügte Enzym Ligase verknüpft dann die DNA-Fragmente kovalent unter Ausbildung einer Phospho-Esterbindung. Auf diese Weise lässt sich ein DNA-Fragment mit einem gewünschten „Gen X“ in ein Plasmid einbringen. Man erhält eine rekombinante DNA. Plasmide dienen als Transportmittel, die ein Gen X in Wirtszellen einschleusen. So ein „Gentaxi“ nennt man Vektor. Der Vorgang des Einschleusens wird als Transformation bezeichnet. Restriktionsenzym 5’ 3’ 5’ G A A T T C C T T A A G A A T A A Die Bakterien produzieren jetzt das Protein, das durch das eingeschleuste Gen codiert wird. Für die Medizin werden auf diese Weise Enzyme und Hormone hergestellt. So hergestelltes Humaninsulin wird zur nebenwirkungsfreien Behandlung von Diabetes eingesetzt. Bei einem Herzinfarkt wird ein gentechnisch hergestelltes Enzym, der Plasminogen Aktivator, in hohen Konzentrationen injiziert, um das Blutgerinnsel im Herzen in Sekundenschnelle aufzulösen. Der Jahresumsatz solchermaßen hergestellter Proteine beläuft sich weltweit auf zweistellige Milliardenbeträge. Die Anwendung der Resistenzgene als Marker nimmt in zunehmendem Maße zugunsten von Genen ab, die für fluoreszierende Proteine codieren. T 3’ C G T T Zugabe eines DNA-Fragments klebrige Enden A Um herauszufinden, welche Bakterienzellen Plasmide mit einem Gen X auch wirklich aufgenommen haben, muss noch eine Selektion durchgeführt werden. Daher verwendet man als Vektor ein Plasmid, das zwei Antibiotikaresistenzen aufweist, zum Beispiel gegen Tetracyclin und gegen Ampicillin. Das Wirtsbakterium weist diese Resistenzen nicht auf. Die Schnittstelle auf dem Plasmid, in die das Gen X eingefügt wird, liegt im Gen für die Ampicillin-Resistenz. Hat das Plasmid das Gen aufgenommen, so wurde die Resistenz gegen Ampicillin durch den Schnitt zerstört. Das rekombinante Bakterium kann also auf Nährböden mit Ampicillin nicht mehr wachsen, wohl aber auf einem Nährboden mit Tetracyclin. Nicht transformierte Wirtszellen können auf keinem der beiden Nährböden wachsen. Nach der Transformation lässt man die Zellen zunächst auf Tetracyclin-haltigem Nährboden wachsen (1) und überträgt dann durch Stempeln die Kolonien auf den Nährboden mit Ampicillin (2). Aus dem Vergleich der Koloniemuster lässt sich die Kolonie ermitteln, die auf Ampicillin nicht mehr wächst (3). Diese enthält das rekombinante Plasmid und wird deshalb in einer Nährflüssigkeit vermehrt (4). unmethylierte DNA G C 5’ EcoR I A T C T G G 3’ C T T A A G A A T T C C T T A A G Ligase 5’ 3’ Restriktionsenzym CH3 5’ 3’ G A A T T C C T T A A G EcoR I methylierte DNA CH3 EcoR I kann die methylierte DNA an den Schnittstellen nicht schneiden. 107.1 Wirkungsweise von Restriktionsenzymen und Ligase Plasmide Gen für Ampicillin-Resistenz Gen für Tetracyclin-Resistenz Restriktionsenzym Fremd-DNA DNA-Ligase Transformation von E.-coli-Zellen DNA der Wirtszelle 1 Kolonie fehlt Abdruck mit Samtstempel 2 3 Vergleich Nährboden mit Ampicillin Nährboden mit Tetracyclin 4 Vermehrung in Nährflüssigkeit 107.2 Prinzip der Selektion Gentechnik 107