Nukleotid Biosynthese
Werbung

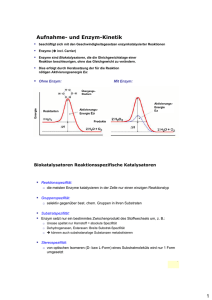
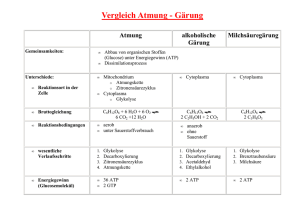
Nucleotid Biosynthese Nucleotide kennen wir als Bausteine der DNA. Doch im Grunde erfüllen sie noch zahlreiche andere Aufgaben: - Aktivierte Vorstufen von Nukleinsäuren, wichtig für Replikation des Genoms & Transkription in RNA - ATP (ein Adeninnucleotid) als universeller Form biologisch verwendbarer Energie - GTP (ein Guaninnucleotid) als Energiequelle für spezifische biol. Prozesse - UDP-Glucose (ein Nucleotidderivat) für Biosynthesen, z.B. Glykogen-Synthese - Wichtig für Signaltransduktion. z.B. cAMP und cGMP als second messenger - ATP dient zudem als Phosphatdonor bei der Proteinkinase Nucleotide sind aufgebaut aus je einem Basenring (Purin bzw. Pyrimidin), einem Zucker (Ribose für RNA, Desoxyribose für DNA) und einer Phosphatgruppe (ohne Phosphatgruppe: Nucleosid). Die Ringsysteme der Nucleotide werden an den Aminosäuren (AS) Glycin und Aspartat aufgebaut. Aspartat dient zudem, zusammen mit Glutamin, als Ammoniakquellen, welche bei der Biosynthese benötigt werden. Der Prozess der Nucleotid-Biosynthese ist äusserst wichtig für die Medizin und Pharmazie. Viele Krebstherapeutika greifen genau diesen Prozess an und verhindern somit die Zellteilung von aus der Kontrolle geratener Zellen. Die Nucleotid-Biosynthese kann in zwei Formen unterteilt werden: - de novo-Synthese - Recycling (engl: „salvage pathways““) Bei de novo-Synthesen werden Nucleotidbasen aus einfacheren Verbindungen hergestellt. Dabei unterscheiden sich noch der Aufbau bei einer Pyrimidinbase und der einer Purinbase: - Pyrimidinbase wird zuerst aufgebaut und anschliessend an einer Ribose angehängt - Purinbase wird direkt an einer Ribose Stück für Stück aufgebaut. Bei der Recycling werden – wie der Name schon sagt – bestehende Basen wiederverwertet und an eine Riboseeinheit angehängt. Durch diese beiden Synthesen entstehen aber erst die Ribonucleotiden, welche Bestandteile der RNA, aber nicht der DNA sind. Die Ribosen werden also danach zuerst zu Desoxyribosen reduziert, bevor sie für die DNA benötigt werden können. Nomenklatur Namen der Basen, Nucleoside und Nucleotide: Base Adenin / Adenin Guanin / Guanin Uracil / Thymin Cytosin / Cytosin RNA / DNA Ribonucleosid /Desoxyr. Ribonucleotid / Desoxyribonucleotid Adenosin / Desoxyadenosin Adenylat (AMP) / Desoxyadenylat (dAMP) Guanosin / Desoxyguanosin Guanylat (GMP) / Desoxyguanylat (dGMP) Uridin / Thymidin Uridylat (UMP) / Thymidylat (TMP) Cytidin / Desoxycitidin Cytidylat (CMP) / Desoxycytidylat (dCMP) Synthese von Pyrimidinbasen Die Pyrimidinringe werden aus Hydrogencarbonat, Asparaginsäure und Glutamin synthetisiert, wobei Glutamin als NH3-Lieferant dient. Aus Hydrogencarbonat und Ammoniak wird Carbamoylphosphat hergestellt Die Reaktion wird mit dem Enzym Carbamoylphosphat-Synthetase (CPS) katalysiert. Dieses Enzym hat insgesamt 3 aktive Zentren. Im ersten Zentrum – ATP-„Greif“-Falte genannt (ATP-grasp fold) – welches sich am N-Terminus befindet, wird Hydrogencarbonat phosphoryliert zu Carboxyphosphat. Somit wird das Molekül aktiviert (Phosphorylierung ist die häufigste Methode, um inaktive Stoffe für biol. Vorgänge zu aktivieren). Anschliessend greift das NH3 am Carboxyphosphat an und bildet Carbaminsäure. Im zweiten ATP-„Greif“-Falte wird das Carbaminsäure wiederum phosphoryliert zu Carbamoylphosphat. Das dritte aktive Zentrum von diesem Enzym hat die Aufgabe, Glutamin durch Hydrolyse in Ammoniak und Glutamat aufzuspalten. Das Ammoniak wird anschliessend für die Herstellung von Carbaminsäure benötigt. Diese Domäne enthält einen Cystein- und einen Histidinrest und ist auch bei der CTP- und GMP-Synthetase zu finden. Zwischenprodukte erreichen die aktiven Zentren durch einen Kanal Die drei aktive Zentren sind durch einen Kanal miteinander verbunden. Zwischenprodukte wandern von einem Zentrum zum anderen, ohne das Enzym zu verlassen. Das Ammoniak, das beim dritten Zentrum hergestellt wird, wandert zum Carbaminsäure-Zentrum, und weiter zum Carbamoylphosphat-Zentrum. Das Kanalsystem hat den Vorteil, dass die Zwischenprodukte auch wirklich für die Reaktion zur Verfügung stehen und nicht „abhauen“, ausserdem bleiben die labilen Zwischenprodukte vor der Hydrolyse geschützt. Uridylat-Synthese Durch eine Reaktion zwischen Carbamoylphosphat und Aspartat entsteht Carbamoylaspartat (beteiligtes Enzym: Aspartat-Transcarbamoylase), welches zum cyclischen Dihydroorotat umgewandelt wird. Dieses wird durch NAD+ zu Orotat oxidiert. Orotat reagiert mit 5Phosphoribosyl-1-pyrophosphat (PRPP), eine aktivierte Riboseform, zum Orotidylat, ein Pyrimidinnucleotid. Die Reaktion wird durch die Pyrimidin-Phosphoribosyltransferase katalysiert, welches eine Hydrolyse des Pyrophosphats bewirkt. Das Orotidylat wird zm Uridylat (UMP) decarboxyliert. (beteiligtes Enzym: Orotidylat-Decarboxylase) - Carbamoylphosphat + Aspartat Carbamoylaspartat Dihydroorotat (cyclisch) Orotat - Orotat + PRPP Orotidylat Uridylat Orotidylat-Decarboxylase ist eines der effektivsten Enzyme, die man zur Zeit kennt. Ohne dieses Enzym würde die Decarboxylierung nämlich 1017 mal langsamer ablaufen (von 1x alle 78 mio. Jahre auf 1x/sek.)! Cytidin-Synthese Für die Synthese von CTP muss zuerst UMP in UTP umgewandelt werden. Dies geschieht durch zwei Enzyme: zum einen die Nucleosidmonophosphat-Kinase und, logisch abgeleitet, die Nucleosiddiphosphat-Kinase. Für die erste Phosphorylierung wird eine Phosphatgruppe des ATP benutzt. Das zweite Kinasenenzym ist allerdings viel unspezifischer. Es kann Phosphatgruppen von beliebige Triphosphate nach Diphosphate transferieren. XDP + YTP XTP + YDP X,Y: beliebiges (Dexoxy-) Ribonucleotid Nach der UTP-Synthese wird eien Carbonylgruppe durch eine NH2-Gruppe ersetzt. Die Reaktion verläuft analog der einer Carbamoylphosphat-Synthese. Zuerst wird ein O-Atom phosphoryliert, danach wird die Phosphatgruppe durch ein Ammoniak ersetzt, welches von einem Glutamin geliefert wird. Das daraus entstehende CTP kann nun bei der RNA-Synthese eingesetzt weden. Synthese von Purinbasen Während Pyrimidinbasen nur durch de novo-Synthesen hergestellt werden können, können Purinbasen sowohl de novo synthetisiert als auch durch recycelt werden. Recycling Freie Purinbasen können mit PRPP Purinnucleosidmonophosphate bilden. Die Reaktion ist analog der Orotidylatbildung. Es gibt zwei Recyclingenzyme, welche Purinbasen wiederverwenden können: - Adenin-Phosphoribosyltransferase katalysiert die Bildung von Adenylat - Hypoxanthin-Guanin-Phosphoribosyltransferase (HGPRT) hingegen ist für die Bildung von Guanylat und Inosinat zuständig (Inosinat = IMP = Inosinmonophosphat, IMP ist eine Vorstufe von Guanylat und Adenylat) De novo-Synthesen von Purinringsysteme Im Gegensatz zur Pyrimidin-Synthese wird der Purinring direkt Schritt für Schritt am Ribosephosphat aufgebaut. Das Pyrophosphat des PRPP wird durch eine Aminogruppe ersetzt. Dabei entsteht 5-Phosphoribosyl-1-amin mit -Konfiguration in der Aminogruppe. Enzym: GlutaminPhosphoribosyl-Amidotransferase. Das Enzym besitzt zwei aktive Zentren. Das erste ist homolog mit dem der Phosphoribosyltransferase, Das zweite erzeugt eine Glutaminhydrolyse, welche Ammoniak für den weiteren Reaktionsverlauf bereitstellt. Anders als bei der Carbamoylphosphat-Synthetase katalysiert ein Cysteinrest am N-Terminus die Glutaminhydrolyse. Das daraus entstandene Ammoniak wird aber auch in diesem Fall durch ein Kanal zum PRPP transportiert. Dieses Enzym wird nur bei der Bildung von PRPP und Glutamin aktiviert. Aufbau des Purinringes Weitere 9 Schritte sind für die Purinring-Synthese erforderlich. Dabei sind die ersten 6 Schritte analog. In jedem Schritt wird ein Carbonylsauerstoffatom druch Phosphorylierung und anschliessend durch ein Ammoniak oder eine Aminogruppe substituiert. 1. Glycin wird ans 5-Phosphoribosyl-1-amin gehängt Glycinamidribonucleotid 2. Ein aktivertes Formiat bindet sich an das Glycinamidribonucleotid Formylglycinamidribonucleotid 3. Die innere Amidgruppe wird mit einer Aminogruppe vom Glutamin verbunden Amidinrest Formylglycinamidinribonucleotid 4. Formylglycinamidinribonucleotid wird zyklisiert 5-Ring (5Aminoimidazolribonucleotid), Reaktion ist irreversibel! 5. Die exozyklische Aminogruppe greift ein Hydrogencarbonat an Umlagerung der Carboxylatgruppe auf Imidazolring Carboxyaminoimidazolribonucleotid 6. Ein Aspartat wird ans Carboxyaminoimidazolribonucleotid gehängt 5-Aminoimidazol-4-(N-succinylcarboxamid-)ribonucleotid Es werden also laufend Glycin, Formiat, Ammoniak, Hydrogencarbonat und Aspartat mithilfe einer analogen Reaktionsmechanismus ans Phosphoribosylamin gehängt. 7. Fumatat wird vom 5-Aminoimidazol-4-(N-succinylcarboxamid-)ribonucleotid abgespalten 5-Aminoimidazol-4-carboxamidribonucleotid 8. Eine Formylgruppe wird auf die Verbindung übertragen 5-Formaminoimidazol-4carboxamidribonucleotid (vgl. Schritt 2) 9. Durch Abspaltung eines H20-Moleküls zyklisiert das letzte Zwischenprodukt zu Inosinat (IMP), welches nun für die weitere Synthese zum Adenylat und Guanylat eingesetzt werden kann (Graphische Darstellung der Reaktionsschritte: Abb. 25.7 und 25.8 im Stryer) AMP und GMP entstehen aus IMP - IMP AMP: Ein Carbonylsauerstoffatom wird durch eine Aminogruppe substituiert. Mechanismus: Addition eines Aspartats und anschliessender Elimination eines Fumarats (vgl. Schritt 6 & 7 der IMP-Synthese). Unterschied: Als Energielieferant dient nicht ATP, sondern GTP. Das beteiligte Enzym (Adenylsuccinat-Synthase) hat keine ATP-„Greif“-Domäne. Übrigens wird dieses Enzym auch für Schritt 7 der IMP-Synthese eingesetzt IMP + GTP + Asp Adenylsuccinat + GDP + Pi Adenylsuccinat AMP + Fumarat - IMP GMP: Addition von einem NH2-Rest am IMP. Mechanismus: Oxidation von IMP mithilfe von NAD+ Xanthylat. Das Sauerstoffatom des Xanthylats wird durch eine Aminogruppe substituiert GMP. Für diese Reaktion ist im Gegensatz zur AMP-Synthese ATP erforderlich. IMP + NAD+ + H2O Xanthylat + NADH + H+ Xanthylat + NH3 + ATP GMP + AMP + PPi Radikalische Reduktion von Ribonucleotide zu Desoxyribonucleotiden Prinzip : Substrat: Red.mittel: Enzym: Die 2’-Hydroxylgruppe der Riboseeinheit wird durch ein H-Atom ersetzt. Ribonucleosiddi- oder –triphosphate, NADPH Ribonucleotid-Reduktase (können z.T. ganz anders aussehen bei verschiedenen Organismen. Der Reaktionsmechanismus ist jedoch derselbe) Mechanismus beim E.coli: Der Riboncleotid-Reduktase der E.coli besteht aus zwei Untereinheiten: R1 und R2. R1: 1 aktives Zentrum und 2 allosterische Kontrollstellen (3 Cystein- + 1 Glutamatrest) R2: 2 Ketten mit je ein stabiles Tyrosylradikal Reaktionsschrittte: 1. 1 Elektron wandert vom Cysteinrest in R1 nach Tyrosylradikal im R2 Cysteinradikal 2. Cysteinradikal nimmt ein H vom C-3’ der Riboseeinheit Radikal am Ribose-C-3’ 3. Ein OH- am C-2’ wird von einem zweiten Cysteinrest protoniert und abgespalten Radikal am Ribose-C-2’ 4. Ein Hydridion (1 Proton mit 2 Elektronen) wandert von einem dritten Cysteinrest nach C-2’ der Riboseeinheit S-S-Brücke und wieder freies Radikal am C-3’ 5. C-3’-Radikal holt H vom ersten Cysteinrest wieder zurück (vgl Schritt 2) Ende der Reduktion 6. Disulfidbrücke der R1-Untereinheit wird gespalten, Enzym wird regeneriert Thymidylat-Synthese Während alle andere Ribonucleotide durch einfache Reduktion in Desoxyribonucleotide überführt werden können, sieht das beim Uracil ein bisschen anders aus. Das Analogon vom Uracil in der DNA ist Thymin, welches eine zusätzliche Methylrest enthält. Dieses Methylrest erleichtert die Identifizierung und Reparatur von DNA-Schäden (Wenn bei der Replikation etwas schief läuft, kann das Reparatursystem den Strang mit Methylgruppen als „Original“ erkennen. Vgl. Kapitel 27). Mechanismus: Desoxyuridylat (dUMP) wird zu Thymidylat (TMP) methyliert. Als Methyldonor dient das N5-N10-Methylentetrahydrofolat, welches zu Dihydrofolat oxidiert wird. Das beteiligte Enzym heisst Thymidylat-Synthase. Reaktion: Ein Thiolat wird ans Thymidylat-Synthase angefügt. Dieser bindet sich anschliessend ans dUMP Aktivierung von dUMP nukleophiler Angriff am Methyldonor Abspaltug einer Methylgruppe vom Methylentetrahydrofolat Abtrennung der EnzymThiolat-Verbindung (Methylentetrahydrofolat besitzt ein Fünfring mit einer Methylengruppe. Die Enzym-ThiolatVerbindung bewirkt die Öffnung des Fünfrings. Die Methylengruppe wird durch die Übertragung eines Hydridions in eine Methylgruppe umgewandelt, welches nun vom aktivierten dUMP angegriffen werden kann) Damit neues Thymidylat hergestellt werden kann, muss das Tetrahydrofolat wieder regeneriert werden. NADPH dient hier als Reduktionsmittel, und katalysiert wird diese Reaktion von der Dihydrofolat-Reduktase. Das daraus entstehende Tetrahydrofolat kann nun in weiteren Schritten wieder in N5-N10-Methylentetrahydrofolat umgewandelt werden. Viele Krebshemmende medikamente blockieren die Thymidylat-Synthese Zellen mit hoher Teilungsrate brauchen viel mehr Thymidylat zur DNA-Synthese und reagieren deshalb besonders empfindlich auf Blockierung der TMP-Synthese. Bei der Chemotherapie sind Thymidylat-Synthase und Dihydrofolat-Reduktase bevorzugte Angriffspunkte: Fluoruracil wird in vivo in Fluordesoxyuridylat (F-dUMP) umgewandelt. Dieses dUMPAnalogon hemmt die Thymidylat-Synthase irreversibel, da sich das Dihydrofolat wegen der hohen Elektronegativität des Fluors nicht vom F-dUMP lösen kann. Die Reaktion wird also auf der Stufe des F-dUMP-Methylentetrahydrofolat-Thiolat-Enzym-Verbindung blockiert. Aminopterin und Methotrexat hemmen die Tetrahydrofolatregenerierung. Sie werden bei schnell wachsende Tumoren eingesetzt wie z.B. akute Leukämie. Der Nachteil dieser Medikamente ist, dass alle schnellwachsnde Zellen angegriffen werden, also auch gesunde Knochenmarkstammzellen, Epithelzellen des Intestinaltraktes und Haarfollikel, was zu Nebenwirkungen wie geschwächtes Immunsystem, Brechreiz und Haarausfall führen. Trimethoprim bindet sich sehr stark an die Dihydrofolat-Reduktase von Bakterien und Protozoen, während sie an jene von Säugern 105-mal schwächer binden. Der Grund dafür sind kleine Konfigurationsunterschiede der Enzyme. Wegen dieser Eigenschaft wird es häufig bei der Infektionsbehandlung eingesetzt. Regulierung der Nucleotidbio-Synthese durch Rückkopplungsmechanismen Die Purinnucleotid-Synthese wird an folgende Stellen durch Rückkopplung kontrolliert: - Umwandlung von PRPP in Phorphoribosylamin, welches am Anfang der Purinribonucleotid-Synthese stattfindet. Gehemmt wird der Vorgang durch AMP und GMP, welche synergistisch (=sich in ihrer Wirkung verstärkend) wirken - AMP hemmt die Umwandlung von IMP in Adenylsuccinat, welches sein direkter Vorläufer ist. Analog hemmt GMP die Umwandlung von IMP in Xanthylat - Wir wissen, dass bei der AMP-Synthese GTP benötigt und bei der GMP-SyntheseATP eingesetzt wird (vgl. Seite 4). Diese reziproke Substratbeziehung sorgt für ein Gleichgewicht in der AMP und GMP-Produktion Die Reduktion von Ribonucleotide zu Desoxyribonucleotide erfolgt über allosterische Wechselwirkungen. Die R1-Untereinheit der Ribonucleotid-Reduktase von E.coli hat 2 allosterische Zentren (vgl. Seite 5): Eines kontrolliert die Gesamtaktivität des Enzyms, das andere die Substratspezifität. - Überschuss an Desoxyribonucleotiden dATP Hemmung der PurinnucleotidSynthesen Pyrimidinnucleotid-Produktion begünstigt Überschuss an Pyrimidindesoxynucleotiden TTP Hemmung der Pyrimidinnucleotid-Reduktion + Förderung der GDP-Reduktion dGTP-Spiegel steigt stimuliert Reduktion von ATP zu dATP NAD+, FAD und Coenzym A werden aus ATP gebildet NAD+-Synthese: Nicotinat + PRPP Nicotinatribonucleotid + PPi Nicotinatribonucleotid + ATP Desamide-NAD+ + PPi Desamide-NAD+ + Glutamin NAD+ + Glutamat Das Nicotinat bindet sich ans PRPP, wobei zwei Phosphatgruppen von PRPP abgespalten werden. Anschliessend wird ein AMP-Rest des ATP auf das daraus entstandene Nicotinatribonucleotid übertragen. Dieses Desamido-NAD+ enthält eine Carboxylgruppe, dessen Sauerstoffatom im letzten Schritt durch eine Aminogruppe ersetzt wird. Das Ammoniak, das dafür benötigt wird, stammt (wie immer) aus Glutamin. (Nicotinat, auch Niacin oder Vitamin B6, leitet sich von Tryptophan ab. Ein Tryptophan- und somit Nicotinatmangel kann zu Pellagra führen. Symptome der Pellagra sind: Dermatitis, Diarrhoe und geistiger Verwirrung) FAD-Synthese: Riboflavin + ATP Riboflavin-5’-phosphat + ADP Riboflavin-5’-phosphat + ATP Flavinadenindinucleotid (FAD) + PPi Eine Phosphatgruppe des ATP wird auf Riboflavin übertragen. Daraus entsteht Riboflavin-5’phosphat (= Flavinmononucleotid FMN). Dieser wird anschliessend mit einer AMP-Einheit eines zweiten ATP zu FAD verbunden. Coenzym A-Synthese: Auch hier wird eine AMP –Einheit aus ATP auf die Phosphatgruppe eines Phosphorylierten Zwischenprodukts übertragen. Daraus entsteht ein Pyrophosphat, welcher dann zu Orthophosphat hydrolysiert wird. Purine werden im Menschen zu Urat abgebaut Die Nucleotide in unseren Zelle werden laufend erneuert. Sie werden durch Nucleotidasen zu Nucleosiden abgebaut und weiter durch Nucleosid-Phosphorylasen in freie Basen und (Desoxy-)Ribose-1-phosphat zerlegt. Ribose-1-phosphat wird durch Ribosephosphat-Mutase in Ribose-5-phosphat umgewandelt, welches für die PRPP-Synthese genutzt werden kann. Einige Basen werden recycelt, andere weiter abgebaut und ausgeschieden. Z.Bsp. wird AMP zu Hypoxanthin abgebaut, welches von Xanthin-Oxidase zu Xanthin und weiter zu Urat (Salz der Harnsäure) oxidiert wird, Dieses kann nun ausgeschieden werden. Ein zu hoher Uratkonzentration im Serum führt zu Gicht (Urat kristallisiert im Körper und schädigt Gelenke und Nieren). Zur Behandlung wird manchmal Allopurinol, ein XanthinOxidase-Inhibitor, eingesetzt. (Der Serumspiegel des Urats beim Menschen liegt gerade unter der Löslichkeitsgrenze, während er bei Halbaffen 10x tiefer liegt. Dies erklärt vielleicht die höhere Lebenserwartung und die geringere Krebshäufigkeit der Menschen im Gegensatz zu anderen Primaten, denn Urat ist, wie die Ascorbinsäure (Vitamin C), ein hervorragender Antioxidans) Das Lesch-nyhan-Syndrom (LNS) Bei Lesch-Nyhan-Syndrom handelt es sich um eine X-Chromosomal rezessiv vererbbare Genmutation, welche zur Folge hat, dass das Recyclingenzym Hypoxanthin-GuaninPhosphoribosyltransferase praktisch ganz fehlt (vgl. Seite 3). Betroffene haben einen zwanghaften Hang zur Selbstzerstörung. Sie knabbern unkontrolliert an Finger und Lippen und würden sie abkauen, wenn man sie nicht daran hinderte. Auch anderen gegenüber verhalten sie sich manchmal aggressiv. Bei schwereren Formen kommen noch geistige Behinderung und spastisches Verhalten dazu. Durch das Fehlen der Hylpoxanthin-Guanin-Phosphoribosyltransferase ist eine erhöhte PRPPKonzentration, eine höhere Geschwindigkeit der de novo-Purinbiosynthese und eine Überproduktion von Urat zu kennzeichnen, was schon früh zu Bildung von Nierensteinen und Symptomen von Gicht führen kann. Der genaue Zusammenhang zwischen dem Fehlen der Transferase und den neurologischen Symptomen ist bislang unbekannt. Doch zeigt diese Krankheit, dass der Weg der Recycling für IMP und GMP wahrscheinlich nicht ohne Grund gewählt wurde.