ppt
Werbung

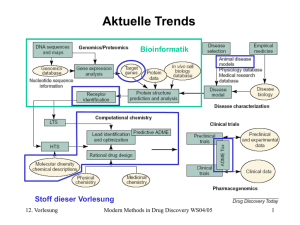
Target Identification and Animal Models Das menschliche Genom könnte nach optimistischen Schätzungen 5000 bis 10000 neue drug targets enthalten. Alle jemals eingesetzten Medikamente zielten auf etwa 500 targets auf molekularer Ebene ab. Aktuell kommen auf alle Medikamente am Markt nur 120 targets. Die 100 meist verkauften Medikamente zielen gar nur auf 43 Proteine ab. Gibt es also nur wenige sog. valid targets ? Gibt es zu wenig Information über sog. drugable targets ? 9. Vorlesung Modern Methods in Drug Discovery WS04/05 1 Innovation vs. „me too“ Neue chemische Verbindungen (new molecular entities) und neuartige targets COX2 Arthritis celecoxib PDE5 Erectile dysfunction sildenafil BCR-ABL Leukämie imantinib Die meisten NMEs zielen auf bereits bekannte targets ab. Aber: müßen besser als bestehende Verbindungen sein, um zugelassen zu werden. Lit: B.P.Zambrowicz & A.T.Sands Nature Rev.Drug Disc. 2 (2003) 38 9. Vorlesung Modern Methods in Drug Discovery WS04/05 2 Typische Targets im Genom Anteil am menschlichen Genom und Pharmaka am Markt Bisher etwa 500 Enzyme als targets benutzt. Möglicherweise 10.000 potentielle targets im Genom 9. Vorlesung Modern Methods in Drug Discovery WS04/05 3 Typische Targets drug targets by biochemical class Enzymes 47% GPCRs 30% Ion Channels 7% DNA 1% Intergrins 1% Miscellaneous 2% Other Receptors 4% Transporters 4% Nuclear Receptors 4% Aufteilung der auf dem Markt befindlichen Pharmaka bezüglich ihrer biochemischen targets Quelle: Hopkins & Groom, Nat.Rev.Drug.Disc. 1 (2002) 727 9. Vorlesung Modern Methods in Drug Discovery WS04/05 4 Validierung von targets Wann ist ein auf genetischer Ebene identifiziertes target tatsächlich auch brauchbar ? Exprimierierung Disease model defined physiological and clinical endpoints Animal model Es muß geklärt werden ob sich das target als therapeutic target eignet und so ein valid target ist. An dieser Stelle setzen dann Proteomics, Metabolomics und Pharmacogenetics / genomics an. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 5 Informationsfluß in einer drug discovery pipeline Bioinformatik 9. Vorlesung Modern Methods in Drug Discovery WS04/05 6 Auf dem Weg zum target (I) Für den Fall einer bekannten Krankheit ist die Identifizierung eines geeigneten targets ein konvergenter Prozeß Lit: M.A.Lindsay Nature Rev.Drug Disc. 2 (2003) 831 9. Vorlesung Modern Methods in Drug Discovery WS04/05 7 Auf dem Weg zum target (II) RNA target protein Exprimierung Modifikationen DNA Verwendete Techniken zur Identifizierung von targets Lit: M.A.Lindsay Nature Rev.Drug Disc. 2 (2003) 831 9. Vorlesung Modern Methods in Drug Discovery WS04/05 8 Auf dem Weg zum target (III) Forward genetics: Screening von Verbindungen gegen Variationen des Phänotyps und Mutationen Lit: M.A.Lindsay Nature Rev.Drug Disc. 2 (2003) 831 9. Vorlesung Modern Methods in Drug Discovery WS04/05 9 Auf dem Weg zum target (IV) orthologues genes Identified gene Animal model Reverse genetics: Veränderung des Genotyps durch selektive Mutationen Lit: M.A.Lindsay Nature Rev.Drug Disc. 2 (2003) 831 9. Vorlesung Modern Methods in Drug Discovery WS04/05 10 Auf dem Weg zum target (V) Der Bioinformatische Ansatz zu neuen targets im Idealfall (Analysten Scenario) In der Praxis stellt sich vor allem die Frage: „Nach welchen Genen muß man suchen ?“ Lit: A.T. Sands Nature Biotech. 21 (2003) 31 9. Vorlesung Modern Methods in Drug Discovery WS04/05 11 Nach was muß man also im Genom suchen? → Ähnlichkeiten mit bereits verwendeten targets Die Chance, innovative targets zu finden sollte die Suche nach den folgenden targets bieten, die bisher unterrepräsentiert sind: Kinasen und Proteasen Transmembranproteine (GPCRs, Ionenkänale, Transporter) DNA, RNA Bindungsstellen Nukleare Rezeptoren (für Hormone) (insb. orphan nuclear receptors siehe Vorlesung 10, bisher nur wenige neue gefunden) Nach vorsichtigen Schätzungen sollten sich etwa 100-150 neue und „wertvolle“ targets (vaild, drugable) finden lassen. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 12 Target validation Wann ist ein target für therapeutische Zwecke tauglich, also valid ? Wenn es in hinreichend kausalem Zusammenhang mit der Krankheit steht: a) Als Enzym, GPCR, Ionenkanal, Rezeptor usw. Überprüfung durch screening mit Leitstrukturen aus focused libraries b) Als target auf DNA, RNA, mRNA Ebene selber Überprüfung durch knockout Mutationen (siehe unten), single point Mutationen (SNPs, siehe unten), und gene silencing mittels durch RNA interference (RNAi) (siehe siRNA) 9. Vorlesung Modern Methods in Drug Discovery WS04/05 13 siRNA for target validation Kurze RNA-Stränge von 11 bis 28 Nucleotiden können an komplementäre mRNA binden und zur deren Degradation durch RNAsen führen. Diese RNA Interferenz (RNAi) dient in Eukaryoten zur Abwehr von viraler RNA. Daraus leitet sich auch deren Bezeichnung als small interfering RNA (siRNA) ab. Dieser Effekt kann sowohl zum Stillegen von mRNA genutzt werden (gene silencing) als auch zum Aufspüren potentieller targets auf mRNA Ebene. Der therapeutische Einsatz von siRNAs ist durch deren Stabilität (Applikationsform) und Selektivität (unspezifische Bindung) begrenzt. Lit: M.A. Lindsay Nature Rev. Drug Disc. 2 (2003) 831. Y.Dorsett & T.Tuschl ibid 3 (2004) 318. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 14 target characterization Im gesammten (menschlichen) Genom treten Variationen auf. Statistisch gesehen 1 Basenpaar pro 1330 Basenpaare macht etwa 3 ∙106 Unterschiede zwischen zwei nicht verwandten Individuen. Auch in den Genabschnitten die tatsächliche oder potentielle targets kodieren, kommen durchschnittlich mehr als 9 solcher Basenaustauschungen vor. Daraus folgt: 1. Nicht jede Variation ist ein Defekt oder bedeutet eine Prädisposition (für eine Krankheit) 2. Die Auswahl potentieller targets wird nicht einfacher 9. Vorlesung Modern Methods in Drug Discovery WS04/05 15 Pharmacogenetics & Pharmacogenomics Die kausale Zuordnung eines klinischen Phänotyps (Allel bzw. Krankheitsbild) zu einer genetischen Ursache wird durch die Vielzahl der möglichen / vorhandenen Variationen des Genotyps erschwert. Als Polymorphismus bezeichnet man Allele die bei mindestens 1% der Population auftreten. D.h. diese Genotypen kommen regulär vor. Im Gegensatz dazu bezeichnet man Veränderungen des Genoms die seltener als 1% auftauchen nur als Mutationen. → Sequenzierung der (in Frage kommenden) Genabschnitte bei möglichst vielen Individuen. Lit: D.B. Goldstein et al. Nature Rev. Genetics 4 (2003) 937. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 16 Single Nucleotide Polymorphism SNPs sind Unterschiede einer einzelnen Base in der DNA die innerhalb einer Population auftauchen. Die Wahrscheinlichkeit durch Sequenzierung SNPs bestimmter Häufigkeit zu finden läßt sich wie folgt abschätzen: Anzahl Individuen 2 5 10 20 40 >1% SNP Häufigkeit >2% >5% >10% >20% 4% 10% 18% 33% 55% 8% 18% 33% 55% 80% 59% 89% 99% >99% >99% 19% 40% 64% 87% 98% 34% 65% 88% 99% >99% Quelle: J.J. McCarthy „Turning SNPs into Useful Markers of Drug Response“ in Pharmacogenomics, J.Licinio & M.-L.Wong (Eds.), Wiley-VCH (2002) pp.35-55. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 17 Multiple SNPs Noch schwieriger ist die kausale Zuordnung einer Reaktion auf ein Medikament, wenn verschiedene von einander unabhängige SNPs vorhanden sind. D.h. wenn keine schlüßige Hypothese vorhanden ist. Die Zahl der zu sequenzierenden Genabschnitte kann dadurch undurchführbar groß werden. Als Beispiele für sog. valid biomarkers hat die US FDA bisher nur den Polymorphismus von CYP2D6 (Cytochrome P450) und TPMT (Thiopurine S-methyl-transferase) herausgestellt. Beide Enzyme sind maßgeblich an der metabolischen Umsetzung vieler Medikamente beteiligt. Mehr zum Polymorphismus von CYP2D6 in Vorlesung 10 Lit. P.C.Sham et al. Am.J.Hum.Genet. 66 (2000) 1616. R.Weinshilboum & L.Wang Nature Rev.Drug Discov. 3 (2004) 739. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 18 Sukzesible Gene Bisher hat man sukzesible Gene im Zusammenhang mit folgenden Krankheitsbildern identifiziert: Herztod Neurodegenerative Krankheiten (Demenz, Alzheimer,...) Epilepsie Schizophrenie Diabetes Arthritis Lungenkrankheiten (Cystische Fibrose) Übergewicht Lit. V.D.Schmith et al. Cell.Mol.Life Sci. 60 (2003) 1636. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 19 Gene Candidate Studies Prinzipielle Vorgehensweise bei potentiellen Gen-Kandidaten Auswahl des pharmazeutischen target Gens, entweder bekanntes target (Enzym, Transporter, pathogenes Gen,..) oder neu identifiziertes Gen aus DNA-Microarrays (auf mRNA Ebene), Proteomics (auf Proteinenebene), Bioinformatik Identifizierung von SNPs im ausgewählten Gen durch SNP-Mapping in größerem Maßstab, Bestimmung der AllelHäufigkeit und ethnischen Verteilung, Analyse der Haplotypen Genotypisierung von SNPs in klinischen Studien Identifizierung der Patientenpopulation, Statistische Analyse Lit. H.Z.Ring & D.L.Kroetz Pharmacogenomics 3 (2002) 47-56. Besonders empfehlenswerter Review 9. Vorlesung Modern Methods in Drug Discovery WS04/05 20 Warum Tiermodelle ? • Zur in vivo Verifikation des Krankheitsmodelles • Zum in vivo screening 9. Vorlesung Modern Methods in Drug Discovery WS04/05 21 Modellorganismen Bevor man Mäuse und andere Säugetiere zum in vivo screening verwendet, werden andere Modellorganismen herangezogen die über entsprechende orthologe Gene verfügen. Größere Zahl an zum Menschen orthologer Gene Zunehmend komplexere Organismen Steigender Aufwand und Kosten bei Experimenten Literatur : R. Knippers Molekulare Genetik 8. Auflage S. 498-503 Modellorganismen, Knockout Technologie 9. Vorlesung Modern Methods in Drug Discovery WS04/05 22 Was leisten animal models ? Zur in vivo Überprüfung eines Krankheitsmodelles können Tiermodelle hilfreich sein. 1. Vergleich des targets in Tier- und menschlichem Genom. 2. Erzeugung von knockout Mutanten / transgenen Tieren Das Vorhandensein eines adäquaten Tiermodelles ist praktisch immer die Voraussetzung für die weitere Entwicklung zum Medikament. Literatur zu transgenen Mäusen: R. Knippers Molekulare Genetik 8. Auflage S. 522 Textbox Plus 18.2 9. Vorlesung Modern Methods in Drug Discovery WS04/05 23 Warum mus musculus als Tiermodell ? (I) • Zu 99% aller Mausgene konnten homologe bzw. orthologe Gene im Menschen identifiziert werden. Lit: Nature 420 (2002) Ausgabe 6915 vom 5.12.2002 Mouse Genome Sequencing Consortium ibid pp.520-562. Vergleich gemeinsamer Elemente in Mensch und Maus Chromosomen 9. Vorlesung Modern Methods in Drug Discovery WS04/05 24 Warum mus musculus als Tiermodell ? (II) • Damit sind von allen in Frage kommenden Modellorganismen Mäuse als Säugetiere am nächsten verwandt • Mäuse haben eine rasche Generationsfolge: Mäuse sind mit 10 bis 12 Wochen geschlechtsreif. 22 bis 24 Tage nach der Paarung kommen 4 bis 8 Junge zur Welt, und das 5 bis 6 Mal im Jahr. Eine einzige Maus kann im Jahr gut 40 Junge haben. • Die verwendeten Zuchtlinien sind genetisch gesehen relativ homogen (hoher Grad an Inzucht) • Bisher gelingt die Erzeugung homozygoter transgener Mäuse leichter als bei Ratten (Rattus norvegicus oder norwegicus) 9. Vorlesung Modern Methods in Drug Discovery WS04/05 25 KO-mouse models (I) Bedeutung von knockout Mausmodellen im pharmazeutischen Bereich: Medizinische Kategorie Umsatz (2001 in Mio.US$) Immunologie Neurologie/Psychatrie Cardiology Gastroenterologie Metabolismus Onkologie Hematologie 20 000 19 000 13 000 12 000 11 000 7 000 7 000 Anzahl targets 8 6 6 2 6 4 2 Anzahl Medikamente 15 13 13 6 10 8 3 Quelle: A.T.Sands Nature Biotech. 21 (2003) 31 9. Vorlesung Modern Methods in Drug Discovery WS04/05 26 KO-mouse models (II) Beispiele für den Einsatz von knockout Mausmodellen in erfolgreichen Medikamenten: Targets Medikament Maus Phänotyp zeigt: Protonenpumpe Lansoprazol Histamine H1-Rezeptor Famotidine neutraler Magen pH Magensäure Sekretion unterdrückt ACE AT1-Rezeptor Enalapril Losartan niedriger Blutdruck dito COX2 COX1 und COX2 Celecoxib Diclofenac verringerte Entzündungen weniger Schmerzen Lit: B.P.Zambrowicz & A.T.Sands Nature Rev.Drug Disc. 2 (2003) 38 9. Vorlesung Modern Methods in Drug Discovery WS04/05 27 Modellorganismen für Bluthochdruck Bei Mäusen ist Bluthochdruck praktisch nicht bekannt. Durch knock-in Mutationen übertrug man die Gene des Renin und Angiotensin Systems von Ratten auf Mäuse (vgl. Vorlesung 2) Lit: H.Ohkubo et al. Proc.Natl.Acad.Sci.USA 87 (1990) 5153. Umgekehrt zeigen knockout Mäuse bei denen das ACE Gen fehlt, niedrigen Blutdruck. Lit: J.H.Krege et al. Nature 375 (1995) 146. Da aber Ratten für funktionelle Studien besser geeignet sind, wurden auch transgene Ratten erzeugt, die das Ren-2 Gen erhielten und dadurch starke Symptome des Bluthochdrucks zeigten die wiederum mittels ACE-Inhibitoren und Angiotensin-II Antagonisten therapierbar waren. Lit: J.J.Mullins et al. Nature 344 (1990) 541. Lit: Li-Na Wei Annu.Rev.Pharmacol.Toxicol. 37 (1997) 119. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 28 Modellorganismen für Krebs In der Krebsforschung spielen zwei Bereiche eine wesentliche Rolle: Der Mechanismus der Entstehung von Krebs und die therapeutische Effizienz der verschiedenen Medikamente. Dabei wurden eine ganze Reihe transgener Mausmodelle entwickelt, die eine erhöhte Sukzeptibilität für bestimmte Krebsarten zeigen. Generell scheinen bei Mäusen aber Tumore sowieso die häufigste Todesursache zu sein, wenn man andere Faktoren bei der Haltung ausschließt. Auf die (ethische) Problematik der Patentierbarkeit transgener Tiere an sich (ohne Anwendungsbezug) sei an dieser Stelle nur hingewiesen. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 29 Weitere Tiermodelle Höhere Säugetiere wie Maus, Ratte, Kaninchen, Hund, Schwein werden häufig auch zum Test auf metabolische und toxikologische Eigenschaften von Verbindungen eingesetzt. Insbesondere der Vergleich von Screening Ergebnissen der metabolischen Umsetzung von Wirkstoffen mit denen aus in E. coli exprimierten CYP P450 Enzymen ist interessant, um das „geeignete“ Tiermodell auszuwählen. Zukünftig dürfen sich allein schon aus Kostengründen transgene Mäuse das bevorzugte Tiermodell bleiben. 9. Vorlesung Modern Methods in Drug Discovery WS04/05 30