Poster-DVG-Tagung2 1
Werbung
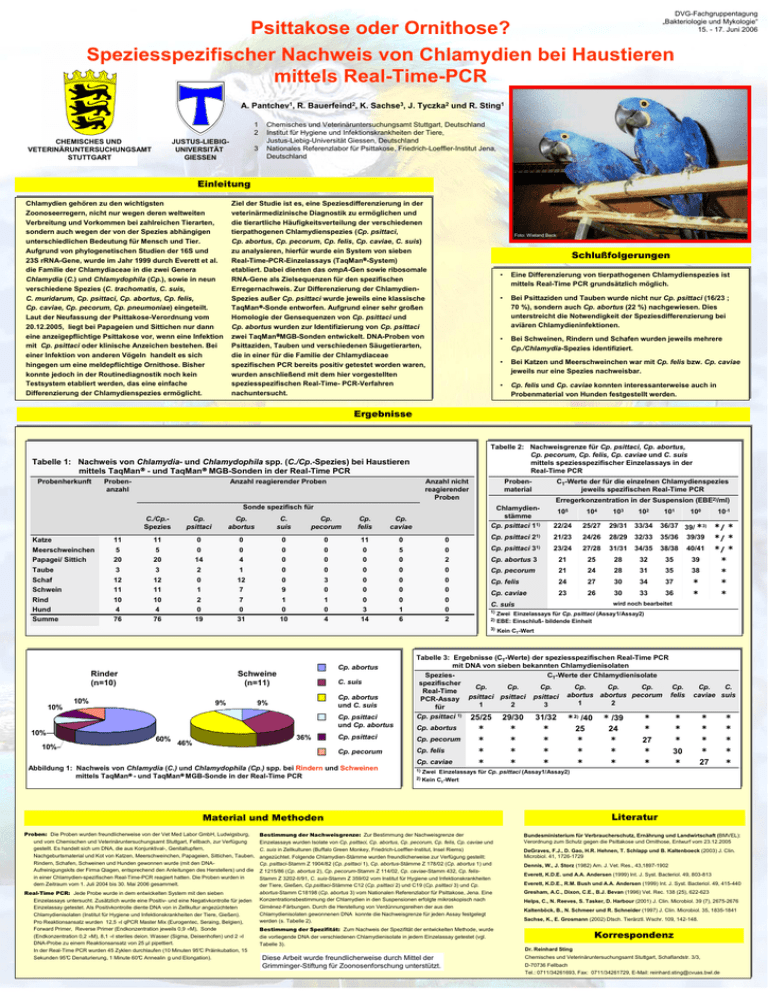
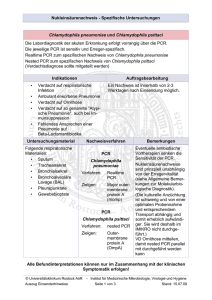
DVG-Fachgruppentagung „Bakteriologie und Mykologie“ 15. - 17. Juni 2006 Psittakose oder Ornithose? Speziesspezifischer Nachweis von Chlamydien bei Haustieren mittels Real-Time-PCR A. Pantchev1, R. Bauerfeind2, K. Sachse3, J. Tyczka2 und R. Sting1 1 2 JUSTUS-LIEBIGUNIVERSITÄT GIESSEN CHEMISCHES UND VETERINÄRUNTERSUCHUNGSAMT STUTTGART 3 Chemisches und Veterinäruntersuchungsamt Stuttgart, Deutschland Institut für Hygiene und Infektionskrankheiten der Tiere, Justus-Liebig-Universität Giessen, Deutschland Nationales Referenzlabor für Psittakose, Friedrich-Loeffler-Institut Jena, Deutschland Einleitung Chlamydien gehören zu den wichtigsten Zoonoseerregern, nicht nur wegen deren weltweiten Verbreitung und Vorkommen bei zahlreichen Tierarten, sondern auch wegen der von der Spezies abhängigen unterschiedlichen Bedeutung für Mensch und Tier. Aufgrund von phylogenetischen Studien der 16S und 23S rRNA-Gene, wurde im Jahr 1999 durch Everett et al. die Familie der Chlamydiaceae in die zwei Genera Chlamydia (C.) und Chlamydophila (Cp.), sowie in neun verschiedene Spezies (C. trachomatis, C. suis, C. muridarum, Cp. psittaci, Cp. abortus, Cp. felis, Cp. caviae, Cp. pecorum, Cp. pneumoniae) eingeteilt. Laut der Neufassung der Psittakose-Verordnung vom 20.12.2005, liegt bei Papageien und Sittichen nur dann eine anzeigepflichtige Psittakose vor, wenn eine Infektion mit Cp. psittaci oder klinische Anzeichen bestehen. Bei einer Infektion von anderen Vögeln handelt es sich hingegen um eine meldepflichtige Ornithose. Bisher konnte jedoch in der Routinediagnostik noch kein Testsystem etabliert werden, das eine einfache Differenzierung der Chlamydienspezies ermöglicht. Ziel der Studie ist es, eine Speziesdifferenzierung in der veterinärmedizinische Diagnostik zu ermöglichen und die tierartliche Häufigkeitsverteilung der verschiedenen tierpathogenen Chlamydienspezies (Cp. psittaci, Cp. abortus, Cp. pecorum, Cp. felis, Cp. caviae, C. suis) zu analysieren, hierfür wurde ein System von sieben Real-Time-PCR-Einzelassays (TaqMan-System) etabliert. Dabei dienten das ompA-Gen sowie ribosomale RNA-Gene als Zielsequenzen für den spezifischen Erregernachweis. Zur Differenzierung der ChlamydienSpezies außer Cp. psittaci wurde jeweils eine klassische TaqMan-Sonde entworfen. Aufgrund einer sehr großen Homologie der Gensequenzen von Cp. psittaci und Cp. abortus wurden zur Identifizierung von Cp. psittaci zwei TaqManMGB-Sonden entwickelt. DNA-Proben von Psittaziden, Tauben und verschiedenen Säugetierarten, die in einer für die Familie der Chlamydiaceae spezifischen PCR bereits positiv getestet worden waren, wurden anschließend mit dem hier vorgestellten speziesspezifischen Real-Time- PCR-Verfahren nachuntersucht. Foto: Wieland Beck Schlußfolgerungen Eine Differenzierung Differenzierung von von tierpathogenen tierpathogenen Chlamydienspezies Chlamydienspezies ist ist •• Eine mittels Real-Time Real-Time PCR PCR grundsätzlich grundsätzlich möglich. möglich. mittels Bei Psittaziden Psittaziden und und Tauben Taubenwurde wurde nicht nicht nur nurCp. Cp. psittaci psittaci (16/23 (16/23 ;; •• Bei 70 %), %), sondern sondern auch auch Cp. Cp. abortus abortus (22 (22 %) %) nachgewiesen. nachgewiesen. Dies Dies 70 unterstreicht die die Notwendigkeit Notwendigkeit der der Speziesdifferenzierung Speziesdifferenzierung bei bei unterstreicht aviären Chlamydieninfektionen. Chlamydieninfektionen. aviären Bei Schweinen, Schweinen, Rindern Rindern und und Schafen Schafenwurden wurdenjeweils jeweilsmehrere mehrere •• Bei Cp./Chlamydia-Spezies identifiziert. identifiziert. Cp./Chlamydia-Spezies Bei Katzen Katzen und undMeerschweinchen Meerschweinchenwar war mit mit Cp. Cp.felis felis bzw. bzw. Cp. Cp. caviae caviae •• Bei jeweilsnur nur eine eine Spezies Spezies nachweisbar. nachweisbar. jeweils Cp. felis felis und und Cp. Cp. caviae caviae konnten konnten interessanterweise interessanterweiseauch auch in in •• Cp. Probenmaterial von von Hunden Hunden festgestellt festgestelltwerden. werden. Probenmaterial Ergebnisse Tabelle 2: Nachweisgrenze für Cp. psittaci, Cp. abortus, Cp. pecorum, Cp. felis, Cp. caviae und C. suis mittels speziesspezifischer Einzelassays in der Real-Time PCR Tabelle 1: Nachweis von Chlamydia- und Chlamydophila spp. (C./Cp.-Spezies) bei Haustieren mittels TaqMan - und TaqMan MGB-Sonden in der Real-Time PCR Probenherkunft Probenanzahl Anzahl reagierender Proben Anzahl nicht reagierender Proben Sonde spezifisch für C./Cp.Spezies Cp. psittaci Cp. abortus C. suis Cp. pecorum Cp. felis Cp. caviae Probenmaterial CT-Werte der für die einzelnen Chlamydienspezies jeweils spezifischen Real-Time PCR Erregerkonzentration in der Suspension (EBE2)/ml) Chlamydienstämme Cp. psittaci 11) 105 104 103 102 22/24 25/27 29/31 33/34 36/37 39/ *3) Cp. psittaci 21) 21/23 24/26 28/29 32/33 35/36 39/39 101 100 Katze 11 11 0 0 0 0 11 0 0 Meerschweinchen Papagei/ Sittich 5 20 5 20 0 14 0 4 0 0 0 0 0 0 5 0 0 2 Cp. psittaci 31) 23/24 27/28 31/31 34/35 38/38 40/41 Cp. abortus 3 21 25 28 32 35 39 Taube Schaf Schwein 3 12 11 3 12 11 2 0 1 1 12 7 0 0 9 0 3 0 0 0 0 0 0 0 0 0 0 Cp. pecorum 21 24 28 31 35 38 Cp. felis 24 27 30 34 37 Cp. caviae 23 26 30 33 36 Rind Hund Summe 10 4 76 10 4 76 2 0 19 7 0 31 1 0 10 1 0 4 0 3 14 0 1 6 0 0 2 * * 2) 3) Rinder (n=10) 10% 10% 10% Cp. abortus Schweine (n=11) 10% 9% 60% C. suis Cp. abortus und C. suis 9% 36% 46% Zwei Einzelassays für Cp. psittaci (Assay1/Assay2) EBE: Einschluß- bildende Einheit Kein CT-Wert Tabelle 3: Ergebnisse (CT-Werte) der speziesspezifischen Real-Time PCR mit DNA von sieben bekannten Chlamydienisolaten Speziesspezifischer Real-Time PCR-Assay für Cp. psittaci und Cp. abortus Cp. psittaci 1) Cp. psittaci Cp. pecorum Cp. pecorum Cp. felis Cp. abortus Cp. caviae Abbildung 1: Nachweis von Chlamydia (C.) und Chlamydophila (Cp.) spp. bei Rindern und Schweinen mittels TaqMan - und TaqMan MGB-Sonde in der Real-Time PCR 1) CT-Werte der Chlamydienisolate Cp. Cp. psittaci psittaci 1 2 Cp. psittaci 3 Cp. Cp. Cp. abortus abortus pecorum 1 2 25/25 * * * * 31/32 * * * * *2) /40 25 * * * 29/30 * * * * Real-Time PCR: Jede Probe wurde in dem entwickelten System mit den sieben Einzelassays untersucht. Zusätzlich wurde eine Positiv- und eine Negativkontrolle für jeden Einzelassay getestet. Als Positivkontrolle diente DNA von in Zellkultur angezüchteten Chlamydienisolaten (Institut für Hygiene und Infektionskrankheiten der Tiere, Gießen). Pro Reaktionsansatz wurden 12,5 µl qPCR Master Mix (Eurogentec, Seraing, Belgien), Forward Primer, Reverse Primer (Endkonzentration jeweils 0,9 µM), Sonde (Endkonzentration 0,2 µM), 8,1 µl steriles deion. Wasser (Sigma, Deisenhofen) und 2 µl DNA-Probe zu einem Reaktionsansatz von 25 µl pipettiert. In der Real-Time PCR wurden 45 Zyklen durchlaufen (10 Minuten 95°C Präinkubation, 15 Sekunden 95°C Denaturierung, 1 Minute 60°C Annealin g und Elongation). * /39 24 * * * * * 27 * * Cp. felis Cp. C. caviae suis * * * 30 * * * * * 27 * * * * * Zwei Einzelassays für Cp. psittaci (Assay1/Assay2) CT-Wert 2) Kein Literatur Material und Methoden Proben: Die Proben wurden freundlicherweise von der Vet Med Labor GmbH, Ludwigsburg, und vom Chemischen und Veterinäruntersuchungsamt Stuttgart, Fellbach, zur Verfügung gestellt. Es handelt sich um DNA, die aus Konjunktival-, Genitaltupfern, Nachgeburtsmaterial und Kot von Katzen, Meerschweinchen, Papageien, Sittichen, Tauben, Rindern, Schafen, Schweinen und Hunden gewonnen wurde (mit den DNAAufreinigungskits der Firma Qiagen, entsprechend den Anleitungen des Herstellers) und die in einer Chlamydien-spezifischen Real-Time-PCR reagiert hatten. Die Proben wurden in dem Zeitraum vom 1. Juli 2004 bis 30. Mai 2006 gesammelt. */ * */ * */ * * * * * wird noch bearbeitet C. suis 1) 10-1 Bestimmung der Nachweisgrenze: Zur Bestimmung der Nachweisgrenze der Einzelassays wurden Isolate von Cp. psittaci, Cp. abortus, Cp. pecorum, Cp. felis, Cp. caviae und C. suis in Zellkulturen (Buffalo Green Monkey, Friedrich-Loeffler-Institut, Insel Riems) angezüchtet. Folgende Chlamydien-Stämme wurden freundlicherweise zur Verfügung gestellt: Cp. psittaci-Stamm Z 1904/82 (Cp. psittaci 1), Cp. abortus-Stämme Z 178/02 (Cp. abortus 1) und Z 1215/86 (Cp. abortus 2), Cp. pecorum-Stamm Z 114/02, Cp. caviae-Stamm 432, Cp. felisStamm Z 3202-II/91, C. suis-Stamm Z 359/02 vom Institut für Hygiene und Infektionskrankheiten der Tiere, Gießen, Cp.psittaci-Stämme C12 (Cp. psittaci 2) und C19 (Cp. psittaci 3) und Cp. abortus-Stamm C18198 (Cp. abortus 3) vom Nationalen Referenzlabor für Psittakose, Jena. Eine Konzentrationsbestimmung der Chlamydien in den Suspensionen erfolgte mikroskopisch nach Giménez-Färbungen. Durch die Herstellung von Verdünnungsreihen der aus den Chlamydienisolaten gewonnenen DNA konnte die Nachweisgrenze für jeden Assay festgelegt werden (s. Tabelle 2). Bundesministerium für Verbraucherschutz, Ernährung und Landwirtschaft (BMVEL): Verordnung zum Schutz gegen die Psittakose und Ornithose, Entwurf vom 23.12.2005 DeGraves, F.J., D. Gao, H.R. Hehnen, T. Schlapp und B. Kaltenboeck (2003) J. Clin. Microbiol. 41, 1726-1729 Dennis, W., J. Storz (1982) Am. J. Vet. Res., 43,1897-1902 Everett, K.D.E. und A.A. Andersen (1999) Int. J. Syst. Bacteriol. 49, 803-813 Everett, K.D.E., R.M. Bush und A.A. Andersen (1999) Int. J. Syst. Bacteriol. 49, 415-440 Gresham, A.C., Dixon, C.E., B.J. Bevan (1996) Vet. Rec. 138 (25), 622-623 Helps, C., N. Reeves, S. Tasker, D. Harbour (2001) J. Clin. Microbiol. 39 (7), 2675-2676 Kaltenböck, B., N. Schmeer und R. Schneider (1997) J. Clin. Microbiol. 35, 1835-1841 Sachse, K., E. Grosmann (2002) Dtsch. Tierärztl. Wschr. 109, 142-148. Bestimmung der Spezifität: Zum Nachweis der Spezifität der entwickelten Methode, wurde die vorliegende DNA der verschiedenen Chlamydienisolate in jedem Einzelassay getestet (vgl. Tabelle 3). Korrespondenz Dr. Reinhard Sting Diese Arbeit wurde freundlicherweise durch Mittel der Grimminger-Stiftung für Zoonosenforschung unterstützt. Chemisches und Veterinäruntersuchungsamt Stuttgart, Schaflandstr. 3/3, D-70736 Fellbach Tel.: 0711/34261693, Fax: 0711/34261729, E-Mail: [email protected]