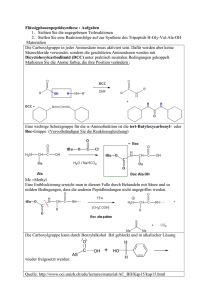
Trotz seiner Einfachheit sind die Vorhersagen dieses Modells recht ge
Werbung

• Trotz seiner Einfachheit sind die Vorhersagen dieses Modells recht genau • So gilt für den mittleren End-zu-End-Abstand: ⌦ 2↵ R = N l2 ,wobei l die Länge eines Kettensegments und N die Zahl der Segmente ist • Mit der Konturlänge L = N l kann man auch schreiben ⌦ 2↵ R = Ll • Der Gyrationsradius, das ist der Radius, den eine Kugelschale mit dem gleichen Trägheitsmoment bei gleicher Masse hätte, beträgt 2 RG = hR2 i Ll = 6 6 • Der hydrodynamische Radius (Stokes-Radius) ist der Radius einer Kugel mit der gleichen Stokes-Reibung RS2 = K Ll 6 , wobei K eine Konstante mit dem Wert von etwa 1,5 ist 29 8.2 Worm-like chain (WLC) • Hier nimmt man an, dass es eine gewisse Steifigkeit der Kette gibt, also die Orientierung der Monomere erst nach einer gewissen Länge lP , der Persistenzlänge, verloren geht • In diesem Fall wird der Ausdruck für den Gyrationsradius komplexer: h ⇣ ⌘i L 4 3 2l 1 exp P 2lp lP LlP 2 RG = lP2 + 2 3 L L • Im Grenzfall L l erhält man 2 RG = LlP Llk = 3 6 , wobei lk = lp /2 die Kuhn-Länge ist, die der Segmentlänge der GaußKette entspricht • Für lange WLC-Ketten erhält man also wieder eine Gauß-Kette 9 Proteine 9.1 9.1.1 Aufbau und Struktur Primärstruktur • Monomere sind die Aminosäuren • Eine Aminosäure besteht aus einer Aminogruppe (basisch), einer CarboxylGruppe (sauer), und einem in der Mitte befindlichen Kohlenstoffatom, dem C-↵ Atom, mit einem Wasserstoffatom und einer Seitenkette • Bei der Polymerisation wird eine Peptidbindung zwischen Carboxylgruppe und Peptidgruppe unter Abspaltung von Wasser gebildet • Die Peptidbindung hat partiellen Doppelbindungscharakter, so dass die Atome O, C, und N in einer Ebene liegen 30 • Es gibt 20 verschiedene natürliche Aminosäuren, die sich in der Seitenkette unterscheiden unpolar/hydrophob basisch Alanin Valin Lysin Methionin Leucin Isoleucin Arginin Prolin Tryptophan Phenylalanin Histidin polar/neutral sauer Threonin Tyrosin Glycin Serin Cystein Glutamin Glutaminsäure Asparagin Asparaginsäure • Die verschiedenen Seitenketten lassen sich zunächst in polare und unpolare unterscheiden, die polaren nochmals in sauer bzw. basisch 9.1.2 Sekundärstruktur • Während die Peptidbindung nicht rotieren kann, ist um die Einfachbindungen am C-↵ Atom zunächst eine Rotation möglich • Rotation wird durch Diederwinkel 31 und beschrieben • Das Vorhandensein verschiedener Seitenketten schränkt die Rotierbarkeit ein • Diese Einschränkung kommt im Ramachandran-Plot zum Ausdruck • Schließlich können sich unter bestimmten Umständen durch Wasserstoffbrückenbindungen stabilisierte Strukturen ausbilden, von denen die beiden wichtigsten die ↵-Helix und das -Faltblatt sind 32 9.1.3 Tertiärstruktur • Die dreidimensionale Struktur einer Polypeptidkette bezeichnet man als Tertiärstruktur • Sie wird durch verschiedene Wechselwirkungen stabilisiert: VdW, Wasserstoffbrückenbindung, elektrostatische Wechselwirkungen (einschließlich Ionenbindung), teilweise auch kovalente Querverbindungen über Disulfidbrücken • Im inneren eines Proteins liegen eher hydrophobe AS, an der Oberfläche hydrophile 9.1.4 Quartärstruktur • Bilden mehrere Polypeptidketten einen Komplex, so spricht man von der Quartärstruktur 33