Molekulare Grundlagen der Vererbung und Entwicklungssteuerung 20
Werbung

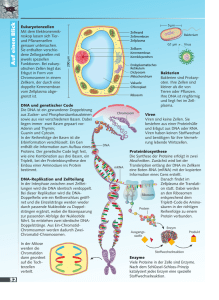
20 Basiswissen Molekulare Grundlagen der Vererbung und Entwicklungssteuerung a. Replikation, Proteinbiosynthese bei Pro- und Eukaryoten, Mutagene und Mutationen (vgl. Aufg. 1, 2, 3) Um die Replikation der DNA erläutern zu können, muss der Aufbau der DNA bekannt sein. Sollten die Kenntnisse über den Aufbau zur Erläuterung der unteren Abbildung nicht mehr ausreichen, müssen diese zuerst erlangt werden. Wichtig ist hier, dass die Nucleotide in der DNA zweifach miteinander verbunden sind, zum einen über die Phosphatgruppen zu einem Strang (hier sind die Bausteine frei kombinierbar und gewährleisten somit die Fähigkeit, Informationen zu speichern) und zum anderen über die Wasserstoffbrücken zwischen den Basen. Hier gibt es nur zwei Kombinationen (zwischen A und T mit zwei, zwischen G und C mit drei Wasserstoffbrücken). Dadurch ist die identische Verdopplung möglich. Helicase DNA-Polymerase Replikationsgabel 3‘ kontinuierliche Synthese 5‘ 3‘ G 5‘ A RNA-Primer Primase DNA-Matrize 5‘ OKAZAKI-Fragment 3‘ 3‘ 5‘ diskontinuierliche Synthese 3‘ 5‘ Nucleotide T C DNA-Ligase 3‘ 5‘ Wanderungsrichtung der Replikationsgabel Schema der DNA-Replikation Nach Öffnung des DNA-Strangs lagern sich an beide offenen Einzelstränge die passenden Nucleotide an. Somit enthält jeder Doppelstrang einen elterlichen Strang und einen Strang aus Einzelnucleotiden, die aus dem Cytoplasma stammen (semi-konservative Replikation). Da die Synthese des neuen Einzelstrangs nur in 5`›3` Richtung möglich ist, kann nur einer der Einzelstränge kontinuierlich gebildet werden, die Synthese des anderen muss diskontinuierlich erfolgen. Die so entstehenden Teilstücke werden OKAZAKI-Fragmente genannt und durch das Enzym Ligase zusammengefügt. Die Umsetzung der Information der DNA in Lebensvorgänge steuernde Proteine (z. B. Enzyme, Carrier, Tunnelproteine, Myofibrillen u.s.w.) wird Proteinbiosynthese genannt. In diesem Verfahren werden entsprechend der Basensequenz der DNA spezifische Aminosäuren zu einer Kette verknüpft. Dabei entspricht ein bestimmtes Basentriplett (eine Folge dreier Basen) einer bestimmten Aminosäure in der zu bildenden Kette. Dieser Zusammenhang wird genetischer Code genannt (vgl. Code-Sonne Genetische und entwicklungsbiologische Grundlagen von Lebensprozessen S. 88/89). Er gilt für fast alle Lebewesen und ist somit universell. Da bei vier verschiedenen Basen und einer Informationseinheit aus insgesamt drei Basen insgesamt 43 = 64 Kombinationsmöglichkeiten bei nur 20 existierenden Aminosäuren zur Verfügung stehen, codieren teilweise mehrere Tripletts für die gleiche Aminosäure: „Der Code ist degeneriert“. Die Proteinbiosynthese läuft bei den Prokaryoten (Zellen ohne Zellkern, z. B. Bakterien) einfacher ab als bei den Eukaryoten (Zellen mit Zellkern). Bei den Prokaryoten geschieht die Umsetzung der Information der DNA in den Aufbau von Proteinen in zwei Schritten: der Transkription, also dem Überschreiben der Information von der DNA in die der mRNA und der Translation, d. h. der „Übersetzung“ der mRNA-Information in die entsprechende Aminosäuresequenz, die dann z. B. als Katalysator (Enzym) oder als Baustein für Zell- und Gewebestrukturen (z. B. Aktin, Keratin) fungiert. DNA Transkription Translation m-RNA Protein Schematische Darstellung der Proteinbiosynthese bei Prokaryoten Transkription (vgl. Aufg. 3, 11) Die DNA öffnet sich nach Bindung der RNA-Polymerase an dem Promotor (Abschnitt mit einer spezifischen Nucleotidsequenz), die Doppelhelixstränge liegen jetzt getrennt vor. An einem der beiden Stränge (codogener Strang) werden die zu diesem Strang komplementären Nucleotide in 5´›3´Richtung angelagert. Danach löst sich der neugebildete Nucleotidstrang (mRNA) von der DNA und wandert zu den Ribosomen. Verlängerung der RNA neu hinzukommendes RNA-Nucleotid Richtung der Transkription DNAEntwindung C DNARückwindung 3‘ G Promotorregion P P P mRNA G 5‘-Ende A Transkription C G RNANucleotide A U A T C U G 5‘ G G C C G 5‘ U C G 3‘ A 3‘ C DNAMatrizenstrang RNA-Polymerase 21