Bakterien sehen Blau – Neues über LOV
Werbung

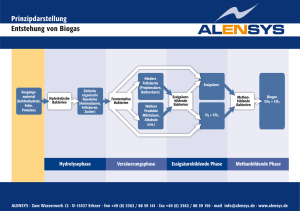
341_379_BIOsp_0407.qxd 12.06.2007 11:32 Uhr Seite 361 361 Bakterielle Photorezeptoren Bakterien sehen Blau – Neues über LOV-Photorezeptoren THOMAS DREPPER, FRANCO CIRCOLONE, ACHIM HECK, ULRICH KRAUSS UND KARL -ERICH JAEGER INSTITUT FÜR MOLEKUL ARE ENZYMTECHNOLOGIE, UNIVERSITÄT DÜSSELDORF, FORSCHUNGSZENTRUM JÜLICH Blaulichtrezeptoren der LOV(light-oxygen-voltage)-Familie sind in Pflanzen und Bakterien weit verbreitet. Detaillierte Kenntnisse über die lichtinduzierten photochemischen Prozesse halfen kürzlich dabei, rekombinante LOV-Proteine zu erzeugen, die als neuartige in vivo-Fluoreszenz-Reporter in anaeroben biologischen Systemen verwendet werden können. ó Licht besitzt einen weitreichenden Einfluss auf unterschiedliche biologische Prozesse: Es ist nicht nur für Pflanzen und photosynthetische Bakterien von zentraler physiologischer Bedeutung, sondern ebenso ein bedeutsames Umweltsignal für verschiedene regulatorische und adaptive Prozesse. Lichtabhängige Prozesse beginnen, genau wie andere signalverarbeitende Abläufe, mit der Wahrnehmung des Signals. Viele Organismen haben zu diesem Zweck Lichtrezeptoren entwickelt. Zu den Rezeptoren, die in pro- und eukaryotischen Taxa identifiziert werden konnten, zählen unter anderem die Rotlichtsensitiven Phytochrome sowie die Flavin-bindenden Blaulichtrezeptoren der BLUF(bluelight sensing using FAD)- und der LOV-Familie. Die bekanntesten Vertreter der LOV-Photorezeptoren sind die Phototropine, die in Pflanzen an zahlreichen lichtabhängigen Prozessen wie dem Phototropismus beteiligt sind[1]. Die Blaulichtrezeptoren der LOV-Familie sind jedoch auch in Bakterien weit verbreitet[2]. Bislang konnten mehr als 80 solcher LOV- Gene in über 60 Genomen Gram-positiver und -negativer Bakterien identifiziert werden[3]. Im Gegensatz zu den pflanzlichen Vertretern der LOV-Familie ist die physiologische Funktion der meisten bakteriellen Blaulichtrezeptoren unbekannt. LOV-Photozyklus und Proteinstruktur Alle bislang untersuchten LOV-Proteine binden Flavin-Mononukleotid (FMN), das den Rezeptoren als Chromophor dient und durch blaues Licht angeregt werden kann. Bisher konnte eine FMN-Bindung für die bakteriellen LOV-Proteine aus Bacillus subtilis (YtvA), Pseudomonas putida (SB2) und Caulobacter crescentus (Q9ABE3) nachgewiesen werden[2, 4, 5]. Die photochemische Charakterisierung der LOV-Proteine zeigte, dass die bakteriellen Sensoren genau wie die PflanzenPhototropine einen lichtinduzierten Photozyklus durchlaufen. Während dieses Vorgangs geht ein konserviertes Cystein eine transiente, kovalente Bindung mit FMN ein, was zur Ausbildung eines FMN-Cystein-C(4a)Thiol-Addukts führt. Dieser photoaktivierte ¯ Abb. 1: Struktur und Photozyklus der YtvALOV-Domäne von B. subtilis. A, Die postulierte Struktur der FMN-bindenden YtvA-LOV-Domäne besteht aus fünf β-Faltblättern, die von zwei αHelices flankiert werden. Das Chromophor FMN ist in Grün dargestellt, das photoaktive Cystein in Rot. B, Nach Anregung mit Licht durchlaufen LOV-Proteine einen Photozyklus. Die lichtaktivierte LOV-Domäne bindet FMN durch Ausbildung einer kovalenten Bindung zwischen dem Flavin C4a und der Thiolgruppe des photoaktiven Cysteins. BIOspektrum | 04.07 | 13. Jahrgang 341_379_BIOsp_0407.qxd 362 12.06.2007 11:32 Uhr Seite 362 WISSENSCHAFT ˚ Abb. 2: FbFP-vermittelte in vivo-Fluoreszenz. A, FbFP-abhängige Fluoreszenz in E. coli. Die grüne Fluoreszenz, die durch die neuen Fluoreszenzproteine (FbFP) hervorgerufen wird, ist mit bloßem Auge gut zu erkennen. YFP: yellow fluorescent protein ; BsFbFP: FMN-basiertes Fluoreszenzprotein aus Bacillus subtilis (=YtvAC62A); EcFbFP: (YtvAC62A-LOV; E. coli-Codon-Gebrauch); PpFbFP: P. putida (=SB2C53A). B, In vivo-Fluoreszenz ohne Sauerstoff. R. capsulatus-Zellen, die entweder YFP oder PpFbFP exprimierten, wurden aerob (+ O2) oder anaerob (– O2) angezogen. Die fluoreszierenden Bakterien wurden mit konfokaler Laser-Scanning-Mikroskopie detektiert, die Anregung erfolgte mit Licht der Wellenlänge 514 nm (YFP) oder 458 nm (PpFbFP), die Fluoreszenzemission wurde bei 530 nm (YFP) oder zwischen 505 und 530 nm (PpFbFP) bestimmt. Als Kontrolle dienten R. capsulatus-Zellen, die keines der beiden Reporterproteine synthetisierten. Zustand geht daraufhin langsam (für YtvA in ∼2600 s) wieder in den Dunkelzustand über. In Abbildung 1 ist der vereinfachte Photozyklus mit FMN im Grundzustand sowie im photoaktivierten Zustand dargestellt. Bislang wurden die Strukturen der LOV1Domäne des Phototropins aus Chlamydomonas reinhardtii und der LOV2-Domäne des Phytochrom-Phototropin-Hybridphotorezeptors phy3 aus dem Farn Adiantum capillusveneris aufgeklärt[6, 7]. Sie ähneln sich sehr und weisen wie alle Proteine der PAS(PerArntSim)-Superfamilie eine typische α/β-Faltung auf. Die Aminosäuresequenzen pflanzlicher und bakterieller LOV-Domänen zeigen eine Übereinstimmung von 40–50 %. Innerhalb der LOV-Sequenz befindet sich das hochkonservierte Sequenzmotiv GXNCRFLQ mit dem photoaktiven Cystein (unterstrichen), welches essenziell für den beschriebenen Photozyklus ist. LOV-Proteine als Fluoreszenzreporter in anaeroben Systemen Autofluoreszierende Proteine wie das grün fluoreszierende Protein (GFP) sind in der biologischen und biomedizinischen Forschung zu unentbehrlichen Markerproteinen geworden. Jedoch weist GFP ebenso wie dessen Farbvarianten (z. B. das gelb fluoreszierende YFP) einen grundsätzlichen Nachteil auf: Die Verwendung ist in vivo auf aerobe biologische Systeme beschränkt, da für die autokatalytische Synthese des GFP-Chromophors molekularer Sauerstoff benötigt wird[8]. Eine völlig neue Gruppe von fluoreszierenden Reporterproteinen, die im Gegensatz zu den Proteinen der GFP-Familie auch unter anaeroben Bedingungen leuchten, wurde kürzlich in unserem Labor entwickelt[9]. Diese neuen Reporterproteine sind Varianten der bakteriellen Blaulichtrezeptoren YtvA und SB2, die den schwach fluoreszierenden Cofaktor Flavin-Mononukleotid (FMN) binden. Um die FMN-abhängige Fluoreszenz der Blaulichtrezeptoren zu erhöhen und somit deren Verwendung als Fluoreszenzmarker zu ermöglichen, wurde das jeweilige photoaktive Cystein durch ein Alanin ersetzt. Durch diese Mutationen wird der Photozyklus unterbrochen, was dazu führt, dass die LOV-vermittelte Autofluoreszenz drastisch erhöht ist (Abb. 2A). Um die Fluoreszenz weiter zu steigern, wurde in einem zweiten Schritt das Cterminale Effektormodul von YtvA deletiert und das nun auf den LOV-kodierenden Bereich reduzierte rekombinante Bacillus-Gen an den E. coli-Codon-Gebrauch angepasst. Dies führte zu einer weiteren Steigerung der in vivo-Fluoreszenzintensität des FMNbasierten fluoreszierenden Proteins (FbFP). Die photochemische Charakterisierung der neuen Markerproteine ergab, dass die Fluoreszenzintensitäten und damit auch die Sensitivitäten der FbFPs mit denen der GFP-Familie annähernd vergleichbar sind. Um die in vivo-Fluoreszenzeigenschaften der FbFPs mit denen von YFP in anaeroben Systemen zu vergleichen, wurden die jeweiligen Reporterproteine im fakultativ anaero- ben Bakterium Rhodobacter capsulatus synthetisiert und deren Fluoreszenz in Gegenwart und Abwesenheit von O2 bestimmt. Wie in Abbildung 2B dargestellt, führte die Expression des FbFPs sowohl unter aeroben als auch unter anaeroben Bedingungen zu einer intensiven Fluoreszenz der Bakterienzellen. Im Gegensatz dazu konnte unter identischen Bedingungen, wie erwartet, keine YFP-Fluoreszenz nachgewiesen werden. Da fakultativ und strikt anaerobe Mikroorganismen bei unterschiedlichen biologischen, biomedizinischen und biotechnologischen Prozessen eine zentrale Rolle spielen, können nun mithilfe der neuen Fluoreszenzreporter erstmals komplexe Vorgänge in lebenden anaeroben Bakterien mittels nicht invasiver Verfahren untersucht werden. Zudem werden die FbFPs dazu beitragen, neue biotechnologische Anwendungsgebiete für Anaerobier zu erschließen. Auf der Basis einer Patentanmeldung[10] soll eine kommerzielle Verwertung der Fluoreszenzreporter durch die neu gegründete Biotech-Firma EVOcatal GmbH mit Sitz im Life Science Center in Düsseldorf erfolgen. Danksagung Die Autoren danken Prof. Dr. Wolfgang Gärtner (Max-Planck-Institut für Bioanorganische Chemie, Mülheim an der Ruhr), Dr. Aba Losi (Department of Physics, Universität Parma, Italien) und Dr. Thorsten Eggert (EVOcatal GmbH, Düsseldorf) für die gute Zusammenarbeit. Teile dieser Arbeit wurden von der BIOspektrum | 04.07 | 13. Jahrgang 341_379_BIOsp_0407.qxd 12.06.2007 11:32 Uhr Seite 363 363 Deutschen Forschungsgemeinschaft gefördert (Forschergruppe „Sensory blue light photoreceptors“ FOR526). ó Literatur [1] Briggs, W. R., Christie, J. M. (2002): Phototropins 1 and 2: versatile plant blue-light receptors. Trends Plant Sci. 7: 204– 210. [2] Losi, A. (2004): The bacterial counterparts of plant phototropins. Photochem. Photobiol. Sci. 3: 566–574. [3] Krauss, U., Losi, A., Gärtner, W., Jaeger, K. E., Eggert, T. (2007): LOV is all around – A phylogenetic study on the origin of LOV-dependent blue light signaling. in preparation. [4] Losi, A., Polverini, E., Quest, B., Gärtner, W. (2002): First evidence for phototropin-related blue-light receptors in prokaryotes. Biophys. J. 82: 2627–2634. [5] Krauss, U., Losi, A., Gärtner, W., Jaeger, K. E., Eggert, T. (2005): Initial characterization of a blue-light sensing, phototropin-related protein from Pseudomonas putida: a paradigm for an extended LOV construct. Phys. Chem. Chem. Phys. 7: 2804–2811. [6] Fedorov, R., Schlichting, I., Hartmann, E., Domratcheva, T., Fuhrmann, M., Hegemann, P. (2003): Crystal structures and molecular mechanism of a light-induced signaling switch: The Phot-LOV1 domain from Chlamydomonas reinhardtii. Biophys. J. 84: 2474–2482. [7] Crosson, S., Moffat, K. (2001): Structure of a flavin-binding plant photoreceptor domain: insights into light-mediated signal transduction. Proc. Natl. Acad. Sci. USA 98: 2995–3000. [8] Tsien, R. Y. (1998): The green fluorescent protein. Annu. Rev. Biochem. 67: 509–544. BIOspektrum | 04.07 | 13. Jahrgang [9] Drepper, T., Eggert, T., Circolone, F., Heck, A., Krauss, U., Guterl, J. K., Wendorff, M., Losi, A., Gärtner, W., Jaeger, K. E. (2007): Reporter proteins for in vivo fluorescence without oxygen. Nat. Biotechnol., 25: 443–445. [10] Eggert, T., Drepper, T., Guterl, J. K., Krauss, U., Heck, A. Jaeger, K.-E. (2005): Fluoreszenzmarker und deren Verwendung. DE 10 2005 048828.5. Korrespondenzadresse: Dr. Thomas Drepper Institut für Molekulare Enzymtechnologie Heinrich-Heine-Universität Düsseldorf Forschungszentrum Jülich Stetternicher Forst D-52426 Jülich Tel.: 02461-614173 Fax: 02461-612490 [email protected] www.iet.uni-duesseldorf.de AUTOREN Von links nach rechts: Ulrich Krauss, Achim Heck, Franco Circolone, Thomas Drepper und Karl-Erich Jaeger. Dr. Thomas Drepper leitet seit 2004 die Gruppe „Bakterielle Photobiotechnologie“ am Institut für Molekulare Enzymtechnologie der Heinrich-Heine-Universität Düsseldorf. Die Gruppe arbeitet vorrangig mit phototrophen Bakterien der Gattung Rhodobacter, wobei sowohl Aspekte der Grundlagenforschung als auch der angewandten Forschung im Mittelpunkt stehen.