Selbst oder Nicht-Selbst – Pflanzliche Immunrezeptoren
Werbung
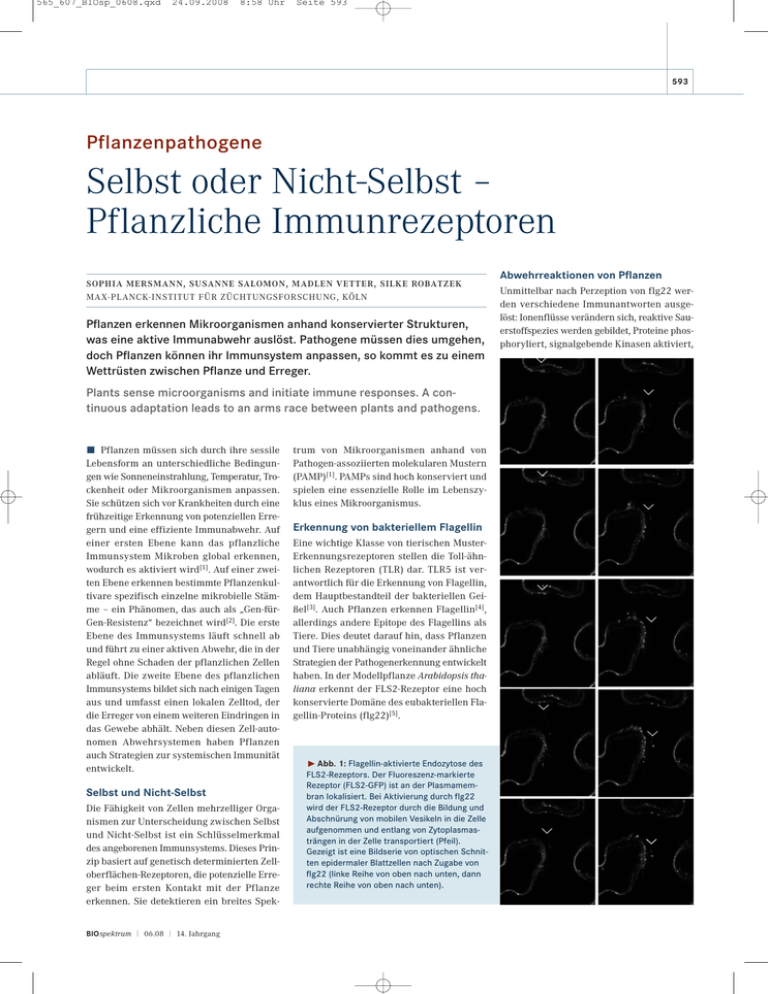
565_607_BIOsp_0608.qxd 24.09.2008 8:58 Uhr Seite 593 593 Pflanzenpathogene Selbst oder Nicht-Selbst – Pflanzliche Immunrezeptoren SOPHIA MERSMANN, SUSANNE SALOMON, MADLEN VETTER, SILKE ROBATZEK MAX-PL ANCK-INSTITUT FÜR ZÜCHTUNGSFORSCHUNG, KÖLN Pflanzen erkennen Mikroorganismen anhand konservierter Strukturen, was eine aktive Immunabwehr auslöst. Pathogene müssen dies umgehen, doch Pflanzen können ihr Immunsystem anpassen, so kommt es zu einem Wettrüsten zwischen Pflanze und Erreger. Plants sense microorganisms and initiate immune responses. A continuous adaptation leads to an arms race between plants and pathogens. ó Pflanzen müssen sich durch ihre sessile Lebensform an unterschiedliche Bedingungen wie Sonneneinstrahlung, Temperatur, Trockenheit oder Mikroorganismen anpassen. Sie schützen sich vor Krankheiten durch eine frühzeitige Erkennung von potenziellen Erregern und eine effiziente Immunabwehr. Auf einer ersten Ebene kann das pflanzliche Immunsystem Mikroben global erkennen, wodurch es aktiviert wird[1]. Auf einer zweiten Ebene erkennen bestimmte Pflanzenkultivare spezifisch einzelne mikrobielle Stämme – ein Phänomen, das auch als „Gen-fürGen-Resistenz“ bezeichnet wird[2]. Die erste Ebene des Immunsystems läuft schnell ab und führt zu einer aktiven Abwehr, die in der Regel ohne Schaden der pflanzlichen Zellen abläuft. Die zweite Ebene des pflanzlichen Immunsystems bildet sich nach einigen Tagen aus und umfasst einen lokalen Zelltod, der die Erreger von einem weiteren Eindringen in das Gewebe abhält. Neben diesen Zell-autonomen Abwehrsystemen haben Pflanzen auch Strategien zur systemischen Immunität entwickelt. Selbst und Nicht-Selbst Die Fähigkeit von Zellen mehrzelliger Organismen zur Unterscheidung zwischen Selbst und Nicht-Selbst ist ein Schlüsselmerkmal des angeborenen Immunsystems. Dieses Prinzip basiert auf genetisch determinierten Zelloberflächen-Rezeptoren, die potenzielle Erreger beim ersten Kontakt mit der Pflanze erkennen. Sie detektieren ein breites SpekBIOspektrum | 06.08 | 14. Jahrgang trum von Mikroorganismen anhand von Pathogen-assoziierten molekularen Mustern (PAMP)[1]. PAMPs sind hoch konserviert und spielen eine essenzielle Rolle im Lebenszyklus eines Mikroorganismus. Erkennung von bakteriellem Flagellin Eine wichtige Klasse von tierischen MusterErkennungsrezeptoren stellen die Toll-ähnlichen Rezeptoren (TLR) dar. TLR5 ist verantwortlich für die Erkennung von Flagellin, dem Hauptbestandteil der bakteriellen Geißel[3]. Auch Pflanzen erkennen Flagellin[4], allerdings andere Epitope des Flagellins als Tiere. Dies deutet darauf hin, dass Pflanzen und Tiere unabhängig voneinander ähnliche Strategien der Pathogenerkennung entwickelt haben. In der Modellpflanze Arabidopsis thaliana erkennt der FLS2-Rezeptor eine hoch konservierte Domäne des eubakteriellen Flagellin-Proteins (flg22)[5]. ˘ Abb. 1: Flagellin-aktivierte Endozytose des FLS2-Rezeptors. Der Fluoreszenz-markierte Rezeptor (FLS2-GFP) ist an der Plasmamembran lokalisiert. Bei Aktivierung durch flg22 wird der FLS2-Rezeptor durch die Bildung und Abschnürung von mobilen Vesikeln in die Zelle aufgenommen und entlang von Zytoplasmasträngen in der Zelle transportiert (Pfeil). Gezeigt ist eine Bildserie von optischen Schnitten epidermaler Blattzellen nach Zugabe von flg22 (linke Reihe von oben nach unten, dann rechte Reihe von oben nach unten). Abwehrreaktionen von Pflanzen Unmittelbar nach Perzeption von flg22 werden verschiedene Immunantworten ausgelöst: Ionenflüsse verändern sich, reaktive Sauerstoffspezies werden gebildet, Proteine phosphoryliert, signalgebende Kinasen aktiviert, 565_607_BIOsp_0608.qxd 594 24.09.2008 8:22 Uhr Seite 594 WISSENSCHAFT konstitutiv zwischen Plasmamembran und Endosomen rezyklieren[8]. Im Falle von FLS2 geht die Lokalisierung an der Plasmamembran verloren, wenn der Rezeptor durch flg22 aktiviert wird[9]. Der aktivierte FLS2-Rezeptor akkumuliert in intrazellulären Vesikeln, was im weiteren Verlauf zu seinem Abbau führt. Die Dynamik der Internalisierung von FLS2-GFP in Vesikel veranschaulicht Abbildung 1. Ähnlich den Befunden zu endosomal lokalisiertem BRI1[8] weisen unsere Ergebnisse darauf hin, dass die Internalisierung von FLS2 an der Signalweiterleitung beteiligt ist. Die Endozytose von FLS2 könnte auch der Aufhebung der Signalweiterleitung dienen. Die Pflanzenzelle könnte sich so vor Selbstschädigung durch Abwehrreaktionen schützen und den transienten Verlauf der meisten PAMP-induzierten Antworten gewährleisten (Abb. 2). Koevolution – Wettlauf um den Vorteil ˚ Abb. 2: Modell zur FLS2-Signalweiterleitung. Nach Bindung von flg22 an den FLS2-Rezeptor wird der Ko-Rezeptor BAK1 in einen aktiven Signalkomplex rekrutiert. In der Folge wird die Endozytose von FLS2 ausgelöst, welche zu einem Abbau von aktiviertem FLS2 führt. Vorstellbar ist, dass der Prozess der Aktivierung von FLS2 bis hin zu dessen Abbau dem Verlauf flg22-induzierter Antworten entspricht. Unmittelbar nach Stimulierung des Rezeptor-Komplexes (Initiierung) werden Abwehrreaktionen ausgelöst, die einem transienten Verlauf folgen und dabei einer Signalverstärkung (Amplifizierung), sowie Signalverminderung (Attenuierung) unterliegen. die Expression von nahezu 1.000 Genen erhöht, das Stresshormon Ethylen gebildet und der Sekundärstoff Callose an der Zellperipherie eingelagert[4, 5]. Kürzlich wurde die Rolle der flg22-Erkennung für den Schutz der Pflanze vor bakteriellem Befall gezeigt[5]. So zeigen Blätter von A. thaliana ohne funktionelles FLS2 eine erhöhte Anfälligkeit gegen das phytopathogene Bakterium Pseudomonas syringae pv. tomato DC3000 (PtoDC3000). Phytopathogene Bakterien dringen häufig über Spaltöffnungen der Blattoberfläche in das Blattgewebe ein und besiedeln den Apoplasten[6]. Die Erkennung von flg22 führt zum Verschluss der Spaltöffnungen, sodass keine weiteren Erreger in das Blattgewebe eindringen können. Der Flagellin-Rezeptor-Komplex Viele Aspekte der Funktionsweise des FLS2Rezeptors sind noch unverstanden. Der durch Bindung von flg22 aktivierte FLS2- Rezeptor bildet einen Komplex mit dem KoRezeptor BAK1[7], wodurch das Immunsystem aktiviert wird. BAK1 ist ein Ko-Rezeptor für multiple Liganden-bindende Rezeptoren und kontrolliert deren aktive Signalweiterleitung[8]. Über die Deregulierung der FLS2Aktivität ist wenig bekannt. Allerdings scheint die Kinase-assoziierte Proteinphosphatase (KAPP) eine Rolle dabei zu spielen. Die Aktivität von Zelloberflächen-Rezeptoren wird aber auch über deren Relokalisierung in endosomale Kompartimente reguliert. Hier setzen unsere eigenen Studien an, die zeigen, dass FLS2 in Abhängigkeit von flg22 endozytiert wird[9]. Subzelluläre Dynamik von FLS2 Die Endozytose von membranständigen Rezeptoren in Pflanzen wurde vor wenigen Jahren beschrieben. Eine ligandenunabhängige Internalisierung von Rezeptoren wurde für BRI1 und BAK1 nachgewiesen, welche Phytopathogene Bakterien müssen die Erkennung ihrer PAMPs durch den Wirtsorganismus verhindern. Dazu können sie in begrenztem Maße ihre PAMPs verändern oder Moleküle sekretieren, die die PAMPs maskieren. Oft injizieren Pathogene mittels Typ3-Sekretionssystemen Effektoren in die Wirtszelle, die zu einer Unterdrückung der Immunabwehr führen. So injiziert der Stamm PtoDC3000 das Effektorprotein AvrPto in die Pflanzenzelle, wo es direkt den Muster-Erkennungsrezeptor FLS2 angreift und seine Aktivität hemmt[10]. Derselbe Stamm produziert das Polyketid-Toxin Coronatin, das dem Pflanzenhormon Jasmonsäure ähnelt und zur Öffnung der durch PAMP-Perzeption geschlossenen Spaltöffnungen führt[6]. Mikroorganismen können sich schnell an wechselnde Umweltbedingungen anpassen und einen großen Selektionsdruck ausüben, sodass Pflanzen ihre Immunabwehr kontinuierlich optimieren müssen. Dies führt zu einer Art Wettrüsten[2], auch in der Evolution der Muster-Erkennungsrezeptoren: Entweder werden Innovation und größtmögliche Vielfalt angestrebt oder eine spezielle Rezeptorvariante favorisiert[11]. So prägen sich effiziente Mechanismen des pflanzlichen Immunsystems aus, die einen maximalen Fortpflanzungserfolg am Standort ermöglichen. Danksagung Unsere Arbeiten werden unterstützt durch die IMPRS Doktorandenschule des MaxPlanck-Instituts für Züchtungsforschung BIOspektrum | 06.08 | 14. Jahrgang 565_607_BIOsp_0608.qxd 24.09.2008 8:22 Uhr (S. M. und S. S.) sowie durch die Deutsche Forschungsgemeinschaft (M. V.). ó Literatur [1] Bittel, P., Robatzek, S. (2007): Microbe-associated molecular patterns (MAMPs) probe plant immunity. Curr. Opin. Plant Biol. 10: 335–341. [2] Jones, J. D., Dangl, J. L. (2006): The plant immune system. Nature 444: 323–329. [3] Hayashi, F., Smith, K. D., Ozinsky, A., Hawn, T. R., Yi, E. C., Goodlett, D. R., Eng, J. K., Akira, S., Underhill, D. M., Aderem, A. (2001): The innate immune response to bacterial flagellin is mediated by Toll-like receptor 5. Nature 410: 1099– 1103. [4] Felix, G., Duran, J. D., Volko, S., Boller, T. (1999): Plants have a sensitive perception system for the most conserved domain of bacterial flagellin. Plant J. 18: 265–276. [5] Zipfel, C., Robatzek, S., Navarro, L., Oakeley, E. J., Jones, J. D., Felix, G., Boller, T. (2004): Bacterial disease resistance in Arabidopsis through flagellin perception. Nature 428: 764– 767. [6] Melotto, M., Underwood, W., Koczan, J., Nomura, K., He, S. Y. (2006): Plant stomata function in innate immunity against bacterial invasion. Cell 126: 969–980. [7] Chinchilla, D., Zipfel, C., Robatzek, S., Kemmerling, B., Nürnberger, T., Jones, J. D., Felix, G., Boller, T. (2007): A flagellin-induced complex of the receptor FLS2 and BAK1 initiates plant defence. Nature 448: 497–500. [8] Geldner, N., Hyman, D. L., Wang, X. L., Schumacher, K., Chory, J. (2007): Endosomal signaling of plant steroid receptor kinase BRI1. Genes Dev. 21: 1598–1602. [9] Robatzek, S., Chinchilla, D., Boller, T. (2006): Ligand-induced endocytosis of the pattern recognition receptor FLS2 in Arabidopsis. Genes Dev. 20: 537–542. [10] Xiang, T., Zong, N., Zou, Y., Wu, Y., Zhang, J., Xing, W., Li, Y., Tang, X., Zhu, L., Chai, J., Zhou, J. M. (2008): Pseudomonas syringae effector AvrPto blocks innate immunity by targeting receptor kinases. Curr. Biol. 18: 74–80. [11] Salvaudon, L., Giraud, T., Shykoff, J. A. (2008): Genetic diversity in natural populations: a fundamental component of plant-microbe interactions. Curr. Opin. Plant Biol. 11: 135– 143. Korrespondenzadresse: Dr. Silke Robatzek Abteilung Molekulare Phytopathologie Max-Planck-Institut für Züchtungsforschung Carl-von-Linné-Weg 10 D-50829 Köln Tel.: 0221-5062-302 Fax: 0221-5062-353 [email protected] AUTOREN Die Arbeitsgruppe von Silke Robatzek erforscht anhand des FLS2-Rezeptors die pflanzliche Immunabwehr. Dabei werden genetische, molekularbiologische, biochemische, zellbiologische und evolutionsbiologische Ansätze verfolgt. Von li. nach re.: Sophia Mersmann (Doktorandin), Thomas Spallek (Doktorand), Nicolas Frei dit Frey (Postdoc), Vera Göhre (Postdoc), Silke Robatzek (Gruppenleiterin), Petra Köchner (technische Assistentin), Susanne Salomon (Doktorandin), Heidrun Häweker (technische Assistentin), Madlen Vetter (assoziierte Doktorandin der Gruppe Juliette de Meaux, MPIZ Köln). BIOspektrum | 06.08 | 14. Jahrgang Seite 595











