Differenzierung, Entwicklung, gewebespezifische Genaktivität, die
Werbung

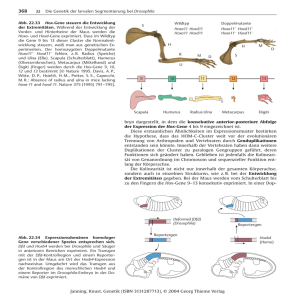
Individualentwicklung/Ontogenese Die Entwicklung eines vielzelligen Organismus (Pflanzen und Tiere, inkl. Mensch) aus der befruchteten Eizelle bedeutet die Durchführung eines kompliziertes, genetisches Entwicklungsprogrammes, durch das bestimmte Gene zeitlich und räumlich ein- oder ausgeschaltet werden. Mithilfe der klassischen und molekularen Genetik wurden Gene identifiziert, die an den Entwicklungsprogrammen beteiligt sind. Während dieses Prozesses sich unterschiedliche Zelltypen und Gewebearten entwickeln, die die Organe und den Körper eines Organismus ausbilden. Während der Individualentwicklung (Ontogenese) entwickeln sich Gewebe und Organe. Zusätzlich, diese Strukturen besitzen bestimmten Ort innerhalb des Körpers des Organismus; deshalb die Zellen oder Gruppe der Zellen „wissen“ es, wofür, wann und wo sich entwickeln müssen. Um diese Frage beantworten zu können, ab Anfang achtziger Jahren Biologen eine systematische Untersuchung der Drosophila Embryonalentwicklung durchgeführt haben. Anschliesslich kamen die Entwicklungsbiologen zu einer erstaunlichen Schlussfolgerung: Die Gene, die die Entwicklung von Drosophila steuern, kommen in nahezu unveränderter Form in den Genomen anderer Arten, einschliesslich den Menschen vor. Die Frühentwicklung des Drosophila Embryo fängt mit Zellkernvermehrungen ohne Furchung und ohne individuelle Zellenbildung an, und ein s.g. syncytiales Blastoderm entsteht. Nach zehn synchronen Kernteilungen wandern die Kerne an die Oberfläche. Schliesslich von aussen nach innen Zellmembranen zwischen den Kernen eingezogen werden, und befindet sich der Embryo im vielzelligem Stadium des zellulären Blastoderms (Abb. 11). Der Anlagenplan / Schicksalskarte (Fate map) eine zytologische Karte ist, die bestimmt, welche blastoderm-Zelle an welchen Körperteil der Larvae entspricht. Wenn die Zelle beschädigt wird, der dementsprechend Körperteil fehlt. In dem zellulären Blastodermstadium setzen Zellbewegungen ein, die als Gastrulation bezeichnet werden. Nach Abschluss dieser Bewegungen beginnt eine Unterteilung des Embryos in aufeinanderfolgende Zellpakete, aus denen sich schliesslich die Segmente der Larve und letzendlich auch der Fliege differenzieren (Abb. 12). Während dieses Prozess verkürzt sich der Embryo durch rückläufiger Gastrulationsbewegung bis die Segmente in der Längsachse angeordnet sind. Ab diesem Stadium kann man die Zellkapete den späteren Segmenten zuordnen, und die weitere Entwicklung zur Larve auch verfolgen kann. Es sind 3 Kopf, 3 Thorax und 9 Abdominalsegmente, von denen die Kopfsegmente bei den Larven nicht zu sehen sind, sie sind nach innen verlagert, die Larve ist azephal (kopflos) (Abb. 12). Auf der ventralen (Bauch) Seite der Larve findet man die Zänchenbänder, jeweils am Vorderrand jedes Thorax- und Abdominalsegments. Ein Segment reicht vom Vorderrand eines Zänchenbändes bis zum Vorderrand des nächsten (Abb. 12). Bei einer jungen Larve kann man innerhalb eines Segments eine weitere Furche erkennen, die das Segment in einen vorderen (anterioren) und einen hinteren (posterioren) Bereich unterteilt. Diese Bereiche heissen Kompartimente. In der Frühentwicklung spielt die Unterteilung in s.g. Parasegmente eine wichtige Rolle. Ein Parasegment aus einem posterioren Kompartiment und aus dem folgenden anterioren Kompariment besteht (Abb. 12). Der Lebenszyklus setzt sich von der Larve zur Puppe fort. Im Verlauf der Metamorphose werden larvale Strukturen abgebaut und Strukturen der adulten Fliege (Imago) aufgebaut. Die adulten Strukturen entstehen aus imaginalen Vorläuferzellen, die entweder als kleine Zellengruppen in den larvalen Geweben zu finden sind, oder als Imaginalscheiben organisiert sind (Abb. 13). Christiane Nüsslein-Vollhard und Erich Wieschaus waren von der plausiblen Überlegung ausgegangen, dass Gene, die an der Segmentierung beteiligt sind, auch mutierbar sein müssen. Aber, wenn die Mutation ein wichtiges Gen im Segmentationsprozess betrifft, sollte die Larve aufgrund der Entwicklungsstörung aus dem Ei nicht zu schlüpfen. Deswegen wurde eine sichtbare Veranderung am Kutikulamuster der Zänchenbänder gewählt, als dieses Muster wird lange vor dem Schlüpfen der Larve differenziert (Abb. 12); damit war es möglich die gefundenen Mutationen der Segmentierungsgene in drei prinzipiell unterschiedlichen Klassen zuordnen, wie folgt: Die Gruppe der Gap-Gene (Lückengene), bei denen ein zusammenhängender Bereich der Segmenten defekt ist; die Gruppe der Segmentpolaritätsgene, bei denen in jedem einzelnen Segment ein Defekt auftritt; in der Gruppe der Paarregelgene jedes zweite Segment fehlt (Abb. 14). Was hier fehlt oder vorhanden ist sind nicht die Segmente, sondern Parasegmente, die in Richtung anterior-posterior die Zänchenbänder begrenzen (Abb. 12). Die nächste wichtige Frage Die Antwort ist ja, und das ftz-Gen enthält eine 9 kbp kodierende Region besteht lautet: Werden die Segmentationsgene zeitlich-räumlich reguliert und exprimiert? fushi-tarazu-Gen (ftz; Japanisch: Wenige Segmente) wird als Beispiel dienen. Das regulatorische Region und eine ~2 kbp transkribierende Region (Abb. 15). Die aus zwei Exons und ein Intron. Die homozygoten ftz-Embryonen den Wildtyp annehmen, wenn das Transgen die gesamte Kontrollregion enthält; das in 5’-Region verkürzte Transgen es nicht leisten konnte (Abb. 15). Die Verteilung der ftz-mRNA in den Embryonen entspricht der ParasegmentSpezifizität. Die Darstellung der Ftz-Proteinverteilung wurde indirekt, mithilfe des lacZ-Reportergen aus E. coli unter der Kontrolle des ftz-Kontrollregion gezeigt, und das Ftz-Proteinmuster als -Galaktosidase Muster sichtbar gemacht (Abb. 15). Das Expressionsmuster der Segmentierungsgene, wie ftz, ermöglicht die Vermutung, dass die Oozyten ein anterior-posteriores Koordinatensystem besitzen, welches schon im synzytialen Cytoplasma die Genexpression aktiviert oder reprimiert. Die an der Etablierung des Koordinatensystems beteiligten, s.g. maternalen Gene produzieren mRNAs, die in der Oozyte in unterschiedlichen Position lokalisiert sind, und erst in der Zygote translatiert wird. Moleküle, die vom Translationsort im Cytoplasma diffundieren und einen Konzentrationsgradienten (Koordinatensystem) bilden, werden als Morphogene bezeichnet. Nach diesem Modell die Morphogene Bicoid und Hunchback ein am anterioren Pol lokalisiertes Proteingradient bilden, und das Gradient von Caudal und Nanos befinden sich am posterioren Pol (Abb. 16). Die mRNAs der Morphogenen werden durch den Nährzellen ins Ei deponiert; während der Reifung der Oozyte die Nährzellen werden abgebaut und verschwinden. Die Translation der morphogenen-mRNAs fängt gleich nach der Eiablage an (Abb. 17). Die dorso-ventrale Achse wird auch während der Oogenese festgelegt, und erfolgt im Übergang von synzytialen zum zellulären Blastodermstadium. Dabei wird ein Dorsal-Gradient in den ventralen Zellkernen aufgebaut (Abb. 18). Das Dorsal-Protein ist ein Transkriptionsfaktor, der den Zellen entlang der dorso-ventralen Achse Positionsinformationen vermittelt. Die dorsal Mutanten sind „dorsalisiert“, weisen keine Zänchenbänder auf. Das Bicoid Protein die Translation von caudal-mRNA verhindert, gibt es Caudal Protein am posterioren Pol. Nanos Protein hemmt die Translation von hunchback-mRNA, so dass der Hunchback-Gradient am anterioren Pol liegt. Dadurch entsteht ein mit Bicoid überlappender Hunchback-Gradient, und ein gegenläufiger, mit Nanos überlappender Caudal-Gradient (Abb. 19). Die ersten zygotisch exprimierten Segmentierungsgene sind die GapGene, wie z.B. Krüppel. Experimentelle Daten zeigen die Beschränkung der Krüppel Expression im Synzytium auf die zentrale Domäne. Obwohl der Morphogenengradient Bicoid und Hunchback krüppel-Transkription aktiviert, die durch auch diesen Gradient aktivierten, benachbarten giant- und knirps-Expression reprimieren anterior und posterior krüppel-Expression. Die krüppel-Kontrollregion besteht aus zahlreichen aktivator- und repressor-Protein-Bindungstellen, die das Expressionsmuster von krüppel steuern (Abb. 20). Dabei entstehen keine scharf abgegrenzte Expressionsbereiche, sondern glockenförmige, überlappende Bereiche (Abb. 19). Die Paarregelgene weisen ein messerscharfes Expressionsmuster in den Parasegmenten auf. Davon fushi tarazu exprimiert in den geradezahligen, even-skipped in den ungeraduzahligen Parasegmenten. Die Aktivatoren Hunchback und Bicoid würden anteriore Expression von even-skipped ermöglichen, aber die als Repressoren wirkenden, schon vorher gebildeten Giant und Krüppel Proteine beschränken die Expression von even-skipped in dem zweiten Streifen des dritten Parasegments. Die Kontrollregion des even-skipped-Gens enthält zahlreiche Aktivator- und Repressor-Bindungsstellen, die die koordinierte Expression ermöglichen (Abb. 21). Die Segmentpolaritätsgene engrailed and wingless wirken in der Stabilisierung der Kompartimentsgrenzen. In den Parasegmenten, welche durch Fushi tarazu und Even-skipped Expression schon etabliert wurden, wird die anteriore Grenze der Parasegmenten Engrailed-Expression definiert, und durch die aktivierung von Wingless die posteriore Parasegmentgrenze festgelegt (Abb. 22). Die Expression von Fushi tarazu und Even-skipped wird beendet, und ein Segment besteht aus einem anterioren und einem posterioren Kompartiment, wo EngrailedExpression stellt die posteriore Segmentgrenze dar, damit das endgültiges Segmentationsmuster entsteht. Ein entwicklungsgenetisches Kompartiment is eine Gruppe von Zellen, die schon genetisch determiniert ist, und ihre mitotische Zellnachkommenschaft ein bestimmtes Entwicklungsschiksal besitzt. Mit anderen Wörter, die genetisch identischen, determinierten somatischen Zellen unterscheiden sich wesentlich phenotypisch. Das im Embryo etablierte Segmentationsmuster wird in der Larve, und später in der adulten fliege wiedergespiegelt, aber wie bekommen die Segmente ihre spezifische genetische Identität? Die Antwort war die Entdeckung der homeotischen Gene. In der bithorax mutanten Fliege wurde das gesamte Thoraxsegment T3 in T2 transformiert, damit eine vierflügige Fliege entstand. Die antennapedia-Fliege trug zwei Beinen anstelle der Antenne (Abb. 23). Die homeotischen Mutationen treffen die homeotischen Selektorgene, oder die Kontrollgene der Segmentdifferenzierung. HOM-C, der Genkomplex der homeotischen Gene aus den Genen von antennapedia-C und bithorax-C besteht (Abb. 24). Die Gene der Komplexen sind in derselben Reihenfolge angeordnet, wie die von anterior bis posterior die Körpersegmente bestimmen. Das erstenmal klonierte homeotische Gen war das Antennapedia-Gen. Das letzte Exon des Gens kodiert für die s.g. Homeobox, eine DNA-bindende Protein-Domäne, die aus 60 Aminosäuren besteht (Abb. 25). Mithilfe dieser Sequenzinformation wurden schliesslich homeotische Gene in Vertebraten, Pflanzen und Pilzen gefunden, welche die hochkonservierte Homeobox aufwiesen (Abb. 26). Die Bindung der Antennapedia-Homeobox an die DNA stellt Abb. 27 dar. In dieser Protein-DNA-Wechselwirkung nehmen Arginin 5 (R5), Isoleucin 47 (I47), Glutamin 50 (Q50) und Methionin 54 (M54) teil (Abb. 27). In den Genomen vieler Tierarten findet man Homeotische Gene, die ebenso wie bei Drosophila in einem Komplex, oder Cluster angeordnet sind und entsprechend dieser Lokalisation ebenso sequenziell entlang der Körperachse exprimiert werden. Im Unterschied zum HOM-C-Cluster von Drosophila werden sie Hox-Cluster in Maus und HOX-Cluster in Mensch genannt. Bei den Säuger gibt es vier Hox-Cluster auf vier verschiedenen Chromosomen. (Maus: 2, 6, 11 und 15; Mensch: 2, 7, 12 und 17) (Abb. 28). Während der Entwicklung der Vorder- und Hinterbeine der Maus verschiedene Bereiche der Extremitäten werden durch Hoxa- und Hoxd-Gene kontrolliert. Ausfallen einige genaktivitäten verursacht schwere Fehlentwicklung (Abb. 29). Die Hoxc-8 -/- homozygot Mutant Maus sich eine extra Rippe entwickelt, und haltet ihre Zähen Faust-ähnlich (Abb. 30). Wegen ihrer Konzervativismus die Segment-Spezifizität homeotischer Gene können in Drosophila und in Maus verglichen werden (Abb. 31, Tab. 1). HOXA11 homeo box A11 7p15p14.2 Radioulnar synostosis with amegakaryocytic thrombocytopenia, 605432 HOXA13 homeo box A13 7p15p14.2 Hand-foot-uterus syndrome Guttmacher syndrome, 176305 HOXD13 homeo box D13 2q31-q32 Synpolydactyly, type II Brachydactyly, type E, 113300 Einige menschliche homeotische Gene und ihrer mutanter Phenotyp. Es ist sicher, dass jeder Körper von mehrzelligen Organismen einem komplizierten Genaktivitätmuster nach gebildet wird, die jetzt nur in einigen Aspekten bekannt ist. Wie hier beschrieben, ist natürlich nur ein kurzer Blick von einem sich rasch entwickelnden Gebiet der Entwicklungsbiologie. Grundanforderung: Wichtige Fachausdrücke Individualentwicklung, Ontogenese syncytiales Blastoderm zelluläres Blastoderm Anlagenplan / Schicksalskarte (Fate map) Segment, Parasegment Segmentierungsgene Zeitlich-räumliche Exprimierung der Segmentationsgene Maternale Gene Anterior-posteriores Koordinatensystem der Oozyten Bicoid, Nanos, Hunchback, Caudal Morphogene Dorsal, Dorsalisierung Paarregelgene Segmentpolaritätsgene Homeotische Selektorgene Segmentdifferenzierung Apikalleiste (apical ectodermal ridge) Sonic hedgehog Fibroblastenwachstumsfaktoren (FGF) polarisierende Zellgruppe (ZPA, zone of polarizing activity) ZPA Einpflanzung, Spiegelsymmetrische Finger HOLT-ORAM SYNDROME Atavismus