Nachweis aller relevanten Genveränderungen an einer Tumorprobe
Werbung

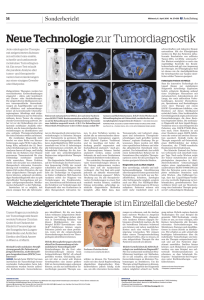
Pharma Report Neue Technologie für die molekulare Tumordiagnostik Nachweis aller relevanten Genveränderungen an einer Tumorprobe © Springer-Verlag GmbH Die zielgerichtete Tumortherapie erfordert eine exakte, rasche und detaillierte Diagnostik. Die klassische molekulare Tumoranalytik mit diagnostischen Methoden der ersten und zweiten Generation – Sequenzierung und FISH – ist dafür oft nicht ausreichend: Bei oftmals widersprüchlichen oder unvollständigen Ergebnissen wird viel Zeit und Tumormaterial benötigt. Mit dem „Hybrid Capturebased Next Generation Sequencing“ ist jetzt ein gewebesparender diagnostischer Test der dritten Generation verfügbar, mit dem sich alle therapierelevanten genomischen Veränderungen des Tumormaterials in einem Schritt nachweisen lassen. Impressum Corporate Publishing (verantwortlich): Ulrike Hafner, Dr. Michael Brysch, Dr. Katharina Finis, Dr. Friederike Holthausen, Sabine Jost, Ann Köbler, Dr. Claudia Krekeler, Inge Kunzenbacher, Dr. Christine Leist, Dr. Sabine Lohrengel, Dr. Ulrike Maronde, Dr. Monika Prinoth, Dr. Annette Schilling, Yvonne Schönfelder, Dr. Petra Stawinski, François Werner, Teresa Windelen Report in „Der Onkologe“ Band 21, Heft 10, Oktober 2015 Mit freundlicher Unterstützung der NEO New Oncology AG, Köln Die Herausgeber der Zeitschrift übernehmen keine Verantwortung für diese Rubrik. Springer Medizin Springer-Verlag GmbH Tiergartenstraße 17, 69121 Heidelberg Springer ist Teil der Fachverlagsgruppe Springer Science + Business Media © Springer-Verlag Berlin Heidelberg 2015 Die Wiedergabe von Gebrauchsnamen, Handelsnamen, Warenbezeichnungen usw. in dieser Zeitschrift berechtigt auch ohne besondere Kennzeichnung nicht zu der Annahme, dass solche Namen im Sinne der Warenzeichen- und Markenschutz-Gesetzgebung als frei zu betrachten wären und daher von jedermann benutzt werden dürfen. Für Angaben über Dosierungsanweisungen und Applikationsformen kann vom Verlag keine Gewähr übernommen werden. Derartige Angaben müssen vom jeweiligen Anwender im Einzelfall anhand anderer Literaturstellen auf ihre Richtigkeit überprüft werden. Treibermutationen: Ziel für die Therapie Im Zentrum der molekularen Tumordiagnostik stehen Treibermutationen, die ursächlich an der Entstehung und Progression eines Tumors beteiligt sind und häufig als therapeutisches Ziel genutzt werden können. So wurden z. B. gegen Mutationen in EGFR (Epidermal Growth Factor Receptor) oder gegen Genfusionen wie EML4-ALK (Echinoderm Microtubule-associated protein-Like 4/ Anaplastic Lymphoma Kinase), wie sie etwa beim nicht-kleinzelligen Lungenkarzinom (NSCLC) vorkommen [1], spezifische Tyrosinkinaseinhibitoren (TKI) entwickelt. Auch in zahlreichen anderen soliden Tumoren wie etwa im malignen Melanom, in CUP (Cancer of Unknown Primary)- Tumoren, im Mamma- oder Ovarialkarzinom sind therapierbare Genveränderungen bekannt. Überlebensvorteil durch personalisierte Therapie Für Patienten ist die personalisierte Therapie von großer Relevanz. Das zeigte z. B. eine Studie, in der 5.000 Patienten mit Lungenkarzinom auf molekulare Veränderungen und Treibermutationen getestet wurden [1]. NSCLC-Patienten mit aktivierenden EGFR-Mutationen, die mit einem EGFR-TKI behandelt wurden, überlebten im Mittel 31,5 Monate, Patienten unter Chemotherapie 9,6 Monate (Abb. 1). Auch Patienten mit nachgewiesener ALK-Genfusion profitierten von der personalisierten Therapie: Unter Crizotinib lag das Gesamtüberleben bei 23 Monaten versus elf Monate unter zytostatischer Therapie. Einen weiteren Beleg für den Nutzen molekularer Tumorana- lysen ergab eine 2012 publizierte Vergleichsstudie [2]: Bei 460 von insgesamt 1.144 Patienten mit fortgeschrittenem Tumor wurden genetische Veränderungen nachgewiesen. Patienten, die daraufhin eine zielgerichtete Therapie erhielten, profitierten im Vergleich zu konventionell behandelten Patienten von einem verbesserten Ansprechen und einer längeren Gesamtüberlebenszeit. Angesichts der zahlreichen Initiativen zur Analyse des Krebsgenoms ist davon auszugehen, dass in den kommenden Jahren weitere therapeutisch relevante genomische Veränderungen identifiziert werden können – was die Entwicklung neuer Medikamente ermöglicht [3]. Damit steigen auch die Anforderungen an eine umfassende molekulare Tumordiagnostik, die eine zunehmende Anzahl therapierelevanter Veränderungen berücksichtigen und testen muss. Abb. 1 NSCLC: Überlebensvorteil durch Therapie mit Tyrosinkinase­ inhibitoren bei Nachweis definierter genomischer Veränderungen Tumor mit EGFR-Mutation EGFR-Inhibitor Chemotherapie 100 60 40 20 0 9,6 0 10 31,5 20 30 40 50 Zeit (Monate) Tumor mit ALK-Fusionsgen Crizotinib Chemotherapie 100 HR 0,169 95%-KI 0,07–0,41 p<0,001 80 60 Gesamtüberleben (%) Bericht: Dr. Katharina Arnheim, Freiburg i. Br. Das Wissen um genetische Veränderungen in Tumorzellen hat im letzten Jahrzehnt deutlich zugenommen. Lungenkrebs beispielsweise wird nicht mehr nur in kleinzellig und nicht-kleinzellig unterteilt, sondern in weitere Untergruppen, die je nach molekularbiologischen Eigenheiten unterschiedliche Prognosen und Ansatzpunkte für Behandlungen bieten. Derzeit sind allein in dieser Indikation mehr als ein Dutzend therapeutisch relevante genetische Veränderungen bekannt. Ähnliches gilt auch für viele andere solide Tumoren. Eine genaue Identifizierung und Beurteilung aller relevanten Genveränderungen im Tumor des einzelnen Patienten ist Voraussetzung für eine zielgerichtete Therapie. Gesamtüberleben (%) „Nachweis aller relevanten Genveränderungen an einer Tumorprobe“ Literaturarbeit HR 0,235 95%-KI 0,074–0,742 p=0,024 80 60 40 20 0 11 0 10 23 20 30 40 50 Zeit (Monate) EGFR= Epidermal Growth Factor Receptor; ALK= Anaplastic Lymphoma Kinase; HR=Hazard Ratio; KI=Konfidenzintervall modifiziert nach [1] 60 Pharma Report Diagnostik aus dem Blut: Nachweis von Resistenzen Ein Patient mit metastasierendem Adenokarzinom der Lunge stellt sich mit einer Vorbehandlung aus EGFR-Inhibitoren, Chemotherapie, Neurochirurgie und Bestrahlung vor. Mit konventioneller Diagnostik konnten keine therapierelevanten Mutationen in einer Tumorprobe nachgewiesen werden. Anhand einer Blutprobe wurden eine EGFR-T790M-Mutation und eine damit vermittelte Resistenz gegen die bisher eingesetzten EGFR-Inhibitoren ermittelt. Der Patient wurde daraufhin mit einem alternativen EGFR-Inhibitor behandelt. Die Erkrankung zeigte schnelles Ansprechen mit Reduktion von Primärtumor und Metastasen und eine deutliche Verbesserung des Allgemeinzustands. Verlaufskontrollen in vierzehntägigem Abstand zeigten eine graduelle Abnahme der Resistenzmutation im Blut. Der Patient ist derzeit klinisch, radiologisch und molekulargenetisch progressionsfrei und in gutem Zustand. Die Behandlung wird fortgesetzt [4]. Herausforderung für die Pathologie Für die molekulardiagnostischen Tests ist jedoch meist nur wenig Tumorgewebe verfügbar, an dem nicht nur einzelne Punktmutationen, sondern zunehmend auch therapierelevante Insertionen, Deletionen, Veränderungen der Kopienzahl einzelner Gene sowie Genfusionen detektiert werden sollen. Die Sanger-Sequenzierung kann dabei nur zum Nachweis von Punktmutationen, kleinen Insertionen und Deletionen genutzt werden. Zwar ist diese Methode kostengünstig, wenn nur eine kleine Zahl von Genabschnitten untersucht wird, die Sensitivität ist jedoch gering. Änderungen der Kopienzahl sowie Genfusionen müssen separat mittels FISH-Analyse (Fluoreszenz-in-situHybridisierung) getestet werden – eine Methode, die fehleranfällig und zeitintensiv ist, da die Testung sequentiell durchgeführt werden muss. In vielen Fällen ist das Tumormaterial aufgebraucht, bevor die Analyse abgeschlossen werden kann [3]. Hingegen können molekulardiagnostische Methoden der zweiten Generation, wie z. B. Realtime-PCR (Polymerase Chain Reaction) oder das PCR-basierte NGS (Next Generation Sequencing), somatische Mutationen parallel und dank höherer Sensitivität auch bei niedriger Allelfrequenz nachweisen. Es ist jedoch nicht möglich, intronische Regionen, in denen die Bruchpunkte von Genfusionen liegen, adäquat mit zu untersuchen. Um Genfusionen detektieren zu können, sind daher zusätzliche FISH-Analysen erforderlich, für die weiteres Tumorgewebe benötigt wird. Neu entdeckte genomische Veränderungen können nur unter größerem Aufwand in die Tests integriert werden: In der Regel muss hierfür ein neuer Assay entwickelt und validiert werden. Zudem kann das Verfahren des PCR-basierten NGS zu fehlerhaften Ergebnissen führen. Insbesondere bei in Formalin fixiertem Gewebe können bei der Amplifikation Fehler auftreten, welche im weiteren Verlauf als falsch-positive Mutationen fehlinterpretiert werden können. Ein Test für alle therapierelevanten Genveränderungen Mit einem molekulardiagnostischen Test der dritten Generation lässt sich das gesamte Spektrum klinisch relevanter genomischer Veränderungen (Mutationen, Insertionen, Deletionen, Änderungen der Kopienzahl und Genfusionen) an einer kleinen Tumorprobe nachweisen. Hierzu wird das „Hybrid Capture-based Next Generation Sequencing“ Verfahren eingesetzt [3]. Für den Einsatz in Europa wurde hierzu der diagnos- tische Test NEO entwickelt. In dem Verfahren werden zunächst alle zu untersuchenden therapierelevanten Genregionen angereichert (Hybrid Capture Verfahren) und im Anschluss mit hoher Abdeckung parallel sequenziert und hochauflösend analysiert (NGS). Im Rahmen dieses Verfahrens werden auch große Intronregionen angereichert und analysiert, sodass Genfusionen verlässlich detektiert werden können. Die gewonnenen Daten werden mittels Computer-Algorithmen ausgewertet, die genomische Treibermutationen von klinisch nicht relevanten PassengerMutationen abgrenzen können. Neu identifizierte, diagnostisch relevante Genveränderungen können zeitnah in den Assay implementiert werden, ohne dass sich die Analysedauer verlängert, der Test kann somit jederzeit an aktuelle wissenschaftliche Erkenntnisse angepasst werden. Mit der Methode des „Hybrid Capture-based Next Generation Sequencing“ sind genomische Veränderungen bei geringer Allelfrequenz detektierbar. Auch bislang unbekannte onkogene Genfusionen können nachgewiesen werden. Die diagnostische Methode der dritten Generation erlaubt daher die Anreicherung und Detektion aller therapierelevanten genomischen Veränderungen in nur einem einzigen Test. Zusätzliche FISH-Analysen oder weitere Sequenzierungen sind nicht nötig, sodass sowohl Tumorgewebe als auch Laborzeit eingespart werden können. Mit dem NEOliquid Test können nun therapierelevante Genveränderungen an einer Blutprobe des Patienten nachgewiesen werden. Dabei werden im Blut des Patienten zirkulierende Bruchstücke von Tumor-DNA ermittelt und untersucht („Flüssigbiopsie“). Der Bluttest stellt damit nicht nur eine Alternative zur klassischen Biopsie dar, sondern ermöglicht auch eine Verlaufskontrolle. Durch die parallele Detektion aller therapierelevanten genomischen Veränderungen an einer einzigen Blutprobe kann der Bluttest eine valide und für den Patienten wenig belastende Grundlage für die weitere Therapieentscheidung bieten. Fazit Die Fortschritte in der Sequenziertechnik und die Entwicklung zielgerichteter Therapeutika haben die Tumortherapie innerhalb weniger Jahre revolutioniert. Die neue Herausforderung besteht darin, therapeutisch relevante genomische Veränderungen bei jedem einzelnen Patienten rasch und zuverlässig zu identifizieren, damit zügig eine zielgerichtete, effektive Behandlung eingeleitet werden kann. Das „Hybrid Capture-based Next Generation Sequencing“ bietet hier eine sensitive und schnelle Methode, mit der das gesamte Spektrum therapierelevanter genomischer Veränderungen anhand geringer Gewebemengen oder im Blut festgestellt werden kann. Bluttestung: nicht-invasive Diagnostik In einigen Fällen kann dennoch nicht genügend Tumorgewebe für eine molekulardiagnostische Untersuchung gewonnen werden. Zudem gestaltet sich die Kontrolle von Therapieerfolg, Metastasierung und möglichem Rezidiv immer noch schwierig – es sei denn, der Patient würde in regelmäßigen Abständen biopsiert. Literatur 1. The Clinical Lung Cancer Genome Project (CLCGP) and Network Genomic Medicine (NGM), Science Translational Medicine 2013, 5:209ra153 2. Tsimberidou AM et al., Clin Cancer Res 2012, 18:6373–6383 3. Heuckmann JM, Thomas RK, Ann Oncol 2015, 26:1830–1837 4. Gautschi O et al., J Thorac Oncol 2015, Epub ahead of print