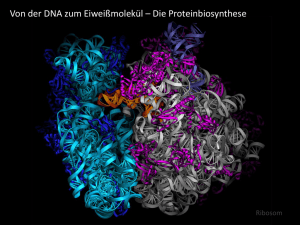
Kombinatorische Selektion funktionaler Peptide durch mRNA
Werbung

Special 201 Kombinatorische Selektion funktionaler Peptide durch mRNA-Display Nikolai A. Raffler und Michael Famulok* * Rheinische Friedrich-Wilhelms-Universität Bonn, Kekulé-Institut für Organische Chemie und Biochemie Einleitung Im menschlichen Körper bilden Antikörper eine wirkungsvolle Maschinerie mit der unter anderem Infektionen bekämpft werden. Durch Rekombination von variablen Domänen kann der Körper etwa einhundert Milliarden (1011) dieser Antikörper mit unterschiedlichsten Funktionalitäten bilden. Wegen ihrer hohen Spezifität und Affinität sind Antikörper mittlerweile zu einem unentbehrlichen Werkzeug in der modernen biologischen Forschung, der Biomedizin und der Diagnostik geworden. In der Forschung wird nun versucht, die natürliche Bildung von Antikörpern im Labor, in vitro nachzuahmen, indem kombinatorische Methoden in der Synthese eingesetzt werden. Evolutive Biotechnologien (Übersichten in Lit.[1,2]) bilden hierzu den Schlüssel, um maßgeschneiderte, hochspezifische und funktionale Moleküle zu generieren. In den achtziger Jahren wurde der Grundstein für die kombinatorische Chemie durch die Entwicklung der Parallelsynthese von Proteinen durch MARIO GEYSEN[3] gelegt. Seit diesen ersten Arbeiten wurde die kombinatorische Chemie auf vielfältige weitere Molekül- Abb. 1: (A) Puromycin-Molekül, (B) 3’-Ende einer Tyrosyl-tRNA Klassen ausgedehnt, die es nun unter anderem erlauben, funktionale Ribonukleinsäuren, Desoxyribonukleinsäuren, modifizierte Nukleinsäuren, Peptide bzw. Proteine aus komplexen Bibliotheken zu isolieren[4]. Beim so genannten Phagen-Display[5] wird die genetische Information des Proteins bzw. Peptids über den Phagen in Bakterien eingeschleust. Infiziert man Bakterien mit einem transformierten Phagen, kann dieser sich vermehren und präsentiert das neu eingeführte Peptid an seiner Oberfläche, während er gleichzeitig die Information für die entsprechende Proteinvariante in Form der DNA in seinem Inneren verpackt enthält. Durch systematische oder zufällige Variation der DNA-Sequenz können Bibliotheken mit einer Komplexität von 107 – 109 Peptiden erzeugt werden. Ähnliche Techniken, die zu den neuen Entwicklungen der evolutiven Special 202 Abb. 2: Mechanismus der Puromycin-vermittelten Fusion von RNA und dem entsprechenden kodierten Peptid. (A) Als Ausgangsmaterial wird eine chimäre mRNA-DNA-Puromycin-Bibliothek eingesetzt. (B) In vitro Translation: das Ribosom bindet die RNA und bewegt sich auf dem Templat in Richtung des 3’Endes, das kodierte Peptid wird synthetisiert (Aminosäuren als dunkle Kreise dargestellt). (C) Das Ribosom erreicht die Verbindungsstelle mRNA-DNA und pausiert, das Puromycin kann in die A-Stelle eintreten, wo es kovalent mit dem Peptid verknüpft wird. (D) Freisetzung der gebildeten Fusionsmoleküle Verfahren gerechnet werden und bei denen Phänotyp und Genotyp eines Peptids oder Proteins direkt miteinander verknüpft werden, sind das Ribosomen- oder PolysomenDisplay[6]. Das Ribosomen-Display ist hierbei als eine Weiterentwicklung des Polysomen-Displays anzusehen und wurde durch die Arbeiten von HANES und PLÜCKTHUN zu einem allgemein anwendbaren System zur in vitro Selektion von funktionellen Peptiden und Proteinen verbessert. Jede mRNA der bis zu 109 verschiedenen Moleküle umfassenden Bibliotheken kodiert für ein spezielles Protein und ist mit diesem über ein einzelnes Ribosom verbunden[7]. Da sowohl Phagen als auch Ribosomen komplexe biologische Systeme darstellen, suchte man zur Optimierung der Methode nach einem kleinen organischen Molekül, das diese hochmolekularen Strukturen substituieren und jedes Mitglied einer Peptidoder Protein-Bibliothek direkt mit seiner kodierenden mRNA verknüpfen sollte. mRNA-Display Eine solche Möglichkeit bietet das mRNA Display[8]. 1997 stellten SZOSTAK und YANAGAWA nahezu gleichzeitig ein Prinzip vor, das es ermöglicht, die mRNA und das durch sie kodierte Peptid kovalent über ein Brückenmolekül miteinander zu verknüpfen. So kann die eindeutige genetische Aussage über die funktionelle Struktur des Proteins erhalten bleiben. Im Zentrum dieser Technologie steht das Antibiotikum Puromycin (Struktur siehe Abb. 1). Das Molekül ahmt in vivo aufgrund einer strukturellen Ähnlichkeit das 3’-Ende einer aminoacylierten tRNA nach (Abbildung 1). Es kann so in die Aminoacyl-tRNA-Bindungsstelle (A-Stelle) des Ribosoms eintreten, wird kovalent an das naszierende Protein gebunden und die Translation bricht ab. Durch organische Synthese kann nun in vitro ein DNA-Puromycin-Oligomer hergestellt werden, welches am 3’-Ende ein kovalent verknüpftes Puromycin-Molekül trägt. Wird durch enzymatische Synthese eine mRNA-Bibliothek mit diesem Linkermolekül verbunden, liegt ein mRNA-DNAPuromycin-Molekül vor, welches für die Translation als Vorlage verwendet werden kann (mRNA display template). Über die 5’Hydroxylgruppe des Puromycin erfolgt eine kovalente Bindung an das 3’-Ende des DNA-Linkers, welcher wiederum mit der für das Peptid kodierenden mRNA verknüpft ist. Wird nun diese Puromycin-gekoppelte mRNA-Bibliothek in eine in vitro Translation eingesetzt, so resultieren letztlich Fusionsmoleküle, die aus dem Protein, Puromycin, dem Linker und der mRNA bestehen (Abb. 2). Während das Ribosom, so der hypothetische Mechanismus, bei Erreichen der Verbindungsstelle zwischen mRNA und DNA pausiert, hat das Puromycin Gelegenheit, die A-Stelle zu besetzen. Durch die PeptidylTransferase-Aktivität des Ribosoms wird daraufhin das Peptid an das Puromycin gebunden (Abb. 3). Somit ist eine stabile kovalente Verbindung zwischen dem Genotyp und dem Phänotyp entstanden. Das gebildete mRNA Display Peptid wird anschließend revers transkribiert, um ein doppelsträngiges RNA-DNA-Hybrid zu gewinnen und sicherzustellen, dass im eigentlichen Selektionsschritt kein RNA-Aptamer isoliert wird. Das so hergestellte Produkt kann für die in vitro Selektion funktioneller Peptide oder Proteine eingesetzt werden. Schematisch ist in Abbildung 4 aufgezeigt, wie eine solche Selektion grundsätzlich aufgebaut ist, und welche Schritte in einem Selektionszyklus durchlaufen werden müssen. Zuerst wird eine DNA-Bibliothek synthetisiert, welche aufgrund von randomisierten Bereichen die Diversität von bis zu 1015 verschiedenen Molekülen in sich birgt. Nach der Umschrift in RNA und der Verknüpfung mit dem Puromycin-Linker wird mit dem erhaltenen mRNA display templaBIOspektrum · 2/04 · 10. Jahrgang Special te eine in vitro Translation durchgeführt. Dabei werden die mRNA Display Peptide generiert und der verknüpfte RNATeil nach der Aufreinigung des Komplexes revers transkribiert. Mit den so hergestellten Molekülen wird der eigentliche Selektionsschritt hinsichtlich der gewünschten Eigenschaft durchgeführt. Nicht gebundene Mitglieder der Bibliothek werden entfernt, die gebundenen spezifisch eluiert und mittels PCR vervielfältigt. Reichern sich nach mehrmaligem Durchlaufen des Selektionszyklus funktionale Sequenzen an, so wird die finale Bibliothek kloniert und die erhaltenen Sequenzen werden analysiert und charakterisiert. Ergebnisse und Perspektiven Mit dieser Methodik sind mittlerweile einige sehr interessante Oligopeptide selektiert worden. Aus komplett randomisierten Proteinbibliotheken wurde eine Sequenz isoliert, die spezifisch ATP binden kann[9]. Hinsichtlich der Wahrscheinlichkeit, eine funktionale Sequenz in 1011 verschiedenen zu finden und der zusätzlichen Entdeckung eines Zn2+-abhängigen Motivs handelt es sich dabei um ein besonders aussagekräftiges Beispiel. Weiterhin wurde ein Streptavidin-bindendes Peptid selektiert, welches mit einer extrem niedrigen Bindungskonstante (Kd = 2 nM) eine hohe Affinität zum Proteintarget besitzt[10]. Das Phänomen des Sequenzraums wurde ebenfalls anhand der Selektion von Peptiden untersucht, die RNA-Motive binden können[11]. Weiterhin wurden durch das Screening von cDNA-Bibliotheken Proteine identifiziert, die an Bcl-X(L)[12] oder phosphoryliertes Tyrosin[13] binden. Von ROBERTS et al. wurden Strategien entwickelt, um nichtnatürliche Aminosäureseitenketten in ein Peptid zu integrieren[14]. So konnten Peptidkonjugate erzeugt werden, die an einer definierten Position einen Penicillinrest tragen[15]. Dies erlaubte die Generierung einer Hybrid-Peptid-Arznei-Bibliothek, welche dazu verwendet wurde, Penicillin-Peptide zu selektieren, welche an das Penicillinbindeprotein 2a (PB2a, S. aureus) binden. Die neuen Substanzen zeigen eine mindestens 100-fach höhere Aktivität als das Penicillinmolekül allein. Dieser Ansatz erlaubt es somit, die Effizient von bereits bestehenden Medikamenten durch die Entdeckung neuartiger Hybrid-Liganden zu erhöhen. Abb. 3: Das mRNA-DNA-Puromycin-Ligationsprodukt wird im Ribosom kovalent an das Peptid geknüpft BIOspektrum · 2/04 · 10. Jahrgang Special 204 [12] Hammond P.W., Alpin J., Rise C.E., Wright M., Kreider B.L.: In vitro selection and characterization of Bcl-X(L)-binding proteins from a mix of tissue-specific mRNA display libraries. J Biol Chem 2001, 276: 20898–20906 [13] Cujec T.P., Medeiros P.F., Hammond P., Rise C., Kreider B.L.: Selection of v-Abl Tyrosine Kinase Substrate Sequences from Randomized Peptide and Cellular Proteomic Libraries Using mRNA Display. Chem Biol 2002, 9: 253–264 [14] Frankel A., Li S., Starck S.R., Roberts R.W.: Unnatural RNA display libraries. Curr Opin Struct Biol. 2003 13: 506–512 [15] Li S., Roberts R.W.: A novel strategy for in vitro selection of peptide-drug conjugates. Chem Biol. 2003, 10: 233–239. [16] Raffler N.A., Schneider-Mergener J., Famulok M.: A novel Class of Small Functional Peptides that Bind and Inhibit Human alpha-Thrombin Isolated by mRNA Display. Chem Biol 2003, 10: 69–79. Abb. 4: Selektionsschema. (A) Ausgehend von der dsDNA der Bibliothek wird die mRNA, die mit dem Puromycin (P) verknüpft ist, erzeugt. (B) Durch in vitro Translation werden die mRNA Display Peptide hergestellt (Aminosäuren als graue Kreise dargestellt). (C) Nach der reversen Transkription wird die Bibliothek in den Selektionsschritt eingesetzt und auf die Eigenschaften gegenüber einem immobilisierten Targetmolekül untersucht. (D) Nach Entfernen der nicht-funktionalen Moleküle werden durch PCR die funktionalen Sequenzen angereichert Wir haben die mRNA Display Technologie auf die Selektion von Peptiden angewendet, die an humanes α-Thrombin binden können[16]. Unsere Experimente belegen, dass die selektierten Peptide in der Lage sind, die biologischen Funktionen von Thrombin in unterschiedlicher Weise zu modulieren. Die Analyse der einzelnen Sequenzen zeigte, dass ein kurzes Motiv aus den Aminosäuren DPGR entscheidend für die Interaktion zwischen Thrombin und dem Peptid ist. Unter den verschiedenen Techniken, die eine eindeutige Verknüpfung zwischen dem Geno- und dem Phänotyp einer Proteinsequenz mit der für sie kodierenden DNA erlauben, wurde das mRNA Display inzwischen stark weiterentwickelt und stellte sich als besonders leistungsstark heraus. Vor allem hinsichtlich der Einfachheit und Stabilität der Verknüpfung, sowie der erzielbaren Komplexität der Bibliotheken bietet es entscheidende Vorteile. Die genannten Beispiele zeigen, welches enorme Potenzial diese Technologie auszeichnet und man kann davon ausgehen, dass weitere bahnbrechende Entwicklungen folgen. Literatur [1] Wilson D.S., Szostak J.W.: In vitro selection of functional nucleic acids. Annu Rev Biochem 1999, 68: 611–647. [2] Kettling U., Koltermann A., Eigen M.: Evolutionary biotechnology – reflections and perspectives. Curr Top Microbiol Immunol 1999, 243: 173–186. Korrespondenzadresse: Prof. Dr. Michael Famulok Rheinische Friedrich-Wilhelms-Universität Bonn Kekulé-Institut für Organische Chemie und Biochemie Gerhard-Domagk-Straße 1 D-53121 Bonn Tel.: 0228-73-5661 Fax: 0228-73-5388 [email protected] [3] Geysen H.M., Meloen R.H., Barteling S.J.: Use of peptide synthesis to probe viral antigens for epitopes to a resolution of a single amino acid. Proc Natl Acad Sci USA 1984, 81: 3998–4002. [4] Schwarz A., Famulok M.: Aptamere und Ribozyme aus kombinatorischen Nukleinsäure-Bibiotheken. Biospektum 2001, 7: 111–114. [5] Smith G.P., Petrenko V.A.: Phage Display. Chem Rev 1997, 97: 391–410. [6] Plückthun A.: Ribosomen Display: In vitro-Selektion und -Evolution von Proteinen. Biospektrum 2000, 5: 374. [7] Hoffmüller U., Schneider-Mergener J.: In vitro evolution and selection of proteins: Ribosome display for larger libraries. Angew Chem Int Ed Engl 1998, 37: 3241–3243. [8] Roberts R.W.: Totally in vitro protein selection using mRNA-protein fusions and ribosome display. Curr Opin Chem Biol 1999, 3: 268–273. [9] Keefe A.D., Szostak J.W.: Functional proteins from a random-sequence library. Nature 2001, 410: 715–718. [10] Wilson D.S., Keefe A.D., Szostak J.W.: The use of mRNA display to select high-affinity protein-binding peptides. Proc Natl Acad Sci USA 2001, 98: 3750–3755. [11] Barrick J.E., Takahashi T.T., Ren J., Xia T., Roberts R.W.: Large libraries reveal diverse solutions to an RNA recognition problem. Proc Natl Acad Sci USA 2001, 98: 12374–12378. BIOspektrum · 2/04 · 10. Jahrgang