molekulare schalter
Werbung

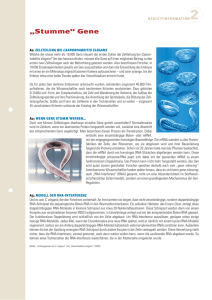
52 ADJUNCT INVESTIGATORS REGULATORISCHE RNA-MOLEKÜLE MOLEKUL ARE SCHALTER BEATRIX SÜSS Beatrix Süß, geboren 1971 in Sonneberg, hat in Greifswald und Erlangen Biologie studiert und an der Friedrich-Alexander-Universität Erlangen-Nürnberg im Jahre 1998 promoviert. Im Anschluss begann sie, ihre eigene Arbeitsgruppe aufzubauen und habilitierte im Jahre 2007 mit dem Thema »Aufbau und molekulare Charakterisierung RNA-basierter konditionaler Genexpressionssysteme«. Forschungsaufenthalte führten sie zu Prof. Renée Schroeder an die Universität Wien und zu Prof. Ron Breaker an die Yale University. 2007 wurde sie auf die Stiftungsprofessur der Aventis Foundation für Chemische Biologie an die Goethe-Universität berufen. Seit 2009 ist Beatrix Süß Adjunct Investigator des CEF. Kristallstruktur eines tetracyclinbindenden RNA-Aptamers, das als synthetischer RNA-Schalter eingesetzt werden kann. Inseriert in den untranslatierten Bereich einer mRNA inhibiert es die Translation, jedoch nur in Anwesenheit des Liganden. Quelle: Xiao et al., Chem Biol. 2008, 15:1125-37. L ange Zeit sah man in den Ribonukleinsäuren (RNA) nur die Überträger der genetischen Information von der DNA, dem Speicher der Erbsubstanz, zu den Ribosomen, dem Syntheseort der Proteine. In den vergangenen Jahren hat sich dieses Bild jedoch dramatisch geändert, als man eine Vielzahl an RNA-Molekülen entdeckte, die wichtige regulatorische Funktionen in biologischen Systemen wahrnehmen. Das Spektrum dieser in den vergangenen Jahren neu entdeckten regulatorischen RNA-Molekülen ist sehr groß. Es gibt kleine RNA-Moleküle, die nicht in ein Protein übersetzt werden. Diese findet man in großer Zahl in allen Domänen des Lebens. Diese, als nichtkodierende RNAs bezeichneten Moleküle können entweder an eine mRNA binden und dadurch verhindern, dass diese in ein Protein übersetzt wird, oder sie binden direkt an ein Protein und inhibieren damit seine Funktion. Neben den kleinen, nichtkodierenden RNA-Molekülen gibt es eine weitere Gruppe regulatorischer RNA-Moleküle, die RNASchalter. RNA-Schalter sind Strukturele- mente, die in der mRNA vor dem Genstart lokalisiert sind und eine hochselektive Bindedomäne für einen Liganden ausbilden. Die Bindung des Liganden führt über Strukturänderungen in der RNA zu einer veränderten Genexpression. Diese Schalter sind einfach aufgebaut und eignen sich deshalb hervorragend als Vorbild für die Entwicklung synthetischer RNA-Schalter. Ziel unserer Forschung ist es, regulatorische RNA-Moleküle zu finden, diese funktional und strukturell zu charakterisieren und das Wissen daraus zu nutzen, um gezielt maßgeschneiderte RNA-basierte Schalterelemente aufzubauen und anzuwenden.