Mutation & Rekombination
Werbung

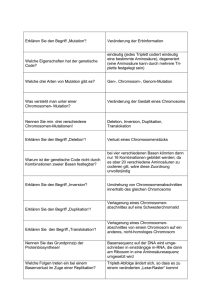
Mutation & Rekombination Aufbau des Vortrages -Grundlagen•Arten von Mutationen •Mechanismus von Mutationen •Mutagene (chem. & phsysikal.) •Reparaturmechanismen Spotlights on... •...warum die DNA Thymin statt Uracil enthält •...was passiert wenn Reparaturmechanismen nicht richtig funktionieren (z.B. Krebs) •...was Trinucleotidwiederholungen sind •...wie man überhaupt feststellt, dass Stoffe karzinogen sind Arten von Mutationen •Substitution eines bp* →am häufigsten •Deletion eines/ mehrerer bp Wegfall des/ der bp •Insertion (=Addition) eines/mehrerer bp des/ der bp Ein bp wird durch ein anderes ersetzt Hinzufügen Die Substitution von bp... ...kann unterteilt werden in: •Transition → Purin wird gegen Purin getauscht •Transversion → Purin gegen Pyrimidin und andersrum (oder Pyrimidin gegen Pyrimidin) Watson & Cricks Vorschlag für den Mechanismus von Mutationen •Vorhandensein von Tautomeren bei den 4 Basen der DNA: R-NH2 ↔ R=NH (Amino-Imino-Tautomerie) C=O ↔ =C-OH • (Keto-Enol-Tautomerie) Anteil der Tautomere: 10-4 bei jeder Base Was passiert nun mit den Tautomeren? •Normal: A---T und C---G •Jetzt: A*---C A*= Iminotautomer des Adenins •→ Thymin wurde gegen Cytosin ausgetauscht; ohne Korrektur liegt eine Mutation vor •Bei nächster Replikation: → paart mit G (falsches bp) Chemische Mutagene Basenanaloga, HNO2 und Acridine A* → A → paart mit T (richtiges bp) C Basenanaloga •Z.B. 5-Bromuracil; 2-Aminopurin •Haben eine noch größere Wahrscheinlichkeit als die normalen Nucleinsäurebasen, die tautomere Form zu bilden, welche (s.o.) zu einer Transition führt •Beispiel 5-Bromuracil: ist ein Thyminanalogon, d.h. Es paart normalerweise mit Adenin; ABER: höhere Enolisierungstendenz wegen des Bromatoms (Br höhere EN als →Folge: Paarung CH3-Gruppe beim Thymin) mit Guanin (statt mit Thymin) und . bei nächster Replikation C—G statt A--T HNO2 •Wirkt durch chemische Modifikation der DNA-Basen •Reagiert mit den Aminogruppen der Basen (sogen. Oxidative Desaminierung) •Adenin Desaminierung Hypoxanthin paart mit C statt mit T Cytosin → Uracil paart mit A statt G Guanin → Xanthin paart mit C •Folge: AT ↔ GC Transitionen Acridine •Flache aromatische Moleküle •Interkalieren in die DNA, d.h. Sie schieben sich zwischen benachbarte bp •Folge: Insertion oder Deletion eines/ mehrer bp •Folge: Das Leseraster bei der Translation verschiebt sich, außer es wurden 3xY bp deletiert/ inseriert •Beispiel? 1 Triplett codiert für 1 Aminosäure... DER BÄR MAG DEN AAL Schieben wir ein A dawzischen DAE RBÄ RMA GDE NAA ... →Leserastermutation Stoffe, die erst durch Enzyme zu Mutagenen werden •Enzyme unseres Körpers können Stoffe in Mutagene umwandeln: •Aflatoxin B1 Cytochrom P450 Hochreaktives Epoxid •Reagiert mit Guanosin •Folge: G—C ↔ A—T Transversion Physikalische Mutagene Am Bsp. UV-Licht UV-Licht als Mutationsfaktor •UV-Licht bewirkt, dass benachbarte Pyrimidinreste in einem DNA-Strang kovalent binden •Folge: Das Pyrimidin-Dimer passt nicht mehr in die Doppelhelix; •Replikation und Genexpression kommen zum Stillstand, bis die Mutation behoben ist Die DNA- Reparatur Direkte Reparatur, Basenexcisionsreparatur, Nucleotidexcisionsreparatur Direkte Reparatur •Bsp. hierfür: photochemische Spaltung von Pyrimidindimeren •Fast alle Zellen des Körpers besitzen es: ein photoreaktivierendes Enzym mit Namen DNA-Photolyase •Das Enzym aus E.coli hat 2 Cofaktoren gebunden: N5,N10Methenyltetrahydrofolat & Flavinadenindinucleotid •Enzym haftet sich an Dimer-Stelle; Lichtenergie wird von einem der Cofaktoren absorbiert; Anregung; Spaltung des Dimers durch genutzte Lichtenergie Basenexcisionsreparatur Bsp. hierfür: Ausschneiden modifizierter Basen (z.B. Methyladenin)aus dem DNA-Strang am Enzym AlkA aus E.coli Ablauf • • • • • • • AlkA bindet an schadenhafte DNA, fehlerhafte Base „springt“ ins aktive Zentrum des Enzyms Glykolasewirkung: Spaltung der glykosidischen Bindung; Freisetzung der fehlerhaften Base. DNA-Rückgrat bleibt erhalten Entstehung eines „Lochs“ = AP-Stelle AP-Endonuklease erkennt AP-Stelle; spaltet DNA-Rückgrat rechts + links von Fehlstelle Desoxyribose-Phosphodiesterase schneidet verbliebene Desoxyribosephosphateinheit heraus DNA-Polymerase I fügt zum Gegenstrang komplementäres Nukleotid ein DNA-Ligase schließt den Strang wieder. Fertig! Struktur eines DNA-Reparaturenzyms: Nucleotidexcisionsreparatur →Herausschneiden eines Pyrimidindimers bei E.coli Ablauf • Enzymkomplex aus Proteinen, die durch uvrABC-Gene codiert werden, entdeckt die Verzerrung im DNA-Strang • Spezielles uvrABC-Enzym (hochspezifische Excinuclease) schneidet die schadhafte Stelle heraus • DNA-Polymerase I führt Reparatursynthese aus; Komplementärstrang dient wie üblich als Matritze • DNA-Ligase verknüpft das neue mit dem alten Stück. Fertig! Excisionsreparatur: Reparaturmechanismen im Überblick: Warum die DNA Thymin statt Uracil enthält... •Beide Basen paaren mit Adenin •Einziger Unterschied: Thymin hat anstelle des H am C5 ein CH3 •Erklärung: es existiert ein Reparatursystem zur Reparatur von desaminiertem Cytosin: Reparatursystem für desaminiertes Cytosin •C desaminiert zu einem merklichen Anteil in der DNA spontan zu Uracil; es käme also bei der Replikation zu U—A statt zu C—G •Ein Reparatursystem erkennt U als fremdes Element der DNA •Das Reparaturenzym Uracil-DNA-Glykolase hydrolysiert die glykosidische Bindung zwischen Uracil- und Desoxyriboseeinheiten •Reparatur durch Einbau von Cytosin in die AP-Stelle •Thyminhaltige Nukleotide werden nicht angegriffen •CH3-Gruppe als Marker, der Nukleotide vor der Uracil-DNA-Glykosylase schützt •Wäre Uracil in der DNA, könnte man „echtes“ nicht von „unechtem“ (=desaminiertes C) unterscheiden •Da RNA nicht repariert wird, kann dort U statt T vorkommen → energetisch nicht so aufwendiger Baustein Reparatur von Uracil: Krebsarten enstehen u.a. durch fehlerhafte DNA-Reparatur •i.A. entsteht Krebs durch Mutationen in Genen, die mit Wachstumskontrolle zu tun haben •Ist ein Reparatursystem defekt, steigt Mutationshäufigkeit →Folge: erhöhtes Krebsrisiko •Bsp. Xeroderma pigmentosum und Lynch-Syndrom Xeroderma pigmentosum •Viele Patienten sterben noch vor dem 30. Lebensjahr an bösartigen Hauttumoren →extreme Lichtempfindlichkeit •Es es entstehen durch UV-Licht Pyrimidindimere (siehe E.coli); Reparaturmechanismus beim Menschen ähnlich •Ursache dieser Krankheit nun geklärt:Die Excinuklease, die schadhafte Pyrimidindimere entfernt, ist defekt. Es werden kaum Dimere entfernt. Lynch-Syndrom •Auch genannt: erbliches, nicht polypöses kolorektales Karzinom = HNPCC •Hierbei ist die DNA-Fehlpaarungsreparatur defekt •Mutierte verantwortliche Gene: hMSH2 und hMLH1 „Trinucleotidwiederholungen“ •Manche genetisch bedingte Erkrankungen entstehen durch die Vermehrung von Wiederholungseinheiten aus 3 Nukleotiden •Bsp.: Huntington-Krankheit Huntington-Krankheit •Nervenleiden •Ein Gen ist mutiert: Sequenzen von CAG wiederholen sich immer wieder •Das hierdurch codierte Protein (Huntingtin) hat daher langen „Schwanz“ von Glutaminresten •Bei der Vererbung wird dieser Schwanz i.d.R. immer länger; Symptome treten bei den Kindern noch früher auf →Antizipation Vermehrung von Triplettwiederholungen: Wie lassen sich potentielle Karzinogene nachweisen? →Bruce Ames entwickelte den AmesTest: Agar ausbringen Salmonellen in •Diese spez. Salmonellen können auf Grund einer Mutation leider kein Histidin selbst synthetisieren man muss es ihnen zum Überleben zur Verfügung stellen ☺ Der Ames-Test: •Gibt man nun ein Mutagen auf die Platte, entstehen viele neue Mutanten – einige von ihnen können können wieder His synthetisieren (Rückmutanten = Reservanten) •Die Teststämme sprechen auf bp-Substitutionen, Deletionen oder Additionen an •Die Empfindlichkeit der Stämme ist erhöht durch a) unvollständige Reparatursysteme Lipopolysaccharidhülle b) unvollständige •Folge: Mutagene können leichter eindringen •Hinzufügen von Säugetierleberhomogenisat (Warum?) •→preiswerter und einfacher Test um mutagenes Risiko vieler Substanzen abzuschätzen ENDE Fragen?