LMU
Werbung

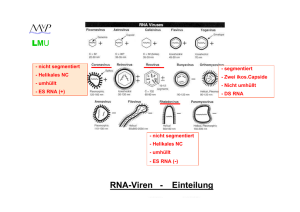
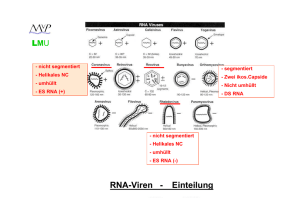
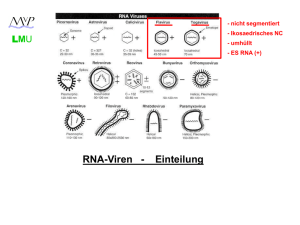
Pflanzenviren Teil II LMU H N 2 0 0 8 LMU PflanzenViren Caulimoviren Geminiviren Comoviren Tobamoviren Potyviren H N 2 0 0 8 Pflanzenviren Teil I LMU Pflanzenviren II Poty-, Gemini,- Caulimo-, Baculoviren Potyviren: große wirtschaftliche Bedeutung, über 100 verschiedene Viren, fast alle Nutzpflanzen können betroffen sein (Protease-Inhibitoren) Geminiviren: einzige bedeutende Familie mit ss-DNA, bidirektionelle Expressions-Strategie, “rolling-circle” Replikation. Caulimoviren: Transkriptase, Regulationselemente für H N 2 0 0 8 “Pararetroviren” im Pflanzenreich, reverse genetische Transformation von Pflanzen Baculoviren: Insektenviren, eukaryontische Genexpression Potyviren LMU Eigenschaften: positive ss-RNA, 9600 nt 700 – 900 nm lang, filamentös (flexible Stäbchen) Durchmesser 12 -15 nm RNA mit 3' poly A und covalent geb. VPg am 5' ein großes ORF ein Polyprotein, das dann proteolytisch gespalten wird (ähnlich Poliovirus) Transmission: aphids (Blattläuse) fungi (Pilze) mite (Milben) H N 2 0 0 8 wirtschaftl. Obwohl meist enges Wirtsspektrum: fast jede Bedeutung: Nutzpflanze kann von Potyviren infiziert werden (> 100 versch. Potyviren). Meist deutliche Symptomausbildung – oft totaler Ernteverlust. Potyviren LMU Copyright ©2002, UF Department of Plant Pathology PO Box 110680, Gainesville, FL 32611-0680 (352) 392-3631, Fax: (352) 392-6532. Plum pox potyvirus induced chlorotic rings/blotches on nectarine. Photograph courtesy P. Gentit, Ctifl, France. H N 2 0 0 8 Potyvirus LMU http://www.uq.edu.au/vdu/VDUPotyvirus.htm H N 2 0 0 8 PROTEIN POSSIBLE FUNCTION(S) P1 Proteinase; Cell-to-cell movement (speculation). HC-Pro Aphid mediated transmission; Proteinase; Cell-to-cell movement (speculation). P3 Unknown (possible role in replication) CI Genome replication (RNA helicase); Membrane attachment; Nucleic acid stimulated ATPase activity; Cell-to-cell movement (speculation). CP RNA encapsidation; Involved in vector transmission; Cell-to-cell movement. NIa-VPg Genome replication (Primer for initiation of RNA synthesis). NIa-Pro Major Proteinase NIb Genome replication (RNA-dependent RNA polymerase [RdRp]). 6K1 & 6K2 Unknown, but possible roles in: - RNA replication; - Regulatory function inhibiting NIa nuclear translocation; - Membrane anchoring of replication machinery. Table 1: Potyvirus proteins and their known and speculated functions. LMU H N 2 0 0 8 Potyvirus Expression Ein großes Polyprotein wird durch Proteasen in funktionelle Proteine gepalten. Potyviren LMU H N 2 0 0 8 Tobacco Etch virus (TEV) LMU Functional Dissection of Naturally Occurring Amino Acid Substitutions in eIF4E That Confers Recessive Potyvirus Resistance in Plants W OA Potyviren Inhwa Yeam,a,1 Jason R. Cavatorta,a Daniel R. Ripoll,b Byoung-Cheorl Kang,a,c and Molly M. Jahna,d,2 a Department of Plant Breeding and Genetics, College of Agriculture and Life Sciences, Cornell University, Ithaca, New York 14853 b Computational Biology Service Unit, Life Sciences Core Laboratories Center, Cornell University, Ithaca, New York 14853 c Department of Plant Science, College of Agriculture and Life Sciences, Seoul National University, Seoul 151-742, Republic of Korea d College of Agricultural and Life Sciences, University of Wisconsin, Madison, Wisconsin 53706 Naturally existing variation in the eukaryotic translation initiation factor 4E (eIF4E) homolog encoded at the pvr1 locus in Capsicum results in recessively inherited resistance against several potyviruses. Previously reported data indicate that the physical interaction between Capsicum-eIF4E and the viral genome-linked protein (VPg) is required for the viral infection in the Capsicum-Tobacco etch virus (TEV) pathosystem. In this study, the potential structural role(s) of natural variation in the eIF4E protein encoded by recessive resistance alleles and their biological consequences have been assessed. Using highresolution three-dimensional structural models based on the available crystallographic structures of eIF4E, we show that the amino acid substitution G107R, found in many recessive plant virus resistance genes encoding eIF4E, is predicted to result in a substantial modification in the protein binding pocket. The G107R change was shown to not only be responsible for the interruption of VPg binding in planta but also for the loss of cap binding ability in vitro, the principal function of eIF4E in the host. Overexpression of the Capsicum-eIF4E protein containing the G107R amino acid substitution in Solanum lycopersicum indicated that this polymorphism alone is sufficient for the acquisition of resistance against several TEV strains. H N 2 0 0 8 (B) View of the molecular surface area of a model of CapsicumeIF4E with the amino acid substitutions colored as follows:o acid substitutions colored as follows: T51A and P66T in green; G107R in red; V67E, L79R, and D109N in yellow; and 7-methylGDP in magenta. Geminiviren LMU als Beispiel für antivirale Strategien: 1. Movement Suppression 2. Defective Interfering Particles (DI) 3. Antisense RNA H N 2 0 0 8 Geminiviren LMU Eigenschaften: positive ss-DNA, 2500 - 3000 nt, sehr kleines zirkuläres Genom Replikation im Zellkern („rolling circle“) Transmission: weisse Fliege ist häufigster Vektor wirtschaftl. Vorkommen in Karibik, Amerika, Südostasien, Bedeutung: Indien, Mittelmeerraum; bevorzugt tropische Nutzpflanzen z.B. Zuckerrohr, aber auch Mais Electron micrograph of plant virus prepared by Dr RG Milne H N 2 0 0 8 Istituto di Fitovirologia Applicata Strada delle Cacce 73 10135 Torino Italy Copyright notice: This notice must accompany any copy of the images. The images must not be used for commercial purpose without the consent of the copyright owners. The images are not in the public domain. The images can be freely used for educational purposes. Geminiviren LMU Dikotyle Pflanzen: Geminiviren meist bipartite Geminiviren meist monopartite Übertragung: weisse Fliege Übertragung: häufig Zikaden Komponente A meist ausreichend für H N 2 0 0 8 Monokotyle Pflanzen: Koponente B für systemische Ausbreitung, Replikation in einer braucht Komponente A zur Zelle, kodiert coat- Replikation Protein Geminiviren LMU H N 2 0 0 8 A simplified version of the geminivirus DNA replication cycle and intercellular movement of viral DNA. Geminivirus DNA replication occurs in two stages. First, the ssDNA is converted into dsDNA with the participation of cellular factors. The dsDNA serves as template for viral gene expression. Secondly, the dsDNA initiates the rolling circle phase, with the participation of viral and cellular factors, to produce new ssDNA products. These can (i) reenter the DNA replication pool, (ii) associate with CP or (iii) be transported outside the nucleus and to the neighbouring cell, most probably through plasmodesmata, with the help of viral MPs. The EMBO Journal (2000) 19, 792–799, doi:10.1093/emboj/19.5.792 Geminiviren LMU www.cals.ncsu.edu/.../geminivirus.jpg H N 2 0 0 8 TGMV and CaLCV both have two components, designated A and B, which are similar in size. The common region (CR) is indicated by black boxes. The CR contains the viral origin of DNA replication and is conserved between the A and B components, but varies between members of the begomoviruses. The A component encodes genes needed for replication, gene expression, and encapsidation. AL1 is also known as AC1(required for replication), AL2 as AC2, and AL3 as AC3. The B component contains two proteins needed for viral movement, BR1 (BV1) and BL1 (BC1). Geminiviren LMU bidirektionelle ExpressionsStrategie: v: virus sense genes c: complentary sense genes H N 2 0 0 8 intranukleäre dsDNA-Formen mit zwei “non-codingRegionen“ 1. Promotoren 2. zwei Polyadenylatoren coat-Protein (V1) nicht notwendig für Replikation aber Transmission. ACMV: African cassava mosaic virus BCTV: Beet curly top virus (single component virus) WDV: Wheat dwarf virus Geminiviren – Movement Supression LMU H N 2 0 0 8 Geminiviren-Movement Supression LMU Testsystem: Koinokulation von TGMV BL1 Gen und DNA B von ACMV führt zur Reduzierung systemischer Symptome. Mechanismus: Einbau des BL1 Proteins (NH2-terminale Sequenz) inhibiert ACMV-movement. Transdominant negative Mutante BC 1 BC 1 H N 2 0 0 8 ACM V BC 1 BC 1 BL 1 BC 1 Geminiviren- Defective Interfering Particles LMU H N 2 0 0 8 Defective Interfering (-RNA/-DNA/-particles/-viruses: DIs haben ein unvollständiges Genom und sind auf “Helferviren” zur Replikation angewiesen. Ihre Genome sind verkürzt und enthalten in der Regel 5' und 3'Enden der entsprechenden Helferviren, sowie ein Verpackungssignal Ω. Sie besitzen auf Grund ihres kurzen Genoms einen Replikationsvorteil und interferieren (negativ) mit der Replikation des Helfervirus. Große Mengen an DIs können bei Infektionsversuchen die Ausbeute an vollständig intakten, replikationsfähigen Viren erheblich senken. Bsp.: Ω Ω DI Helfervirus DIs LMU Positivkontrolle H N 2 0 0 8 Protektion durch DIs Geminiviren- Defective Interfering Particles LMU Defective Interfering (-RNA/-DNA/-particles/-viruses): Bsp.: African cassava mosaic virus (vorheriges Bild) Defekte DNAs leiten sich ausschließlich von DNA B her (BV 1 komplett deletiert, BC 1 Carboxy-Terminus deletiert). DIs inhibieren generell die Produktion von DNA A und B. Da aber DNA A und B für die systemische Infektion notwendig sind, ist es bei geringer Konzentration der DNA B wenig wahrscheinlich, dass es unter diesen Bedingungen zur gleichzeitigen Infektion einer Zelle mit A und B kommen kann. H N 2 0 0 8 Dies führt zu einer Reduktion der systemischen Ausbreitung. DIs interferieren auf der Ebene der DNA-Replikation DIs kompetieren mit Helfervirus um transaktivierende Proteine Antisense RNA LMU Inhibition der Virusreplikation durch Antisense-RNA: Prinzip: Zur viralen RNA komplementäre RNA soll die Replikation inhibieren (Störung der Polymerase, ...). Häufig werden hierbei Antisense-RNAs eingesetzt, deren Bindung sich gegen Sequenzen viraler Replikasen richtet, da diese Gene essentiell sind und deren Expression nur auf relativ geringem Niveau erfolgt. Probleme: Antisense RNAs werden nur in relativ geringer Menge transkribiert, ihre Expression ist genetisch nicht stabil, schnelle “Rückmutation”. H N 2 0 0 8 Praktisches Beispiel für Antisense-RNA-Strategie: Entsprechende, gentechnisch veränderte Pflanzen, zeigen bei Infektion mit dem Tomato Golden Mosaic Virus eine Reduktion der Zahl von Pflanzen mit Symptomen einer Virusinfektion. Retroviren und Pararetroviren LMU H N 2 0 0 8 4 Typen von Viren mit Reverser Transkriptase Aktivität: Retroviren Hepadnaviren (HBV) Badnaviren (Bacilliforme Pflanzenviren, wenig untersucht) Caulimoviren Genom: RNA “echte” Retroviren Genom: DNA Pararetroviren Integration in das Zellgenom Mehrere nicht integrierte “Minichromosomen” im ZK Beiden Gruppen gemeinsam: gag und pol Proteine env: bei Caulimoviren nicht vorhanden (unbehüllt), hier wurden „Reste“ von env durch Sequenzhomologie identifiziert ORF VI (scaffolding ?) LMU Caulimoviren Viren mit reverser Transkription (Pararetroviren) ds-DNA, 7800 – 8200 bp, zirkulär keine Hülle, dafür ein mehrschichtiges Kapsid Ikosaedrisch, Durchmesser 45 – 50 nm Übertragung häufig durch Blattläuse (wie z.B. CaMV) DNA Replikation durch RT eines „full-length“-Transkriptes Priming der DNA-Synthese durch t-RNAMet H N „strand-switching“ 2 0 0 8 Genome: Quelle für Regulationselemente in der Gentechnik Genom mit ungewöhnlicher Topologie (3 Formen) Caulimoviren – Symptome bei Infektion LMU Vergilbung der Blätter Mosaikartige Blattaufhellungen, Streifenbildung Aufhellung der Blattadern Reduziertes Wachstum eingerollte Blätter Farbveränderungen (Abnahme der grünen Blattpigmente) Blattränder erscheinen gewellt, „ausgefranst“ Verkürzte Internodien Uncharakteristisches, buschiges Wachstum H N 2 0 0 8 Verzweigung verändert Auftreten von Nekrosen (absterbende Gewebebereiche) Caulimoviren – Genom LMU ORF I Movement protein ORF II Insect transmission factor ORF III minor struct. Pr. ORF IV Capsid protein VII VI II ORF V Protease, reverse transcriptase and RnaseH CaMV 8000 bp ORF VI Translational activator / Inclusion H N 2 0 0 8 body protein ORF VII Unknown (dispensable) I V III IV Cauliflower Mosaic Virus - Genom LMU 3 Genom-Formen 1. Twisted Virus-DNA Drei Lücken in der DNA an defin. Stellen: eine im (-) strang zwei im (+) strang 2. Supercoiled DNA, „Minichromosom“ Transkriptionstemplate Expression durch zwei Promotoren kontrolliert (P) einmal für genomische 35S RNA + subgenom. 19S RNA H N 2 0 0 8 3. 35S RNA Template für Translation und Reverse Transkript. CaMV - Proteine LMU ORF Protein I P1, cell to cell movement (durch Plasmodesmata) II P2, transmission III minor structural protein P3 gag IV major coat protein P4 (phosphoryliert) gag V Rev. Transkr. (PR aber keine INT), Aspartat-PR pol VI P6 „verkrüppeltes“ env, Transaktiv. der Translat. “env” VII kodiert vermutlich kein Protein, involviert in Expression der 35S polycistronischen mRNA H N 2 0 0 8 LMU CaMVLebenszyklus beide CaMV RNAs werden posttranskriptionell polyadenyliert (AATAAA) 35S RNA: 600 Basen leader Sequenz Genprodukt ORF VI stabilisiert 5'-Ende der 35S-RNA und erhöht Translation H N 2 0 0 8 Insertionen in Genprodukt VI attenuieren Schwere der Symptome bei Infektion LMU H N 2 0 0 8 CaMV – Genregionen mit Einfluss auf die Symptomausbildung LMU Viren bei Invertebraten Baculoviren H N 2 0 0 8 Baculoviren LMU 400 verschiedene Baculoviren bekannt ds-DNA, ca. 129.000 bp, zirkulär, supercoiled, DNA im Kern behüllt Ikosaedrisch, Durchmesser 45 – 50 nm Zuerst: Immediate Early Gene Transkription (zelluläre Polymerasen) Immediate Early Genes aktivieren Delayed Early Genes, danach folgt die DNA-Replikation (12 h p.i.) Late Genes (z.B. Glycoprotein gp41, Kapsid Protein p24, ...) H N 2 0 0 8 Very Late Genes (18 h p.i.), p10, u. Polyhedrin- Expression, Polyhedrin kann in Zellkultur deletiert und Fremdgene stattdessen einkloniert werden. Baculoviren – Autographa californica LMU Am besten untersuchter Vertreter der Baculoviren: Autographa californica nuclear polyhedrosis virus Wirte: Arthropoden, Insekten, besonders Schmetterlinge sonst keine bekannten Wirte Für die Larven von Insekten verläuft die Infektion fatal, hohe Virustiter H N 2 0 0 8 Insektenlarven „verflüssigen“ sich auf Grund der Infektion, daher z.T. Einsatz als Insektizid. Baculoviren - Genexpression LMU Baculoviren interessant wegen der Möglichkeit der Expression rekombinanter Proteine. Vorteile: Modifikation von Proteinen in eukaryontischem System im Gegensatz zu E. coli, hier: Glycosilierung, post-translational processing, trafficking (Sekretion), native Proteine mit entsprechenden antigenen und immunogenen Eigenschaften H N Hohe Expressionsrate (bis zu 50% des Gesamt-Zell-Proteins) 2 0 0 8 Gene bis zu ca. 10.000 bp sind exprimierbar Baculoviren – 2 Phänotypen LMU Beim Durchlaufen eines kompletten Lebenszyklus treten zwei völlig unterschiedliche Phänotypen des Virus auf. 1. PDV (Polyhedra-derived-virus) Initiation der Infektion von Larven, die Virus z.B. durch Nahrung aufnehmen. Parakristalline Proteinstruktur. Großteil des Materials ist Polyhedrin („occlusion bodies“). Sehr stabil, 1-5 µm, lichtmikroskopisch sichtbar. H N 2 0 0 8 BV (oder EV), budded virus / extracellular virus Für Zell-zu-Zell Transport im Wirt. Budding von Zellmembran als einzelnes Nukleokapsid, lose Hülle aus env gp64, surface projections, nie „occluded forms“ LMU Baculovirus Polhedra Lichtmikroskopische Darstellung H N 2 0 0 8 Baculoviren - BV/PDV LMU H N 2 0 0 8 Beide Formen genetisch völlig identisch, aber morphologisch und in Bezug auf Infektiosität und Antigenität völlig verschieden Oral appliziert: PDV 25000 x infektiöser als BV in Zellkultur: BV (EV) 1000 x infektiöser als PDV Baculovirus - Lebenszyklus LMU H N 2 0 0 8 Baculovirus – rekombinante Expression LMU Hohe Expressionsraten (Effizienz des Promotors?) Wegen der Größe des Genoms: Einbringen von Fremdgenen oft über Plasmide/Rekombination Wichtig ist eine 12 nt Transkriptions-Initiations-Sequenz, aber diese Sequenz findet sich auch bei anderen Genen mit geringeren Expressionsraten Zytopathogenese wird durch „shut-down“ der Synthese von Makromolekülen des Wirtes getriggert H N 2 0 0 8 Radikale Restrukturierung des Zytoskeletts („Verflüssigung“) LMU Baculovirus Expressions Systeme Diverse kommerzielle Systeme verfügbar H N 2 0 0 8 Baculovirus – Transfer - Vectors LMU H N 2 0 0 8