1 Struktur von Biomolekülen
Werbung

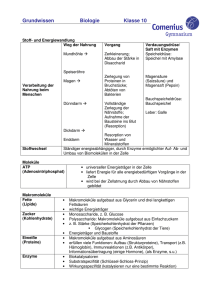
1 Struktur von Biomolekülen Modul 10-202-2208 Bioinformatik von RNA- und Proteinstrukturen Jana Hertel Lehrstuhl Bioinformatik 8. April 2013 Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 1 / 21 Organisatorisches Vorlesung 08.04.2013 - 03.06.2013, Montags 12:45 - 16:00 Uhr Seminar 24.06.2013 - 28.06.2013, Nachmittags/Abends Praktikum 28.06.2013 - Vorbesprechung 01.07.2013 - 12.07.2013, ganztägig (ca.8h) Alle Veranstaltungen nden im Raum 109, Härtelstr. 16-18 statt. (Auÿnahmen werden auf unserer Website o. in der Vorlesung bekannt gegeben) Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 2 / 21 Inhalt heute 1 4 Molekülstrukturen Experimentell Allgmeines 2 Strukturbestimmung Berechnung Struktur von RNA Vorhersage Primärstruktur Sekundärstruktur Tertiärstruktur 3 Struktur von Proteinen Primärstruktur Sekundärstruktur Stukturen höherer Ordnung Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 3 / 21 1 Molekülstruktur .. räumliche relative Anordnung der Atome in einem Molekül. Wie erhält man diese? → experimentell, präzise Berechnung, Vorhersage Beschreibung durch: kartesische Koordinaten (x , y , z ) interne Koordinaten (Bindungslängen, Bindungswinkel) → wohldenierte Zusammensetzung Grundzustand = Zustand mit niedrigster Energie. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 4 / 21 2 Struktur von RNA Primärstruktur - Bausteine der RNA: Basen Purine (Adenin, Guanin) Pyrimidine (Cytosin, Uracil)) Ribose Phosphat Verbindung 2er Nucleotide durch Phosphatgruppe → negative Ladung am Phosphatrest Human miRNA let-7a1: UGGGAUGAGGUAGUAGGUUGUAUAGUUUUAGGGUCACACCCACCACUGGGAGAUAACUAUACAAUCUACUGUCUUUCCUA Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 5 / 21 Sekundärstruktur von RNA .. Interaktionen zwischen den Basen der Nukleotide Wasserstobrückenbindungen + Stapelwechselwirkungen nicht-benachbarte Basen benachbarte Basen(paare) Energiegewinn gering energetisch sehr günstig Jede der 4 Basen kann mit einer anderen Base ≤3 H2 -brückenbindungen bilden. RNA einzelsträngig, faltet auf sich selbst. Kanonische (Watson-Crick, WC) Paare: Purin mit Pyrimidin; AU und GC Wobble-Basenpaar: GU und AC Basiselemente der Sekundärstruktur sind: Helices, hairpin-loops, Bulges/Interne Loops und Verzweigungsloops Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 6 / 21 Loops Hairpin-Loop Loop, der Helix überbrückt Spezialfall:extra-stabile Tetraloops thermodynam. am stabilsten Bulges, Interne Loops unterbrechen Helices an nicht-komplemenären Bereichen einseitig → Bulge, zweiseitig biegen, knicken Helices → → Interner Loop - Einuss auf Tertiärstruktur Verzweigungsloops verbinden >2 Helices können ungepaarte Basen enthalten Destabiliserende Wirkung von Loops abhängig von deren Gröÿe (Anzahl ungepaarter Basen), Sequenz und Art der Randbasenpaare. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 7 / 21 Tertiärstruktur von RNA .. zusätzlicher H2 -brückenbindungen und Stapelwechselwirkungen werden gebildet für biologische Aktivität vieler RNAs unabdingbar häug Wechselwirkungen zwischen Loops, o. zw. Loop und freien Enden schnelle Bildung der Sekundärstruktur aus Primärstruktur vs. langsamere Entstehung der Tertiärstruktur Annahme: Tertiärstruktur wird aus Sekundärstruktur gebildet ohne diese maÿgebliche zu beeinussen. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 8 / 21 3 Struktur von Proteinen Proteine sind Polymere, deren Bausteine 20 verschiedene Aminosäuren (As): Cα -Atom mit verbundenem Proton Seitenkette −H −R Carboxylgruppe Aminogruppe −COOH −NH2 Primärstruktur .. lineare Abfolge der As vom N-terminalen zum C-terminalen Ende Synthese katalysiert durch Ribosomen Hydrolyse enzymatisch o. durch Kochen in HCl o. NaOH katalyisert Struktur und Funktion ergibt sich aus chem. Eigenschaften der Kette. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 9 / 21 Peptidbindung .. Verbindung 2er Aminosäuren; Kondensationsreaktion katalysiert durch Ribosomen Carboxylgruppe der einen As reagiert mit Aminogruppe der anderen: Rotation um Peptidbindung ist eingeschränkt (cis, trans). Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 10 / 21 Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 11 / 21 Sekundärstruktur α-Helix rechts gedrehte Spirale, 3,6 As pro Windung stabilisert durch H2 -Brückenbindungen zw. 1. und 4. As (in Windung) Seitenketten zeigen nach auÿen sehr stabil β -Faltblatt 2 Pp-ketten zieharmonikaartig verknüpft antiparallel, parallel Peptidgruppen - Fächern, C-Atome - Kanten Stabilisierung durch H2 Bb zw. Peptidbindungen Seitenketten sehr nah beieinander Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 12 / 21 Stukturen höherer Ordnung Supersekundärstruktur β -Hairpin - verbindet 2 antiparallele αα-Hairpin - verbindet 2 antiparallele Coiled-coil β -Faltblätter α-Helix α-Helices → - Superhelix aus 2 Helix-Turn-Helix Motife α-Helices Tertiärstruktur Verdichtung von Sekundärstrukturelementen zu Domains Quartärstruktur Zusammengesetzt aus Tertiärstrukturelementen, desselben o. versch. Proteine Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 13 / 21 4 Strukturbestimmung Wie kann man die Strukturen von RNA oder Proteinen ermitteln? Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 14 / 21 Experimentelle Ermittlung von Molekülstrukturen Optische Methoden UV-, CD- o. Raman Spektroskopie → thermodynamische/kinetische Eigenschaften Hydrodynamische Eigenschaften Ultrazentrifugation → grobe Form der Struktur Form/Ladungstrennung Gelelektrophorese → Atomkoordinaten Strukturverteilungen, thermodynam. Parameter Kernmagnetische Resonanzspektroskopie und Kristallstrukturanalyse chem./molekularbiol. Methoden (RNA) Enzymat./chem. Mapping, Nucleotidsubstitutionen → Unterscheidung zw. gestapelten, gepaarten u. ungepaartem Zustand einzelner Nucleotide Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 15 / 21 Gelelektrophorese .. Methode zur Trennung unterschiedlich groÿer Moleküle Mischung aus zu trennenden, elektr. geladenen Molekülen (RNAs) wandert unter Einuss eines elektr. Feldes durch ein Gel Geschwindigkeit abhaengig von Gröÿe des Moleküls (kurz → schnell, lang → langsam) Reibung wirkt Fluss entgegen → Moleküle bleiben stehen, wenn beide Kräfte ausgeglichen Gröÿe an Kontrolle ablesbar Moleküle sortiert, können spezisch extrahiert werden Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 16 / 21 Kernmagnetische Resonanzspektroskopie (NMR) .. misst Energieabsorption von Kernen beim Übergang zw. benachbarten kernmagnet. Spin-Niveaus. elektronische Umgebung einzelner Atome + Wechselwirkungen mit Nachbaratomen benötigt Moleküle in gelöstem Zustand (hohe Konzentration) + hohe Auösung → genauere Strukturen - unerwünschte Strukturänderung in hochkonzentr. Moleküllösungen - auf kleine Moleküle beschränkt (50nt bzw. 100aa) Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 17 / 21 Kristallstrukturanalyse .. bestimmt atomare und molekulare Struktur eines Kristalls mittels (Röntgen)-Strahlung. Kristallatome beugen Strahlen in spezische Richtungen Winkel und Intensitäten sind messbar → 3D Bild von Elektronen eines Kristalls ⇒ mittlere Position von Atomen u. chem. Bindungen Primärstruktur wird aufgelegt - Atome zugeordnet + mächtigste Technik zur Strukturaufklärung + beliebig groÿe Moleküle analysierbar - Kristallisation von Molekülen kann native Struktur verändern - enorme Datenmengen auszuwerten - Vorzeichenfehler durch quadrat. Amplitudenmessung Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 18 / 21 Präzise Berechnung von Molekülstrukturen 1 bestimme Gesamtenergie E aller möglichen Geometrien eines Aggregates aus Ionen und Elektronen 2 Grundzustand ist die Geometrie mit geringster Energie + Exakte Bestimmung des räumlichen Aufbaus - bei N >2 Atomen zu viele Geometrien möglich, Globales Energieminimum schwierig zu nden, da Anzahl lokaler Minima sehr groÿ werden kann. - der Zwang vereinfachte Formen interatomarer Kräfte zu benutzen schränkt Aussagekraft der Rechnung stark ein. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 19 / 21 Vorhersage von Molekülstrukturen Valenzschalen-Elektronenpaar-Abstoÿungsmodell (VSEPR/EPA-Modell) .. Elektronenwolken in der Valenzschale eines Atoms entfernen sich so weit wie möglich → Molekülgestalt ist Resultat der gegenseitigen Abstoÿung von Elektronenwolken der Atome ◦ vs. 90◦ im Quadrat! Tetraederwinkel: 109.5 Für groÿe Moleküle nicht eindeutig, da zu viele versch. Geometrien für gleiche Anzahl Atome möglich.. ⇒ Vorhersage der Sekundärstruktur von Nukleinsäuren u. Proteine mittels: Graphentheorie, Informationstheorie und genetischen Algorithmen Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 20 / 21 Referenzen Bilder Sämtliche Bilder wurden Wikipedia oder der Rfam Database entnommen und unterliegen somit keinem Copyright. Jana Hertel (Lehrstuhl Bioinformatik) 1 Struktur von Biomolekülen 8. April 2013 21 / 21